Method Article
Eine Lipid-Extraktions- und Analysemethode zur Charakterisierung von Boden-Mikroben in Experimenten mit vielen Proben
In diesem Artikel
Zusammenfassung
Der Artikel beschreibt eine Methode, die den Durchsatz erhöht, während der Aufwand und die Genauigkeit für die Extraktion von Lipiden aus den Zellmembranen von Mikroorganismen zur Verwendung bei der Charakterisierung sowohl der Gesamtlipide als auch der relativen Häufigkeit von Indikatorlipiden zur Bestimmung der mikrobiellen Bodenstruktur in Studien mit vielen Proben ausgeglichen werden.
Zusammenfassung
Mikrobielle Gemeinschaften sind wichtige Treiber und Regulatoren für Ökosystemprozesse. Um zu verstehen, wie das Management von Ökosystemen mikrobielle Gemeinschaften beeinflussen kann, ist eine relativ präzise, aber aufwendungsintensive Technik zur Untersuchung der mikrobiellen Gemeinschaftszusammensetzung die Phospholipidfettsäure (PLFA) -Analyse. PLFA wurde entwickelt, um Phospholipid-Biomarker zu analysieren, die als Indikatoren für mikrobielle Biomasse und die Zusammensetzung von breiten funktionellen Gruppen von Pilzen und Bakterien verwendet werden können. Es wurde häufig verwendet, um Böden unter alternativen Pflanzengemeinschaften, Ökologie und Management-Regime zu vergleichen. Die PLFA-Methode hat sich als empfindlich erwiesen, um Verschiebungen in der mikrobiellen Gemeinschaftszusammensetzung zu detektieren.
Eine alternative Methode, Fettsäuremethylester-Extraktion und Analyse (MIDI-FA) wurde für die schnelle Extraktion von Gesamtlipiden ohne Trennung der Phospholipidfraktion aus reinen Kulturen als mikrobielle Identifikationstechnik entwickelt. Diese Methode istSchnell, ist aber für Bodenproben weniger geeignet, da es anfänglich anfängt, die Bodenpartikel zu trennen und stattdessen mit einer Verseifungsreaktion zu beginnen, die wahrscheinlich Artefakte aus dem Hintergrund organischer Substanz im Boden hervorbringt.
Dieser Artikel beschreibt eine Methode, die den Durchsatz erhöht, während der Aufwand und die Genauigkeit für die Extraktion von Lipiden aus den Zellmembranen von Mikroorganismen zur Verwendung bei der Charakterisierung sowohl der Gesamtlipide als auch der relativen Häufigkeit von Indikatorlipiden zur Bestimmung der mikrobiellen Bodenstruktur in Studien mit vielen Proben ausgeglichen werden. Die Methode kombiniert die durch die PLFA-Profilierung erzielte Genauigkeit durch Extraktion und Konzentration von Bodenlipiden als ersten Schritt und eine Verringerung des Aufwands durch Verseifung des organischen Materials, das mit dem MIDI-FA-Verfahren extrahiert und verarbeitet wird, als zweiten Schritt.
Einleitung
Angesichts der Schlüsselrolle der Mikroorganismen im Nährstoffkreislauf 1 , der Veränderung der Pflanzengemeinschaftszusammensetzung 2 , der Regulierung der Pflanzenproduktivität 3 und der Zersetzung der organischen Substanz 4 ist das Verständnis der mikrobiellen Bodengemeinschaften für das Verständnis der terrestrischen Ökosysteme von entscheidender Bedeutung.
Wegen ihrer relativ hohen Häufigkeit im Boden und ihrer chemischen Signatur können Lipid-Biomarker verwendet werden, um die dominierenden ökologischen Gruppen mit bodenmikrobiellen Gemeinschaften zu profilieren 5 . Durch die Quantifizierung von Lipid-Biomarkern, die für verschiedene mikrobielle Gruppen charakteristisch sind, können wir die Gesamtlipide abschätzen und dann diese Lipide in ökologisch relevante Gruppen wie Gram-positive (Gm +) und Gram-negative (Gm-) Bakterien, arbuskuläre Mykorrhiza (AM) und saprotrophische trennen Pilze und Aktinomyceten 5 , Ss = "xref"> 6 , 7 , 8 .
Es gibt viele Methoden zur Charakterisierung von Aspekten mikrobieller Gemeinschaften. Die PLFA-Methode ist eine häufig verwendete, um grundlegende mikrobielle Gemeinschaftsstruktur zu verstehen. Es ist ein effektiver Weg, um die relative Häufigkeit von mikrobiellen Gruppen sowie die gesamte mikrobielle Biomasse zu beurteilen. Aufgrund des schnellen Lipidumsatzes ermöglicht die PLFA-Profilierung auch eine relativ schnelle Erkennung von Veränderungen in der mikrobiellen Bodengemeinschaft und gibt Informationen, die einen Vergleich der Ökosystemfunktion ermöglichen, z. B. Pilz: Bakterienverhältnisse zur Bewertung der Nährstoffzyklen 1 , 9 , 10 . Während die PLFA-Extraktionsmethode jedoch zeitgeprüft und gut respektiert ist, ist sie auch zeitaufwendig und eignet sich nicht gut für Ökosystem-Skala-Studien, die eine große Anzahl von Proben aus Feldskalen-Replikaten erfordernF "> 11 , 12 .
Im Gegensatz dazu hat das Fettsäuremethylester-Extraktionsverfahren (MIDI-FA) das Potenzial, einen schnellen Durchsatz zu ermöglichen. Bei dieser Methode werden die Proben verseift, in FAME umgewandelt, extrahiert und dann analysiert. Die MIDI-FA-Methode ist schnell, aber weniger diskriminierend als PLFA, die die Extraktion von Lipiden mit der Trennung der verschiedenen Lipidklassen 13 (Phospholipide, neutrale Lipide und Glykolipide) kombiniert.
In diesem Protokoll beschreiben wir eine Methode, die Elemente von PLFA und MIDI-FA Lipid Profiling kombiniert. Es verwendet die Extraktion von Lipiden unter Verwendung der anfänglichen Chloroform-Extraktionsschritte der modifizierten Bligh- und Dyer-Methode und dann der Verseifung und Umwandlung in FAMEs. Dies stellt einen robusten Weg zur Erkennung mikrobieller Gemeinschaftsstruktur bereit, während ein Großteil des Hintergrundrauschens aus nichtmikrobiellem Material 5 , 14 ausgeschlossen ist . Diese Methode wurde ein Gleichgewicht zwischen den beiden PLFA und MIDI-FA - Protokolle erreichen entwickelt, dh die meisten der Genauigkeit beibehalten , während der Durchsatz erhöht wird es logistisch zu machen und wirtschaftlich machbar ist 15 Lipide aus großen Studien mit vielen Proben zu analysieren. Durch die Durchführung der anfänglichen Extraktion und Isolierung der organisch-löslichen Komponenten ( z. B. Lipide) vor der Durchführung von MIDI-FA und dem Abschließen dieser mit einem Reinigungsschritt bietet das Protokoll ein Gleichgewicht zwischen Geschwindigkeit und Präzision.
Protokoll
HINWEIS: Tragen Sie während des gesamten Verfahrens immer eine angemessene persönliche Schutzausrüstung (PSA). Um eine mögliche Kontamination der Proben zu vermeiden, darf man keine Gläser mit bloßen Händen anfassen. Bei der Durchführung von Protokollschritten, die die Handhabung von Chloroform erfordern, geeignete Handschuhe tragen.
1. Vorbereitungen (2 Tage für ~ 40 Proben)
- Sammle den Boden in sterile Beutel und transportiere aus dem Feld in einem Kühler mit Eis. Wenn es nicht möglich ist, frischen Boden zu säen und sofort trocken zu trocknen, lagern Sie die Proben in einem Gefrierschrank von -80 ° C bis zum Beginn der Analyse.
- Vorbereiten des Bodens durch Homogenisierung. Wurzeln und Steine entfernen und Klumpen durch Grobsiebung ( zB 2 mm) aufteilen.
- Vorbereiten zum Gefriertrocknen, indem man die Proben in einem geeigneten Behälter gemäß den Anweisungen des Gefriertrockners manuell einlegt und so schnell wie möglich einfriert. Sobald die Böden gefriergetrocknet sind, lagern Sie in einem verschlossenen Behälter mit Trockenmittel bis zur Extraktion. Es ist am besten zu sRissgefriergetrockneten Boden bei mindestens -20 ° C, vorzugsweise jedoch bei -80 ° C.
- In Vorbereitung auf die Extraktion, entfernen Sie gefriergetrocknete Böden von der Lagerung und schleifen zu einer mehlartigen Konsistenz. Verfahren zum Schleifen sind Kugelmühle, Kugelschläger und Mörser und Stößel. Nach dem Schleifen des Bodens in einem Gefrierschrank lagern (siehe 1.3).
ANMERKUNG: Die für die Extraktion verwendete Probenmenge hängt von ihrem Gehalt an organischen Stoffen ab. Eine allgemeine Richtlinie ist die Verwendung von 0,5 bis 1 g eines Bodens, der 12 bis 18 Gew .-% Kohlenstoff und 3 bis 5 g für einen Boden von 1 bis 3 Gew .-% Kohlenstoff beträgt. - 30 ml Zentrifugenröhrchen mit Hexan spülen. Füge etwa 2 bis 3 ml Hexan zu Röhrchen hinzu und um 5 Sek. Vortexen. Dekantieren Hexan zu einem anderen Röhrchen und vortexen. Hexan (2 bis 3 ml) kann verwendet werden, um sechs Röhrchen seriell zu spülen. Hexan-gespülte Röhrchen, die in der Dunstabzugshaube umgekehrt sind, aufbewahren und gebrauchte Hexan in einem geeigneten Abfallbehälter entsorgen.
- Wickeln Sie die Glasware in 2 bis 3 Schichten Aluminiumfolie und legen Sie sie in den Muffelofen. Backen(Muffel) Glasware bei 450 ° C für 4,5 h.
- Reagenzien vorbereiten
- Für die eingesetzte Bodenmenge fügen Sie Chemikalien in einem Volumenverhältnis von 0,8: 1: 2 für Phosphatpuffer (P-Puffer) hinzu: CHCl 3 : MeOH.
- P-Pufferlösung vorbereiten: Phosphat-Puffer: 0,1 M, pH 7,0.
- Füge 61 ml 1M K 2 HPO 4 Lager hinzu, steril (Chemikalie sollte ACS zertifiziert oder besser sein). Add 39 mL von 1 M KH 2 PO 4 Lager, steril (Chemikalie sollte ACS zertifiziert Grad oder besser). Füllung auf 1000 ml mit Typ 1 Wasser. Den pH-Wert auf 7,0 mit NaOH oder HCl einstellen. Unbenutzte Lösung für bis zu 7 d bei Raumtemperatur oder 30 Tage im Kühlschrank aufbewahren.
- Alternativ wiegen wir 10,62 g K 2 HPO 4 und 5,31 g KH 2 PO 4 ; Auf 1 l mit Typ 1 Wasser verdünnen. PH überprüfen, ggf. einstellen
- Reagenz 1, Verseifungsreagenz vorbereiten.
- 300 ml Wasser des Typs 1 geben.Füge 300 ml CH 3 OH (HPLC-Klasse oder höher) hinzu. Füge 90,00 g NaOH hinzu (zertifiziert ACS oder besser). Zugabe von NaOH-Pellets zu der Lösung unter Rühren. Rühren, bis sich die Pellets auflösen. Es wird empfohlen, diese Lösung nicht länger als 30 d zu speichern.
- Reagenz 2, Methylierungsreagenz vorbereiten.
- 275 ml CH 3 OH (HPLC-Klasse oder höher) abgeben. Füge 325 ml zertifiziertes 6,00 N HCL unter Rühren hinzu. Es wird empfohlen, diese Lösung nicht länger als 30 d zu speichern.
- Reagenz 3, Extraktionsreagenz vorbereiten: 50% C 6 H 14 (Hexan), 50% C 5 H 12 O (MTBE).
- In der Dunstabzugshaube 200 mL C 6 H 14 (HPLC-Klasse oder höher) und 200 ml C 5 H 12 O (HPLC-Klasse oder höher) kombinieren. Gut mischen; Kappe fest. Im brennbaren Schrank oder Abzugshaube für nicht länger als 1 Jahr aufbewahren.
- Reagenz vorbereiten 4, Base wash Reagenz.
- Dispense 900Ml von 1 Wasser zum Becher. 10,80 g NaOH (zertifiziert ACS oder besser) unter Rühren zugeben. Rühren, bis sich die Pellets auflösen. Es wird empfohlen, diese Lösung nicht länger als 30 d zu speichern.
- Vorbereitung der Lagerlösung des internen Standards.
- Wiegen Sie 100 mg von 19: 0 EE-Standard. Zu 50 ml Hexan-gespültem Messkolben geben. Füge ~ 15 ml Hexan-MTBE-Lösung (Reagenz 3) in den Kolben ein, um sich aufzulösen. Mit Hexan-MTBE-Lösung (Reagenz 3) auf 50 ml abkühlen lassen. Cap und Swirl zu mischen. Übertragen auf Hexan-gespülte Glasrohre mit PTFE-gefütterten Kappen und Lagerung bei -20 ° C.
2. TAG 1 - Extraktion von Fettsäuren aus dem Boden (4 bis 5 h für ~ 40 Proben)
- Bereiten Sie drei 5 bis 10 ml Dosierpipetten für P-Puffer, Chloroform und Methanol vor.
HINWEIS: Weniger gefährliche Alternativen zu Chloroform wurden als Extraktionsmittel vorgeschlagen. Diese führen in äquivalenten Lipidwiederfindungsraten 16 , 17 , werden aber in diesem Protokoll nicht ausgewertet. - Gefriergetrocknete Böden in 30 ml Zentrifugenröhrchen wiegen und Bodenmasse aufzeichnen.
- Machen Sie zwei Leerzeichen und beinhalten einen Prüfstandard (ein Boden, der zuvor extrahiert wurde).
- In der Dunstabzugshaube die Reagenzien dem Boden im Zentrifugenröhrchen in folgender Reihenfolge zugeben: P-Puffer, CHCl 3 und MeOH (Tabelle 1). Lassen Sie die Böden nach dem P-Pufferzugabe vor dem Zugabe der CHCl 3 abwischen. Führen Sie die Zentrifugenröhrchen fest und bedecken Sie sie, um vor Licht zu schützen.
- Legen Sie sie auf den Shaker horizontal, um sicherzustellen, dass sie gut gesichert sind. Mit der Geschwindigkeitseinstellung auf 280 U / min schütteln Sie 1 h 18 .
- Vorbereiten von zwei 16 mm x 150 mm Glasröhren für jede Probe wie folgt: Beschriften Sie das Röhrchen und fügen Sie das gleiche Volumen an CHCl 3 und ein gleiches Volumen an P-Puffer hinzu.
- Die Zentrifugenröhrchen aus dem Schüttler entfernen und 10 min bei 1.430 zentrifugierenXg und 25 ° C. Die Phasentrennung sollte im Glasrohr sichtbar sein.
- In der Dunstabzugshaube dekantieren Sie den Überstand aus dem Zentrifugenröhrchen in eines der in Schritt 2.6 hergestellten Röhrchen. Wiederholen Sie die Schritte 2.4 bis 2.5 und dekantieren den Überstand in das zweite Röhrchen.
- Sichern Sie alle 16 mm × 150 mm Glasrohre mit PTFE-gefütterten Kappen und umdrehen Sie 10 x zum Mischen.
- Lassen Sie die Proben über Nacht ungestört stehen, um die Trennung der beiden Phasen abzuschließen. Um dies zu tun, halten Sie die Proben in einem dunklen Schrank / Raum oder bedeckt in Aluminiumfolie bei Raumtemperatur. Es ist akzeptabel, dass sich die Auszüge über das Wochenende trennen lassen.
- Alternativ, wenn man sich direkt zum nächsten Schritt bewegen möchte oder wenn Proben am nächsten Tag gestört werden, werden die Proben für 10 min bei 1000 xg und 25 ° C zentrifugiert.
HINWEIS: Betreffe alle Samples innerhalb einer Vergleichsgruppe zur gleichen Phasentrennungszeit.
- Alternativ, wenn man sich direkt zum nächsten Schritt bewegen möchte oder wenn Proben am nächsten Tag gestört werden, werden die Proben für 10 min bei 1000 xg und 25 ° C zentrifugiert.
3 TAGE2 - Isolierung von Lipiden (3 bis 4 h für ~ 40 Proben)
- Verdampfungsapparatur und Lösemittelfalle einschalten. Den Verdampfungsapparat auf 33 ° C stellen und vorheizen.
- Stellen Sie einen Vakuumsauger in die Motorhaube ein: ein Seitenarmkolben, der mit einer Vakuumpumpe verbunden ist, eine Länge von hochreinem Schlauch und eine Glaspipette.
- Die obere Schicht und die Grenzfläche (ca. 2/3 des Weges nach unten) der beiden Glasröhrchen aufnehmen und die wässrige Schicht zurückhalten. Wiederholen Sie den Vorgang mit einer sauberen Pipette für jede Probe.
- Kombinieren Sie die wässrigen Extraktionsschichten aus dem zweiten Röhrchen mit dem im ersten Röhrchen durch sorgfältiges Dekantieren. Vergewissern Sie sich, dass der Schlauch, der dekantiert, frei von Kratzern oder Rissen ist.
- Sobald die CHCl 3 -Extrakte kombiniert sind, kontrollieren Sie die Flüssigkeit - es sollte klar sein. Wenn man MeOH nicht hinzufügt,
- Trocknen Sie alle Proben mit dem Vakuumverdampfungsgerät ab. Start mit Einstellungen von Temperatur 33 ° C, Wirbelgeschwindigkeit 26% und Druck 400 mbar.
- PDie Proben im Verdunstungssystem spitzen und nach 10 min langsam den Druck auf 350 mbar senken.
- Überwachen Sie den Fortschritt und wenn der Flüssigkeitsspiegel im Rohr den Füllstand des Heizblocks erreicht hat, verringern Sie den Druck auf 300 mbar.
- Fahren Sie fort, die Höhe der verbleibenden Flüssigkeit zu überwachen und wenn es ungefähr 1 cm in der Tiefe ist, verringern Sie den Druck auf 200 mbar.
- Nachdem die Proben getrocknet sind, Proben entfernen und die Pumpe für 10 zusätzliche Minuten laufen lassen, um Dämpfe zu löschen.
- Die Röhrchen fest einfüllen und die Lipide in einem Gefrierschrank bei -80 ° C aufbewahren.
- Entsorgen Sie die angesaugte Flüssigkeit gemäß den geltenden Chemikaliensicherheitsbestimmungen.
4. TAG 3 - Verseifung und Methylierung (6 bis 7 h für ~ 40 Proben)
- Schalte die Wasserbäder ein. Bad 1 bis 95 ° C und Bad 2 bis 80 ° C einstellen.
- Stellen Sie die Pipettenspender ein und stellen Sie sicher, dass sie die richtige Lautstärke verteilen.
Reagenz 1: 1 ml pro Probe
Reagenz 2: 2 ml pro Probe
Reagenz 3: 1,25 ml pro Probe
Reagenz 4: 3 ml pro Probe
HINWEIS: Der Heizvorgang baut Druck im Rohr auf. Verwenden Sie keine zerkratzten, rissigen oder abgebrochenen Röhrchen. - Mit einem Pipettenspender 1,0 ml Reagenz 1 zu den getrockneten Lipiden geben. Mütze fest, für 5 s vortexen und in ein Gestell legen.
- Legen Sie das Rack der Probenröhrchen in das 95 ° C Wasserbad für 5min. Entfernen Sie die Zahnstange aus dem Bad und überprüfen Sie die Rohre auf Lecks, angezeigt durch Blasen steigen in der Tube. Ziehen oder ersetzen Sie die Kappen von austretenden Rohren und überprüfen Sie erneut auf Risse oder Chips. Setzen Sie die Röhren im Wasserbad für weitere 10 Minuten weiter ein.
- Proben aus Bad 1 entfernen und in Bad 2 (80 ° C) stellen und die Inkubation für weitere 15 min fortsetzen.
- Entfernen Sie die Röhrchen und kühlen Sie die Proben ab, indem Sie das Gestell in eine Pfanne mit Leitungswasser zum Kühlen legen.
- Füge 2 ml Reagenz 2 zu jeder Probe hinzu. Kappe fest und vortexen für 5 bis 10s.
- Legen Sie die Zahnstange in das 80 ° C Wasserbad und inkubieren für 10 min.
- Entfernen Sie die Zahnstange aus dem Wasserbad und legen Sie sie in eine Pfanne mit Leitungswasser zum Kühlen. Bewegen Sie die Zahnstange, um den Kühlvorgang zu beschleunigen.
HINWEIS: Zeit und Temperatur nicht überschreiten. Zu viel Erhitzung könnte FAMEs verschlechtern. - Mit dem Pipettenspender werden 1,25 ml Reagenz 3 zu jedem Probenröhrchen gegeben, um die Fettsäuremethylester zu extrahieren. Kappe fest und die Röhrchen für 10 min auf den Schüttler stellen.
- Nach dem Schütteln, lassen Sie die Rack von Röhren sitzen für 10 min für die Phasen zu trennen. Übertragen Sie die organische Phase (Deckschicht) auf ein 16 mm x 100 mm Glasreagenzglas mit einer Glaspipette.
- Wiederholen Sie die Extraktion durch erneutes Hinzufügen von Reagenz 3, Schütteln, so dass Phasen zu trennen, und Übertragung der oberen Phase.
HINWEIS Übertragen Sie keine der unteren Phase. Es ist gut, eine kleine Menge von Top-Phase in der Tube zu verlassen. - Mit dem Pipettenspender 3 ml Reagenz 4 zu th gebenE 16 mm x 100 mm Rohre.
- Führen Sie die Reagenzgläser fest und vortexen für 20-30 s.
- Nach dem Vortexen wird 3 min bei 1000 x g zentrifugiert.
- Mit einer sauberen Glaspipette die obere organische Phase abspülen und in eine 4- mL-Bernstein-Durchstechflasche überführen.
- Verdampfungsapparatur und Lösemittelfalle einschalten. Den Verdampfungsapparat auf 30 ° C einstellen, die Wirbelgeschwindigkeit auf 26%, das Vakuum auf 200 mbar und die Vorwärmung drücken.
- Überwachen Sie den Fortschritt. Nachdem die Proben getrocknet sind, Proben entfernen und die Pumpe für 10 zusätzliche Minuten laufen lassen, um Dämpfe zu löschen.
- Entfernen Sie die Pipette aus den Reagenzflaschen 1 und 4 und pumpen Sie durch etwas verdünnte Säure ( zB 1% HCl), gefolgt von DI-Wasser. Spülen Sie die für Reagenz 2 verwendete Pipette durch Pumpen durch DI-Wasser. In der Haube das Lösungsmittel aus der für Reagens 3 eingesetzten Pipette in einen geeigneten Behälter geben und in der Haube aufbewahren, bis die Lösungsmittelrückstände verdampft sind.
- Lagern Sie die Pipetten umgedreht, mit den Kolben entfernt, um Stickin zu verhindernG der Rückschlagventile
- Glaspipetten einmal verwenden und dann in einem geeigneten Behälter entsorgen.
- In der Haube lassen Lösungsmittelreste auf Glaswaren verdampfen.
- Spritzguss mit sauberem Wasser und Spülmittel löslich machen.
5. TAG 4 - Vorbereitung der Arbeitslösung und Übertragung von FAME auf die GC-Durchstechflaschen (2-3 h für ~ 40 Proben)
- Sammeln von Materialien: 4 ml Bernsteinfläschchen mit getrockneten Proben; Bernsteinfarbene 2 mL GC-Durchstechflaschen; 400 μL flache Bodeneinsätze und Kappen; 500 μl Glasspritze; 50 mL Messkolben; Stock-Lösung - Ethyl nonadecanoat (19: 0 EE interner Standard) in 50/50 Hexan / MTBE (Reagenz 3).
- Unter Verwendung der Glasspritze werden 500 μl Ethylnonadecanoat (19: 0 EE) Stammlösung in einen 50-mL-Messkolben gegeben.
- Füllkolben zur volumetrischen Partitur mit Reagenz 3 füllen.
- Cap Flasche und Invert 5x zu mischen.
- Übertragen Sie 3 ml auf eine saubere 4 mL Durchstechflasche für eine Arbeitslösung Reservoir.
- Mit thE Glasspritze, fügen Sie 300 μl der Arbeitslösung zu jeder der 4 ml-Durchstechflaschen hinzu, die die getrockneten Fettsäuremethylester und die Kappe enthalten.
- Vortex die Probe für 15 s und beiseite legen für 15 min stehen.
- Mit einer Glaspasteurpipette die suspendierten Fettsäuremethylester sorgfältig auf eine 2 ml GC-Durchstechflasche mit dem 400 μl-Einsatz übertragen.
- Dichtung GC-Durchstechflaschen in einem Gefrierschrank von -20 ° C vor der Analyse aufbewahren.
- Setzen Sie Proben für die GC-Analyse ein.
HINWEIS: Die Analyse muss mit einer bestimmten GC-Spalte und Bedingungen durchgeführt werden, die in der Ergänzenden Datei S1 beschrieben sind . Es ist am besten, dass die GC-Analyse innerhalb von 2 Wochen nach der Methylierung abgeschlossen ist.
Ergebnisse
Die Datentabellen aus den Berichten können in eine Tabellenkalkulation oder Datenbank zusammengefasst werden. Nach dem Anpassen des Ansprechfaktors (ein Korrekturfaktor, der die Reaktion für unterschiedliche Kettenlängen normalisiert), können Peakflächen mit der Peakfläche von externen oder internen Standards verglichen werden, um zu einer Konzentration im Extrakt zu gelangen. Durch die Teilung durch die Masse des extrahierten Bodens können die Daten als Masse von FAME pro Gramm Boden ausgedrückt werden, oder unter Verwendung des Molekulargewichts jedes FAME die üblicherweise gemeldeten nmol pro Gramm Boden. Die Summe der mikrobiellen FAME ist ein Hinweis auf die gesamte mikrobielle Biomasse und kann unter den Behandlungen verglichen werden ( Abbildung 1 ). Bestimmte FAMEs können auch mit bestimmten mikrobiellen Gruppen wie Gram-positiven oder gramnegativen Bakterien, Actinomyceten, arbuskulären Mykorrhizapilzen und saprotrophischen Pilzen 19 , 20 , 21 , 22 , 23 , 24 . Die Verhältnisse der Masse der spezifischen Biomarker können die relative Häufigkeit dieser Gruppen widerspiegeln ( Abbildung 2 ). Insgesamt FAME-Häufigkeitsmuster erzeugen Fingerabdrücke auf Gemeinschaftsebene, die den Vergleich mikrobieller Gemeinschaftsuniversität durch multivariate Techniken wie die Ordination ermöglichen ( Abbildung 3 ). Im Gegensatz zu den meisten DNA-basierten Ansätzen können Lipiddaten auf Gemeinschaftsebene entweder als relative oder absolute Häufigkeit analysiert werden. Wenn sich die Gesamtbiomasse im wesentlichen zwischen den Proben unterscheidet, ergeben sich diese beiden Ansätze sehr unterschiedliche Ergebnisse; Die ökologischen Fragen, die dem Experiment zugrunde liegen, sollten bestimmen, welcher Ansatz verwendet wird.
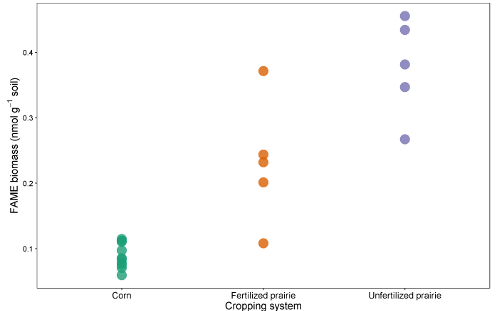
F Igure 1 : Gesamt-FAME-Biomasse Vergleich der gesamten FAME Biomarker Biomasse (nmol g -1 Boden) aus kontinuierlichen Mais, befruchtete Prärie und unbefruchtete Prärie. Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
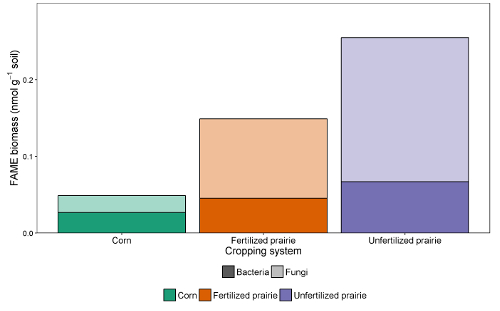
Abbildung 2 : FAME Biomasse von Bakterien und Pilzen. Behandlungsvergleich von Bakterien und Pilz FAME Biomarker Biomasse (nmol g -1 Boden) aus kontinuierlichen Mais, befruchtete Prärie und unbefruchtete Prärie.Fungal und Bakterienmasse aus absoluter Fülle. Durchschnitt (Summe f / Summe b). Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
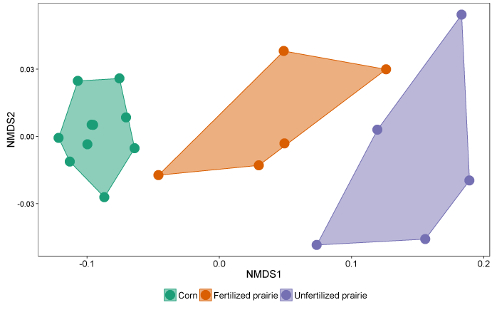
Abbildung 3 : Nichtmetrische Dimensionsskalierung von FAME-Lipid-Biomarkern. Vergleich der gesamten mikrobiellen Gemeinschaft mit absoluten Häufigkeitsprofilen aller mikrobiellen FAME-Lipid-Biomarker. In diesem Beispiel sind fortlaufende Mais- und unbefruchtete Präriegemeinschaften getrennt und sind sehr weit auseinander, während einige befruchtete Prärieproben mikrobielle Gemeinschaften haben, die denen von Mais ähneln, und andere ähneln der unbefruchteten Prärie. Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
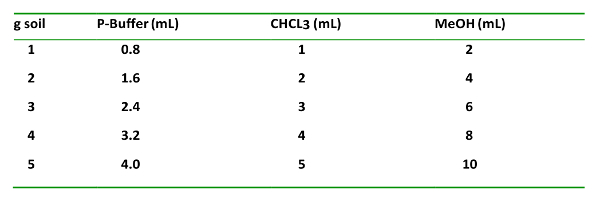
Tabelle 1: T h e soVen l t - Typ, propor ti on s hinzugefügt g -1 Boden, ein t d h EIR orde r o f o n iti hinzuzufügen. Dies ist wichtig für die richtige Extraktion und Trennung der organischen und wässrigen Phasen.
Diskussion
Für die Untersuchung von mehreren Proben aus Experimenten mit vielen Wiederholungen und / oder experimentellen Einheiten können Forscher Phospholipid Fettsäure-Analyse (PLFA) finden in Bezug auf Zeit und Materialien 25 unerschwinglich zu sein. Mit dem PLFA-Verfahren werden Zellmembran-Phospholipide extrahiert, gereinigt und unter Verwendung der modifizierten Blight- und Dyer- 26 -Zweiphasen-wässrig-organischen Extraktion identifiziert. Darauf folgt eine Festphasen-Siliciumdioxid-Chromatographie, um die Lipide durch Polarität zu trennen, und eine alkalische Methylierung von Phospholipidfettsäuren in Fettsäuremethylester. In PLFA kann die Profilierung der Lipidausbeute gering sein, aber von einer sehr hohen Reinheit. Die mikrobielle ID führte eine alternative Methode ein, das Fettsäuremethylesterverfahren (MIDI-FA). Bei der MIDI-FA-Methode werden alle Lipide direkt aus Reinkulturen oder Boden- / Sedimentproben 11 , 12 , 27 durch extrahiertVerseifung Diese Methode hat einen geringeren Lipidverlust und ist schnell, weil sie keine der Konzentrations- oder Reinigungsschritte der PLFA-Methode aufweist. Während die MIDI-FA-Methode jedoch schnell und kostengünstiger ist, weil sie ursprünglich für die Identifizierung von Organismen in der reinen Kultur konzipiert wurde, gibt es keine anfänglichen Extraktions- oder Reinigungsschritte. So kann es auch lipidähnliche Verbindungen enthalten, die aus der organischen organischen Substanz extrahiert sind, die die gemeinschaftliche Signatur 27 , 28 , 29 verzerren. Da diese Einbindung auch Biomasse-Maßnahmen verzerren kann, wurde MIDI-FA typischerweise nur zur grob qualitativen Beschreibung von Bodenlipiden eingesetzt 13 .
Das hier beschriebene Verfahren kombiniert das Beste aus den beiden separaten Extraktionsverfahren: 1) eine Extraktion und Konzentration von Lipiden nach dem modifizierten Bligh- und Dyer- 26- Verfahren und 2) dem Fettsäuremethylester saPonifizierung, Methylierung, Extraktion und Grundwaschgang im Handel entwickelt. Diese Methode wurde entwickelt , um die Vorteile beider dieser Protokolle zu erreichen , während die Nachteile zu minimieren 15. Durch die Durchführung der anfänglichen Extraktion und der Isolierung der organisch-löslichen Komponenten ( z. B. Lipide) vor der Durchführung von MIDI-FA und deren Vervollständigung mit einem Reinigungsschritt bietet dieses Protokoll ein Gleichgewicht zwischen Geschwindigkeit und Präzision. Obwohl dieses Verfahren nicht geeignet ist, wenn eine hohe Reinheit erforderlich ist ( dh für 13 C PLFA-Analysen) oder bei der Analyse von Phospholipiden und neutralen Lipiden separat, erlaubt es in vielen Fällen die Erkennung von mikrobiellen Gemeinschaftsreaktionen auf Umweltbedingungen mit größerer Empfindlichkeit als DNA-basiert Verfahren 30 , 31 , 32 , 33 . Membranlipide zersetzen sich schnell nach dem Zelltod, der ihnen erlaubtSpiegeln die lebende mikrobielle Gemeinschaft zum Zeitpunkt der Probenahme 5 , 7 , im Gegensatz zu Umwelt-DNA, in der ein Großteil der Informationen von toten oder inaktiven Organismen stammt 34 . In Anbetracht 35 die hohe Rate der Dormanz beobachtet unter Bodenmikroorganismen kann die Charakterisierung von lebende Biomasse verwendet werden können , verstehen zeitliche Pflanzen-Mikroben - Interaktionen mit einer relativ feinen zeitlichen Skala und Lipid - Biomarkern verwendet werden 7 den physiologischen Zustand der mikrobiellen Gemeinschaft zu untersuchen. Es wurde gezeigt, dass hohe Durchsatzmethoden erforderlich sind, um die mikrobielle Antwort in großen Feldeinstellungen 25 zu beurteilen, und während die Methode, die wir hier vorschlagen, nicht die Genauigkeit der PLFA-Biomarker-Profilierung repliziert, erhöht sie den Durchsatz und minimiert die Variabilität, die mit dem MIDI-FA realisiert wird Verfahren. Die Methode hat sich als wirksames Instrument bei der Adressierung erwiesenFragen im Zusammenhang mit der mikrobiellen Gemeinschaftsdynamik auf einer breiten Palette von Böden in groß angelegten landwirtschaftlichen und ökosystemischen Studien 36 , 37 , 38 , 42 , 43 , 44 , 45 , 46 , 47 , 48 , 49 , 50 .
Lipidklassen werden mit dieser Methode kombiniert, und es kann ein Verlust der in diesen separaten Klassen 22 , 39 enthaltenen Informationen geben, aber die Kombination der Lipidklassen kann die Kraft verstärken, den arbuskulären Mykorrhizalpilzursprung des 16: 1 ω5c von beiden Phospho zu detektieren - und neutrale Lipide 40 . Darüber hinaus, während tDie Anzahl der unbekannten Fettsäuren (die sich aus nicht-lebender organischer Substanz ableiten lassen), kann mit dieser Methode höher sein, es wurde gezeigt, dass sie niedriger als MIDI-FA ist und ermöglicht einen Behandlungsvergleich von Lipidprofilen aus Studien mit vielen Proben, bei denen die Probe vorliegt Durchsatzkapazität ist ein Anliegen 15 . Es wird allgemein angenommen, daß die neutrale Fraktion hauptsächlich aus Lagerlipiden stammt, die durch Pilze erzeugt werden, obwohl es auch einen kleineren Beitrag aus der Bodenfauna geben kann 41 . Angesichts dessen kann das hier beschriebene Verfahren zu Ergebnissen führen, die einen größeren Beitrag der Pilzlipide 18: 2 ω 6,9c und 18: 1 ω 9c als PLFA zeigen. Die anderen Lipide, die in der neutralen Fraktion auftauchen, schließen einige der gesättigten Fettsäuren ein, z. B. 16: 0, 18: 0, 20: 0.
Es gibt verschiedene Möglichkeiten, Lipiddaten auszudrücken und zu analysieren. Die häufigsten Darstellungen sind Fülle (nmol g -1 Boden), MaulbruchstelleN (nmol einzelnes Lipid nmol -1 Gesamtlipid) und Molprozent (Molenbruch * 100). Normalisiert durch die Gesamtlipide in einer Probe, Mol-Fraktion und Mol-Prozent sind Maßnahmen der relativen Häufigkeit eines gegebenen Lipids. Nach einer geeigneten Transformation, z. B. arcsinischer Quadratwurzel, ist der Molenbruch zur Verwendung bei der Analyse durch Hauptkomponenten oder Redundanzanalyse-Ordination geeignet. Fülle ist die absolute Menge eines gegebenen Lipids, das pro Gramm Boden extrahiert wird. Weil die Menge an Lipid pro Zelle vernünftigerweise konstant ist und die Lipid-Extraktion hoch effizient und umfassend ist, ist die Gesamthäufigkeit eine gute Schätzung der Gesamtlipide und die Fülle der Schlüsselindikatoren spiegelt die Biomasse der ökologischen Gruppe wider, die sie darstellt 17 . Schließlich ist ein guter Weg, um mikrobielle Gemeinschaftszusammensetzung zu betrachten, multivariate Analysemethoden 16 zu verwenden , zB Ordinationsmethoden wie nichtmetrische multidimensionale Skalierung (NMDS -Die keine Datentransformation benötigt) oder Hauptkomponentenanalyse (PCA), kann nützlich sein, um die relative Häufigkeit aller Lipidbiomarker zu vergleichen.
Offenlegungen
Die Autoren haben nichts zu offenbaren.
Danksagungen
Diese Arbeit wurde zum Teil von der DOE Great Lakes Bioenergy Research Center (DOE BER Büro der Wissenschaft DE-FC02-07ER64494) und DOE OBP Büro für Energieeffizienz und erneuerbare Energie (DE-AC05-00RL01830) finanziert. Die Autoren möchten Anders Gurda für seinen geduldigen und geschickten Beitrag zur Videographie und Bearbeitung ansprechen.
Materialien
| Name | Company | Catalog Number | Comments |
| RapidVap | Labconco | 7900000 | Evaporative system |
| RapidVap | Labconco | 7491400 | Sample block |
| Chem-resistant vacuum pump | Labconco | 7584000 | Vacuum pump |
| Chemical trap cannister | Labconco | 7815300 | Trap cannister |
| Solvent insert | Labconco | 7515200 | Solvent trap insert |
| Diaphragm pump | Welch | 7398000 | DryFast vacuum pump |
| Water bath | Fisher | 15-462-15Q | 2 well water bath |
| Gas chromatograph | Various | N/A | For sample analysis |
| Centrifuge | Various | N/A | For sample separation |
| Freeze Dryer | Various | N/A | For sample lyophilization |
| Repipet (6) | BrandTech | 4720440 | For dispensing reagents |
| Vortex | Fisher | 02-215-365 | Analog vortex mixer |
| Teflon centrifuge tubes | Thermo/Nalgene | 3114-0030 | Teflon sample tubes |
| Caps | Thermo/Nalgene | DS3131-0020 | Caps for teflon tubes |
| Test tube | Corning | 9825-16 | 16x100mm tubes |
| Test tube | Corning | 9825-16xx | 16x150mm tubes |
| Caps | Corning | 9998-15 | 15-415 thread black phenolic caps w/PTFE liner |
| Pasteur pipets | Fisher | 13-678-20B | 14.6cm |
| 500 uL glass syringe | Fisher | 13684106LC | Hamilton 81217 |
| Amber vials | Agilent | 5182-0716 | 4mL Amber vials |
| Caps | Agilent | 5182-0717 | Blue screw caps |
| Inserts | Agilent | 5181-3377 | 400uL flat bottom glass inserts |
| Standards | |||
| 19:0 ethyl nonadecanoate (Ethyl nonadecanoate) | VWR | TCN0459-5G | Analytical standard |
| Chemicals | |||
| Dipotassium phosphate (K2HPO4 - ACS grade or better) | Various | N/A | For making Phosphate-Buffer |
| Potassium phosphate monobasic (KH2PO4 - ACS grade or better) | Various | N/A | For making Phosphate-Buffer |
| Methanol (CH3OH - HPLC grade or better) | Various | N/A | For making Reagents 1 & 2 |
| Sodium hydroxide (NaOH - ACS grade or better) | Various | N/A | For making Reagents 1 & 4 |
| Hydrochloric acid (6N HCL) | Various | N/A | For making Reagent 2 |
| Hexane (HPLC grade or better) | Various | N/A | For making Reagent 3 |
| MTBE (Methyl tert-butyl ether - HPLC grade or better) | Various | N/A | For making Reagent 3 |
Referenzen
- Van Der Heijden, M. G. a., Bardgett, R. D., Van Straalen, N. M. The unseen majority: Soil microbes as drivers of plant diversity and productivity in terrestrial ecosystems. Ecol Lett. 11 (3), 296-310 (2008).
- Bever, J. D., et al. Rooting theories of plant community ecology in microbial interactions. Trends Ecol Evol. 25 (8), 468-478 (2010).
- Suleiman, A. K. A., Manoeli, L., Boldo, J. T., Pereira, M. G., Roesch, L. F. W. Shifts in soil bacterial community after eight years of land-use change. Syst Appl Microbiol. 36 (2), 137-144 (2013).
- Sayer, E. J., et al. Grassland management influences spatial patterns of soil microbial communities. Soil Biol Biochem. 61, 61-68 (2013).
- Fernandes, M. F., Saxena, J., Dick, R. P. Comparison of Whole-Cell Fatty Acid (MIDI) or Phospholipid Fatty Acid (PLFA) Extractants as Biomarkers to Profile Soil Microbial Communities. Microbial Ecol. 66 (1), 145-157 (2013).
- van der Heijden, M. G. A., Wagg, C. Soil microbial diversity and agro-ecosystem functioning. Plant Soil. 363 (1-2), 1-5 (2013).
- Allison, V. J., Miller, R. M., Jastrow, J. D., Matamala, R., Zak, D. R. Changes in soil microbial community structure in a tallgrass prairie chronosequence. Soil Sci Soc Am J. 69 (5), 1412-1421 (2005).
- Kowalchuk, G. A., Buma, D. S., de Boer, W., Klinkhamer, P. G. L., van Veen, J. A. Effects of above-ground plant species composition and diversity on the diversity of soil-borne microorganisms. A van Leeuw. 81 (1-4), 509-520 (2002).
- Bardgett, R. D., Hobbs, P. J., Frostegard, A. Changes in soil fungal:bacterial biomass ratios following reductions in the intensity of management of an upland grassland. Biol Fert Soils. 22 (3), 261-264 (1996).
- Frostegård, &. #. 1. 9. 7. ;., Tunlid, A., Bååth, E. Use and misuse of PLFA measurements in soils. Soil Biol Biochem. 43 (8), 1621-1625 (2011).
- Haack, S. K., Garchow, I. H., Odelson, D. A., Forney, L. J., Klug, M. J. Accuracy , Reproducibility , and Interpretation of Fatty Acid Methyl Ester Profiles of Model Bacterial Communitiest. Appl Environ Microbiol. 60 (7), 2483-2493 (1994).
- Drenovsky, R. E., Elliott, G. N., Graham, K. J., Scow, K. M. Comparison of phospholipid fatty acid ( PLFA ) and total soil fatty acid methyl esters ( TSFAME ) for characterizing soil microbial communities. Soil Biol Biochem. 36, 1793-1800 (2004).
- Cavigelli, M. A., Robertson, G. P., Klug, M. Fatty acid methyl ester ( FAME ) profiles as measures of soil microbial community structure. Plant Soil. 170, 99-113 (1995).
- Determination of microbial community structure using phospholipid fatty acid profiles. Molecular microbial ecology manual Available from: https://www.researchgate.net/profile/Robert_Findlay2/publication/281208148_Determination_of_microbial_community_structure_using_phospholipid_fatty_acid_profiles/links/55db4f0b08aec156b9afe776.pdf (2004)
- Balser, T. C., Liang, C., Gutknecht, J. L. Linking microbial community analysis and ecosystem studies: A rapid lipd analysis protocol for high throughput. Biol Fert Soils. , (2017).
- Findlay, R. H., King, G. M., Watling, L., Watling, L. E. S. Efficacy of Phospholipid Analysis in Determining Microbial Biomass in Sediments Efficacy of Phospholipid Analysis in Determining Microbial Biomass in Sedimentst. Appl Environ Microbiol. 55 (11), 2888-2893 (1989).
- Willers, C., Jansen van Rensburg, P. J., Claassens, S. Microbial signature lipid biomarker analysis - an approach that is still preferred, even amid various method modifications. J Appl Microbiol. 118 (6), 1251-1263 (2015).
- Zelles, L., Bai, Q. Y., Beck, T., Beese, F. Signature fatty-acids in phospholipids and lipopolysachharides as indicators of microbial biomass and community structure in agricultural soils. Soil Biol Biochem. 24 (4), 317-323 (1992).
- Frostegard, A., Tunlid, A., Baath, E. Phospholipid fatty-acid composition, biomass, and activity of microbial communities from 2 soil types experimentally exposed to different heavy-metals. Appl Environ Microbiol. 59 (11), 3605-3617 (1993).
- Federle, T. W., Dobbins, D. C., Thorntonmanning, J. R., Jones, D. D. Microbial biomass, activity, and community structure in subsurface soils. Ground Water. 24 (3), 365-374 (1986).
- Vestal, J. R., White, D. C. Lipid analysis in microbial ecology - quantitative approaches to the study of microbial communities. BioScience. 39 (8), 535-541 (1989).
- Wilkinson, S. G. Gram negative bacteria. Microbial Lipids. , 299-488 (1988).
- Balser, T. C., Treseder, K., Ekenler, M. Using lipid analysis and hyphal length to quantify AM and saprotrophic fungal abundance along a soil chronosequence. Soil Biol Biochem. 37 (3), 601-604 (2005).
- Buyer, J. S., Sasser, M. High throughput phospholipid fatty acid analysis of soils. Appl Soil Ecol. 61, 127-130 (2012).
- Bligh, E. G., Dyer, W. J. A rapid method of total lipid extraction and purification. Can J Biochem Phys. (37), 911-917 (1959).
- Schutter, M. E., Dick, R. P. Comparison of Fatty Acid Methyl Ester (FAME) Methods for Characterizing Microbial Communities. Soil Sci Soc Am J. 64 (5), 1659-1668 (2000).
- Jandl, G., Leinweber, P., Schulten, H., Ekschmitt, K. Contribution of primary organic matter to the fatty acid pool in agricultural soils. Soil Biol Biochem. 37, 1033-1041 (2005).
- Nielsen, P., Petersen, S. O. Ester-linked polar lipid fatty acid profiles of soil microbial communities: a comparison of extraction methods and evaluation of interference from humic acids. Soil Biol Biochem. 32, 1241-1249 (2000).
- Duncan, D. S., Jewell, K. A., Suen, G., Jackson, R. D. Detection of short-term cropping system-induced changes to soil bacterial communities differs among four molecular characterization methods. Soil Biol Biochem. 96, 160-168 (2016).
- Liang, C., et al. Switchgrass rhizospheres stimulate microbial biomass but deplete microbial necromass in agricultural soils of the upper Midwest, USA. Soil Biol Biochem. 94, 173-180 (2016).
- Jangid, K., et al. Land-use history has a stronger impact on soil microbial community composition than aboveground vegetation and soil properties. Soil Biol Biochem. 43 (10), 2184-2193 (2011).
- Ritz, K., et al. Spatial structure in soil chemical and microbiological properties in an upland grassland. FEMS Microbiol Ecol. 49 (2), 191-205 (2004).
- Carini, P., et al. Relic DNA is abundant in soil and obscures estimates of soil microbial diversity. J Chem Inf Model. 53 (9), 1689-1699 (2013).
- Lennon, J. T., Jones, S. E. Microbial seed banks: The ecological and evolutionary implications of dormancy. Nat Rev Microbiol. 9 (2), 119-130 (2011).
- Gutknecht, J. L. M., Field, C. B., Balser, T. C. Microbial communities and their responses to simulated global change fluctuate greatly over multiple years. Glob Change Biol. 18, 225-269 (2012).
- Pei, Z., et al. Soil and tree species traits both shape soil microbial communities during early growth of Chinese subtropical forests. Soil Biol Biochem. 96, 180-190 (2016).
- Oates, L. G., Duncan, D. S., Sanford, G. R., Liang, C., Jackson, R. D. Bioenergy cropping systems that incorporate native grasses stimulate growth of plant-associated soil microbes in the absence of nitrogen fertilization. Ag Ecosys Environ. 233, 396-403 (2016).
- Bååth, E. The use of neutral lipid fatty acids to indicate the physiological conditions of soil fungi. Microbial Ecol. 45 (4), 373-383 (2003).
- Ngosong, C., Gabriel, E., Ruess, L. Use of the signature Fatty Acid 16:1ω5 as a tool to determine the distribution of arbuscular mycorrhizal fungi in soil. J Lipids. 2012, 236807 (2012).
- Sharma, M. P., Buyer, J. S. Comparison of biochemical and microscopic methods for quantification of arbuscular mycorrhizal fungi in soil and roots. Appl Soil Ecol. 95, 86-89 (2015).
- Oates, L. G., Balser, T. C., Jackson, R. D. Subhumid pasture soil microbial communities affected by presence of grazing, but not grazing management. Appl Soil Ecol. 59, 20-28 (2012).
- Liang, C. Potential legacy effects of biofuel cropping systems on soil microbial communities in southern Wisconsin, USA. Ag Sci. 2 (2), 131-137 (2011).
- Herzberger, A. J., Duncan, D. S., Jackson, R. D. Bouncing Back Plant-Associated Soil Microbes Respond Rapidly to Prairie Establishment. PloS One. 9 (12), 1-14 (2014).
- Fraterrigo, J. M., Balser, T. C., Turner, M. G. Microbial community variation and its relationship with nitrogen mineralization in historically altered forests. Ecology. 87 (3), 570-579 (2006).
- Kao-Kniffin, J., Balser, T. C. Elevated CO2 differentially alters belowground plant and soil microbial community structure in reed canary grass-invaded experimental wetlands. Soil Biol Biochem. 39 (2), 517-525 (2007).
- Mentzer, J. L., Goodman, R. M., Balser, T. C. Microbial response over time to hydrologic and fertilization treatments in a simulated wet prairie. Plant Soil. 284 (1-2), 85-100 (2006).
- Ushio, M., Wagai, R., Balser, T. C., Kitayama, K. Variations in the soil microbial community composition of a tropical montane forest ecosystem: Does tree species matter. Soil Biol Biochem. 40 (10), 2699-2702 (2008).
- Bartelt-Ryser, J., Joshi, J., Schmid, B., Brandl, H., Balser, T. Soil feedbacks of plant diversity on soil microbial communities and subsequent plant growth. Perspect Plant Ecol. 7 (1), 27-49 (2005).
- Fichtner, A., von Oheimb, G., Härdtle, W., Wilken, C., Gutknecht, J. L. M. Effects of anthropogenic disturbances on soil microbial communities in oak forests persist for more than 100 years. Soil Biol Biochem. 70, 79-87 (2014).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten