Method Article
Split-BioID: Análisis proteómico de complejos de proteínas específicos de contexto en su entorno celular nativo
En este artículo
Resumen
Proporcionamos un protocolo paso a paso para split-BioID, un ensayo de complementación de fragmentos de proteína basado en la técnica de etiquetado de proximidad BioID. Activa en la interacción de dos proteínas determinadas, permite el análisis de Proteómica de los complejos de la proteína dependiente de contexto en su entorno celular nativo. El método es simple, rentable y sólo requiere equipo normal de laboratorio.
Resumen
Para complementar la actual purificación de la afinidad (AP) enfoques para la identificación de las interacciones proteína-proteína (PPI), se han introducido las enzimas que permiten el etiquetado de proximidad dependiente de proteínas en células vivas. Una tal enzima, BirA * (utilizado en el enfoque BioID), media el biotinylation de proteínas dentro de un rango de aproximadamente 10 nm. Por lo tanto, cuando unida a una proteína de interés y expresado en las células, permite el etiquetado de proteínas proximal en su ambiente nativo. En contraposición a la AP que se basa en la purificación de complejos de proteína ensambladas, BioID detecta las proteínas que se han marcado dentro de las células no importa si ellos todavía interactúan con la proteína de interés cuando están aislados. Puesto que biotinylates proximal proteínas, uno puede además aprovechar la excepcional afinidad de streptavidin para biotina aislarlos de manera muy eficiente. Mientras BioID realiza mejor que AP para identificación transitoria o interacciones débiles, ambos métodos de espectrometría de masa AP y BioID proporcionan una visión general de todas las interacciones posibles de que una proteína dada puede tener. Sin embargo, no proporcionan información sobre el contexto de cada PPI identificado. De hecho, la mayoría de las proteínas suelen parte de varios complejos, correspondientes a pasos de maduración distintos o diferentes unidades funcionales. Para solucionar esta limitación común de ambos métodos, hemos diseñado un ensayo de complementación de fragmentos de proteína basado en la BirA * enzima. En este ensayo, pueden volver a montar en una enzima activa cuando cerca de dos proteínas de interacción a la que se fusionan dos fragmentos inactivos de la BirA. El análisis de split-BioID resultante así permite el etiquetado de proteínas que se montan alrededor de un par de interacción de proteínas. Siempre estos dos sólo interactúan en un contexto determinado, split-BioID permite el análisis de las unidades funcionales de dependiente del contexto específicos en su entorno celular nativo. Presentamos un protocolo paso a paso para probar y aplicar BioID split a un par de interacción de proteínas.
Introducción
Como funciones más celulares son realizadas por proteínas que dinámicamente complejos macromoleculares, la identificación de las interacciones proteína-proteína (PPI) es un esfuerzo importante en la investigación biomédica. De hecho, PPI son a menudo desregulado en la enfermedad y representan potenciales dianas terapéuticas1. El más utilizado método para la identificación de PPI es el enfoque de purificación (AP) de afinidad en la que, tras la lisis celular, una proteína de interés específicamente se purifica en una matriz y las proteínas asociadas posteriormente identificadas por espectrometría de masas (MS). AP-MS es un enfoque potente, por lo general no realiza bien en complejos poco solubles de la proteína, interacciones muy transitorias o PPI que requieren una estructura subcelular intacta. Por otra parte, la naturaleza dinámica de las redes PPI, puede complicar la interpretación de los datos como una única proteína es a menudo parte de varios complejos de distintas proteínas.
Recientemente se desarrollaron técnicas de etiquetado de proximidad como BioID2 o3,de APEX24 para abordar algunas de las limitaciones de los enfoques de AP-MS. En BioID, la enzima BirA * (que corresponde a una variante G115R de la enzima de e. coli de tipo salvaje) cataliza la formación de lábil biotinil AMP (bio-AMP) que puede reaccionar con aminas primarias. A diferencia de la enzima de tipo salvaje, que conserva bio-AMP en su centro activo, BirA * libera bio-AMP permitiendo su difusión a su entorno vecino. Por lo tanto, unida a una proteína de interés y expresado en las células, proteínas proximal puede biotinilado dentro de un rango estimado de 10 nm5. Estos marcan proximal proteínas son aisladas por estreptavidina telecine e identificadas por MS. A diferencia de AP-MS, BioID requiere la expresión de una proteína de fusión. Se puede así sólo aplicar a las proteínas cuya función no es obstaculizado por etiquetado. Por otra parte, la velocidad de etiquetado es lenta, por lo general 6-24 h2,6, haciendo difícil la detección de proteínas de vida corta. Sin embargo, comparado con el AP-MS, MS BioID ofrece varias ventajas claves: primero, su captura las interacciones en su entorno celular nativo; proteínas en segundo lugar, el etiquetado, en lugar de complejos montados se aíslan tras la lisis celular; en tercer lugar, jalones de estreptavidina permiten el uso de búferes de desnaturalización y las condiciones de lavado áspero. Por lo tanto, el método es más sensible para detectar transitorios o interacciones débiles7 o interacciones que se producen en un específico y difícil de aislar la estructura subcelular8.
Sin embargo, la mayoría de las proteínas generalmente forman parte de complejos más grandes que puede remodelar según señales de celulares o a la función que debe realizar. Por lo tanto, una sola proteína suele ser parte de varios complejos, correspondientes a distintas unidades funcionales, que implican distintas o superposición de PPI. Ambos enfoques dan un resumen de todas las asociaciones que puede tener una determinada proteína, pero no abordar el contexto de cada PPI. Para aumentar la resolución de este último, hemos diseñado un ensayo de complementación de fragmentos de proteína (ACP) en la cuales dos fragmentos inactivos de BirA * (NBirA *, que contiene el dominio catalítico, y CBirA * que puede ser visto como el dominio de reactivación) puede volver a montar en una enzima activa cuando cerca de dos interactuar las proteínas9. El ensayo resultante de split-BioID enfoca el biotinylation de proximidad dependiente proteínas que montan alrededor de un par de interacción de proteínas y permite así la identificación del contexto ensamblados dependientes de la proteína. Recientemente hemos demostrado el poder de resolución excepcional de split-BioID por resolución de dos complejos de distintas proteínas implicadas en la mediada por miRNA-silenciamiento génico vía9.
En conjunto, en un análisis simple y sencillo, split-BioID permite descubrir y asignar específicamente PPI a unidades funcionales definidas en el cual una determinada proteína es implicado, proporciona que una proteína de interacción adicional de la proteína correspondiente complejo es conocida.
Protocolo
Nota: Un resumen del método se muestra en la figura 1.
1. planificación de la estrategia de clonación
- Seleccione dos proteínas supuestamente interactúan para ser probado.
Nota: Cada una de las dos proteínas se fundieron a un fragmento de fractura BioID: NBirA * o CBirA *. Como control negativo, se probarán las proteínas de fusión CBirA * con NBirA * fusionado a la proteína verde fluorescente (NBirA *-GFP). Como un control adicional, el NBirA * fusiones pueden analizarse solo, sin cognado CBirA * fusión o en combinación con un CBirA * unida a una proteína sin relación. Es recomendable no hacer uso de CBirA-GFP como control negativo ya que ha demostrado consistentemente conducen a mayor fondo combinando en cualquier NBirA * fusión proteína9. La causa de esta observación puede ser el nivel de expresión de la CBirA-GFP, mucho mayor que cualquier otro CBirA * proteínas de fusión que hemos utilizado hasta ahora, que pueden conducir a significativo reasociación con el fragmento del NBirA *. -
Una vez seleccionadas las dos proteínas, revise la literatura para encontrar si ambos han ya sido con éxito marcados en estudios funcionales (por ejemplo como una proteína de fusión GFP-etiquetado).
- Si existen tales estudios, observe la posición de la etiqueta (en N - o C-terminal) y la misma orientación para etiquetar las proteínas de interés con los fragmentos de fractura BioID.
- Si no existe ningún tal estudio, plan de codificación para ambas proteínas etiquetadas en la N - y c-terminal y plan de un ensayo para probar la funcionalidad de las proteínas de fusión (por ejemplo, un experimento de rescate de una línea de celular agotada para la proteína WT).
Nota: Cuando se están emparejando dos proteínas de fusión con la plásmidos se describe en la figura 2 que codifican a enlazadores flexibles larga, la orientación de las proteínas de fusión (BirA * fragmentos fundidos aguas arriba o aguas abajo de las proteínas de interés) generalmente no importa . De hecho usando FRB y FKBP como proteínas de modelo, se demostró que todos cuatro iteraciones posibles (ambos fragmentos en N-termini, en el termini de C, uno en el N-terminal y c-terminal y viceversa) rendimiento comparable Biotinilación9.
- Diseño de cebadores para amplificar los ORFs de las dos proteínas de interés para la clonación en la plásmidos BioID split. Preste atención que los marcos de la traducción son las mismas que los fragmentos de BirA *. Si usa la plásmidos que se describe en la figura 2, use las enzimas de restricción PmeI y PacI construir CBirA * fusión y las enzimas ClaI y MluI para la fusión de NBirA *.
Nota: La plásmidos representada en la figura 2 llevar un elemento sensible al tet bidireccional flanqueado por sitios de recombinación F3/FRT. Esto permite que la expresión cooperación regulada aproximadamente al mismo nivel tanto de proteínas de fusión desde el mismo plásmido10y la posibilidad de construir rápidamente líneas celulares estables de intercambio mediado por recombinación cassette (RMCE). En estos plásmidos, NBirA * tiene una etiqueta de myc CBirA * tiene una etiqueta de bandera. Los ORFs supuesto también pueden ser clonados en plásmidos individuales con promotores constitutivos.
Paso crítico: Cuando dos proteínas interactuantes, se recomienda comparar unida a proteína 1 combinada con CBirA * fusionada a la proteína 2 de NBirA * y NBirA * fusionada a la proteína 2 combinada con CBirA * unida a proteína 1. De hecho, se observa con frecuencia que uno iteración funciona mejor que el otro.
2. clonación los ORFs de los Genes de interés en el plásmido BioID Split
Nota: En este ejemplo, se consideran dos proteínas que pueden ser etiquetadas en el N-terminal. Cuatro condiciones serán probadas y comparadas a las células transfectadas no (tabla 1).
- Utilizar la reacción en cadena de polimerasa (PCR) para amplificar los ORFs de las dos proteínas a probarse con los cebadores adecuados. Diseño de cartillas que el ClaI y MluI sitios de restricción para las proteínas de fusión de NBirA * y los sitios para las proteínas de fusión CBirA * PacI y PmeI.
Nota: La PCR puede realizarse con cualquier comercial, preferentemente corrección, polimerasa de la DNA. Seguir el protocolo estándar del manual y adaptarlo a la temperatura de fusión de los cebadores y la longitud de la ORF amplificado según las directrices del fabricante. -
Subclone primer ORF
- Digerir el plásmido (2 μg) y ORF1 polimerización en cadena-amplificado (para fusionar a la NBirA) con ClaI y MluI. Realizar reacciones de digestión con 1 μL de cada enzima, mezclado con el volumen adecuado de DNA y reacción tampón en un volumen total de 20 μL en un tubo de 1,5 mL por 1 h a 37 ° C.
- Ejecutar ambas muestras de la digestión en gel 1% agarosa TAE ADN. Suprimir las bandas correspondientes a plásmido digerido y ORF1 con un bisturí limpio y transferencia a tubos de 1,5 mL.
- Purificar ambas bandas usando un kit de extracción de ADN estándar.
- Ligar el plásmido digerido y ORF1 reactivos estándar.
- Si utilizando el kit de ligadura indicado en la Tabla de materiales, ligar 100 ng de ADN que contienen exceso molar de tiempo de tres a cinco Inserte sobre plásmido en un volumen total de 4.5 μL. Añadir 5 μL de tampón de ligasa de x T4 2 y 0,5 μL de T4 ADN ligasa del kit de la ligadura. Realizar la reacción de la ligadura en un tubo de 1,5 mL durante 10 minutos a temperatura ambiente (RT).
- Convertir en estándar DH-5α e. coli células competentes (preparadas según el método de Inoue11).
- Mezcla 3 μL de la reacción de la ligadura con 50 μL de células competentes de 1,5 mL tubo en hielo e incubar durante 30 min transferencia de las células a un calor bloquean conjunto a 42 ° C por 30-45 s y luego incuban en hielo por 2 minutos Añadir 250 μL de precalentado (37 ° C) Lisogenia caldo (LB) medio y placa 100 μL de las células en una placa de LB-agar precalentado (37 ° C) que contiene la ampicilina. Incube las células durante la noche a 37 ° C.
- Al día siguiente, tomar cuatro a seis colonias e incubar a 37 ° C, 180 rpm, durante la noche en 3 mL de medio LB con ampicilina de-1 μg⋅mL 100 en un tubo de 15 mL.
- Aislar la plásmidos de las colonias escogidas usando un kit estándar de la DNA MiniPrep.
- Verificar la corrección de la plásmidos por Sanger secuenciación utilizando la cartilla reversa 2 cassettes (tabla 2).
- Una vez que se ha identificado un plásmido que contiene el primer ORF, subclone segunda ORF en ese plásmido siguiendo los mismos pasos como paso 2.2 pero usando las enzimas PmeI y PacI.
- La secuencia de la plásmidos resultante usando la cartilla reversa 1 Cassette (tabla 2).
3. prueba de las proteínas de fusión
Nota: Las siguientes instrucciones son para la plásmidos de expresión inducible dual (figura 2) y HeLa-11ht las células, una línea de células HeLa CCL2 subclonal, expresando estable el activador de transcripción reversa controlada por tetraciclina rtTA-M2 y que contiene un lugar geométrico para RMCE12. El medio de crecimiento de estas células es modificado Eagle Medium (DMEM de Dulbecco) que contiene 10% de suero bovino fetal libre de tetraciclina (FBS). Cuando se utiliza otro tipo de célula, exactas condiciones de siembra y crecimiento medio tendrá que ser adaptado.
-
Transitorios de la transfección
- Células de semilla en una concentración de 1 x 105 células en 2 mL por pocillo de una placa de la pozo de seis un día antes de la transfección e incubar las células durante la noche a 37 ° C, 5% CO2 en una incubadora de cultivo celular.
- En el día de la transfección, retire el medio de cada pozo y reemplazar con 2 mL de medio fresco.
- Prepare las cuatro reacciones de transfección según tabla 1. Para que cada pocillo transfectar, añadir 6 μg de polietilenimina a 3 μg de plásmido ADN en un tubo estéril de 1.5 mL y llenar a 500 μl con medio DMEM sin suero.
- Incubar cada mezcla de transfección para por lo menos 5 min a temperatura ambiente antes de agregar la gota sabios a cada pocillo. Incube las células durante la noche a 37 ° C, 5% CO2.
-
Inducción y proximidad-etiquetado
- El día después de la transfección, quitarlo del medio y reemplazar con suplementado con biotina a 50 μm para estimular la Biotinilación y doxiciclina en 200 ng⋅mL−1 para inducir la expresión de las proteínas de fusión. Incube las células durante al menos 20 h a 37 ° C, 5% CO2.
- Para hacer una solución de biotina, disolver biotina en 50 mg⋅mL-1 (corresponde a aprox. 200 mM) en hidróxido de amonio de 2 M. Una vez que esté completamente disuelta, diluir hasta 50 mM de 500 mM Hepes, pH 7,4, luego ajustar el pH a 7.4 con HCl. alícuota y almacenar el resultado 1.000 x solución a-20 ° C. Disolver doxiciclina 10 mg⋅mL-1 en etanol al 70% y tienda en un microtubo tapón a-20 ° C en la oscuridad.
- El día después de la transfección, quitarlo del medio y reemplazar con suplementado con biotina a 50 μm para estimular la Biotinilación y doxiciclina en 200 ng⋅mL−1 para inducir la expresión de las proteínas de fusión. Incube las células durante al menos 20 h a 37 ° C, 5% CO2.
-
Preparación de lisado celular
- Lavar las células una vez con solución fisiológica 1 mL de frío (4 ° C) con tampón fosfato (PBS).
- A cada pocillo, añada 100 μl de tampón de lisis (50 mM Tris, pH 7.4, 150 mM NaCl, 2 mM EDTA, 0,5% NP-40, 0,5 mM DTT e inhibidores de la proteasa).
- Cosechar las células con un scrapper de célula y transferencia a tubos de 1,5 mL.
- Centrifugar las muestras a 14.000 x g por 10 min a 4 ° C para eliminar restos celulares.
- Transferir el sobrenadante a tubos de frescos y coloque en hielo.
- Determinar cantidades de proteína con un ensayo de Bradford.
-
Electroforesis del gel de SDS-poliacrilamida (PAGE) y Western Blot
- Preparar un gel de SDS-poliacrilamida.
- Para cada uno despejado lisado, preparar una muestra de página de 30 μg (mínimo 15 μg) proteína en un total de 30 μl de tampón de carga SDS. Luego cargar cantidades iguales de cada muestra (20 μL/pocillo) sobre el gel de SDS-poliacrilamida y proceder a la electroforesis.
- Después de la electroforesis, transferencia de las proteínas fraccionadas a una membrana de blot PVDF bajo fluorescencia usando cualquier protocolo estándar.
Nota: Para el análisis de proteína biotinylation, un tiempo de transferencia de 10 minutos con el programa "alta MW" de una rápida transferencia semi-seco Western Blot dispositivo generalmente funciona bien. - Después de la transferencia, bloquear la membrana en leche en polvo 5% en PBS por 30 min a TA.
- Incubar la membrana por 30 – 60 min a temperatura ambiente con fluorescente estreptavidina conjugada 1:15,000 diluido en PBS con BSA 2% y 0,1% Tween-20.
- Lavar la membrana tres veces, cada 10 min, con PBS que contenga 0,1% Tween-20 y luego una vez más con PBS.
- La exploración la membrana en un sistema de imágenes de escáner de fluorescencia.
Nota: una típica mancha blanca /negra occidental se muestra en la figura 3. Este procedimiento describe la detección fluorescente base pero una detección basada en luminiscencia con estreptavidina acoplado a HRP funciona igualmente bien.
4. Split-BioID estudios de proteómica
Crítica: Para el análisis de espectrometría de masa final, todos los pasos siguientes deben realizarse en condiciones libres de queratina, todo el material y los reactivos deben ser como queratina-libre como sea posible.
- Transitorios de la transfección
- El día antes de la transfección, semilla 10 cm tres o cuatro placas para cada condición en una concentración de 8 x 105 células en 10 mL por placa e Incube las células durante la noche a 37 ° C, 5% CO2 en una incubadora de cultivo celular.
- Al día siguiente, preparar una mezcla de transfección maestro para cada condición de transfección: por tres placas por condición, 36 μg de polietilenimina y 18 μg de plásmido ADN disuelven en 900 μl DMEM libre de suero. Incubar cada mezcla principal por al menos 5 min a TA.
- Mientras tanto, sustituir el medio de cada plato con medio fresco y luego añadir 300 μL de la gota de mezclas de transfección para cada placa.
- Incube las células durante la noche a 37 ° C, 5% CO2.
- Inducción y proximidad-etiquetado
- El día después de la transfección, transferir las células a los platos de 15 cm. Para cada plato, quitarlo del medio, lavan las células con 7 mL de PBS, añadir 1,5 mL de una solución de tripsina-EDTA e incube durante 5 min a TA. Añadir 3.5 mL de medio de cultivo para resuspender las células y transferir la suspensión de células a un plato de 15 cm con 20 mL de medio de cultivo complementado con biotina a 50 μm para estimular la Biotinilación y doxiciclina en 200 ng⋅mL−1 (concentraciones finales) para inducir la expresión de las proteínas de fusión.
- Incube las células durante al menos 20 h a 37 ° C, 5% CO2.
- Cosecha y almacenamiento de las células
- Lavan las células dos veces con PBS, luego añadir 1,5 mL de PBS a cada placa y cosechar las células con un scrapper.
- Las células cosechadas corresponde a una condición a un tubo de 15 mL de transferencia y cosecha por centrifugación a 1.200 x g, 5 min, 4 ° C.
- Eliminar el sobrenadante y snap congelar las pelotillas en nitrógeno líquido y luego tienda a 80 ° C hasta el procesamiento posterior.
Nota: Alternativamente, las células pueden separadas por tripsinización, cosechadas en medio del crecimiento, transfirió a un tubo de 15 mL y lavar tres veces con PBS antes de congelarlos.
- Preparación de lysates de la célula
- Vuelva a suspender los pellets de células en 1 mL de tampón de lisis (50 mM Tris, pH 7.4, 500 mM NaCl, 0,4% de SDS, 5 mM EDTA, 1 mM TDT, 1 x inhibidor de la proteasa completa) en paso RT. las células de 10-20 veces (cinco a diez carreras) a través de una aguja de 25 G.
- Someter a ultrasonidos las muestras con un dispositivo de sonificación.
Nota: Con el dispositivo de sonificación indicado en la Tabla de materiales, el siguiente programa puede ser utilizado: cuatro ciclos a alta intensidad, 30 s por ciclo en un frío (4 ° C) baño de agua. Cualquier otro dispositivo de sonificación es adecuado pero los parámetros que tenga que ajustarse en consecuencia. - Agregue Triton X-100 para el recuperado sonicada lisado para alcanzar una concentración final de 2% (normalmente, añada 100 μL del 20% Tritón X-100 para 900 μL sonicada lysate de la célula) y luego 2,3 mL de 50 mM Tris, pH = 7.4 por 1 mL de lisado para ajustar la concentración de NaCl hasta 150 m m antes de b inding para granos de estreptavidina acoplado.
Crítica: Concentraciones de sal superiores a menudo resultan en Unión mucho menos eficiente a los granos. - Distribuir los lisados ajustados en tubos de 1,5 mL (ca. 1.1 mL en tres tubos) y centrifugar a 16.000 x g, 10 min, 4 ° C.
- Transferir el sobrenadante (ca. 3,2 mL) a un tubo de 15 mL y mantener 50 – 100 μL como material de entrada.
- Medir la concentración de cada muestra con un ensayo de Bradford y equivalente del contenido de proteínas de 3 a 3.5 mg para el telecine de estreptavidina.
- Desplegable de estreptavidina
Nota: Un resumen del procedimiento de telecine se muestra en la figura 4. Es casi idéntico del protocolo original publicado por Roux y colegas2. La primera vez que se realiza la desconexión, las muestras de flujo y lavado 1 – 4 podrán ponerse a un lado análisis de Western blot asegurar el procedimiento funcionado correctamente (figura 5).- Para cada condición, transfiera 200 μL de suspensión de granos magnéticos estreptavidina acoplado a un tubo de 1,5 mL. Colocar los tubos en una parrilla magnética, espere hasta que los granos se adhieren al lado de los tubos (ca. 1 min.) y retire el buffer de almacenamiento.
- Lavar los granos mezclando suavemente con 1 mL de tampón de equilibrado (50 mM de Tris pH 7 – 4, 150 mM NaCl, 0,05% Tritón X-100 y 1 mM TDT).
- Enviar las cuentas equilibradas en cantidad igual en el número necesario de tubos (generalmente cuando a partir de 3 a 3.5 mg de proteína contenido, que los lisados de cada condición pueden ser despachados en dos a cuatro tubos de 1.5 mL) y el lugar en la rejilla magnética.
- Eliminar el tampón de equilibrado y resuspender cada conjunto de granos con cantidades iguales de los lysates de la célula correspondiente del paso 4.4.7. Incubar durante una noche a 4 ° C en un disco giratorio.
- Al día siguiente, colocar los tubos de 1,5 mL en una parrilla magnética, espere hasta que los granos del palillo al lado de los tubos y transferir el sobrenadante a un tubo de 15 mL etiquetado como flujo a través.
Nota: de ahora en adelante, todos los pasos se realizan a temperatura ambiente a menos que se indique lo contrario. - Suspender los granos en cada tubo con 200 μL de tampón de lavado 1 (SDS 2% en agua) y combinar cada conjunto de granos resuspendidos corresponde a una condición en tubos de 1,5 mL.
- Lavar los granos dos veces durante 8 minutos en una rueda de rotación con 1 mL de tampón de lavado 1.
- Lavar los granos dos veces durante 8 minutos en una rueda de rotación con 1 mL de tampón de lavado 2 (50 mM de pH HEPES 7.4, 1 mM EDTA, 500 mM NaCl, 1% Tritón X-100 y 0.1% de desoxicolato de Na).
- Lavar los granos dos veces durante 8 minutos en una rueda de rotación con 1 mL de tampón de lavado 3 (10 mM Tris, pH 8, 250 mM LiCl, 1 mM EDTA, 0,5% NP-40 y 0.5% desoxicolato de Na).
- Lavar los granos dos veces durante 8 minutos en una rueda de rotación con 1 mL de tampón de lavado 4 (50 mM Tris, pH 7.4, 50 mM NaCl, 0,1% NP-40).
- Para asegurarse de que el tampón de lavado se elimina totalmente después de la última colada paso, eliminar la mayor parte del sobrenadante y luego girar por las muestras. Colocar en la rejilla magnética, espere hasta que los granos del palillo al lado de los tubos y retire el buffer restante.
- Añadir 30 μL de tampón de elución (10 mM de Tris pH 7.4, biotina de 2 mM, SDS 2% y 5% β-mercaptoetanol) para los granos. Incubar a 98 ° C durante 15 minutos, luego retire inmediatamente los granos en un soporte magnético.
- Transferir la muestra eluída a un nuevo tubo y conservar a-20 ° C hasta el procesamiento posterior.
- SDS-PAGE y Western Blot
Nota: Antes del análisis de espectrometría de masas, se recomienda evaluar el éxito de la Biotinilación y telecine por SDS-PAGE y Western blot. Si no se observa ningún patrón de Biotinilación uno puede considerar dox o biotina no fue agregada al medio o que una de las dos soluciones madre se ve comprometida.- Para cada entrada muestra, preparar una muestra de página mezclando cantidades iguales de la muestra de proteína con el volumen adecuado de amortiguador de carga SDS x 3 en un total de 28 μL. Preparar página muestras mezclando 5 μl de cada muestra de elución con 2,5 μl de tampón de carga SDS x 3.
- Carga de las muestras (25 μL/pocillo de entradas, 7 μL/pocillo para las muestras de elución) en un gel de SDS-poliacrilamida. Proceder a electroforesis y Western blot como se describe en la sección 3.4.
- En caso de desconexión por primera vez, preparar página muestras para cada paso de telecine (entrada de flujo a través, elución de 1 – 4, lavado) mezclando 20 μl de cada muestra (entrada de flujo a través, de lavado de 1 a 4) con 10 μl de 3 x de buffer de carga SDS o 5 μl (elución) con 2,5 μl de 3 x S DS-carga de búfer y proceder como en paso 4.6.4 (figura 5).
- SDS-PAGE para análisis de MS
Nota: Para minimizar la contaminación potencial de la queratina, geles prefabricados y tampón de carga comercial pueden ser utilizados.- Añadir 6,25 μL de tampón de muestra de 4 x a 18,75 μL de cada muestra de elución y correr las muestras en un gel de SDS 4-20% prefabricado hasta migran 2 – 3 cm en el gel.
- Manchar el gel coloidal G250 azul brillante de Coomassie tinción13 en un plato de petri de 15 cm.
- Utilice un bisturí limpio para suprimir los carriles enteros para cada muestra, sin incluir la banda de estreptavidina (correr en aprox. 17 kDa, figura 6) y para transferir las bandas suprimidas a tubos de 1,5 mL.
- Enviar estas muestras a un centro de proteómica para su posterior análisis.
Nota: Resultados MS típica pueden verse en la referencia 9 (figura 3 y figura 7y tablas complementarias). Los conjuntos de datos enteros están disponibles en el repositorio de orgullo (número PXD005005).
Resultados
Para ilustrar cómo funciona este método, los marcos de lectura abierta (ORFs) de las proteínas antes2, TNRC6C y Dicer (todos los involucrados en el vía de silenciamiento del gen mediada por miRNA) se clonaron en split-BioID plásmidos. Antes2 es conocida para interactuar con TNRC6C dentro de un inducido por miRNA silenciamiento complejo (miRISC) que reprime la traducción y estimular el decaimiento del objetivo mRNAs14. Antes de montar el miRISC, antes2 interactuar con Dicer, la enzima que produce miRNAs maduros, dentro de un complejo en el que puede llegar cargado con un miRNA15. Por lo tanto split BioID se aplicó al par antes2/Dicer o el par de antes2/TNRC6C. Para cada par de proteínas probadas, antes2 fue bien fundido a NBirA * o CBirA * utilizar nuestros plásmidos de split-BioID (figura 2), y Dicer y TNRC6C a la correspondiente cognada BirA * fragmento. Además, cada proteína fue fundido a la CBirA y emparejado con un NBirA *-fusión de GFP como control negativo. El resultado en la prueba cuatro iteraciones para cada par de proteínas probadas (tabla 1).
Para probar si BioID split se activa con la interacción del par de proteínas probadas, hemos seguido el esquema representado en la figura 1. La plásmidos fueron transfectada transitoriamente en una línea celular HeLa compatible de tet-sistema. La expresión de las proteínas de fusión fue inducida con doxiciclina (dox) y Biotinilación fue estimulada por agregar biotina exceso al medio de crecimiento. Después de un tiempo de incubación de 20 h con dox y biotina, las células fueron lisadas y analizadas por Western Blot utilizando conjugado estreptavidina para detectar proteínas biotiniladas. En células de mamíferos, dos bandas principales son detectados típicamente por la estreptavidina conjugada en la muestra untransfected (figura 3, estrellas) y corresponden a endógeno biotinilado proteínas (carboxilasas mitocondriales probablemente). Estas dos bandas están presentes en todas las muestras y pueden ser utilizadas convenientemente como controles de carga interna, por lo tanto, la detección de una proteína de limpieza para controlar la carga de cantidades de proteína igual es superflua. Típico de un experimento BioID/split-BioID, las bandas principales adicionales que se pueden observar son las proteínas de fusión que tiene uno mismo-biotinilado. Aunque es vista no hay otra proteína biotinilada, detección de Biotinilación de las proteínas de fusión en esta etapa ya indica que las dos proteínas probadas interactuaron en las células. En el experimento representado en la figura 3, está claro que tener un NBirA *-proteína de fusión antes2 con CBirA * fusiones TNRC6C o Dicer es más eficaz que las combinaciones opuestas en que CBirA *-antes2 está emparejado a las NBirA * fusiones de los otros dos proteínas (figura 3, panel superior, comparar las intensidades de 2 a 3 carriles a carriles 6 y 7). Por otra parte, la activación fue específica como podría activar ninguna de las fusiones CBirA * NBirA *-proteína de la fusión de GFP control a niveles apreciables (figura 3, comparar los carriles 1, 4-5 en el carril 8 que corresponde a las células untransfected). Ya que en los plásmidos, NBirA * tiene una etiqueta de myc y CBirA * tiene una etiqueta de bandera (figura 2), los niveles de expresión de cada proteína de fusión pueden ser analizados con los anticuerpos contra estos dos etiquetas (figura 3, panel inferior).
Cuando se observa la Biotinilación inducida por la interacción, el experimento puede ampliarse, y las proteínas biotiniladas aislaron en granos de estreptavidina acoplado como se indica en el párrafo 4 del Protocolo (figura 4). Al realizar el aislamiento de la primera vez, todos los pasos de la purificación pueden ser analizados por Western Blot (figura 5). Por lo general, vinculante a los granos debe ser casi cuantitativa y prácticamente fugas a través de no deben ser observados en los lavados. Antes de procesar las muestras para espectrometría de masas, se recomienda ejecutar un Western blot para asegurar inducida-Biotinilación trabajado como se esperaba y que las proteínas de fusión fueron expresadas. Es la falta de expresión de las proteínas de fusión debido a la eficiencia de transfección deficiente o defectuosa dox inducción. Si se expresan las proteínas de fusión pero no Biotinilación es observado, compruebe si exceso biotina (50 μM) realmente fue agregada al medio y que la biotina stock aún está activa. Cuando el material eluído se analiza en un gel teñido de Coomassie de la proteína (figura 6), por lo general, la banda más fuerte para ser observado funciona en alrededor de 17 kDa y corresponde a estreptavidina monomérica. También se pueden observar bandas correspondientes a las proteínas endógenas biotinilado y las proteínas de fusión. Suele suprimir la zona del carril de la muestra por encima de la banda de estreptavidina hasta la carga bien (figura 6). La banda suprimida puede ser almacenada en un tubo de 1,5 mL y enviada a un centro de espectrometría de masas. Alternativamente, encuadernadas proteínas también pueden ser digerido por la tripsina en las cuentas de estreptavidina acoplado y los digeridos péptidos eluidos forman la columna. Habitualmente utilizamos el software de MaxQuant16 (usando sobre todo los parámetros por defecto y añadiendo lisina Biotinilación como una posible modificación poste-de translación, ver referencia 9 para más detalles y resultados típicos de MS) para analizar los datos raws de MS y la Perseus suite17 para el análisis estadístico posterior, ambos son software libre. Las muestras normalmente se ejecutan en tres réplicas biológicas. Mediante cuantificación de etiqueta-libre, específicamente las proteínas enriquecidas pueden ser identificadas por las condiciones de control. Para filtrar endógeno biotinilado las proteínas y las proteínas que están etiquetados no específicamente por la enzima BirA *, consideramos sólo las proteínas que se enriquecen significativamente en golpes de seis conjuntos de datos generados con seis proteínas no relacionadas. Además, consideramos sólo hits que se enriquecen por un conjunto de datos BioID split en el que las proteínas de fusión de NBirA * han sido reemplazadas por NBirA *-GFP. Se han propuesto otras estrategias de análisis de datos en particular utilizando isótopos estables de etiquetado con aminoácidos en cultura de célula (SILAC) para proteómica cuantitativa18. Además, se han descrito diversas estrategias para el aislamiento directo de péptidos biotinilados usando una variante de estreptavidina con débil afinidad biotina18, las condiciones de elución especial usando disolventes orgánicos19 o biotina específicos anticuerpos20,21. Mientras que no necesariamente conduce al descubrimiento de más proteínas, la identificación de sitios de Biotinilación añadir más confianza en cuanto a la especificidad de los golpes y es útil cuando a la topología de una interacción.
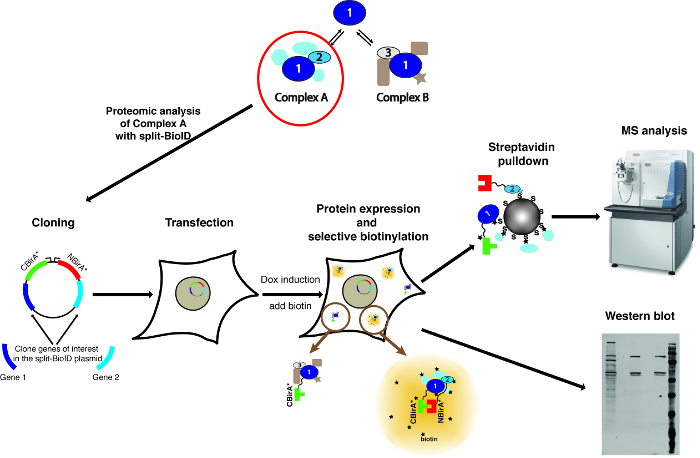
Figura 1: Resumen del procedimiento split BioID. 1 la proteína interactúa con la proteína 2 como parte del complejo A, o con proteína 3 como parte del complejo B. A prueba específicamente la composición del complejo, split-BioID puede aplicarse a las proteínas 1 y 2. La fotografía del espectrómetro de masas está bajo una licencia Creative Commons Atribución Compartir igual 3.0 Unported y fue descargada de https://commons.wikimedia.org con nombre de archivo de ThermoScientificOrbitrapElite.JPG. Haga clic aquí para ver una versión más grande de esta figura.
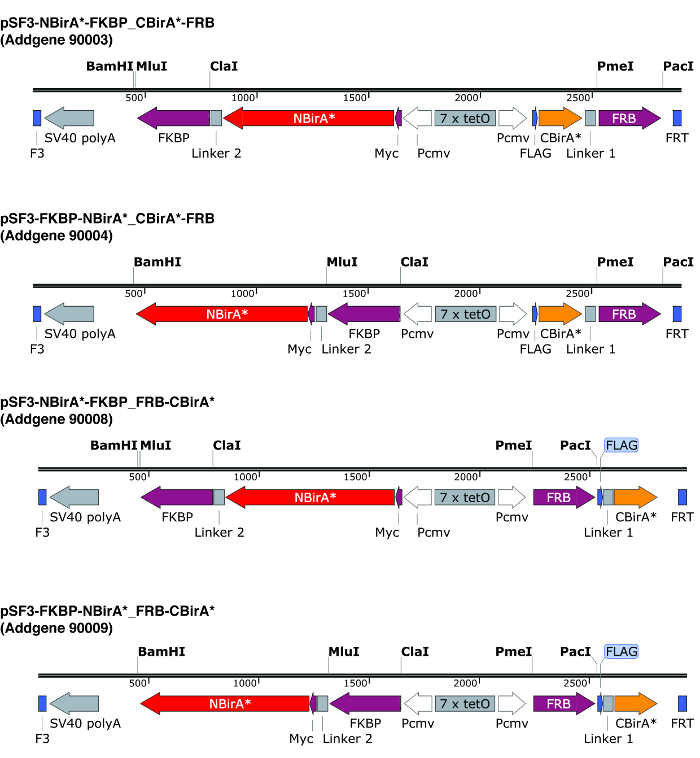
Figura 2: cassettes de expresión de la plásmidos de split-BioID. Ofrecemos cuatro plásmidos para realizar las pruebas de todas las combinaciones de NBirA * y CBirA * proteínas de fusión. Los plásmidos y mapas completos están disponibles en addgene.org bajo el número indicado. La plásmidos tienen un elemento sensible al tet (7 x tetO) y puede ser empleado en una línea celular que sea compatible con el sistema de expresión de TTE. También cuenta que en plásmidos todos los ORFs de FKBP y FRB se fusionan a la NBirA * CBirA * fragmentos respectivamente. Estas dos proteínas interactúan sólo en presencia de la rapamicina y por lo tanto, la plásmidos pueden utilizarse para probar rápidamente el sistema de la presencia o ausencia de este químico9. Los sitios de restricción indicada son únicos. Haga clic aquí para ver una versión más grande de esta figura.
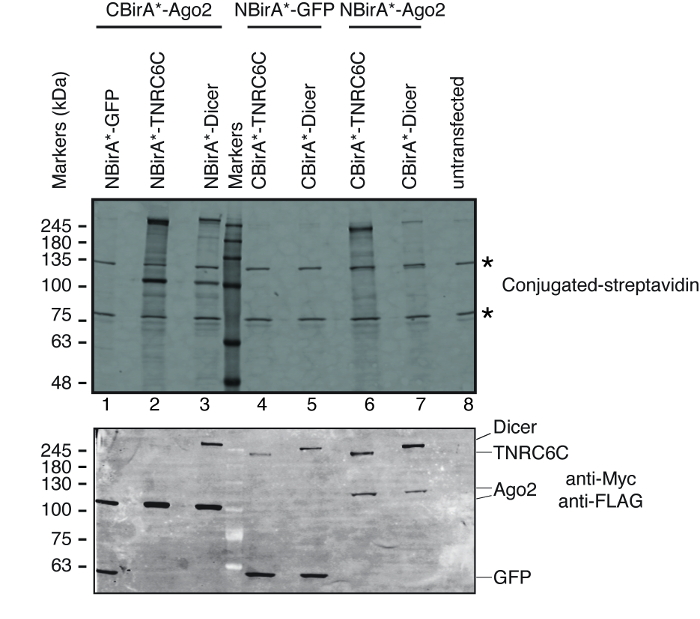
Figura 3: típico Western blot para un experimento de split-BioID. Panel superior: detección de proteínas biotiniladas con estreptavidina marcada fluorescencia. Panel inferior: detección de las proteínas de fusión con los anticuerpos anti-Myc y de anti-FLAG. Se probaron dos pares de proteínas: antes2/TNRC6C y antes2/Dicer. En los carriles 2 y 3, antes2 fue anexa al fragmento de CBirA *. En los carriles 6 y 7, antes2 fue anexado al fragmento de NBirA *. Ninguna señal significativa fue observada cuando cualquiera de las tres proteínas se combinaron con NBirA *-GFP (carriles 1, 4-5). Las estrellas indican las bandas correspondientes a endógeno biotinilado proteínas que pueden servir como controles de carga interna. Esta figura es una adaptación de la figura 5B de Schopp et al. 9 bajo una licencia Creative Commons Atribución 4.0 International. Haga clic aquí para ver una versión más grande de esta figura.
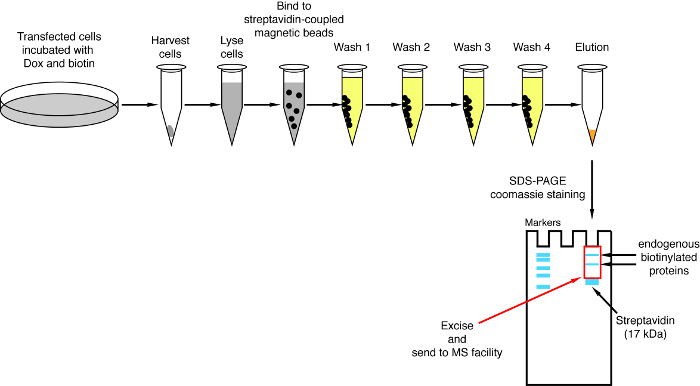
Figura 4: Resumen del procedimiento de desconexión de estreptavidina. Se representan pasos importantes para el aislamiento de proteínas biotiniladas para el análisis de espectrometría de masas. Haga clic aquí para ver una versión más grande de esta figura.
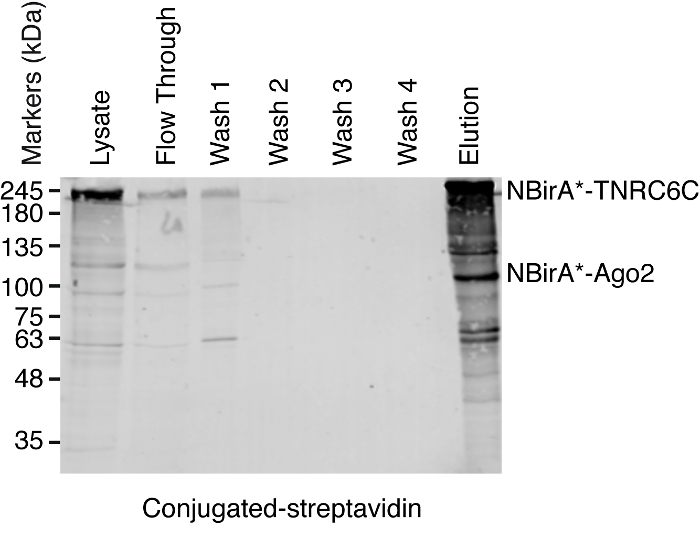
Figura 5: típico Western blot para un experimento de telecine estreptavidina. Iguales volúmenes de cada muestra indicado fueron cargadas en un gel de SDS-poliacrilamida. Tras borrar occidental, proteínas biotiniladas fueron detectadas con streptavidin HRP-juntado. Bandas correspondientes a la NBirA-TNRC6C y CBirA *-se indican antes2. Haga clic aquí para ver una versión más grande de esta figura.
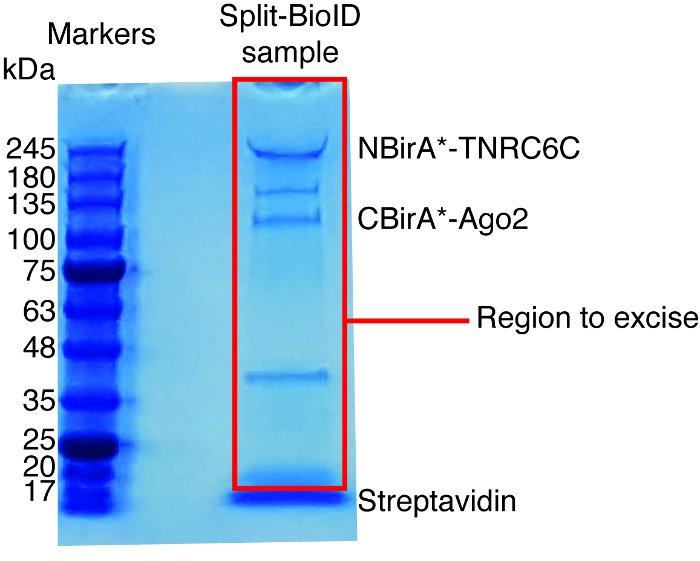
Figura 6: gel de proteína teñido de Coomassie típica para el análisis de espectrometría de masas. La muestra eluída de granos estreptavidina acoplado fue cargada en un gel de proteínas prefabricados y hasta que la muestra migra 2-3 cm. La banda principal en cerca de 17 kDa es estreptavidina. El área directamente sobre esa banda es suprimido y enviado a un centro de espectrometría de masas. Bandas correspondientes a la NBirA-TNRC6C y CBirA *-se indican antes2. Haga clic aquí para ver una versión más grande de esta figura.
| Muestra de transfección | Condición de prueba | ||
| 1 | NBirA *-protein1 / CBirA *-protein2 | ||
| 2 | CBirA *-protein1 / NBirA *-protein2 | ||
| 3 | NBirA *-GFP / CBirA *-protein1 | ||
| 4 | NBirA *-GFP / CBirA *-protein2 | ||
| 5 | no la transfección | ||
Tabla 1: Prueba típicamente condiciones al aplicar BioID split a dos proteínas.
| Cartilla de la secuencia | secuencia de |
| Primer revés de cassette 1 (CBirA * fusión) | TATACTTTCTAGAGAATAGGAAC |
| Primer revés de cassette 2 (NBirA * fusión) | GTGGTTTGTCCAAACTCATC |
Tabla 2: Secuencia cartillas para el split BioID plásmidos.
Discusión
El procedimiento describe cómo clonar genes de interés en split-BioID plásmidos probar la Biotinilación inducida por la interacción y aislar las proteínas biotiniladas para el análisis de espectrometría de masas. Aquí describimos un procedimiento basado en la transfección transitoria. Mientras que la expresión de las proteínas de fusión puede ser afinada por la cantidad de dox añadido al medio, transitorios de la transfección pueden conducir a la expresión de la proteína no homogéneo con algunas células que sobreexpresan groseramente las proteínas de fusión comparado con el endógeno contrapartes. Esto puede conducir a distorsiones de la interactomes correspondiente y a PPI que no reflejan fielmente las interacciones que implican las proteínas endógenas. Así es generalmente recomendable para construir líneas celulares estables una vez BioID split se ha establecido con el sistema transitorio. La plásmidos son compatible con el sistema de recombinación mediada por el Flp y coloque ambos genes de interés en la regulación de un mismo elemento sensible al TTE. Si es necesario y cuando se usa con compatible de células de mamífero, que permiten la fácil creación de líneas celulares inducibles estable. Por ejemplo, utilizamos la línea HeLa-EM2-11 que expresa el activador de transcripción activado por tetraciclina rtTA y un único locus genómico targetable de que expresión génica mediada por la tetraciclina puede ser bien regulado12. Usando esta línea celular y recombinación mediada por Flp, líneas celulares estables que contienen solamente una copia del transgén pueden obtenerse dentro de dos o tres semanas. Alternativamente, uno puede usar técnicas actuales de la edición del genoma para introducir los fragmentos de BirA * en los loci genómicos nativos de los genes de interés.
Como en cualquier análisis que se basa en la selección de una proteína, es necesario considerar si la proteína de fusión resultante es funcional. Los datos disponibles en que las proteínas de interés fueron etiquetadas (por ejemplo GFP para estudios por imágenes) y funcionalmente probados son útiles decidir si los BirA * fragmentos clonados aguas arriba o aguas abajo los genes de interés. Si estos datos no están disponibles, uno debe probar N-terminal o con la etiqueta proteínas C-terminal en un análisis funcional. Por ejemplo, la actividad de las proteínas de fusión se puede probar en una línea celular en el cual la proteína endógena ha sido golpeado-hacia fuera y en comparación con la situación de tipo salvaje. Si las proteínas de interés toleran ambas etiquetas N - y C-terminal, ambos deben analizarse. De hecho, en experimentos BioID, la orientación de la proteína de fusión puede influir en la eficacia del etiquetado22. Además, hemos observado al aplicar BioID split a un par de proteínas, que de las dos proteínas se anexa al ya sea el NBirA * o CBirA * fragmento también influencia la eficacia del etiquetado9. En el split-BioID plásmidos, lo 16 aminoácidos largo glicina/serina rico enlazadores acoplamiento de las proteínas de interés en los BirA * fragmentos fueron tomadas de otra PCA23 y trabajó para nosotros todas las proteínas interactúan hemos probado hasta ahora. Sin embargo, uno debe considerar que algunos pares de proteínas podrían funcionar mejor con enlazadores más cortos o más largo. De la nota final, un ensayo fue descrito por el Bollen grupo24. En este ensayo, BirA * se divide en otro sitio (e140 con/Q141) que el nuestro (E256/G257). Hemos probado ambos split BioID sabores por lado y encontró que E256/G257, descrito en el presente Protocolo, conduce a la reactivación más fuerte cuando junto a dos proteínas de interacción9.
Una desventaja general de este método es la lenta velocidad de etiquetado. Por lo general, tiempo de incubación de 6 a 24 h con la biotina es necesaria para obtener Biotinilación apreciable6, imposibilitando el uso de esta técnica para estudiar la dinámica remodelación de complejos de la proteína. Mientras que este ensayo aborda parcialmente esta advertencia sólo es activado cuando interactúan dos proteínas, la lenta velocidad de etiquetado imposibilita su uso para el estudio de respuesta a procesos muy dinámicos o para analizar proteínas de corta vida. La peroxidasa ingeniería APEX2 es conocido por promover el etiquetado eficiente de proteínas proximal dentro de 1 minuto3. Un AAC basado en APEX2 así podría abordar las limitaciones de la velocidad de etiquetado lento de ensayos derivados BioID. Un estudio de prueba de principio había descrito tal split APEX2 ensayo25. Sin embargo, aunque una proteína homodimerizing con éxito fue biotinilado, si el ensayo también puede utilizarse para etiquetar e identificar proteínas que alrededor de un par de interacción de proteínas queda por demostrarse. Muy recientemente, evolución dirigida se utiliza para crear TurboID y miniTurbo, dos variantes de la BirA con actividad mejorada que permiten etiquetado mucho más corto tiempo windows, hasta 10 minutos26. Adaptación de split-BioID a estas nuevas variantes más se extenderá el uso de esta técnica a un campo más amplio de aplicaciones.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este trabajo fue financiado por el Consejo de investigación alemán (DFG) a través de la iniciativa de excelencia alemán (CellNetworks DFG-EXC 81) y un financiamiento parcial por el centro de investigación en colaboración SFB638.
Materiales
| Name | Company | Catalog Number | Comments |
| Acetic acid (glacial) | VWR | 20104.298 | To make TAE buffer |
| Agarose | Sigma | A9539 | Take TAE-agarose gels for DNA analysis and extraction |
| Ammonia solution 25% NH3 | Bernd Kraft | 6012 | To dissolve biotin |
| Ampicillin | Sigma | A9518 | To select transformed bacteria |
| Bioruptor plus sonification device | Diagenode | B01020001 | Other sonification devices are also ok |
| Biotin | Sigma | B4639 | To be added to the growth medium to stimulate efficient biotinylation |
| Bovine serum albumin fraction V | Carl Roth | 8076 | Used in Western blot buffers and a protein standard in Bradford assays |
| Bradford Ultra reagent | Expedeon | BFU05L | Any other method/kit for protein determination is fine, this particular reagent is more tolerant to detergent than other Bradford reagents |
| Cell scrappers | TPP | 99002 | Any other model is also fine |
| ClaI | New England Biolabs | R0197 | Restriction enzyme for cloning into the split-BioID plasmids (NBirA* fusion) |
| DMEM medium | Sigma | D6046 | If using another cell line, use the corresponding optimal growth medium |
| DNA miniprep kit | Sigma | PLN350 | Any other kit is also fine |
| Doxycycline | Applichem | A2951 | Dox is light sensitive |
| DTT | Applichem | A2948 | Make 1M stock solution, store at -20 °C and always use fresh |
| DyLight 680-conjugated streptavidin | Thermo scientific | 21848 | to use with a LiCor Western blot scanning device |
| Dynabeads MyOne Streptavidin C1 | Invitrogen | 65002 | The C1 beads are not BSA coated which is preferable for downstream MS applications (no leakthrough of BSA in the final elution) |
| EDTA | Applichem | A5097 | Make a 500 mM stock, adjust pH to 8 while dissolving the EDTA powder |
| Ethanol | Sigma | 32205 | Make a 70% stock solution in which Doxycycline can be dissolved at 10 mg.mL-1 |
| Fastgene Gel/PCR DNA Extraction Kit | Nippon Genetics | FG-91302 | Any other kit is also fine |
| HCl 37% | Merck | 1.00317.1000 | To adjust pH of biotin stock solution |
| HEPES | Carl Roth | 6763 | Make a 500 mM stock solution, adjust the pH to 7.4 |
| Immobilon-FL PVDF membrane, 0.45 µm | Millipore | IPFL00010 | This membrane shows minimal autofluorescence when used with a LiCor Western blot scanning device |
| LiCl | Grüssing GmbH | 12083 | Make a 5M stock solution |
| Odyssey CLx imaging system | LI-COR | N/A | To scan Western blot membrane decorated with fluorophore-labeled antibody |
| Linear polyethylenimine (PEI) | Polysciences | 23966-2 | Any other transfection reagent is also fine |
| Milk powder | Carl Roth | T145 | To block Western blot membranes |
| MluI-HF | New England Biolabs | R3198 | Restriction enzyme for cloning into the split-BioID plasmids (NBirA* fusion) |
| Na-deoxycholate | Sigma | 30970 | Make a 10% (w/v) stock solution |
| NaCl | Sigma | 31434 | Make a 5M stock solution |
| NP-40 (Nonidet P40 substitute) | Sigma | 74385 | Make a 20% (v/v) stock solution |
| PacI | New England Biolabs | R0547 | Restriction enzyme for cloning into the split-BioID plasmids (CBirA* fusion) |
| Phosphate buffer saline (PBS) | Sigma | 806552 | To wash cells before scrapping |
| PmeI | New England Biolabs | R0560 | Restriction enzyme for cloning into the split-BioID plasmids (CBirA* fusion) |
| Protease inhibitor cocktail | Roche | 4693132001 | Added to the lysis buffer to prevent protein degradation |
| pSF3-Flag-CBir-FRB_Myc-NBir-FKBP | Addgene | 90003 | Split-BioID plasmid, mediates the co-expression of NBirA*-FKBP and CBirA*-FRB, FKBP and FRB can be replaced by two other ORFs |
| pSF3-Flag-CBir-FRB_FKBP-Myc-Nbir | Addgene | 90004 | Split-BioID plasmid, mediates the co-expression of FKBP-NBirA* and CBirA*-FRB, FKBP and FRB can be replaced by two other ORFs |
| pSF3-Flag-FRB-Cbir_Myc-NBir-FKBP | Addgene | 90008 | Split-BioID plasmid, mediates the co-expression of NBirA*-FKBP and FRB-CBirA*, FKBP and FRB can be replaced by two other ORFs |
| pSF3-Flag-FRB-Cbir_FKBP-Myc-Nbir | Addgene | 90009 | Split-BioID plasmid, mediates the co-expression of FKBP-NBirA* and FRB-CBirA*, FKBP and FRB can be replaced by two other ORFs |
| Q5 High-Fidelity PCR kit | New England Biolabs | E0555S | To amplify the ORF coding for the proteins to be tested. Any other thermostable DNA polymerase is fine. |
| Quick ligation kit | New England Biolabs | M2200S | To ligate DNA fragments into the split-BioID plasmids, any other DNA ligation system is fine. |
| RunBlue 4-20% SDS precast gels | Expedeon | BCG42012 | To use when running samples for MS analysis |
| RunBlue LDS Sample Buffer | Expedeon | NXB31010 | Running buffer for the RunBlue precast gels |
| SDS | Sigma | 5030 | Comes as a 20% stock solution |
| tet-free serum | Biowest | S181T | we use tet-free serum to minimize basal expression of the fusion proteins |
| Trans-Blot Turbo Transfer system | Bio-Rad | 1704150 | High speed Western blotting transfer system, any other transfer system is also fine |
| Tris | Carl Roth | 4855 | Make 1M stock solutions with adequate pH (7.4 and 8) |
| Triton X-100 | Applichem | A4975 | Make a 20% (v/v) stock solution |
| Tween-20 | Carl Roth | 9127 | Used in Western blot buffers, Tween 20 leads to high background fluorescence and should be omitted in the blocking and last wash step |
Referencias
- Scott, D. E., Bayly, A. R., Abell, C., Skidmore, J. Small molecules, big targets: drug discovery faces the protein-protein interaction challenge. Nat Rev Drug Discov. 15 (8), 533-550 (2016).
- Roux, K. J., Kim, D. I., Raida, M., Burke, B. A promiscuous biotin ligase fusion protein identifies proximal and interacting proteins in mammalian cells. J Cell Biol. 196 (6), 801-810 (2012).
- Lam, S. S., et al. Directed evolution of APEX2 for electron microscopy and proximity labeling. Nat Methods. 12 (1), 51-54 (2015).
- Rhee, H. W., et al. Proteomic mapping of mitochondria in living cells via spatially restricted enzymatic tagging. Science. 339 (6125), 1328-1331 (2013).
- Kim, D. I., et al. Probing nuclear pore complex architecture with proximity-dependent biotinylation. P Natl Acad Sci USA. 111 (24), E2453-E2461 (2014).
- Kim, D. I., et al. An improved smaller biotin ligase for BioID proximity labeling. Mol Biol Cell. 27 (8), 1188-1196 (2016).
- Lambert, J. P., Tucholska, M., Go, C., Knight, J. D., Gingras, A. C. Proximity biotinylation and affinity purification are complementary approaches for the interactome mapping of chromatin-associated protein complexes. J Proteomics. 118, 81-94 (2015).
- Morriswood, B., et al. Novel bilobe components in Trypanosoma brucei identified using proximity-dependent biotinylation. Eukaryot Cell. 12 (2), 356-367 (2013).
- Schopp, I. M., et al. Split-BioID a conditional proteomics approach to monitor the composition of spatiotemporally defined protein complexes. Nat Commun. 8, (2017).
- Béthune, J., Artus-Revel, C. G., Filipowicz, W. Kinetic analysis reveals successive steps leading to miRNA-mediated silencing in mammalian cells. EMBO Rep. 13 (8), 716-723 (2012).
- Inoue, H., Nojima, H., Okayama, H. High efficiency transformation of Escherichia coli with plasmids. Gene. 96 (1), 23-28 (1990).
- Weidenfeld, I., et al. Inducible expression of coding and inhibitory RNAs from retargetable genomic loci. Nucleic Acids Res. 37 (7), e50 (2009).
- Dyballa, N., Metzger, S. Fast and sensitive colloidal coomassie G-250 staining for proteins in polyacrylamide gels. J Vis Exp. (30), (2009).
- Jonas, S., Izaurralde, E. Towards a molecular understanding of microRNA-mediated gene silencing. Nat Rev Genet. 16 (7), 421-433 (2015).
- MacRae, I. J., Ma, E., Zhou, M., Robinson, C. V., Doudna, J. A. In vitro reconstitution of the human RISC-loading complex. P Natl Acad Sci USA. 105 (2), 512-517 (2008).
- Cox, J., Mann, M. MaxQuant enables high peptide identification rates, individualized p.p.b.-range mass accuracies and proteome-wide protein quantification. Nat Biotechnol. 26 (12), 1367-1372 (2008).
- Tyanova, S., et al. The Perseus computational platform for comprehensive analysis of (prote)omics data. Nat Methods. 13 (9), 731-740 (2016).
- Opitz, N., et al. Capturing the Asc1p/Receptor for Activated C Kinase 1 (RACK1) Microenvironment at the Head Region of the 40S Ribosome with Quantitative BioID in Yeast. Mol Cell Proteomics. 16 (12), 2199-2218 (2017).
- Mackmull, M. T., et al. Landscape of nuclear transport receptor cargo specificity. Mol Syst Biol. 13 (12), 962 (2017).
- Kim, D. I., et al. BioSITe: A Method for Direct Detection and Quantitation of Site-Specific Biotinylation. J Proteome Res. , (2017).
- Udeshi, N. D., et al. Antibodies to biotin enable large-scale detection of biotinylation sites on proteins. Nat Methods. 14 (12), 1167-1170 (2017).
- Chapat, C., et al. Cap-binding protein 4EHP effects translation silencing by microRNAs. P Natl Acad Sci USA. 114 (21), 5425-5430 (2017).
- Luker, K. E., et al. Kinetics of regulated protein-protein interactions revealed with firefly luciferase complementation imaging in cells and living animals. P Natl Acad Sci USA. 101 (33), 12288-12293 (2004).
- De Munter, S., et al. Split-BioID: a proximity biotinylation assay for dimerization-dependent protein interactions. FEBS Lett. 591 (2), 415-424 (2017).
- Xue, M., et al. Optimizing the fragment complementation of APEX2 for detection of specific protein-protein interactions in live cells. Sci Rep. 7 (1), 12039 (2017).
- Branon, T. C., et al. Directed evolution of TurboID for efficient proximity labeling in living cells and organisms. bioRxiv. , (2017).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados