Method Article
ネイティブな携帯電話環境におけるコンテキスト固有蛋白質複合体の分割-BioID-プロテオーム解析
要約
分割-BioID、近接ラベリング手法 BioID を用いたタンパク質断片否定回路試金のステップ バイ ステップのプロトコルを提供します。与えられた 2 つのタンパク質の相互作用活性化、彼らのネイティブ セルラ環境での文脈依存的タンパク質のプロテオミクス解析をことができます。メソッドは、シンプルでコスト効果の高い、標準的な実験装置を必要なだけ。
要約
タンパク質間相互作用 (PPI) の識別のため近づくと既存の親和性の浄化 (AP) を補完するために、生きている細胞の蛋白質の近接依存ラベリングができる酵素が導入されています。1 つそのような酵素・ ビラ * (BioID アプローチで使用される)、約 10 の範囲内のタンパク質のビオチン化を仲介する nm。したがって、興味の蛋白質に溶けるし、細胞で発現できますネイティブな環境で近位蛋白質の分類します。AP 組み立てタンパク質複合体の精製に依存しているのではなく BioID かどうかまだ通信している興味の蛋白質と分離できたらどんな細胞内のマークされているタンパク質が検出されます。それ以来 biotinylates 近位蛋白質 1 つはさらに非常に効率的に、それらを分離するためにビオチンをストレプトアビジンの例外的な親和性を活用できます。BioID は識別する一時的なまたは弱い相互作用の AP より実行、している間両方の AP-BioID 質量分析法によるアプローチは特定の蛋白質があるかもしれないすべての可能な相互作用の概要を示します。ただし、それぞれの特定の PPI のコンテキストに情報は提供しない彼ら。確かに、ほとんどの蛋白質は、通常異なる成熟手順やさまざまな部門単位に対応するいくつかの複合体の一部です。この両方のメソッドの一般的な制限に対処するため我々 は BirA * 酵素に基づくタンパク質断片否定回路試金を設計しました。このアッセイの BirA * 2 つのアクティブでないフラグメントは活性酵素が融合する 2 つの相互作用の蛋白質によって近くになったときに再構成できます。結果の分割 BioID アッセイでは、相互作用の蛋白質のまわりを組み立てる蛋白質の分類したがってことができます。これらの 2 つの特定のコンテキストでのみ対話、提供分割 BioID は、ネイティブ細胞における特定の文脈依存機能単位の分析をできます。ここでは、テストし、相互作用するタンパク質のペアに分割 BioID を適用する手順を追ってプロトコルを提供します。
概要
ほとんどの携帯電話の機能は、動的に組み立てる高分子複合体蛋白質によって実行される、のでタンパク質間相互作用 (PPI) の同定は生物医学研究における主要な試みです。確かに、PPI は病気で自由化頻繁および1の治療のための潜在的なターゲットを表します。最も広くメソッドの PPI の識別は、アフィニティ精製 (AP) アプローチのセル換散、行列に興味の蛋白質を精製具体的と関連付けられているタンパク質を質量分析法によって識別されますその後(MS)。AP MS 強力なアプローチですが、通常は行わない難溶性タンパク質複合体、非常に一時的な相互作用または無傷の細胞内構造を必要とする PPI でも。また、データの解釈は、単一蛋白質はしばしばいくつかの明瞭な蛋白質の複合体の一部として PPI ネットワークの動的な性質を合併することができます。
BioID2または APEX23,4などの近接ラベリング手法は AP MS アプローチの制限のいくつかに対処する最近開発されました。BioID、酵素・ ビラ * (野生型大腸菌の酵素の G115R の亜種に対応する) は不安定なビオチニル-アンプ (バイオ AMP) アミンと反応できるの形成を触媒します。野生型酵素活性中心の生体アンプを保持するのではなく・ ビラ * 生体アンプ、近隣環境への拡散を許可するを解放します。したがって、興味の蛋白質に融合し、細胞で発現、近位蛋白質は 10 nm5の推定範囲内ビオチン化をすることができます。これらの近位マーク蛋白質は、ストレプトアビジン プルダウンによって隔離され、MS によって識別されます。AP-MS ではなく BioID 融合タンパク質の発現が必要です。それがこうしてにのみ適用蛋白質の機能が、タグ付けによって妨げられていません。また、ラベリングの速度が遅い、通常 6-24 h2,6、短命の蛋白質の検出の挑戦を作るします。BioID MS AP MS と比較して、いくつかの重要な利点を提供してまだ、: 最初に、そのキャプチャ ネイティブな携帯電話環境における相互作用組み立ての複合体ではなく、2 つ目のラベルが付いたタンパク質は次のセル換散; 分離第三に、ストレプトアビジン プルダウンにより変性バッファーと洗う過酷な条件を使用します。したがって、メソッドはより一時的なまたは弱い相互作用7または特定の発生する相互作用を検出する敏感な細胞内構造8を分離するは難しい。
ただし、ほとんどの蛋白質は通常携帯電話合図に従って、または実行する必要があります関数を改造できるより大きい複合体の部分であります。したがって、単一のタンパク質は通常いくつかの複合体、異なる機能単位に対応する、異なるを含むおよび/または PPI を重複の一部です。両方のアプローチは特定の蛋白質があるかもしれません、すべてのアソシエーションの概要を与えるが、彼らは個々 の PPI のコンテキストに対処するため失敗します。後者の解像度を上げる、我々 は BirA * (触媒ドメインを含む NBirA *、CBirA * 再活性化ドメインとして見ることができる) の 2 つ使用頻度の低いフラグメントことができますのタンパク質断片否定回路試金 (PCA) を設計しています。活性酵素 2 近くになったときに再蛋白質9の相互作用。結果として得られる分割 BioID 試金を当ててタンパク質相互作用の蛋白質のまわりを組み立てる、従って依存性蛋白質アセンブリ コンテキストの識別ができるように近接依存ビオチン化。我々 は最近、miRNA を介した遺伝子サイレンシング経路9に関与する 2 つの明瞭な蛋白質の複合体を解決することによって分割 BioID の卓越した解像力を示した。
完全に、単一、簡単な試験で分割 BioID により発見し、具体的に定義された機能単位特定の蛋白質が関与、対応するタンパク質複合体の追加相互作用の蛋白質は知られている提供する PPI を割り当てます。
プロトコル
メモ: メソッドの概要については、図 1に表示されます。
1. クローン作成戦略の計画
- テストする 2 つの推定の相互作用の蛋白質を選択します。
注: 2 つの蛋白質の各は分割 BioID フラグメントが 1 つに融合する、: NBirA * または CBirA *。ネガティブ コントロールとして CBirA * 融合蛋白質を持つ NBirA * 緑色蛍光タンパク質融合テストされます (NBirA *-GFP)。同種 CBirA * 融合なしまたは無関係な蛋白質に溶ける CBirA * との組み合わせで、付加的な制御として NBirA * 融合だけでは、テスト可能性があります。CBirA * を使用しないことをお勧め-それはネガティブ コントロールとして GFP 一貫して任意 NBirA * 融合タンパク質9に結合されたとき主要な背景につながることが示されました。この観測の原因 CBirA * の発現レベルがあります-GFP、任意他 CBirA * の融合蛋白質我々 はこれまでのところ、使用している NBirA のフラグメントと重要な再結合する可能性がありますよりもはるかに高い。 -
2 つのタンパク質を選んだら、文学かどうか両方既に正常にタグ付けされている機能の研究 (例えばとして GFP タグ融合タンパク質) を見つけることを確認してください。
- このような研究が存在する場合 (N- または C 末端で) タグの位置に注意してくださいし、分割 BioID フラグメントの興味の蛋白質にタグを同じ方向を使用します。
- このような研究が存在しない場合、両蛋白質のためのコーディング計画構造はタグ付きの N 末端と C 領域でと (たとえば、WT タンパク質用消耗したセル行のレスキュー実験) の融合蛋白質の機能をテストするアッセイを計画します。
注:図 2で説明したプラスミド長く柔軟なリンカーがエンコードを使用して 2 つの融合蛋白質を対にする場合融合蛋白質 (BirA * フラグメントの融合上流または下流に興味の蛋白質) の向き一般に問題ではないです。.確かに FRB と FKBP モデル蛋白質として、それが示された、すべて可能なイテレーションが 4 (N テルミニ C テルミニ N 末端と C 末端で 1 つの両方の両方の断片と逆) 匹敵するビオチン化9をもたらします。
- 分割 BioID プラスミドにクローニングに興味の 2 つの蛋白質の Orf を増幅するプライマーを設計します。翻訳フレームは、・ ビラのフラグメントと同じ注意を払います。図 2で説明したプラスミドを使用している場合は、CBirA * の融合と NBirA の融合酵素 ClaI および MluI を構築する制限酵素 PmeI とパーチを使用します。
注:図 2にプラスミド F3/FRT 再結合サイトが並ぶ双方向テト応答要素を運ぶ。これは、同じプラスミド10組換えを介したカセット交換 (RMCE) による安定したセルラインを急速に構築する可能性からの両方の融合蛋白質のほぼ同じレベル規制共発現をことができます。これらのプラスミド NBirA * は、myc タグ CBirA * フラグの札があるとき。Orf は、もちろんは構成のプロモーターと個々 のプラスミドの複製もことができます。
重要なステップ:NBirA * CBirA * 2、蛋白質に溶けると結合蛋白質 1 に溶けるを比較する勧めします 2 つの相互作用の蛋白質をテストするとき、NBirA * CBirA * 1 蛋白質に溶けると結合蛋白質 2 に溶けます。確かに、イテレーションは 1 つが他よりよく動作することはよく見られます。
2 分割 BioID プラスミドへの興味の遺伝子の Orf のクローニング
注: この例では、N 末端タグすることができます 2 つの蛋白質と見なされます。4 つの条件は、テストし、非 transfected セル (表 1) に比較されます。
- ポリメラーゼの連鎖反応 (PCR) を使用して、適切なプライマーでテストする 2 つのタンパク質の Orf を増幅します。CBirA * の融合蛋白質のパーチと PmeI サイト NBirA * 融合蛋白質のため ClaI および MluI 制限のサイトを紹介するプライマーを設計します。
注: PCR 可能任意の商業的に優先的に校正、DNA ポリメラーゼです。ハンドブックから標準的なプロトコルに従うし、プライマーの溶ける温度と製造元のガイドラインに従って増幅される ORF の長さに適応。 -
最初の ORF を subclone します。
- プラスミド (約 2 μ g) と ClaI と MluI (NBirA * に融合される) を PCR 増幅 ORF1 を消化します。適切な量の総量が 37 ° C で 1 時間 1.5 mL チューブで 20 μ L の DNA と反応バッファーと混合、各酵素の 1 μ L を使用して消化反応を実行します。
- 1% のアガロース TAE DNA ゲルの両方消化サンプルを実行します。きれいなメスと 1.5 mL チューブへの転送消化プラスミッドおよび ORF1 に対応するバンドを切除します。
- 標準的な DNA 抽出キットを使用して両方のバンドを浄化します。
- 消化されたプラスミッドおよび ORF1 を縛る標準試薬を用いるします。
- ライゲーション キットを使用して材料の表に示されている場合、結紮 100 ng の DNA を含む 3 〜 5 時間モル過剰 4.5 μ L の総ボリュームでプラスミドに挿入します。ライゲーション キットから 2 x T4 リガーゼ バッファーの 5 μ L と T4 DNA リガーゼの 0.5 μ L を追加します。部屋の温度 (RT) で 10 分の 1.5 mL チューブの ligation の反作用を実行します。
- (井上の方法11によると準備) 標準の DH 5 αエシェリヒア属大腸菌の有能なセルに変換します。
- 1.5 mL の有能なセルの 50 μ L の ligation の反作用のミックス 3 μ L 氷の上管および転送熱細胞ブロック 30 45 s 42 ° C までのセットとに予め温めておいた (37 ° C) の 2 分を追加 250 μ L の氷の上に戻ってインキュベーションして 30 分間インキュベートホストゲノム スープ (LB) 中小企業プレート 100 μ L に予め温めておいた (37 ° C) アンピシリン含有 LB 寒天培地プレート上のセルの。37 ° C で一晩細胞をインキュベートします。
- 次の日に 4 ~ 6 のコロニーを拾うと 37 ° C, 180 rpm でそれらを孵化させなさい、15 mL チューブに 100 μg⋅mL-1アンピシリンを含む LB 培地 3 mL に一晩します。
- 標準的な DNA の MiniPrep キットを使用して選ばれた植民地からプラスミドを分離します。
- サンガー カセット 2 逆プライマー (表 2) を用いたシーケンスによってプラスミドの正しさを確認します。
- 最初の ORF を含んでいるプラスミッドを特定したら、次の 2.2 の手順と同じ手順が、PmeI、パーチの酵素を使用してそのプラスミドで 2 番目の ORF を subclone します。
- カセット 1 逆プライマー (表 2) を使用して生成されたプラスミドをシーケンスします。
3. 融合蛋白質のテスト
注: 次の手順は、デュアル誘導発現プラスミド (図 2) と HeLa 11ht セル、subclonal CCL2 HeLa 細胞株、逆テトラサイクリン制御転写活性化因子の情報を頼むこと M2 を安定に発現するを含む、RMCE12の軌跡。これらの細胞の成長媒体はダルベッコ変更イーグル培地 (DMEM) 10% ウシ テトラサイクリン無料胎児血清 (FBS) を含みます。別のセルのタイプを使用する正確な播種条件と成長媒体は適応する必要があります。
-
トランスフェクション
- 種子 1 x 10 の5の濃度で細胞は細胞のトランスフェクション前日 6 ウェル プレートのウェルあたり 2 mL と 37 ° C、5% CO2細胞文化のインキュベーターで一晩でセルを孵化させなさい。
- トランスフェクションの日、各井戸の中を削除し、新鮮な培地 2 mL に置き換えます。
- 表 1に従って 4 つのトランスフェクション反応を準備します。使っても、ポリエチレンイミンの 6 μ g を 1.5 mL 滅菌チューブと血清なし DMEM 培地で 500 μ L に塗りつぶしのプラスミッド DNA の 3 μ g に追加します。
- 賢明なドロップを各ウェルに追加する前に室温で少なくとも 5 分ごとのトランスフェクション ミックスを孵化させなさい。一晩で 37 ° C、5% CO2セルを孵化させなさい。
-
誘導と近接ラベリング
- トランスフェクション後、日はメディアを削除し、ビオチン化を刺激するために 50 μ M でビオチンと融合タンパク質の発現を誘導するために 200 の ng⋅mL−1でドキシサイクリン培に置き換えます。37 ° C、5% CO2で少なくとも 20 時間のセルを孵化させなさい。
- ビオチンの原液をするためには、50 の mg⋅mL-1でビオチンを解散 ( caに対応します。 200 mM) 2 M 水酸化アンモニウム。それを完全に溶解、一度 50 mm 500 mM Hepes、pH 7.4 では、希釈し、塩酸因数で 7.4 に pH を調整して結果 1,000 x 貯蔵液-20 ° C で保存70% エタノールで 10 mg⋅mL-1と暗闇の中での-20 ° C でスクリュー キャップ チューブ内のストアでドキシサイクリンを溶解します。
- トランスフェクション後、日はメディアを削除し、ビオチン化を刺激するために 50 μ M でビオチンと融合タンパク質の発現を誘導するために 200 の ng⋅mL−1でドキシサイクリン培に置き換えます。37 ° C、5% CO2で少なくとも 20 時間のセルを孵化させなさい。
-
細胞ライセートの調製
- 一度風邪 (4 ° C) の 1 mL のリン酸緩衝生理食塩水 (PBS) とセルを洗います。
- 各ウェルに 100 μ L の換散バッファー (50 mM Tris pH 7.4 では、150 mM の NaCl、2 ミリメートルの EDTA、0.5 %np-40 0.5 mM DTT、プロテアーゼ阻害剤) を追加します。
- セル スクラッパーと 1.5 mL チューブへの転送セルを収穫します。
- 14,000 x g細胞の残骸を削除する 4 ° C で 10 分間のサンプルを遠心します。
- 培養上清を新しいチューブに転送し、氷の上に配置します。
- ブラッドフォードの試金とタンパク質の量を定めます。
-
SDS ポリアクリルアミドゲル電気泳動 (ページ) と西部にしみが付くこと
- SDS ポリアクリルアミドのゲルを準備します。
- 各クリア ライセート、SDS の読み込みバッファーの 30 μ L の合計で 30 μ g (最小 15 μ g) 蛋白質のページのサンプルを準備します。SDS ポリアクリルアミドのゲルの各サンプル (20 μ L/ウェル) の同量をロードし、電気泳動に進みます。
- 電気泳動後に、任意の標準的なプロトコルを使用して、低蛍光 PVDF のしみの膜に分別された蛋白質を転送します。
メモ: ビオチン化タンパク質を解析、高速「高 MW」プログラムで 10 分転送時間セミドライ デバイスを通常ウエスタンブロットがうまく転送します。 - 転送後、ブロック 5% 室温 30 分間 PBS で乾燥したミルクの膜
- 2 %bsa および 0.1% を含む PBS で蛍光ストレプトアビジン共役希釈 1:15,000 と RT で 30-60 分のための膜をインキュベート トゥイーン 20。
- 0.1% Tween 20、その後 PBS でより多くを含む PBS で 3 回、各 10 分間、膜を洗浄します。
- 蛍光スキャナーのイメージング システムの膜をスキャンします。
注: A 典型的な西部のしみは、図 3に表示されます。上記の手順は、蛍光検出を説明しますが、HRP 結合ストレプトアビジンを使用して発光検出が均等にうまきます。
4. 分割-BioID プロテオミクス研究
重要なメモ:すべてのケラチン無料条件で実行する次の手順は、最終の質量分析のすべての材料および試薬として可能な限りケラチン無料はずです。
- トランスフェクション
- トランスフェクション、前日プレートあたり 10 mL の 8 x 10 の5セルの濃度条件ごとに 3 〜 4 10 cm シードし、37 ° C、5% CO2細胞文化のインキュベーターで一晩でセルを孵化させなさい。
- 次の日、トランスフェクション条件ごとにマスター トランスフェクション ミックスを準備: 1 つの条件 3 つの板は、ポリエチレンイミン、36 μ g、18 μ g のプラスミド DNA は 900 μ L に溶解した無血清 DMEM。各マスターのミックス ルートで少なくとも 5 分間のインキュベートします。
- 一方で、それぞれの料理の中を新鮮な媒体に置き換えて、各プレートに賢明なトランスフェクション混合ドロップの 300 μ L を追加します。
- 一晩で 37 ° C、5% CO2セルを孵化させなさい。
- 誘導と近接ラベリング
- トランスフェクション後、日は、15 cm の皿にセルを転送します。各料理は、メディアを取り出して、7 ml の PBS のセルを洗浄して、トリプシン EDTA 溶液 1.5 mL を追加、細胞を再懸濁し、細胞懸濁液を 20 ml 成長培地の 15 cm 皿に転送する成長媒体のルート追加 3.5 mL で 5 分間インキュベートビオチン化を刺激するために 50 μ M でビオチンとドキシサイクリン 200 ng⋅mL− 1 (最終濃度) 融合タンパク質の発現を誘導することで補った。
- 37 ° C、5% CO2で少なくとも 20 時間のセルを孵化させなさい。
- 細胞の収穫・
- PBS で 2 回細胞を洗浄し、各プレートに 1.5 mL の PBS を追加、スクラッパーとセルを収穫します。
- 15 mL チューブに 1 つの条件に対応する収穫のセルを転送し、1,200 x g、5 分、4 ° C での遠心分離によってそれらを収穫
- 培養上清を外し、スナップは、さらに処理まで液体窒素 80 ° c 店でペレットを凍結します。
注: また、セルも trypsinization によって取り外される、成長培地で収穫された、15 mL チューブに転送でき凍結前に PBS で 3 時間を洗浄します。
- セル lysates の準備
- 再懸濁します換散バッファー (50 mM トリス pH 7.4 では、500 mM の NaCl、0.4 %sds、5 ミリメートルの EDTA、1 mM DTT、1 完全なプロテアーゼ阻害剤) RT。 峠での 1 mL の細胞ペレット セル 10-20 倍 (5 〜 10 ストローク) 25 G 注射針で。
- サンプル音声化デバイスと超音波照射します。
注: 可聴デバイス材料の表に示されている、次のプログラム使用できます: 高強度、30 で 4 サイクル (4 ° C) 冷サイクル毎秒水浴。他の音声化デバイスが適していますが、パラメーターがそれに応じて調整する必要があります。 - 回復したにトリトン X-100 が 2% の最終濃度に到達するライセート超音波処理を追加 (通常は、100 μ L を追加 20 %900 μ L にトリトン X-100 は細胞ライセートを超音波処理) と 50 mm、トリス、2.3 mL pH = b の前に 150 mm NaCl 濃度を調整する溶解液の 1 mL あたり 7.4ストレプトアビジン結合ビーズに inding。
重要なメモ:高い塩濃度は、ビーズにはるかに少ない効率的なバインディングではよくあります。 - 1.5 mL チューブの調整の lysates を配布 (ca3 つのチューブで 1.1 mL) 16,000 × g、10 分、4 ° c で遠心分離機のことと。
- 培養上清を転送 (ca. 3.2 mL) 15 mL チューブ、および入力材料として保つ 50-100 μ L に。
- ブラッドフォードの試金の各サンプルの濃度を測定し、ストレプトアビジン プルダウン 3 〜 3.5 mg タンパク質含有量に相当します。
- ストレプトアビジン プルダウン
注: プルダウンの手順の概要については、図 4に表示されます。ルーと同僚2発行元のプロトコルとほぼ同じです。プルダウンを実行すると、最初に流れると洗浄 1-4 のサンプルが後回しにする西部のしみの分析のため正しく働いてプロシージャ (図 5)。- 各条件について、ストレプトアビジン結合磁性体ビーズ懸濁液 200 μ L を 1.5 mL チューブに転送します。磁気ラックにチューブを置き、管の側に固執するビーズまで待つ (ca。 1 分) およびストレージ バッファーを削除します。
- 優しく平衡バッファー (50 mM Tris pH 7-4、150 mM NaCl、0.05% トリトン X-100 と 1 mM DTT) の 1 mL を混合することによってビーズを洗浄します。
- 磁気ラック背面チューブ (通常起動時 3-3.5 mg タンパク質のコンテンツ、各状態から溶解液を 2 ~ 4 1.5 mL チューブにディスパッチできます) および場所の必要な数の等しい量の平衡のビーズをディスパッチします。
- 平衡バッファーを削除し、ステップ 4.4.7 から対応するセル lysates の同量とビーズの各セットを再懸濁します。回転ホイールに 4 ° C で一晩インキュベートします。
- 次の日、磁気ラックに 1.5 mL チューブを置き、ビーズ、チューブの側に固執し、フローというレッテルを 15 mL チューブに培養上清を転送するを待ちます。
注: これからのすべての手順を行った室温で別段 - 各管を洗浄バッファー 1 (水 2 %sds) 200 μ l でビードを再停止しなさい、1.5 mL チューブの 1 つの条件に対応する再懸濁のビーズの各セットを組み合わせます。
- 1 ml の洗浄バッファー 1 の回転ホイールに 8 分間 2 回ビーズを洗います。
- 1 mL の洗浄バッファー 2 回転ホイールに 8 分間 2 回ビーズを洗浄 (50 mM HEPES pH 7.4 では、1 mM EDTA、500 mM の NaCl、1% トリトン X-100, 0.1 %na デオキシ コール酸)。
- 1 mL の洗浄バッファー 3 回転ホイールに 8 分間 2 回ビーズを洗う (10 mM Tris pH 8、250 mM LiCl、1 mM EDTA、0.5 %np-40、および 0.5 %na デオキシ コール酸)。
- 1 mL の洗浄バッファー 4 回転ホイールに 8 分間 2 回ビーズを洗浄 (50 mM Tris pH 7.4 では、50 mM NaCl 0.1% NP 40)。
- 最後のステップを洗浄後、洗浄バッファーが完全に削除されていることを確認するには、上澄みの大部分を削除して後、スピンダウン サンプル。マグネッ トラックに戻ってそれらを置く、ビーズ チューブの側面に固執し、バッファーの残りを削除するを待ちます。
- 30 μ L の溶出バッファーを追加 (10 mM Tris pH 7.4 では、2 mM ビオチン、5% β-メルカプトエタノール、2 %sds) ビーズに。98 の ° C、15 分間インキュベートし、磁気ラックのビーズはすぐに削除します。
- さらに処理まで溶出サンプルを新鮮なチューブ、-20 ° C でストアに転送します。
- SDS のページおよび西部にしみが付くこと
注: 質量分析の前に勧めしますビオチン化やプルダウンの成功を評価するために SDS-PAGE と西部のしみ。1 つはことを考慮可能性がありますビオチン化パターンが観測されない場合、dox またはビオチンが媒体に追加されませんでしたまたは株式の 2 つのソリューションのいずれかが侵害されました。- それぞれの入力サンプルを 28 μ L の合計で 3 x SDS の読み込みバッファーの適切なボリュームと蛋白質のサンプルの同じ量を混合することによってページのサンプルを準備します。3 x SDS の読み込みバッファーの 2.5 μ L で各溶出サンプルの 5 μ L を混合することによって、ページのサンプルを準備します。
- SDS ポリアクリルアミドのゲルのサンプル (入力のため 25 μ L/ウェル、溶出サンプル 7 μ L/ウェル) をロードします。電気泳動、ウエスタンブロット セクション 3.4 で説明するように進みます。
- 初めてのプルダウンを行う場合準備ページ プルダウン (入力、流れ、洗浄 1-4 溶出) の各ステップのサンプル各サンプル (入力、流れ、洗浄 1-4) SDS 読み込みバッファーまたは 3 の 2.5 μ L、5 μ L (溶出) x 3 の 10 μ L の 20 μ L を混合することによって S xDS 読み込みバッファーし、4.6.4 (図 5) の手順に進みます。
- MS 分析用 SDS ページ
注: ケラチンの潜在的な汚染を最小限に抑えるプレキャスト ゲルと商業サンプル読み込みバッファー可能性があります使用。- 各溶出サンプルの 18.75 μ L に 4 x サンプルバッファーの 6.25 μ L を追加し、ゲルに 2-3 cm を移行するまで、プレキャスト 4-20 %sds ゲルのサンプルを実行します。
- 13 15 cm のシャーレを染色コロイド Coomassie の鮮やかなブルー G250 とゲルを染色します。
- (約 17 kDa、図 6で実行)、ストレプトアビジン バンドを除く、各サンプルの全体のレーンを切除し、摘出バンドを 1.5 mL チューブに転送するには、きれいなメスを使用します。
- さらに分析のためのプロテオミクス施設にこれらのサンプルを送信します。
注: 参照 9 (図 3および図 7、および補足テーブル) の典型的な MS の結果を見ることができます。プライド リポジトリ (加入番号 PXD005005) に全体のデータセットがあります。
結果
このメソッドがどのように動作するか、Ago2 タンパク質の開いたリーディング ・ フレーム (ORFs) を説明するために TNRC6C およびダイサー (すべてのマイクロ Rna を介した遺伝子サイレンシング経路の関係者) は分割 BioID プラスミッドの複製だった。Ago2 翻訳を抑圧する miRNA によるサイレンシング複合 (miRISC) 内で TNRC6C と対話し、ターゲット Mrna14の崩壊を刺激する知られています。MiRISC、Ago2 をアセンブルする前に、ダイサー、複合施設では、miRNA15にロードを取得可能性があります内の成熟 Mirna を生成する酵素と対話します。したがって分割 BioID は Ago2/ダイサー ペアまたは Ago2/TNRC6C ペアに適用されました。テストされた蛋白質の各ペア、Ago2 だった NBirA * または CBirA * 私たちの分割 BioID プラスミド (図 2) を使用していずれかの融合、ダイサーや TNRC6C に対応する同種・ ビラ * フラグメントします。さらに、各蛋白質を融合した CBirA * され、NBirA * と対になって-ネガティブ コントロールとして GFP 融合。テスト蛋白質 (表 1) のペアごとに 4 つのイテレーションのテストでこの結果します。
分割 BioID がテストされた蛋白質のペアが相互にアクティブ化されるかどうかをテストするのには図 1に描かれているスキームを追ったプラスミッドは一過性テト システム互換性のある HeLa 細胞ラインに導入させた。ドキシサイクリン (dox) で誘導された融合タンパク質の発現とビオチン化は成長媒体に過剰なビオチンを追加することによって刺激されました。Dox とビオチン 20 h 培養時間、続いて細胞分離, ビオチン化タンパク質を検出する共役ストレプトアビジンを用いたウェスタンブロッテイングにより分析した.哺乳類セルの 2 つの主要なバンドは、通常融合のサンプル (図 3星) の共役ストレプトアビジンで検出、内生ビオチン化タンパク質 (最もおそらくミトコンドリア カルボキシラーゼ) に対応します。これらの 2 つのバンドがすべてのサンプルに存在し内部のローディング コントロールとして便利に使用ことができます、したがって、同じ蛋白質量の読み込みを制御するハウスキーピング蛋白の検出は不要です。BioID/分割-BioID の実験のために典型的、観察することができます追加の主要なバンド、自己ビオチン標識を持って融合蛋白質であります。他のビオチン化タンパク質を見てない場合でもはこの段階で既に融合タンパク質のビオチン化を検出テストの 2 つの蛋白質が細胞の相互作用を示します。図 3に描かれている実験ではオフに NBirA * を持つ-CBirA * TNRC6C またはダイサー融合と共に Ago2 融合蛋白質がどの CBirA * で逆の組み合わせよりも効率的-Ago2 NBirA * 他の 2 つの融合にペアになっています(図 3、上部のパネル比較レーン 6 7 2 3 車線の強度) 蛋白質。NBirA * CBirA * 融合のどれもを有効にできるようまた、活性化された特定のかなりのレベル (図 3、比較車線 1、融合細胞に対応するレーン 8 4 5) に GFP 制御融合タンパク質。私たちのプラスミッドの NBirA * が myc タグ、CBirA * フラグの札 (図 2) には、それぞれ融合タンパク質の発現レベルはこれらの 2 つのタグ (図 3底板) に対する抗体と分析できます。
相互作用に基づくビオチン化を観察すると、実験をスケール アップすることができます、プロトコル (図 4) の 4 項に示されているように、ストレプトアビジン結合ビーズにビオチン化タンパク質が分離されました。分離の最初の時間を実行すると、西部 (図 5) あぶらとりによって浄化のすべての手順を分析する可能性があります。通常、ビーズに結合がほぼ定量的にする必要があります、事実上を通じてリークする必要がありますも見られなくなる洗浄中。事前処理の質量分析のためのサンプル、誘導ビオチン化想定どおり、融合蛋白質を表現したように西部のしみを実行します。融合蛋白質の表現の欠如は、貧しいトランスフェクション効率か障害のある dox 誘導です。融合蛋白質を表現したビオチン化が認められなかった場合は、余分なビオチン (50 μ M) がメディアに実際に追加されたかどうか、在庫のビオチンがまだアクティブであるを確認します。通常、Coomassie 染色蛋白質ゲル (図 6) の溶出の材料を分析観察する最強のバンドは約 17 kDa で実行され、単量体ストレプトアビジンに対応します。内因性ビオチン化タンパク質との融合タンパク質に対応するバンドも観察することがあります。我々 は通常読み込みも (図 6) までストレプトアビジン バンド上記サンプル車線部を切除します。摘出したバンドを 1.5 mL チューブに格納して質量分析施設に送信できます。または、バインドされた蛋白質はストレプトアビジン結合ビーズのトリプシン消化もあります、消化ペプチドの溶出が列を形成します。我々 は定期的に MaxQuant ソフトウェア16 (ほとんどの既定のパラメーターを使用して、参照 9 典型的な MS の結果の詳細についてを参照してください可能な翻訳後修飾としてリジン ビオチン化を追加する、) を使用して MS の生データを分析して、ペルセウス スイート17以降の統計解析では、両方ともフリー ソフトウェア。サンプルは、3 つの生物学的複製で通常実行されます。無料のラベルの数量を使用して、制御条件を具体的に濃縮タンパク質を識別できます。内生ビオチン化タンパク質とタンパク質・ ビラ * 酵素によりラベル付けされた非具体的にフィルターを適用するには、と六つの無関係な蛋白質で生成された六つのデータセットからのヒットは、豊かさを大幅蛋白質のみ考えています。さらに、我々 はのみによって NBirA * NBirA * の融合蛋白質が置き換えられて分割 BioID データセットに濃縮されてヒットを考慮-GFP。特に定量的プロテオミクス18細胞培養 (SILAC) のアミノ酸組成と安定同位体標識を使用して他のデータ解析方法が提案されています。さらに、様々 な戦略は、ビオチン化ペプチド有機溶剤19またはビオチン固有を使用して特別な溶出条件ビオチン18、弱体化した親和性ストレプトアビジン バリアントを使用しての直接分離について記載されています。抗体20,21。必ずしも多くのタンパク質の発見につながる、ながらビオチン化部位の同定は、ヒットの特異性に関してもっと自信を追加、役に立つときは、相互作用のトポロジに対処します。
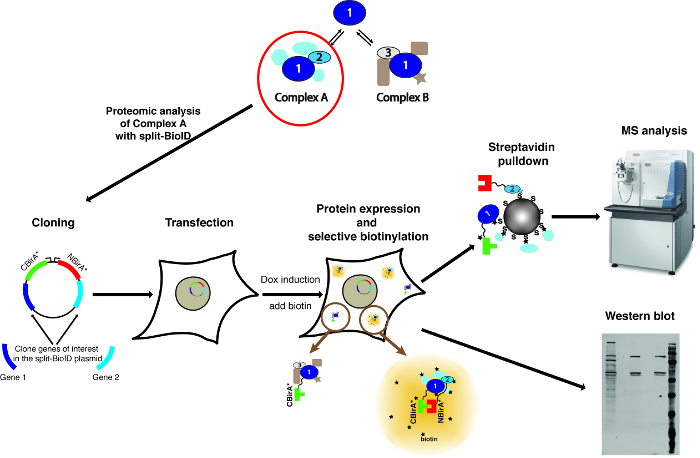
図 1: 分割 BioID プロシージャの概要。タンパク質 1 対話タンパク質 2 複雑な A の一部として、複雑な B の一部としてタンパク質 3特に体の組成を調べる、分割 BioID は 1 と 2 の蛋白質に適用できます。質量分析計の写真はクリエイティブ ・ コモンズ帰属-シェアも 3.0 unported 派生ライセンスの下で、ThermoScientificOrbitrapElite.JPG のファイル名を https://commons.wikimedia.org からダウンロードしました。この図の拡大版を表示するのにはここをクリックしてください。
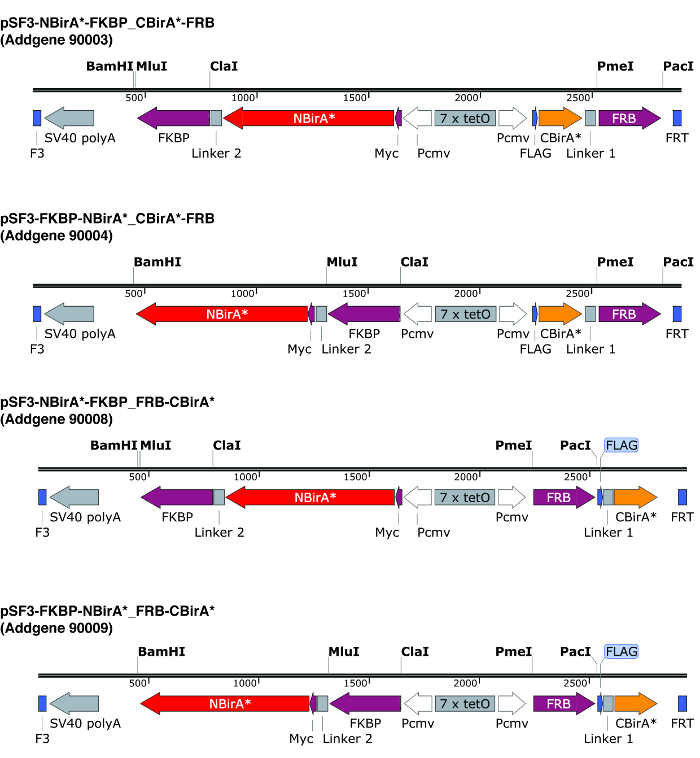
図 2: 分割 BioID プラスミドの式カセット。NBirA *、CBirA * の融合蛋白質のすべての組み合わせをテストできるように 4 つのプラスミドを提供します。指定された番号の下 addgene.org でプラスミドと完全なマップがあります。プラスミッドはテト応答要素 (7 x テト) とテト式システムと互換性がある細胞ラインで使用する必要があります。またすべてのプラスミッドの Orf の FKBP と FRB は NBirA * に融合し、CBirA * それぞれの断片を注意してください。ラパマイシンの存在下でこれら 2 つのタンパク質が相互作用し、したがって、プラスミドはこの化学9の有無でシステムをすばやく確認する使用ことができます。指定された制限のサイト、ユニークです。この図の拡大版を表示するのにはここをクリックしてください。
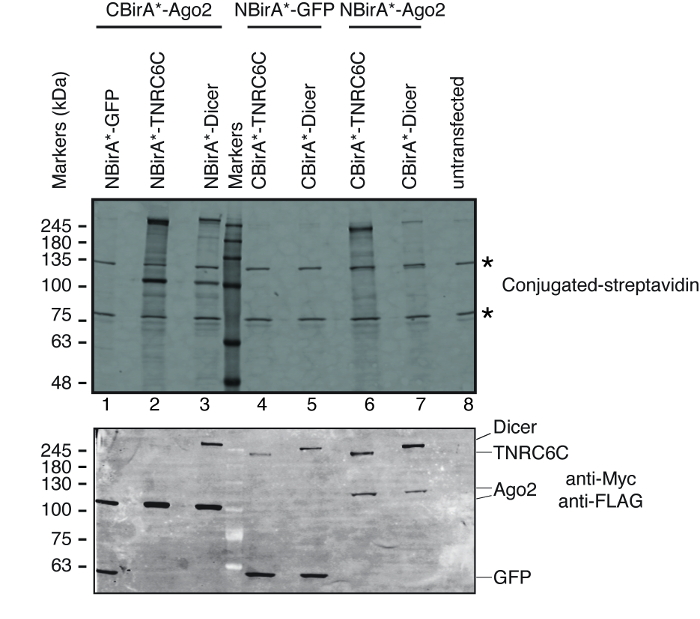
図 3: 分割 BioID 実験のための典型的な西部のしみ方。上部パネル: 蛍光標識ストレプトアビジンとビオチン化タンパク質の検出。下部のパネル: 反 Myc および反フラグ抗体との融合蛋白質の検出。蛋白質の 2 つのペアがテストされた: Ago2/TNRC6C と Ago2/ダイサー。2 & 3 の車線、Ago2 が CBirA の断片に追加されました。6 & 7 の車線、Ago2 が NBirA の断片に追加されました。3 つのタンパク質のいずれかが NBirA * と結合されたとき重要な信号が認められなかった-GFP (レーン 1、4-5)。星は、内部のローディング コントロールとして使用できるビオチン化タンパク質内生に対応するバンドを示します。この図は図 5 b出ないでらから適応9クリエイティブ ・ コモンズ帰属 4.0 国際ライセンスの下で。この図の拡大版を表示するのにはここをクリックしてください。
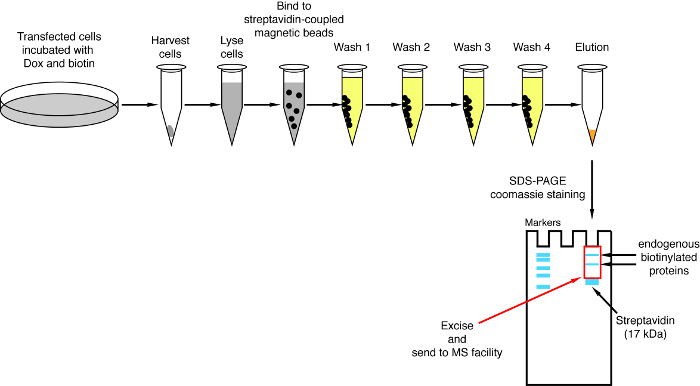
図 4: ストレプトアビジン プルダウン手順の概要です。ビオチン化タンパク質質量分析のための分離のための主な手順が描かれています。この図の拡大版を表示するのにはここをクリックしてください。
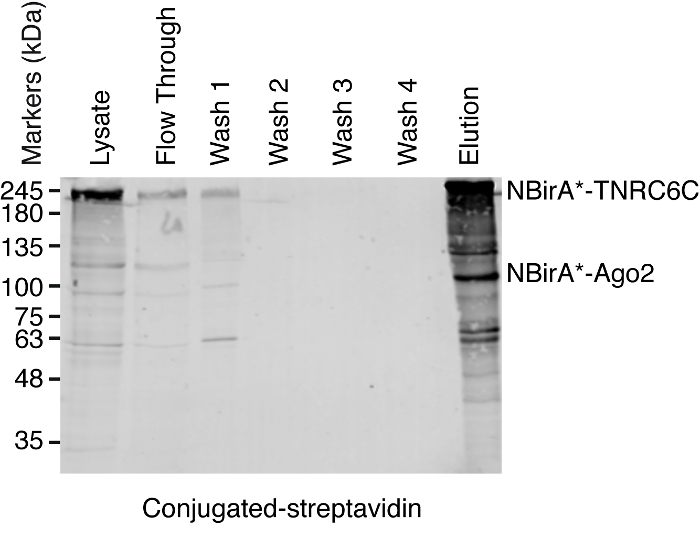
図 5: ストレプトアビジン プルダウン実験のための典型的な西部のしみ方。それぞれの指定されたサンプルのボリュームを等しく SDS ポリアクリルアミドのゲルの読み込まれました。次の西部のしみが付くこと、HRP 結合ストレプトアビジンとビオチン化タンパク質が検出されました。NBirA * に対応するバンド-TNRC6C と CBirA *-Ago2 が示されます。この図の拡大版を表示するのにはここをクリックしてください。
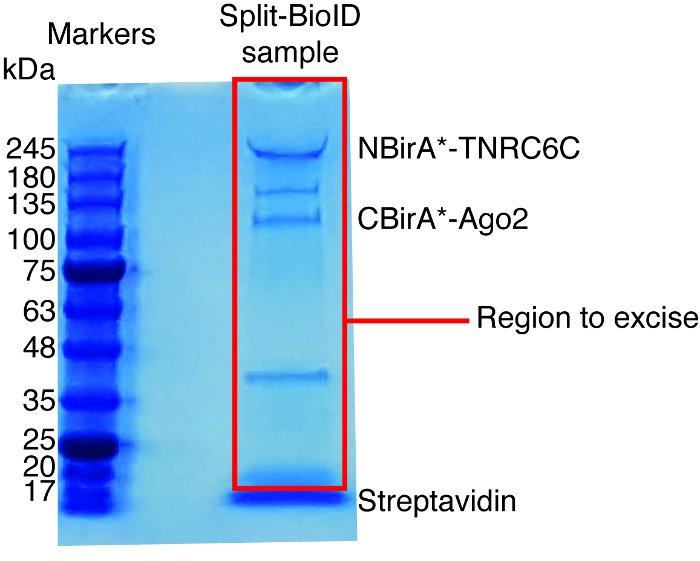
図 6: 質量分析の典型的な Coomassie 染色蛋白質ゲル。ストレプトアビジン結合ビーズから溶出サンプルがプレキャスト蛋白質ゲルのロードされ、サンプル 2-3 cm に移行までを実行します。約 17 kDa で見られる主要なバンドは、ストレプトアビジンです。そのバンドの直接上の領域、質量分析施設に送信されます。NBirA * に対応するバンド-TNRC6C と CBirA *-Ago2 が示されます。この図の拡大版を表示するのにはここをクリックしてください。
| トランスフェクション サンプル | テスト条件 | ||
| 1 | NBirA *-protein1/CBirA *-protein2 | ||
| 2 | CBirA *-protein1/NBirA *-protein2 | ||
| 3 | NBirA *-GFP/CBirA *-protein1 | ||
| 4 | NBirA *-GFP/CBirA *-protein2 | ||
| 5 | ないトランスフェクション | ||
表 1: は通常分割 BioID を 2 つの蛋白質に適用する場合の条件をテストしました。
| シーケンス入門 | シーケンス |
| カセット 1 逆プライマー (CBirA * 融合) | TATACTTTCTAGAGAATAGGAAC |
| カセット 2 逆プライマー (NBirA * 融合) | GTGGTTTGTCCAAACTCATC |
表 2: 配列分割 BioID プラスミッドのためのプライマー。
ディスカッション
記載の手順では、プラスミド、相互作用に基づく講演をテストする方法および質量分析のためのビオチン化タンパク質を特定する方法の分割 BioID に興味のある遺伝子のクローンを作成する方法について説明します。トランスフェクションに基づいて手順をご紹介します。融合タンパク質の発現は、培地に添加 dox の量によって調整できるトランスフェクション著しく低下、内因性と比較した場合の融合蛋白質が癌細胞と非均質なタンパク質発現につながる可能性があります。対応。これは対応する interactomes の歪みと PPI 内因性のタンパク質を含む相互作用を忠実に反映していない可能性があります。つまり、一般的に分割 BioID を確立する一時的なシステムで安定したセルラインを構築することをお勧め。プラスミッドは Flp を介する組み換えシステムと互換性があり、同じテト応答要素の規則の下で興味の両方の遺伝子を配置します。必要な場合、互換性のある哺乳類セルで使用した場合、彼らは安定した誘導性細胞を簡単に作成できます。たとえば、情報を頼むことテトラサイクリン アクティブ化される転写活性化因子およびテトラサイクリンを介した遺伝子発現が厳しく規制された12をすることができますユニークな対象 genomic 位置を表す HeLa EM2 11 行を使用します。この細胞ラインと Flp を介する組み換えを使用して、遺伝子のコピーを 1 つだけを含む安定したセルラインは 2-3 週間以内に取得できます。また、興味のある遺伝子のネイティブのゲノム遺伝子座の BirA * 断片を紹介するのに現行ゲノム編集技術を使用することも 1 つ。
タンパク質のタグ付けに依存する任意のアッセイのように結果の融合蛋白質が機能している考慮する 1 つ必要があります。利用可能なデータを興味の蛋白質が付いた (たとえばイメージング研究の GFP) と機能的テスト役・ ビラのフラグメントであるべきかどうかを決定するクローン上流または下流遺伝子の発現。このようなデータが利用できない場合、N 末期 1 つテストする必要があります。 または C 末端タグ付きタンパク質の機能アッセイに。たとえば、する内因性のタンパク質ノックアウトされ野生タイプの状況と比較してセル行の融合蛋白質の活動をテストできます。興味の蛋白質は両方の N 末端と C 末端のタグを容認する場合、両方テストする必要があります。確かに、BioID 実験の融合蛋白質の向きは22のラベル付けの効率を影響します。分割 BioID を蛋白質のペアに適用し、2 つのタンパク質のうち、どちらかの NBirA * に追加されますまたは CBirA * フラグメントも影響9のラベル付けの効率とさらに、我々 はそれを観察しました。分割 BioID プラスミッドの 16 アミノ酸長いグリシン/セリン豊かなリンカー ・ ビラのフラグメントに興味の蛋白質を結合は別の PCA23から撮影され、私たちのすべての相互作用の蛋白質の仕事我々 テストしているところです。ただし、いくつかのタンパク質のペアが短いまたは長いリンカーとよりよく働くかもしれない 1 つをお勧めします。最終的な注記のもう一つの試金は Bollen グループ24によって記述されていた。このアッセイの BirA * で分割されます我々 (E256/G257) より別のサイト (E140/Q141)。我々 は両方分割 BioID 味サイド バイ サイドをテストし、E256/G257 このプロトコルで記述されているがより強力な再活性化 2 つの相互作用の蛋白質に結合するときにつながることを発見9。
このメソッドの 1 つの一般的な欠点は、ラベリングの遅い速度です。通常、ビオチンと 6 ~ 24 時間培養時間はかなりビオチン化6、タンパク質複合体の動的な改造を勉強のためこのテクニックの使用は除外を得る必要です。この試金は部分的にそれは 2 つのタンパク質が相互作用するときにのみアクティブ化としてこの警告をアドレス、ラベルの低速は非常にダイナミックなプロセスまたは短命の蛋白質を分析する応答を勉強するための使用を排除します。設計のペルオキシダーゼ APEX2 は 1 分3で近位蛋白質の効率的な分類を促進するために知られています。APEX2 に基づく PCA したがって BioID 由来の試金の遅いラベリング速度の制限に対処可能性があります。原理の実証研究では、このような分割 APEX2 アッセイ25をについて説明します。ただし、homodimerizing 蛋白質は正常にビオチン化の示されるべきままラベルし、相互作用の蛋白質のまわりを組み立てる蛋白質を識別する試金することができます使用もかどうか。非常に最近では、進化は、TurboID と miniTurbo、2 つの亜種の BirA * 活性の増強とはるかに短いラベルの時間ウィンドウを 10 分26まで許可するを作成に使用されました。これらの新しい亜種に分割 BioID を適応させると、アプリケーションの広範な分野にこの技術の使用をさらに拡張します。
開示事項
著者が明らかに何もありません。
謝辞
この作品は、ドイツのエクセレンス イニシアチブ (CellNetworks DFG EXC 81) と共同研究センター SFB638 によって部分的な融資を通じてドイツの研究議会 (DFG) によって融資されました。
資料
| Name | Company | Catalog Number | Comments |
| Acetic acid (glacial) | VWR | 20104.298 | To make TAE buffer |
| Agarose | Sigma | A9539 | Take TAE-agarose gels for DNA analysis and extraction |
| Ammonia solution 25% NH3 | Bernd Kraft | 6012 | To dissolve biotin |
| Ampicillin | Sigma | A9518 | To select transformed bacteria |
| Bioruptor plus sonification device | Diagenode | B01020001 | Other sonification devices are also ok |
| Biotin | Sigma | B4639 | To be added to the growth medium to stimulate efficient biotinylation |
| Bovine serum albumin fraction V | Carl Roth | 8076 | Used in Western blot buffers and a protein standard in Bradford assays |
| Bradford Ultra reagent | Expedeon | BFU05L | Any other method/kit for protein determination is fine, this particular reagent is more tolerant to detergent than other Bradford reagents |
| Cell scrappers | TPP | 99002 | Any other model is also fine |
| ClaI | New England Biolabs | R0197 | Restriction enzyme for cloning into the split-BioID plasmids (NBirA* fusion) |
| DMEM medium | Sigma | D6046 | If using another cell line, use the corresponding optimal growth medium |
| DNA miniprep kit | Sigma | PLN350 | Any other kit is also fine |
| Doxycycline | Applichem | A2951 | Dox is light sensitive |
| DTT | Applichem | A2948 | Make 1M stock solution, store at -20 °C and always use fresh |
| DyLight 680-conjugated streptavidin | Thermo scientific | 21848 | to use with a LiCor Western blot scanning device |
| Dynabeads MyOne Streptavidin C1 | Invitrogen | 65002 | The C1 beads are not BSA coated which is preferable for downstream MS applications (no leakthrough of BSA in the final elution) |
| EDTA | Applichem | A5097 | Make a 500 mM stock, adjust pH to 8 while dissolving the EDTA powder |
| Ethanol | Sigma | 32205 | Make a 70% stock solution in which Doxycycline can be dissolved at 10 mg.mL-1 |
| Fastgene Gel/PCR DNA Extraction Kit | Nippon Genetics | FG-91302 | Any other kit is also fine |
| HCl 37% | Merck | 1.00317.1000 | To adjust pH of biotin stock solution |
| HEPES | Carl Roth | 6763 | Make a 500 mM stock solution, adjust the pH to 7.4 |
| Immobilon-FL PVDF membrane, 0.45 µm | Millipore | IPFL00010 | This membrane shows minimal autofluorescence when used with a LiCor Western blot scanning device |
| LiCl | Grüssing GmbH | 12083 | Make a 5M stock solution |
| Odyssey CLx imaging system | LI-COR | N/A | To scan Western blot membrane decorated with fluorophore-labeled antibody |
| Linear polyethylenimine (PEI) | Polysciences | 23966-2 | Any other transfection reagent is also fine |
| Milk powder | Carl Roth | T145 | To block Western blot membranes |
| MluI-HF | New England Biolabs | R3198 | Restriction enzyme for cloning into the split-BioID plasmids (NBirA* fusion) |
| Na-deoxycholate | Sigma | 30970 | Make a 10% (w/v) stock solution |
| NaCl | Sigma | 31434 | Make a 5M stock solution |
| NP-40 (Nonidet P40 substitute) | Sigma | 74385 | Make a 20% (v/v) stock solution |
| PacI | New England Biolabs | R0547 | Restriction enzyme for cloning into the split-BioID plasmids (CBirA* fusion) |
| Phosphate buffer saline (PBS) | Sigma | 806552 | To wash cells before scrapping |
| PmeI | New England Biolabs | R0560 | Restriction enzyme for cloning into the split-BioID plasmids (CBirA* fusion) |
| Protease inhibitor cocktail | Roche | 4693132001 | Added to the lysis buffer to prevent protein degradation |
| pSF3-Flag-CBir-FRB_Myc-NBir-FKBP | Addgene | 90003 | Split-BioID plasmid, mediates the co-expression of NBirA*-FKBP and CBirA*-FRB, FKBP and FRB can be replaced by two other ORFs |
| pSF3-Flag-CBir-FRB_FKBP-Myc-Nbir | Addgene | 90004 | Split-BioID plasmid, mediates the co-expression of FKBP-NBirA* and CBirA*-FRB, FKBP and FRB can be replaced by two other ORFs |
| pSF3-Flag-FRB-Cbir_Myc-NBir-FKBP | Addgene | 90008 | Split-BioID plasmid, mediates the co-expression of NBirA*-FKBP and FRB-CBirA*, FKBP and FRB can be replaced by two other ORFs |
| pSF3-Flag-FRB-Cbir_FKBP-Myc-Nbir | Addgene | 90009 | Split-BioID plasmid, mediates the co-expression of FKBP-NBirA* and FRB-CBirA*, FKBP and FRB can be replaced by two other ORFs |
| Q5 High-Fidelity PCR kit | New England Biolabs | E0555S | To amplify the ORF coding for the proteins to be tested. Any other thermostable DNA polymerase is fine. |
| Quick ligation kit | New England Biolabs | M2200S | To ligate DNA fragments into the split-BioID plasmids, any other DNA ligation system is fine. |
| RunBlue 4-20% SDS precast gels | Expedeon | BCG42012 | To use when running samples for MS analysis |
| RunBlue LDS Sample Buffer | Expedeon | NXB31010 | Running buffer for the RunBlue precast gels |
| SDS | Sigma | 5030 | Comes as a 20% stock solution |
| tet-free serum | Biowest | S181T | we use tet-free serum to minimize basal expression of the fusion proteins |
| Trans-Blot Turbo Transfer system | Bio-Rad | 1704150 | High speed Western blotting transfer system, any other transfer system is also fine |
| Tris | Carl Roth | 4855 | Make 1M stock solutions with adequate pH (7.4 and 8) |
| Triton X-100 | Applichem | A4975 | Make a 20% (v/v) stock solution |
| Tween-20 | Carl Roth | 9127 | Used in Western blot buffers, Tween 20 leads to high background fluorescence and should be omitted in the blocking and last wash step |
参考文献
- Scott, D. E., Bayly, A. R., Abell, C., Skidmore, J. Small molecules, big targets: drug discovery faces the protein-protein interaction challenge. Nat Rev Drug Discov. 15 (8), 533-550 (2016).
- Roux, K. J., Kim, D. I., Raida, M., Burke, B. A promiscuous biotin ligase fusion protein identifies proximal and interacting proteins in mammalian cells. J Cell Biol. 196 (6), 801-810 (2012).
- Lam, S. S., et al. Directed evolution of APEX2 for electron microscopy and proximity labeling. Nat Methods. 12 (1), 51-54 (2015).
- Rhee, H. W., et al. Proteomic mapping of mitochondria in living cells via spatially restricted enzymatic tagging. Science. 339 (6125), 1328-1331 (2013).
- Kim, D. I., et al. Probing nuclear pore complex architecture with proximity-dependent biotinylation. P Natl Acad Sci USA. 111 (24), E2453-E2461 (2014).
- Kim, D. I., et al. An improved smaller biotin ligase for BioID proximity labeling. Mol Biol Cell. 27 (8), 1188-1196 (2016).
- Lambert, J. P., Tucholska, M., Go, C., Knight, J. D., Gingras, A. C. Proximity biotinylation and affinity purification are complementary approaches for the interactome mapping of chromatin-associated protein complexes. J Proteomics. 118, 81-94 (2015).
- Morriswood, B., et al. Novel bilobe components in Trypanosoma brucei identified using proximity-dependent biotinylation. Eukaryot Cell. 12 (2), 356-367 (2013).
- Schopp, I. M., et al. Split-BioID a conditional proteomics approach to monitor the composition of spatiotemporally defined protein complexes. Nat Commun. 8, (2017).
- Béthune, J., Artus-Revel, C. G., Filipowicz, W. Kinetic analysis reveals successive steps leading to miRNA-mediated silencing in mammalian cells. EMBO Rep. 13 (8), 716-723 (2012).
- Inoue, H., Nojima, H., Okayama, H. High efficiency transformation of Escherichia coli with plasmids. Gene. 96 (1), 23-28 (1990).
- Weidenfeld, I., et al. Inducible expression of coding and inhibitory RNAs from retargetable genomic loci. Nucleic Acids Res. 37 (7), e50(2009).
- Dyballa, N., Metzger, S. Fast and sensitive colloidal coomassie G-250 staining for proteins in polyacrylamide gels. J Vis Exp. (30), (2009).
- Jonas, S., Izaurralde, E. Towards a molecular understanding of microRNA-mediated gene silencing. Nat Rev Genet. 16 (7), 421-433 (2015).
- MacRae, I. J., Ma, E., Zhou, M., Robinson, C. V., Doudna, J. A. In vitro reconstitution of the human RISC-loading complex. P Natl Acad Sci USA. 105 (2), 512-517 (2008).
- Cox, J., Mann, M. MaxQuant enables high peptide identification rates, individualized p.p.b.-range mass accuracies and proteome-wide protein quantification. Nat Biotechnol. 26 (12), 1367-1372 (2008).
- Tyanova, S., et al. The Perseus computational platform for comprehensive analysis of (prote)omics data. Nat Methods. 13 (9), 731-740 (2016).
- Opitz, N., et al. Capturing the Asc1p/Receptor for Activated C Kinase 1 (RACK1) Microenvironment at the Head Region of the 40S Ribosome with Quantitative BioID in Yeast. Mol Cell Proteomics. 16 (12), 2199-2218 (2017).
- Mackmull, M. T., et al. Landscape of nuclear transport receptor cargo specificity. Mol Syst Biol. 13 (12), 962(2017).
- Kim, D. I., et al. BioSITe: A Method for Direct Detection and Quantitation of Site-Specific Biotinylation. J Proteome Res. , (2017).
- Udeshi, N. D., et al. Antibodies to biotin enable large-scale detection of biotinylation sites on proteins. Nat Methods. 14 (12), 1167-1170 (2017).
- Chapat, C., et al. Cap-binding protein 4EHP effects translation silencing by microRNAs. P Natl Acad Sci USA. 114 (21), 5425-5430 (2017).
- Luker, K. E., et al. Kinetics of regulated protein-protein interactions revealed with firefly luciferase complementation imaging in cells and living animals. P Natl Acad Sci USA. 101 (33), 12288-12293 (2004).
- De Munter, S., et al. Split-BioID: a proximity biotinylation assay for dimerization-dependent protein interactions. FEBS Lett. 591 (2), 415-424 (2017).
- Xue, M., et al. Optimizing the fragment complementation of APEX2 for detection of specific protein-protein interactions in live cells. Sci Rep. 7 (1), 12039(2017).
- Branon, T. C., et al. Directed evolution of TurboID for efficient proximity labeling in living cells and organisms. bioRxiv. , (2017).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved