Method Article
Split-BioID — L’analyse protéomique des Complexes de protéines spécifiques au contexte dans leur environnement natif cellulaire
Dans cet article
Résumé
Nous fournissons un protocole étape par étape pour split-BioID, une analyse de fragments-complémentation protéique basée sur la technique de marquage proximité BioID. Activé sur l’interaction de deux protéines donnés, elle permet l’analyse protéomique de complexes protéiques contextuelle dans leur environnement cellulaire native. La méthode est simple, rentable et ne nécessite que le matériel de laboratoire standard.
Résumé
Purification d’affinité existant (AP) en complément des approches pour l’identification des interactions protéine-protéine (PPI), enzymes ont été introduites qui permettent l’étiquetage de proximité dépendant des protéines en cellules vivantes. Une telle enzyme, BirA * (utilisés dans la méthode BioID), intervient dans la biotinylation des protéines dans un rayon d’environ 10 nm. Par conséquent, quand fusionné à une protéine d’intérêt et exprimés dans les cellules, il permet le marquage des protéines proximales dans leur milieu d’origine. Plutôt que de l’AP qui s’appuie sur la purification de complexes protéiques assemblé, BioID détecte les protéines qui ont été marqués dans les cellules, peu importe si elles sont toujours en interaction avec la protéine d’intérêt lorsqu’ils sont isolés. Car il biotinylates protéines proximale, un peut plus capitaliser sur l’affinité exceptionnelle de streptavidine pour la biotine pour les isoler très efficacement. Tandis que BioID exécute mieux que AP pour l’identification transitoire ou des interactions faibles, ces deux approches de spectrométrie de masse-AP et BioID donnent un aperçu de toutes les interactions possibles, qu'une protéine donnée peut avoir. Toutefois, ils ne fournissent pas d’informations sur le contexte de chaque PPI identifié. En effet, la plupart des protéines font généralement partie de plusieurs complexes, correspondant à des étapes distinctes de maturation ou de différentes unités fonctionnelles. Pour remédier à cette limitation commune de ces deux méthodes, nous avons conçu une analyse de complémentation de fragments protéiques basée sur l’enzyme BirA *. Dans cet essai, deux fragments inactifs de la BirA peuvent remonter dans une enzyme active lorsque présentée à proximité de deux protéines qui interagissent à laquelle ils sont fusionnés. Le test de split-BioID qui en résulte permet le marquage de protéines qui se réunir autour d’une paire de protéines qui interagissent. Condition que ces deux seulement interagir dans un contexte donné, split-BioID permet ensuite l’analyse des unités fonctionnelles spécifiques dépendant du contexte dans leur environnement cellulaire native. Ici, nous fournissons un protocole étape par étape afin de tester et d’appliquer des split-BioID à une paire de protéines qui interagissent.
Introduction
Comme plus cellulaires sont assumées par les protéines qu’assemblent dynamiquement des complexes macromoléculaires, l’identification des interactions protéine-protéine (PPI) est un effort majeur dans la recherche biomédicale. En effet, les PPI sont souvent déréglés dans la maladie et représentent des cibles potentielles pour la thérapeutique1. Le plus largement utilisé la méthode pour l’identification des IPP est la démarche de purification (AP) d’affinité dans lequel, suite à la lyse des cellules, une protéine d’intérêt est plus particulièrement épurée sur une matrice et protéines associées sont par la suite identifiés par spectrométrie de masse (MS). AP-MS est une approche puissante, il généralement n’effectue pas bien sur les complexes de protéine peu solubles, des interactions très transitoires ou PPI qui nécessitent une structure subcellulaire intacte. En outre, l’interprétation des données peut être compliquée par la nature dynamique des réseaux de l’IPP, car une seule protéine fait souvent partie de plusieurs complexes de protéines distinctes.
Techniques de proximité-étiquetage comme BioID2 ou3,de APEX24 ont été récemment mis au point pour répondre à certaines des limitations des approches AP-MS. Dans BioID, l’enzyme BirA * (correspondant à une variante de G115R de l’enzyme d’e. coli de souche sauvage) catalyse la formation de labile biotinyl-AMP (bio-AMP) qui peut réagir avec des amines primaires. Plutôt que de l’enzyme de type sauvage, qui conserve bio-AMP en son centre actif, BirA * libère bio-AMP permettant sa diffusion à son environnement voisin. Par conséquent, quand fusionné à une protéine d’intérêt et exprimés dans les cellules, protéines proximales peuvent être biotinylé dans une fourchette estimée de 10 nm5. Ces marqués proximales protéines sont ensuite isolés par la Streptavidine pulldown et identifiés par SM. Par opposition à AP-MS, BioID nécessite l’expression d’une protéine de fusion. Il peut donc seulement être appliqué aux protéines dont la fonction n’est pas gênée par le marquage. En outre, la vitesse de marquage est lente, généralement 6 à 24 h2,6, rendant la détection des protéines éphémères difficiles. Pourtant, comparativement à AP-MS, BioID-MS offre plusieurs avantages principaux : tout d’abord, ses captures les interactions dans leur environnement natif cellulaire ; Deuxièmement, étiquetés protéines plutôt que complexes assemblés sont isolés après la lyse cellulaire ; en troisième lieu, streptavidine pulldowns permettent à l’aide de tampons de dénaturation et de conditions de lavage dure. Par conséquent, la méthode est plus sensible pour détecter les transitoires, les interactions faibles7 ou les interactions qui se produisent sur un spécifique et difficile d’isoler la structure subcellulaire8.
Cependant, la plupart des protéines font généralement partie des plus grands complexes qui peuvent remodeler selon les signaux cellulaires ou à la fonction qui doit être effectuée. Par conséquent, une seule protéine appartient généralement plusieurs complexes, correspondant à des unités fonctionnelles distinctes, mettant en cause distinct et/ou chevauchement PPI. Les deux approches donnent un aperçu de toutes les associations, qu'une protéine donnée peut avoir, mais ils ne parviennent pas à traiter le cadre du PPI individuel. Pour augmenter la résolution de ce dernier, nous avons conçu une analyse de complémentation de fragments protéiques (APC) dans lequel deux fragments inactifs de BirA * (NBirA *, qui contient le domaine catalytique, et CBirA * qui peut être considéré comme du domaine de réactivation) peut montons dans une enzyme active lorsque présentée à proximité de deux protéines9en interaction. Le test de split-BioID qui en résulte concentre la biotinylation de proximité dépendant sur des protéines qui se réunir autour d’une paire de protéines qui interagissent et permet ainsi l’identification du contexte d’assemblys dépendants de protéine. Nous avons récemment démontré le pouvoir de résolution exceptionnelle de split-BioID en résolvant deux protéines distinctes complexes impliqués dans la médiation miRNA silençage génique voie9.
Au total, dans un essai unique et simple, split-BioID permet de découvrir et plus précisément en assignant PPI à des unités fonctionnelles définies dans laquelle une protéine donnée est impliqué, a fourni qu'une protéine additionnelle d’interaction de la protéine correspondante complexe est connue.
Protocole
Remarque : Un aperçu de la méthode est montré sur la Figure 1.
1. planification de la stratégie de clonage
- Sélectionnez deux protéines putativement interagissants à tester.
NOTE : Chacun des deux protéines va être fusionnée à un fragment de split-BioID : NBirA * ou CBirA *. Comme témoin négatif, les protéines de fusion CBirA * seront testés avec NBirA * fondue à la protéine fluorescente verte (NBirA *-GFP). Comme un contrôle supplémentaire, les fusions NBirA * peuvent être testées seul, sans objet apparenté CBirA * fusion ou en combinaison avec un CBirA * fusionné à une protéine non apparentée. Il est déconseillé d’utiliser CBirA *-GFP comme témoin négatif car il s’est avéré toujours conduire à fond majeur lorsqu’il est combiné à n’importe quel NBirA * fusion protéines9. La cause de cette observation peut être le niveau d’expression de la CBirA-GFP, beaucoup plus élevé que n’importe quel autres CBirA * protéines de fusion nous avons utilisé jusqu'à présent, pouvant conduire à la réassociation significative avec le fragment de NBirA *. -
Une fois que les deux protéines sont sélectionnés, vérifiez la littérature pour trouver si les deux ont déjà été correctement étiquetés dans les études fonctionnelles (par exemple comme une protéine de fusion de GFP-étiquetée).
- Si de telles études existent, noter la position de la balise (à l’extrémité N - ou C-terminale) et utilisez la même orientation pour baliser les protéines d’intérêt avec les fragments de split-BioID.
- Si aucune étude n’existe, des constructions de plan codant pour deux protéines tag à la N - et C-terminale et planifier un test afin de tester la fonctionnalité des protéines de fusion (par exemple, une expérience de sauvetage dans une lignée de cellules épuisée pour la protéine WT).
Remarque : Lorsque vous appariez deux protéines de fusion en utilisant des plasmides décrits à la Figure 2 qui codent pour les linkers flexibles long, l’orientation des protéines de fusion (fragments BirA * fusible en amont ou en aval des protéines d’intérêt) en général n’est pas grave . En effet en utilisant FRB et FKBP comme protéines de modèle, on a montré que tous les quatre itérations possibles (les deux morceaux à la N-termini, tous deux aux extrémités C-terminales, l’un à l’extrémité N-terminale et l’autre l’extrémité C-terminale et vice versa) rendement comparables biotinylation9.
- Conception des amorces pour amplifier l’ORF de deux protéines d’intérêt pour le clonage dans les plasmides split-BioID. Faire attention que les cadres de la traduction sont les mêmes que les fragments de BirA *. Si vous utilisez les plasmides décrits à la Figure 2, utilisez les enzymes de restriction PmeI et PacI pour construire la fusion CBirA * et les enzymes ClaI et MluI pour la fusion de NBirA *.
Remarque : Les plasmides illustrées au tableau 2 comportent un élément de tet-réactif de bi-directionelle flanqué de sites de recombinaison F3/FRT. Cela permet la co-expression réglementée à peu près le même niveau de ces deux protéines de fusion depuis le même plasmide10et la possibilité de construire rapidement des lignées cellulaires stables par échange de recombinaison-mediated cassette (RMCE). Dans ces plasmides, NBirA * a une balise de myc tandis que CBirA * a une étiquette de drapeau. L’ORF peut bien sûr également être cloné sur des plasmides différents avec des promoteurs constitutifs.
Étape critique : Lorsque vous testez deux protéines qui interagissent, il est recommandé de comparer fondue à protéine 1 combinée avec fondue à protéine 2 CBirA * NBirA * et NBirA * fondue à protéine 2 combiné avec CBirA * fondue à protéine 1. En effet, on constate fréquemment qu’une itération fonctionne mieux que l’autre.
2. clonage l’ORF des gènes d’intérêt dans le plasmide de Split-BioID
Remarque : Dans cet exemple, deux protéines qui peuvent être marqués à l’extrémité N-terminale sont considérés. Quatre conditions seront testées et comparées aux cellules non transfectées (tableau 1).
- Réaction en chaîne par polymérase (PCR) permet d’amplifier l’ORF de deux protéines à tester avec les amorces appropriées. Concevoir des amorces qui introduisent les ClaI et MluI sites de restriction pour les protéines de fusion de NBirA * et les sites PacI et PmeI pour les protéines de fusion CBirA *.
Remarque : La PCR peut être effectuée avec un commercial, préférentiellement relecture, ADN polymérase. Suivez le protocole standard du manuel et l’adapter à la température de fusion des amorces et la longueur de l’ORF à amplifier selon les directives du fabricant. -
Subclone le premier FRO
- Digérer les plasmides (environ 2 μg) et ORF1 amplifié par PCR (pour être fusionnée à NBirA *) avec ClaI et MluI. Effectuer des réactions de digestion à l’aide de 1 μL de chaque enzyme, mélangé avec le volume approprié de l’ADN et la réaction du tampon dans un volume total de 20 μL dans un tube de 1,5 mL pour 1 h à 37 ° C.
- Exécutez les deux échantillons de digestion sur un gel d’agarose-TAE d’ADN de 1 %. Les bandes correspondant à plasmide digéré et ORF1 avec un bistouri propre et le transfert aux tubes de 1,5 mL de l’accise.
- Purifier les deux bandes à l’aide d’un kit standard d’extraction d’ADN.
- Ligaturer le plasmide digéré et ORF1 en utilisant des réactifs standards.
- Si en utilisant le kit de ligature indiquées dans la Table des matières, ligaturer 100 ng d’ADN contenant des excès molaire de trois à cinq fois insérer sur le plasmide dans un volume total de 4,5 μL. Ajouter 5 μl de tampon de ligase x T4 2 et 0,5 μL de T4 DNA ligase du kit de ligature. Effectuer la réaction de ligature dans un tube de 1,5 mL pour 10 min à température ambiante (RT).
- Transformer en cellules compétentes standard DH-5α e. coli (préparés selon méthode11 de Inoue).
- Mix 3 μL de la réaction de ligature avec 50 μl de cellules compétentes dans un 1,5 mL tube sur la glace et incuber pendant 30 min. transfert des cellules à une chaleur bloquent ensemble à 42 ° C pendant 30 à 45 s et incuber puis retour sur la glace pour 2 min. ajouter 250 μL de pré chauffé (37 ° C) lysogénie bouillon (LB) support et plaque 100 μL des cellules sur une plaque de LB-gélose contenant de l’ampicilline préchauffé (37 ° C). Incuber les cellules durant la nuit à 37 ° C.
- Le lendemain, prélever des colonies de quatre à six et les incuber à 37 ° C, 180 tr/min, du jour au lendemain dans 3 mL de milieu LB contenant 100 μg⋅mL-1 ampicilline dans un tube de 15 mL.
- Isoler les plasmides des colonies prélevées à l’aide d’un kit de DNA MiniPrep standard.
- Vérifier l’exactitude des plasmides par Sanger sequencing utilisant l’amorce inverse Cassette 2 (tableau 2).
- Une fois un plasmide contenant le premier ORF a été identifié, subclone le deuxième ORF dans ce plasmide suivant les mêmes étapes comme étape 2.2, mais en utilisant les enzymes PmeI et PacI.
- Séquencer les plasmides qui en résulte, à l’aide de l’apprêt inverse 1 Cassette (tableau 2).
3. essai du système les protéines de Fusion
Remarque : Les instructions suivantes concernent les double expression inductible plasmides (Figure 2) et HeLa-11ht cellules, une lignée de cellules HeLa-CCL2 subclonal, exprimant de manière stable la transcription inverse contrôlée par tétracycline activateur rtTA-M2 et contenant un locus de RMCE12. Le milieu de croissance de ces cellules est modifié Eagle (DMEM de Dulbecco) contenant 10 % sérum exempt de tétracycline fœtal (SVF). Lorsque vous utilisez un autre type de cellule, conditions exactes de semis et de milieu de croissance devra être adapté.
-
Transfection transitoire
- Les cellules à une concentration de 1 x 105 graines la veille de la transfection des cellules dans 2 mL par puits d’une plaque de six puits et incuber les cellules pendant la nuit à 37 ° C, 5 % de CO2 dans un incubateur de culture cellulaire.
- Le jour de la transfection, retirez le support de chaque puits et remplacez par 2 mL de milieu frais.
- Préparer les quatre réactions de transfection selon le tableau 1. Pour chaque puits à transfecter, ajoutez 6 µg de polyéthylènimine à 3 µg d’ADN plasmidique dans un tube stérile de 1,5 mL et le remplir à 500 µL avec milieu DMEM sans sérum.
- Incuber chaque mélange de transfection pendant au moins 5 min à température ambiante avant d’ajouter goutte sage à chaque puits. Incuber les cellules pendant la nuit à 37 ° C, 5 % de CO2.
-
Induction et proximité-étiquetage
- Le jour après la transfection, retirez le support et remplacez par additionné de biotine à 50 µM pour stimuler la biotinylation et doxycycline à 200 ng⋅mL−1 pour induire l’expression des protéines de fusion. Incuber les cellules pendant au moins 20 h à 37 ° C, 5 % de CO2.
- Pour apporter une solution mère de biotine, dissoudre biotine à 50 mg⋅mL-1 (correspond à ca. 200 mM) à 2 M d’hydroxyde d’ammonium. Une fois qu’elle est complètement dissoute, diluer à 50 mM à 500 mM Hepes, pH 7,4, puis ajuster le pH à 7,4 avec HCl. aliquote et stocker le résultat 1 000 x solution stock à-20 ° C. Dissoudre la doxycycline à 10 mg⋅mL-1 dans l’éthanol à 70 % et l’entreposer dans un microtube de bouchon à vis à-20 ° C dans l’obscurité.
- Le jour après la transfection, retirez le support et remplacez par additionné de biotine à 50 µM pour stimuler la biotinylation et doxycycline à 200 ng⋅mL−1 pour induire l’expression des protéines de fusion. Incuber les cellules pendant au moins 20 h à 37 ° C, 5 % de CO2.
-
Préparation de lysat cellulaire
- Laver les cellules une fois avec une solution saline de 1 mL de froid (4 ° C) tamponnée au phosphate (PBS).
- Dans chaque puits, ajouter 100 µL de tampon de lyse (50 mM Tris pH 7,4, 150 mM NaCl, 2 mM EDTA, 0,5 % NP-40, 0. 5 mM DTT et inhibiteurs de la protéase).
- Récolter les cellules avec un scrapper de cellule et le transfert aux tubes de 1,5 mL.
- Centrifuger les échantillons à 14 000 x g pendant 10 min à 4 ° C pour éliminer les débris cellulaires.
- Transférer les surnageants à tubes fraîches et placer sur la glace.
- Déterminer les quantités de protéines avec une analyse de Bradford.
-
L’électrophorèse sur gel SDS-polyacrylamide (PAGE) et Western Blot
- Préparer un gel SDS-polyacrylamide.
- Pour chacune effacées lysat, préparer un échantillon de PAGE de 30 µg (minimum 15 µg) protéine dans un total de 30 µL de tampon de chargement du SDS. Puis chargez une quantité égale de chaque échantillon (20 µL/puits) sur le gel de polyacrylamide SDS et aller de l’avant à une électrophorèse.
- Après électrophorèse, transférer les protéines fractionnées sur une membrane de tache PVDF faible fluorescence à l’aide de n’importe quel protocole standard.
Remarque : Pour l’analyse des protéines biotinylation, un temps de transfert de 10 min avec le programme « haute MW » d’un rapide transfert semi-sec Western blotting périphérique habituellement fonctionne bien. - Après le transfert, bloquer la membrane dans le lait sec 5 % dans du PBS pendant 30 min à température ambiante.
- Incuber la membrane durant 30 à 60 min à ta avec fluorescent streptavidine conjuguée 1:15,000 dilué dans du PBS contenant 2 % de BSA et 0,1 % Tween-20.
- Laver la membrane trois fois, chacune pendant 10 min, avec du PBS contenant 0,1 % de Tween-20 et puis une fois plus avec du PBS.
- Scan de la membrane sur un système d’imagerie scanner de fluorescence.
La tache occidentale typique NOTE : une est indiquée sur la Figure 3. La procédure ci-dessus décrit une détection par fluorescence, mais une détection basée sur luminescence à l’aide de streptavidine couplé HRP fonctionne tout aussi bien.
4. Split-BioID pour études protéomique
NOTE critique : Pour l’analyse par spectrométrie de masse finale, toutes les étapes suivantes doivent être exécutées dans des conditions sans kératine, tout le matériel et réactifs doivent être que la kératine-libérer que possible.
- Transfection transitoire
- La veille de la transfection, semences de trois à quatre cm 10 plaques pour chaque condition à une concentration de 8 x 10 cellules de5 à 10 mL par plaque et incuber les cellules pendant la nuit à 37 ° C, 5 % de CO2 dans un incubateur de culture cellulaire.
- Le lendemain, préparer un mélange de transfection maître pour chaque condition de transfection : pour trois plaques par condition, 36 µg de polyéthylènimine et 18 µg d’ADN plasmidique dissous dans 900 µL DMEM sans sérum. Incuber à chaque mélange principal pendant au moins 5 min à température ambiante.
- En attendant, remplacer le milieu de chaque plat avec un milieu frais et puis ajouter 300 µL de la goutte de mélanges de transfection sage de chaque plaque.
- Incuber les cellules pendant la nuit à 37 ° C, 5 % de CO2.
- Induction et proximité-étiquetage
- Le jour après la transfection, les cellules de transfert aux plats de 15 cm. Pour chaque plat, retirez le support, laver les cellules avec 7 mL de PBS, ajouter 1,5 mL d’une solution de trypsine-EDTA et incuber pendant 5 min à RT. Ajouter 3,5 mL de milieu de culture pour remettre en suspension les cellules et transférer la suspension cellulaire dans un plat de 15 cm rempli de 20 mL de milieu de croissance additionné de biotine à 50 µM pour stimuler la biotinylation et doxycycline à 200 ng⋅mL−1 (concentration finale) pour induire l’expression des protéines de fusion.
- Incuber les cellules pendant au moins 20 h à 37 ° C, 5 % de CO2.
- Récolte et conservation des cellules
- Laver les cellules deux fois avec du PBS, puis ajouter 1,5 mL de PBS à chaque plaque et récolter les cellules avec un racloir.
- Transférer les cellules récoltées correspondant à un État et un tube de 15 mL et leur capture par centrifugation à 1 200 x g, 5 min, 4 ° C.
- Retirez les surnageants et snap geler les boulettes dans l’azote liquide, puis conserver à 80 ° C jusqu'à ce que la poursuite de la procédure.
Remarque : Vous pouvez également cellules peuvent également détachés par trypsinisation, récoltés dans le milieu, transférées dans un tube de 15 mL et laver trois fois avec du PBS avant la congélation.
- Préparation des lysats cellulaires
- Les granules cellulaires dans 1 mL de tampon de lyse (50 mM Tris pH 7,4, 500 mM NaCl, 0,4 % SDS, 5 mM EDTA, 1 millimètre DTT, 1 x inhibiteur de protéase complet) au col de la RT. de Resuspendre les cellules de 10 à 20 fois (cinq à dix coups) à travers une aiguille 25 G.
- Laisser agir les échantillons avec un dispositif de sonication.
Remarque : Le dispositif de la sonification indiqué dans la Table des matières, le programme suivant peut être utilisé : quatre cycles à haute intensité, 30 s par cycle dans un froid (4 ° C) bain-marie. Tout autre dispositif sonification convient mais paramètres devrez peut-être être ajustées en conséquence. - Ajouter X-100 Triton pour le récupéré sonication lysat d’atteindre une concentration finale de 2 % (en général, ajouter 100 μL de 20 % X-100 Triton à 900 μL sonication lysat cellulaire) et ensuite 2,3 mL 50 mm Tris, pH = 7,4 pour 1 mL de lysat pour ajuster la concentration de NaCl 150 mm avant b constat de perles streptavidine couplé.
NOTE critique : Des concentrations plus élevées de sels entraînent souvent beaucoup moins économe en liaison aux talons. - Distribuer les lysats ajustés en tubes de 1,5 mL (ca. 1,1 mL dans trois tubes) et les centrifuger à 16 000 x g, 10 min, 4 ° C.
- Transférer les surnageants (ca. 3,2 mL) à un tube de 15 mL et gardez 50 – 100 μL comme matériau d’apport.
- Mesurer la concentration de chaque échantillon avec une analyse de Bradford et utiliser l’équivalent de la teneur en protéines 3 à 3,5 mg pour le menu déroulant de streptavidine.
- Streptavidine pulldown
Remarque : Une vue d’ensemble de la procédure de conversion est montré sur la Figure 4. Il est presque identique au protocole original publié par Roux et ses collègues2. La première fois que le menu déroulant est effectué, échantillons d’écoulement à travers et lavage 1 à 4 peuvent être mis de côté pour analyse par transfert Western assurer la procédure a fonctionné correctement (Figure 5).- Pour chaque condition, transférer 200 μl de suspension de billes magnétiques streptavidine couplé à un tube de 1,5 mL. Placer les tubes sur un support magnétique, attendez que les perles s’en tenir au côté des tubes (ca. 1 min) et enlever le tampon de stockage.
- Laver les billes en mélangeant doucement avec 1 mL de tampon d’équilibration (50 mM Tris pH 7 – 4, NaCl 150 mM, 0,05 % Triton X-100 et 1 millimètre DTT).
- Dispatcher les perles équilibrés en quantité égale du nombre nécessaire de tubes (habituellement lors du démarrage avec 3 – 3,5 mg de protéine contenu, que les lysats de chaque condition peuvent être expédiés en deux à quatre tubes de 1,5 mL) et de la place sur la grille magnétique.
- Retirer le tampon de l’équilibration et remettre en suspension de chaque ensemble de perles avec une quantité égale des correspondante lysats de cellules de l’étape 4.4.7. Incuber une nuit à 4 ° C sur une roue en rotation.
- Le lendemain, placer les tubes de 1,5 mL sur un support magnétique, attendez que les perles s’en tenir au côté des tubes et transférer les surnageants dans un tube de 15 mL étiqueté comme couler à travers.
Remarque : à l’avenir, toutes les mesures sont effectuées à température ambiante sauf indication contraire. - Remettre en suspension les perles dans chaque tube avec 200 μL de tampon de lavage 1 (SDS 2 % dans l’eau) et combiner chaque jeu de billes remises en suspension, correspondant à un État en tubes de 1,5 mL.
- Laver les perles deux fois pendant 8 min sur une roue de rotation avec 1 mL de tampon de lavage 1.
- Laver les perles deux fois pendant 8 min sur une roue de rotation avec 1 mL de tampon de lavage 2 (50 millimètres HEPES pH 7,4, 1 mM EDTA, 500 mM NaCl, 1 % Triton X-100 et 0,1 % Na-désoxycholate).
- Laver les perles deux fois pendant 8 min sur une roue de rotation avec 1 mL de tampon de lavage 3 (10 mM Tris pH 8, 250 mM LiCl, 1 mM EDTA, 0,5 % NP-40 et 0,5 % Na-désoxycholate).
- Laver les perles deux fois pendant 8 min sur une roue de rotation avec 1 mL de tampon de lavage 4 (50 mM Tris pH 7,4, 50 mM NaCl, 0,1 % NP-40).
- Pour s’assurer que le tampon de lavage est complètement enlevé après la dernière étape de lavage, enlever la majeure partie du liquide surnageant et puis tournez en bas les échantillons. Mettez-les sur la grille magnétique, attendez que les perles s’en tenir au côté des tubes et puis retirez le tampon restant.
- Ajouter 30 μL de tampon d’élution (10 mM Tris pH 7,4, SDS 2 % et 5 % de β-mercaptoéthanol 2 mM biotine) aux talons. Incuber à 98 ° C pendant 15 min, puis retirer immédiatement les perles sur un support magnétique.
- Transférer l’échantillon éluée dans un nouveau tube et conserver à-20 ° C jusqu'à ce que la poursuite de la procédure.
- SDS-PAGE et Western Blot
NOTE : Avant l’analyse de spectrométrie de masse, il est recommandé d’évaluer le succès de la biotinylation et menu déroulant par SDS-PAGE et Western blot. Si aucun schéma biotinylation n’est observée, on peut considérer que dox ou biotine n’était pas ajoutée au milieu ou que l’une des deux solutions stocks est compromise.- Pour chaque échantillon d’entrée, préparer un échantillon de PAGE en mélangeant des quantités égales d’échantillon de protéine avec le volume suffisant de tampon de SDS-charge x 3 sur un total de 28 μL. Préparer les échantillons de PAGE en mélangeant 5 µL de chaque échantillon d’élution avec 2,5 µL de tampon de SDS-charge x 3.
- Charger les échantillons (25 µL/puits pour les entrées, 7 µL/puits d’échantillons d’élution) sur un gel SDS-polyacrylamide. Passez à l’électrophorèse et Western blotting tel que décrit à la section 3.4.
- Si vous effectuez le menu déroulant pour la première fois, préparer les PAGE échantillons pour chaque étape de l’avancement du film (entrée, traversent, élution 1 – 4, lavage) en mélangeant 20 µL de chaque échantillon (entrée, traversent, lavage 1 – 4) avec 10 µL de 3 x SDS-chargement tampon ou 5 µL (élution) avec 2,5 µL de 3 x S DS-chargement de la mémoire tampon et aller de l’avant comme à l’étape 4.6.4 (Figure 5).
- SDS-PAGE pour analyse par SM
Remarque : Afin de minimiser la contamination potentielle de la kératine, gels préfabriqués et tampon de charge échantillon commercial peuvent servir.- Ajouter 6,25 μL de tampon de 4 x à 18,75 μL de chaque échantillon d’élution et exécuter les exemples sur un 4 – 20 % préfabriqué SDS-gel jusqu'à ce qu’ils migrent le gel de 2 à 3 cm.
- Détachant le gel colloïdal Coomassie G250 de bleu brillant coloration13 dans un plat de Pétri de 15 cm.
- Utiliser un scalpel propre pour exciser les voies entières pour chaque échantillon, à l’exclusion de la bande de streptavidine (en cours d’exécution à env. 17 kDa, Figure 6) et transférer les bandes excisées pour tubes de 1,5 mL.
- Envoyer ces échantillons à un centre de protéomique pour une analyse ultérieure.
NOTE : Résultats MS typique peuvent être vu en référence 9 (Figure 3 et Figure 7et tables supplémentaires). Les ensembles de données entières sont disponibles sur le référentiel de fierté (numéro PXD005005).
Résultats
Pour illustrer le fonctionne de cette méthode, les cadres ouverts de lecture (ORF) des protéines Ago2, TNRC6C et Dicer (tous impliqués dans les gènes miRNA silencieux voie) ont été clonées dans split-BioID plasmides. Ago2 est connu pour interagir avec TNRC6C dans un induite par miRNA silencieux complexe (miRISC) qui réprime la traduction et de stimuler la dégradation d’ARNm cible14. Avant d’assembler le miRISC, Ago2 interagir avec Dicer, l’enzyme qui produit des miARN matures, au sein d’un complexe dans lequel il peut obtenir chargé avec un miRNA15. C’est pourquoi split-BioID a été appliqué à la paire Ago2/Dicer ou la paire Ago2/TNRC6C. Pour chaque paire de protéines testées, Ago2 est soit fondue à NBirA * ou CBirA * à l’aide de notre split-BioID plasmides (Figure 2) et Dicer et TNRC6C à la correspondante apparenté BirA * fragment. En outre, chaque protéine a été fusionnée à CBirA * et jumelé avec un NBirA *-fusion de GFP comme témoin négatif. Cela se traduit dans l’essai quatre itérations pour chaque paire de protéines testées (tableau 1).
Pour tester si split-BioID est activé sur l’interaction de la paire de protéines testées, nous avons suivi le schéma représenté sur la Figure 1. Les plasmides ont été transfectées transitoirement en ligne compatible aux cellules HeLa tet-système. L’expression des protéines de fusion a été induite avec la doxycycline (dox) et biotinylation est stimulée par l’ajout de biotine excès dans le milieu de croissance. Après un temps d’incubation de 20 h avec dox et biotine, les cellules ont été lysées et analysés par Western Blot utilisant conjuguée streptavidine pour détecter les protéines biotinylées. Dans les cellules de mammifères, deux bandes principales sont généralement détectés par la Streptavidine conjuguée dans l’échantillon celllulaire (Figure 3étoiles) et correspondent aux protéines de façon endogène biotinylé (très probablement mitochondriales carboxylases). Ces deux bandes sont présents dans tous les échantillons et peuvent être utilisées comme témoins de charge interne, par conséquent, la détection d’une protéine de ménage pour contrôler le chargement des quantités égales de protéines est superflue. Typique pour une expérience BioID/split-BioID, grandes bandes supplémentaires qui peuvent être observées sont les protéines de fusion qui s’est auto-biotinylé. Même si aucune autre protéine biotinylé n’est considérée, détection biotinylation des protéines fusion à ce stade déjà indique que les deux protéines testées ont interagi dans les cellules. Dans l’expérience représentée sur la Figure 3, il est clair que d’avoir un NBirA *-protéine de fusion Ago2 jumelé avec CBirA * fusions TNRC6C ou Dicer est plus efficace que les combinaisons opposés dans laquelle CBirA *-Ago2 est couplé aux NBirA * fusions des deux autres protéines (Figure 3, panneau supérieur, comparer les intensités des voies 2 et 3 voies 6 et 7). En outre, l’activation est spécifique comme aucun d'entre les fusions CBirA * pourrait activer le NBirA *-protéine de fusion GFP contrôle à un niveau appréciable (Figure 3, comparez les voies 1, 4-5 pour lane 8 qui correspond aux cellules celllulaire). Comme dans nos plasmides, NBirA * a une balise de myc et CBirA * a une étiquette de drapeau (Figure 2), les niveaux d’expression de chaque protéine de fusion peuvent être analysées avec les anticorps dirigés contre ces deux balises (Figure 3, panneau inférieur).
Lorsque induite par l’interaction biotinylation est observée, l’expérience peut évoluer vers le haut, et les protéines biotinylées isolement sur des billes de streptavidine couplé comme indiqué au paragraphe 4 du protocole (Figure 4). Lorsque vous effectuez l’isolement, la première fois, toutes les étapes de la purification peuvent être analysés par Western Blot (Figure 5). En général, liaison aux talons devrait être presque quantitatif et pratiquement sans fuite par devrait être observé dans les lavages. Avant traitement des échantillons pour la spectrométrie de masse, nous vous recommandons d’exécuter une tache occidentale afin d’assurer la biotinylation induit a fonctionné comme prévu et que les protéines de fusion ont été exprimées. L’absence d’expression des protéines de fusion est soit due à l’efficacité de transfection pauvres ou induction dox défectueux. Si les protéines de fusion ont été exprimées, mais aucun biotinylation n’est observée, vérifiez si excès biotine (50 μM) était en fait ajouté au milieu et que la biotine stock est toujours active. Lorsque le matériau éluée est analysé sur un gel teinté bleu de Coomassie protéine (Figure 6), en général, la bande plus forte à observer fonctionne à environ 17 kDa et correspond au monomère streptavidine. Bandes correspondant aux protéines biotinylées endogènes et les protéines de fusion peuvent également être observées. Nous avons généralement exciser la zone de la voie de l’échantillon au-dessus de la bande de streptavidine jusqu’au puits de chargement (Figure 6). La bande excisée peut être stockée dans un tube de 1,5 mL et envoyée à une installation de spectrométrie de masse. Sinon, protéines liés peuvent également être digéré la trypsine sur les perles de streptavidine couplé et les peptides digérées éluées forment la colonne. Nous utilisons systématiquement le MaxQuant logiciel16 (en utilisant principalement les paramètres par défaut et l’ajout de lysine biotinylation comme une éventuelle modification poteau-de translation, consultez la référence 9 pour plus de détails et pour obtenir les résultats MS typiques) pour analyser les données brutes de MS et la Persée suite17 pour l’analyse statistique ultérieure, les deux sont des logiciels libres. Les échantillons sont généralement exécutés dans trois réplicats biologiques. À l’aide de quantification exempte d’étiquette, spécifiquement enrichi de protéines peuvent être identifiées sur les conditions de contrôle. Pour filtrer de manière endogène des protéines biotinylées et pour les protéines qui sont étiquetés non spécifique de l’enzyme BirA *, nous considérons que les protéines qui sont considérablement enrichis au cours de visites de six ensembles de données générés avec six protéines non reliées. En outre, nous considérons que les hits qui sont enrichissent sur un split-BioID dataset dans lequel les protéines de fusion NBirA * ont été remplacés par NBirA *-GFP. Autres stratégies d’analyse de données ont été proposées, notamment à l’aide d’isotope stable étiquetant avec des acides aminés dans la culture de cellules (SILAC) pour la protéomique quantitative18. En outre, les différentes stratégies ont été décrites pour l’isolement direct des peptides biotinylés en utilisant une variante de streptavidine avec affinité affaiblie à la biotine18, conditions spéciales élution utilisant des solvants organiques19 ou biotine spécifiques anticorps20,21. Alors qu’il mène pas nécessairement à la découverte de plus de protéines, l’identification des sites biotinylation ajouter plus de confiance quant à la spécificité des hits et utile quand aborde la topologie d’une interaction.
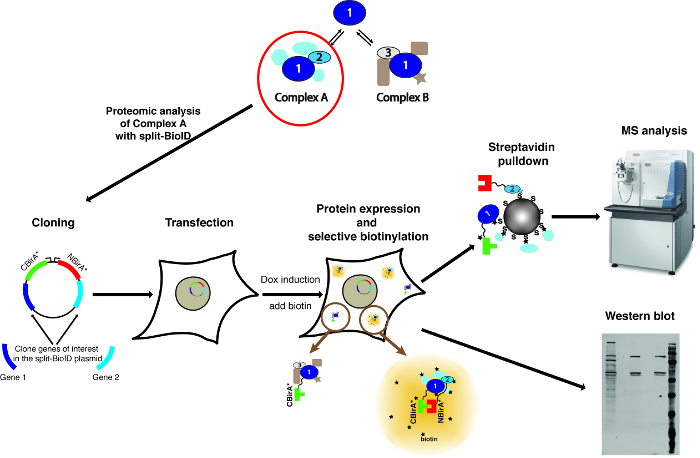
Figure 1 : vue d’ensemble de la procédure de scission-BioID. Protéine 1 interagit avec la protéine 2 dans le cadre d’un Complex ou protéine 3 dans le cadre du complexe B. Pour spécifiquement sonder la composition du complexe A, split-BioID peut être appliquée aux protéines 1 et 2. La photo du spectromètre de masse est sous une licence Creative Commons Attribution-Share Alike 3.0 Unported et a été téléchargée à partir https://commons.wikimedia.org avec le nom du fichier de ThermoScientificOrbitrapElite.JPG. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
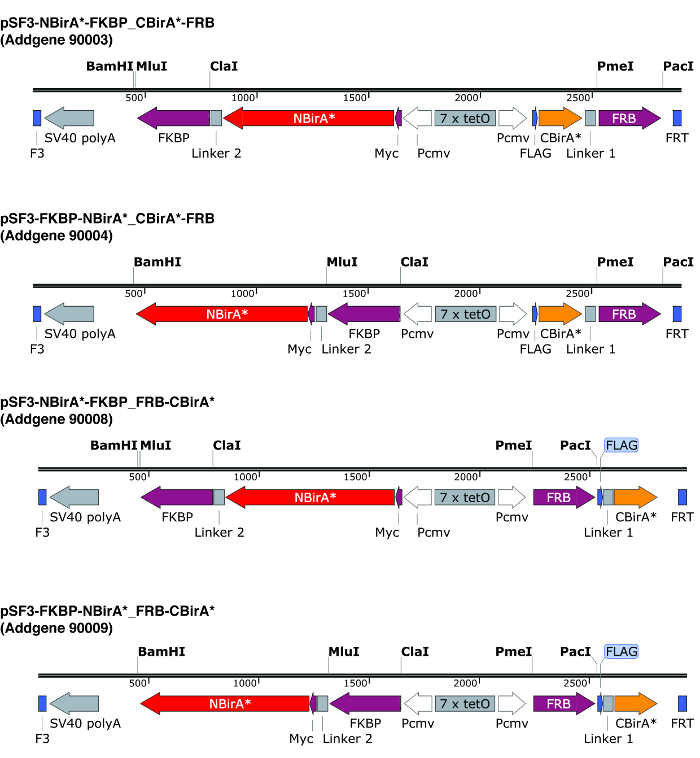
Figure 2 : cassettes d’Expression des plasmides split-BioID. Nous offrons quatre plasmides pour permettre de tester toutes les combinaisons de NBirA * et CBirA * protéines de fusion. Les plasmides et les cartes complètes sont disponibles à addgene.org sous les numéros indiqués. Les plasmides possèdent un élément favorisant le tet (7 x tetO) et doivent être utilisés dans une lignée cellulaire qui est compatible avec le système d’expression de tet. Notez également que dans tous les plasmides l’ORF de FKBP et FRB sont fondues à la NBirA * et CBirA * des fragments respectivement. Ces deux protéines n'interagissent qu’en présence de la rapamycine et donc les plasmides peuvent être utilisés pour tester rapidement le système en présence ou en absence de ce chimique9. Les sites de restriction indiquée sont uniques. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
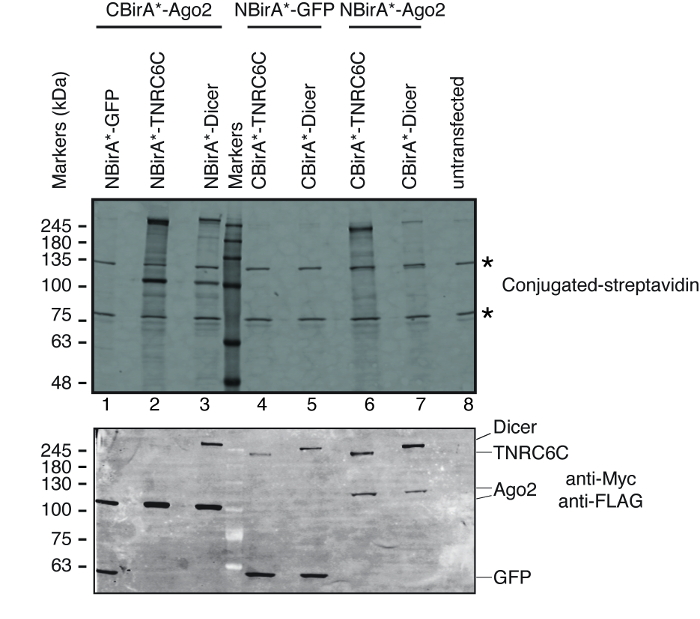
Figure 3 : type Western blot pour une expérience de split-BioID. Panneau supérieur : détection de protéines biotinylées avec streptavidine fluorescent étiquetée. Panneau inférieur : détection des protéines de fusion avec les anticorps anti-Myc et Anti-Flag. Deux paires de protéines ont été testés : Ago2/TNRC6C et Ago2/Dicer. Dans les couloirs, 2 & 3, Ago2 était annexée au fragment CBirA *. Dans les couloirs 6 & 7, Ago2 était annexée au fragment NBirA *. Aucun signal significatif a été observé lorsque une des trois protéines étaient combinée avec NBirA *-GFP (pistes 1, 4-5). Les étoiles indiquent les bandes correspondant à endogène des protéines biotinylées pouvant servir de témoins de charge interne. Ce chiffre est une adaptation de la Figure 5 b de Schopp et al. 9 sous une licence Creative Commons Attribution 4.0 International. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
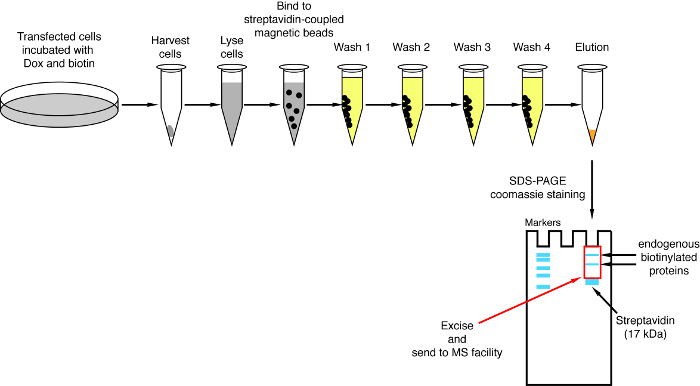
Figure 4 : vue d’ensemble de la procédure de conversion de streptavidine. Des mesures importantes pour l’isolement des protéines biotinylées pour analyse par spectrométrie de masse sont représentés. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
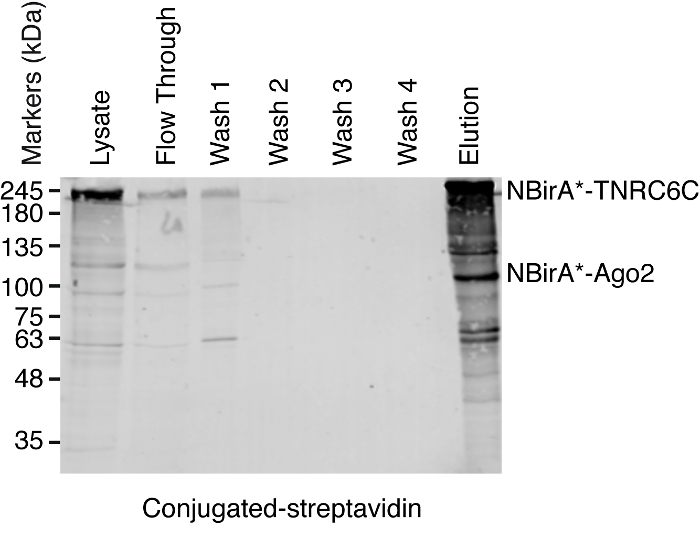
Figure 5 : typique Western blot pour une expérience de pulldown streptavidine. Volumes de chaque échantillon indiqué égaux ont été chargés sur un gel SDS-polyacrylamide. Suite Éponger occidental, protéines biotinylées ont été détectés avec la Streptavidine couplé HRP. Les bandes correspondant à NBirA *-TNRC6C et CBirA *-Ago2 sont indiqués. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
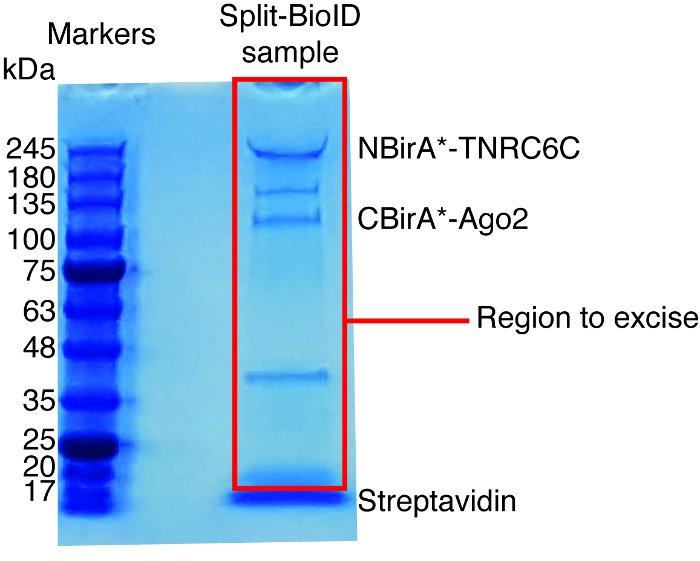
Figure 6 : gel de protéine teinté bleu de Coomassie typique pour analyse par spectrométrie de masse. L’échantillon éluée de perles streptavidine couplé a été chargé sur un gel de protéine préfabriqué et exécuté jusqu'à ce que l’échantillon migrent 2-3 cm. La principale bande observée à environ 17 kDa est streptavidine. La zone directement au-dessus de cette bande est excisée et envoyée à une installation de spectrométrie de masse. Les bandes correspondant à NBirA *-TNRC6C et CBirA *-Ago2 sont indiqués. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
| Échantillon de transfection | Conditions testées | ||
| 1 | NBirA *-protein1 / CBirA *-protein2 | ||
| 2 | CBirA *-protein1 / NBirA *-protein2 | ||
| 3 | NBirA *-GFP / CBirA *-protein1 | ||
| 4 | NBirA *-GFP / CBirA *-protein2 | ||
| 5 | aucun transfection | ||
Tableau 1 : Testé en général des conditions lors de l’application split-BioID à deux protéines.
| Apprêt de séquençage | séquence |
| Amorce de marche arrière cassette 1 (CBirA * fusion) | TATACTTTCTAGAGAATAGGAAC |
| Apprêt d’inverse (NBirA * fusion) de la cassette 2 | GTGGTTTGTCCAAACTCATC |
Tableau 2 : Séquençage des amorces pour les plasmides split-BioID.
Discussion
La procédure décrite décrit comment copier des gènes d’intérêt dans le split-BioID plasmides, comment tester biotinylation induite par l’interaction et la façon d’isoler les protéines biotinylées pour analyse par spectrométrie de masse. Nous décrivons une procédure basée sur la transfection transitoire. Alors que l’expression des protéines de fusion peut être réglée par la quantité de dox ajouté au milieu, transfection transitoire peut conduire à l’expression de la protéine non homogène avec certaines cellules qui surexpriment grossièrement les protéines de fusion par rapport à l’endogène homologues. Cela peut conduire à des distorsions de l’interactomes correspondante et à PPI qui ne reflètent pas fidèlement les interactions impliquant les protéines endogènes. Ainsi, il est généralement conseillé de construire des lignées cellulaires stables une fois split-BioID a été établie avec le système transitoire. Les plasmides sont compatibles avec le système de recombinaison induite par le Flp et placer les deux gènes d’intérêt en vertu du règlement d’un même élément tet-sensible. Si nécessaire et lorsqu’il est utilisé avec des cellules de mammifères compatibles, ils permettent la création facile de lignées cellulaires inductible stable. Par exemple, nous utilisons la ligne de HeLa-EM2-11 qui exprime l’activateur de transcription activés par tétracycline rtTA et un locus génomique targetable unique, d'où l’expression génétique induite par la tétracycline peut être étroitement réglementé12. À l’aide de cette lignée cellulaire et la recombinaison induite par le Flp, lignées cellulaires stables qui ne contiennent qu’une seule copie du transgène peuvent être obtenues dans les deux à trois semaines. On pourrait également utiliser, techniques d’édition actuel du génome d’introduire les fragments BirA * dans les natifs locus génomiques des gènes d’intérêt.
Comme dans toute analyse qui s’appuie sur le marquage d’une protéine, il faut examiner si les protéines de fusion qui en résultent sont fonctionnels. Les données disponibles dans lequel les protéines d’intérêt ont été marqués (par exemple avec GFP pour les études d’imagerie) et fonctionnellement testés sont utiles pour déterminer si les fragments BirA * doivent être cloné en amont ou en aval des gènes d’intérêt. Si aucune donnée n’est disponible, on doit tester en position N-terminale ou le tag terminalement protéines dans un test fonctionnel. Par exemple, l’activité des protéines de fusion peut être testée dans une lignée cellulaire dans lequel la protéine endogène a été frappé-out et par rapport à la situation de type sauvage. Si les protéines d’intérêt tolèrent les deux balises N - et C-terminale, tous deux doivent être testés. En effet, dans les expériences de BioID, l’orientation de la protéine de fusion peut influencer l’efficacité de l’étiquetage22. En outre, nous avons observé que lorsque application split-BioID à une paire de protéines, qui des deux protéines est accolé à l’autre NBirA * ou CBirA * fragment aussi influence l’efficacité du marquage9. Dans les plasmides split-BioID, la 16 acides aminés longs glycine/sérine linkers riches de couplage des protéines d’intérêt aux fragments BirA * ont été pris d’un autre PCA23 et a travaillé pour nous pour toutes les protéines qui interagissent nous avons testé jusqu'à présent. Cependant, on devrait considérer que quelques paires de protéines pourraient mieux fonctionner avec les linkers plus ou moins longues. De la note finale, un autre test a été décrit par le groupe de Bollen24. Dans cet essai, BirA * est répartie sur un autre site (E140/Q141) que le nôtre (E256/G257). Nous avons testé les deux split-BioID saveurs-by-side et trouvé que E256/G257, décrites dans le présent protocole, conduit à la réactivation plus forte lorsqu’elle est associée à deux protéines qui interagissent9.
Un inconvénient général de cette méthode est la lenteur de l’étiquetage. En général, temps d’incubation de 6 à 24 h avec de la biotine est nécessaire pour obtenir biotinylation appréciable6, excluant l’utilisation de cette technique pour étudier la dynamique de remodelage de complexes protéiques. Bien que cet essai aborde partiellement cette mise en garde car il n’est activé que lorsque deux protéines interagissent, la lenteur de l’étiquetage empêche son utilisation pour étudier la réponse à des processus très dynamiques ou à analyser les protéines éphémères. La peroxydase machinée APEX2 est connu pour promouvoir un étiquetage efficace des protéines proximales dans 1 min3. Un APC basé sur APEX2 pourrait ainsi pallier les lacunes de la faible vitesse d’étiquetage de dosages BioID dérivés. Une étude de démonstration des principes décrits tel un split-APEX2 dosage25. Cependant, bien qu’une protéine de homodimerizing a été correctement biotinylé, si le dosage peut également servir d’étiqueter et d’identifier les protéines qui se réunir autour d’une paire de protéines qui interagissent reste à démontrer. Très récemment, évolution dirigée a été utilisée pour créer TurboID et miniTurbo, deux variantes de la BirA avec augmentation de l’activité qui permettent aux fenêtres de temps étiquetage beaucoup plus courts, jusqu'à 10 min26. Adaptation de split-BioID à ces nouvelles variantes s’étendra davantage l’utilisation de cette technique à un plus large champ d’applications.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Ce travail a été financé par le Conseil de la recherche allemande (DFG) par le biais de l’initiative d’excellence allemande (CellNetworks DFG-EXC 81) et un financement partiel par le centre de recherche en collaboration SFB638.
matériels
| Name | Company | Catalog Number | Comments |
| Acetic acid (glacial) | VWR | 20104.298 | To make TAE buffer |
| Agarose | Sigma | A9539 | Take TAE-agarose gels for DNA analysis and extraction |
| Ammonia solution 25% NH3 | Bernd Kraft | 6012 | To dissolve biotin |
| Ampicillin | Sigma | A9518 | To select transformed bacteria |
| Bioruptor plus sonification device | Diagenode | B01020001 | Other sonification devices are also ok |
| Biotin | Sigma | B4639 | To be added to the growth medium to stimulate efficient biotinylation |
| Bovine serum albumin fraction V | Carl Roth | 8076 | Used in Western blot buffers and a protein standard in Bradford assays |
| Bradford Ultra reagent | Expedeon | BFU05L | Any other method/kit for protein determination is fine, this particular reagent is more tolerant to detergent than other Bradford reagents |
| Cell scrappers | TPP | 99002 | Any other model is also fine |
| ClaI | New England Biolabs | R0197 | Restriction enzyme for cloning into the split-BioID plasmids (NBirA* fusion) |
| DMEM medium | Sigma | D6046 | If using another cell line, use the corresponding optimal growth medium |
| DNA miniprep kit | Sigma | PLN350 | Any other kit is also fine |
| Doxycycline | Applichem | A2951 | Dox is light sensitive |
| DTT | Applichem | A2948 | Make 1M stock solution, store at -20 °C and always use fresh |
| DyLight 680-conjugated streptavidin | Thermo scientific | 21848 | to use with a LiCor Western blot scanning device |
| Dynabeads MyOne Streptavidin C1 | Invitrogen | 65002 | The C1 beads are not BSA coated which is preferable for downstream MS applications (no leakthrough of BSA in the final elution) |
| EDTA | Applichem | A5097 | Make a 500 mM stock, adjust pH to 8 while dissolving the EDTA powder |
| Ethanol | Sigma | 32205 | Make a 70% stock solution in which Doxycycline can be dissolved at 10 mg.mL-1 |
| Fastgene Gel/PCR DNA Extraction Kit | Nippon Genetics | FG-91302 | Any other kit is also fine |
| HCl 37% | Merck | 1.00317.1000 | To adjust pH of biotin stock solution |
| HEPES | Carl Roth | 6763 | Make a 500 mM stock solution, adjust the pH to 7.4 |
| Immobilon-FL PVDF membrane, 0.45 µm | Millipore | IPFL00010 | This membrane shows minimal autofluorescence when used with a LiCor Western blot scanning device |
| LiCl | Grüssing GmbH | 12083 | Make a 5M stock solution |
| Odyssey CLx imaging system | LI-COR | N/A | To scan Western blot membrane decorated with fluorophore-labeled antibody |
| Linear polyethylenimine (PEI) | Polysciences | 23966-2 | Any other transfection reagent is also fine |
| Milk powder | Carl Roth | T145 | To block Western blot membranes |
| MluI-HF | New England Biolabs | R3198 | Restriction enzyme for cloning into the split-BioID plasmids (NBirA* fusion) |
| Na-deoxycholate | Sigma | 30970 | Make a 10% (w/v) stock solution |
| NaCl | Sigma | 31434 | Make a 5M stock solution |
| NP-40 (Nonidet P40 substitute) | Sigma | 74385 | Make a 20% (v/v) stock solution |
| PacI | New England Biolabs | R0547 | Restriction enzyme for cloning into the split-BioID plasmids (CBirA* fusion) |
| Phosphate buffer saline (PBS) | Sigma | 806552 | To wash cells before scrapping |
| PmeI | New England Biolabs | R0560 | Restriction enzyme for cloning into the split-BioID plasmids (CBirA* fusion) |
| Protease inhibitor cocktail | Roche | 4693132001 | Added to the lysis buffer to prevent protein degradation |
| pSF3-Flag-CBir-FRB_Myc-NBir-FKBP | Addgene | 90003 | Split-BioID plasmid, mediates the co-expression of NBirA*-FKBP and CBirA*-FRB, FKBP and FRB can be replaced by two other ORFs |
| pSF3-Flag-CBir-FRB_FKBP-Myc-Nbir | Addgene | 90004 | Split-BioID plasmid, mediates the co-expression of FKBP-NBirA* and CBirA*-FRB, FKBP and FRB can be replaced by two other ORFs |
| pSF3-Flag-FRB-Cbir_Myc-NBir-FKBP | Addgene | 90008 | Split-BioID plasmid, mediates the co-expression of NBirA*-FKBP and FRB-CBirA*, FKBP and FRB can be replaced by two other ORFs |
| pSF3-Flag-FRB-Cbir_FKBP-Myc-Nbir | Addgene | 90009 | Split-BioID plasmid, mediates the co-expression of FKBP-NBirA* and FRB-CBirA*, FKBP and FRB can be replaced by two other ORFs |
| Q5 High-Fidelity PCR kit | New England Biolabs | E0555S | To amplify the ORF coding for the proteins to be tested. Any other thermostable DNA polymerase is fine. |
| Quick ligation kit | New England Biolabs | M2200S | To ligate DNA fragments into the split-BioID plasmids, any other DNA ligation system is fine. |
| RunBlue 4-20% SDS precast gels | Expedeon | BCG42012 | To use when running samples for MS analysis |
| RunBlue LDS Sample Buffer | Expedeon | NXB31010 | Running buffer for the RunBlue precast gels |
| SDS | Sigma | 5030 | Comes as a 20% stock solution |
| tet-free serum | Biowest | S181T | we use tet-free serum to minimize basal expression of the fusion proteins |
| Trans-Blot Turbo Transfer system | Bio-Rad | 1704150 | High speed Western blotting transfer system, any other transfer system is also fine |
| Tris | Carl Roth | 4855 | Make 1M stock solutions with adequate pH (7.4 and 8) |
| Triton X-100 | Applichem | A4975 | Make a 20% (v/v) stock solution |
| Tween-20 | Carl Roth | 9127 | Used in Western blot buffers, Tween 20 leads to high background fluorescence and should be omitted in the blocking and last wash step |
Références
- Scott, D. E., Bayly, A. R., Abell, C., Skidmore, J. Small molecules, big targets: drug discovery faces the protein-protein interaction challenge. Nat Rev Drug Discov. 15 (8), 533-550 (2016).
- Roux, K. J., Kim, D. I., Raida, M., Burke, B. A promiscuous biotin ligase fusion protein identifies proximal and interacting proteins in mammalian cells. J Cell Biol. 196 (6), 801-810 (2012).
- Lam, S. S., et al. Directed evolution of APEX2 for electron microscopy and proximity labeling. Nat Methods. 12 (1), 51-54 (2015).
- Rhee, H. W., et al. Proteomic mapping of mitochondria in living cells via spatially restricted enzymatic tagging. Science. 339 (6125), 1328-1331 (2013).
- Kim, D. I., et al. Probing nuclear pore complex architecture with proximity-dependent biotinylation. P Natl Acad Sci USA. 111 (24), E2453-E2461 (2014).
- Kim, D. I., et al. An improved smaller biotin ligase for BioID proximity labeling. Mol Biol Cell. 27 (8), 1188-1196 (2016).
- Lambert, J. P., Tucholska, M., Go, C., Knight, J. D., Gingras, A. C. Proximity biotinylation and affinity purification are complementary approaches for the interactome mapping of chromatin-associated protein complexes. J Proteomics. 118, 81-94 (2015).
- Morriswood, B., et al. Novel bilobe components in Trypanosoma brucei identified using proximity-dependent biotinylation. Eukaryot Cell. 12 (2), 356-367 (2013).
- Schopp, I. M., et al. Split-BioID a conditional proteomics approach to monitor the composition of spatiotemporally defined protein complexes. Nat Commun. 8, (2017).
- Béthune, J., Artus-Revel, C. G., Filipowicz, W. Kinetic analysis reveals successive steps leading to miRNA-mediated silencing in mammalian cells. EMBO Rep. 13 (8), 716-723 (2012).
- Inoue, H., Nojima, H., Okayama, H. High efficiency transformation of Escherichia coli with plasmids. Gene. 96 (1), 23-28 (1990).
- Weidenfeld, I., et al. Inducible expression of coding and inhibitory RNAs from retargetable genomic loci. Nucleic Acids Res. 37 (7), e50 (2009).
- Dyballa, N., Metzger, S. Fast and sensitive colloidal coomassie G-250 staining for proteins in polyacrylamide gels. J Vis Exp. (30), (2009).
- Jonas, S., Izaurralde, E. Towards a molecular understanding of microRNA-mediated gene silencing. Nat Rev Genet. 16 (7), 421-433 (2015).
- MacRae, I. J., Ma, E., Zhou, M., Robinson, C. V., Doudna, J. A. In vitro reconstitution of the human RISC-loading complex. P Natl Acad Sci USA. 105 (2), 512-517 (2008).
- Cox, J., Mann, M. MaxQuant enables high peptide identification rates, individualized p.p.b.-range mass accuracies and proteome-wide protein quantification. Nat Biotechnol. 26 (12), 1367-1372 (2008).
- Tyanova, S., et al. The Perseus computational platform for comprehensive analysis of (prote)omics data. Nat Methods. 13 (9), 731-740 (2016).
- Opitz, N., et al. Capturing the Asc1p/Receptor for Activated C Kinase 1 (RACK1) Microenvironment at the Head Region of the 40S Ribosome with Quantitative BioID in Yeast. Mol Cell Proteomics. 16 (12), 2199-2218 (2017).
- Mackmull, M. T., et al. Landscape of nuclear transport receptor cargo specificity. Mol Syst Biol. 13 (12), 962 (2017).
- Kim, D. I., et al. BioSITe: A Method for Direct Detection and Quantitation of Site-Specific Biotinylation. J Proteome Res. , (2017).
- Udeshi, N. D., et al. Antibodies to biotin enable large-scale detection of biotinylation sites on proteins. Nat Methods. 14 (12), 1167-1170 (2017).
- Chapat, C., et al. Cap-binding protein 4EHP effects translation silencing by microRNAs. P Natl Acad Sci USA. 114 (21), 5425-5430 (2017).
- Luker, K. E., et al. Kinetics of regulated protein-protein interactions revealed with firefly luciferase complementation imaging in cells and living animals. P Natl Acad Sci USA. 101 (33), 12288-12293 (2004).
- De Munter, S., et al. Split-BioID: a proximity biotinylation assay for dimerization-dependent protein interactions. FEBS Lett. 591 (2), 415-424 (2017).
- Xue, M., et al. Optimizing the fragment complementation of APEX2 for detection of specific protein-protein interactions in live cells. Sci Rep. 7 (1), 12039 (2017).
- Branon, T. C., et al. Directed evolution of TurboID for efficient proximity labeling in living cells and organisms. bioRxiv. , (2017).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon