Method Article
Split-BioID — Proteomic análise de complexos de proteínas específicas do contexto em seu ambiente nativo do celular
Neste Artigo
Resumo
Nós fornecemos um protocolo passo a passo para dividir-BioID, um ensaio de fragmentos-complementação de proteínas com base na técnica de criação de etiquetas de proximidade BioID. Ativado na interação das duas proteínas determinadas, permite a análise proteômica de contexto-dependente da proteína em seu ambiente nativo do celular. O método é simples e econômica e só requer equipamentos de laboratório padrão.
Resumo
Para complementar a purificação da afinidade existente (AP) abordagens para a identificação das interações da proteína-proteína (PPI), foram introduzidas as enzimas que permitem que a rotulagem de proximidade-dependente de proteínas nas células vivas. Uma tal enzima, BirA * (usado na abordagem BioID), Medeia a biotinylation de proteínas dentro de um intervalo de aproximadamente 10 nm. Daí, quando fundiu-se a uma proteína de interesse e expressos nas células, permite que a rotulagem das proteínas proximais em seu ambiente nativo. Ao contrário do AP que depende da purificação de complexos de proteínas montado, BioID detecta proteínas que foram marcadas dentro das células, não importa se eles ainda estão interagindo com a proteína de interesse, quando estão isolados. Uma vez que biotinylates proteínas proximal, um Além disso pode capitalizar a afinidade excepcional de streptavidin para biotina para muito eficientemente isolá-los. Enquanto BioID executa melhor AP para identificação transiente ou interações fracas, ambas as abordagens de espectrometria de massa de AP e BioID fornecem uma visão geral de todas as possíveis interacções que pode ter uma determinada proteína. No entanto, eles não fornecem informações sobre o contexto de cada PPI identificado. Na verdade, a maioria das proteínas são tipicamente parte de vários complexos, correspondendo a etapas de maturação distintas ou diferentes unidades funcionais. Para resolver essa limitação comum de ambos os métodos, temos engenharia um ensaio de complementação de fragmentos da proteína baseado a enzima BirA *. Neste ensaio, dois fragmentos inativos de BirA * podem montar em uma enzima ativa quando trouxe nas proximidades por duas proteínas interagindo ao qual eles são fundidos. O ensaio de separação-BioID resultante permite, assim, a rotulagem de proteínas que reunir em torno de um par de interação de proteínas. Desde que estes dois só interagem em um determinado contexto, split-BioID, em seguida, permite a análise das unidades funcionais específicas do contexto-dependente no seu ambiente nativo do celular. Aqui, nós fornecemos um protocolo passo a passo para testar e aplicar BioID-divisão de um par de interação de proteínas.
Introdução
Como funções mais celulares são executadas por proteínas que montam dinamicamente complexos macromoleculares, a identificação das interações da proteína-proteína (PPI) é um grande esforço na investigação biomédica. Com efeito, o PPI são frequentemente desregulamentado em doença e representam potenciais alvos para terapêutica1. O mais amplamente utilizado método para a identificação de PPI é a abordagem de purificação (AP) de afinidade em que, na sequência de lise celular, especificamente, uma proteína de interesse é purificada em uma matriz e proteínas associadas são posteriormente identificadas por espectrometria de massa (MS). Enquanto AP-MS é uma abordagem poderosa, ele normalmente não executa bem em complexos da proteína pouco solúvel, interações muito transitórias ou PPI que exigem uma estrutura subcelular intacta. Além disso, a interpretação dos dados pode ser complicada pela natureza dinâmica das redes PPI, como uma única proteína geralmente é parte de vários complexos de proteínas distintas.
Técnicas de criação de etiquetas de proximidade como BioID2 ou APEX23,4 recentemente foram desenvolvidas para resolver algumas das limitações das abordagens AP-MS. Em BioID, a enzima BirA * (correspondente a uma variante do G115R da enzima tipo selvagem Escherichia coli ) catalisa a formação de lábil biotinyl AMP (bio-AMP) que pode reagir com aminas primárias. Em oposição a enzima do tipo selvagem, que mantém em seu centro ativo da bio-AMP, BirA * libera bio-AMP, permitindo a sua difusão ao seu ambiente vizinho. Daí, quando fundiu-se a uma proteína de interesse e expressos nas células, proteínas proximais podem ser biotinilado dentro de um intervalo estimado de 10 nm5. Estas marcadas proximais proteínas são então isoladas por streptavidin pulldown e identificadas pelo MS. Ao contrário de AP-MS, BioID requer a expressão de uma proteína de fusão. É, portanto, só pode ser aplicado às proteínas cuja função não é dificultada por marcação. Além disso, a velocidade de rotulagem é lento, normalmente 6 – 24 h2,6, tornando a detecção de proteínas de curta duração desafiador. Mas, comparado com AP-MS, BioID-MS oferece várias vantagens: primeiro, sua captura as interações em seu ambiente nativo e celular; segunda, etiquetadas proteínas, ao invés de complexos montados são isolados após a lise celular; em terceiro lugar, pulldowns streptavidin permitem usar buffers de desnaturação e lavagem duras condições. Portanto, o método é mais sensível para detectar transiente ou interações fracas7 ou interações que ocorrem em um específico e difícil de isolar subcellular estrutura8.
No entanto, a maioria das proteínas são geralmente parte de complexos maiores que pode remodelar de acordo com sugestões de celulares ou para a função que precisa ser executada. Portanto, uma única proteína é normalmente parte de vários complexos, correspondente às unidades funcionais distintas, envolvendo distintos e/ou sobrepostas PPI. Ambas as abordagens dar uma visão geral de todas as associações que pode ter uma determinada proteína, mas deixam de abordar o contexto de PPI individual. Para aumentar a resolução do último, nós projetamos um ensaio de complementação de proteína-fragmentos (PCA) em que dois fragmentos inativos de BirA * (NBirA *, que contém o domínio catalítico, e CBirA * que pode ser visto como o domínio de reativação) pode remontagem em uma enzima ativa quando trouxe nas proximidades por dois interagindo proteínas9. O ensaio de separação-BioID resultante enfoca o dependente da proximidade biotinylation proteínas que reunir em torno de um par de interação de proteínas e, portanto, permite a identificação do contexto de módulos (assemblies) dependentes da proteína. Nós recentemente demonstrou o poder de resolução excelente de split-BioID resolvendo dois complexos de proteínas distintas envolvidas na mediada por miRNA-silenciamento via9.
No total, em um único e simples ensaio, split-BioID permite descobrir e atribuir especificamente PPI para unidades funcionais definidas com uma determinada proteína é envolvido, desde que uma proteína de interação adicional da proteína correspondente complexa é conhecida.
Protocolo
Nota: Uma visão geral do método é mostrada na Figura 1.
1. planejamento da estratégia de clonagem
- Selecione duas proteínas presumidamente interagindo para ser testado.
Nota: Cada uma das duas proteínas vai ser fundida a um fragmento de split-BioID: NBirA * ou CBirA *. Como um controle negativo, as proteínas de fusão CBirA * serão testadas com NBirA * fundidos com a proteína verde fluorescente (NBirA *-GFP). Como um controle adicional, as NBirA * fusões podem ser testadas em paz, sem cognato CBirA * fusão ou em combinação com um CBirA * fundido a uma proteína independente. É aconselhável não usar CBirA *-GFP como controlo negativo como foi mostrado consistentemente conduzir a grande fundo quando combinado a qualquer NBirA * fusão proteína9. A causa desta observação pode ser o nível de expressão de CBirA *-GFP, muito maior do que qualquer outras CBirA * proteínas da fusão temos usado até agora, isso podem levar a reassociação significativa com o fragmento de NBirA. -
Uma vez que duas proteínas são selecionadas, verificar a literatura para encontrar-se os dois já foram com sucesso identificados em estudos funcionais (por exemplo, como uma proteína de fusão de GFP-etiquetado).
- Se tais estudos, observe a posição da marca (em N - ou C-terminal) e usar a mesma orientação para as proteínas de interesse com os fragmentos de split-BioID.
- Se tal estudo não existir, plano construções codificação para ambas as proteínas marcados no N - e C-terminal e planejar um ensaio para testar a funcionalidade das proteínas de fusão (por exemplo, um experimento de resgate em uma linha de celular esgotado para a proteína WT).
Nota: Quando emparelhar duas proteínas de fusão usando o plasmídeo descrito na Figura 2 que codificam linkers longo e flexíveis, a orientação das proteínas de fusão (BirA * fragmentos fundidos o montante ou a jusante das proteínas de interesse) geralmente não importa . Na verdade usando FRB e FKBP como proteínas do modelo, foi mostrado que todos os quatro iterações possíveis (os dois fragmentos no N-termini, ambos no C-termini, um no N-terminal e o outro do C-terminal e vice-versa) rendimento comparável biotinylation9.
- Desenho de primers para amplificar as ORFs das duas proteínas de interesse para clonagem em plasmídeos o split-BioID. Preste atenção que os quadros de tradução são os mesmos que os fragmentos de BirA. Se usando o plasmídeo descrito na Figura 2, use as enzimas de restrição PmeI e PacI para construir a fusão CBirA * e as enzimas ClaI e MluI para a NBirA * fusão.
Nota: Os plasmideos representados na Figura 2 carregam um elemento tet-responsivo de bi-direcional, ladeado por sites de recombinação F3/FRT. Isso permite que a expressão regulamentada co mais ou menos o mesmo nível de ambas as proteínas da fusão do mesmo plasmídeo10e a possibilidade de construir rapidamente linhas celulares estável pelo exchange mediada por recombinação gaveta (RMCE). Nestes plasmídeos, NBirA * tem uma marca de myc enquanto CBirA * tem uma marca de bandeira. As ORFs claro também podem ser clonados em plasmídeos individuais com promotores constitutivos.
Etapa crítica: Ao testar duas proteínas de interação, recomenda-se comparar fundido a proteína 1 combinada com CBirA * fundido a proteína 2 NBirA * e NBirA * fundido a proteína 2 combinada com CBirA * fundido a proteína 1. Com efeito, frequentemente observa-se que uma iteração funciona melhor do que o outro.
2. clonagem do ORFs dos Genes de interesse para o plasmídeo Split-BioID
Nota: Neste exemplo, são consideradas duas proteínas que podem ser marcadas no N-terminal. Quatro condições vão ser testadas e comparadas às células não transfectadas (tabela 1).
- Use a reação em cadeia da polimerase (PCR) para amplificar as ORFs das duas proteínas a serem testadas com os primers adequados. Projetar primers que introduzem os ClaI e MluI locais de restrição para as proteínas de fusão NBirA * e os sites PacI e PmeI para as proteínas de fusão CBirA *.
Nota: O PCR pode ser realizada com qualquer comercial, preferencialmente revisão, DNA polimerase. Siga o protocolo padrão de manual e adaptá-lo para a temperatura de fusão dos primers e o comprimento da ORF para ser amplificado de acordo com as orientações do fabricante. -
Subclone o primeiro ORF
- Digeri tanto plasmídeo (cerca de 2 μg) e PCR amplificados ORF1 (para ser fundido ao NBirA *) com ClaI e MluI. Realizar reações de digestão utilizando 1 μL de cada enzima, misturada com o volume apropriado de DNA e reação buffer em um volume total de 20 μL em um tubo de 1,5 mL para 1 h a 37 ° C.
- Execute ambas as amostras de digestão em um gel de DNA de TAE-agarose 1%. Excisar as bandas correspondentes do plasmídeo digerido e ORF1 com um bisturi limpo e transferência para tubos de 1,5 mL.
- Purifica as duas bandas usando um kit padrão de extração de DNA.
- Ligate plasmídeo digerido e ORF1 usando reagentes padrão.
- Se usando o kit de ligadura indicada na Tabela de materiais, ligate 100 ng de DNA contendo excesso molar de três a cinco tempo insira sobre plasmídeo em um volume total de 4,5 μL. Adicione 5 μL de tampão, ligase x T4 2 e 0,5 μL de T4 DNA ligase do kit de ligadura. Realize a reação de ligadura em um tubo de 1,5 mL por 10 min à temperatura ambiente (RT).
- Transforme-se em padrão de DH-5 α Escherichia coli competentes células (preparadas de acordo com método11 do Inoue).
- Mix 3 μL de reação da ligadura com 50 μL de células competentes em um 1,5 mL tubo no gelo e incube por 30 min. transferência das células para um calor bloqueiam o conjunto a 42 ° C por 30-45 s e então incubam no gelo por 2 min. Adicione 250 μL de pré-aquecido (37 ° C) lisogenia caldo (LB) médio e placa 100 μL de células numa placa de LB-ágar contendo ampicilina pré-aquecido (37 ° C). Incubar as células durante a noite a 37 ° C.
- No dia seguinte, escolhe quatro a seis colônias e os incubar a 37 ° C, 180 rpm, durante a noite em 3 mL de meio LB contendo 100 μg⋅mL-1 ampicilina em um tubo de 15 mL.
- Isole o plasmídeo das colônias escolhidos usando um kit padrão de DNA MiniPrep.
- Verificar a regularidade do plasmídeo por Sanger sequenciamento usando o primer reverso de gaveta 2 (tabela 2).
- Uma vez que foi identificado um plasmídeo contendo o primeiro ORF, subclone o segundo ORF nesse plasmídeo seguindo os mesmos passos como passo 2.2, mas usando as enzimas PmeI e PacI.
- Sequenciar os plasmideos resultantes usando o primer reverso 1 gaveta (tabela 2).
3. teste das proteínas de fusão
Nota: As instruções a seguir são para o plasmídeo de expressão inducible dual (Figura 2) e HeLa-11ht células, uma linhagem de células HeLa-CCL2 subclonal, estàvel expressando a transcrição reversa controlado tetraciclina ativador rtTA-M2 e contendo um Locus para RMCE12. O meio de crescimento para estas células é modificado águia médio (DMEM de Dulbecco) contendo 10% de soro bovino fetal livre de tetraciclina (FBS). Ao usar outro tipo de célula, condições exatas de semeadura e crescimento médio precisará ser adaptado.
-
Transfecção transiente
- Células de sementes em uma concentração de 1 x 105 em 2 mL por bem de uma placa de seis células na véspera do transfection e incubar as células durante a noite a 37 ° C, 5% de CO2 em uma incubadora de cultura de células.
- No dia do transfection, remover o meio de cada poço e substituir com 2 mL de meio fresco.
- Prepare as quatro reações de Transfeccao de acordo com a tabela 1. Para cada poço de transfect, adicione 6 µ g do polyethylenimine a 3 µ g do ADN do plasmídeo em um tubo estéril de 1,5 mL e encha a 500 µ l com meio DMEM sem soro.
- Incube cada mistura de transfeccao pelo menos 5 min à temperatura ambiente antes de Adicionar gota sábio a cada poço. Incube as células durante a noite a 37 ° C, 5% de CO2.
-
Indução e criação de etiquetas de proximidade
- O dia depois de transfeccao, remover o meio e substitua suplementado com biotina em 50 µM para estimular biotinylation e doxiciclina 200 ng⋅mL−1 para induzir a expressão das proteínas de fusão. Incube as celulas pelo menos 20 h a 37 ° C, 5% de CO2.
- Para fazer uma solução stock de biotina, dissolver biotina em 50 mg⋅mL-1 (corresponde a ca. 200 mM) em 2 M de hidróxido de amônio. Uma vez que é completamente dissolvido, diluir a 50 mM em 500 mM Hepes, pH 7,4, em seguida, ajustar o pH para 7,4 com HCl. alíquota e armazenar o resultante 1.000 x solução estoque a-20 ° C. Dissolva a doxiciclina em 10 mg⋅mL-1 em etanol a 70% e a loja em um microtubo de tampa de rosca a-20 º C no escuro.
- O dia depois de transfeccao, remover o meio e substitua suplementado com biotina em 50 µM para estimular biotinylation e doxiciclina 200 ng⋅mL−1 para induzir a expressão das proteínas de fusão. Incube as celulas pelo menos 20 h a 37 ° C, 5% de CO2.
-
Preparação de lisado celular
- Lave as células uma vez com soro 1 mL de frio (4 ° C) tamponada fosfato (PBS).
- Para cada poço, adicione 100 µ l de tampão de lise (50 mM Tris pH 7,4, 150 mM de NaCl, 2 mM EDTA, 0,5% NP-40, 0.5 mM DTT e inibidores da protease).
- Colha as células com um sucateiro de célula e transferência para tubos de 1,5 mL.
- Centrifugar as amostras a 14.000 x g durante 10 minutos a 4 ° C para remover os resíduos de célula.
- Transfira os sobrenadantes para tubos frescos e no gelo.
- Determine quantidades de proteína com um ensaio de Bradford.
-
Eletroforese em gel de SDS-poliacrilamida (PAGE) e mancha ocidental
- Prepare um gel de poliacrilamida-SDS.
- Para cada desmarcada lisado, preparar uma página de amostra de 30 µ g (mínimo 15 µ g) proteína, num total de 30 µ l de tampão de carregamento SDS. Em seguida, carregar quantidades iguais de cada amostra (20 µ l/poço) sobre o gel de poliacrilamida-SDS e proceder a eletroforese.
- Após a electroforese, transferi as proteínas fracionadas para uma membrana de borrão PVDF baixa fluorescência usando qualquer protocolo padrão.
Nota: Para a análise de proteína biotinylation, um tempo de transferência de 10 minutos com o programa "alta MW" de um rápido transferi semi seco Western mancha dispositivo geralmente funciona bem. - Após a transferência, bloquear a membrana no leite seco 5% em PBS durante 30 min à RT
- Incubar a membrana durante 30 a 60 min à RT com fluorescente estreptavidina conjugada 1:15,000 diluídos em PBS contendo 2% de BSA e 0.1% Tween-20.
- Lave a membrana três vezes, cada um por 10 min, com PBS contendo 0,1% de Tween-20 e então uma vez mais com PBS.
- Varredura da membrana em um sistema da imagem latente do varredor de fluorescência.
Borrão ocidental típico de nota: um é mostrado na Figura 3. O procedimento acima descreve uma detecção fluorescente mas uma detecção baseada em luminescência usando streptavidin HRP-acoplado funciona igualmente bem.
4. Split-BioID para estudos de proteômica
Nota crítica: Para a análise final de espectrometria de massa, todas as etapas a seguir devem ser executadas em condições de livre de queratina, todo o material e reagentes devem ser como queratina-livre quanto possível.
- Transfecção transiente
- No dia anterior a transfeccao, semente 10 cm três ou quatro pratos para cada condição em uma concentração de 8 x 105 células em 10 mL por placa e incubar as células durante a noite a 37 ° C, 5% de CO2 em uma incubadora de cultura de células.
- No dia seguinte, preparar uma mistura de transfeccao mestre para cada condição de transfeccao: para três placas por condição, 36 µ g do polyethylenimine e 18 µ g do ADN do plasmídeo dissolvido em 900 µ l soro livre DMEM. Incube cada mestre mistura durante pelo menos 5 min à RT
- Entretanto, substituir o meio de cada prato com meio fresco e em seguida, adicione 300 µ l de transfeccao misturas drop sábio para cada placa.
- Incube as células durante a noite a 37 ° C, 5% de CO2.
- Indução e criação de etiquetas de proximidade
- O dia depois de transfeccao, transferi as células para pratos de 15 cm. Para cada prato, retire o meio, lavar as células com 7 mL de PBS, adicionar 1,5 mL de uma solução de tripsina-EDTA e incubar durante 5 min à RT. adicionar 3,5 mL de meio de crescimento para Ressuspender as células e transferir a suspensão de células para um prato de 15 cm, preenchido com 20 mL do meio de crescimento suplementado com biotina em 50 µM para estimular biotinylation e doxiciclina em 200 ng⋅mL− 1 (concentrações finais) para induzir a expressão das proteínas de fusão.
- Incube as celulas pelo menos 20 h a 37 ° C, 5% de CO2.
- Colheita e armazenamento das células
- Lavar as células duas vezes com PBS, em seguida, adicionar 1,5 mL de PBS a cada prato e colher as células com uma scrapper.
- Transferir as células colhidas, correspondendo a uma condição para um tubo de 15 mL e colhê-las por centrifugação a 1.200 x g, 5 min, 4 ° C.
- Remover os sobrenadantes e snap congelar as bolinhas em nitrogênio líquido e, em seguida, loja a 80 ° C até o processamento adicional.
Nota: Como alternativa, células podem também ser desanexadas por tripsinização, colhidas em meio de crescimento, transferidas para um tubo de 15 mL e lavadas três vezes com PBS antes do congelamento.
- Preparação de lisados celulares
- Resuspenda as pelotas de células em 1 mL de tampão de lise (50 mM Tris pH 7,4, 500 mM de NaCl, 0,4% SDS, 5 mM EDTA, 1 milímetro DTT, 1 x inibidor da protease completa) em RT. Pass as células 10 – 20 vezes (cinco a dez cursos) através de uma agulha de 25 G.
- Proceda à sonicação das amostras com um dispositivo de sonorização.
Nota: Com o dispositivo de sonorização indicado na Tabela de materiais, o programa a seguir pode ser usado: quatro ciclos em alta intensidade, 30 s por ciclo em uma fria (4 ° C) banho de água. Qualquer outro dispositivo de sonorização é adequado mas parâmetros talvez precise ser ajustado em conformidade. - Adicionar X-100 Triton para o recuperado lisados lisado para atingir uma concentração final de 2% (normalmente, adicionar 100 μL de 20% Triton X-100 para 900 μL lisados lisado celular) e em seguida 2,3 mL de 50 mM Tris, pH = 7,4 por 1 mL de lisado para ajustar a concentração de NaCl 150 mm antes de b ulação de grânulos streptavidin-acoplado.
Nota crítica: Altas concentrações de sal muitas vezes resultam em muito menos eficiente vinculação aos talões. - Distribuir os lysates ajustados em tubos de 1,5 mL (ca. 1,1 mL em três tubos) e centrifugá-los a 16.000 x g, 10 min, 4 ° C.
- Transferir os sobrenadantes (ca. 3,2 mL) para um tubo de 15 mL e manter 50 – 100 μL como material de entrada.
- Medir a concentração de cada amostra com um ensaio de Bradford e usar o equivalente do teor de proteínas de 3 a 3,5 mg para o pulldown estreptavidina.
- Streptavidin pulldown
Nota: Uma visão geral do procedimento de pulldown é mostrada na Figura 4. É quase idêntico do protocolo original publicado por Roux e colegas2. A primeira vez que o pulldown é executado, amostras de fluir e lavagem 1 – 4 podem ser pôr de lado para análise ocidental do borrão garantir o procedimento funcionou corretamente (Figura 5).- Para cada condição, transferi 200 μL de suspensão de grânulos magnético streptavidin-acoplado a um tubo de 1,5 mL. Coloque os tubos em um rack magnético, aguarde até que os grânulos ficar ao lado dos tubos (ca. 1 min) e remover o buffer de armazenamento.
- Lave os grânulos misturando suavemente com 1 mL de tampão de equilíbrio (50 mM Tris pH 7 – 4, 150 mM NaCl, 0,05% Triton X-100 e 1 milímetro DTT).
- Despache os grânulos equilibrados em quantidade igual do número necessário de tubos (geralmente quando começando com 3 e 3,5 mg de proteína conteúdo, que os lysates de cada condição pode ser despachados em dois a quatro tubos de 1,5 mL) e lugar volta sobre o rack magnético.
- Retire o tampão de equilíbrio e ressuspender cada conjunto de grânulos com quantidades iguais dos correspondente lisados celulares da etapa 4.4.7. Incube durante uma noite a 4 ° C em uma roda giratória.
- No dia seguinte, coloque os tubos de 1,5 mL em um rack magnético, aguarde até que os grânulos ficar ao lado dos tubos e transfira os sobrenadantes para um tubo de 15 mL rotulado como fluir através de.
Nota: de agora em diante, todas as etapas são executadas à temperatura salvo indicação em contrário. - Resuspenda os grânulos em cada tubo com 200 μL de tampão de lavagem 1 (SDS 2% em água) e combinar cada conjunto de grânulos ressuspensão, correspondendo a uma condição em tubos de 1,5 mL.
- Lave os grânulos duas vezes por 8 min em uma roda de rotação com 1 mL de tampão de lavagem 1.
- Lave os grânulos duas vezes por 8 min em uma roda de rotação com 1 mL de tampão de lavagem 2 (pH HEPES 50mm 7.4, 1 mM EDTA, 500 mM de NaCl, 1% Triton X-100 e 0.1% Deoxycholate at-do).
- Lave os grânulos duas vezes por 8 min em uma roda de rotação com 1 mL de tampão de lavagem 3 (pH de Tris 10 mM 8, 250mm LiCl, 1 mM EDTA, 0,5% NP-40 e 0,5% Deoxycholate at-do).
- Lave os grânulos duas vezes por 8 min em uma roda de rotação com 1 mL de tampão de lavagem 4 (pH de Tris 50mm 7.4, 50mm NaCl, 0,1% NP-40).
- Para certificar-se de que o tampão de lavagem é completamente removido depois que o último passo de lavar, remover a maior parte do sobrenadante e então gire para baixo as amostras. Colocá-los de volta na prateleira magnética, aguarde até que os grânulos ficar ao lado dos tubos e em seguida, retire o restante do buffer.
- Adicionar 30 μL de tampão de eluição (pH de 10 mM Tris 7.4, SDS 2%, 5% β-Mercaptoetanol e 2mm biotina) para as contas. Incubar a 98 ° C por 15 min e, em seguida, remova imediatamente as contas em um rack magnético.
- Transferi a amostra eluted para um tubo fresco e armazenar a-20 ° C até o processamento adicional.
- SDS-PAGE e mancha ocidental
Nota: Antes da análise por espectrometria de massa, é recomendável para avaliar o sucesso do biotinylation e suspenso por SDS-PAGE e Western blot. Se nenhum teste padrão biotinylation observa-se se pode considerar que dox ou biotina não foi adicionada ao meio ou que uma das duas soluções estoque está comprometida.- Para cada amostra de entrada, prepare uma amostra de página através da mistura de quantidades iguais de amostra de proteína com o volume apropriado de tampão de carregamento SDS x 3 em um total de 28 μL. Prepare amostras de página através da mistura de 5 µ l de cada amostra de eluição com 2,5 µ l de tampão de carregamento SDS x 3.
- Carrega as amostras (25 µ l/poço para entradas, 7 µ l/poço para amostras de eluição) em um gel de poliacrilamida-SDS. Proceda à eletroforese e mancha conforme descrito na seção 3.4 ocidental.
- Se executar o pulldown pela primeira vez, preparar página amostras para cada etapa do pulldown (entrada, fluir, eluição de 1 – 4, lavagem) misturando-se 20 µ l de cada amostra (entrada, fluxo, através de lavagem 1 – 4), com 10 µ l de 3x do amortecedor do SDS-carregamento ou 5 µ l (eluição) com 2,5 µ l de 3 x S DS-carregamento do buffer e proceder como no passo 4.6.4 (Figura 5).
- SDS-PAGE para análise de MS
Nota: Para minimizar o potencial de contaminação de queratina, geles de pré-moldado e amortecedor de carregamento da amostra comercial podem ser usados.- Adicionar 6,25 μL de tampão de amostra 4 x 18,75 μL de cada amostra de eluição e executar os exemplos em um gel de SDS de 4 – 20% pré-moldado até eles migram de 2 – 3 cm no gel.
- Manche o gel com coloidal Coomassie brilhante azul G250 coloração13 em uma placa de Petri 15 cm.
- Use um bisturi limpo para excisar as pistas inteira para cada amostra, excluindo a banda streptavidin (rodando a ca. 17 kDa, Figura 6) e transferir as bandas extirpadas para tubos de 1,5 mL.
- Envie estas amostras para uma instalação de proteômica para posterior análise.
Nota: Resultados de MS típico podem ser vistos na referência 9 (Figura 3 e Figura 7e quadros suplementares). Os conjuntos de dados inteiro estão disponíveis no repositório do orgulho (número de adesão PXD005005).
Resultados
Para ilustrar como funciona esse método, os frames de leitura abertos (ORFs) das proteínas Ago2, TNRC6C e Dicer (todos os envolvidos no caminho de silenciamento do gene mediada por miRNA) foram clonados em split-BioID plasmídeos. Ago2 é conhecido por interagir com TNRC6C dentro de um induzida por miRNA silencioso complexo (miRISC) que reprime a tradução e estimular a decadência do alvo mRNAs14. Antes de montar o miRISC, Ago2 interagir com Dicer, a enzima que produz os miRNAs maduros, dentro de um complexo em que ele pode ficar carregado com miRNA15. Portanto, split-BioID foi aplicado para o par de Ago2/Dicer ou o par Ago2/TNRC6C. Para cada par de proteínas testadas, Ago2 foi também fundidos para NBirA * ou CBirA * usando nosso separação-BioID plasmídeo (Figura 2), e Dicer e TNRC6C para o BirA cognato correspondente * fragmentam. Além disso, cada proteína foi fundida ao CBirA * e emparelhada com um NBirA *-fusão de GFP como controlo negativo. Isso resulta em teste quatro iterações para cada par de proteínas testadas (tabela 1).
Para testar se o split-BioID é ativado mediante a interação do par de proteínas testadas, seguimos o esquema descrito na Figura 1. Os plasmídeos foram transfectados transitoriamente em uma linhagem de células de HeLa compatível de tet-sistema. A expressão das proteínas de fusão foi induzida com doxiciclina (dox) e biotinylation foi estimulada pela adição de biotina em excesso ao meio de crescimento. Após um tempo de incubação de 20 h com dox e biotina, as células foram lysed e analisadas pela mancha ocidental usando estreptavidina conjugada para detectar proteínas biotinilado. Em células de mamíferos, duas grandes bandas normalmente são detectadas pela estreptavidina conjugada na amostra untransfected (Figura 3, estrelas) e correspondem às proteínas de biotinilado endogenamente (mais provavelmente mitocondriais carboxylases). Estas duas bandas estão presentes em todas as amostras e podem ser convenientemente usadas como controles de carregamento interno, assim, a detecção de uma proteína de limpeza para controlar o carregamento de quantidades iguais de proteínas é supérflua. Típicos para um experimento de BioID/split-BioID, as bandas principais adicionais que podem ser observadas são as proteínas de fusão que tem autobiotinilado. Mesmo se nenhuma outra proteína biotinilado é vista, detectar biotinylation das proteínas de fusão nesta fase já indica que as duas proteínas testadas interagiam nas células. No experimento representado na Figura 3, é claro que ter um NBirA *-proteína de fusão Ago2 emparelhado com CBirA * fusões de TNRC6C ou Dicer é mais eficiente do que as combinações opostas no qual CBirA *-Ago2 está emparelhado com NBirA * fusões dos outros dois proteínas (Figura 3, painel superior, comparar as intensidades de lanes 2-3 para faixas 6-7). Além disso, a ativação foi específica como nenhum das fusões CBirA * poderia ativar o NBirA *-proteína da fusão de controle GFP para níveis apreciáveis (Figura 3, comparar faixas 1, 4-5 a lane 8 que corresponde às células untransfected). Desde que em nossos Plasmideos, NBirA * tem uma marca de myc e CBirA * tem uma marca de bandeira (Figura 2), os níveis de expressão de cada proteína de fusão podem ser analisados com anticorpos contra estas duas marcas (Figura 3, painel inferior).
Quando biotinylation induzida pela interação é observado, o experimento pode ser ampliado e as proteínas biotinilado isolaram em grânulos streptavidin-acoplados conforme indicado no n. º 4 do protocolo (Figura 4). Ao executar o isolamento pela primeira vez, todas as etapas da purificação podem ser analisadas pela mancha ocidental (Figura 5). Normalmente, vinculação aos talões deve ser quase quantitativa e praticamente nenhum vazamento através de deve ser observado nas lavagens. Prévia de processamento das amostras para espectrometria de massa, recomendamos um Western blot para garantir induzida-biotinylation funcionou como esperado e que as proteínas de fusão foram expressos em execução. Ou devido à eficiência de transfeccao pobre ou indução dox com defeito é a falta de expressão das proteínas de fusão. Se manifestaram-se a proteínas da fusão, mas não biotinylation é observado, verifique se o excesso biotina (50 μM) na verdade foi adicionada ao meio e que o estoque biotina ainda está ativa. Quando o material eluted é analisada em um gel de proteína manchadas de Coomassie (Figura 6), normalmente, a banda mais forte a ser observado é executado em cerca de 17 kDa e corresponde ao streptavidin monomérico. Bandas correspondentes as proteínas endógenas biotinilado e as proteínas de fusão também podem ser observadas. Nós normalmente excisar a área da faixa de amostra acima da faixa de estreptavidina até o carregamento bem (Figura 6). A banda extirpada pode ser armazenada em um tubo de 1,5 mL e enviada para uma instalação de espectrometria de massa. Alternativamente, ligado a proteínas podem também ser digerido tripsina sobre os grânulos streptavidin-acopladas e os peptídeos digeridos eluídos formam a coluna. Rotineiramente usamos o software de MaxQuant16 (usando principalmente parâmetros padrão e adicionando biotinylation lisina como uma possível modificação pós-traducional, consulte referência 9 para mais detalhes e a típica MS resultados) para analisar os dados brutos do MS e o Perseu suíte17 para posterior análise estatística, ambos são software livre. Amostras são normalmente executadas em três réplicas biológicas. Usando quantificação rótulo livre, especificamente enriquecidas proteínas podem ser identificadas sobre condições de controle. Para filtrar por endogenamente biotinilado proteínas e de proteínas que não específica são rotuladas pela enzima BirA *, consideramos apenas as proteínas que são significativamente enriquecidas mais correspondências a partir de seis conjuntos de dados gerados com seis proteínas independentes. Além disso, consideramos apenas hits que são enriquecidos com um conjunto de dados de divisão-BioID em que as proteínas de fusão NBirA * foram substituídas por NBirA *-GFP. Outras estratégias de análise de dados têm sido propostas, nomeadamente utilizando o isótopo estável etiquetando com aminoácidos na cultura de pilha (SILAC) para proteômica quantitativa18. Além disso, várias estratégias têm sido descritas para o isolamento direto de peptídeos biotinilado usando uma variante do streptavidin com afinidade enfraquecida a biotina18, condições de eluição especial utilizando solventes orgânicos19 ou biotina-específicos anticorpos de20,21. Enquanto não necessariamente conduz à descoberta de mais proteínas, a identificação dos sítios biotinylation adicionar mais confiança quanto a especificidade dos hits e é útil quando abordar a topologia de uma interação.
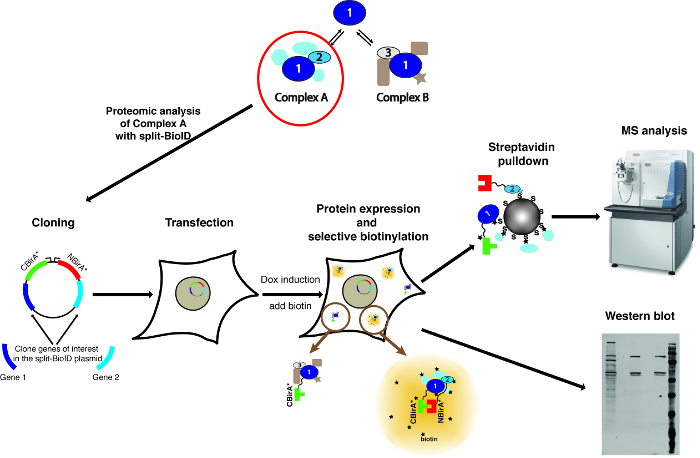
Figura 1: visão geral do processo de separação-BioID. Proteína 1 interage com a proteína 2 como parte do complexo A, ou com proteína 3 como parte do complexo B. Para sondar especificamente a composição do complexo A, divisão-BioID pode ser aplicada a proteínas 1 e 2. A fotografia do espectrômetro de massa está sob uma licença Creative Commons Attribution-Share Alike 3.0 Unported e foi baixada do https://commons.wikimedia.org com o nome do arquivo de ThermoScientificOrbitrapElite.JPG. Clique aqui para ver uma versão maior desta figura.
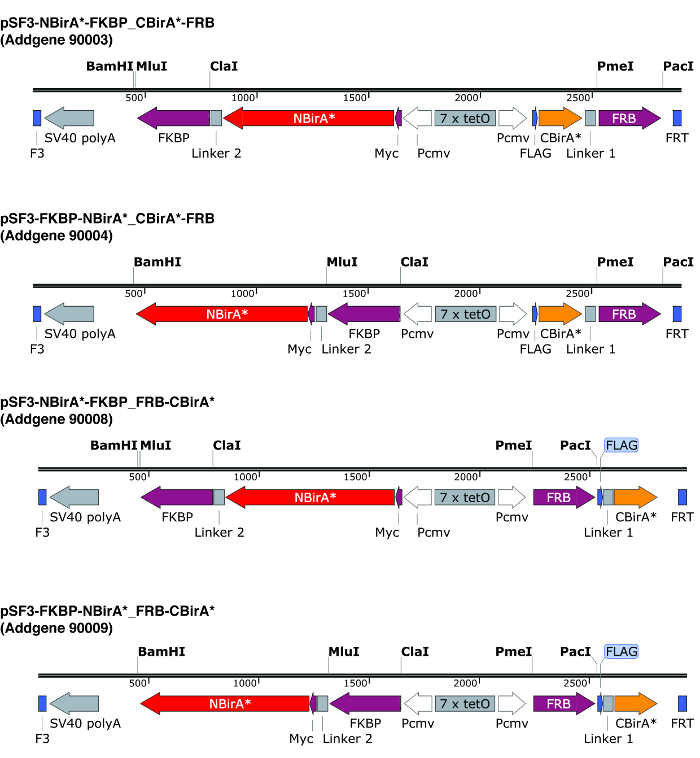
Figura 2: fitas de expressão de plasmídeos o split-BioID. Nós fornecemos quatro plasmídeos para permitir testar todas as combinações de NBirA * e CBirA * proteínas da fusão. Os plasmídeos e mapas completos estão disponíveis no addgene.org sob os números indicados. Os plasmideos têm um elemento tet-responsivos (7 x tetO) e precisam ser usado em uma linha de celular que é compatível com o sistema de expressão de tet. Observe também que em plasmídeos todas as ORFs de FKBP e FRB são fundidos para o NBirA * e CBirA * fragmentos respectivamente. Estas duas proteínas interagem somente na presença de rapamicina e daí o plasmídeo pode ser usado para testar rapidamente o sistema a presença ou ausência desta química9. Os locais de restrição indicada são únicos. Clique aqui para ver uma versão maior desta figura.
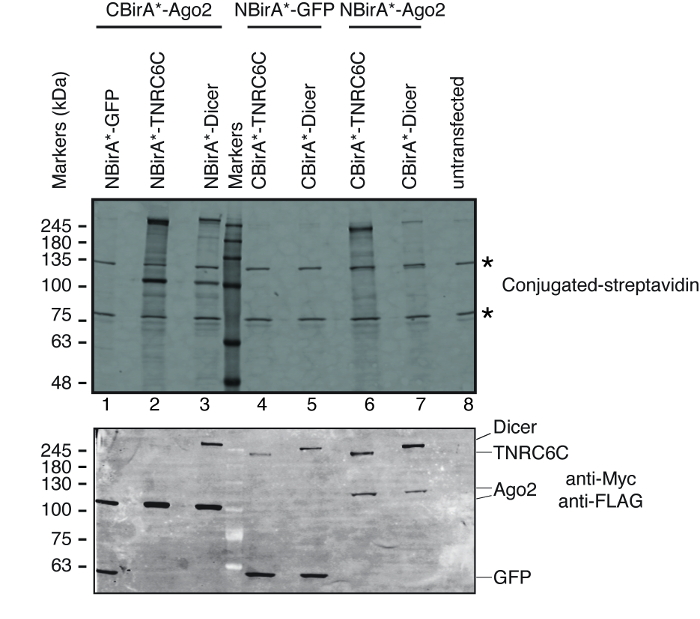
Figura 3: borrão ocidental típico para um experimento de divisão-BioID. Painel superior: deteção de proteínas biotinilado com estreptavidina fluorescente etiquetada. Painel inferior: deteção das proteínas de fusão com anticorpos anti-Myc e anti-FLAG. Dois pares de proteínas foram testados: Ago2/TNRC6C e Ago2/Dicer. Nas pistas, 2 & 3, Ago2 foi acrescentado o fragmento de CBirA. Em pistas 6 & 7, Ago2 foi acrescentado o fragmento de NBirA. Nenhum sinal significativo foi observado quando qualquer uma das três proteínas foram combinados com o NBirA-GFP (faixas 1, 4-5). As estrelas indicam as bandas correspondentes a endogenamente biotinilado proteínas que podem servir como controles de carregamento interno. Esta figura é uma adaptação de Figura 5B de Schopp et al 9 sob uma licença Creative Commons Attribution 4.0 internacional. Clique aqui para ver uma versão maior desta figura.
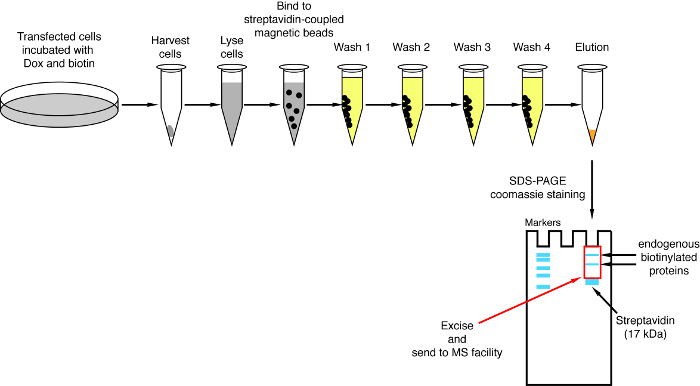
Figura 4: visão geral do processo de pulldown streptavidin. Passos importantes para o isolamento de proteínas biotinilado para análise de espectrometria de massa são retratados. Clique aqui para ver uma versão maior desta figura.
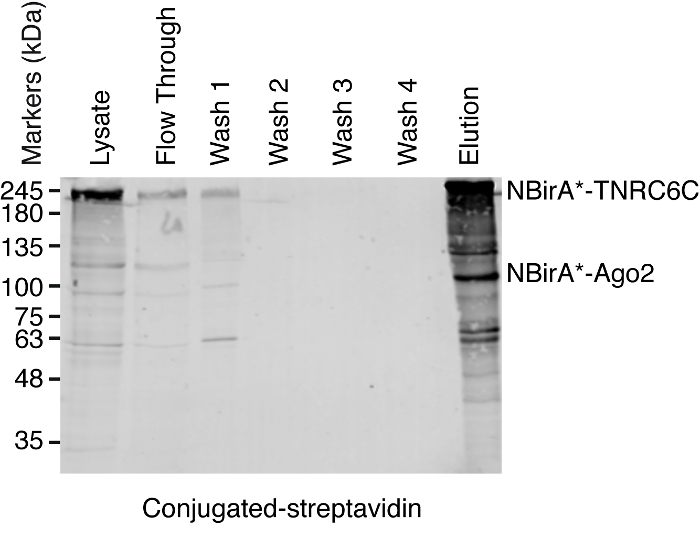
Figura 5: borrão ocidental típico para um experimento de pulldown streptavidin. Volumes de cada amostra indicada iguais foram carregados em um gel de poliacrilamida-SDS. Mancha ocidental, na sequência de proteínas biotinilado foram detectadas com streptavidin HRP-acoplado. Bandas correspondentes a NBirA *-TNRC6C e CBirA *-Ago2 são indicados. Clique aqui para ver uma versão maior desta figura.
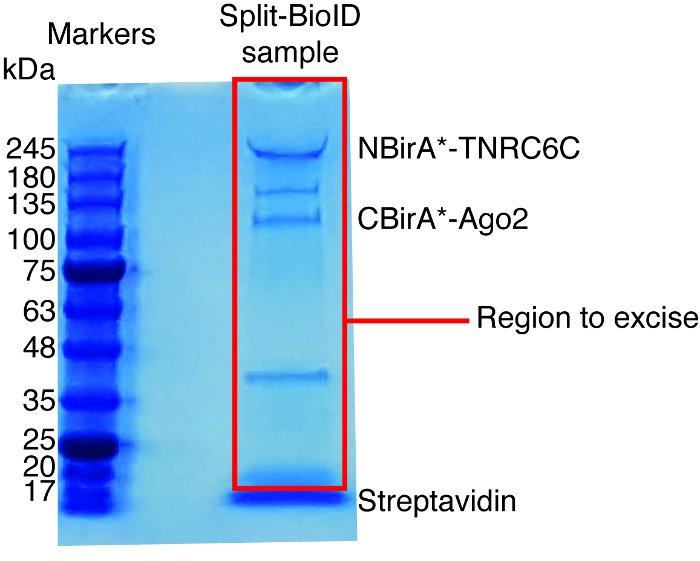
Figura 6: gel de proteína manchadas de Coomassie típico para análise de espectrometria de massa. A amostra eluted de missanga streptavidin-acoplado foi carregada em um gel de proteína pré-moldado e correr até a amostra migrar de 2-3 cm. A banda importante vista em cerca de 17 kDa é estreptavidina. A área diretamente acima essa banda é excisada e enviada para uma instalação de espectrometria de massa. Bandas correspondentes a NBirA *-TNRC6C e CBirA *-Ago2 são indicados. Clique aqui para ver uma versão maior desta figura.
| Amostra de transfeccao | Condição testada | ||
| 1 | NBirA *-protein1 / CBirA *-protein2 | ||
| 2 | CBirA *-protein1 / NBirA *-protein2 | ||
| 3 | NBirA *-GFP / CBirA *-protein1 | ||
| 4 | NBirA *-GFP / CBirA *-protein2 | ||
| 5 | Não transfecção | ||
Tabela 1: Testado normalmente condições ao aplicar o split-BioID duas proteínas.
| Cartilha de sequenciamento | sequência de |
| Cartilha de gaveta 1 reversa (CBirA * fusão) | TATACTTTCTAGAGAATAGGAAC |
| Cartilha de gaveta 2 reversa (NBirA * fusão) | GTGGTTTGTCCAAACTCATC |
Tabela 2: Sequenciamento primers para os divisão-BioID plasmídeos.
Discussão
O procedimento descrito descreve como clonar genes de interesse para a divisão-BioID plasmídeos, como testar biotinylation induzida pela interação e como isolar proteínas biotinilado para análise de espectrometria de massa. Descrevemos aqui um procedimento baseado na transfecção transiente. Enquanto a expressão das proteínas de fusão pode ser ajustada pela quantidade de dox adicionado ao meio, transfecção transiente pode levar a expressão de proteínas não homogénea, com algumas células que overexpress grosseiramente as proteínas de fusão quando comparado com o endógeno homólogos. Isto pode conduzir a distorções de interactomes o correspondente e a PPI que não refletem fielmente as interações que envolvem as proteínas endógenas. Assim, é geralmente aconselhável construir linhas celulares estável, uma vez dividida-BioID foi estabelecida com o sistema transitório. Os plasmídeos são compatíveis com o sistema de recombinação mediada por Flp e coloque ambos os genes de interesse nos termos do Regulamento do mesmo elemento responsivo-tet. Se necessário e quando usado com células de mamíferos compatíveis, eles permitem a fácil criação de linhas de células inducible estável. Por exemplo, nós usamos a linha HeLa-EM2-11 que expressa o ativador de transcrição ativado por tetraciclina rtTA e um locus genômico targetable exclusivo do qual expressão gênica mediada por tetraciclina pode ser fortemente regulamentado12. Usando esta linha celular e recombinação mediada por Flp, linhas de célula estável que contêm somente uma cópia do transgene podem ser obtidas dentro de duas a três semanas. Alternativamente, pode também usar técnicas de edição atual do genoma para introduzir os BirA * fragmentos o nativo genomic loci dos genes de interesse.
Como em qualquer ensaio que baseia-se na marcação de uma proteína, é preciso considerar se as proteínas de fusão resultante são funcionais. Dados disponíveis, em que as proteínas de interesse foram marcadas (por exemplo com as boas práticas agrícolas para estudos de imagem) e funcionalmente testados são úteis decidir se os fragmentos de BirA devem ser clonado o montante ou a jusante dos genes de interesse. Se tais dados não estão disponíveis, um deve testar N-terminal ou C-terminal tagged proteínas em um ensaio funcional. Por exemplo, a atividade das proteínas de fusão pode ser testada em uma linha de celular, em que a proteína endógena foi eliminado e em relação à situação do tipo selvagem. Se as proteínas de interesse toleram as duas etiquetas N - e C-terminal, ambos devem ser testados. De fato, em experimentos de BioID, a orientação da proteína de fusão pode influenciar a eficiência de rotulagem22. Além disso, observamos que quando aplicar o split-BioID um par de proteínas, qual das duas proteínas é acrescentado ou o NBirA * ou CBirA * fragmento também influência a eficiência de rotulagem9. Nos Divisão-BioID plasmídeos, a 16 aminoácido longa glicina/serina ricos linkers acoplamento as proteínas de interesse para os fragmentos de BirA foram tiradas de outro PCA23 e trabalhou para nós para todas as proteínas interagindo nós testamos até agora. No entanto, deve-se considerar que alguns pares de proteína podem funcionar melhor com os linkers menores ou mais. Da nota final, outro ensaio foi descrito pelo grupo Flickr24. Neste ensaio, BirA * é dividido em outro site (E140/Q141) do que o nosso (E256/G257). Nós temos testado tanto dividir-BioID sabores-lado e encontrou que o E256/G257, descrito neste protocolo, leva a mais forte reativação quando acoplado a duas proteínas interagindo9.
Uma desvantagem geral deste método é a lenta velocidade de rotulagem. Normalmente, o tempo de incubação de 6 a 24 h com biotina é necessário obter apreciável biotinylation6, impedindo a utilização desta técnica para o estudo de remodelação dinâmica dos complexos de proteína. Enquanto este ensaio aborda parcialmente esta ressalva como ele só é ativado quando duas proteínas interagem, a lenta velocidade de rotulagem impede seu uso para estudar a resposta de processos muito dinâmicos ou analisar proteínas de curta duração. A peroxidase de engenharia APEX2 é conhecido por promover eficiente rotulação de proximais proteínas dentro de 1 min3. Um PCA, com base em APEX2 assim poderia abordar as limitações da velocidade lenta rotulagem dos ensaios BioID-derivado. Um estudo prova de princípio descrito tal ensaio um split-APEX225. No entanto, apesar de uma proteína homodimerizing com êxito foi biotinilado, se o ensaio também pode ser usado para rotular e identificar as proteínas que reunir em torno de um par de interação de proteínas permanece para ser demonstrado. Muito recentemente, a evolução dirigida foi usada para criar TurboID e miniTurbo, duas variantes de BirA * com atividade reforçada que permitir que o windows muito mais curto de tempo a rotulagem, até 10 min26. Split-BioID para estas novas variantes de adaptação mais estenderá o uso desta técnica para um campo mais amplo de aplicações.
Divulgações
Os autores não têm nada para divulgar.
Agradecimentos
Este trabalho foi financiado pelo Conselho de pesquisa alemã (DFG) através da iniciativa de excelência alemão (CellNetworks DFG-EXC 81) e um financiamento parcial pelo centro de investigação em colaboração SFB638.
Materiais
| Name | Company | Catalog Number | Comments |
| Acetic acid (glacial) | VWR | 20104.298 | To make TAE buffer |
| Agarose | Sigma | A9539 | Take TAE-agarose gels for DNA analysis and extraction |
| Ammonia solution 25% NH3 | Bernd Kraft | 6012 | To dissolve biotin |
| Ampicillin | Sigma | A9518 | To select transformed bacteria |
| Bioruptor plus sonification device | Diagenode | B01020001 | Other sonification devices are also ok |
| Biotin | Sigma | B4639 | To be added to the growth medium to stimulate efficient biotinylation |
| Bovine serum albumin fraction V | Carl Roth | 8076 | Used in Western blot buffers and a protein standard in Bradford assays |
| Bradford Ultra reagent | Expedeon | BFU05L | Any other method/kit for protein determination is fine, this particular reagent is more tolerant to detergent than other Bradford reagents |
| Cell scrappers | TPP | 99002 | Any other model is also fine |
| ClaI | New England Biolabs | R0197 | Restriction enzyme for cloning into the split-BioID plasmids (NBirA* fusion) |
| DMEM medium | Sigma | D6046 | If using another cell line, use the corresponding optimal growth medium |
| DNA miniprep kit | Sigma | PLN350 | Any other kit is also fine |
| Doxycycline | Applichem | A2951 | Dox is light sensitive |
| DTT | Applichem | A2948 | Make 1M stock solution, store at -20 °C and always use fresh |
| DyLight 680-conjugated streptavidin | Thermo scientific | 21848 | to use with a LiCor Western blot scanning device |
| Dynabeads MyOne Streptavidin C1 | Invitrogen | 65002 | The C1 beads are not BSA coated which is preferable for downstream MS applications (no leakthrough of BSA in the final elution) |
| EDTA | Applichem | A5097 | Make a 500 mM stock, adjust pH to 8 while dissolving the EDTA powder |
| Ethanol | Sigma | 32205 | Make a 70% stock solution in which Doxycycline can be dissolved at 10 mg.mL-1 |
| Fastgene Gel/PCR DNA Extraction Kit | Nippon Genetics | FG-91302 | Any other kit is also fine |
| HCl 37% | Merck | 1.00317.1000 | To adjust pH of biotin stock solution |
| HEPES | Carl Roth | 6763 | Make a 500 mM stock solution, adjust the pH to 7.4 |
| Immobilon-FL PVDF membrane, 0.45 µm | Millipore | IPFL00010 | This membrane shows minimal autofluorescence when used with a LiCor Western blot scanning device |
| LiCl | Grüssing GmbH | 12083 | Make a 5M stock solution |
| Odyssey CLx imaging system | LI-COR | N/A | To scan Western blot membrane decorated with fluorophore-labeled antibody |
| Linear polyethylenimine (PEI) | Polysciences | 23966-2 | Any other transfection reagent is also fine |
| Milk powder | Carl Roth | T145 | To block Western blot membranes |
| MluI-HF | New England Biolabs | R3198 | Restriction enzyme for cloning into the split-BioID plasmids (NBirA* fusion) |
| Na-deoxycholate | Sigma | 30970 | Make a 10% (w/v) stock solution |
| NaCl | Sigma | 31434 | Make a 5M stock solution |
| NP-40 (Nonidet P40 substitute) | Sigma | 74385 | Make a 20% (v/v) stock solution |
| PacI | New England Biolabs | R0547 | Restriction enzyme for cloning into the split-BioID plasmids (CBirA* fusion) |
| Phosphate buffer saline (PBS) | Sigma | 806552 | To wash cells before scrapping |
| PmeI | New England Biolabs | R0560 | Restriction enzyme for cloning into the split-BioID plasmids (CBirA* fusion) |
| Protease inhibitor cocktail | Roche | 4693132001 | Added to the lysis buffer to prevent protein degradation |
| pSF3-Flag-CBir-FRB_Myc-NBir-FKBP | Addgene | 90003 | Split-BioID plasmid, mediates the co-expression of NBirA*-FKBP and CBirA*-FRB, FKBP and FRB can be replaced by two other ORFs |
| pSF3-Flag-CBir-FRB_FKBP-Myc-Nbir | Addgene | 90004 | Split-BioID plasmid, mediates the co-expression of FKBP-NBirA* and CBirA*-FRB, FKBP and FRB can be replaced by two other ORFs |
| pSF3-Flag-FRB-Cbir_Myc-NBir-FKBP | Addgene | 90008 | Split-BioID plasmid, mediates the co-expression of NBirA*-FKBP and FRB-CBirA*, FKBP and FRB can be replaced by two other ORFs |
| pSF3-Flag-FRB-Cbir_FKBP-Myc-Nbir | Addgene | 90009 | Split-BioID plasmid, mediates the co-expression of FKBP-NBirA* and FRB-CBirA*, FKBP and FRB can be replaced by two other ORFs |
| Q5 High-Fidelity PCR kit | New England Biolabs | E0555S | To amplify the ORF coding for the proteins to be tested. Any other thermostable DNA polymerase is fine. |
| Quick ligation kit | New England Biolabs | M2200S | To ligate DNA fragments into the split-BioID plasmids, any other DNA ligation system is fine. |
| RunBlue 4-20% SDS precast gels | Expedeon | BCG42012 | To use when running samples for MS analysis |
| RunBlue LDS Sample Buffer | Expedeon | NXB31010 | Running buffer for the RunBlue precast gels |
| SDS | Sigma | 5030 | Comes as a 20% stock solution |
| tet-free serum | Biowest | S181T | we use tet-free serum to minimize basal expression of the fusion proteins |
| Trans-Blot Turbo Transfer system | Bio-Rad | 1704150 | High speed Western blotting transfer system, any other transfer system is also fine |
| Tris | Carl Roth | 4855 | Make 1M stock solutions with adequate pH (7.4 and 8) |
| Triton X-100 | Applichem | A4975 | Make a 20% (v/v) stock solution |
| Tween-20 | Carl Roth | 9127 | Used in Western blot buffers, Tween 20 leads to high background fluorescence and should be omitted in the blocking and last wash step |
Referências
- Scott, D. E., Bayly, A. R., Abell, C., Skidmore, J. Small molecules, big targets: drug discovery faces the protein-protein interaction challenge. Nat Rev Drug Discov. 15 (8), 533-550 (2016).
- Roux, K. J., Kim, D. I., Raida, M., Burke, B. A promiscuous biotin ligase fusion protein identifies proximal and interacting proteins in mammalian cells. J Cell Biol. 196 (6), 801-810 (2012).
- Lam, S. S., et al. Directed evolution of APEX2 for electron microscopy and proximity labeling. Nat Methods. 12 (1), 51-54 (2015).
- Rhee, H. W., et al. Proteomic mapping of mitochondria in living cells via spatially restricted enzymatic tagging. Science. 339 (6125), 1328-1331 (2013).
- Kim, D. I., et al. Probing nuclear pore complex architecture with proximity-dependent biotinylation. P Natl Acad Sci USA. 111 (24), E2453-E2461 (2014).
- Kim, D. I., et al. An improved smaller biotin ligase for BioID proximity labeling. Mol Biol Cell. 27 (8), 1188-1196 (2016).
- Lambert, J. P., Tucholska, M., Go, C., Knight, J. D., Gingras, A. C. Proximity biotinylation and affinity purification are complementary approaches for the interactome mapping of chromatin-associated protein complexes. J Proteomics. 118, 81-94 (2015).
- Morriswood, B., et al. Novel bilobe components in Trypanosoma brucei identified using proximity-dependent biotinylation. Eukaryot Cell. 12 (2), 356-367 (2013).
- Schopp, I. M., et al. Split-BioID a conditional proteomics approach to monitor the composition of spatiotemporally defined protein complexes. Nat Commun. 8, (2017).
- Béthune, J., Artus-Revel, C. G., Filipowicz, W. Kinetic analysis reveals successive steps leading to miRNA-mediated silencing in mammalian cells. EMBO Rep. 13 (8), 716-723 (2012).
- Inoue, H., Nojima, H., Okayama, H. High efficiency transformation of Escherichia coli with plasmids. Gene. 96 (1), 23-28 (1990).
- Weidenfeld, I., et al. Inducible expression of coding and inhibitory RNAs from retargetable genomic loci. Nucleic Acids Res. 37 (7), e50(2009).
- Dyballa, N., Metzger, S. Fast and sensitive colloidal coomassie G-250 staining for proteins in polyacrylamide gels. J Vis Exp. (30), (2009).
- Jonas, S., Izaurralde, E. Towards a molecular understanding of microRNA-mediated gene silencing. Nat Rev Genet. 16 (7), 421-433 (2015).
- MacRae, I. J., Ma, E., Zhou, M., Robinson, C. V., Doudna, J. A. In vitro reconstitution of the human RISC-loading complex. P Natl Acad Sci USA. 105 (2), 512-517 (2008).
- Cox, J., Mann, M. MaxQuant enables high peptide identification rates, individualized p.p.b.-range mass accuracies and proteome-wide protein quantification. Nat Biotechnol. 26 (12), 1367-1372 (2008).
- Tyanova, S., et al. The Perseus computational platform for comprehensive analysis of (prote)omics data. Nat Methods. 13 (9), 731-740 (2016).
- Opitz, N., et al. Capturing the Asc1p/Receptor for Activated C Kinase 1 (RACK1) Microenvironment at the Head Region of the 40S Ribosome with Quantitative BioID in Yeast. Mol Cell Proteomics. 16 (12), 2199-2218 (2017).
- Mackmull, M. T., et al. Landscape of nuclear transport receptor cargo specificity. Mol Syst Biol. 13 (12), 962(2017).
- Kim, D. I., et al. BioSITe: A Method for Direct Detection and Quantitation of Site-Specific Biotinylation. J Proteome Res. , (2017).
- Udeshi, N. D., et al. Antibodies to biotin enable large-scale detection of biotinylation sites on proteins. Nat Methods. 14 (12), 1167-1170 (2017).
- Chapat, C., et al. Cap-binding protein 4EHP effects translation silencing by microRNAs. P Natl Acad Sci USA. 114 (21), 5425-5430 (2017).
- Luker, K. E., et al. Kinetics of regulated protein-protein interactions revealed with firefly luciferase complementation imaging in cells and living animals. P Natl Acad Sci USA. 101 (33), 12288-12293 (2004).
- De Munter, S., et al. Split-BioID: a proximity biotinylation assay for dimerization-dependent protein interactions. FEBS Lett. 591 (2), 415-424 (2017).
- Xue, M., et al. Optimizing the fragment complementation of APEX2 for detection of specific protein-protein interactions in live cells. Sci Rep. 7 (1), 12039(2017).
- Branon, T. C., et al. Directed evolution of TurboID for efficient proximity labeling in living cells and organisms. bioRxiv. , (2017).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados