Method Article
Cuantitativa de polimerasa análisis reacción-basado cadena de la Microbiota Intestinal murino después del tratamiento antibiótico Oral
En este artículo
Resumen
Aquí ofrecemos protocolos detallados para la administración oral de antibióticos a ratones, colección de muestras fecales, la extracción de ADN y cuantificación de coliformes fecales por qPCR.
Resumen
La microbiota intestinal tiene una influencia central en la salud humana. Disbiosis microbiana está asociado con muchos immunopathologies comunes como la enfermedad inflamatoria intestinal, asma y artritis. Por lo tanto, entender los mecanismos subyacentes de la interferencia del sistema inmune de la microbiota es de crucial importancia. La administración de antibióticos, mientras que ayudar a la remoción de patógenos, también induce cambios drásticos en el tamaño y composición de comunidades bacterianas intestinales que pueden tener un impacto en la salud humana. Tratamiento con antibióticos en ratones recapitula el impacto y a largo plazo cambios en la microbiota humana de antibióticos pacientes tratados y permite la investigación de los vínculos mecanicistas entre cambios en las comunidades microbianas y función inmune de la célula. Si bien se han descrito varios métodos para el tratamiento antibiótico de los ratones, algunos de ellos inducen deshidratación severa y pérdida de peso que complica la interpretación de los datos. Aquí, le ofrecemos dos protocolos para la administración oral de antibióticos que puede utilizarse para el tratamiento a largo plazo de ratones sin inducir pérdida de peso importante. Estos protocolos hacen uso de una combinación de antibióticos dirigidos tanto Gram-positivas y gram-negativas bacterias y pueden ser proporcionados o ad libitum en el agua potable o por sonda oral. Por otra parte, se describe un método para la cuantificación de la densidad microbiana en muestras fecales por qPCR que puede utilizarse para validar la eficacia del tratamiento antibiótico. La combinación de estos enfoques ofrece una metodología confiable para la manipulación de la microbiota intestinal y el estudio de los efectos del tratamiento antibiótico en ratones.
Introducción
La mucosa gastrointestinal de mamífera es un único entorno colonizado por una mezcla muy compleja de microorganismos que establecen una relación mutualista con el host. El sistema de defensa de la mucosa intestinal compone de una capa epitelial y una gran cantidad de células inmunes que restringen comensales en el intestino conservando su número y diversidad. Por el contrario, microorganismos comensales son necesarios para el desarrollo de un sistema inmune completamente funcional. Mientras que las interacciones entre huésped y las bacterias comensales son normalmente beneficiosas, es cada vez más claro que altera microbiota sistema inmune interferencia puede favorecer el desarrollo de enfermedades inflamatorias crónicas, tal asinflammatory intestinal enfermedad, artritis o asma1,2.
La microbiota intestinal puede ser alterada por diversos factores, pero quizás los cambios más drásticos son inducidos por el tratamiento antibiótico que altera gravemente el tamaño y composición de comunidades bacterianas3,4. Mientras que los beneficios de los antibióticos para tratar infecciones son incuestionables, los cambios de la microbiota inducidos por la exposición de antibióticos en los seres humanos también pueden modificar defensas inmunológicas que pueden llevar a efectos perjudiciales para la salud. Por ejemplo, tratamiento con antibióticos en los seres humanos se ha relacionado con un mayor riesgo de Clostridium difficile-inducida por la diarrea, el asma y ciertos tipos de cáncer3. Tratamiento con antibióticos en ratones recapitula el impacto y a largo plazo alteraciones encontradas las comunidades de intestino de pacientes tratados con antibióticos y ha permitido la investigación de los vínculos mecanicistas entre cambios en las comunidades microbianas y función inmune de la célula. Sin embargo, varios informes han demostrado que la administración de antibióticos en el agua potable ad libitum resulta en pérdida de peso muy notable como ratones abstengan de beber agua, probablemente debido a su mal gusto5,6. Así, en estos modelos la deshidratación severa concomitante a la administración oral de antibióticos puede complicar la interpretación de los experimentos con el objetivo de identificar el efecto del tratamiento antibiótico en función de las células inmunes.
Varios enfoques se pueden utilizar para explorar el tamaño y la composición de las comunidades microbianas en los compartimento intestinal7. Próxima generación tecnologías de secuenciación han proporcionado datos invaluables sobre esta materia8, sin embargo, estos métodos son relativamente caros y requieren expertos bioinformática análisis para la interpretación de los datos. Por otra parte, métodos microbiológicos tradicionales de la cultura permiten la detección de especies bacterianas, pero tienen baja sensibilidad y una gran parte de las bacterias comensales (particularmente anaerobios) son muy difíciles o imposibles de cultivar con métodos disponibles en la actualidad8. Técnicas de reacción en cadena (qPCR) cuantitativa de polimerasa se utilizan cada vez más para la cuantificación e identificación de especies bacterianas fecales, ya que proporcionan una medida independiente de la cultura rápida y confiable de la carga microbiana total. Por consiguiente, qPCR métodos han demostrado ser útiles para estudiar cambios en la microbiota asociada con la edad o con la progresión de varias enfermedades incluyendo enfermedad de intestino inflamatoria9,10. En consonancia con esto, qPCR métodos proporcionan un enfoque rápido y rentable para validar el efecto de varios tratamientos (incluyendo antibióticos) en cargas de bacterias fecales y microbiota composición10,11,12.
Aquí, presentamos un relato paso a paso detallado de dos protocolos distintos para la administración de antibiótico oral a ratones, recolección de muestras fecales, la extracción de ADN, elaboración de normas y cuantificación de bacterias en muestras de heces por qPCR. Estos protocolos proporcionan un método confiable para manipular la microbiota intestinal en ratones y al estudio de los efectos del tratamiento antibiótico en la homeostasis intestinal y la enfermedad.
Protocolo
Experimentos descritos aquí se realizaron mediante 6-8 semanas ratones del viejo tipo salvaje (C57BL6/J) mantenidas en una instalación (SPF) libre de patógenos específicos. Todos los experimentos en animales fueron aprobados por College de Londres el King y el bienestar de los animales Instituto Francis Crick y órgano de examen de ética y el Ministerio del interior de Reino Unido. Antes de comenzar cualquier procedimiento animal, garantizar la obtención de los permisos apropiados a través de la institución u organización local.
1. la administración de antibióticos
Nota: Se proporcionan dos métodos alternativos para el tratamiento antibiótico: oral por sonda nasogástrica (paso 1.1) y la administración de antibióticos a través de agua potable (paso 1.2).
- Oral por sonda nasogástrica
- Preparar las existencias de antibióticos individuales mediante la disolución en agua esterilizada en las siguientes concentraciones: ampicilina 100 mg/ml, gentamicina 100 mg/ml, neomicina 100 mg/ml, metronidazol 10 mg/ml y vancomicina 100 mg/ml. Filtro de esterilización usando un filtro de 0.45 μm. Alícuota y almacenar a-20 ° C.
- Preparar un cóctel de antibióticos mezclando la acción preparada por encima. Para un volumen de 1 mL mezclar 50 μl de la ampicilina (final 5 mg/mL), 50 μl de gentamicina (5 mg/mL final), 50 μl de la neomicina (final 5 mg/mL), 500 μl de metronidazol (5 mg/mL final), 25 μl de vancomicina (2,5 mg/mL final) y 325 μl de agua. Preparar el cóctel fresco antes de usarlo.
- Cargar la mezcla de antibióticos en una jeringa estéril de 1 mL y fijar una aguja de por sonda nasogástrica de calibre apropiado (20 G para los ratones 15 – 20 g), eliminando las burbujas.
- Ratones por sonda nasogástrica con 200 μL de la mezcla antibiótica.
- Agarrar firmemente la piel sobre el hombro del ratón, extienden la cabeza y el cuello para enderezar el esófago. Dirigir la punta de bola de la aguja de alimentación a lo largo del techo de la boca y hacia la parte posterior de la faringe. Luego, suavemente pase en el esófago e inyectar la solución de 200 μL.
- Administrar una vez cóctel antibiótica cada día durante la duración del experimento.
- Antibióticos en el agua potable
PRECAUCIÓN: Vigilar cuidadosamente peso ratón diario para las dos primeras semanas de la administración de antibióticos en el agua potable.- Preparar un cóctel de antibióticos mediante la disolución de lo siguiente en 1 L de agua esterilizada: 1 g de ampicilina (1 g/L final), 1 g de neomicina (1 g/L final), 1 g de metronidazol (1 g/L final), 0,5 g de vancomicina (final 0.5 g/L) y 8 sobres (0,75 g) de artificial edulcorante (60 g/L final). Agite hasta que se disuelva. Almacenar a 4 ° C.
Nota: Se agrega edulcorante para ocultar el sabor de los antibióticos y evitar la deshidratación de ratones. Mientras que varias marcas de edulcorantes pueden funcionar, la concentración final requerida puede ser diferente para cada marca específica. - Llene el cóctel antibiótico en una botella de agua (~ 100 mL/botella) y coloque la botella en una jaula del ratón.
Nota: Usar botellas marrones o cubrir las botellas con papel de aluminio para proteger a antibióticos de la luz. - Vuelva a colocar el cóctel antibiótico con acción fresca dos veces a la semana durante la duración del experimento.
- Preparar un cóctel de antibióticos mediante la disolución de lo siguiente en 1 L de agua esterilizada: 1 g de ampicilina (1 g/L final), 1 g de neomicina (1 g/L final), 1 g de metronidazol (1 g/L final), 0,5 g de vancomicina (final 0.5 g/L) y 8 sobres (0,75 g) de artificial edulcorante (60 g/L final). Agite hasta que se disuelva. Almacenar a 4 ° C.
2. colección de muestras fecales de heces, contenido íleon y pared de íleon
- Pesar y etiquetar 2 mL autoclave tubos para recogida de muestras.
- Para la recogida de muestras de heces frescas, colocar cada ratón en un limitador y recoger pellets fecales directamente del ano de un tubo de recogida.
Nota: Las muestras pueden obtenerse también colocando ratones en una jaula limpia esterilizada, y recoger muestras de heces con pinzas esterilizadas limpiamos. - Eutanasia a los ratones con CO2 asfixia seguida por dislocación cervical.
- Pone un canal de ratón con el abdomen completamente descubierto y rocíe el área abdominal con etanol al 70%.
- Usando tijeras y pinzas esterilizadas, haga una incisión transversal en el abdomen para exponer el peritoneo sin dañar cualquier tejidos internos. Levantar el peritoneo y hacer una incisión para exponer los intestinos.
- Quitar los intestinos (de colon estómago) con las pinzas y tijeras y colocar en una placa Petri estéril.
- Cuidadosamente mediante fórceps para el intestino (SI) de las arterias mesentéricas y grasa. Extender el intestino y colocar en un paño limpio de laboratorio.
- Con una regla, medir y cortar 4 cm de íleo distal de SI (la parte más cercana al ciego). Corte y deseche el 1 cm de intestino proximal para el ciego. Habrá una porción de 3 cm de íleon que se utilizarán para recoger las bacterias intestinales a la izquierda.
- Sujete la porción de íleon (3 centímetros) sobre un tubo estéril de 2 mL. Recoger el contenido intestinal por sacar directamente el intestino y el cobro de la muestra en el tubo. Esta muestra tendrá bacterias del contenido del íleon.
- Preparar una jeringa de 20 mL con tampón frío fosfato salino (PBS) y eliminar la porción de íleon (ignorar el paso).
- Ponga la porción de íleon en una toalla de papel limpia y abrir longitudinalmente con tijeras.
- Raspe el interior de la pared de íleon con un bisturí.
- Recoger cualquier bacteria en el bisturí por el bisturí con 1 mL de PBS sobre un tubo de centrífuga limpio de lavado. Girar a 8.000 × g por 5 min a las bacterias de la pelotilla y descarte el sobrenadante. Esta muestra contiene bacterias de la pared del íleon.
Nota: Las muestras fecales de heces, contenido del íleon y la pared pueden ser congeladas y almacenadas a-80 ° C hasta su uso. - Pesan los tubos con las muestras de contenido de heces e íleon y restar el peso de los tubos de vacíos (del paso 2.1) para obtener el peso fecal en cada muestra.
- Extracto de ADN bacteriano de heces, contenido de íleon y muestras de la pared de íleon usando kits disponibles en el mercado. Almacenar muestras de ADN a-20 ° C hasta su uso.
3. cuantificación de Microbiota Intestinal por qPCR
Nota: Este procedimiento incluye la generación de un estándar (paso 3.1) y el método para la disposición de qPCR para muestras fecales y estándar (paso 3.2)
- Generación de un estándar para qPCR
- Utilizar la reacción en cadena de polimerasa (PCR) para amplificar el gene del rRNA 16S de la DNA genomic extraída de una cultura bacteriana usando los reactivos13 y PCR condiciones detalladas en la tabla 1.
- Ejecutar el producto PCR en un gel de agarosa al 1.5% y purificar la banda de ADN usando un kit de extracción de ADN disponible en el mercado.
- Ligar el purificado ADN fragmento utilizando una clonación kit (véase la Tabla de materiales, utilizar un vector que contiene los marcadores de resistencia a los antibióticos y la fusión del gen de β-galactosidasa (LacZ) para la selección de Colonia azul/blanco) y transformar competente DH10 E. coli siguiendo las instrucciones del fabricante. Placa de transformación a ampicilina (100 μg/mL), placas de agar de Luria Bertani (LB) X-gal (20 μg/mL) (10 G/l triptona, 5 g/L Extracto de levadura, 10 g/L de NaCl, Agar de 15 g/L). Incubar durante la noche (O/N) a 37 ° C.
- Seleccione una colonia solo positivo (blanco) de la placa. Inocular en caldo LB de 5 mL que contienen 100 μg/mL de ampicilina y O/N de incubar a 37 ° C, agitación a 250 rpm.
- De la cultura de O/N, aislar y purificar el plásmido usando un kit comercial miniprep según instrucciones del fabricante.
Nota: Es importante la secuencia de la inserción del plásmido en esta etapa, para asegurar que el plásmido contiene una única copia del gene del rRNA 16S y determinar su longitud en pares de bases (PB). - Alinear el plásmido con una enzima de restricción que corta el plásmido solamente una vez.
- Purificar el plásmido lineal usando un kit disponible comercialmente.
- Determinar la concentración del plásmido mediante la medición de absorbancia a 260 nm usando un espectrofotómetro.
- Calcular el número de copias de plásmido/μl de la muestra utilizando la fórmula siguiente14:
número de copias = (cantidad * 6.022 × 1023) / (longitud * 1 × 109 * 650)
donde cantidad es la concentración de ADN obtenida en el paso 3.1.8 (ng/μL); y longitud es la longitud total del plásmido (con inserto) en bp. Se obtiene el número de copias como copias/μl.
Nota: Existen herramientas en línea basados en la fórmula anterior que permiten el cálculo fácil del número de copias del plásmido. - Alícuota y tienda el estándar según sea necesario en-20 ° C hasta su uso.
- qPCR preparación para estándar y muestras de heces
- Descongelar la qPCR estándar (del paso 3.1.10), ADN de muestras fecales (del paso 2.15) y qPCR reactivos (de un kit disponible comercialmente) en hielo.
Nota: qPCR reactivos utilizados en este ejemplo son la mezcla de reacción de qPCR de SYBR Green y cartillas de avance y retroceso (tabla 2). - Diluir el estándar en agua estéril libre de ADN en un rango de 107 a 102 copias/μl (por ejemplo, 1/5 diluciones seriadas de 107 a 6.4 x 102 copias/μL). Diluir muestra fecal DNA 1/2, 1/5, 1/10.
- Hacer una reacción de la mezcla principal para el número total de reacciones y 1 (tabla 2).
- Mezcla 30 μl de la mezcla principal y 5 μl de plantilla (estándar, muestra o agua para el control negativo)
- Añadir 10 μl de esta mezcla a cada pocillo de una placa de 384 pozos óptico qPCR. Realice cada reacción por triplicado.
- Sellar el lugar de la qPCR, centrifugar brevemente y la placa de la máquina de qPCR programada con las siguientes condiciones de ciclo de carga: 95 ° C por 20 min seguido por 40 ciclos de 95 ° C por 15 s y 60 ° C durante 1 minuto.
- Obtener los valores de CT de estándar y las muestras.
- Generar una curva estándar mediante la representación de los valores de CT para los estándares versus el logaritmo del número de copia del plásmido (según lo calculado en el paso 3.1.9). Realice regresión lineal de la curva estándar.
- Calcular los números de copia de 16S rDNA para muestras de heces por interpolación valores de CT (obtenidos en paso 3.2.2) en la curva estándar.
Nota: números de copia de ADNr 16S para cada muestra deberán corregirse teniendo en cuenta el factor de dilución de la muestra (como preparado en el paso 3.2.2), el volumen final los ácidos nucleicos que se eluyen (en el paso 2.15) y la cantidad de muestra fecal (calculado en el paso de 2.14) para obtener copias de genes por gramo de heces.
- Descongelar la qPCR estándar (del paso 3.1.10), ADN de muestras fecales (del paso 2.15) y qPCR reactivos (de un kit disponible comercialmente) en hielo.
Resultados
Aquí ofrecemos dos protocolos alternativos para el tratamiento antibiótico oral de ratones. La figura 1 muestra el porcentaje de peso corporal (relacionado con el peso original de la línea de base para cada animal) en ratones tratados con antibióticos orales por sonda nasogástrica (rojo) o en el agua potable (azul) durante 10 días consecutivos. No hay pérdida de peso notable se encuentra en ratones que reciben antibióticos por sonda oral. Sin embargo, cuando los ratones son tratados con antibióticos ad libitum en el agua potable, bajar de peso (~ 10%) dentro de los primeros días de la administración de antibióticos, pero recuperar la ganancia de peso normal después de eso (figura 1). Sin embargo, unos 5-10% de los ratones recibiendo antibióticos en el agua potable puede llegar a > 20% pérdida de peso durante la primera semana de tratamiento, en cuyo caso son sacrificados.
La cuantificación de bacterias en muestras de heces requiere el uso de una adecuada curva estándar que se obtiene trazando el registro del número de copias para el estándar (según lo calculado en el paso 3.2.2) versus los valores de CT obtenidos de qPCR (paso 3.2.7). Figura 2A muestra un ejemplo representativo de una curva estándar que cumple con los criterios de rendimiento de la curva estándar con un valor de2 R de 0.99827, una pendiente de-3.09 y una eficacia ((-1 + 10^(-1/slope))*100) de 110%. Se prefieren los valores de2 R de 0.99 y eficiencias de la PCR dentro del rango de 90 a 110%. Dentro del rango lineal, la ecuación del análisis de regresión permite la cuantificación de la abundancia de ADNr 16S en las muestras fecales. Figura 2B muestra el número de copias de 16S rDNA en heces fecales, SI contenido y pared SI. En la figura 2B se muestran datos como 16S rDNA copias/g de muestra fecal de heces fecales y SI contenido. Para la pared SI, se presentan datos como el número total de ejemplares de ADNr 16S Obtenido de la bacteria que se recuperó de los 3 cm de pared SI (como la cantidad de material de partida es demasiado pequeña para obtener un peso exacto).
Para evaluar el efecto de antibióticos sobre la densidad de bacterias en muestras de heces, ratones fueron tratados con antibióticos por sonda oral diariamente durante 10 días (días 1 a 10) y se colectaron muestras de heces antes (día 0), en diferentes momentos durante el tratamiento con antibióticos ( días 5 y 10) y 7 días después de suspender la administración de antibióticos (día 17; Figura 3A). Como se muestra en la figura 3B, tratamiento antibiótico induce una fuerte disminución en el número de 16S rDNA copias/g de heces detectada en los días 5 y 10, mientras que la densidad de bacterias en las heces que se recuperó a los niveles normales (comparables a un tratamiento previo) 1 semana después la administración de antibióticos fue detenida (día 17).
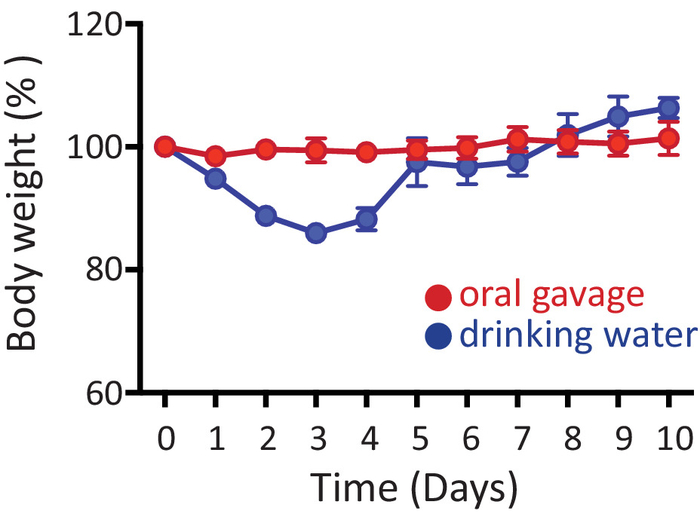
Figura 1: administración de antibióticos. Ratones recibieron antibióticos oral por sonda nasogástrica (rojo) o en el agua potable (azul) durante 10 días consecutivos. Muestra los pesos de los ratones a lo largo de la duración de los experimentos en relación con el peso original antes de la administración de antibióticos (día 0). Datos se muestran como media ± SEM. haga clic aquí para ver una versión más grande de esta figura.
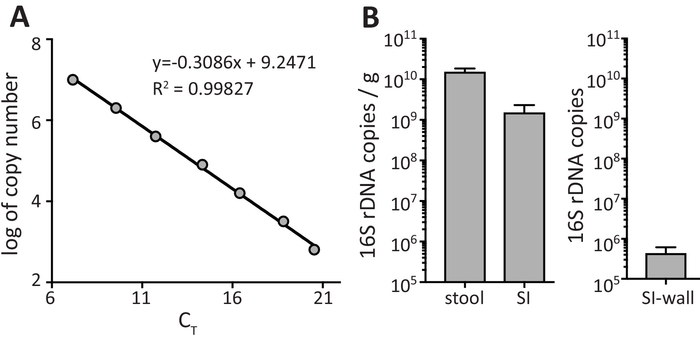
Figura 2: amplificación de qPCR del gen de 16S rRNA de los estándares y muestras de heces. (A) regresión lineal de la curva estándar con descriptores de la curva estándar. (B) cálculo de abundancia de genes de muestras fecales. Datos se muestran como media ± SEM. haga clic aquí para ver una versión más grande de esta figura.
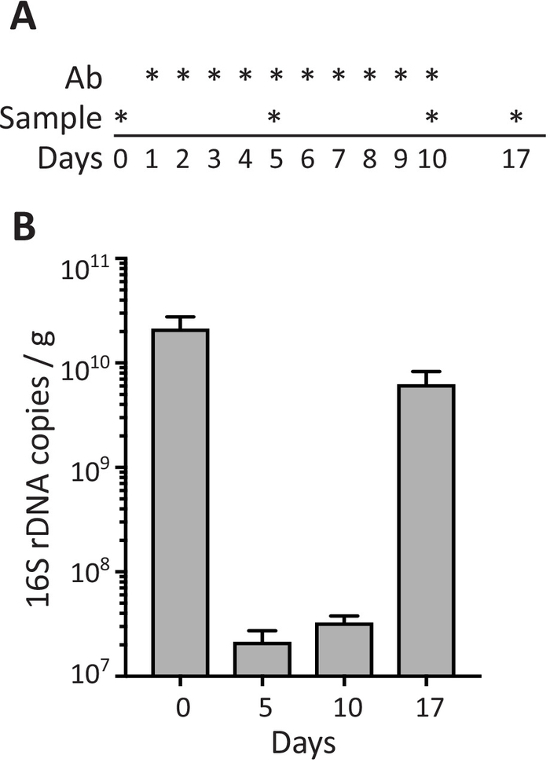
Figura 3: bacterias fecales durante el tratamiento antibiótico. (A) esquema de la programación para la administración de antibióticos oral por sonda nasogástrica (Ab) y recogida de la muestra como con *. (B) 16S rDNA ejemplares por gramo de heces en las muestras de heces de los ratones en los días indicados. Datos se muestran como media ± SEM. haga clic aquí para ver una versión más grande de esta figura.
| Reactivo de | Volumen | |
| DNA | 8 ΜL | |
| Tampón 10 X | 2 ΜL | |
| dNTPs (10mM) | 0.4 ΜL | |
| Eubacterias - primer F (10 mM) | 1 ΜL | |
| Eubacterias - primer R (10 mM) | 1 ΜL | |
| Taq polimerasa | 0.2 ΜL | |
| H2O | 7,4 ΜL | |
| Volumen total | 20 ΜL | |
| Secuencias de la cartilla | ||
| Eubacterias - cartilla de F | 5' ACTCCTACGGGAGGCAGCAGT 3' | |
| Eubacterias - primer R | 5' ATTACCGCGGCTGCTGGC 3' | |
| Condiciones de ciclismo | ||
| Temperatura | hora | Ciclos de |
| 94 ° C | 5 min | 1 x |
| 94 ° C | 30 s | 30 x |
| 60 ° C | 30 s | 30 x |
| 72 ° C | 1 min | 30 x |
| 72 ° C | 5 min | 1 x |
| 4 ° C | ∞ | 1 x |
Tabla 1: PCR reactivos y condiciones de. Esta tabla proporciona los reactivos y condiciones ciclo PCR para amplificar el gen de rRNA 16S de cultivo bacteriano para la generación de un estándar a utilizar en los ensayos de qPCR. Secuencias de la cartilla fueron publicadas originalmente por Kruglov et al. 13.
| Reactivo de | Volumen |
| Mezcla maestra SYBR Green (2 x) | 17.5 ΜL |
| Eubacterias - primer F (10 mM) | 0,7 ΜL |
| Eubacterias - primer R (10 mM) | 0,7 ΜL |
| H2O | 11,1 ΜL |
Tabla 2: mix master qPCR. Los volúmenes de muestra (μL de volumen final 35) son para que una sola muestra para ejecutarse en triplicado (10 μl) en una placa de 384 pozos qPCR (contabilidad para 5 μl adicionales para error de pipeteo). La cantidad puede ampliarse según el número de muestras a analizar.
Discusión
Aquí ofrecemos protocolos experimentales para la administración oral de antibióticos a ratones y cuantificación de coliformes fecales por qPCR. La combinación de antibióticos utilizados en los objetivos de este protocolo (contiene ampicilina, gentamicina, neomicina, metronidazol y vancomicina) bacterias Gram positivas y gram negativas, con actividad bactericida contra un amplio espectro de bacterias. Oral por sonda nasogástrica y administración de antibióticos en el agua potable reducen bacterias fecales carga5,6,12. Además, ambos tratamientos tienen un profundo efecto en el fenotipo de los ratones que se desarrollan varias características típicas de los ratones libres de gérmenes como ciego agrandado bazo reducido tamaño. La selección de un método en particular para la administración de antibióticos puede posiblemente depende de la longitud del experimento como el método oral por sonda nasogástrica requiere administración diaria de antibióticos, siendo más intensivas y posiblemente causando más molestias a los animales en el largo plazo.
Para la administración de antibióticos en el agua potable, debe tener cuidado con la adición de edulcorante para la mezcla de antibióticos ya que es un factor crucial para mantener los ratones de la deshidratación. Varios grupos han demostrado cómo la administración de antibióticos en el agua potable (sin adición de edulcorante) conduce a pérdida de peso muy severa y rápida con todos los ratones perder más del 20% del peso corporal inicial en los primeros días del experimento5 , 6. en nuestro protocolo, el uso de este edulcorante sacarina base parecía ser suficiente para enmascarar el sabor de antibiótico en el agua y los ratones perdida peso en los primeros días después de la administración de antibióticos, pero sus pesos se recuperó rápidamente después de eso ( Figura 1). Sin embargo, en nuestros experimentos 5-10% de los ratones todavía alcanzar el punto final humano de > 20% de pérdida de línea de base el peso corporal y debían ser sacrificados. También hemos probado edulcorantes sucralosa-basado que falló completamente para evitar la deshidratación de ratones (100% de los ratones perdidas > 20% del peso) mientras que otros autores han publicado fracasos similares para edulcorantes basados en aspartame5,6. Sumado a esto, la edad, genética y estado de salud general de los ratones utilizados para los experimentos se deben considerar, ya que pueden influir en la pérdida de peso y el bienestar animal durante el tratamiento con antibióticos. Por lo tanto, el monitoreo cuidadoso del peso de ratones y estado de salud general debe realizarse diariamente durante las dos primeras semanas de la administración oral de antibióticos.
qPCR métodos proporcionan un enfoque rápido y rentable para la cuantificación del 16S rRNA en muestras de heces. Sin embargo, deben considerarse algunas limitaciones con respecto a esta técnica como: i) la exigencia de un estándar de calidad confiable; II) el diseño y la eficiencia de los iniciadores de la qPCR; III) el hecho de que los microorganismos pueden tener números diferentes de copia del gene del rRNA 16S, así copias del gen no sea directamente igual celular cuenta15. Sin embargo, qPCR es un método sensible y robusto que permite un análisis rápido de muestras de heces. Este método puede ser especialmente útil para validar rápidamente el efecto de diversos tratamientos (incluyendo antibióticos) en cargas bacterianas fecales como aquí detallada. Por otra parte, aunque proporcionamos un protocolo para la cuantificación del total 16S rRNA, este método puede ser fácilmente adaptado (mediante el diseño de iniciadores específicos16) a permitir la identificación de taxones bacterianos individuales, proporcionando tanto cuantitativa y información cualitativa sobre la composición y tamaño del microbioma.
En Resumen, hemos incluido dos protocolos para el tratamiento antibiótico oral de ratones y de un método basado en qPCR para cuantificar los cambios inducidos por antibióticos en bacterias fecales. Aunque estos protocolos pueden optimizados y combinados con otros enfoques según necesidades experimentales, pueden servir como herramientas rápidas, rentables y confiables para manipular la microbiota intestinal murina y para el estudio de los efectos del tratamiento antibiótico en la homeostasis intestinal y la enfermedad.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este trabajo fue financiado por el Consejo de investigación médica de Reino Unido (beca al Señor P.B./L008157/1); R.J. fue apoyado por una beca intraeuropeas Marie Curie de Marie Curie (H2020-MSCA-IF-2015-703639); P.m.b. fue apoyado por una beca del Medical Research Council de Reino Unido y College Londres sociedad del rey de formación Doctoral en ciencias biomédicas (Señor/N013700/1).
Materiales
| Name | Company | Catalog Number | Comments |
| Ampicillin sodium salt | Sigma-Aldrich (Merck) | A9518 | |
| Neomycnin trisulfate salt hydrate | Sigma-Aldrich (Merck) | N1876 | |
| Metronidazole | Sigma-Aldrich (Merck) | M3761 | |
| Vancomycin hydrochloride | Sigma-Aldrich (Merck) | V2002 | |
| Gentamicin sulfate salt | Sigma-Aldrich (Merck) | G3632 | |
| Tryptone | Sigma-Aldrich (Merck) | T7293 | |
| Yeast Extract | Sigma-Aldrich (Merck) | Y1625 | |
| NaCL | Sigma-Aldrich (Merck) | S7653 | |
| Sweetener Sweet'n Low | Sweet'N Low | Available in the UK from Amazon.co.uk | |
| X-Gal (5-brom-4-chloro-3-indoyl B-D-galactopyranoside) | Fisher scientific | 10234923 | |
| Phosphate Buffered Saline | Thermo Fisher Scientific (Gibco) | 10010023 | |
| Ultrapure Agarose | Thermo Fisher Scientific (Invitrogen) | 16500500 | |
| RT-PCR grade water | Thermo Fisher Scientific (Invitrogen) | AM9935 | |
| Phusion High-Fidelity DNA Polymerase | New England BioLabs | M0530 | |
| Deoxynucleotide (dNTP) Solution Mix | New England BioLabs | N0447 | |
| iTaq Universal SYBR Green Supermix | Bio-Rad | 1725124 | with ROX |
| TOPO TA cloningTM for sequencing | Thermo Fisher Scientific (Invitrogen) | 450030 | |
| QIAamp fast DNA Stool mini kit | Qiagen | 51604 | |
| QIAprep spin Miniprep kit | Qiagen | 27106 | |
| QIAquick gel extraction kit | Qiagen | 28704 | |
| Syringe filter 0.45 µm | Fisher scientific | 10460031 | |
| Swann-MortonTM Carbon steel sterile scalpel blades | Fisher scientific | 11792724 | |
| Syringe (1 mL) | BD Plastipak | 303172 | |
| Syringe (20 mL) | BD Plastipak | 300613 | |
| 1.5 mL Crystal clear microcentriguge tube | StarLab | E1415-1500 | |
| 2 mL Ultra high recovery microcentrifuge tube | StarLab | I1420-2600 | |
| Oral dosing needles 20 G x 38 mm curved (pk/3) | Vet-Tech | DE008A | |
| Sterilin petri dish 50 mm | Scientific Laboratory Supplies | PET2020 | |
| Absolute qPCR plate seals | Thermo Fisher Scientific | AB1170 | |
| MicroAmpTM optical 384-well plate | Thermo Fisher Scientific (Applied Biosystems) | 4309849 | |
| ViiA7TM 7 real-time PCR system with 384-well block | Thermo Fisher Scientific (Applied Biosystems) | 4453536 | |
| Spectrophotometer (Nanodrop 1000) | Thermo Fisher Scientific | ND-1000 | |
| Labnet Prism microcentrifuge | Labnet | C2500 | |
| MultiGene Optimax Thermal cycler | Labnet | TC9610 |
Referencias
- Belkaid, Y., Hand, T. W. Role of the microbiota in immunity and inflammation. Cell. 157 (1), 121-141 (2014).
- Hooper, L. V., Littman, D. R., Macpherson, A. J. Interactions between the microbiota and the immune system. Science. 336 (6086), 1268-1273 (2012).
- Ubeda, C., Pamer, E. G. Antibiotics, microbiota, and immune defense. Trends in Immunology. 33 (9), 459-466 (2012).
- Dethlefsen, L., Huse, S., Sogin, M. L., Relman, D. A. The pervasive effects of an antibiotic on the human gut microbiota, as revealed by deep 16S rRNA sequencing. PLoS Biology. 6 (11), e280 (2008).
- Reikvam, D. H., et al. Depletion of murine intestinal microbiota: effects on gut mucosa and epithelial gene expression. PLoS One. 6 (3), e17996 (2011).
- Hill, D. A., et al. Metagenomic analyses reveal antibiotic-induced temporal and spatial changes in intestinal microbiota with associated alterations in immune cell homeostasis. Mucosal Immunology. 3 (2), 148-158 (2010).
- Fraher, M. H., O'Toole, P. W., Quigley, E. M. Techniques used to characterize the gut microbiota: a guide for the clinician. Nature Reviews Gastroenterology & Hepatology. 9 (6), 312-322 (2012).
- Eckburg, P. B., et al. Diversity of the human intestinal microbial flora. Science. 308 (5728), 1635-1638 (2005).
- Sokol, H., et al. Low counts of Fecalibacterium prausnitzii in colitis microbiota. Inflammatiry Bowel Diseases. 15 (8), 1183-1189 (2009).
- Bartosch, S., Fite, A., Macfarlane, G. T., McMurdo, M. E. Characterization of bacterial communities in feces from healthy elderly volunteers and hospitalized elderly patients by using real-time PCR and effects of antibiotic treatment on the fecal microbiota. Applied Environmental Microbiology. 70 (6), 3575-3581 (2004).
- Ubeda, C., et al. Vancomycin-resistant Enterococcus domination of intestinal microbiota is enabled by antibiotic treatment in mice and precedes bloodstream invasion in humans. Journal of Clinical Investigation. 120 (12), 4332-4341 (2010).
- Saez de Guinoa, J., et al. CD1d-mediated lipid presentation by CD11c(+) cells regulates intestinal homeostasis. EMBO Journal. 37 (5), (2018).
- Kruglov, A. A., et al. Nonredundant function of soluble LTalpha3 produced by innate lymphoid cells in intestinal homeostasis. Science. 342 (6163), 1243-1246 (2013).
- Wilhelm, J., Pingoud, A., Hahn, M. Real-time PCR-based method for the estimation of genome sizes. Nucleic Acids Research. 31 (10), e56 (2003).
- Kembel, S. W., Wu, M., Eisen, J. A., Green, J. L. Incorporating 16S gene copy number information improves estimates of microbial diversity and abundance. PLoS Computational Biology. 8 (10), e1002743 (2012).
- Yang, Y. W., et al. Use of 16S rRNA Gene-Targeted Group-Specific Primers for Real-Time PCR Analysis of Predominant Bacteria in Mouse Feces. Applied Environmental Microbiology. 81 (19), 6749-6756 (2015).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados