Method Article
Quantitative Polymerase Kettenreaktion-basierte Analysen der murinen intestinale Mikrobiota nach Behandlung mit oralen Antibiotika
In diesem Artikel
Zusammenfassung
Hier bieten wir ausführliche Protokolle für die orale Verabreichung von Antibiotika für Mäuse, Sammlung von Stuhlproben, DNA-Extraktion und Quantifizierung von fäkalen Bakterien durch qPCR.
Zusammenfassung
Der Darm Microbiota hat einen zentralen Einfluss auf die menschliche Gesundheit. Mikrobielle Dysbiose ist verbunden mit vielen gemeinsamen Immunopathologies wie chronisch entzündliche Darmerkrankungen, Asthma und Arthritis. So ist das Verständnis der Mechanismen, die Mikrobiota Immunsystem Übersprechen von entscheidender Bedeutung. Antibiotikum Verwaltung, während Beihilfe Erreger Abfertigung, induziert auch drastische Veränderungen in der Größe und Zusammensetzung der intestinalen bakteriellen Gemeinschaften, die sich auf die menschliche Gesundheit auswirken kann. Antibiotische Behandlung bei Mäusen rekapituliert die Auswirkungen und die langfristige Veränderungen der menschlichen Mikrobiota von Antibiotika behandelten Patienten und ermöglicht die Untersuchung der mechanistischen Verbindungen zwischen Veränderungen in der mikrobiellen Gemeinschaften und Immunzelle Funktion. Während mehrere Methoden zur antibiotischen Behandlung von Mäusen beschrieben worden, einige von ihnen induzieren, schwere Dehydratation und Gewichtsabnahme erschwert die Interpretation der Daten. Hier bieten wir zwei Protokolle für orale Antibiotika Einnahme, die für die Langzeitbehandlung von Mäusen verwendet werden kann, ohne daß große Gewichtsabnahme. Diese Protokolle stellen eine Kombination von Antibiotika grampositive und gramnegative Bakterien verwenden und entweder Ad Libitum im Trinkwasser oder durch orale Magensonde bereitgestellt werden können. Darüber hinaus beschreiben wir eine Methode zur Quantifizierung der mikrobiellen Dichte in Stuhlproben von qPCR die verwendet werden können, um die Wirksamkeit der Antibiotika-Behandlung zu überprüfen. Die Kombination dieser Ansätze bietet eine bewährte Methodik für die Manipulation der intestinalen Mikrobiota und die Studie über die Auswirkungen der antibiotischen Behandlung bei Mäusen.
Einleitung
Die Säugetier-Magen-Darm-Schleimhaut ist eine einzigartige Umgebung, die kolonisiert durch eine hochkomplexe Mischung von Mikroorganismen, die eine symbiotische Beziehung mit dem Host zu etablieren. Das Abwehrsystem der Darmschleimhaut besteht aus einer Epithelschicht und eine Fülle von Immunzellen, die Kommensalen innerhalb des Darms zu beschränken, unter Wahrung ihrer Zahl und Vielfalt. Im Gegensatz dazu sind Kommensale Organismen für die Entwicklung eines funktionsfähigen Immunsystems erforderlich. Während Interaktionen zwischen Host und Kommensalen Bakterien normalerweise vorteilhaft sind, wird es immer deutlicher, dass das Dysregulated Immunsystem-Mikrobiota Übersprechen die Entwicklung von chronischen Entzündungskrankheiten, solche Asinflammatory Darm begünstigen können Krankheit, Arthritis oder Asthma1,2.
Der Darm Microbiota kann durch verschiedene Faktoren verändert werden, aber vielleicht sind die drastischsten Veränderungen induziert durch antibiotische Behandlung, die die Größe und Zusammensetzung der bakteriellen Gemeinschaften3,4stark verändert. Während die Vorteile von Antibiotika zur Behandlung von Infektionen unbestritten sind, können die Mikrobiota induziert durch Antibiotika Exposition beim Menschen auch Immunabwehr Änderungen nachteilige Auswirkungen auf die Gesundheit führen kann. Zum Beispiel Antibiotika-Behandlung bei Menschen mit einem erhöhten Risiko von Clostridium Difficileverknüpft wurde-induzierte, Durchfall, Asthma und bestimmte Arten von Krebs3. Antibiotische Behandlung bei Mäusen rekapituliert die Auswirkungen und die langfristige Veränderungen im Darm Gemeinschaften der mit Antibiotika behandelten Patienten gefunden und Untersuchung der mechanistischen Verbindungen zwischen Veränderungen in der mikrobiellen Gemeinschaften und Immunzelle Funktion aktiviert hat. Mehrere Berichte haben jedoch gezeigt, dass die Verabreichung von Antibiotika auf das Trinkwasser Ad Libitum in sehr deutlicher Gewichtsverlust führt, wie Trinkwasser, vermutlich wegen seiner schlechten Geschmack5,6Mäuse unterlassen. So kann die schwere Dehydratation, orale antibiotische Verabreichung begleitenden in diesen Modellen die Interpretation der Experimente mit dem Ziel, die Wirkung der antibiotischen Behandlung in Immunzelle Funktion identifizieren erschweren.
Mehrere Ansätze können verwendet werden, um die Größe und Zusammensetzung mikrobieller Gemeinschaften in den Darm Fach7zu erkunden. Nächste Generation Sequenziertechnologien lieferten wertvolle Daten auf dieser Angelegenheit8, aber diese Methoden relativ teuer sind und benötigen kompetente bioinformatische Analysen für die Interpretation der Daten. Auf der anderen Seite, traditionelle mikrobiologische Kulturmethoden ermöglichen die Erkennung von Bakterienarten, aber sie haben geringen Empfindlichkeit und ein großer Teil der Kommensalen Bakterien (insbesondere Anaerobier) sind sehr schwer oder gar nicht mit pflegen derzeit verfügbaren Methoden8. Quantitative Polymerase-Kettenreaktion (qPCR) Techniken werden zunehmend zur Quantifizierung und Identifizierung von fäkalen Bakterien-Spezies eingesetzt, da sie eine schnelle und zuverlässige kulturunabhängig Maßnahme des gesamten mikrobiellen Belastung bieten. Dementsprechend haben qPCR Methoden, Veränderungen in der Mikrobiota, verbunden mit dem Alter oder mit einer Progression von mehreren Krankheiten einschließlich entzündliche Darm-Krankheit9,10studieren nützlich erwiesen. Im Einklang mit dieser bieten qPCR Methoden einen schnellen und kostengünstigen Ansatz um die Wirkung der verschiedenen Behandlungen (einschließlich Antibiotika) zu überprüfen in fäkale bakterielle Belastungen und Mikrobiota Zusammensetzung10,11,12.
Hier präsentieren wir Ihnen Schritt für Schritt detailliert über zwei unterschiedliche Protokolle für die orale Verabreichung der Antibiotika, Mäuse, Kotprobe Sammlung, DNA-Extraktion, Erarbeitung von Normen und Quantifizierung von Bakterien in Stuhlproben von qPCR. Diese Protokolle stellen eine zuverlässige Methode, die intestinale Mikrobiota bei Mäusen zu manipulieren und die Auswirkungen der antibiotischen Behandlung bei intestinalen Homöostase und Krankheit zu studieren.
Protokoll
Hier beschriebenen Experimente wurden durchgeführt mit ca. 6-8 Wochen alte Wildtyp (C57BL6/J) Mäusen in einer spezifischen Erreger frei (SPF) Anlage gepflegt. Alle Tierversuche wurden von der Kings College London und Francis Crick Institut Animal Welfare und ethische Nachprüfungsstelle das Vereinigte Königreich Innenministerium genehmigt. Vor Beginn jedes Tier Verfahren, stellen Sie sicher, dass die entsprechenden Berechtigungen durch lokale Institution/Organisation gewonnen werden.
1. Gabe von Antibiotika
Hinweis: Werden zwei alternative Methoden zur Behandlung mit Antibiotika zur Verfügung gestellt: orale Magensonde (Schritt 1.1) und Verabreichung von Antibiotika über das Trinkwasser (Schritt 1.2).
- Orale Magensonde
- Bestände der einzelnen Antibiotika durch lösen sie im Autoklaven Wasser in den folgenden Konzentrationen vorzubereiten: Ampicillin bei 100 mg/mL, Gentamicin bei 100 mg/mL, Neomycin bei 100 mg/mL, bei 10 mg/mL Metronidazol und Vancomycin bei 100 mg/mL. Filter mit einem 0,45 µm-Filter zu sterilisieren. Aliquoten und Store bei-20 ° C.
- Bereiten Sie einen Cocktail von Antibiotika durch Mischen der Bestände über vorbereitet. Mischen Sie für ein Volumen von 1 mL 50 µL Ampicillin (5 mg/mL final), 50 µL Gentamicin (5 mg/mL final), 50 µL Neomycin (5 mg/mL final), 500 µL von Metronidazol (5 mg/mL final), 25 µL von Vancomycin (2,5 mg/mL final) und 325 µL Wasser. Der cocktail frisch vor Gebrauch zubereiten.
- Laden Sie die antibiotische Mischung in eine sterile 1 mL Spritze und beheben Sie eine entsprechende Nadel der Magensonde (20 G für 15 – 20 g Mäuse) während die Beseitigung eventuell Luftblasen zu.
- Magensonde Mäuse mit 200 µL des Antibiotika-Mix.
- Schnappen Sie sich die Haut über die Maus Schulter fest, Dehnen des Kopfes und Halses, die Speiseröhre zu begradigen. Direkt dem Kugelende der Fütterung Nadel auf das Dach des Mundes und in Richtung der Rückseite des Rachens. Dann vorsichtig in die Speiseröhre überliefern und 200 μl Lösung zu injizieren.
- Verwalten Sie die antibiotische cocktail einmal täglich für die Dauer des Experiments.
- Antibiotika im Trinkwasser
Achtung: Überwachen Sie sorgfältig Maus Gewicht täglich für die ersten zwei Wochen der antibiotische Verabreichung im Trinkwasser.- Bereiten Sie einen Cocktail von Antibiotika durch Auflösen in 1 L Wasser autoklaviert Folgendes: 1 g Ampicillin (1 g/L Finale), 1 g Neomycin (1 g/L Finale), 1 g Metronidazol (1 g/L Finale), 0,5 g Vancomycin (0,5 g/L Finale) und 8 Sachets (0,75 g) des künstlichen Süßstoff (60 g/L Finale). Schütteln Sie, bis aufgelöst. Shop bei 4 ° C.
Hinweis: Süßstoff wird hinzugefügt, den Geschmack von Antibiotika zu verstecken und Mäuse Austrocknung zu verhindern. Während mehrere Süßstoff Marken arbeiten können, kann die Endkonzentration erforderlich für jede spezifische Marke abweichen. - Den Antibiotika-Cocktail in eine Flasche Wasser füllen (ca. 100 mL/Flasche) und stellen Sie die Flasche auf eine Maus Käfig.
Hinweis: Verwenden Sie braune Flaschen oder decken Sie die Flaschen mit Folie zu Antibiotika vor Licht zu schützen. - Ersetzen des Antibiotika-Cocktails mit frischer Vorrat zweimal in der Woche für die Dauer des Experiments.
- Bereiten Sie einen Cocktail von Antibiotika durch Auflösen in 1 L Wasser autoklaviert Folgendes: 1 g Ampicillin (1 g/L Finale), 1 g Neomycin (1 g/L Finale), 1 g Metronidazol (1 g/L Finale), 0,5 g Vancomycin (0,5 g/L Finale) und 8 Sachets (0,75 g) des künstlichen Süßstoff (60 g/L Finale). Schütteln Sie, bis aufgelöst. Shop bei 4 ° C.
2. Sammlung von Kotproben von Hocker, Ileum Inhalt und Ileum Wand
- Wiegen und label 2 mL autoklaviert Rohre für die Probenahme.
- Für die Sammlung von frischen Stuhlproben jede Maus in eine einschränkende und sammeln Sie fäkale Pellets direkt aus dem Anus in ein Sammelrohr zu.
Hinweis: Proben können auch erhalten werden, indem Mäuse in einem Käfig sauber autoklaviert und Stuhlproben mit sauberen sterilisierte Pinzette zu sammeln. - Einschläfern Sie die Mäuse mit CO2 Erstickung gefolgt von zervikale Dislokation.
- Legen Sie eine Maus-Karkasse mit den Bauch voll ausgesetzt und sprühen Sie Bauchbereich mit 70 % Ethanol.
- Mit sterilisierten Pinzette und Schere, machen Sie eine Quereinschnitt im Bauch, das Peritoneum verfügbar zu machen, ohne irgendwelche internen Gewebe zu beschädigen. Heben Sie das Peritoneum und einen Einschnitt, den Darm verfügbar zu machen.
- Entfernen Sie den Darm (aus Darm, Magen) mit Zange und Schere und legen Sie sie in eine sterile Petrischale.
- Verwenden Sie sorgfältig Zange um den Dünndarm (SI) zu necken abseits der mesenterialen Arterien und Fett. Erweitern Sie den Darm und legen Sie es auf eine saubere Lab wischen.
- Messen Sie mit einem Lineal und schneiden Sie 4 cm des distalen Ileum des SI (der nächste Teil, der Blinddarm). Schneiden Sie und entsorgen Sie die 1 cm des Darmes proximal zu den Blinddarm. Es wird ein 3 cm Teil Ileum verlassen, die verwendet wird, um die Darmbakterien zu sammeln.
- Halten Sie den Krummdarm Teil (3 cm) über eine 2 mL steriles Röhrchen. Sammeln Sie den Darminhalt durch Extrudieren direkt den Darm und Sammelreise der Probe in der Röhre. In diesem Beispiel werden Bakterien aus dem Ileum Inhalt haben.
- Vorbereiten einer 20 mL Spritze mit kalten Phosphat gepufferte Kochsalzlösung (PBS) und Ileum Teil (Verwerfen der durchströmten) spülen.
- Setzen Sie den Ileum Teil auf einem sauberen Papiertuch und längs mit einer Schere öffnen.
- Kratzen Sie die Innenseite der Krummdarm Wand mit einem Skalpell.
- Sammeln Sie alle Bakterien auf das Skalpell durch das Skalpell mit 1 mL PBS über eine saubere Zentrifugenröhrchen waschen. Drehen Sie mit 8.000 × g für 5 min pellet der Bakterien und den Überstand verwerfen. In diesem Beispiel enthält Bakterien aus dem Ileum-Wand.
Hinweis: Stuhlproben von Hocker, Ileum Inhalt und Wand können eingefroren und bei-80 ° C bis zum Gebrauch gespeichert. - Wiegen Sie die Rohre, die Proben aus dem Hocker und Ileum Inhalte enthält, und subtrahieren Sie das Gewicht aus der leere Rohre (aus Schritt 2.1) um das fäkale Gewicht in jeder Probe zu erhalten.
- Hocker, Ileum Inhalt und Ileum Wand Proben mit handelsüblichen Kits extrahieren Sie bakteriellen DNA. DNA-Proben bei-20 ° C bis zu seiner Verwendung zu speichern.
3. Quantifizierung der intestinalen Mikrobiota von qPCR
Hinweis: Dieses Verfahren umfasst die Generierung von Standard (Schritt 3.1) und die Methode für qPCR-Set-up für Standard- und fäkalen Proben (Schritt 3.2)
- Generierung eines Standards für qPCR
- Verwenden Sie Polymerase-Kettenreaktion (PCR), der 16 s rRNA-Gen von der genomic DNA extrahiert aus einer Bakterienkultur mit den Reagenzien13 und in Tabelle 1aufgeführten PCR-Bedingungen zu verstärken.
- Führen Sie das PCR-Produkt auf einem 1,5 % Agarosegel und reinigen Sie die DNA-Band mit einem im Handel erhältlichen DNA Extraktion Kit.
- Die gereinigte DNA Fragment mit einem Klonen kit (siehe die Tabelle der Materialien, verwenden Sie einen Vektor mit Antibiotikaresistenz-Marker und β-Galaktosidase (LacZ) gen Fusion für blau/weiße Kolonie Auswahl) verbinden und transformieren DH10 zuständigen E. Coli nach den Anweisungen des Herstellers. Transformation auf Ampicillin-Platte (100 µg/mL), X-gal (20 µg/mL) Luria Bertani (LB) Agarplatten (10 g/L Tryptone, 5 g/L Hefeextrakt, 10 g/L NaCl, 15 g/L Agar). Inkubation über Nacht (O/N) bei 37 ° C.
- Wählen Sie eine einzelne Positive (weiß) Kolonie von der Platte. In 5 mL LB Brühe enthält 100 µg/mL Ampicillin zu impfen Sie und inkubieren Sie O/N bei 37 ° C, bei 250 u/min schütteln.
- Isolieren Sie von der O/N Kultur und reinigen Sie das Plasmid mit einem kommerziellen Miniprep Kit nach Herstellerangaben.
Hinweis: Es ist wichtig, die Reihenfolge des Plasmid-Einsatzes in diesem Stadium, um sicherzustellen, dass das Plasmid nur eine Kopie der 16 s rRNA-Gen enthält und seiner Länge in Basenpaaren (bp) zu bestimmen. - Das Plasmid mit einem Restriktionsenzym schneidet das Plasmid nur einmal zu linearisieren.
- Reinigen Sie die linearisierte Plasmid mit einem kommerziell erhältlichen Kit.
- Bestimmen Sie die Konzentration des Plasmids durch Messung der Absorption bei 260 nm mit einem Spektralphotometer.
- Berechnen Sie die Anzahl der Kopien/µL Plasmid Probe mithilfe der folgenden Formel14:
Anzahl der Kopien = (Menge * 6.022 × 10-23) / (Länge * 1 × 109 * 650)
wo ist die DNA-Konzentration erwarb Schritt 3.1.8 (in ng/µL); und Länge beträgt die Gesamtlänge des Plasmids (mit Insert) in bp. Die Anzahl der Kopien werden als Kopien/µL bezogen werden.
Hinweis: Es gibt Online-Tools, die auf Grundlage der oben genannten Formel, die einfache Berechnung der Anzahl der Plasmid-Kopien zu ermöglichen. - Aliquoten und laden den Standard Bedarf bei-20 ° C bis zur Verwendung.
- qPCR Set-up für Standard- und Stuhlproben
- Die qPCR standard (aus Schritt 3.1.10), Stuhlproben DNA (aus Schritt 2.15) und qPCR Reagenzien (aus einem handelsüblichen Bausatz) auf Eis auftauen.
Hinweis: in diesem Beispiel verwendete qPCR-Reagenzien sind die SYBR Green qPCR Reaktion Mix und forward und reverse Primer (Tabelle 2). - Verdünnen Sie den Standard in der DNA-freie Sterilwasser in einem Bereich von 107 bis 102 Kopien/µL (z. B. 1/5 serielle Verdünnungen von 107 bis 6,4 x 102 Kopien/µL). Verdünnen Sie Kotprobe DNA, 1/2, 1/5, 1/10.
- Machen Sie eine master-Mix-Reaktion für die Gesamtzahl der Reaktionen plus 1 (Tabelle 2).
- Mischung 30 µL der master-Mix und 5 µL der Vorlage (Standard, Probe oder Wasser für die negativ-Kontrolle)
- Fügen Sie 10 µL dieser Mischung in jede Vertiefung in einer optischen 384-Well qPCR-Platte. Führen Sie jede Reaktion in dreifacher Ausfertigung.
- Versiegeln den qPCR-Ort, kurz Zentrifugieren und laden Sie die Platte auf die qPCR-Maschine programmiert mit folgenden Radsport Bedingungen: 95 ° C für 20 min gefolgt von 40 Zyklen von 95 ° C für 15 s und 60 ° C für 1 min.
- Erhalten Sie CT -Werte für Standard- und Proben.
- Generieren Sie eine Standardkurve durch Auftragen der CT -Werte für Ansprüche gegen den Logarithmus der Anzahl Kopie des Plasmids (wie in Schritt 3.1.9 berechnet). Durchführen Sie lineare Regression der Standardkurve.
- Berechnen Sie die 16 s rDNA Heftnummern für Stuhlproben durch Interpolation CT -Werte (in Schritt 3.2.2 erhaltenen) in der Standardkurve.
Hinweis: 16 s rDNA Heftnummern für jede Probe sollte korrigiert werden unter Berücksichtigung den Verdünnungsfaktor der Probe (wie in Schritt 3.2.2 vorbereitet), das Endvolumen Nukleinsäuren, die (in Schritt 2.15) eluiert waren und die Quantität der Kotprobe (berechnet in Schritt 2.14) zu erhalten gen-Kopien pro Gramm Kot.
- Die qPCR standard (aus Schritt 3.1.10), Stuhlproben DNA (aus Schritt 2.15) und qPCR Reagenzien (aus einem handelsüblichen Bausatz) auf Eis auftauen.
Ergebnisse
Hier bieten wir zwei alternative Protokolle für orale antibiotische Behandlung von Mäusen. Abbildung 1 zeigt den Prozentsatz des Körpergewichts (bezogen auf die ursprüngliche Grundlinie Gewicht für jedes Tier) bei Mäusen, die mit Antibiotika behandelt, entweder durch orale Magensonde (rot) oder im Trinkwasser (blau) für 10 aufeinanderfolgende Tage. Keine spürbaren Gewichtsverlust findet sich bei Mäusen, die Antibiotika erhalten von oralen Magensonde. Jedoch wenn Mäuse mit Antibiotika Ad Libitum im Trinkwasser behandelt werden, verlieren Gewicht (~ 10 %) innerhalb der ersten paar Tage Antibiotika Verwaltung, sondern normale Gewichtszunahme danach (Abbildung 1) zu erholen. Dennoch, etwa 5-10 % der Mäuse erhalten Antibiotika im Trinkwasser erreichen > 20 % Gewichtsverlust innerhalb der ersten Woche der Behandlung, die in diesem Fall sind sie eingeschläfert.
Die Quantifizierung von Bakterien in Stuhlproben erfordert den Einsatz von einer angemessenen Standardkurve die gewonnen wird durch das Protokoll Kopienzahl für den Standard (wie in Schritt 3.2.2 berechnet) im Vergleich zu den CT Werte aus qPCR (Schritt 3.2.7) auftragen. Abbildung 2A zeigt ein repräsentatives Beispiel aus einer Standardkurve entspricht die Standardkurve Leistungskriterien mit einem R-2 -Wert von 0.99827, einer Neigung von-3.09 und einen Wirkungsgrad ((-1 + 10^(-1/slope))*100) von 110 %. R2 Werte von 0,99 und PCR-Effizienz im Bereich von 90 bis 110 % werden bevorzugt. Innerhalb des linearen Bereichs ermöglicht die Analyse Regressionsgleichung Quantifizierung der 16 s rDNA Fülle innerhalb der Stuhlproben. Abbildung 2 b zeigt die Anzahl der 16 s rDNA Kopien im fäkalen Hocker, SI-Gehalt und SI Wand. In Abbildung 2 b werden Daten als 16 s rDNA Kopien/g der Kotprobe für fäkale Hocker und SI-Gehalt angezeigt. Für SI-Wand werden Daten als Gesamtzahl der 16 s rDNA Kopien erhalten von Bakterien erholte sich von den 3 cm von SI-Wand (wie die Menge des Ausgangsmaterials zu klein, um eine genaue Gewicht zu erhalten ist) dargestellt.
Um die Wirkung von Antibiotika auf die Dichte der Bakterien in Stuhlproben zu bewerten, Mäuse wurden behandelt mit Antibiotika durch orale Magensonde täglich für 10 Tage (Tage 1 bis 10) und Stuhlproben wurden vor (Tag 0), zu verschiedenen Zeitpunkten während der Behandlung mit Antibiotika () Tag 5 und 10), und 7 Tage nach Beendigung der antibiotischen Verwaltung (Tag 17; ( Abb. 3A). Wie in Abbildung 3 bgezeigt, induziert antibiotischer Behandlung einen starken Rückgang der Zahl von 16 s rDNA Kopien/g Kot am Tag 5 und 10, während die Dichte der Bakterien im Kot erholte sich auf ein normales Niveau (vergleichbar mit Vorbehandlung) 1 Woche nach erkannt Antibiotika-Verwaltung wurde (Tag 17) gestoppt.
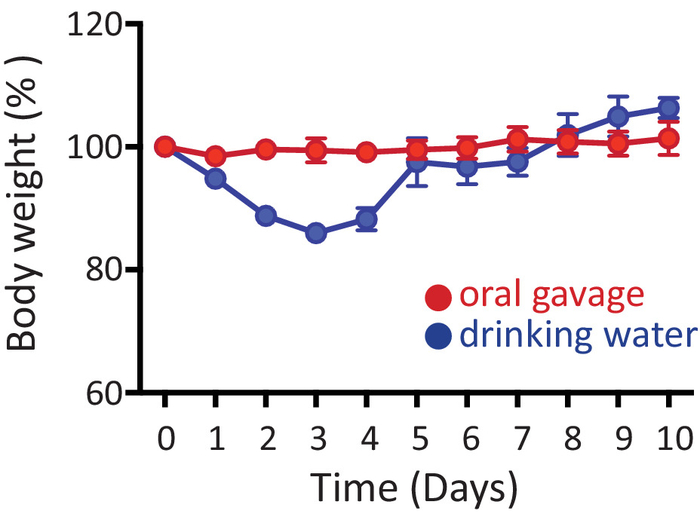
Abbildung 1: Verabreichung von Antibiotika. Mäuse erhalten Antibiotika, orale Magensonde (rot) oder im Trinkwasser (blau) für 10 aufeinanderfolgende Tage. Gewichte von Mäusen während der gesamten Dauer der Experimente im Vergleich zu ursprünglichen Gewicht vor der antibiotische Verabreichung (Tag 0) zeigt. Daten sind als Mittelwert ± SEM gezeigt Klicken Sie bitte hier, um eine größere Version dieser Figur.
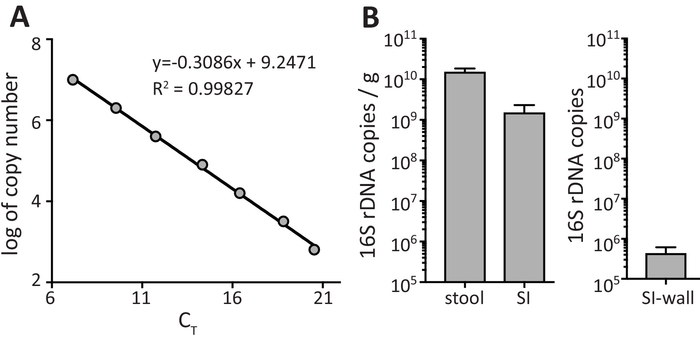
Abbildung 2: 16 s rRNA Gens qPCR Verstärkung des Standards und Stuhlproben. (A) lineare Regression der Standardkurve Standardkurve Deskriptoren. (B) Berechnung der gen Häufigkeiten von Stuhlproben. Daten sind als Mittelwert ± SEM gezeigt Klicken Sie bitte hier, um eine größere Version dieser Figur.
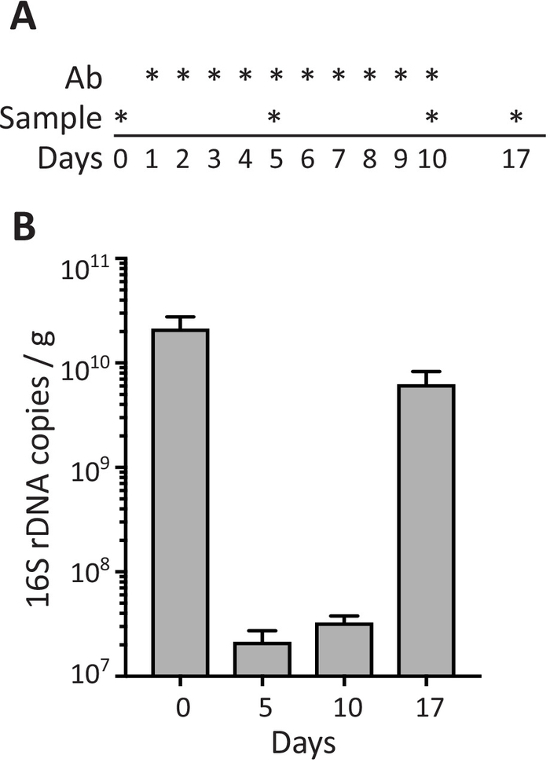
Abbildung 3: fäkalen Bakterien während der Behandlung mit Antibiotika. (A) schematische Darstellung der Zeitplan für die antibiotische Verwaltung durch orale Magensonde (Ab) und Entnahme von Proben mit dargestellt *. (B) 16 s rDNA Kopien pro Gramm Kot in Stuhlproben von Mäusen, die an den angegebenen Tagen gesammelt. Daten sind als Mittelwert ± SEM gezeigt Klicken Sie bitte hier, um eine größere Version dieser Figur.
| Reagenz | Volumen | |
| DNA | 8 ΜL | |
| Puffer-10 X | 2 ΜL | |
| dNTPs (10mM) | 0.4 ΜL | |
| Eubakterien - F Grundierung (10 mM) | 1 ΜL | |
| Eubakterien - R Grundierung (10 mM) | 1 ΜL | |
| Taq Polymerase | 0,2 ΜL | |
| H2O | 7.4 ΜL | |
| Gesamtvolumen | 20 ΜL | |
| Primer-Sequenzen | ||
| Eubakterien - F-Grundierung | 5' ACTCCTACGGGAGGCAGCAGT 3' | |
| Eubakterien - R Grundierung | 5' ATTACCGCGGCTGCTGGC 3' | |
| Radfahren-Bedingungen | ||
| Temperatur | Zeit | Zyklen |
| 94 ° C | 5 min | 1 x |
| 94 ° C | 30 s | 30 x |
| 60 ° C | 30 s | 30 x |
| 72 ° C | 1 min | 30 x |
| 72 ° C | 5 min | 1 x |
| 4 ° C | ∞ | 1 x |
Tabelle 1: PCR Reagenzien und Bedingungen. Diese Tabelle enthält die Reagenzien und PCR-Radsport-Zustände, 16 s rRNA-Gen von Bakterienkultur zur Erzeugung von einem Standard in der qPCR-Assays verwenden zu verstärken. Primer-Sequenzen wurden ursprünglich von Kruglov Et Al. veröffentlicht. 13.
| Reagenz | Volumen |
| SYBR Green-master-Mix (2 X) | 17.5 ΜL |
| Eubakterien - F Grundierung (10 mM) | 0,7 ΜL |
| Eubakterien - R Grundierung (10 mM) | 0,7 ΜL |
| H2O | 11.1 ΜL |
Tabelle 2: qPCR-master-Mix. Die Volumes angezeigt (Endvolumen 35 µL) sind für ein einzelnes Sample in dreifacher Ausfertigung (10 µL) auf einer qPCR 384-Well-Platte (Buchhaltung für extra für Pipettier Fehler 5 µL) ausgeführt werden. Der Betrag kann je nach Anzahl der zu analysierenden Proben hochskaliert.
Diskussion
Hier bieten wir experimentelle Protokolle für die orale Verabreichung von Antibiotika für Mäuse und Quantifizierung von fäkalen Bakterien durch qPCR. Die Kombination von Antibiotika verwendet in diesem Protokoll (mit Ampicillin, Gentamicin, Neomycin, Metronidazol und Vancomycin) Ziele grampositive und gramnegative Bakterien, bakterizide Aktivität gegen ein breites Spektrum an Bakterien bietet. Orale Magensonde und Verabreichung von Antibiotika im Trinkwasser stark verringern fäkalen Bakterien laden5,6,12. Darüber hinaus haben beide Behandlungen eine tiefgreifende Wirkung auf den Phänotyp der Mäuse, wie sie typisch für keimfreie Mäuse einschließlich reduzierter Milz Größe und vergrößerte Blinddarm mehrere Merkmale zu entwickeln. Die Auswahl eines bestimmten Verfahrens zur antibiotischen Verwaltung kann möglicherweise auf die Länge des Experiments abhängen, wie die orale Magensonde Methode tägliche Verabreichung von Antibiotika erfordert, als arbeitsintensiver und möglicherweise mehr Unbehagen an der Tiere auf lange Sicht.
Für die Verabreichung von Antibiotika im Trinkwasser muss Vorsicht mit dem Zusatz des Süßstoffes, die antibiotische Mischung genommen werden, da dies ein entscheidender Faktor, um Mäuse vor Austrocknung zu halten. Mehrere Gruppen haben gezeigt, wie Verabreichung von Antibiotika im Trinkwasser (ohne Zusatz von Süßstoff) führt zu sehr starken und schnellen Gewichtsverlust mit alle Mäuse verlieren mehr als 20 % des ursprünglichen Körpergewichts in den ersten Tagen des Experiment5 , 6. in unserem Protokoll die Verwendung von Süßstoff Saccharin-basierte schien ausreichen, um den Antibiotika Geschmack im Wasser und Mäuse abgenommen in den ersten Tagen nach der Verabreichung der Antibiotika zu maskieren, aber ihr Gewicht hat sich schnell erholt nach diesem ( Abbildung 1). Dennoch in unseren Experimenten ca. 5-10 % der Mäuse noch erreichen den menschlichen Endpunkt von > 20 % Verlust der Grundlinie Körpergewicht und musste eingeschläfert werden. Wir haben auch getestet, Sucralose-basierte Süßungsmittel, die völlig versäumt, Mäuse Austrocknung zu verhindern (100 % der Mäuse verloren > 20 % des Gewichts) während andere Autoren ähnliche Fehler für Süßungsmittel Aspartam-basierten5,6veröffentlicht haben. Hinzu kommt, sollten Alter, genetische Hintergrund und allgemeine Gesundheitszustand der Mäuse für die Experimente verwendet berücksichtigt werden da sie Gewichtsverlust und Wohlbefinden der Tiere während der Behandlung mit Antibiotika beeinflussen können. Daher sollten sorgfältige Überwachung der Mäuse Gewicht und allgemeine Gesundheitszustand täglich während der ersten zwei Wochen der oralen Verabreichung der Antibiotika erfolgen.
qPCR Methoden bieten einen schnellen und kostengünstigen Ansatz zur Quantifizierung der 16 s rRNA in Stuhlproben. Jedoch einige Einschränkungen bezüglich dieser Technik einschließlich betrachtet werden: ich) die Voraussetzung für eine zuverlässige Qualitätsstandard; (II) die Design und Effizienz der qPCR Primer; (III) die Tatsache, dass Mikroorganismen verschiedene Heftnummern der 16 s rRNA-Gen haben können, können somit gen Kopien nicht direkt Zelle zählt15gleich. Dennoch, qPCR ist eine robuste und empfindliche Methode ermöglicht schnelle Analyse der Stuhlproben. Diese Methode ist besonders nützlich, um schnell die Wirkung der verschiedenen Behandlungen (einschließlich Antibiotika) in fäkale bakterielle Belastungen wie detaillierte hier überprüfen. Darüber hinaus obwohl wir ein Protokoll für die Quantifizierung von insgesamt 16 s bieten rRNA, diese Methode kann leicht angepasst werden (indem Sie entwerfen spezifische Primer16) ermöglicht die Identifizierung der einzelnen bakterielle Taxa, wodurch sowohl quantitative und qualitative Informationen über Microbiome Größe und Zusammensetzung.
Zusammenfassend lässt sich sagen haben wir zwei Protokolle für orale antibiotische Behandlung von Mäusen und eine qPCR basierende Methode, Antibiotika-induzierte Veränderungen im fäkalen Bakterien zu quantifizieren bereitgestellt. Obgleich diese Protokolle weiter optimiert und können in mit anderen Ansätzen, nach experimentellen Bedürfnissen Kombination, können sie als schnelle, kostengünstige und zuverlässige Werkzeuge die murine intestinale Mikrobiota manipulieren und um die Auswirkungen zu studieren dienen. der antibiotischen Behandlung bei intestinalen Homöostase und Krankheit.
Offenlegungen
Die Autoren haben nichts preisgeben.
Danksagungen
Diese Arbeit wurde von der UK Medical Research Council (Finanzhilfe für P.B MR/L008157/1), finanziert. R.j. wurde unterstützt durch eine Marie Curie Intra Fellowship (H2020-MSCA-IF-2015-703639); P.M.B. wurde durch eine Zugehörigkeit von UK Medical Research Council und Kings College London Doctoral Training Partnerschaft in den biomedizinischen Wissenschaften (MR/N013700/1) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Ampicillin sodium salt | Sigma-Aldrich (Merck) | A9518 | |
| Neomycnin trisulfate salt hydrate | Sigma-Aldrich (Merck) | N1876 | |
| Metronidazole | Sigma-Aldrich (Merck) | M3761 | |
| Vancomycin hydrochloride | Sigma-Aldrich (Merck) | V2002 | |
| Gentamicin sulfate salt | Sigma-Aldrich (Merck) | G3632 | |
| Tryptone | Sigma-Aldrich (Merck) | T7293 | |
| Yeast Extract | Sigma-Aldrich (Merck) | Y1625 | |
| NaCL | Sigma-Aldrich (Merck) | S7653 | |
| Sweetener Sweet'n Low | Sweet'N Low | Available in the UK from Amazon.co.uk | |
| X-Gal (5-brom-4-chloro-3-indoyl B-D-galactopyranoside) | Fisher scientific | 10234923 | |
| Phosphate Buffered Saline | Thermo Fisher Scientific (Gibco) | 10010023 | |
| Ultrapure Agarose | Thermo Fisher Scientific (Invitrogen) | 16500500 | |
| RT-PCR grade water | Thermo Fisher Scientific (Invitrogen) | AM9935 | |
| Phusion High-Fidelity DNA Polymerase | New England BioLabs | M0530 | |
| Deoxynucleotide (dNTP) Solution Mix | New England BioLabs | N0447 | |
| iTaq Universal SYBR Green Supermix | Bio-Rad | 1725124 | with ROX |
| TOPO TA cloningTM for sequencing | Thermo Fisher Scientific (Invitrogen) | 450030 | |
| QIAamp fast DNA Stool mini kit | Qiagen | 51604 | |
| QIAprep spin Miniprep kit | Qiagen | 27106 | |
| QIAquick gel extraction kit | Qiagen | 28704 | |
| Syringe filter 0.45 µm | Fisher scientific | 10460031 | |
| Swann-MortonTM Carbon steel sterile scalpel blades | Fisher scientific | 11792724 | |
| Syringe (1 mL) | BD Plastipak | 303172 | |
| Syringe (20 mL) | BD Plastipak | 300613 | |
| 1.5 mL Crystal clear microcentriguge tube | StarLab | E1415-1500 | |
| 2 mL Ultra high recovery microcentrifuge tube | StarLab | I1420-2600 | |
| Oral dosing needles 20 G x 38 mm curved (pk/3) | Vet-Tech | DE008A | |
| Sterilin petri dish 50 mm | Scientific Laboratory Supplies | PET2020 | |
| Absolute qPCR plate seals | Thermo Fisher Scientific | AB1170 | |
| MicroAmpTM optical 384-well plate | Thermo Fisher Scientific (Applied Biosystems) | 4309849 | |
| ViiA7TM 7 real-time PCR system with 384-well block | Thermo Fisher Scientific (Applied Biosystems) | 4453536 | |
| Spectrophotometer (Nanodrop 1000) | Thermo Fisher Scientific | ND-1000 | |
| Labnet Prism microcentrifuge | Labnet | C2500 | |
| MultiGene Optimax Thermal cycler | Labnet | TC9610 |
Referenzen
- Belkaid, Y., Hand, T. W. Role of the microbiota in immunity and inflammation. Cell. 157 (1), 121-141 (2014).
- Hooper, L. V., Littman, D. R., Macpherson, A. J. Interactions between the microbiota and the immune system. Science. 336 (6086), 1268-1273 (2012).
- Ubeda, C., Pamer, E. G. Antibiotics, microbiota, and immune defense. Trends in Immunology. 33 (9), 459-466 (2012).
- Dethlefsen, L., Huse, S., Sogin, M. L., Relman, D. A. The pervasive effects of an antibiotic on the human gut microbiota, as revealed by deep 16S rRNA sequencing. PLoS Biology. 6 (11), e280 (2008).
- Reikvam, D. H., et al. Depletion of murine intestinal microbiota: effects on gut mucosa and epithelial gene expression. PLoS One. 6 (3), e17996 (2011).
- Hill, D. A., et al. Metagenomic analyses reveal antibiotic-induced temporal and spatial changes in intestinal microbiota with associated alterations in immune cell homeostasis. Mucosal Immunology. 3 (2), 148-158 (2010).
- Fraher, M. H., O'Toole, P. W., Quigley, E. M. Techniques used to characterize the gut microbiota: a guide for the clinician. Nature Reviews Gastroenterology & Hepatology. 9 (6), 312-322 (2012).
- Eckburg, P. B., et al. Diversity of the human intestinal microbial flora. Science. 308 (5728), 1635-1638 (2005).
- Sokol, H., et al. Low counts of Fecalibacterium prausnitzii in colitis microbiota. Inflammatiry Bowel Diseases. 15 (8), 1183-1189 (2009).
- Bartosch, S., Fite, A., Macfarlane, G. T., McMurdo, M. E. Characterization of bacterial communities in feces from healthy elderly volunteers and hospitalized elderly patients by using real-time PCR and effects of antibiotic treatment on the fecal microbiota. Applied Environmental Microbiology. 70 (6), 3575-3581 (2004).
- Ubeda, C., et al. Vancomycin-resistant Enterococcus domination of intestinal microbiota is enabled by antibiotic treatment in mice and precedes bloodstream invasion in humans. Journal of Clinical Investigation. 120 (12), 4332-4341 (2010).
- Saez de Guinoa, J., et al. CD1d-mediated lipid presentation by CD11c(+) cells regulates intestinal homeostasis. EMBO Journal. 37 (5), (2018).
- Kruglov, A. A., et al. Nonredundant function of soluble LTalpha3 produced by innate lymphoid cells in intestinal homeostasis. Science. 342 (6163), 1243-1246 (2013).
- Wilhelm, J., Pingoud, A., Hahn, M. Real-time PCR-based method for the estimation of genome sizes. Nucleic Acids Research. 31 (10), e56 (2003).
- Kembel, S. W., Wu, M., Eisen, J. A., Green, J. L. Incorporating 16S gene copy number information improves estimates of microbial diversity and abundance. PLoS Computational Biology. 8 (10), e1002743 (2012).
- Yang, Y. W., et al. Use of 16S rRNA Gene-Targeted Group-Specific Primers for Real-Time PCR Analysis of Predominant Bacteria in Mouse Feces. Applied Environmental Microbiology. 81 (19), 6749-6756 (2015).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten