Method Article
Polimerase quantitativos baseados na reação em cadeia análises de murino Microbiota Intestinal após o tratamento antibiótico Oral
Neste Artigo
Resumo
Aqui nós fornecemos protocolos detalhados para a administração oral de antibióticos para ratos, coleta de amostras fecais, extração de DNA e quantificação de bactérias fecais por qPCR.
Resumo
A microbiota do intestino tem uma influência central na saúde humana. Disbiose microbiana é associado com muitos immunopathologies comuns, tais como artrite, asma e doença inflamatória intestinal. Assim, compreender os mecanismos subjacentes a interferência do sistema imunológico microbiota é de importância crucial. Administração de antibióticos, enquanto ajudava o afastamento do agente patogénico, também induz mudanças drásticas no tamanho e composição das comunidades bacterianas intestinais que podem ter um impacto sobre a saúde humana. Tratamento antibiótico em camundongos recapitula o impacto e a longo prazo mudanças na microbiota humana de antibióticos pacientes tratados e permite que a investigação das ligações entre alterações em comunidades microbianas e função imune celular mecanicistas. Enquanto vários métodos de tratamento antibiótico de ratos têm sido descritos, algumas delas induzem severa desidratação e perda de peso, complicando a interpretação dos dados. Aqui, nós fornecemos dois protocolos para administração de antibiótica oral, que pode ser usada para tratamento a longo prazo dos ratos sem induzir a maior perda de peso. Estes protocolos de fazem uso de uma combinação de antibióticos que visam tanto Gram-positiva e gram-negativa bactérias e podem ser fornecidos ou ad libitum na água potável ou por gavagem oral. Além disso, descrevemos um método para a quantificação da densidade microbiana em amostras fecais por qPCR, que pode ser usado para validar a eficácia do tratamento com o antibiótico. A combinação dessas abordagens fornece uma metodologia confiável para a manipulação da microbiota intestinal e o estudo dos efeitos do tratamento com antibiótico em camundongos.
Introdução
A mucosa gastrointestinal dos mamíferos é um ambiente único, colonizado por uma mistura altamente complexa de microorganismos que estabelecem uma relação mutualista com o host. O sistema de defesa da mucosa intestinal é composto por uma camada epitelial e uma infinidade de células do sistema imunológico que restringem os comensais dentro do intestino, preservando o seu número e diversidade. Por outro lado, organismos comensais são necessários para o desenvolvimento de um sistema imune completamente funcional. Enquanto as interações entre host e bactérias comensais são normalmente benéficas, torna-se cada vez mais claro que esse diagrama de sistema imunológico microbiota desregulação pode favorecer o desenvolvimento de doenças inflamatórias crônicas, tal asinflammatory do intestino artrite asma ou doença1,2.
A microbiota do intestino pode ser alterada por vários fatores, mas talvez as alterações mais drásticas são induzidas por tratamento com antibióticos que severamente altera o tamanho e a composição de comunidades bacterianas3,4. Enquanto os benefícios de antibióticos para tratar infecções são inquestionáveis, as alterações da microbiota induzidas pela exposição aos antibióticos nos seres humanos também podem modificar as defesas imunitárias que podem levar a efeitos nocivos sobre a saúde. Por exemplo, o tratamento com antibióticos nos seres humanos tem sido associado a um risco aumentado de Clostridium difficile-induzida por certos tipos de câncer3, asma e diarreia. Tratamento antibiótico em camundongos recapitula o impacto e a longo prazo alterações encontradas em comunidades de intestino de pacientes tratados com antibióticos e permitiu a investigação das ligações entre alterações em comunidades microbianas e função imune celular mecanicistas. No entanto, vários relatos têm demonstrado que a administração de antibióticos sobre a água potável ad libitum resulta em perda de peso muito perceptível como ratos abster-se de água potável, presumivelmente devido ao seu mau gosto5,6. Assim, nestes modelos a desidratação grave concomitante à administração de antibióticos oral pode complicar a interpretação de experimentos com o objetivo de identificar o efeito do tratamento com antibiótico na função imune celular.
Várias abordagens podem ser usadas para explorar o tamanho e a composição de comunidades microbianas no compartimento intestinal7. Próxima geração tecnologias de sequenciamento apresentaram dados inestimáveis sobre esta questão8, no entanto, estes métodos são relativamente caros e exigem análises de bioinformatic perito para interpretação dos dados. Por outro lado, métodos tradicionais de cultura microbiológica permitam a detecção das espécies bacterianas, mas eles têm baixa sensibilidade e uma grande fração de bactérias comensais (particularmente anaeróbios) são muito difíceis ou impossíveis de se cultivar com métodos atualmente disponíveis8. Técnicas de reação em cadeia (qPCR) polimerase quantitativos estão sendo usadas cada vez mais para quantificação e identificação de espécies bacterianas fecais, que proporcionam uma rápida e confiável independente de cultura medida da carga microbiana total. Por conseguinte, qPCR métodos provaram útil para estudar as mudanças na microbiota associada com a idade ou com a progressão de várias doenças, incluindo a doença de entranhas inflamatório9,10. Dessa forma, qPCR métodos fornecem uma abordagem rápida e econômica para validar o efeito de diferentes tratamentos (incluindo antibióticos) em cargas de bactérias fecais e microbiota composição10,11,12.
Aqui, apresentamos um relato detalhado passo a passo de dois protocolos distintos para administração de antibiótico oral para ratos, coleta de amostra fecal, extração de DNA, elaboração de normas e quantificação de bactérias em amostras fecais por qPCR. Esses protocolos fornecem um método confiável para manipular a microbiota intestinal em camundongos e para estudar os efeitos do tratamento com antibiótico na homeostasia intestinal e doença.
Protocolo
Foram realizados experimentos descritos aqui, usando de 6 a 8 semanas velhos ratos de tipo selvagem (C57BL6/J) mantidos em uma instalação do patógeno específico livre (SPF). Todos os experimentos com animais foram aprovados pelo College de Londres do rei e o Francis Crick Instituto bem-estar Animal e corpo de revisão ética e o Home Office do Reino Unido. Antes de começar qualquer procedimento animal, certifique-se de que as permissões apropriadas são obtidas através da instituição/organização local.
1. a administração de antibióticos
Nota: Dois métodos alternativos de tratamento antibiótico são fornecidos: gavagem oral (etapa 1.1) e administração de antibióticos através da água potável (etapa 1.2).
- Gavagem oral
- Preparar ações de antibióticos individuais, dissolvendo-os em água esterilizada às seguintes concentrações: ampicilina 100 mg/ml, gentamicina a 100 mg/mL, neomicina 100mg/ml, metronidazol a 10 mg/mL e vancomicina em 100 mg/mL. Filtro de esterilizar usando um filtro de 0,45 µm. Alíquota e loja a-20 ° C.
- Prepare um coquetel de antibióticos misturando os estoques preparados acima. Para um volume de 1 mL, misture 50 µ l de ampicilina (5 mg/mL final), 50 µ l de gentamicina (5 mg/mL final), 50 µ l de neomicina (5 mg/mL final), 500 µ l de metronidazol (5 mg/mL final), 25 µ l de vancomicina (2,5 mg/mL final) e 325 µ l de água. Prepare o coquetel fresco antes da utilização.
- Carregar a antibiótica mistura em uma seringa de 1ml estéril e fixar uma agulha de gavagem de calibre adequado (20 G para ratos de 15 – 20 g) ao eliminar quaisquer bolhas.
- Ratos de gavage com 200 µ l de mistura de antibiótica.
- Pegue a pele por cima do ombro do mouse firmemente, esticar a cabeça e o pescoço para endireitar o esôfago. Direcionar a bola-ponta da agulha alimentação ao longo do teto da boca e para trás da parede posterior da faringe. Em seguida, delicadamente passá-lo para baixo para o esôfago e injecte a solução de 200 μL.
- Administre a antibiótica cocktail vez diariamente para a duração do experimento.
- Antibióticos na água potável
Cuidado: Monitorize cuidadosamente peso rato diariamente para as duas primeiras semanas de administração de antibiótica na água potável.- Preparar um coquetel de antibióticos dissolvendo-se o seguinte em 1 litro de água esterilizada: 1 g de ampicilina (1G/L final), 1 g de neomicina (1 g/L final), 1 g de metronidazol (1G/L final), 0,5 g de vancomicina (0,5 g/L final) e 8 sachês (0,75 g cada) de artificial adoçante (60 g/L final). Agite até dissolver. Loja a 4 ° C.
Nota: O adoçante é adicionado para esconder o sabor de antibióticos e prevenir a desidratação de ratos. Enquanto várias marcas de adoçantes podem funcionar, a concentração final necessária pode ser diferente para cada marca específica. - Preencher o coquetel de antibióticos dentro de uma garrafa de água (~ 100 mL/frasco) e coloque a garrafa em uma gaiola do rato.
Nota: Usar garrafas marrons ou cobrir as garrafas com papel alumínio para proteger os antibióticos de luz. - Substitua o coquetel de antibióticos com estoque fresco duas vezes por semana durante a duração do experimento.
- Preparar um coquetel de antibióticos dissolvendo-se o seguinte em 1 litro de água esterilizada: 1 g de ampicilina (1G/L final), 1 g de neomicina (1 g/L final), 1 g de metronidazol (1G/L final), 0,5 g de vancomicina (0,5 g/L final) e 8 sachês (0,75 g cada) de artificial adoçante (60 g/L final). Agite até dissolver. Loja a 4 ° C.
2. coleta de amostras fecais de fezes, conteúdo do íleo e parede do íleo
- Pesar e etiquetar mL 2 tubos esterilizados para coleta de amostra.
- Recolha de amostras de fezes frescas, coloque cada rato em uma retenção e recolher pelotas fecais diretamente do ânus em um tubo de coleta.
Nota: Amostras também podem ser obtidas colocando ratos em uma gaiola limpa autoclavada e coletando amostras de fezes com Pinças esterilizadas limpas. - Eutanásia em ratos com CO2 asfixia seguida por deslocamento cervical.
- Colocar uma carcaça do mouse com o abdômen completamente exposto e pulverizar a área abdominal com etanol a 70%.
- Usando uma tesoura e pinça esterilizada, fazer uma incisão transversa no abdômen para expor o peritônio sem danificar qualquer tecidos internos. Levante o peritônio e fazer uma incisão para expor os intestinos.
- Remover os intestinos, (do cólon, estômago), com a pinça e a tesoura e coloque-o em uma placa de Petri estéril.
- Com cuidado, use fórceps para arreliar o intestino delgado (SI) longe das artérias mesentéricas e gordura. Estender o intestino e colocá-lo em uma limpeza de laboratório limpo.
- Com uma régua, meça e corte 4 cm do íleo distal de SI (a parte mais próxima ao ceco). Corte e elimine a 1 cm do intestino proximal ao ceco. Vai haver uma porção de 3 cm de íleo deixada que será usado para coletar as bactérias intestinais.
- Mantenha a porção do íleo (3 cm) sobre um tubo estéril de 2 mL. Recolha o conteúdo intestinal por extrusão diretamente do intestino e colecionando a amostra no tubo. Esta amostra terá bactérias do conteúdo do íleo.
- Preparar uma seringa de 20 mL com soro fisiológico frio tamponado de fosfato (PBS) e liberar a porção do íleo (descarte o escoamento).
- Coloque a porção de íleo em uma toalha de papel limpa e abri-lo no sentido longitudinal, com uma tesoura.
- Raspe o interior da parede íleo com um bisturi.
- Recolha todas as bactérias sobre o bisturi lavando o bisturi com 1 mL de PBS sobre um tubo de centrífuga limpo. Girar a 8.000 × g por 5 min para as bactérias de pelotas e descartar o sobrenadante. Este exemplo irá conter bactérias da parede do íleo.
Nota: Amostras fecais de fezes, conteúdo do íleo e parede podem ser congeladas e armazenadas a-80 ° C até o uso. - Pesar os tubos contendo as amostras de fezes e íleo de conteúdo e subtrair o peso dos tubos de vazio (da etapa 2.1) para obter o peso fecal em cada amostra.
- Extrai ADN bacteriano de fezes, conteúdo do íleo e amostras de parede do íleo usando kits comercialmente disponíveis. Armazenar as amostras de DNA a-20 º C até o uso.
3. quantificação da Microbiota Intestinal por qPCR
Nota: Este procedimento inclui a geração de um padrão (passo 3.1) e o método de afinação de qPCR para amostras fecais e padrão (passo 3.2)
- Geração de um padrão para qPCR
- Use a reação em cadeia da polimerase (PCR) para amplificar o DNA genômico extraído de uma cultura bacteriana usando os reagentes13 e as condições PCR detalhadas no quadro 1do gene 16S rRNA.
- Executar o produto do PCR em um gel de agarose 1,5% e purificar a banda de DNA usando um kit de extração de DNA disponível comercialmente.
- Ligate fragmento do DNA purificado usando uma clonagem kit (veja a Tabela de materiais, usar um vetor contendo marcadores de resistência aos antibióticos e fusão de gene β-galactosidase (LacZ) para a seleção de colônia azul/branco) e transformar DH10 competentes Escherichia coli seguindo as instruções do fabricante. Placa de transformação para ampicilina (100 µ g/mL), placas de ágar de Luria Bertani (LB) X-galão (20 µ g/mL) (10 g/L triptona, 5g/L de extrato de levedura, 10 g/L de NaCl, 15 g/L de ágar). Incubar durante a noite (O/N) a 37 ° C.
- Selecione uma colônia única positivo (branco) da placa. -Inocular em caldo LB de 5 mL contendo 100 mg/mL ampicilina e incubar O/N a 37 ° C, agitação a 250 rpm.
- Da cultura O/N, isolar e purificar o plasmídeo usando um kit miniprep comercial de acordo com as instruções do fabricante.
Nota: É importante para a inserção do plasmídeo de sequências nesta fase, para garantir que o plasmídeo contém somente uma cópia do gene 16S rRNA e determinar seu comprimento em pares de bases (bp). - Linearizar o plasmídeo com uma enzima de restrição que corta o plasmídeo de uma só vez.
- Purifica o plasmídeo linear usando um kit disponível comercialmente.
- Determinar a concentração do plasmídeo por medir a absorvância a 260 nm, utilizando um espectrofotômetro.
- Calcule o número de cópias do plasmídeo / µ l de amostra usando a seguinte fórmula14:
número de cópias = (quantidade * 6.022 × 1023) / (comprimento * 1 × 109 * 650)
onde a quantidade é a concentração de DNA obtida na etapa 3.1.8 (em µ ng/l); e o comprimento é o comprimento total do plasmídeo (com a inserção) no bp. O número de cópias serão obtidos como cópias / µ l.
Nota: Existem ferramentas on-line com base na fórmula acima que permitem o fácil cálculo do número de cópias do plasmídeo. - Alíquota e armazenar o padrão conforme necessário a-20 º C até o uso.
- qPCR set-up para Standard e amostras fecais
- Descongele o qPCR padrão (da etapa 3.1.10), DNA de amostras fecais (da etapa 2.15) e qPCR reagentes (a partir de um kit comercialmente disponível) no gelo.
Nota: qPCR reagentes usados neste exemplo incluem a mistura de reação SYBR Green qPCR e primers para diante e reversos (tabela 2). - Diluir o padrão em água estéril livre de DNA em uma escala de 107 a 102 cópias / µ l (diluições em série, por exemplo, 1/5 de 107 a 6,4 x 102 cópias / µ l). Diluir a amostra fecal DNA para 1/2, 1/5, 1/10.
- Fazer uma reação de mistura de mestre para o número total de reações mais 1 (tabela 2).
- Mix de 30 µ l de mistura principal e 5 µ l de modelo (norma, amostra ou água para controle negativo)
- Adicione 10 µ l desta mistura para cada poço em um prato de qPCR 384-bem óptico. Realize cada reação em triplicado.
- Selar o lugar de qPCR, centrifugue brevemente e carregar a placa na máquina qPCR programada com as seguintes condições de ciclismo: 95 ° C por 20 min, seguido de 40 ciclos de 95 ° C por 15 s e 60 ° C por 1 min.
- Obter valores CT padrão e amostras.
- Gera uma curva de calibração plotando os valores de CT para os padrões versus o logaritmo do número de cópia do plasmídeo (como calculado na etapa 3.1.9). Realize a regressão linear da curva padrão.
- Calcule o número de cópias de rDNA 16S para amostras fecais por interpolação valores CT (obtidos na etapa 3.2.2) na curva padrão.
Nota: 16S rDNA, número de cópias para cada amostra deve ser corrigido Considerando o factor de diluição da amostra (preparada no passo 3.2.2), os volume final os ácidos nucleicos que foram eluídos (na etapa 2.15) e a quantidade de amostra fecal (calculado na etapa 2.14) para obter cópias do gene por grama de fezes.
- Descongele o qPCR padrão (da etapa 3.1.10), DNA de amostras fecais (da etapa 2.15) e qPCR reagentes (a partir de um kit comercialmente disponível) no gelo.
Resultados
Aqui nós fornecemos dois protocolos alternativos de tratamento antibiótico oral de ratos. A Figura 1 mostra o percentual de peso corporal (relacionado com peso de linha de base original para cada animal) em ratos tratados com antibióticos por gavagem oral (vermelha) ou na água potável (azul) durante 10 dias consecutivos. Nenhum peso-perda perceptível é encontrada nos ratos que recebem antibióticos por gavagem oral. No entanto, quando os ratos são tratados com antibióticos ad libitum na água potável, perder peso (~ 10%) dentro os primeiros dias da administração de antibióticos, mas recuperar o ganho de peso normal depois disso (Figura 1). No entanto, por volta das 5-10% dos ratos recebendo antibióticos na água potável pode chegar > 20% perda de peso na primeira semana de tratamento, caso em que eles são sacrificados.
A quantificação das bactérias em amostras fecais requer o uso de uma curva padrão adequado que é obtido traçando o log do número de cópia para o padrão (como calculado no passo 3.2.2) versus os valores de CT obtidos qPCR (etapa 3.2.7). Figura 2A mostra um exemplo representativo de uma curva padrão que satisfaça os critérios de desempenho de curva padrão com um valor de2 R de 0.99827, uma inclinação de-3.09 e uma eficiência ((-1 + 10^(-1/slope))*100) de 110%. R2 valores de 0,99 e eficiências PCR dentro do intervalo de 90 a 110% são preferidos. Dentro da escala linear, a equação de regressão análise permite a quantificação da 16S rDNA abundância dentro de amostras fecais. Figura 2B mostra o número de cópias de 16S rDNA em fezes fecal, conteúdo de SI e da parede de SI. Na Figura 2B dados são mostrados como 16S rDNA cópias/g de amostra fecal de fezes fecal e conteúdo de SI. Para a parede de SI, os dados são apresentados como o número total de 16S rDNA cópias obtidas a partir de bactérias recuperadas a 3 cm da parede de SI (como a quantidade de material começar é muito pequena para obter um peso exato).
Para avaliar o efeito de antibióticos na densidade de bactérias em amostras fecais, os ratos foram tratados com antibióticos por gavagem oral diariamente por 10 dias (dias 1 a 10) e amostras de fezes foram coletadas antes (dia 0), nos pontos de tempo diferentes durante o tratamento com antibióticos ( dias 5 e 10) e 7 dias depois de parar a administração de antibióticos (dia 17; Figura 3A). Como mostrado na Figura 3B, tratamento antibiótico induz uma forte diminuição no número de cópias de 16S rDNA/g de fezes detectado nos dias 5 e 10, enquanto a densidade de bactérias nas fezes recuperadas para níveis normais (comparáveis ao pré-tratamento) 1 semana após Administração de antibióticos foi interrompida (dia 17).
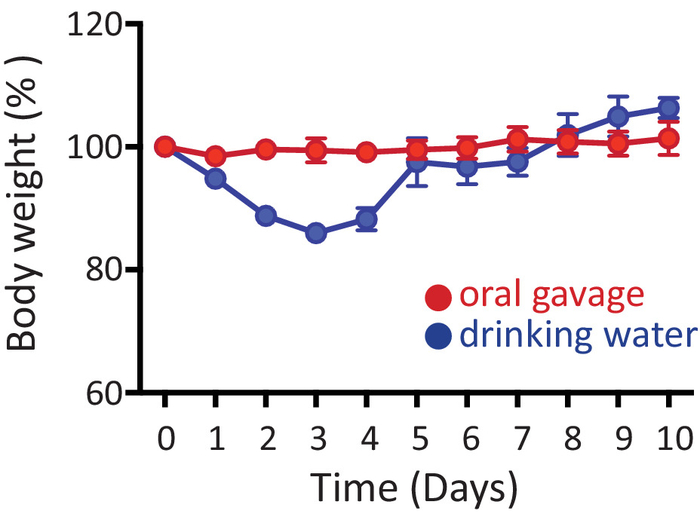
Figura 1: administração de antibióticos. Os ratos receberam antibióticos por gavagem oral (vermelha) ou na água potável (azul) durante 10 dias consecutivos. Enredo mostra os pesos dos ratos durante toda a duração dos experimentos em relação ao peso original antes da administração de antibióticos (dia 0). Dados são mostrados como média ± SEM. clique aqui para ver uma versão maior desta figura.
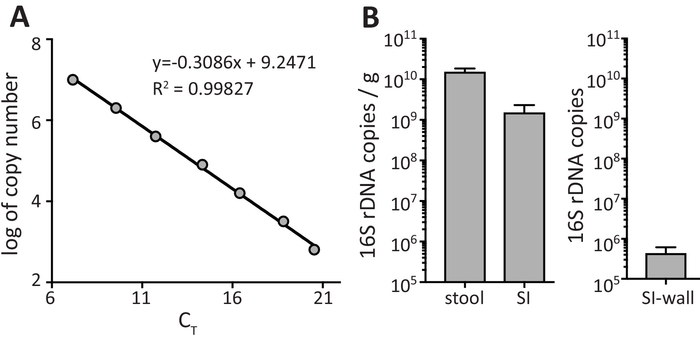
Figura 2: amplificação de qPCR de gene 16S rRNA de padrões e amostras fecais. (A) Regressão Linear da curva de calibração com descritores de curva padrão. (B) cálculo das abundâncias de gene de amostras fecais. Dados são mostrados como média ± SEM. clique aqui para ver uma versão maior desta figura.
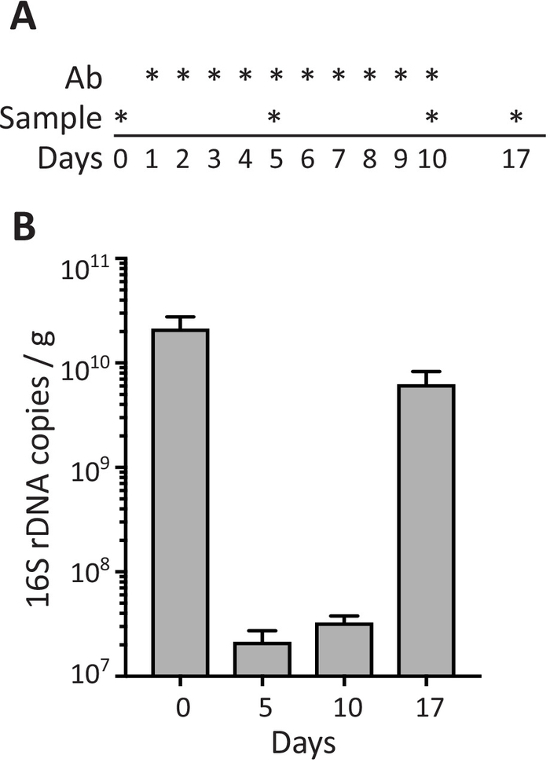
Figura 3: bactérias fecais durante o tratamento com antibióticos. (A) diagrama esquemático da agenda para a administração de antibióticos por gavagem oral (Ab) e coleta de amostra como retratado com *. (B) 16S rDNA copia por grama de fezes em amostras de fezes de ratos recolhidos no dias indicados. Dados são mostrados como média ± SEM. clique aqui para ver uma versão maior desta figura.
| Reagente | Volume de | |
| DNA | 8 Μ l | |
| Tampão 10 X | 2 Μ l | |
| dNTPs (10mM) | 0,4 Μ l | |
| Eubactérias - cartilha F (10 mM) | 1 Μ l | |
| Eubactérias - R primeira demão (10 mM) | 1 Μ l | |
| Taq Polimerase | 0,2 Μ l | |
| H2O | 7.4 Μ l | |
| Volume total | 20 Μ l | |
| Sequências da primeira demão | ||
| Eubactérias - cartilha de F | 5' ACTCCTACGGGAGGCAGCAGT 3' | |
| Eubactérias - cartilha de R | 5' ATTACCGCGGCTGCTGGC 3' | |
| Condições de ciclismo | ||
| Temperatura | Tempo | Ciclos de |
| 94 ° C | 5 min | 1 x |
| 94 ° C | 30 s | 30 x |
| 60 ° C | 30 s | 30 x |
| 72 ° C | 1 min | 30 x |
| 72 ° C | 5 min | 1 x |
| 4 ° C | ∞ | 1 x |
Tabela 1: PCR reagentes e condições. Esta tabela fornece os reagentes e condições de ciclagem do PCR para amplificar o gene de rRNA 16S de cultura bacteriana para a geração de um padrão para usar em ensaios da qPCR. Sequências da primeira demão foram originalmente publicadas por Kruglov et al 13.
| Reagente | Volume de |
| Mistura de mestre SYBR Green (2x) | Μ l 17,5 |
| Eubactérias - cartilha F (10 mM) | 0,7 Μ l |
| Eubactérias - R primeira demão (10 mM) | 0,7 Μ l |
| H2O | 11.1 Μ l |
Tabela 2: mistura de mestre qPCR. Os volumes mostrados (volume final 35 µ l) são para uma única amostra ser executado em triplicado (10 µ l de cada) em um prato de 384-bem qPCR (contabilização de 5 µ l extra para pipetagem erro). A quantidade pode ser ampliada de acordo com o número de amostras a analisar.
Discussão
Aqui nós fornecemos protocolos experimentais para a administração oral de antibióticos para ratos e quantificação de bactérias fecais por qPCR. A combinação de antibióticos usados em alvos deste protocolo (contendo ampicilina e gentamicina, neomicina, metronidazol, vancomicina) bactérias gram-positivas e Gram-negativas, oferecendo atividade bactericida contra um espectro completo de bactérias. Tanto a gavagem oral e a administração de antibióticos na água potável diminuem consideravelmente bacteriana fecal carregar5,6,12. Além disso, ambos os tratamentos têm um profundo efeito sobre o fenótipo dos ratos como eles desenvolvem várias características típicas dos ratos germ-free incluindo tamanho reduzido do baço e alargada do ceco. A seleção de um método específico para administração de antibióticos pode possivelmente depende da duração do experimento como o método de gavagem oral requer a administração diária de antibióticos, sendo mais trabalhosas e possivelmente causando mais desconforto para o animais em longo prazo.
Para a administração de antibióticos na água potável, cuidado deve ser tomado com a adição de adoçante à mistura de antibióticos como este é um fator crucial para manter os ratos da desidratação. Vários grupos têm mostrado como a administração de antibióticos em água potável (sem adição de adoçante) conduz à muito grave e rápida perda de peso com todos os mouses perder mais de 20% do peso corporal inicial dentro dos primeiros dias do experimento5 , 6. em nosso protocolo, o uso de adoçante sacarina baseado parecia ser suficiente para mascarar o sabor de antibiótico na água e os ratos perdida peso nos primeiros dias após a administração de antibióticos, mas recuperou seus pesos rapidamente após essa ( Figura 1). No entanto, em nossos experimentos 5-10% dos ratos ainda alcançar o ponto de extremidade humano de > 20% perda de linha de base de peso corporal e precisava ser sacrificado. Nós também testamos adoçantes sucralose-baseado que falharam completamente para evitar a desidratação de camundongos (100% dos ratos perdidos > 20% do peso) enquanto outros autores publicaram falhas semelhantes para adoçantes aspartame-baseado5,6. Somado a isso, a idade, fundo genético e estado de saúde geral dos ratos utilizados para os experimentos devem ser consideradas, como eles podem influenciar a perda de peso e bem-estar animal, durante o tratamento com antibióticos. Assim, o acompanhamento atento dos ratos peso e estado de saúde geral deve ser realizada diariamente durante as duas primeiras semanas de administração oral de antibióticos.
qPCR métodos fornecem uma abordagem rápida e econômica para quantificação de 16S rRNA em amostras fecais. No entanto, algumas limitações devem ser consideradas em relação esta técnica incluindo: eu) a exigência de um padrão confiável de alta qualidade; II) o design e a eficiência dos primers qPCR; III) o fato de que os microorganismos podem ter números diferentes de cópia do gene 16S rRNA, assim cópias do gene não podem diretamente ser igual de contagens de célula15. Não obstante, qPCR é um método robusto e sensível, que permite a rápida análise de amostras fecais. Esse método pode ser especialmente útil para validar rapidamente o efeito de diversos tratamentos (incluindo antibióticos) em cargas de bactérias fecais como detalhada aqui. Além disso, embora nós fornecemos um protocolo para a quantificação do total 16S rRNA, esse método pode ser facilmente adaptado (por projetar primers específicos16) para permitir a identificação dos táxons bacterianas individuais, proporcionando assim tanto quantitativa e informações qualitativas sobre microbiome tamanho e composição.
Em resumo, nós fornecemos dois protocolos de tratamento antibiótico oral de ratos e um método baseado em qPCR quantificar alterações induzida por antibióticos em bactérias fecais. Embora estes protocolos podem ser ainda mais otimizados e combinados com outras abordagens de acordo com as necessidades individuais de experimentais, podem servem como ferramentas de rápida, econômicas e confiáveis para manipular a microbiota intestinal murino e para estudar os efeitos do tratamento com antibiótico na homeostasia intestinal e doença.
Divulgações
Os autores não têm nada para divulgar.
Agradecimentos
Este trabalho foi financiado pelo Conselho de pesquisa médica UK (subvenção ao senhor p. b/L008157/1); R.J. foi apoiado por uma bolsa de Marie Marie Curie (H2020-ACEM-IF-2015-703639); P.M.B. foi apoiado por uma bolsa de estudo do Conselho de pesquisa médica do Reino Unido e faculdade Londres doutorado parceria do rei formação em ciências biomédicas (Sr/N013700/1).
Materiais
| Name | Company | Catalog Number | Comments |
| Ampicillin sodium salt | Sigma-Aldrich (Merck) | A9518 | |
| Neomycnin trisulfate salt hydrate | Sigma-Aldrich (Merck) | N1876 | |
| Metronidazole | Sigma-Aldrich (Merck) | M3761 | |
| Vancomycin hydrochloride | Sigma-Aldrich (Merck) | V2002 | |
| Gentamicin sulfate salt | Sigma-Aldrich (Merck) | G3632 | |
| Tryptone | Sigma-Aldrich (Merck) | T7293 | |
| Yeast Extract | Sigma-Aldrich (Merck) | Y1625 | |
| NaCL | Sigma-Aldrich (Merck) | S7653 | |
| Sweetener Sweet'n Low | Sweet'N Low | Available in the UK from Amazon.co.uk | |
| X-Gal (5-brom-4-chloro-3-indoyl B-D-galactopyranoside) | Fisher scientific | 10234923 | |
| Phosphate Buffered Saline | Thermo Fisher Scientific (Gibco) | 10010023 | |
| Ultrapure Agarose | Thermo Fisher Scientific (Invitrogen) | 16500500 | |
| RT-PCR grade water | Thermo Fisher Scientific (Invitrogen) | AM9935 | |
| Phusion High-Fidelity DNA Polymerase | New England BioLabs | M0530 | |
| Deoxynucleotide (dNTP) Solution Mix | New England BioLabs | N0447 | |
| iTaq Universal SYBR Green Supermix | Bio-Rad | 1725124 | with ROX |
| TOPO TA cloningTM for sequencing | Thermo Fisher Scientific (Invitrogen) | 450030 | |
| QIAamp fast DNA Stool mini kit | Qiagen | 51604 | |
| QIAprep spin Miniprep kit | Qiagen | 27106 | |
| QIAquick gel extraction kit | Qiagen | 28704 | |
| Syringe filter 0.45 µm | Fisher scientific | 10460031 | |
| Swann-MortonTM Carbon steel sterile scalpel blades | Fisher scientific | 11792724 | |
| Syringe (1 mL) | BD Plastipak | 303172 | |
| Syringe (20 mL) | BD Plastipak | 300613 | |
| 1.5 mL Crystal clear microcentriguge tube | StarLab | E1415-1500 | |
| 2 mL Ultra high recovery microcentrifuge tube | StarLab | I1420-2600 | |
| Oral dosing needles 20 G x 38 mm curved (pk/3) | Vet-Tech | DE008A | |
| Sterilin petri dish 50 mm | Scientific Laboratory Supplies | PET2020 | |
| Absolute qPCR plate seals | Thermo Fisher Scientific | AB1170 | |
| MicroAmpTM optical 384-well plate | Thermo Fisher Scientific (Applied Biosystems) | 4309849 | |
| ViiA7TM 7 real-time PCR system with 384-well block | Thermo Fisher Scientific (Applied Biosystems) | 4453536 | |
| Spectrophotometer (Nanodrop 1000) | Thermo Fisher Scientific | ND-1000 | |
| Labnet Prism microcentrifuge | Labnet | C2500 | |
| MultiGene Optimax Thermal cycler | Labnet | TC9610 |
Referências
- Belkaid, Y., Hand, T. W. Role of the microbiota in immunity and inflammation. Cell. 157 (1), 121-141 (2014).
- Hooper, L. V., Littman, D. R., Macpherson, A. J. Interactions between the microbiota and the immune system. Science. 336 (6086), 1268-1273 (2012).
- Ubeda, C., Pamer, E. G. Antibiotics, microbiota, and immune defense. Trends in Immunology. 33 (9), 459-466 (2012).
- Dethlefsen, L., Huse, S., Sogin, M. L., Relman, D. A. The pervasive effects of an antibiotic on the human gut microbiota, as revealed by deep 16S rRNA sequencing. PLoS Biology. 6 (11), e280 (2008).
- Reikvam, D. H., et al. Depletion of murine intestinal microbiota: effects on gut mucosa and epithelial gene expression. PLoS One. 6 (3), e17996 (2011).
- Hill, D. A., et al. Metagenomic analyses reveal antibiotic-induced temporal and spatial changes in intestinal microbiota with associated alterations in immune cell homeostasis. Mucosal Immunology. 3 (2), 148-158 (2010).
- Fraher, M. H., O'Toole, P. W., Quigley, E. M. Techniques used to characterize the gut microbiota: a guide for the clinician. Nature Reviews Gastroenterology & Hepatology. 9 (6), 312-322 (2012).
- Eckburg, P. B., et al. Diversity of the human intestinal microbial flora. Science. 308 (5728), 1635-1638 (2005).
- Sokol, H., et al. Low counts of Fecalibacterium prausnitzii in colitis microbiota. Inflammatiry Bowel Diseases. 15 (8), 1183-1189 (2009).
- Bartosch, S., Fite, A., Macfarlane, G. T., McMurdo, M. E. Characterization of bacterial communities in feces from healthy elderly volunteers and hospitalized elderly patients by using real-time PCR and effects of antibiotic treatment on the fecal microbiota. Applied Environmental Microbiology. 70 (6), 3575-3581 (2004).
- Ubeda, C., et al. Vancomycin-resistant Enterococcus domination of intestinal microbiota is enabled by antibiotic treatment in mice and precedes bloodstream invasion in humans. Journal of Clinical Investigation. 120 (12), 4332-4341 (2010).
- Saez de Guinoa, J., et al. CD1d-mediated lipid presentation by CD11c(+) cells regulates intestinal homeostasis. EMBO Journal. 37 (5), (2018).
- Kruglov, A. A., et al. Nonredundant function of soluble LTalpha3 produced by innate lymphoid cells in intestinal homeostasis. Science. 342 (6163), 1243-1246 (2013).
- Wilhelm, J., Pingoud, A., Hahn, M. Real-time PCR-based method for the estimation of genome sizes. Nucleic Acids Research. 31 (10), e56 (2003).
- Kembel, S. W., Wu, M., Eisen, J. A., Green, J. L. Incorporating 16S gene copy number information improves estimates of microbial diversity and abundance. PLoS Computational Biology. 8 (10), e1002743 (2012).
- Yang, Y. W., et al. Use of 16S rRNA Gene-Targeted Group-Specific Primers for Real-Time PCR Analysis of Predominant Bacteria in Mouse Feces. Applied Environmental Microbiology. 81 (19), 6749-6756 (2015).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados