Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Ingeniería de genoma CRISPR Cas9 basado en generar modelos de Jurkat reportero para infección por VIH-1 con los sitios seleccionados integración Proviral
En este artículo
Resumen
Presentamos un flujo de trabajo de ingeniería de genoma por la generación de nuevos modelos in vitro para la infección por VIH-1 que recapitular integración proviral en sitios genómicos. Ataques contra reporteros derivados del VIH es facilitada por la manipulación del genoma mediada por Cas9 CRISPR, site-specific. Cuentan con protocolos detallados unicelular clon generación, detección y verificación dirigida a corregir.
Resumen
Virus de inmunodeficiencia humana (VIH) se integra su ADN proviral en el genoma celular de host en sitios recurrentes y puntos calientes genómicas no-al azar. Aquí presentamos un protocolo detallado para la generación de nuevos modelos in vitro para la infección por el VIH con los sitios de integración genómica solicitadas usando tecnología ingeniería CRISPR Cas9 basado en genoma. Con este método, una secuencia de reportero de elección puede integrarse en un locus genómico específico, solicitado, reflejando sitios clínicamente relevantes de la integración.
En el protocolo, el diseño de un reportero derivados del VIH y la elección de una secuencia Diana de sitio y gRNA se describen. Un vector apuntado con armas de homología se construye y transfected en las células de Jurkat T. La secuencia del reportero está dirigida al sitio genómico seleccionado por recombinación homóloga facilitado por una rotura de doble cadena mediaron Cas9 en el sitio de destino. Sola célula clones generadas y proyectadas para apuntar eventos por citometría de flujo y PCR. Clones seleccionados se expanden, y orientación correcta se verifica mediante PCR, secuenciación y borrar meridional. Se analizan los potenciales eventos fuera del objetivo de la ingeniería del genoma mediada por Cas9 CRISPR.
Mediante el uso de este protocolo, sistemas de cultivo celular novela puede generarse la infección por VIH modelo en sitios clínicamente relevantes de la integración. Aunque la generación de clones de unicelular y verificación de integración de secuencia correcto periodista son desperdiciador de tiempo, las líneas clonales que son herramientas poderosas para analizar funcionalmente la elección del sitio de integración proviral.
Introducción
Integración del ADN proviral en el genoma del anfitrión a la infección es un paso crítico en el ciclo vital del virus de inmunodeficiencia humana (VIH). Tras la integración, el VIH persiste mediante el establecimiento de latencia en subconjuntos de células T CD4 + duraderos como células de memoria T CD4 +. Integración del VIH parece ser no-al azar1,2. Un número de hotspots genomic ADN proviral integrado recurrentemente se ha detectado en varios estudios a través de la secuenciación de los sitios de integración en los individuos infectados agudo y crónico2,3,4 ,5,6,7,8. Curiosamente, en algunos de estos sitios de integración, mismo lugar geométrico fue detectado en una gran parte de las células infectadas, llevando a la idea de que integración en sitios recurrentes podría afectar positivamente la extensión clónica1.
Para avanzar en nuestra comprensión de la importancia de los sitios de integración recurrente, elección del sitio de integración proviral debe ser explorada. Sin embargo, varios aspectos técnicos dificultan integración VIH estudio sitio de elección y las consecuencias. Ampliamente utilizado modelos de cultivo celular para la latencia del VIH como líneas de celulares JLat no reflejan integración recurrente clínicamente relevantes sitios9. Estudios sobre las células primarias derivados del paciente, por un lado, permiten la descripción del paisaje del sitio de integración por la secuencia pero no permiten análisis funcionales. A nuestro conocimiento, no hay un modelo experimental adecuado está disponible para analizar funcionalmente las sitios de integración clínicamente relevantes.
Aquí presentamos un detallado flujo de trabajo para generar nuevos modelos para la infección por VIH utilizando tecnología ingeniería CRISPR Cas9 basado en genoma. El flujo de trabajo descrito en este documento se puede utilizar para generar líneas de células de reportero derivados de la célula de T que la infección por VIH, llevando un reportero proviral genómicamente integrado en un sitio elegido de la integración del modelo. Por lo tanto están sirviendo como herramientas para explorar cómo el sitio de integración proviral puede afectar Biología del VIH y cómo el provirus responde a diferentes estrategias de tratamiento (p. ej., inducibilidad de, por los agentes de inversión de latencia). Nuestro método utiliza las ventajas de la ingeniería CRISPR Cas9 basado en genoma, en que integración del reportero de secuencia por la recombinación homóloga es facilitado por una rotura de doble cadena inducida por la nucleasa de Cas9 en el sitio de destino. Sitios de destino para su integración son seleccionados según la proximidad a los sitios de integración recurrente descrito de los estudios en personas infectadas por el VIH y la presencia de motivos de PAM adecuados para la ingeniería del genoma mediaron Cas9.
En los resultados de nuestros ejemplares, nos hemos centrado en el BACH2 Lugar geomtrico del gene, que codifica el regulador transcripcional de BTB y homología de CNC 2. En los individuos crónicamente infectados por el VIH en tratamiento antirretroviral, BACH2 es uno de los lugares geométricos que enriquecimiento del integrado VIH-1 secuencias3,6,7,8,10. Hemos elegido mínimo derivados del VIH reportero de VIH-1-derivado largo terminal de repetición (LTR), secuencia de codificación de tdTomato y hormona de crecimiento bovino (BGH) señal de poliadenilación (PA), nos hemos dirigido a dos sitios específicos en el BACH2 intrón 5. El protocolo presentado está optimizado para Jurkat células, una humana CD4 + línea celular derivada de la célula de T de la suspensión, pero otras células pueden utilizarse líneas y el protocolo adaptado en consecuencia. Presentamos un detallado flujo de trabajo para la selección del sitio de destino, construcción del vector blanco con brazos de homología, CRISPR Cas9-mediada por ataques contra el reportero en el lugar escogido genómica, generación y selección de líneas clonales y completa de verificación de líneas celulares de reportero recién generada, destino.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. segmentación estrategia de genoma ingeniería y orientación diseño vectorial (tv)
Nota: El primer paso de la ingeniería del genoma implica selección y generación de las herramientas necesarias para mediado de Cas9 CRISPR apuntar. Selección de un lugar del sitio de integración genómica, elección del tipo de célula para apuntar y el diseño de un reportero derivados del VIH para la integración deben preceder a este paso. Este protocolo describe la focalización de un reportero mínimo VIH-LTR_tdTomato_BGH-PA en las células de Jurkat blanco. Un diagrama de flujo del flujo de trabajo para CRISPR Cas9 basado en targeting, generación, investigación y verificación de líneas clonales se representa en la figura 1. La estrategia de focalización descrita utiliza el Cas9 de S. pyogenes (SpCas9) para generar dsDNA gRNA dirigida se rompe en un sitio de integración seleccionada. El reportero es entonces concentrado en el locus genómico solicitado a través de recombinación homóloga proporcionando un no lineal targeting vector (tv) que contiene la secuencia de reportero flanqueada por supuesto 5' y 3' homología brazos (HA)11.
-
Elección del locus específicos, gRNA y targeting vector diseño
- Elije thegenomic locus a orientarse con la pregunta científica individual. Uso de listas publicadas de los sitios de integración recurrente del VIH encontradas en pacientes en los diferentes estudios2,3,4,5,6,7,8 como un pauta. Extracto in silico de la secuencia genómica del locus genómico deseada dirigida (secuencia del gen completo) o por lo menos 5 kb de secuencia genómica genoma UCSC navegador (http:// genome.edu.ucsc.edu).
- Seleccione Guía RNAs (gRNAs) de 20 nt para apuntar del locus genómico solicitado utilizando el E-quebradizo webtool (http://www.e-crisp.org).
- Seleccione "GRCh38 Homo sapiens" como el organismo. Entrada 2.000 bp de la secuencia genómica que cubre el deseado locus genómico extraído en el paso 1.1.1.
- Iniciar una búsqueda de gRNA utilizando la configuración de la aplicación media (cualquier PAM, cualquier 5' base, de objetivos toleran las discrepancias, y se excluyen las islas CPG/intrones). Aparecerá una lista con gRNA posibles diseños, ranking de mayor a bajar partituras para especificidad y eficiencia.
- Seleccione un gRNA que preferiblemente muestra una puntuación alta especificidad y eficiencia y que es lo más cercano posible al deseada locus genómico para orientarse.
Nota: Un compromiso entre la proximidad al deseada locus genómico y el diseño de gRNA específica y eficiente tiene que ser encontrado.
- Explosión de la secuencia de gRNA solicitadas contra el genoma de referencia usando el browser de blast NCBI (https://blast.ncbi.nlm.nih.gov) para verificar la unicidad del sitio de unión del gRNA.
- Seleccione "humano" como el genoma. Entrada de la secuencia de gRNA como la secuencia de consulta. Seleccione "secuencias altamente similares" (megablast) como el programa. Asegúrese de que la secuencia de gRNA es única. Si no, elegir un gRNA diferentes paso 1.1.2.3 y explosión otra vez.
- Una vez escogida la gRNA secuencia, selecciónelo en silico 1.000 PB upstream y downstream de gRNA secuencia secuencia genomic extraída en el paso 1.1.1 como 5' y 3' HA por consiguiente.
Nota: Las gRNAs debe ser homólogo al lugar geométrico del sitio de integración genómica solicitadas y situado junto a un motivo adyacente de protospacer (PAM, por ejemplo,, NGG para SpCas9) (Figura 2a). La televisión contiene la secuencia de reportero que es 5' y 3' flanqueado por ha. Tiene cubierta 1000 bp aguas arriba y aguas abajo de la gRNA secuencia11. La secuencia de gRNA completo no se incluyera en la HA. Una superposición de hasta 5 nt es aceptable (Figura 2a).
-
Generación de gRNA y targeting de vectores
Nota: Para programas de vectores, ver figura 2b.- Para generar un vector para la expresión de SpCas9 y gRNA, utilice pX330-U6-Chimeric_BB-cBh-hSpCas9 como la columna vertebral que SpCas9 y la guía simple de RNA (sgRNA) pueden ser expresados simultáneamente. Para clonar la secuencia de gRNA en la columna vertebral, usar los títulos BbsI restricción sitios12.
- Para generar el televisor, elija un plásmido de alto copia como columna vertebral (pMK de, o cDNA3.1).
- En primer lugar, montar el reportero (en este protocolo: LTR_tdTomato_BGH-PA) en la columna vertebral de la construcción por montaje de Gibson clong kit13 usa un clonación comercial e introducir 5' y 3' que flanquean sitios de restricción (por ejemplo,, 5' PacI y 3' SmaI) para posterior restricción digestión clonación de la HAs.
- Amplificar bp de 1000 de los fragmentos HA elegido en el paso 1.1.4 de DNA genómico (gDNA) el tipo de células a ser objetivo (en este protocolo: células Jurkat) usando una ADN polimerasa con actividad de corrección (ver cuadros 1 y 2 para los ingredientes de la polimerización en cadena y condiciones de ciclismo). Luego, introducir el reportero que flanquean sitios de restricción en los extremos 5' y 3' de cada HA (PacI en 5' HA en ambos extremos, SmaI en 3' HA en ambos extremos).
- Secuencialmente el clon tiene en columna vertebral de la construcción ya que el reportero (generado en el paso 1.2.2.1) por enzima de restricción, clonación de14,15. En primer lugar, la copia en 5' HA entonces con sitios de restricción de PacI, clonado en 3' HA utilizando sitios de restricción SmaI.
Nota: Si la columna vertebral de la tv contiene un reportero fluorescente adicional, integración de la columna vertebral no deseados puede evaluarse mediante citometría de flujo (ver pasos 3.2.2 y 3.2.3). Si la columna vertebral de la tv no contiene ningún reportero fluorescente, integración de la columna vertebral debe evaluarse mediante PCR (ver paso 3.2.8).
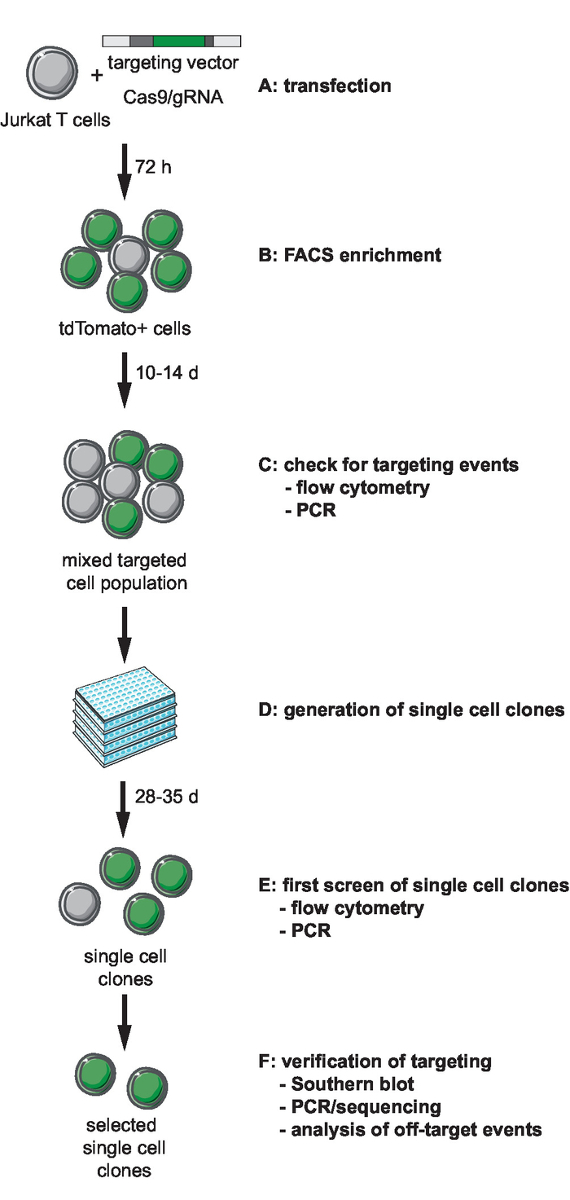
Figura 1: líneas de flujo de trabajo mediada por Cas9 CRISPR dirigido a, la generación y selección de reportero clonal con sitio de integración definidos. (A) generar el vector objetivo y transducir las células Jurkat T vector blanco y plásmido de expresión Cas9/gRNA. (B) enriquecen la transfección de post 72 h las células transfected por FACS. (C) dejar que las células crecen de 10 a 14 días y confirman la ocurrencia de apuntar eventos por PCR y citometría de flujo. (D) generar clones unicelular limitando clones dilución y dejar crecen durante 3 semanas. (E) pantalla los clones para apuntar correcto por PCR y citometría en formato de 96 pozos de flujo. Ampliar clones seleccionados. (F) verificar correcto en clones seleccionados por Southern blot, PCR, secuenciación y análisis de eventos fuera del objetivo de la actividad de endonucleasa Cas9. Haga clic aquí para ver una versión más grande de esta figura.
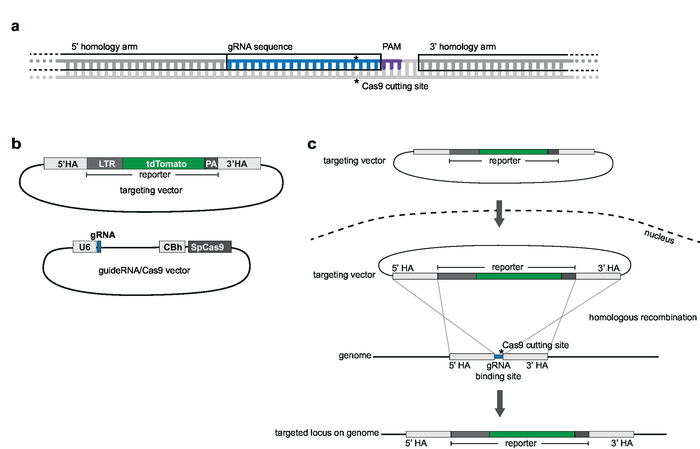
Figura 2: A estrategia y vector diseño. (un) gRNA y opción de brazos de homología. 20 nt gRNA es homólogo al sitio de destino genómica solicitadas y situado junto a una homología de identificación armas son complementarios a 1.000 bp arriba y aguas abajo de la gRNA y no deben incluir la secuencia de gRNA. (b) esquemas de focalización vector y gRNA/Cas9. El vector de objetivo consiste en la secuencia de reportero solicitadas que es 5' y 3', flanqueado por los brazos de homología. El vector de gRNA/Cas9 se basa en la columna vertebral pX330-U6-Chimeric_BB-cBh-hSpCas9. (c) esquema de focalización por recombinación homóloga. Vector objetivo y guideRNA/Cas9 son transfected en las células Jurkat. Cas9 media una rotura de doble cadena en el sitio de destino genómica (indique *) y facilita la recombinación homóloga y la integración de la secuencia de reportero en el lugar de destino genómica. Haga clic aquí para ver una versión más grande de esta figura.
2. CRISPR Cas9 basado en objetivos de células Jurkat
-
Transfección de células Jurkat
- 24 h antes de la transfección, placa de 1,25 x 106 T Jurkat células en 2,5 mL de RPMI 1640 complementado con 10% (v/v) de suero fetal de ternero (FCS) y 4 mM L-glutamina [denominado "Antibióticos de w.o. RPMI (AB)"] por pozo de una placa de cultivo celular 6-bien. Para un solo experimento objetivo, preparar una placa de 6 pozos completa (es decir,, 6 pozos cada uno con 2,5 mL de la suspensión celular).
- Al día siguiente, co transfectar las células con circular tv y pX330-U6-Chimeric_BB-cBh-hSpCas9/gRNA usando un específico reactivo de transfección de células Jurkat.
- Añadir 2 μg de tv circular y 2 μg de pX330-U6-Chimeric_BB-cBh-hSpCas9/gRNA por pozo a 250 μl de Medio RPMI comercial con concentración sérica reducida optimizada para transfección (RPMI con reducción del 50% en suero) en un tubo de reacción y mezclar bien.
- Añadir 12 μl de reactivo de transfección lentamente al medio del ADN sin tocar la pared del tubo y agitar. Deja la mezcla incubar por 15 min y añadir gota a gota a un pozo de las células. Incube las células a 37 ° C y 5% CO2.
Nota: La preparación de la reacción de transfección puede ampliarse. No hay cambio de medio se requiere después de transfección.
-
Enriquecimiento de células transfected por celular activado por fluorescencia (FACS) de clasificación
- piscina de 72 h después de la transfección, las células transfected, contarlos y preparar para el enriquecimiento por FACS. Recoger las células en un tubo cónico de 50 mL, centrifugar a 300 x g y temperatura ambiente (RT) por 4 min., lavar las células una vez en PBS, centrifugar nuevamente, suspender el sedimento en una cantidad apropiada de tampón FACS (PBS suplementado con 1% FCS + 1 mM EDTA) en 1 x 10 7 células/mL y finalmente transferir a un tubo de FACS.
- Someter las células a FACS y ordenar aquellos que expresan el reportero fluorescente de la tv (e.g., tdTomato en este protocolo). Recoger las células en RPMI 1640 suplementado con 10% FCS, 4 mM L-glutamina y 50 U/mL de penicilina y estreptomicina (denominado "RPMI con AB").
- Después de FACS clasificar, lavar las células una vez añadiendo 20 mL de RPMI con AB a las células clasificadas y centrifugar a 300 x g durante 4 min a RT. Resuspender el pellet celular en una cantidad apropiada de RPMI con AB y placa las células en un pozo de una placa de cultivo celular con la volumen adecuado según el número de células post-FACS.
Nota: Cultura clasificar las células en un volumen pequeño (e.g., 24-bien), como considerables niveles de muerte celular se han observado en la primera semana post-targeting (hasta 80-90%). - Ampliar la población objetivo mezclado de la célula hasta una densidad de 1 x 106 células/mL en un matraz de cultivo celular de 75 cm². Esto le llevará alrededor de 10 – 14 días post-FACS clasificación.
-
Confirmación de apuntar eventos mediante citometría de flujo en la población objetivo mezclado de la célula
- Después de 10 a 14 días de expansión (cuando las células han alcanzado una densidad de 1 x 106 células/mL en un matraz de cultivo celular de 75 cm2 ), dos pozos de la placa con 1 x 106 células de la mixta dirigida a población de células en 1 mL de RPMI con AB en un cultivo celular de 12 pozos placa.
- Inducir el LTR del VIH del reportero (generación de tv se describe en el paso 1.2.2.1.) en uno de los pozos mediante la adición de 50 ng/mL forbol 12-miristato 13-acetato (PMA) y 1 μm Ionomycin (denominado PMA-Iono). Utilice las células en el segundo pozo que el control no inducida. De la cultura el inducido y las células no inducidas por 24 h.
- Tomar 0,5 mL de la suspensión celular de células inducidas y no inducidas (cada uno), lavarlos una vez en PBS y suspender a cada uno en 200 μL de tampón FACS.
- Analizar las 100.000 células por citometría de flujo. Las células solo viables basadas en el tamaño en dispersión hacia delante y hacia un lado de la puerta y analizar la expresión del gen reportero fluorescente.
Nota: En este paso (10 – 14 días post-FACS clasificación), la expresión del reportero fluorescente por la transfección transitoria no debería ser detectable. Expresión de fluorescente en este momento indica integración genomic de la secuencia de reportero.
-
Detección de apuntar eventos por PCR el ADN genómico de la población objetivo mezclado de la célula
Nota: Para detectar eventos dirigidos a través de PCR, diseño dos pares de imprimación específica para la integración (int.) ensambladura de 5' y 3' cruce int.. Para la 5' Unión PCR int., debe enlazar el primer avance aguas arriba del 5' HA y el primer revés en el litro de la reportera (iniciadores P1 y P2 en la figura 3a). El par de la cartilla para el cruce 3' int PCR debe abarcan desde el PA del reportero a 100 – 200 bp aguas abajo de los 3' HA (cartillas P3 y P4 en la figura 3a). Primers P1 y P4 también servirá para la amplificación del alelo en la población objetivo mixto no dirigidos. Para un esquema, vea la figura 3a.- Preparar gDNA de 2 mL de la suspensión celular de la población de mezclado de la célula específica del paso 2.2.4. Utilice un kit de extracción gDNA según protocolo del fabricante. Preparar a continuación gDNA de células no dirigidos como control.
- Realizar cruce int. PCRs (iniciadores P1/P2 y P3/P4 5' y 3' cruce int., respectivamente) y no dirigidas alelo PCR (iniciadores P1/P4) usando una ADN polimerasa de alta fidelidad (ver tablas 3 y 4 para PCR ingredientes y condiciones de ciclismo) . Analizar 5 μl de los productos PCR en gel de agarosa/TAE 1.5%.
Nota: Si la población objetivo mixta contiene células que han sido sometidos a ingeniería del genoma, un producto PCR específico debe observarse que no es detectable en gDNA de células no dirigidos (control negativo). Para el alelo no orientada por PCR, se debe observar un producto del mismo tamaño para ambos las células dirigidas y no dirigidas (control positivo para genómicas iniciadores P1 y P4). Si observan no hay bandas, considere alterando condiciones ciclo de PCR por aumentar el número de ciclos o alterar el tampón de PCR (por ejemplo a través de la adición de DMSO o mayores cantidades de Mg2 +), o cambiando la polimerasa.
3. generación de líneas clonales y proyección para apuntar al blanco correcto
Nota: Después de la confirmación de los eventos dirigidos a la población de mezclado de la célula dirigida por citometría de flujo y PCR (secciones 2.2 – 2.4), generar clones de unicelular (duración: 28 a 35 días) y pantalla para la correcta integración de la secuencia de reportero.
- Generación de clones de unicelulares a través de placas de la dilución
- Preparar medio de Jurkat-condicionada de antemano: sacar RPMI con medio AB de células Jurkat T sanas, sin tratar a 1 x 106 células/mL, centrifugar durante 5 min a 300 x gy filtrar el sobrenadante usando una unidad de filtro de jeringa de 0,22 μm.
Nota: Mantener el medio condicionado a 4 ° C para almacenamiento a corto plazo o a-20 ° C para el almacenamiento de más de 1 semana. Preparar 20 a 30 mL de medio condicionado antes de galjanoplastia de dilución. - Contar las células apuntadas de paso 2.2.4 después de 10 a 14 días de expansión y los diluye en RPMI con AB a una concentración de 1 x 105 células/mL. Tomar 100 μl de solución de 1 x 105 células/mL y diluir con 9,9 mL de medio para alcanzar una concentración de 1.000 células/mL. Tomar 1 mL de solución de 1.000 células/mL y diluir con 9 mL de medio para alcanzar una concentración de 100 células/mL.
- Placa de 96 pocillos platos contiene 1 célula por pozo y 2 células por pocillo. 1 células por pocillo, tomar 1 mL de solución de 100 células/mL y mezclar suavemente con 5 mL de medio condicionado y 4 mL de medio fresco en un reservorio de reactivo estéril.
- Para las 2 células por pocillo, tomar 2 mL de 100 células/mL de la solución y mezclar suavemente con 5 mL de medio condicionado y 3 mL de medio fresco. Placas placa de 96 pocillos de fondo redondo con 100 μl de la dilución de respectivas células por pocillo con una pipeta multicanal.
Nota: 5 a 10 placas de 96 pocillos por dirigidos a construir son suficientes para obtener clones para proyección. - Apilar las placas de 96 pocillos, cubrir cada pila con una placa de 6 pozos que contienen 3 mL de PBS en cada pocillo e incubar las placas a 37 ° C en un incubador de cultura celular humidificada con 5% CO2 durante 3 semanas.
Nota: No cambie el medio de cultivo celular durante este tiempo. No abra la incubadora más de una vez o dos veces a la semana. Los mejores resultados se observan en las incubadoras con depósito de agua abierto. - Después de 3 semanas de incubación, confirmar visualmente la presencia de colonias crecidas usando microscopia ligera (4 aumentos) y marque los pozos con colonias crecidas por lo que son visibles como puntos en la parte inferior de los pozos.
- Preparar una placa de 96 pocillos de fondo redondo con 100 μl de RPMI con AB por pozo. Resuspender suavemente las células de un marcado bien por pipeteo. Transferir 100 μl de la suspensión celular en un pocillo de la placa de 96 pocillos nueva ya que contiene 100 μl de RPMI con AB, luego mezclar suavemente mediante pipeteo. Transferir 100 μl de esta suspensión celular en un vacío segunda placa de 96 pocillos de fondo redondo para duplicar la placa.
- Continuar con todos los pocillos marcados con colonias crecidas. Rellenar los pocillos del blanco con 200 μL de RPMI con medio de AB. Incubar las placas a 37° C y 5% CO2.
Nota: Una de estas placas servirán para la expansión de clones unicelular "(placa de stock") y el otro como un"duplicado" para proyección.
- Preparar medio de Jurkat-condicionada de antemano: sacar RPMI con medio AB de células Jurkat T sanas, sin tratar a 1 x 106 células/mL, centrifugar durante 5 min a 300 x gy filtrar el sobrenadante usando una unidad de filtro de jeringa de 0,22 μm.
- Selección de clones de unicelular mediante citometría de flujo y PCR
Nota: Mientras que los clones de células solo se están expandiendo, utilice la placa duplicada de paso 3.1.8 se reproduce sola celda de pantalla para presencia de reportero secuencia por PCR (medidas 3.2.4–3.2.12) y la expresión del reportero fluorescente mediante citometría de flujo (medidas 3.2.2–3.2.3) (Figura 3C).- Deje la placa duplicada incubar durante 24-48 h y duplicar la placa otra vez. Para hacer esto, añadir 100 μl de RPMI con AB a cada pocillo, mezclar suavemente mediante pipeteo y transfiera 100 μL en una placa de 96 pocillos de fondo redondo nuevo utilizando una pipeta multicanal. Utilizar una placa para proyección de citometría de flujo y el otro basado en la PCR.
- Para la detección de citometría de flujo, estimular las células con PMA-Iono. Preparar un mastermix de 0,1 μl de Ionomocin (1 mM stock), 0.1 μl de PMA (50 μg/μl comunes), y 4.8 μl de RPMI con AB por número de pozos, luego añadir 5 μl del mastermix por pozo.
Nota: Inducción es necesaria identificar con éxito los clones, donde el litro puede ser transcripcionalmente silenciosa y por lo tanto no se expresa el reportero fluorescente. - Dejar que las células incubar durante 24 h y preparar las células para citometría de flujo como se describe en el paso 2.3.3. Cualquier solo células viables, basadas en tamaño de dispersión hacia adelante y hacia un lado de la puerta y análisis de la expresión del gen reportero fluorescente por citometría de flujo (por ejemplo los resultados, ver figura 3 c). Si la columna vertebral de la tv contiene un segundo reportero fluorescente con el promotor (por ejemplo, GFP), pantalla también cualquier clones para la expresión del reportero de columna vertebral (ver paso 1.2.2 y la nota siguiente para explicación).
Nota: Columna vertebral reportero expresión indica integración no deseado de las secuencias de la columna vertebral. - Una vez que los clones en la segunda placa duplicado han crecido lo suficiente (generalmente 24 a 48 h después de la duplicación de la placa de 96 pocillos), preparar los lysates de la célula que contiene gDNA para la detección de PCR. Centrifugar la placa por 10 min a 300 x g en RT. cuidadosamente saque el sobrenadante sin perturbar el sedimento celulares.
Nota: Todos los pasos para la preparación de lisados y reacciones de PCR se pueden realizar con pipetas multicanales. - Lavar las células con 100 μl de PBS por pipeteo suave y centrifugar la placa por 5 min a 300 x g a TA. Sacar el PBS y añadir 200 μL de tampón de lisis [200 mM NaCl, 100 mM Tris-HCl pH 8-8.5, 5 mM EDTA, 0.1% SDS; luego agregar 250 – 1.000 μg/mL de proteinasa K (liofilizado en polvo, pesaje de recién)] por pozo. Mezclar suavemente mediante pipeteo y transferir la suspensión a una placa PCR de nuevo.
- Sellar la placa con la película de parafina e incubar 1 h a 55 ° C en un termociclador. Centrifugar a velocidad máxima durante 10 minutos (3.000 x g) girar por restos celulares y transferir el sobrenadante a un nuevo plato de PCR.
Nota: Lysates de la célula en placas pueden almacenarse en esta etapa a 4 ° C hasta su uso posterior. - Preparar una placa PCR de 96 pocillos con 110 μl de dH2O y añadir 10 μl de lisado de células (1:12 dilución). Lysates de la célula puede ser viscoso y difícil de pipetear. Use por lo menos puntas de pipeta de 20 μl.
- Inactivar la proteinasa K por la incubación durante 10 min a 99 º C en un termociclador. Posteriormente utilice los lysates de la célula inactivado y diluido para la detección de PCR.
- Diseño de cebadores para la detección de PCR (P5 y P6) basado en la secuencia de reportero solicitadas para amplificar 500 – 800 bp de la secuencia de reportero. Para el control positivo de PCR, utilizar cartillas P7 y P8 que amplifican 630 bp de un locus genómico tipo salvaje, no dirigidos (genNUP188 ) (figura 3 c y tabla 5). Diseño de un tercer par de cartilla que amplifica 500 – 600 bp de la columna vertebral de la tv como un control para la integración de secuencias de tv columna vertebral (espina dorsal PCR).
- Para la proyección, control y la columna vertebral PCR, utilizar un comercial mastermix PCR (ver cuadros 6 y 7 para PCR ingredientes y condiciones de ciclismo). Utilice 2 μl del inactivado y diluido lisado elaborado en el paso 3.2.8 como plantilla y PCR para 38 a 40 ciclos de amplificación por PCR en formato de 96 pozos.
- Analizar 5 μl de los productos PCR en gel de agarosa/TAE 1.5%.
Nota: Para el control de la polimerización en cadena, una banda específica de 630 bp debe ser observado para cada muestra, confirmando que la calidad de los lysates de la célula es adecuada para PCR. Una banda específica de detección PCR (500 – 800 bp según primer diseño) indica la integración de la secuencia de reportero. Una banda específica para columna vertebral PCR (500 – 600 bp, dependiendo del diseño de la cartilla) indica no deseado integración de secuencias de la columna vertebral de tv (por ejemplo los resultados, ver figura 3 c). - Combinar los resultados de la citometría de flujo (paso 3.2.3) y el análisis polimerización en cadena-basado (paso 3.2.12). Clones selectos que muestran tamaños correctos de los productos PCR en la detección de PCR y control positivo de PCR y la expresión del reportero fluorescente después de la inducción con PMA-Iono en citometría de flujo. Excluir clones que cualquier producto de la polimerización en cadena en espina dorsal PCR o expresión de tv codificada en columna vertebral proteína fluorescente, indicando la integración inespecífica de secuencia de columna vertebral de tv.
- Gradualmente ampliar clones seleccionados de la placa de 96 pocillos stock de formatos muy grandes (48/24/12/6 pocillos) hasta lograr la forma T75 célula cultura matraz mediante la adición de medio fresco cada 2 a 3 días. Mantener una densidad celular entre 1 x 105 y 1 x 106 células/mL.
- Asegúrese de preparar acciones celulares de los clones durante la expansión: cuenta las celdas, centrifugar a 300 x g durante 5 min a temperatura ambiente, desechar el sobrenadante y suspender las células en FCS + 10% DMSO 5 x 106 células/ml. Alicuotar en viales criogénicos y envase de uso un cryo-congelación para congelar las células a 80 ° C a 1 ° C por minuto. Para almacenamiento a largo plazo, transferir al líquido N2.
Nota: Es recomendable mantener un frasco de cultivo celular T75 (es decir,, alrededor de 1 x 107 células) durante la expansión en la preparación del gDNA para la verificación de la focalización por borrar meridional (véase la sección 3.4).
- Verificación de sitios de integración por la PCR/secuenciación de clones seleccionados
Nota: ensambladuras int. 5' y 3' de los clones seleccionados son PCR amplificado y enviado a Sanger secuenciación para verificar corregir dirigidos a nivel de la secuencia de ADN.- Preparar gDNA de los clones seleccionados y células de tipo salvaje de Jurkat utilizando un kit de extracción comercial gDNA.
- Utilice pares de primer atar el extremo 5' de la periodista y aguas arriba de 5' HA de 5' Unión interior (iniciadores P1 y P2) y el extremo 3' de la periodista y aguas abajo de 3' HA por 3' cruce int. (cartillas P3 y P4) como se describe en el paso 2.4. Utilizar iniciadores P1 y P4 para ampliar el sitio de integración específica en el alelo sin reportero integrante (figura 4a).
- Preparar reacciones de PCR con 100 – 200 ng del gDNA como plantilla y realizar PCR utilizando una polimerasa con actividad de corrección (ver tablas 1 y 2 para PCR ingredientes y condiciones de ciclismo).
Nota: Si vendas no se observaron, considere la posibilidad de alterar las condiciones del ciclo de PCR aumentando el número de ciclos o alterar el tampón de PCR (por ejemplo, mediante la adición de DMSO o mayores cantidades de Mg2 +), o cambiando la polimerasa. - Analizar 5 μl de los productos PCR en gel de agarosa/TAE 1.5%. Si se observan los tamaños de banda correcto, purificar el restante producto PCR utilizando un kit comercial y sujeto a Sanger secuenciación. Verificar secuencias de int. ensambladura de 5', 3' cruce int. y lugar deseado del alelo sin reportero integrante alineándolas con las secuencias esperadas.
Nota: El alelo homólogo donde el reportero no ha integrado probablemente mostrará mediaron Cas9 cambios en el sitio de destino, tales como inserciones de nucleótidos o eliminaciones (figura 4a). - Para los clones que muestran int. correcto secuencias de unión después de la alineación, realizar una PCR amplificando el reportero todo objetivo y sujeto a Sanger secuenciación para verificar la correcta secuencia de los integrantes.
- Análisis de la mancha blanca /negra meridional para la verificación de la focalización en los clones seleccionados
Nota: Análisis de Southern blot de clones seleccionados se requiere verificar la correcta orientación y excluir a eventos de recombinación mediaron Cas9 que pudieran haber ocurrido en el sitio de integración específicas.- Desarrollar una estrategia para la digestión apropiada gDNA y probe el diseño antes de comenzar el experimento.
- Seleccione una enzima de restricción restricción gDNA que genera fragmentos correspondientes de longitud de 2 a 10 kb en el lugar deseado. Ciertas enzimas de restricción, como Asp718, BamHI, BglI, BglII, EcoRV, HindIII, NcoI, PstI, PvuII, Scaque Stu, y SST Yo he utilizado con éxito para digerir gDNA de alto peso molecular.
- Diseño de dos diferentes sondas sur: un reportero-específico sonda y sonda genómica. El reportero-específico sonda hibridiza una secuencia dentro de la reportera (es decir,, tdTomato-específico punta de prueba). La sonda genómica cruza por hibridación a una región genómica cerca (pero no superpuestas) uno HA.
- Elija la sonda genómica que el fragmento generado por la digestión gDNA que será detectada por el atascamiento de sonda genómica diferencia en longitud (más de 2 kb) de los alelos dirigidos y no dirigidas (Figura 4b). Una longitud de sonda de 400 a 1.000 bp se recomienda.
- Diseño de primers PCR para amplificar las dos puntas de prueba requeridas. Amplificar la sonda específica de reportero de plantilla de tv usando una ADN polimerasa de alta fidelidad (ver tablas 3 y 4 para PCR ingredientes y condiciones de ciclismo).
- Amplificar la sonda genómica del salvaje-tipo Jurkat gDNA preparado con un kit de extracción comercial gDNA usando una ADN polimerasa con actividad de corrección (ver tablas 1 y 2 para PCR ingredientes y condiciones de ciclismo). Purificar los productos PCR en un gel de agarosa/TAE y extraer los fragmentos usando un kit de extracción de gel comercial según las instrucciones del fabricante.
- Extraiga el gDNA de alto peso molecular de 1 x 107 células de las células de Jurkat de tipo salvaje y clones seleccionados de paso 3.2.14.
- Sedimenten las células por centrifugación a 300 x g durante 5 min a temperatura ambiente, lavar una vez con PBS y suspender el sedimento en 4 mL de tampón de lisis [200 mM NaCL, 100 mM Tris-HCl pH 8, 5 mM EDTA, 0.1% SDS; añada proteinasa de 250-1.000 μg/mL K (polvo liofilizado peso en fresco)]. O/n a 55 ° C, agitando a 350 rpm en una mesa Termomezcladores de incubar.
- Añadir 4 mL de isopropanol y mezclar por inversión 10 a 20 veces. El gDNA debe quedar visible como precipitado blanco. Carrete del gDNA precipitado sobre la fina punta de una pipeta de vidrio, lavar emergiendo en 750 μl de 70% EtOH y deje seco a temperatura ambiente (5 a 10 min).
- Arrojar el precipitado en un tubo de reacción de 1,5 mL conteniendo 500 μl de tampón de TE de 1 x (10 mM Tris-HCl pH 8.0, 1 mM EDTA) y dejar para disolver o/n a 4 ° C, agitación de 350 RPM. Cualquier uso del gDNA de esta etapa debe realizarse con punta ancha para evitar el corte.
Nota: De alto peso molecular gDNA son esenciales para el análisis de Southern blot, y kits de preparación gDNA comercialmente disponibles no son adecuados.
- Digest (dos veces) 15 μg del gDNA de los clones seleccionados y células de Jurkat de tipo salvaje con las enzimas de restricción (ver paso 3.4.1.1) en una reacción de 60 μL con 6 μl de enzima (20 unidades/μL): en primer lugar, añadir ADN, tampón de digestión y ddH2O, incuban o/n a 37 ° C , luego añadir enzima e incubar o/n en la temperatura de digestión enzimática específica. 15 ug de gDNA digerido se requiere sonda sur.
- Usar 7 μl de la recopilación de la restricción de 60 μL para electroforesis analítica en gel de agarosa/TAE 1%. Un borrón de transferencia indica la digestión completa y buena calidad de ADN para el análisis de Southern blot.
- Precipitar el digest de restricción restante mediante la adición de 1:10 acetato de sodio de 3 M y 2 volúmenes 100% EtOH, luego incubar durante 1 h a-80 ° C y centrifugar por 30 min a 15.600 x g a 4 ° C.
- Deseche el sobrenadante y lavar el pellet con el 70% EtOH. Centrifugar durante 15 min a 15.600 x g a 4 ° C, eliminar el sobrenadante, dejó la pelotilla seca brevemente en la RT y disolver en 20 μl de ddH2O.
- Ejecute 1% agarosa/TAE Blot gel, 20 uL del gDNA digerido por carril de carga. Correr el gel por 2 h a 60 V, 400 mA.
Nota: El porcentaje de agarosa gel y funcionamiento tiempo/tensión puede ajustarse según el tamaño del fragmento esperado para la detección de la mancha blanca /negra meridional calculada en el paso 3.4.1.1. Los siguientes pasos de análisis de Southern blot se describen en detalle en un protocolo adicional al Protocolo (pasos 1 a 18). Estos pasos comprenden: lavado del gel Blot, borrar en una membrana de nylon, generación de radiactivo de la sonda, sonda de hibridación y desarrollo de película autoradiograph. Comparar el patrón de bandas obtenido después del desarrollo de autoradiograph en paso 18 (Protocolo adicional) con el patrón esperado según estrategia meridional (por ejemplo los resultados, ver Figura 4b).
- Desarrollar una estrategia para la digestión apropiada gDNA y probe el diseño antes de comenzar el experimento.
- Análisis de los eventos de fuera de objetivo
Nota: Ya que la ingeniería del genoma mediada por Cas9 CRISPR puede generar efectos off-target, PCR-amplificar los diez más altos del ranking en silico-predijo sitios objetivo de seleccionaron clones y someterlas a Sanger secuenciación.- Utilizar CCTop16 (http://crispr.cos.uni-heidelberg.de) para generar una lista de los diez más altos del ranking en silico predice secuencias objetivo.
- Entrada de la secuencia de gRNA incluyendo PAM como para apuntar como una secuencia de consulta. Seleccione "NGG" PAM como "Genoma humano" como la referencia para la predicción del objetivo.
- Máximos total desajustes "4" y el objetivo de establecer sitio longitud a la longitud de la gRNA sin PAM. El archivo de salida proporcionará una lista ordenada de sitios objetivo genómicos para gRNA respectivo.
- In silico Extraiga el bp de secuencia genómica 500 aguas arriba y aguas abajo de cada uno de los diez más alto del ranking objetivo hits usando el Browser del genoma de UCSC (http://genome.edu.ucsc.edu) y la posición del golpe de la lista de resultados de la CCTop de blanco.
- Para cada uno de los sitios de destino para analizar, diseño par de una cartilla PCR que amplifica un fragmento de 600 a 700 bp de longitud incluyendo el sitio de destino previsto.
- Extraiga el gDNA de los clones seleccionados y células de tipo salvaje de Jurkat utilizando un kit de extracción comercial gDNA. Para cada sitio de fuera de objetivo, realizar una polimerización en cadena usando una ADN polimerasa con actividad de corrección (ver tablas 1 y 2 para PCR ingredientes y condiciones de ciclismo) en el tipo salvaje y la respectiva gDNA derivados de clones.
- Analizar 5 μl de los productos PCR en gel de agarosa/TAE 1.5%. Si se observan los tamaños de banda correcto, purificar el restante producto PCR utilizando un kit de purificación PCR comercial y sujeto a Sanger secuenciación. Comparar secuencias de los sitios de fuera de objetivo en las células de Jurkat y los clones específicos.
- Utilizar CCTop16 (http://crispr.cos.uni-heidelberg.de) para generar una lista de los diez más altos del ranking en silico predice secuencias objetivo.
Access restricted. Please log in or start a trial to view this content.
Resultados
En este experimento representativo hemos elegido a un reportero de VIH-1-derivado mínimo que consta de un litro, tdTomato codificación secuencia y secuencia señal de polyA para dos loci en el intrón 5 BACH2 gen17. Los loci para apuntar al blanco fueron escogidos según la proximidad a sitios de integración recurrente publicado encontrado en diversos estudios sobre las células T primarias de pacientes VIH-infectados2,
Access restricted. Please log in or start a trial to view this content.
Discusión
Aquí, describimos un protocolo para generar modelos de reportero de VIH-1-derivadas de Jurkat con sitios de integración proviral elegido aplicar ingeniería CRISPR Cas9 basado en genoma.
Varios puntos del protocolo requieren especial atención durante la fase de planificación. En primer lugar, el lugar geométrico de a debería ser elegido cuidadosamente, como algunos loci podrían ser más fáciles al destino que otros (por ejemplo,, dependiendo del estado de la cromatina de la re...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Agradecemos a Britta Weseloh y Bettina Abel para asistencia técnica. También agradecemos a Arne Düsedau y Jana Hennesen (plataforma de tecnología de citometría de flujo, Instituto de Heinrich Pette) para obtener asistencia técnica.
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| pX330-U6-Chimeric_BB-cBh-hSpCas9 | Addgene | 42230 | vector for expression of SpCas9 and gRNA |
| pMK | GeneArt | mammalian expression vector for cloning | |
| cDNA3.1 | Invitrogen | V79020 | mammalian expression vector for cloning |
| BbsI | New England Biolabs | R0539S | restriction enzyme |
| NEBuilder Hifi DNA Assembly Cloning Kit | New England Biolabs | E5520S | Assembly cloning kit used for target vector generation |
| TaqPlus Precision PCR System | Agilent Technologies | 600210 | DNA polymerase with proofreading activity used for amplification of homology arms (step 1.2.2.2), verification of integration site and reporter sequence (step 3.3.3 and 3.3.5), generation of genomic probe for Southern blot (step 3.4.1.5) and analysis of off-target events (step 3.5.4) |
| 96-well tissue culture plate (round-bottom) | TPP | 92097 | tissue culture plates for dilution plating |
| Phusion High-Fidelity DNA polymerase | New England Biolabs | M0530 L | DNA polymerase used for detection of targeting events (step 2.4.2) and generation ofreporter-specific probe for Southern blot (step 3.4.1.4) |
| Dimethyl sulfoxide (DMSO) | Sigma-Aldrich | D9170 | dimethyl sulfoxide as PCR additive |
| Magnesium Chloride (MgCl2) Solution | New England Biolabs | B9021S | MgCl2 solution as PCR additive |
| Deoxynucleotide (dNTP) Solution Mix | New England Biolabs | N0447S | dNTP mixture with 10 mM of each nt for PCR reactions |
| 5PRIME HotMasterMix | 5PRIME | 2200400 | ready-to-use PCR mix used for screening PCR (step 3.2.11) |
| QIAamp DNA blood mini kit | Qiagen | 51106 | DNA isolation and purification kit |
| QIAquick PCR Purification Kit | Qiagen | 28106 | PCR Purification Kit |
| RPMI 1640 without glutamine | Lonza | BE12-167F | cell culture medium |
| Fetal Bovine Serum South Africa Charge | PAN Biotech | P123002 | cell culture medium supplement |
| L-glutamine | Biochrom | K 0282 | cell culture medium supplement |
| Penicillin/Streptomycin 10.000 U/mL/ 10.000 µg/mL | Biochrom | A 2212 | cell culture medium supplement |
| Gibco Opti-MEM Reduced Serum Media | Thermo Fisher Scientific | 31985062 | cell culture medium with reduced serum concentration optimized for transfection |
| TransIT-Jurkat | Mirus Bio | MIR2125 | transfection reagent |
| phorbol 12-myristate 13-acetate | Sigma-Aldrich | P8139-1MG | cell culture reagent |
| Ionomycin | Sigma-Aldrich | I0634-1MG | cell culture reagent |
| Syringe-driven filter unit, PES membrane, 0,22 µm | Millex | SLGP033RB | filter unit for sterile filtration |
| Heracell 150i incubator | Thermo Fisher Scientific | 51026280 | tissue culture incubator |
| Amershan Hybond-N+ | GE Healthcare | RPN1520B | positively charged nylon membrane for DNA and RNA blotting |
| Stratalinker 1800 | Stratagene | 400072 | UV crosslinker |
| High Prime | Roche | 11585592001 | kit for labeling of DNA with radioactive dCTP using random oligonucleotides as primers |
| illustra ProbeQuant G-50 Micro Columns | GE Healthcare | 28-9034-08 | chromatography spin-columns for purification of labeled DNA |
Referencias
- Hughes, S. H., Coffin, J. M. What Integration Sites Tell Us about HIV Persistence. Cell Host and Microbe. 19 (5), 588-598 (2016).
- Marini, B., Kertesz-Farkas, A., et al. Nuclear architecture dictates HIV-1 integration site selection. Nature. 521 (7551), 227-231 (2015).
- Cesana, D., Santoni de Sio, F. R., et al. HIV-1-mediated insertional activation of STAT5B and BACH2 trigger viral reservoir in T regulatory cells. Nature Communications. 8 (1), 498(2017).
- Cohn, L. B., Silva, I. T., et al. HIV-1 Integration Landscape during Latent and Active Infection. Cell. 160 (3), 420-432 (2015).
- Han, Y., Lassen, K., et al. Resting CD4+ T cells from human immunodeficiency virus type 1 (HIV-1)-infected individuals carry integrated HIV-1 genomes within actively transcribed host genes. Journal of Virology. 78 (12), 6122-6133 (2004).
- Ikeda, T., Shibata, J., Yoshimura, K., Koito, A., Matsushita, S. Recurrent HIV-1 integration at the BACH2 locus in resting CD4+ T cell populations during effective highly active antiretroviral therapy. The Journal of Infectious Diseases. 195 (5), 716-725 (2007).
- Wagner, T. A., Mclaughlin, S., et al. Proliferation of cells with HIV integrated into cancer genes contributes to persistent infection. Science. 345 (6196), 570-573 (2014).
- Maldarelli, F., Wu, X., et al. Specific HIV integration sites are linked to clonal expansion and persistence of infected cells. Science. 345 (6193), 179-183 (2014).
- Jordan, A., Bisgrove, D., Verdin, E. HIV reproducibly establishes a latent infection after acute infection of T cells in vitro. The EMBO Journal. 22 (8), 1868-1877 (2003).
- Mack, K. D., Jin, X., et al. HIV insertions within and proximal to host cell genes are a common finding in tissues containing high levels of HIV DNA and macrophage-associated p24 antigen expression. Journal of Acquired Immune Deficiency Syndromes. 33 (3), 308-320 (2003).
- Byrne, S. M., Ortiz, L., Mali, P., Aach, J., Church, G. M. Multi-kilobase homozygous targeted gene replacement in human induced pluripotent stem cells. Nucleic Acids Research. 43 (3), 1-12 (2014).
- ZhangLab. CRISPR Genome Engineering Toolbox: Target Sequence Cloning Protocol. Addgene website. , Available from: https://www.addgene.org/static/cms/filer_public/e6/5a/e65a9ef8-c8ac-4f88-98da-3b7d7960394c/zhang-lab-general-cloning-protocol.pdf (2013).
- Moser, F. Addgene. Gibson Assembly Protocol. Addgene website. , Available from: https://www.addgene.org/protocols/gibson-assembly/ (2009).
- Addgene Plasmid Cloning by PCR. Addgene website. , Available from: https://www.addgene.org/protocols/pcr-cloning/ (2014).
- Addgene Plasmid Cloning by Restriction Enzyme Digest (aka Subcloning). Addgene website. , Available from: https://www.addgene.org/protocols/subcloning/ (2013).
- Stemmer, M., Thumberger, T., Del Sol Keyer, M., Wittbrodt, J., Mateo, J. L. CCTop: An intuitive, flexible and reliable CRISPR-Cas9 target prediction tool. Public Library of Science (PLoS) ONE. 10 (4), (2015).
- Lange, U. C., Bialek, J. K., Walther, T., Hauber, J. Pinpointing recurrent proviral integration sites in new models for latent HIV-1 infection. Virus Research. 249, (2018).
- Bialek, J. K., Dunay, G. A., et al. Targeted HIV-1 Latency Reversal Using CRISPR-Cas9-Derived Transcriptional Activator Systems. PloS ONE. 11 (6), e0158294(2016).
- Lee, C. M., Davis, T. H., Bao, G. Examination of CRISPR-Cas9 design tools and the effect of target site accessibility on Cas9 activity. Experimental Physiology. 103 (4), 456-460 (2018).
- Jensen, K. T., Fløe, L., et al. Chromatin accessibility and guide sequence secondary structure affect CRISPR-Cas9 gene editing efficiency. FEBS Letters. 591 (13), 1892-1901 (2017).
- Simonetti, F. R., Sobolewski, M. D., et al. Clonally expanded CD4 + T cells can produce infectious HIV-1 in vivo. Proceedings of the National Academy of Sciences. 113 (7), 1883-1888 (2016).
- Chen, H. C., Martinez, J. P., Zorita, E., Meyerhans, A., Filion, G. J. Position effects influence HIV latency reversal. Nature Structural and Molecular Biology. 24 (1), 47-54 (2017).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados