Method Article
Microarrays de ADN y ARN de alta densidad - Síntesis fotolitográfica, hibridación y preparación de grandes bibliotecas de ácido nucleico
En este artículo
Resumen
En este artículo, presentamos y discutimos nuevos desarrollos en la síntesis y aplicaciones de microarrays de ácido nucleico fabricados in situ. Específicamente, mostramos cómo los protocolos para la síntesis de ADN se pueden extender al ARN y cómo se pueden utilizar los microarrays para crear bibliotecas de ácido nucleico recuperables.
Resumen
La fotolitografía es una potente técnica para la síntesis de oligonucleótidos de ADN en diapositivas de vidrio, ya que combina la eficiencia de las reacciones de acoplamiento de fosforamidia con la precisión y densidad de la luz UV reflejada a partir de espejos del tamaño de un micrometro. La fotolitografía produce microarrays que pueden acomodar desde cientos de miles hasta varios millones de secuencias de ADN diferentes, 100 o más, en sólo unas pocas horas. Con este espacio de secuencia muy grande, los microarrays son plataformas ideales para explorar los mecanismos de las interacciones de ácido nucleico, que son particularmente relevantes en el caso del ARN. Recientemente informamos sobre la preparación de un nuevo conjunto de fosamiditas de ARN compatibles con la fotolitografía in situ y que posteriormente se utilizaron para cultivar oligonucleótidos de ARN, homopolímeros, así como secuencias de base mixta. Aquí, ilustramos en detalle el proceso de fabricación de microarrays de ARN, desde el diseño experimental, hasta la configuración instrumental, la síntesis de matrices, la desprotección y el ensayo de hibridación final utilizando una secuencia de plantilla 25mer que contiene las cuatro bases como ejemplo. En paralelo, vamos más allá de los experimentos basados en la hibridación y explotamos la fotolitografía de microarrays como una puerta de entrada de bajo costo a las complejas bibliotecas de ácido nucleico. Para ello, los microarrays de ADN de alta densidad se fabrican en un monómero sensible a la base que permite que el ADN se corte y recupere convenientemente después de la síntesis y la desprotección. El protocolo de fabricación está optimizado para limitar el número de errores sintéticos y, en ese sentido, se introduce una capa de solución de caroteno para absorber fotones UV que de otro modo podrían reflejarse de nuevo en los sustratos de síntesis. Describimos paso a paso el proceso completo de preparación de la biblioteca, desde el diseño hasta el escote y la cuantificación.
Introducción
El uso práctico de microarrays de ADN ha sido tradicionalmente en el estudio de las variaciones en los niveles de expresión génica entre dos poblaciones celulares, utilizando hebras complementarias y fluorescencia como método de detección1. Ocasionalmente, los microarrays de ADN se aventuran en eventos de unión con ligandos ácidos no nucleicos, como proteínas, con una estrategia de permutación sistemática de la secuencia que ofrece una visión global del paisaje de unión2,3, 4,5. Este enfoque transforma eficazmente los microarrays de meras superficies de hibridación en plataformas con una amplia cobertura de secuencias, lo que sería un activo para el estudio del mundo más rico y complejo de la estructura y función del ARN. Apoyados por la reacción de acoplamiento de fosforamidida extremadamente eficiente6, las matrices de ADN sintetizadas in situ ahora también pueden considerarse como una fuente barata de ADN7,que se está volviendo particularmente relevante teniendo en cuenta la creciente demanda de material de ácido nucleico para ensamblaje genético8,9, nanoestructuras basadas en ADN10, almacenamiento de información o secuenciación11,12. Del mismo modo, es probable que las tecnologías de secuenciación se beneficien del desarrollo de métodos que produzcan mezclas muy complejas de oligonucleótidos de ARN13. En este contexto, los protocolos de fabricación de matrices que permiten sintetizar oligonucleótidos in situ y a alta densidad están en una posición ideal para satisfacer las necesidades del campo en rápida expansión de la biotecnología de ácido nucleico. Sin embargo, con un campo tan diverso como la biotecnología, el propósito de cada aplicación puede requerir que el ADN en microarray se produzca ya sea a alto rendimiento o con una cantidad muy baja de errores sintéticos14,15,o ambos, que requieren un más de cerca a los protocolos de síntesis de microarrays de ADN que, históricamente, se han optimizado principalmente para ensayos de hibridación. Mientras tanto, la síntesis in situ de microarrays de ARN se ha encontrado como un esfuerzo desafiante, con la mayor parte de la dificultad asociada con el grupo protector para la función 2'-OH, por lo general una mitad de silyl en la síntesis de fase sólida estándar que se elimina con reactivos a base de flúor, productos químicos incompatibles con superficies de vidrio o silicio. Esos problemas y desafíos en la síntesis de microarrays de ADN y ARN han sido últimamente objeto de un gran número de trabajo, en particular con el enfoque de fotolitografía16.
La fotolitografía utiliza luz UV para desbloquear oligonucleótidos antes del acoplamiento y requiere máscaras para construir un patrón de exposición UV, organizando y controlando espacialmente el crecimiento de oligonucleótidos. Las máscaras físicas han sido sustituidas por microespejos controlados por ordenador cuya inclinación refleja selectivamente la luz UV sobre el sustrato de microarray17,18,19. Como fuente UV, utilizamos 365 nm de luz de una fuente LED de alta potencia20. Las configuraciones fotolitográficas actuales están equipadas con matrices de microespejos que contienen 1024 x 768 espejos, correspondientes amás de 780.000 puntos direccionables individualmente ("características") en un área pequeña de sólo 1,4 cm 2, o 1080p con matrices de 1920 a 1080, o >2 millones de espejos. Por lo tanto, cada uno de los espejos del dispositivo tiene control directo sobre la secuencia cultivada en la función correspondiente. Con la excepción de la luz UV, la fotolitografía funciona como una técnica de síntesis de fase sólida y adopta la química de fosfomidito basada en ciclos. Sólo se requiere una estrategia de protección completamente diferente para que la síntesis de ARN tenga éxito. Desarrollamos una nueva serie de fosamiditas de ARN sensibles a la luz con grupos de protección de hidrazina-lábil21. Estos monómeros permiten desproteger el ARN en condiciones leves que no afectan la integridad de la superficie. Un primer paso de desprotección utiliza trietilamina para eliminar los grupos de protección de fosfodiester cianoetilo, mientras que la hidrazina se utiliza en un segundo paso separado para eliminar los de las funciones de amina 2'-OH y exocíclico. Al hacerlo, los oligonucleótidos de ARN de 30 nt de longitud y de cualquier secuencia ahora se pueden sintetizar in situ en microarrays22,23. Paralelamente, recientemente también comenzamos a abordar las cuestiones de rendimiento, calidad y velocidad en la fotolitografía de ADN y ARN. Medimos las eficiencias de acoplamiento >99% para todos los ADN y ARN amidites (Figura1) e investigamos cada paso individual en el ciclo de elongación de oligonucleótidos, desde el tiempo de oxidación, hasta la elección del activador y hasta la exposición UV óptima24 , 25. Hemos traído nuevos grupos de protección de 5' sensibles a la luz que se pueden eliminar sólo en segundos, transformando la síntesis de cientos de miles de 100 mers en un proceso de unas pocas horas de duración26. También hemos duplicado el rendimiento de la fabricación de matrices al exponer dos sustratos simultáneamente27. Por último, hemos introducido un fosamidito dT que contiene un grupo de saccinil sensible a la base como una forma conveniente de aplacar, recoger y analizar oligonucleótidos de ADN y ARN, que es fundamental para la preparación de la biblioteca28.
A pesar del aspecto relativamente mundano de la síntesis de fase sólida de ADN y ARN, especialmente para los químicos de ácido nucleico, la fotolitografía de microarray sigue siendo una actualización no trivial que requiere una configuración compleja, un control cuidadoso y una supervisión del proceso, y instrucciones separadas para el manejo post-sintético dependiendo de la naturaleza del oligonucleótido y el tipo de aplicación. En este artículo, queremos presentar detalladamente todo el procedimiento paso a paso de la síntesis in situ de microarrays de ADN y ARN por fotolitografía, desde el diseño experimental hasta el análisis de datos, con énfasis en la preparación de instrumentos y Consumibles. A continuación, describimos los métodos de desprotección postsintéticos que corresponden al propósito previsto de la fabricación de microarrays (es decir, la hibridación o la recuperación de bibliotecas de ácido nucleico).
Protocolo
1. Diseño de microarray
- Escriba las secuencias que se van a sintetizar en un editor de texto, 5 '-3', una línea por secuencia. Para el control de calidad 25mer, utilice la secuencia "GTCATCATCATGAACCACCCTTC". Utilice letras A, C, G y T para nucleótidos de ADN y números 5, 6, 7 y 8 para los nucleótidos de ARN.
- En el caso de la preparación de la biblioteca, añada un número adicional (es decir, 9) al final de 3' de cada secuencia. Esto corresponderá al acoplamiento del monómero sensible a la base.
- Cada secuencia debe ir seguida de una coma. Asigne un nombre a cada secuencia después de la coma y compruebe que cada carril sigue el formato [sequence,#sequence_name] (sin corchetes). Guarde la lista de secuencias como un archivo .txt.
- Inicie MatLab y cargue el programa ChipDesign.m. Ejecute el programa.
- En la ventana Panel de propiedades, cargue el archivo de diseño EntireChip de microarreglo. Cargue el archivo de diseño MilliChip si el objetivo es sintetizar toda la matriz en cuatro ubicaciones idénticas.
- En la ventana Panel de propiedades, en Especificaciónde chip , seleccione Seleccionar contenedor y, a continuación, Zona de síntesis. En Patrón, seleccione el patrón que produce la cantidad correcta de entidades direccionables, contando el número de secuencias , número de réplicas. Para el control de calidad 25mer, seleccione el patrón 25:36.
- En Seleccionar contenedor, seleccione Fiduciario. Las características fiduciales generalmente se sintetizan en las esquinas del área de síntesis y se utilizan para extraer datos de hibridación. Con Fiducial seleccionado, escriba una secuencia (5'a 3') que se sintetizará en las características fiduciales y que puede actuar como un control positivo. Para el experimento de 25mer, utilice la misma secuencia de ADN.
- En Secuencia, cargue el archivo de texto que contiene todas las secuencias escritas. Asegúrese de que Randomize esté seleccionado. Dar un título al experimento en El título del proyecto y, en Secuencia del vinculador, escribir TTTTT (correspondiente a un vinculador T5).
- Presione Generary, a continuación, busque los archivos de diseño y las máscaras de matriz (Figura 2) en la carpeta MaskGen_delta_rc1/Designs. Asegúrese de que contiene un script de visualización,una secuencia de flujo y un archivode diseño.
- Para la preparación de la biblioteca, abra el script de visualización. Agregue una línea adicional en la parte inferior del script de visualización que copie exactamente la primera línea del script (por ejemplo, mostrar First_Mask.bmp 150). Esto eliminará el grupo de fotoprotección terminal en el extremo de 5' de todos los oligonucleótidos.
- Inicie el programa AutoJob del creador de trabajos, cargue una plantilla haciendo clic en Cargar plantillay, a continuación, haga clic en Mostrar script para cargar el archivo de script de visualización generado por MatLab. Pulse Generar. Este paso creará una serie de instrucciones, llamadas jobfile, que controlarán la comunicación del ordenador con los microespejos y el sintetizador de ADN.
2. Preparación y funcionalización de diapositivas
- Taladre una diapositiva en dos posiciones correspondientes a la ubicación del tubo de entrada y salida en la celda de síntesis. Utilice una broca de diamante de 0,9 mm en un router CNC para taladrar con precisión y ficapacidad. Enjuague los portaobjetos perforados con agua ultrapura y colóquelos en una portaobjetos.
- Limpie las superficies sonicando los portaobjetos en un baño de agua que contenga el 5% de un limpiador de propósito especial a base de amoníaco durante 30 minutos a 35 oC. Enjuague el portaobjetos con H2O de doble destilado y transfieralo en un estante limpio y seco.
- Organice portaobjetos perforados y sin perforar en un portaobjetos. En un cilindro grande graduado, preparar la solución de funcionalización mezclando 475 ml de etanol (EtOH) con 25 ml de ddH2O, 10 g de reactivo silanizante (N-(3-triethoxysilylpropyl)-4-hidroxibutiramida) y 1 ml de ácido acético. Revuelva bien hasta que sea homogéneo y luego transfiera en un recipiente adecuado y cerrado.
- Coloque el bastidor cargado en el recipiente, cierre la tapa y deje que el recipiente se balancee suavemente sobre un agitador orbital durante 4 horas a temperatura ambiente.
- Después de 4 h, deseche la solución de funcionalización y sustitúyala por 500 ml de solución de lavado, consistente en 475 ml de EtOH, 25 ml de ddH2O y 1 ml de ácido acético. Agitar lentamente durante 20 minutos a temperatura ambiente, luego deseche y reemplace con 500 ml de solución de lavado fresco.
- Después de 20 minutos adicionales a temperatura ambiente, deseche la solución, seque los portaobjetos con una corriente de argón y cúnelos en un horno de vacío precalentado a 120 oC. Después de 2 h, apague el horno y la bomba de vacío, pero deje los portaobjetos bajo presión reducida durante la noche. A continuación, vuelva a poner el horno a la presión atmosférica y guarde los portaobjetos en un desecador hasta su uso posterior.
3. Preparación de reactivos de síntesis y reactivos
- Lleve los polvos de fosforamidito (Figura1) desde su temperatura de almacenamiento (-25 o -45 oC) a temperatura ambiente en un desecador.
- Cuando las fosfosamiditas alcanzaran la temperatura ambiente, disolver el polvo con un volumen de acetonitrilo ultraseco (<30 ppm H2O) para alcanzar una concentración de 30 mM para el ADN estándar y fosfosamiditas de ARN, y 50 mM para el monómero dT sensible a la base ( preparación de la biblioteca). Agregue una pequeña bolsa de tamices moleculares para atrapar cualquier rastro de humedad.
- Preparar una solución de 1% (p/p) imidazol en DMSO disolviendo 11 g de imidazol en 1 L de DMSO seco. Agitar bien hasta que se disuelva por completo. Conecte la solución al puerto auxiliar del sintetizador de ADN. Este será el disolvente de exposición necesario para la eliminación completa del grupo fotoprotector de 5o.
- Para la síntesis de las bibliotecas, prepare una solución de 1% (p/v) de caroteno en diclorometano diclorometano disolviendo 100 mg de caroteno en 10 ml de diclorometano. Agitar bien en una botella de vidrio ámbar y luego envolver en papel de aluminio.
4. Preparación y seguimiento de la síntesis de microarrays.
- Registre la temperatura y la humedad en la sala de fabricación de microarrays y asegúrese de que el sintetizador de ADN esté bajo suficiente presión de helio.
- Encienda el UV-LED y su ventilador de refrigeración. Fije un medidor de intensidad UV en el plano focal de la luz UV entrante y enciéndalo (Figura3A).
- En el equipo, inicie el software de controlador de archivos jobfile/micromirror/synthesis (denominado WiCell). Active y, a continuación, inicialice el dispositivo microespejo y cargue un archivo de máscara blanco haciendo clic con el botón derecho en DMDy, a continuación, seleccionando Cargar imagen.
- Haga clic con el botón derecho en el icono UVS y seleccione Obturador UV Abierto. Lea el valor de potencia (en mW/cm2) en el medidor de intensidad y cuente 60 s. Después de 60 s, lea el valor de potencia de nuevo y anote los valores inicial y final. Cierre el obturador seleccionando Uv Shutter Close y apague el medidor de intensidad. Calcular el valor medio de intensidad UV en mW/cm2.
- Calcular el tiempo de exposición necesario para alcanzar una energía radiante de 6 J/cm2 para la fotodesprotección 5'-NPPOC (microarrays de ADN
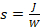 y ARN) y 3 J/cm2 para 5'-BzNPPOC fotodeprotection (bibliotecas de ADN), simplemente siguiendo la relación.
y ARN) y 3 J/cm2 para 5'-BzNPPOC fotodeprotection (bibliotecas de ADN), simplemente siguiendo la relación. - En el software DNA synthesizer Workstation, cree un archivo de secuencia en el Editor de secuencias copiando y pegando el contenido de la secuencia de flujo generada por MatLab. Agregue dos pasos de lavado adicionales en el extremo de 3' (letra de ciclo predeterminada: "s") para lavar la superficie de los sustratos antes de pasar al primer acoplamiento. Guarde y exporte el archivo de secuencia.
- En el Editor de protocolos del software Workstation, cree un protocolo que contenga un ciclo dedicado con el nombre de cada una de las letras y números presentes en el archivo de secuencia (por ejemplo, si un archivo de secuencia contiene solo letras A, C, G y T, el archivo de protocolo debe contienen cuatro ciclos, denominados A, C, G y T).
- Establezca el tiempo de acoplamiento para las fosfosamiditas de ADN (ciclos A, C, G y T) en 15 s, en 120 s para la fosamidito rU (ciclo 8) y en 300 s para las fosforamiditas rA, rC y rG (ciclos 5, 6 y 7). Para la preparación de la biblioteca, ajuste el acoplamiento del monómero dT escleable y sensible a la base a 2 x 120 s (Tabla 1 y Tabla 2).
- Asegúrese de que el tiempo de espera entre dos eventos de comunicación de salida del evento 2 en cada ciclo corresponde al tiempo de exposición UV calculado para alcanzar la energía radiante necesaria. En cuanto al archivo de secuencia, guarde y exporte el archivo de protocolo.
- En el software WiCell, cargue los archivos de trabajo, secuencia y protocolo en sus respectivas subventanas y, a continuación, haga clic en Enviar para enviar los archivos de secuencia y protocolo al sintetizador de ADN.
- En el archivo de secuencia, cuente el número de acoplamientos para cada fosforamidia.
- Para medir el volumen requerido (en L) de la solución de fosforamidia necesaria para realizar cada síntesis, multiplique el número de acoplamientos por 60. Añadir 250 l a cada volumen para la seguridad y el cebado de la línea en el sintetizador de ADN. Utilice un volumen base de 250 ml para fosforamiditas que requieran un solo acoplamiento.
- Transfiera rápidamente la solución a los viales del sintetizador de ADN correspondiente a la letra/número de puerto en los ciclos de protocolo.
- Prepara los puertos con las soluciones de fosforamidia con 10 pulsos primos cada uno para llenar las líneas con reactivo. Prepara la línea de lavado de acetonitrilo antes de conectar la célula de reacción.
- Para ensamblar la célula de síntesis, coloque primero una junta de perfluoroelastómero (FFKM) gruesa (250 m) en el bloque de cuarzo de la célula. Coloque una diapositiva de microscopio funcionaly perforada en la parte superior de la primera junta y verifique que los orificios de las diapositivas se conecten con el tubo de entrada y salida de la celda de síntesis.
- Coloque una segunda junta de politetrafluoroetileno (PTFE) delgada (50 m) sobre la corredera perforada, que rodea los dos orificios. Por último, coloque una segunda diapositiva funcionalizada pero no perforada sobre la segunda junta (Figura3B). Coloque un marco de metal de 4 tornillos en la parte superior de la celda de doble sustrato montada y apriete el tornillo a la misma fuerza de sujeción (0,45 Nm) con un destornillador de torsión.
- Fije el tubo de entrada y salida al sintetizador de ADN. Prepara la línea de lavado de acetonitrilo y verifica el flujo adecuado de acetonitrilo (ACN) a través de los sustratos. Desmontar y volver a montar la célula si se puede observar alguna fuga de ACN en esta etapa. Mida el volumen de ACN en la línea de residuos después de pasar por 7 ciclos de cebado ACN. Este volumen debe ser de 2 ml.
- Fije la celda de síntesis en el plano focal de la luz UV entrante. En el caso de la preparación de la biblioteca, coloque una línea de entrada y salida adicional en la parte posterior de la celda y llene la cámara posterior con la solución de caroteno (2 ml de solución es suficiente). Asegúrese de que no haya fugas (Figura3C).
- Inicie la síntesis haciendo clic primero en Ejecutar en el software WiCell. En el primer comando WAIT del archivo de trabajo, pulse Iniciar en el sintetizador de ADN.
- Durante la síntesis, verifique regularmente que la visualización de los archivos de máscara coincide con la exposición UV y la apertura del obturador.
- Después de la síntesis de microarrays regulares, desconecte la célula del sintetizador, desmonte la celda y use una pluma de diamante para grabar el número de síntesis en las diapositivas de vidrio. Etch el número en la cara no sintetizada de cada diapositiva. Transfiera los portaobjetos a tubos centrífugos de 50 ml y guárdelos en un área desecada hasta su uso posterior.
- Después de la síntesis de microarrays de la biblioteca, primero drenar la solución de caroteno de la cámara y luego lavarla fluyendo 2 x 5 ml de CH2Cl2y luego drenar. Continúe con el desmontaje según el paso 4.21. La matriz sintetizada puede ser visible a simple vista (Figura4).
5. Desprotección de microarray de ADN
- Llenar un frasco de vidriera con 20 ml de EtOH y 20 ml de etilendiamina (EDA). Coloque los microarrays solo de ADN verticalmente en el frasco, cierre la tapa y deje que los portaobjetos se desprotejan durante 2 h a temperatura ambiente.
ADVERTENCIA: La AEC es un líquido agudamente tóxico, corrosivo e inflamable. Trabaje con guantes en una campana de humo bien ventilada. - Después de 2 h, recupere las correderas con pinzas y enjuáguelas bien con H2O de doble destilado.
- Seque los portaobjetos en una centrífuga de microarray durante unos segundos y luego guárdelos en un desecador.
6. Desprotección del microarray de ARN
- En un tubo centrífugo de 50 ml, prepare una solución seca de 2:3 trietilamina/ACN (20 ml y 30 ml de cada uno, respectivamente). Transfiera una diapositiva de microarray de ARN al tubo centrífugo, cierre la tapa y luego envuelva con película de sellado de plástico. Agitar suavemente el tubo centrífugo en un agitador orbital durante 1h y 30 min a temperatura ambiente.
- Después de 1h y 30 min, retire el portaobjetos, lave con 2 x 20 ml de ACN seco y luego seque en una centrífuga de microarray durante unos segundos.
ADVERTENCIA: La trietilamina es un líquido agudamente tóxico, corrosivo e inflamable. El acetonitrilo es tóxico e inflamable. Trabaje con guantes en una campana de humo bien ventilada.
NOTA: Primer paso de desprotección completado. - Preparar una solución hidrahidrata de 0,5 M en 3:2 piridina/ácido acético. En primer lugar, mezclar 20 ml de ácido acético y 30 ml de piridina en un cilindro graduado. Espere a que la solución se enfríe antes de añadir 1,21 ml de hidrazina hidrahidrato. Transfiera alrededor de 40 ml de la solución resultante a un tubo centrífugo de 50 ml.
ADVERTENCIA: El hidrato de hidrazina es un líquido agudamente tóxico y corrosivo. La piridina es altamente inflamable y agudamente tóxica. El ácido acético es inflamable y corrosivo. Trabaje con guantes en una campana de humo bien ventilada. - Después del primer paso de desprotección, transfiera la diapositiva de ARN a la solución de hidrazina hidrahidrato, cierre la tapa y envuelva con película de sellado de plástico. Agitar suavemente el tubo en un agitador orbital durante 2 h a temperatura ambiente. Después de 2 h, retire el portaobjetos, lave con 2 x 20 ml de ACN seco y luego seque en una centrífuga de microarray durante unos segundos.
NOTA: Segundo paso de desprotección completado. - Si el microarray de ARN también contiene nucleótidos de ADN, proceda con un tercer paso de desprotección. En un tubo centrífugo de 50 ml, mezcle 20 ml de EDA con 20 ml de EtOH. Agregue el microarray DNA/RNA a la solución 1:1 EDA/EtOH y déjela a temperatura ambiente durante 5 min.
- Después de 5 min, retire el portaobjetos, lave con 2 x 20 ml de agua estéril y luego seque en una centrífuga de microarray y guárdelo en un desecador.
7. Hibridación con una hebra complementaria con etiqueta fluorescente
- Descongelar la albúmina sérica bovina acetilada (10 mg/ml) y la hebra complementaria con etiqueta Cy3 (100 nM) y calentar hasta temperatura ambiente.
- En un tubo de microcentrífuga estéril de 1,5 ml, mezcle 150 ml de 2 tampón MES (200 mM 2-(N-morpholino)ácido etanosulfónico; 1,8 M NaCl; 40 mM EDTA; 0,02% Tween-20) con 26,7 l de ADN etiquetado en Cy3, 13,4 ml de BSA acetilado y 110 l de mezcla estéril De2O. Vórtice. Doble el volumen si ambas diapositivas se van a utilizar para la hibridación.
- Precaliente un horno de hibridación a una temperatura por debajo de la Tm del dúplex pero lo suficientemente alta como para garantizar una buena discriminación entre secuencias de coincidencia completa y no igualadas. Para el control de calidad 25mer, ajustela temperatura a 42 oC (Tm de 59 oC).
- Coloque cuidadosamente una cámara de hibridación autoadhesiva de 300 l sobre el área de síntesis de cada diapositiva y pipeta en la solución de hibridación preparada anteriormente. Cubra los orificios de la cámara con puntos adhesivos y envuelva toda la corredera en papel de aluminio.
- Coloque la diapositiva del microarray en el horno de hibridación, cubra y deje que gire suavemente a la temperatura de hibridación seleccionada durante 2 h.
- Después de 2 h, desenganche la corredera, retire la lámina de aluminio y retire cuidadosamente la cámara de hibridación. Transfiera las correderas a un tubo centrífugo que contenga 30 ml de tampón de lavado no estricto (NSWB; 0,9 M NaCl, 0,06 M de fosfato, 6 mM EDTA, 0,01% Tween20, pH 7,4) y agitar vigorosamente durante 2 minutos a temperatura ambiente.
- Transfiera la corredera en un tubo centrífugo que contenga 30 ml de tampón de lavado estricto (SWB; 100 mM MES, 0,1 M NaCl, 0,01% Tween20) y agítese vigorosamente durante 1 min.
- Finalmente, transfiera la corredera en un tubo centrífugo que contenga 30 ml de tampón de lavado final (FWB; 0,1x citrato de solución salina sódica) y agite durante unos segundos. Seque el portaobjetos en una centrífuga de microarray.
- Coloque el microarray seco, área de síntesis hacia abajo, en el portaobjetos del escáner de microarray. Para dúplex con etiqueta Cy3, escanee a una resolución de 5 m con una longitud de onda de excitación de 532 nm, un filtro de 575 nm y un fotomultiplicador de 350. Guarde el análisis de alta resolución como un archivo de imagen .tif (figura5A).
8. Extracción y análisis de datos
- Antes de la extracción de datos, gire el escaneo de matriz guardado en un editor de imágenes para situar la cadena más larga de entidades fiduciales en la esquina superior izquierda del escaneo. Guarde la imagen girada.
- Inicie NimbleScany, a continuación, pulse Archivo . Abra y cargue el análisis de la matriz. A continuación, haga clic en Examinar en la subsección Archivo de diseño, cargue el archivo . ndf archivo de diseño que se generó automáticamente durante el diseño del experimento de microarray. A continuación, haga clic en Abrir.
- En Vista, haga clic en Contraste/Brillo automático . Haga clic en el icono Alinear manualmente encima del escaneo. Coloque cuatro marcadores de forma cuadrada en las cuatro esquinas del escaneo y, a continuación, vuelva a hacer clic en el icono verde. Extraiga los datos de hibridación haciendo clic en Análisis, Informes y, a continuación, Informe de sondeo.
- Abra el archivo . archivo de informe de sondeo en un editor de hojas de cálculo. Mantenga las columnas B e I y descarte el resto antes de proceder con el cálculo de los valores medios y la desviación estándar de los datos extraídos (Figura5B).
9. Desprotección, escote y recuperación de la biblioteca
- Para desproteger y cortar las bibliotecas de ADN, prepare una solución de 1:1 EDA/tolueno seco en un tubo centrífugo de 50 ml. Sumerja la corredera en la solución de escisión, cierre la corredera y envuelva con película de sellado de plástico, luego gire suavemente en un agitador orbital durante 2 h a temperatura ambiente.
- Después de 2 h, retire el portaobjetos y lave con 2 x 20 ml de ACN escrupulosamente seco. Retire la corredera y déjela secar al aire.
- Con una pipeta, aplique 100 ml de H2O estéril sobre el área de síntesis ahora discernible. Pipeta la solución hacia arriba y hacia abajo unas cuantas veces antes de transferirla a un tubo de microcentrífuga de 1,5 ml. Repita el proceso y combine el eluivar en el mismo tubo (Figura6).
- Evaporar los 2 x 100 l de eluido de viruta a la sequedad y luego volver a disolver en 10 l de H2O libre de nucleasas.
- Deslízate la biblioteca de ADN sobre una punta de pipeta de 10 l equipada con resina C18. En primer lugar, transforme el eluido de virutas en una solución tamponada de acetato de trietilammonio de 0,1 M (TEAA).
- Humedezca la resina aspirando 3 x 10 l de H2O/ACN 1:1, y equilibre la resina lavando con 3 x 10 l de tampón TEAA de 0,1 M. Enlazar el ADN mediante la pipeteado del eluido de viruta 10 veces arriba y abajo a través de la resina. Lavar la resina con 3 x 10 l de tampón TEAA de 0,1 M, 3 x 10 l de H2O.
- Eluye el ADN desalado de la punta lavando la resina con 10 sl de H2O/ACN 1:1. Secar la solución dessalada y volver a disolver en 10 ml de H2Oestéril. Evaporar la biblioteca a la sequedad y almacenar a -20 oC hasta su uso posterior.
Resultados
Hibridación de microarrayde ADN y ARN
La Figura 5 muestra los resultados de un ensayo de hibridación realizado en un microarray que contiene las versiones de ADN y ARN de una secuencia de 25 mer (5'-GTCATCATCATGAACCACCCTTC-3' en forma de ADN). El escaneo en la Figura 5A aparece en un formato de escala verde correspondiente al espectro de excitación/emisión de fluorescencia Cy3, con intensidad de fluorescencia registrada en unidades arbitrarias entre 0 y 65536. El diseño de la matriz siguió el diseño de la característica 25:36 descrito en la sección del protocolo. El escaneo se muestra después de la orientación adecuada de la matriz, con la esquina superior izquierda poblada con la cadena más larga de entidades fiduciales. Aquí, las características fiduciales contienen la versión de ADN del 25mer y, en principio, siempre deben dar una señal de fluorescencia positiva con el fin de realizar la alineación del escaneo y la extracción de datos. El microarray hibridado debe parecer uniformemente brillante, con los bordes del área de síntesis siendo sin embargo generalmente más brillante que el centro (hasta un 50% más brillante). La gran cantidad de réplicas de secuencia, distribuidas aleatoriamente por toda la zona, reduce el impacto de los artefactos espaciales. Aquí, cada secuencia (ADN y ARN) fue sintetizada en 2.000 ubicaciones aleatorias. Por lo general, hay un bajo ruido de fluorescencia (fondo), <50 a.u., que conduce a una relación señal/ruido en el orden de 200:1 a 800:1 en ensayos de hibridación. Después de la extracción de los datos, las intensidades de fluorescencia se promedian y se trazan sD.
Hay una variabilidad significativa en los valores absolutos de fluorescencia entre experimentos. Aquí, mostramos los resultados de tres síntesis independientes usando los mismos parámetros de fabricación y el mismo manejo post-sintético. El ADN de 25mer, cuando se hibrida a su cadena de ADN con etiqueta Cy3 complementaria, producirá señales de fluorescencia que van desde 20.000 hasta 30.000, muy raramente por encima o por debajo. El ARN de 25 mer, cuando se hibrida al mismo complemento de ADN etiquetado con Cy3, dará intensidades de fluorescencia en las características correspondientes que van de 15.000 a 20.000. Sin embargo, la intensidad de la fluorescencia de los dúplex de ARN/ADN ocasionalmente caerá por debajo de 8.000, cuando los dúplex de ADN/ADN correspondientes seguirán fluorado dentro del rango de 20.000-30.000. En tales casos, los resultados del ARN pueden considerarse subóptimos. Una falla de síntesis o hibridación, ya sea para ADN o ARN, será inmediatamente perceptible durante el escaneo de la evidente falta de fluorescencia. Hay múltiples oportunidades para que la síntesis de ARN fracase o tenga éxito parcialmente y se esbozarán en la parte de discusión.
Desprotección, escote y recuperación de bibliotecas
Dependiendo de la complejidad y la densidad de la biblioteca, la forma y el contorno de la matriz sintetizada se pueden ver sin aumento pero bajo la iluminación adecuada (Figura4), con el ADN todavía en forma protegida. Después de la desprotección con EDA/tolueno, y antes de la adición de agua para recoger la biblioteca de sin levadura, el área de síntesis que contiene los oligonucleótidos ahora desprotegidos puede destacar como una zona hidrófila, cuando el resto del tobogán de vidrio aparecerá cubierto con una capa hidrófoba turbia. La observación directa del área de síntesis depende de la superficie total utilizada para sintetizar oligonucleótidos: un mayor uso del área de síntesis corresponderá a una mayor probabilidad de una clara distinción entre las regiones hidrófilas e hidrófobas en la superficie. Por el contrario, las bibliotecas sintetizadas con menos espejos y con características más pequeñas pueden no ser observables inmediatamente.
Del mismo modo, la cantidad de ADN recuperado después de la desalación es directamente proporcional a la superficie total utilizada para la síntesis. Si todas las características se utilizan para la síntesis de oligonucleótidos, el procedimiento de escisión y recuperación debe producir entre 25 y 30 pmol de ADN. Por lo tanto, un uso del 10% del área de síntesis sólo permitirá alrededor de 3 pmol de ADN.
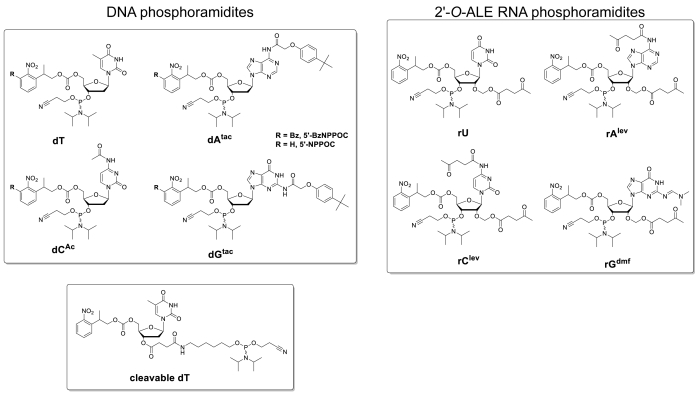
Figura 1 . Estructuras químicas del ADN y ARN fosamiditas utilizadas en la síntesis de oligonucleótidos por fotolitografía de microarray. Los grupos de protección fotosensibles estándar de nitrophenylpropyloxycarbonyl (NPPOC) en el 5'-OH se utilizan en la síntesis regular de ADN y microarray de ARN para fines de hibridación. Para la síntesis de bibliotecas de ADN complejas, más fotolabile-benzyl-NPPOC (BzNPPOC) se prefieren en el 5'-OH, ya que BzNPPOC se elimina dos veces más rápido que NPPOC, lo que reduce significativamente el tiempo total de síntesis de microarray. Los oligonucleótidos de ADN para bibliotecas también requieren el acoplamiento de un monómero dT escleable en el extremo de 3'. Este monómero, que lleva una función de éster de sacapinilo, será cleavedd durante la desprotección, lo que permite que el ADN sea recogido del microchip. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2 . Ejemplo de una máscara como un archivo de imagen enviado al dispositivo microespejo durante la exposición UV. Los píxeles blancos corresponden a los espejos que se inclinarán en la posición "ON", reflejando la luz UV en la celda de síntesis. Los píxelesnegros corresponden a los espejos "OFF", donde la luz UV se reflejará lejos de la celda. Por lo tanto, los píxeles blancos permitirán el acoplamiento del siguiente fosforamidia entrante en los oligonucleótidos que se encuentran en las características correspondientes en los sustratos de vidrio. Oligonucleótidos sintetizados en las características cuyos espejos correspondientes son, en este archivo de máscara, los píxeles negros permanecerán sin embargo inertes durante el próximo evento de acoplamiento. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3 . Fotografías de la configuración óptica y de síntesis de fotolitografíade microarray. (A) Circuito óptico para la exposición UV. La luz UV del UV-LED se homogeneiza primero a través de un tubo de luz rectangular de sección transversal y luego se refleja en los microespejos. Los microespejos que se han inclinado en una posición "OFF" reflejarán la luz UV lejos de la celda de síntesis, pero los microespejos en la posición "ON" reflejarán la luz en la celda de síntesis, situada en el plano focal, al pasar primero a través de un relé Offner 1:1 sistema de imágenes. (B) La celda de síntesis, una vez montada, consiste en una corredera perforada colocada en primer lugar sobre el bloque de cuarzo de la célula, separada por una junta de PTFE gruesa (no se muestra). A continuación, se coloca una segunda diapositiva no perforada sobre la diapositiva perforada, separada por una junta de PTFE delgada. Un marco metálico (no se muestra) mantiene el ensamblaje unido. (C). Para la preparación de la biblioteca, una vez que la célula de síntesis está unida en el plano focal de la luz UV entrante, la cámara situada entre el bloque de cuarzo y la corredera perforada se llena con una solución del 1% de caroteno en CH2Cl2. Para ello, se une un tubo de entrada y salida adicional al bloque de cuarzo y la solución naranja fluye de la posición más a la derecha a la posición más a la izquierda. El flujo de reactivos y disolventes para la síntesis se muestra en flechas blancas. Haga clic aquí para ver una versión más grande de esta figura.

Figura 4 . El área de síntesis es generalmente visible a simplevista. Aquí, una biblioteca de ADN se puede ver en la superficie de vidrio justo después de la síntesis, con el ADN todavía en forma protegida. Haga clic aquí para ver una versión más grande de esta figura.
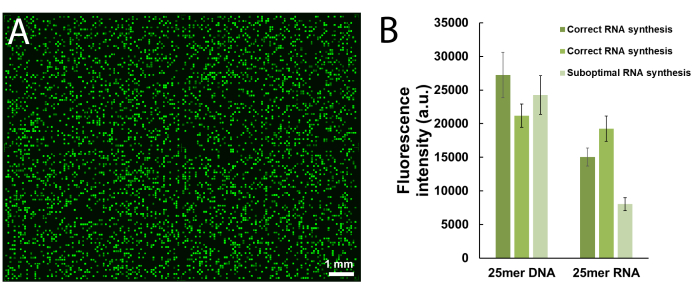
Figura 5 . Ensayos de hibridación a las secuencias de ADN y ARN de 25mer sintetizadas in situ en microarrays. (A) Escaneo de fluorescencia de todo el ADN hibridado y microarray de ARN. Los oligonucleótidos de ADN y ARN de 25mer se hibridan en sus hebras complementarias etiquetadas con Cy3. La matriz fue escaneada con un láser a una longitud de onda de excitación de 532 nm, a una resolución de 5 m. (B) Intensidades de fluorescencia (unidades arbitrarias) de los dúplex DNA:DNA y RNA:DNA en tres experimentos separados. Los datos verdes claros de los oligonucleótidos de ARN sintetizados in situ pueden considerarse subóptimos, en comparación con la intensidad de fluorescencia de las secuencias de ADN correspondientes. Las barras de error son SD. Por favor, haga clic aquí para ver una versión más grande de esta figura.
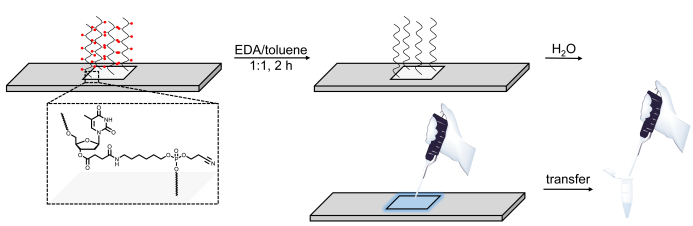
Figura 6 . Representación esquemática del procedimiento de desprotección, escisión y recuperación de las bibliotecas de ADN sintetizadas en microarrays. Las secuencias de ADN se cultivan en un nucleósido dT escinible sensible a la base (que se muestra en el área ampliada). Después de la síntesis, la desprotección de los oligonucleótidos de ADN (los grupos protectores de bases se representan como esferas rojas) en EDA/tolueno deja el material desprotegido unido electrostáticamente a la superficie y luego se puede pipetear aplicando una pequeña agua en el área sintetizada. Haga clic aquí para ver una versión más grande de esta figura.
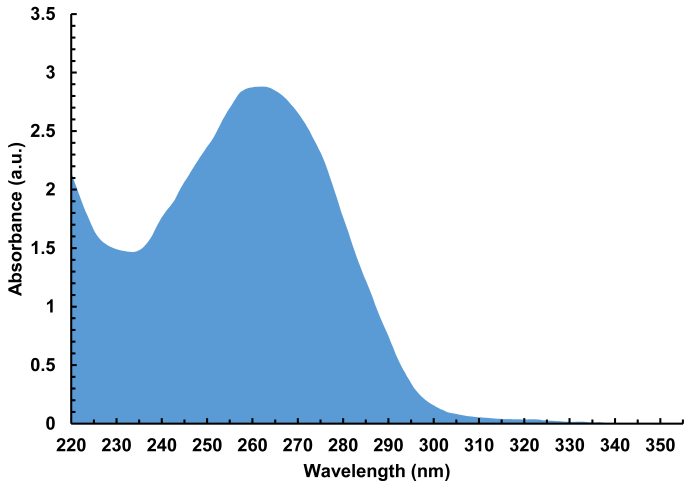
Figura 7 . Espectro de absorbancia representativo (220 - 350 nm) de una biblioteca de ADN desalinada y sin levadura que contiene 4.000 secuencias diferentes, de 100 nt de longitud. Un total de 940 ng de ADN se aisló de una sola síntesis de matriz, correspondiente a 30 pmol de ADN total, o 15 pmol por sustrato de vidrio. Haga clic aquí para ver una versión más grande de esta figura.
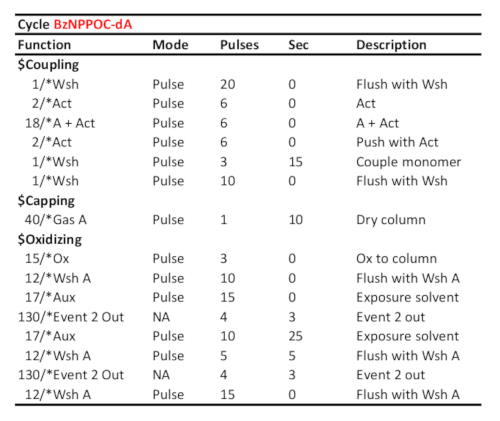
Tabla 1. Protocolo de ciclo representativo para el acoplamiento/oxidación/fotodesprotección de 5'-BzNPPOC-dA, suponiendo que el fosforamidito correspondiente se cargó en el puerto "A". El tiempo de acoplamiento (en segundos) se muestra en la línea "couple monomer". El tiempo de protección de fotodesprotección UV, aquí correspondiente a una energía radiante de 3 J/cm2 (fotoquímica BzNPPOC), se calcula como el tiempo transcurrido entre las dos señales de comunicación "Evento 2 Out".
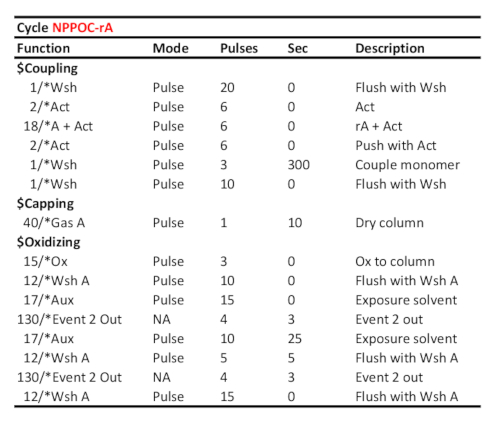
Cuadro 2. Protocolo de ciclo representativo para el acoplamiento/oxidación/fotodesprotección de 5'-NPPOC-rA, suponiendo que el fosamidito de ARN correspondiente se cargó en el puerto "A". El tiempo de acoplamiento (en segundos) se muestra en la línea "couple monomer". El tiempo de protección de fotodesprotección UV, aquí correspondiente a una energía radiante de 6 J/cm2 (fotoquímica NPPOC), se calcula como el tiempo transcurrido entre las dos señales de comunicación "Evento 2 Out".
Discusión
La síntesis de ADN y ARN en fase sólida es el pan y la mantequilla de todos los laboratorios de química de ácido nucleico, y aunque la adición del componente de fotolitografía es ciertamente una operación compleja, la fabricación de microarrays mediada por la luz UV es también un proceso muy confiable . Es, además, el único método disponible para la síntesis de ARN in situ en microarrays. Sin embargo, como en cualquier procedimiento experimental de varias etapas, hay un amplio espacio para el error humano.
Tal vez el paso más crítico es el acoplamiento de una fosforamidia, ya que necesita ser una reacción química de alto rendimiento constantemente con el fin de permitir oligonucleótidos con pocos errores sintéticos. En nuestro protocolo de síntesis de microarrays, el acoplamiento de fosforamidia es aún más fundamental para la calidad de síntesis general, ya que el proceso de fabricación evita el taponamiento y evita la purificación de oligonucleótidos. Ocasionalmente se han calculado eficiencias de acoplamiento escalonadas superiores al 99% para todas las fosfosamiditas fotosensibles de ADN y ARN, incluso para tiempos de acoplamiento muy cortos (15 s)24, pero ocasionalmente pueden producirse rendimientos de acoplamiento más bajos, especialmente en el caso de las dG amiditas. La estabilidad de las fosfosamiditas solubilizadas a temperatura ambiente se ha investigado antes y se demostró que dependía de la naturaleza de la nucleobase, con fosfosamiditas de guanosina propensas a una degradación extensiva en cuestión de días29, 30. Pero cuando se almacena a -25 oC, se encontró que las fosfosamiditas dG se disolvieron en ACN, ya que la solución de 30 mM era estable durante varias semanas. Sin embargo, la relativa inestabilidad de las soluciones de fosforamidia dG a temperatura ambiente significa que no deben mantenerse unidos al sintetizador de ADN durante varios días.
En el caso de las fosfosamiditas de ARN, el rendimiento del acoplamiento depende en gran medida de la calidad de la fosforamidia (que puede evaluarse mediante espectroscopia de RMN de 31P) y del tiempo de acoplamiento. Los tiempos de acoplamiento de 5 minutos para rA, rG, rC y 2 min para rU parecen necesarios. De hecho, descubrimos que acortar el tiempo de condensación a 2 min para todas las fosfosamiditas de ARN condujo a señales de hibridación significativamente más bajas.
El sintetizador de ADN en sí, así como los reactivos y disolventes, sin duda necesita ser lo más limpio posible con el fin de lograr el mayor rendimiento de síntesis de oligonucleótidos. Sin embargo, el material insoluble, sales o partículas, puede acumularse con el tiempo en las líneas y tubos del sistema de suministro, lo que conduce a una disminución gradual en el consumo de reactivos y reactivos. Cuando una limpieza general del sintetizador no resuelve un volumen de salida bajo, un aumento en el número de pulsos puede ser una solución alternativa. Especialmente útil en el caso de bajo consumo de fosforamidia, la línea del protocolo de acoplamiento correspondiente al bombeo de una mezcla de fosforamidia y activador (tercera línea de la subsección de acoplamiento en los cuadros 1 y 2)puede ser modificado, de 6 a 9 pulsos sin ningún efecto negativo apreciable en la calidad de síntesis. Además, el número de pulsos de activador necesarios para llevar la mezcla de amidito/activador al sustrato de síntesis (actualmente 6, cuarta línea en la subsección de acoplamiento, ver Tabla 1 y Tabla 2) depende del propio sintetizador de ADN, así como del propio sintetizador de ADN, así como de longitud del tubo en la célula de síntesis. Este número se puede ajustar después de reemplazar el fosforamidito con una solución de color y contar el número de pulsos necesarios para empujar la mezcla de colores al sustrato de vidrio para el acoplamiento.
El método descrito en este documento permite que la síntesis de ADN y ARN continúe simultáneamente, en el mismo microarray. Los híbridos de ADN y ARN también se pueden preparar sin ningún cambio en los protocolos de fabricación de arreglos de discos, y siempre y cuando se siga el protocolo de desprotección en tres pasos. Sin embargo, cabe señalar que los microarrays sólo de ARN sólo requieren una desprotección en dos pasos: una decyanoetilación primero con Et3N seguida de hidroxilo y desprotección de la base con hidrazina. Se encontró que las nucleobases de ADN estaban incompletamente desprotegidas en esas condiciones, y necesitaban el paso adicional en LA AED para llevar a cabo la eliminación completa de los grupos de fenoxiacetil (Pac). Este tratamiento adicional con EDA es más corto (5 min) que para la desprotección estándar de los microarrays de ADN31,pero es suficiente para llevarlo a término después de los tratamientos de trietilamina e hidrazina. Además, un corto tiempo de reacción con EDA limita la exposición de un oligonucleótido de ARN totalmente desprotegido a condiciones básicas.
Una ventaja de la síntesis in situ de la matriz de ARN sobre métodos alternativos como la mancha o transcripción de ADN32,33,34 es la capacidad de almacenar el microchip de ARN sintetizado en forma protegida hasta su uso, evitando así el riesgo de degradación potencial del ARN. Los procedimientos postsintéticos para el ARN implican, por otro lado, que los consumibles y reactivos se mantienen estériles y que la manipulación se realiza en condiciones libres de RNase. Cabe destacar que encontramos que la adición de inhibidor de RNase a la mezcla de hibridación no produjo señales de hibridación más fuertes para las características del ARN.
La síntesis de bibliotecas de ADN en un monómero sensible a la base es más compleja que la síntesis de unas pocas secuencias de control en una superficie, y como tal es ciertamente más propensa a errores de diseño. Sin embargo, suponiendo que el diseño de la secuencia (es decir, la naturaleza y el número de secuencias) es correcto, transformar esta lista en una colección de máscaras de exposición y una serie ordenada de ciclos de acoplamiento sigue siendo un proceso sencillo. Sin embargo, existen variaciones importantes de la síntesis de microarrays estándar que son fundamentales para una fabricación exitosa de una matriz de bibliotecas de alta densidad.
En primer lugar, un monómero dT sensible a la base se acopla como el primer fosforamidito después de la síntesis del vinculador. Se encontró que el rendimiento de acoplamiento de este monómero (Figura1) era relativamente bajo, alrededor del 85%28,por lo que se hacen esfuerzos para mejorar su tasa de incorporación, ya sea aumentando su concentración en ACN de 30 mM a 50 mM, o repitiendo la paso de acoplamiento: dos reacciones de acoplamiento consecutivas utilizando monómeros frescos, o dos ciclos de acoplamiento separados pero consecutivos.
El segundo cambio es la adición de una solución de caroteno en la cámara posterior de la célula de síntesis, que absorbe convenientemente la luz de 365 nm. Esta es una modificación importante de la configuración de la fotolitografía, ya que evita que la luz UV se refleje de nuevo en el sustrato de la matriz. De hecho, después de atravesar el medio intersticial entre los sustratos, la luz UV entrante sale a través de la diapositiva perforada y llega al bloque de cuarzo de la célula. Las ecuaciones de Fresnel predicen que el 4 % de la luz UV perpendicularmente incidente se reflejará desde cada una de las tres interfaces de aire-vidrio aguas abajo (lado de salida del 2o sustrato y ambos lados del bloque de cuarzo) y de nuevo al sustrato de síntesis, exposición involuntaria de oligonucleótidos fotoprotegidos. La difracción y la dispersión también contribuyen a la fotodesprotección "fuera del objetivo" y, por lo tanto, a la inserción de nucleótidos, lo que afecta directamente a la tasa de error de síntesis, pero estas contribuciones son mucho más pequeñas que las reflexiones y pueden ser abordadas principalmente por reducción de la densidad de síntesis (dejando huecos entre las entidades). Hemos encontrado que el nivel de solución de caroteno en la cámara inferior de la célula tiende a disminuir ligeramente sólo durante los primeros minutos de síntesis de matriz, y por lo tanto necesita ser monitoreado y reajustado.
Por último, el tercer cambio es la solución de desprotección, reemplazando etOH por tolueno, que mantiene la biblioteca de ADN desculada unida a la superficie, presumiblemente a través de interacciones electrostáticas. La aplicación de una pequeña cantidad de agua en el área de síntesis después del lavado a ACN permite que la biblioteca se recopile convenientemente. Sin embargo, el proceso sólo tiene éxito si el contenido de agua en EDA y tolueno es mínimo, haciendo que el ácido nucleico sea totalmente insoluble en el cóctel de desprotección. Alternativamente, las bibliotecas de ADN pueden ser arrancadas del chip usando amoníaco9,10,14,35, y luego desprotegidas calentando la solución de amoníaco acuoso que contiene ADN a 55 oC durante la noche. Sin embargo, la recuperación de bibliotecas de ADN que utilizan amoníaco no es compatible con el ARN. Los oligonucleótidos de ARN sobre un sustrato escleable en base se pueden eluir de la superficie utilizando el mismo procedimiento EDA/tolueno descrito anteriormente, pero sólo en la penúltima etapa después de la estrategia de desprotección de dos pasos Et3N e hidrazina28.
Las estrategias alternativas para recuperar piscinas de oligonucleótidos de microarrays sin necesidad de un tratamiento básico específico existen, son en principio compatibles con la fotolitografía y se basan en el uso de enzimas. Por ejemplo, un solo nucleótido desoxiuracilo puede ser el objetivo de la glicosilasa de ADN uracilo (UDG) y extirpado del resto de la secuencia de ADN, o una sola unidad de ARN puede ser reconocida por las enzimas RNase H tipo 2 y el enlace de fosfodiester 5' al ARN , liberando la parte23de ADN de 5' .
Ahora tenemos un método potente, confiable y de alta densidad para la síntesis de ADN, ARN y microarrays híbridos de ADN/ARN. Estos no sólo pueden servir como plataformas para la hibridación o ensayos de unión36,sino que también representan una manera rápida y económica de producir bibliotecas complejas de ácido nucleico. Para el almacenamiento de datos digitales basado en EL ADN, la fotolitografía de microarray puede convertirse en una solución potencial para el cuello de botella de "escritura" (es decir, para la codificación de la información por síntesis). El éxito en la codificación digital en el ADN y en el ensamblaje del gen de novo depende de la fidelidad de la secuencia que, a nivel de síntesis, se traduce en la tasa de error. Los errores sintéticos y ópticos en nuestros protocolos actuales de fabricación de matrices serán discutidos e informados en otros lugares. Paralelamente, se están realizando esfuerzos para aumentar aún más la escala y el rendimiento de fabricación.
Divulgaciones
Los autores certifican que no tienen afiliación con ninguna organización con fines de lucro.
Agradecimientos
Este trabajo fue apoyado por el Fondo de Ciencia de Austria (el FWF concede a P23797, P27275 y P30596) y a la Fundación Nacional Suiza para la Ciencia (Grant #PBBEP2_146174).
Materiales
| Name | Company | Catalog Number | Comments |
| Slide functionalization | |||
| Acetic acid >99.8% | Sigma | 33209 | For RNA deprotection |
| CNC router | Stepcraft | 300 CK | |
| Ethanol absolute | VWR | 1.07017.2511 | For deprotection and functionalization |
| N-(3-triethoxysilylpropyl)-4-hydroxybutyramide | Gelest | SIT8189.5 | Silanizing reagent |
| Nexterion Glass D microscope slides | Schott | 1095568 | |
| Polymax 1040 | Heidolph | Orbital shaker | |
| Proclean 507 Ultrasonic water bath | Ulsonix | To clean slides after drilling | |
| Tickopur RW 77 Special Purpose Cleaner | Sigma | Z860086 | To clean slides after drilling |
| Microarray synthesis | |||
| 0.25 M dicyanoimidazole in ACN | Biosolve | 0004712402BS | Activator |
| 0.7 XGA DMD | Texas Instruments | Digital Micromirror Device | |
| 20 mM I2 in pyridine/H2O/THF | Sigma | L860020-06 | Oxidizer |
| 250 μm thick Chemraz 584 perfluoroelastomer | FFKM | Lower teflon gasket | |
| 2'-O-ALE RNA phosphoramidites | ChemGenes | ||
| 365 nm high-power UV-LED | Nichia | NVSU333A | |
| 5'BzNPPOC DNA phosphoramidites | Orgentis | ||
| 5'NPPOC DNA phosphoramidites | FlexGen | ||
| Acetonitrile | Biosolve | 0001205402BS | For DNA synthesis |
| Amidite Diluent for DNA synthesis | Sigma | L010010 | For dissolving phosphoramidites |
| Cleavable dT | ChemGenes | Base-sensitive monomer for library preparation | |
| DMSO | Biosolve | 0004474701BS | As exposure solvent |
| DNA and RNA microarray deprotection | |||
| Ethylenediamine >99.5% | Sigma | 3550 | For deprotection |
| Expedite 8909 | PerSeptive Biosystems | DNA synthesizer | |
| Hydrazine hydrate 50-60% hydrazine | Sigma | 225819 | For RNA deprotection |
| Imidazole | Sigma | 56750 | |
| Industrial Strength lower-density PTFE tape | Gasoila | Thin, upper teflon gasket | |
| Pyridine >99% | Sigma | P57506 | For RNA deprotection |
| Triethylamine >99% | Sigma | T0886 | For RNA deprotection |
| β-carotene | Sigma | C9750 | For library preparation |
| Hybridization and scanning | |||
| 20x Sodium Saline Citrate | Roth | 1054.1 | |
| 5'Cy3-labelled complementary strand | Eurogentec | For duplex hybridization | |
| Biopur Safe-Lock microcentrifuge tube | Eppendorf | ||
| BSA (10 mg/mL) | Promega | R3961 | |
| EDTA molecular biology grade | Promega | H5031 | |
| GenePix 4100A | Molecular Devices | Microarray scanner | |
| Hybridization oven | Boekel Scientific | 230500 | |
| MES monohydrate | Sigma | 69889 | |
| MES sodium | Sigma | M3058 | |
| NaCl >99.5% | Sigma | 71376 | |
| SecureSeal SA200 hybridization chamber | Grace BioLabs | 623503 | |
| Spectrafuge mini | Labnet | C1301 | Microarray centrifuge |
| Tween-20 molecular biology grade | Sigma | P9416 | |
| Data extraction | |||
| Excel | Microsoft | For data extraction | |
| MatLab | MathWorks | Microarray design | |
| NimbleScan 2.1 | Roche NimbleGen | ||
| Desalting and quantification | |||
| NanoDrop One Spectrophotometer | Thermo Scientific | ||
| Toluene | Merck | ||
| ZipTip C18 | Millipore | ZTC18s008 | Desalting pipet tips |
Referencias
- Bumgarner, R. . Current protocols in molecular biology. 101, 22 (2013).
- Berger, M. F., Bulyk, M. L. Universal protein-binding microarrays for the comprehensive characterization of the DNA-binding specificities of transcription factors. Nature Protocols. 4 (3), 393-411 (2009).
- Tietjen, J. R., Donato, L. J., Bhimisaria, D., Ansari, A. Z. Sequence-specificity and energy landscapes of DNA-binding molecules. Methods in enzymology. 497, 3-30 (2011).
- Katilius, E., Flores, C., Woodbury, N. W. Exploring the sequence space of a DNA aptamer using microarrays. Nucleic Acids Research. 35 (22), 7626-7635 (2007).
- Franssen-van Hal, N. L. W., et al. Optimized Light-Directed Synthesis of Aptamer Microarrays. Analytical Chemistry. 85 (12), 5950-5957 (2013).
- Matteucci, M. D., Caruthers, M. H. Nucleotide Chemistry .4. Synthesis of Deoxyoligonucleotides on a Polymer Support. Journal of the American Chemical Society. 103 (11), 3185-3191 (1981).
- Eroshenko, N., Kosuri, S., Marblestone, A. H., Conway, N., Church, G. M. Gene Assembly from Chip-Synthesized Oligonucleotides. Current Protocols in Chemical Biology. 2012, (2012).
- Kosuri, S., et al. Scalable gene synthesis by selective amplification of DNA pools from high-fidelity microchips. Nature Biotechnology. 28 (12), 1295 (2010).
- Richmond, K. E., et al. Amplification and assembly of chip-eluted DNA (AACED): a method for high-throughput gene synthesis. Nucleic Acids Research. 32 (17), 5011-5018 (2004).
- Schmidt, T. L., et al. Scalable amplification of strand subsets from chip-synthesized oligonucleotide libraries. Nature Communications. 6, (2015).
- Grass, R. N., Heckel, R., Puddu, M., Paunescu, D., Stark, W. J. Robust Chemical Preservation of Digital Information on DNA in Silica with Error-Correcting Codes. Angewandte Chemie International Edition. 54 (8), 2552-2555 (2015).
- Erlich, Y., Zielinski, D. DNA Fountain enables a robust and efficient storage architecture. Science. 355 (6328), 950-953 (2017).
- Garalde, D. R., et al. Highly parallel direct RNA sequencing on an array of nanopores. Nature Methods. 15 (3), 201-206 (2018).
- Cleary, M. A., et al. Production of complex nucleic acid libraries using highly parallel in situ oligonucleotide synthesis. Nature Methods. 1 (3), 241-248 (2004).
- LeProust, E. M., et al. Synthesis of high-quality libraries of long (150mer) oligonucleotides by a novel depurination controlled process. Nucleic Acids Research. 38 (8), 2522-2540 (2010).
- Lietard, J., Damha, M. J., Somoza, M. M., Fernández-Lucas, J. . Enzymatic and Chemical Synthesis of Nucleic Acid Derivatives. , (2018).
- Fodor, S. P. A., et al. Spatially Addressable Parallel Chemical Synthesis. Science. 251 (4995), 767-773 (1991).
- Singh-Gasson, S., et al. Maskless fabrication of light-directed oligonucleotide microarrays using a digital micromirror array. Nature Biotechnology. 17 (10), 974-978 (1999).
- Pease, A. C., et al. Light-Generated Oligonucleotide Arrays for Rapid DNA-Sequence Analysis. Proceedings of the National Academy of Sciences of the United States of America. 91 (11), 5022-5026 (1994).
- Holz, K., Lietard, J., Somoza, M. M. High-Power 365 nm UV LED Mercury Arc Lamp Replacement for Photochemistry and Chemical Photolithography. ACS Sustainable Chemistry & Engineering. 5 (1), 828-834 (2017).
- Lackey, J. G., Mitra, D., Somoza, M. M., Cerrina, F., Damha, M. J. Acetal Levulinyl Ester (ALE) Groups for 2′-Hydroxyl Protection of Ribonucleosides in the Synthesis of Oligoribonucleotides on Glass and Microarrays. Journal of the American Chemical Society. 131 (24), 8496-8502 (2009).
- Lackey, J. G., Somoza, M. M., Mitra, D., Cerrina, F., Damha, M. J. In-situ chemical synthesis of rU-DNA chimeras on chips and enzymatic recognition. Chimica Oggi-Chemistry Today. 27 (6), 30-33 (2009).
- Lietard, J., Ameur, D., Damha, M., Somoza, M. M. High-density RNA microarrays synthesized in situ by photolithography. Angewandte Chemie International Edition. 57 (46), 15257-15261 (2018).
- Agbavwe, C., et al. Efficiency, Error and Yield in Light-Directed Maskless Synthesis of DNA Microarrays. Journal of Nanobiotechnology. 9, (2011).
- Sack, M., et al. Express photolithographic DNA microarray synthesis with optimized chemistry and high-efficiency photolabile groups. Journal of Nanobiotechnology. 14, (2016).
- Kretschy, N., Holik, A. K., Somoza, V., Stengele, K. P., Somoza, M. M. Next-Generation o-Nitrobenzyl Photolabile Groups for Light-Directed Chemistry and Microarray Synthesis. Angewandte Chemie International Edition. 54 (29), 8555-8559 (2015).
- Sack, M., Kretschy, N., Rohm, B., Somoza, V., Somoza, M. M. Simultaneous Light-Directed Synthesis of Mirror-Image Microarrays in a Photochemical Reaction Cell with Flare Suppression. Analytical Chemistry. 85 (18), 8513-8517 (2013).
- Lietard, J., et al. Base-cleavable microarrays for the characterization of DNA and RNA oligonucleotides synthesized in situ by photolithography. Chemical Communications. 50 (85), 12903-12906 (2014).
- Krotz, A. H., et al. Solution stability and degradation pathway of deoxyribonucleoside phosphoramidites in acetonitrile. Nucleosides Nucleotides & Nucleic Acids. 23 (5), 767-775 (2004).
- Hargreaves, J. S., Kaiser, R., Wolber, P. K. The Degradation of Dg Phosphoramidites in Solution. Nucleosides Nucleotides & Nucleic Acids. 34 (10), 691-707 (2015).
- McGall, G. H., et al. The efficiency of light-directed synthesis of DNA arrays on glass substrates. Journal of the American Chemical Society. 119 (22), 5081-5090 (1997).
- Collett, J. R., et al. Functional RNA microarrays for high-throughput screening of antiprotein aptamers. Analytical Biochemistry. 338 (1), 113-123 (2005).
- Buenrostro, J. D., et al. Quantitative analysis of RNA-protein interactions on a massively parallel array reveals biophysical and evolutionary landscapes. Nature Biotechnology. 32 (6), 562-568 (2014).
- Wu, C. -. H., Holden, M. T., Smith, L. M. Enzymatic Fabrication of High-Density RNA Arrays. Angewandte Chemie International Edition. 53 (49), 13514-13517 (2014).
- Tian, J., et al. Accurate multiplex gene synthesis from programmable DNA microchips. Nature. 432 (7020), 1050-1054 (2004).
- Lietard, J., et al. Mapping the affinity landscape of Thrombin-binding aptamers on 2'F-ANA/DNA chimeric G-Quadruplex microarrays. Nucleic Acids Research. 45 (4), 1619-1632 (2017).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados