Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Dinámica de oligomerización de receptores de superficie celular en células vivas mediante la microscopía de fluorescencia de reflexión interna total combinada con análisis de número y brillo
En este artículo
Resumen
Describimos un enfoque de imagen para la determinación del estado oligomérico promedio de los oligómeros receptores etiquetados con mEGFP inducidos por la unión de ligandos en la membrana plasmática de las células vivas. El protocolo se basa en la microscopía de Fluorescencia de Reflexión Interna Total (TIRF) combinada con el análisis Número y Brillo (N&B).
Resumen
A pesar de la importancia y la ubicuidad de la oligomerización de receptores, pocos métodos son aplicables para detectar eventos de agrupación en clústeres y medir el grado de agrupación en clústeres. Aquí, describimos un enfoque de imagen para determinar el estado oligomérico promedio de los homocomplejos de receptores etiquetados con mEGFP en la membrana de las células vivas. El protocolo se basa en la microscopía de Fluorescencia de Reflexión Interna Total (TIRF) combinada con el análisis Número y Brillo (N&B). N&B es un método similar a la espectroscopia de fluorescencia-correlación (FCS) y el histograma de recuento de fotones (PCH), que se basan en el análisis estadístico de las fluctuaciones de la intensidad de fluorescencia de los fluoróforos que se difunden dentro y fuera de una iluminación volumen durante un tiempo de observación. En particular, N&B es una simplificación de PCH para obtener información sobre el número medio de proteínas en mezclas oligoméricas. Las amplitudes de fluctuación de intensidad se describen por el brillo molecular del fluoróforo y el número medio de fluoróforos dentro del volumen de iluminación. Por lo tanto, N&B considera sólo el primer y segundo momento de la distribución de amplitud, a saber, la intensidad media y la varianza. Esta es, al mismo tiempo, la fuerza y la debilidad del método. Debido a que sólo se consideran dos momentos, N&B no puede determinar la fracción molar de los oligómeros desconocidos en una mezcla, pero sólo estima el estado medio de oligomerización de la mezcla. Sin embargo, se puede aplicar a series temporales relativamente pequeñas (en comparación con otros métodos de momento) de imágenes de células en vivo píxel por píxel, simplemente monitoreando las fluctuaciones de tiempo de la intensidad de la fluorescencia. Reduce el tiempo efectivo por píxel a unos pocos microsegundos, lo que permite la adquisición en el intervalo de tiempo de segundos a milisegundos, lo que es necesario para la cinética de oligomerización rápida. Por último, se pueden explorar grandes áreas celulares, así como compartimentos subcelulares.
Introducción
Describimos un enfoque de imagen de Fluorescencia-Número de Fluorescencia Y Brillo de Reflexión Interna Total (TIRF-N&B) para determinar el estado oligomérico promedio de las moléculas receptoras en la membrana plasmática de las células vivas, con el objetivo de vincular el conjunto del receptor dinámica a la función biológica de las proteínas (Figura 1).
Tras la unión de ligandoextra extracelular, los receptores inician la transducción de la señal intracelular dependiendo de su conformación, oligomerización, posibles coreceptores y composición de la membrana. A pesar de la importancia y ubicuidad de la oligomerización de receptores, reconocido como un evento clave en la señalización celular1,2,3,4,5,6, 7, pocos métodos pueden detectar eventos de agrupación en clústeres y medir el grado de agrupación en clústeres experimentalmente8,9. El volumen confocal (x,y a 300 nm, z a 900 nm) no está suficientemente resuelto para probar la interacción molecular y la estequiometría, incluso después de la optimización por algoritmos de restauración de imágenes10. La composición de la subunidad de los oligómeros proteicos no puede resolverse sobre una base puramente espacial incluso mediante métodos de superresolución a resolución x,y de 20-70 nm como PALM11,STORM12y STED13. Además, su resolución temporal (en el orden de minutos por imagen) no puede seguir la cinética en el rango de segundos. El blanqueamiento escalonado de una sola molécula resuelve la estequiometría de los oligómeros proteicos sólo si están inmóviles14.
Uno de los métodos más versátiles para medir la densidad y el oligomerización de proteínas etiquetadas fluorescentes dentro de imágenes individuales es el análisis de distribución de intensidad espacial (SpIDA), que se basa en el muestreo espacial. Es aplicable tanto a células químicamente fijas como vivas, y permite el análisis de varias regiones de interés de la célula simultáneamente utilizando la microscopía de fluorescencia estándar15. Alternativamente, los métodos de momento, tales como espectroscopia de fluorescencia-correlación (FCS)16, histograma de recuento de fotones (PCH)17,y Número y brillo (N&B)18,19, son adecuados para el oligomer cuantitativo Medidas. Estos métodos analizan las fluctuaciones de intensidad de fluorescencia que se pueden observar en el tiempo cuando los fluoróforos se difunden dentro y fuera de un volumen de iluminación. Las amplitudes de las fluctuaciones de intensidad pueden describirse de forma única por el brillo molecular del fluoróforo y el número medio de fluoróforos (n) dentro del volumen de iluminación17 (Figura 2). Típicamente, el coeficiente de difusión de los fluoróforos y el número medio de moléculas (inversamente relacionadas con el valor G(0)) dentro del volumen de iluminación se pueden obtener por FCS20. Sin embargo, dado que el tiempo de difusión sólo se escala con la raíz cúbica de la masa, el FCS no es lo suficientemente sensible para detectar cambios en la masa molecular21. En la práctica, fcS de un solo color no puede detectar la dimerización de los receptores de membrana. PCH resuelve mezclas de diferentes oligómeros con precisión. Utilizando más de dos momentos de la distribución de amplitud, detecta moléculas de diferente brillo que ocupan el mismo volumen de iluminación. Escaneando FCS22 y desarrollos, como la interesante correlación par del enfoque23del brillo molecular (pCOMB), introducido para ampliar el rango de aplicabilidad de los métodos de correlación de fluorescencia en los sistemas biológicos24 , siguen siendo métodos de un solo punto que carecen de la capacidad de mediciones rápidas en un área grande de una celda, lo que requiere muchas observaciones consecutivas en cada píxel y la adquisición de datos en el orden de segundos.
N&B es una versión simplificada de PCH que considera sólo el primer y segundo momento de la amplitud de la distribución de la fluorescencia, a saber, la intensidad media, , y la varianza, n.o2 (Figura 2)18,19 y, debido a eso, no puede determinar la fracción molar de los oligómeros desconocidos en una mezcla, pero sólo estima el estado medio de oligomerización de la mezcla. Sin embargo, N&B tiene la ventaja de trabajar con series temporales relativamente más pequeñas de imágenes de células en vivo que PCH en una base de píxel a píxel, simplemente monitoreando las fluctuaciones en el tiempo de la intensidad de la fluorescencia. Debido a que N&B reduce el tiempo por píxel a unos pocos microsegundos, puede seguir la cinética de oligomerización rápida en áreas de celdas grandes, lo que permite la adquisición de imágenes en una escala de tiempo de segundos en microscopía de escaneo ráster (por ejemplo, confocal, 2 fotones) y milisegundos de milisegundos microscopía basada en cámara (por ejemplo, TIRFM).
Varios informes han demostrado la capacidad de N&B para cuantificar el número de subunidades en los grupos de proteínas mediante imágenes de regiones celulares extendidas. Se detectaron grupos de Paxillin-EGFP en los sitios de adhesión en las células CHO-K125,y la agregación intracelular del péptido patógeno Httex1p se describió en las células COS-726. N&B se aplicó para seguir la oligomerización impulsada por ligando sorda del receptor ErbB27, y el efecto del ligando FGF21 en Klothob (KLB) y FGFR1c en las células De LaLa28. La combinación de imágenes TIRF y análisis N&B se utilizó para mostrar que el dinamina-2 es principalmente tetramírico en toda la membrana celular29. Aplicamos N&B tanto a las imágenes de escaneo ráster como a las imágenes TIRF para probar la dimerización impulsada por ligando sin efecto de los receptores de membrana celular uPAR y FGFR130,31.
Los métodos de correlación de fluorescencia, como N&B, FCS y PCH, se basan en la noción de que en un volumen abierto el número de ocupación de partículas sigue una distribución de Poisson. Debido a que sólo se pueden detectar los fotones que emiten los fluoróforos, el valor 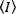 medio de una intensidad de fluorescencia medida frente al tiempo en un píxel de la imagen, , es el producto del número medio de fluoróforos en el volumen de iluminación, n, y su brillo molecular,17:
medio de una intensidad de fluorescencia medida frente al tiempo en un píxel de la imagen, , es el producto del número medio de fluoróforos en el volumen de iluminación, n, y su brillo molecular,17:
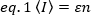
donde se expresa como el número de fotones emitidos por unidad de tiempo (convencionalmente por segundo) por molécula cuando la molécula está en el centro del volumen de iluminación.
El brillo es una propiedad de cada fluoróforo en una adquisición determinada establecida, mientras que la intensidad es la suma de todas las contribuciones de todos los fluoróforos. En los concursos biológicos, el brillo aumentará con el aumento del número de fluoróforos que fluctúan juntos, dando información sobre el estado de oligomerización de la proteína etiquetada fluorescentemente. Las amplitudes de fluctuación en un píxel dado se miden a partir de la varianza de la señal de fluorescencia,n.o 2:
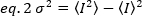
Donde la media del cuadrado 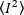 de intensidad, , y el
de intensidad, , y el 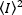 cuadrado de la media de intensidad, , se calculan a partir de los valores de intensidad individuales en cada píxel de cada fotograma:
cuadrado de la media de intensidad, , se calculan a partir de los valores de intensidad individuales en cada píxel de cada fotograma:
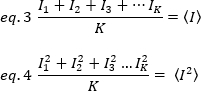
donde K es el número total de fotogramas de la serie temporal. Experimentalmente, es necesario calcular para toda la serie de imágenes la varianza que describe la dispersión de los valores de intensidad individuales en cada píxel de una sola imagen alrededor del valor de intensidad media. La varianza incluye todas las fluctuaciones de diferentes orígenes. En una primera aproximación, la varianza debida a las partículas difusoras en el volumen de iluminación,20, se puede separar de la varianza debido al ruido de disparo del detector,2d. Las dos varianzas son independientes; por lo tanto, la desviación total se da por su suma:
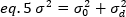
La varianza, debido a las fluctuaciones moleculares dentro y fuera del volumen de detección, depende linealmente del brillo molecular y la intensidad:
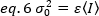
Reorganización eq. 6 según eq. 1:
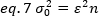
Según el concepto típico en espectroscopía de correlación de fluorescencia, la ecuación 7 establece que la varianza debida al número de fluctuaciones depende del cuadrado del brillo de las partículas.
Entonces, la varianza debida a las fluctuaciones del detector es una función lineal de la intensidad detectada, bajo el supuesto de que el detector se opera por debajo de su límite de saturación19:
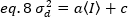
En el caso de los detectores de recuento de fotones a unnúmero 1 y ca 0, por lo tanto, la varianza del detector es igual a la intensidad media:
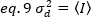
Para aplicar estos conceptos a mediciones reales en celdas vivas, Gratton y sus colegas18 definen el brillo aparente, B, para cada píxel como la proporción de la varianza sobre la intensidad media:
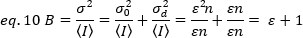
B es el parámetro que se mide experimentalmente. En este trabajo, las imágenes de series temporales de receptores FGFR1 en la membrana plasmática de las células HeLa son capturadas por la microscopía TIRF y el brillo aparente promedio, B, está determinado por el análisis N&B. Luego, después de la adición de FGF2, series temporales consecutivas se capturan para seguir los cambios en el autoensamblaje de las moléculas receptoras en la superficie de la membrana después de la estimulación del receptor con el ligando canónico.
Sin embargo, dado que el detector del microscopio TIRF es una cámara EMCCD, la expresión para el brillo aparente debe modificarse como19:
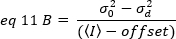
donde el desplazamiento es el desplazamiento de intensidad de la electrónica de detección que es una característica de la configuración del detector. La varianza y la intensidad media de un detector analógico se dan respectivamente por:
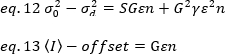
donde G es la ganancia analógica en niveles digitales (DL/fotones), S, los niveles digitales por foton19,se da por la pendiente de una intensidad frente a la varianza de la gráfica para una fuente de luz con intensidad constante (sin fluctuaciones temporales). El factor de la unidad está relacionado con la forma del volumen de detección de píxeles. Según Hassler et al.32, el factor de á es igual a 0,3 para las imágenes TIRF que funcionan con la ganancia máxima de la cámara de detección19. Los parámetros offset, S y G son características de la cámara y del microscopio. El brillo aparente, B, se obtiene reorganizando eq. 11 según eq. 12 y 13:
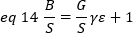
Experimentalmente, es una función compleja de la intensidad del láser y la eficiencia de detección del sistema. Sin embargo, puesto que B/S depende linealmente de la variable, sólo es importante determinar el valor relativo de la opción para un modo de detección determinado:
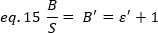
donde es proporcional a . Aún así, una calibración se realiza utilizando una referencia interna.
Protocolo
1. Preparación de muestras
- Día 1. Células HeLa de semillaen en medio completo a una concentración de 100,000-200,000 células / ml en platos con fondo de vidrio. Semilla 6-8 replicar platos.
NOTA: En este ejemplo, el medio se complementa con 10% de suero bovino fetal (FBS) inactivado por calor, piruvato sódico de 1 mM, 100 U/100 g de penicilina/estreptomicina. Se preparan varios platos replicados. - Día 2-3. Cuando las células están en subconfluencia, transfectar la mitad de los platos con el plásmido proteico y la segunda mitad con plásmidos de referencia (monómero y dimer), en medio libre de suero.
NOTA: La transfección se realiza en medio libre de suero complementado con antibióticos, 0,1% de albúmina sérica bovina y tampón HEPES de 25 mM, sin color rojo fenol. - Día 3-4. Compruebe que las células transtrósperas son adherentes a la parte inferior de los platos y que la membrana celular es fluorescente. Deseche los platos con células cubiertas o con muy baja fluorescencia.
NOTA: No deje que las células crezcan en exceso. Las células deben estar bien distribuidas y adherirse al área de vidrio del plato(Figura 1A). Los platos de fondo de vidrio precoatados se pueden utilizar para favorecer la adhesión celular. El cultivo celular se prueba para detectar la contaminación por micoplasma antes de cualquier experimento. En este ejemplo, las células están infectadas con un plásmido (A207K)mEGFP-FGFR1 y las células de referencia están infectadas con un GPI-(A207K)mEGFP y un GPI-(A207K)mEGFP-(A207K)mEGFP plásmidos utilizando protocolos estándar. Para la microscopía de células vivas, se recomienda un medio libre de indicadores; Se añade un tampón HEPES de 25 mM para evitar cambios de pH durante la toma de imágenes.
2. TIRF Imaging — Alineación de la línea láser y optimización de la iluminación TIRF
- Cuatro horas antes del experimento, active la incubadora de temperatura del microscopio a 37oC.
- Encienda el microscopio, las computadoras y las cámaras y espere a que las cámaras alcancen la temperatura de trabajo adecuada.
NOTA: La temperatura de trabajo de la cámara utilizada en este estudio es de -75 oC. - Coloque una gota de aceite en el objetivo. Ponga un plato de muestra en su lugar. Cierre las puertas de la incubadora y deje que la temperatura del plato se equilibre (10 min).
- Encienda la lámpara de epifluorescencia y el láser de 488 nm.
- Seleccione el modo de contraste de epifluorescencia para explorar la muestra, buscando una célula para enfocar desde el ocular.
NOTA: El uso de una lámpara fluorescente para buscar células a través del ocular no es obligatorio. En su lugar, se puede utilizar una línea láser adecuada. - Seleccione el filtro adecuado para recoger la emisión verde a través de la cámara del microscopio (Band Pass Ex 490/20 (500) Band Pass Em 525/50, o similar.
- Cambie del puerto ocular al puerto de la cámara (#1 de cámara en la Figura 1)en modo de epifluorescencia, refinar el enfoque y cambiar al modo TIRF. Los modos de epifluorescencia y TIRF pueden ser nombrados con una nomenclatura diferente dependiendo de la marca del microscopio.
NOTA: Puede haber problemas para enfocar o alinear el láser si no hay marcadores fluorescentes en la interfaz de cubreobjetos. Para alinear el láser correctamente (esencial para un buen TIRF), concéntrese en la cubierta. A menudo es muy difícil determinar si el cubreobjetos está enfocado. Como sugerencia, concéntrese en los bordes de las celdas. - Active la alineación automática siguiendo las instrucciones del microscopio TIRF.
NOTA: Brevemente, para los pasos de 2.4 a 2.8, primero encuentre las células a través del ocular y concéntrese en ellas, luego envíe la emisión al puerto de la cámara del microscopio TIRF, vuelva a enfocar las células en la pantalla del ordenador del microscopio y active el procedimiento para la alineación láser. La alineación consiste en encontrar el ángulo crítico en el que la iluminación se convierte en evanescente(Figura 3). Los microscopios comerciales pueden tener protocolos de alineación ligeramente diferentes y también estar totalmente automatizados; otros podrían tener una pequeña cámara para facilitar la visualización de las condiciones de ángulo crítico. - Elija una profundidad de iluminación adecuada y optimice la dirección del campo evanescente(Figura 3).
NOTA: La profundidad de penetración se mantiene constante para todos los controles y muestras.
3. TIRF Imaging: Captura de la serie temporal
- Defina una región de interés (ROI) de al menos 256 x 256 píxeles.
NOTA: En esta configuración, la captura se realiza con la cámara #2 bajo software que controla directamente sólo la cámara (vea la leyenda de la figura 1). - Establezca la exposición a 1 ms y la ganancia EM en 1.000 (este es el factor G en eq. 12 y 13). A tal velocidad, podría ser necesario ajustar o aumentar la potencia del láser. Aquí la potencia del láser es de 0,5 mW.
NOTA: Dependiendo del tipo de cámara y de los límites impuestos por el coeficiente de difusión de la proteína, la intensidad de la fluorescencia y el fondo, los criterios generales para ajustar la potencia del láser son no saturar el detector, minimizar el fotoblanqueo y capturar como lo más rápido posible en un S/N razonable. La ganancia EM siempre se establece en el máximo de la cámara (consulte Introducción). - Ejecute una primera secuencia de prueba en condiciones iniciales y calcule aproximadamente el valor S/N. Las condiciones son aceptables en S/N a 2-3 o superior, medida en el primer fotograma de la primera serie temporal.
- Utilice el control deslizante del sistema de división de emisiones que conecta la cámara #2 al microscopio para enmascarar un lado de la imagen(Figura 1B, Figura 4A-B)
NOTA: En esta configuración se instala un conector de imágenes multicanal en la cámara #2 para permitir la adquisición simultánea de dos imágenes espacialmente idénticas. El sistema está equipado con portaobjetos para el montaje de diferentes filtros de emisión. Uno de los controles deslizantes monta una máscara negra para cubrir un lado de la imagen. El área enmascarada se utiliza para la calibración interna de cada serie temporal, para determinar los parámetros de la cámara (eq. 12 y eq. 13). De esta manera no hay necesidad de un paso de calibración independiente y, lo que es más importante, la calibración se realiza en paralelo a la captura de cada serie temporal. En ausencia de este sistema, la cámara se puede calibrar aplicando los protocolos publicados33. - Seleccione la opción de guardado automático del archivo de cámara.
- Inicie la adquisición de la serie de imágenes. Adquiera un mínimo de 700 cuadros con una relación S/N mínima de 2.
NOTA: El número de fotogramas necesarios para el análisis depende de la estabilidad de la muestra para el fotoblanqueo y de la dispersión de los datos. Por lo tanto, la calidad de cada serie temporal se evalúa durante el análisis de N&B. - Sin sacar el plato del microscopio, agregue el ligando.
- Seleccione una célula con una membrana de fluorescencia brillante e inicie rápidamente la primera serie temporal de la carrera cinética.
NOTA: Si la adición del ligando se realiza rápidamente, esta primera captura establece el punto 0 tiempo de la cinética de ligando. El software registra la hora exacta de la captura. - Busque una segunda célula y adquiera el segundo punto de tiempo de la cinética.
NOTA: Las rutinas de visitas puntuales están disponibles en algunos microscopios equipados con etapas motorizadas x,y,z. Estos permiten la memorización de múltiples posiciones en la placa de la celda, y pueden ayudar a mantener un intervalo de tiempo más constante entre las series de imágenes en diferentes celdas. - Captura una nueva celda para cada punto de tiempo de la ejecución cinética.
NOTA: Después de capturar, una celda se fotoblanquea parcialmente y no se puede volver a crear una imagen. Debido a eso, la cinética se obtiene mediante la combinación de series temporales de muchas células, cada una capturada en un punto de tiempo diferente. - Para cada plato nuevo, repita el protocolo del paso 2.3 al 3.9.
NOTA: Para platos de referencia, añadir un volumen del vehículo (PBS complementado con 0.01% albúmina sérica bovina) equivalente al utilizado para el ligando.
4. Número y brillo (N&B): Comprobación de calidad de la serie temporal
- Convertir y guardar como . TIFF los archivos adquiridos con el software de la cámara (archivos .sif en este ejemplo).
- Importación. TIFF en la rutina de software de análisis mediante la activación de la interfaz gráfica de usuario N&B (GUI) MATLAB.
NOTA: Aquí se utiliza una rutina de N&B ejecutable de Matlab personalizada (análisis De N&B en https://www.cnic.es/en/investigacion/2/1187/tecnologia). Al abrir un archivo . TIFF, la rutina genera la imagen de intensidad media, el perfil de intensidad media y permite inspeccionar la serie fotograma a fotograma(Figura suplementaria 1). Otros programas están disponibles para el análisis de N&B (por ejemplo, el software SimFCS). - Descartar series para las que el perfil de intensidad media muestra más del 10% de fotoblanqueo, y series en las que ha habido una distorsión o traducción evidente de la membrana celular durante la adquisición.
- Recortar marcos que evidentemente están desenfocados.
NOTA: Una herramienta de recorte se implementa en la rutina para descartar uno o varios fotogramas dentro de la serie de imágenes. Esta operación se permite porque el tiempo de fotograma a fotograma no es crítico, mientras que el tiempo de permanencia del píxel (tiempo de exposición) es (consulte Discusión). - Conservar para el análisis solo series con al menos 500 marcos de tiempo.
5. Número y brillo (N&B): Determinación de los parámetros de la cámara (Desplazamiento, s y S)
- Active la rutina Calibrar cámara.
- Seleccione un área de al menos 20 x 50 píxeles en la región de ruido del detector(Figura 4).
NOTA: La rutina origina un histograma de los valores (también definido Digital Level, DL) y devuelve una gráfica de ritmo del Frequency versus Digital Levels. - En el gráfico Log Frequency versus Digital level, mueva el cursor rojo lineal para delimitar la parte gaussiana y la parte lineal de la curva.
NOTA: El cursor rojo divide las dos secciones de la curva y activa la rutina que devuelve el desplazamiento, que es el centro de la función gaussiana de la respuesta de la cámara, el ajuste de la unidad gaussiana y el factor S, que es la pendiente de la parte lineal de los responses de la cámara e (Figura 4C-D).
6. Número y brillo (N&B): Cálculo de los valores B en la región de interés seleccionada (ROI)
- Active la tecla B.
NOTA: Esta acción genera la imagen de intensidad media(Figura 5, primera columna) y la imagen B en la que cada valor B individual está asociado al píxel relacionado en la imagen(Figura suplementaria 1). - Aplique un binning mínimo (2 2) para reducir la dispersión de los datos y para generar el histograma B-I(Figura 5,segunda columna).
NOTA: El histograma B-I representa la distribución de los valores B de todos los píxeles de la imagen frente a la intensidad del píxel. Y - B/S; X á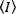 ( - offset)/S(Figura suplementaria 1 y eq. 11 y 15).
( - offset)/S(Figura suplementaria 1 y eq. 11 y 15). - Inspeccione el histograma B-I utilizando el cursor cuadrado interactivo.
- Seleccione un ROI cuadrado para el análisis(Figura 5,tercera columna).
NOTA: El cursor sincroniza una máscara móvil en la imagen de intensidad media, resaltando los píxeles seleccionados dentro del área del cursor cuadrado(Figura suplementaria 1). Mediante esta inspección, es posible excluir del análisis el fondo y las áreas con muy baja intensidad. - Genere el mapa B del ROI seleccionado(Figura 5, cuarta columna).
- Guarde el archivo ASCII de los valores B asociados a la selección.
- Importe el archivo ASCII en un software gráfico para calcular la distribución de frecuencia de los datos y obtener el valor B medio : S.E(Figura 5,quinta columna).
NOTA: Si los datos son homogéneos, la distribución de frecuencia de los valores B se aproxima a una distribución gaussiana. - Aplique eq. 15 para
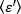 derivar
derivar 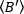 el brillo promedio - 1 [(cuentas/molécula) por tiempo de permanencia] para cada célula en cada punto de tiempo de la ejecución cinética. Normalizar los datos según:
el brillo promedio - 1 [(cuentas/molécula) por tiempo de permanencia] para cada célula en cada punto de tiempo de la ejecución cinética. Normalizar los datos según: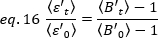
donde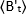 se mide el valor B promedio en el
se mide el valor B promedio en el 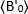 momento "t" después de la adición de ligando, y es el valor B promedio medido en el momento t-0 (10-20 s después de la adición de ligando).
momento "t" después de la adición de ligando, y es el valor B promedio medido en el momento t-0 (10-20 s después de la adición de ligando).
NOTA: La normalización de los resultados permite la comparación directa de experimentos que se llevan a cabo en diferentes días. Compensa las diferencias en el brillo medido debido a la potencia del láser y las fluctuaciones técnicas. - Trazar el brillo medio normalizado frente al tiempo de adquisición para construir la ejecución cinética(Figura 6).
Resultados
Los resultados de dos células representativas de HeLa-mEGFP-FGFR1 sembradas en el mismo plato de cultivo se muestran en la Figura 5 y en la Tabla Suplementaria 1. Las dos celdas fueron capturadas en el tiempo 0 min(Figura 5A, superior) y 7 min(Figura 5A,inferior) después de la adición del ligando FGF2.
Discusión
N&B requiere varias precauciones en la elección del modelo celular y la estrategia de etiquetado. Solo se puede aplicar a las celdas vivas que permanecen establemente adheridas durante el tiempo de captura de imágenes. Las fluctuaciones adicionales debidas al desplazamiento rígido de toda la celda podrían manejarse con los enfoques adecuados de restauración de imágenes38. Sin embargo, generalmente cuando una célula se mueve, la membrana celular también se deforma, y la deformación de la e...
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
El CNIC cuenta con el apoyo del Ministerio de Ciencia, Innovacion y Universidades y la Fundación Pro CNIC, y es un Centro de Excelencia Severo Ochoa (SEV-2015-0505). También contamos con el apoyo del Fondo Europeo de Desarrollo Regional (FEDER) "Una manera de hacer Europa". La UC reconoce el apoyo de la Associazione Italiana Ricerca sul Cancro, la Asociación para la Investigación Internacional del Cáncer (ahora conocida como Worldwide Cancer Research) y el Ministerio de Salud italiano. A.T. reconoce a la "Fondazione Banca del Monte di Lombardia" por apoyar parcialmente su trabajo con la Beca FOTO "Progetto Profesionalitá Ivano Becchi" 2011-2012.
Materiales
| Name | Company | Catalog Number | Comments |
| 3-Colour Fast TIRF Leica AM TIRF MC inverted microscope, with smi-automatic TIRF alignment. The microscope is equipped with a diode 488 nm laser, a 100x 1.46 oil TIRF objective, Ex/Em Bandpass filters at 490/20 and 525/50, temperature/CO2 incubator and Andor DU 8285 VP EMCCD camera. The microscope is operated by Leica LIF software. | Leica Microsystems, Wetzlar, Germany | ||
| Albumin from Bovine Serum 98% minimun | Sigma-Aldrich, St. Louis, MI, USA | A7906-100G | |
| DMEM without Phenol Red with 25 mM HEPES | GIBCO Thermo Fisher Scientific,Waltham, MA, USA | 21063029 | Used serum free for microscopy |
| DMEM high-glucose GlutaMAX I | GIBCO Thermo Fisher Scientific,Waltham, MA, USA | 10566-016 | Used for complete medium |
| Dulbecco's Phosphate Buffered Saline 10x (PBS) | Biowest, Nuaillé, France | X0515-500 | |
| Emission splitting system Photometrics DV2 | TeledynePhotometrics, Tucson, AZ, USA | ||
| Fetal Bovine Serum, qualified, Brazil | GIBCO Thermo Fisher Scientific,Waltham, MA, USA | 10270106 | 10% inactivated supplement for complete medium |
| Glass bottom 35 mm sterile 1.5 dishes | MatTek, Ashland, MA, USA | P35G-0.170-14-C | uncoated, glass thickness 0.17 microns |
| GraphPad Prism | GraphPad Software Inc., San Diego, CA, USA | ||
| Human cervical carcinoma (HeLa), serum-free animal component (AC) cells | Millipore-Sigma ECACC, Darmstadt, Germany | CB_08011102 | |
| iXonEM+ 897 EMCCD (back-illuminated) ANDOR camera controlled by ANDOR Solis software | Oxford Instruments, Andor TM Technology, Abingdon-on-Thames, UK | This camera, installed in an additional port of the microscope, is used for acquiring the N&B time series | |
| Matlab Executable N&B routine | Unit of Microscopy and Dynamic Imaging, CNIC, Madrid, Spain | download at https://www.cnic.es/en/investigacion/2/1187/tecnologia | |
| MatLab v.2018b | The MathWorks, Inc. Natick, MA, USA | https://www.mathworks.com/products/matlab.html | |
| Penicillin:Streptomycin for tissue culture 100x | Biowhittaker Inc. Walkersville, MD, USA | LONZA 17-602E | supplement for medium at Penicillin/Streptomycin 100 U/100µg. |
| pN1-mEGFP-FGFR1 expression vector | Unit of Gynecological Oncology Research, European Institute of Oncology IRCCS, Milan, Italy | Zamai et al., 2019 | |
| pN1-N-Gly-mEGFP-GPI expression vector | Unit of Microscopy and Dynamic Imaging, CNIC, Madrid, Spain | Hellriegel et al., 2011 | |
| pN1-N-Gly-mEGFP-mEGFP-GPI expression vector | Unit of Microscopy and Dynamic Imaging, CNIC, Madrid, Spain | Hellriegel et al., 2011 | |
| Recombinant FGF2 | PeproTech EC, Ltd., London, UK | Ligand solution: 20 ng/mL of FGF2 in PBS supplemented with 0.01%BSA. | |
| Sodium pyruvate GIBCO | ThermoFisher Scientific | 11360070 | 1 mM supplement for medium |
| TransIt-LT1 Transfection Reagent | MirusBio LLC, Madison, WI, USA | MIR 2300 | |
| Trypsin-EDTA (0.25%), phenol red | GIBCO Thermo Fisher Scientific,Waltham, MA, USA | 25200056 | |
| Type F Immersion liquid 10 mL | Leica Microsystems, Wetzlar, Germany | 11513 859 | |
| UltraPure BSA (50 mg/mL) | ThermoFisher Scientific | AM2618 | 0.1% supplement for medium without phenol red used for transfections |
Referencias
- Agwuegbo, U. C., Jonas, K. C. Molecular and functional insights into gonadotropin hormone receptor dimerization and oligomerization. Minerva Ginecologica. 70 (5), 539-548 (2018).
- Ferre, S., et al. G protein-coupled receptor oligomerization revisited: functional and pharmacological perspectives. Pharmacological Reviews. 66 (2), 413-434 (2014).
- Marsango, S., Ward, R. J., Alvarez-Curto, E., Milligan, G. Muscarinic receptor oligomerization. Neuropharmacology. 136 (Pt C), 401-410 (2018).
- Oishi, A., Cecon, E., Jockers, R. Melatonin Receptor Signaling: Impact of Receptor Oligomerization on Receptor Function. International Review of Cell and Molecular Biology. 338, 59-77 (2018).
- Thelen, M., Munoz, L. M., Rodriguez-Frade, J. M., Mellado, M. Chemokine receptor oligomerization: functional considerations. Current Opinion in Pharmacology. 10 (1), 38-43 (2010).
- Van Craenenbroeck, K. GPCR oligomerization: contribution to receptor biogenesis. Subcellular Biochemistry. 63, 43-65 (2012).
- Wnorowski, A., Jozwiak, K. Homo- and hetero-oligomerization of beta2-adrenergic receptor in receptor trafficking, signaling pathways and receptor pharmacology. Cell Signaling Technology. 26 (10), 2259-2265 (2014).
- Fricke, F., Dietz, M. S., Heilemann, M. Single-molecule methods to study membrane receptor oligomerization. Chemphyschem. 16 (4), 713-721 (2015).
- Vidi, P. A., Ejendal, K. F., Przybyla, J. A., Watts, V. J. Fluorescent protein complementation assays: new tools to study G protein-coupled receptor oligomerization and GPCR-mediated signaling. Molecular and Cellular Endocrinology. 331 (2), 185-193 (2011).
- Trussell, H. J., et al., Trussell, J., et al. . Academic Press Library in Signal Processing. 4, 3-9 (2014).
- Betzig, E., et al. Imaging intracellular fluorescent proteins at nanometer resolution. Science. 313 (5793), 1642-1645 (2006).
- Rust, M. J., Bates, M., Zhuang, X. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM). Nature Methods. 3 (10), 793-795 (2006).
- Nagerl, U. V., Willig, K. I., Hein, B., Hell, S. W., Bonhoeffer, T. Live-cell imaging of dendritic spines by STED microscopy. Proceedings of the National Academy of Sciences of the United States of America. 105 (48), 18982-18987 (2008).
- Tsekouras, K., Custer, T. C., Jashnsaz, H., Walter, N. G., Presse, S. A novel method to accurately locate and count large numbers of steps by photobleaching. Molecular Biology of the Cell. 27 (22), 3601-3615 (2016).
- Godin, A. G., et al. Revealing protein oligomerization and densities in situ using spatial intensity distribution analysis. Proceedings of the National Academy of Sciences of the United States of America. 108 (17), 7010-7015 (2011).
- Qian, H., Elson, E. L. Distribution of molecular aggregation by analysis of fluctuation moments. Proceedings of the National Academy of Sciences of the United States of America. 87 (14), 5479-5483 (1990).
- Chen, Y., Muller, J. D., So, P. T., Gratton, E. The photon counting histogram in fluorescence fluctuation spectroscopy. Biophysical Journal. 77 (1), 553-567 (1999).
- Dalal, R. B., Digman, M. A., Horwitz, A. F., Vetri, V., Gratton, E. Determination of particle number and brightness using a laser scanning confocal microscope operating in the analog mode. Microscopy Research and Technique. 71 (1), 69-81 (2008).
- Unruh, J. R., Gratton, E. Analysis of molecular concentration and brightness from fluorescence fluctuation data with an electron multiplied CCD camera. Biophysical Journal. 95 (11), 5385-5398 (2008).
- Hess, S. T., Huang, S., Heikal, A. A., Webb, W. W. Biological and chemical applications of fluorescence correlation spectroscopy: a review. Biochemistry. 41 (3), 697-705 (2002).
- Muller, J. D., Chen, Y., Gratton, E. Fluorescence correlation spectroscopy. Methods in Enzymology. 361, 69-92 (2003).
- Levi, V., Ruan, Q., Kis-Petikova, K., Gratton, E. Scanning FCS, a novel method for three-dimensional particle tracking. Biochemical Society Transactions. 31 (Pt 5), 997-1000 (2003).
- Hinde, E., et al. Quantifying the dynamics of the oligomeric transcription factor STAT3 by pair correlation of molecular brightness. Nature Communications. 7, 11047 (2016).
- Waithe, D., et al. Optimized processing and analysis of conventional confocal microscopy generated scanning FCS data. Methods. 140-141, 62-73 (2018).
- Digman, M. A., Dalal, R., Horwitz, A. F., Gratton, E. Mapping the number of molecules and brightness in the laser scanning microscope. Biophysical Journal. 94 (6), 2320-2332 (2008).
- Ossato, G., et al. A two-step path to inclusion formation of huntingtin peptides revealed by number and brightness analysis. Biophysical Journal. 98 (12), 3078-3085 (2010).
- Nagy, P., Claus, J., Jovin, T. M., Arndt-Jovin, D. J. Distribution of resting and ligand-bound ErbB1 and ErbB2 receptor tyrosine kinases in living cells using number and brightness analysis. Proceedings of the National Academy of Sciences of the United States of America. 107 (38), 16524-16529 (2010).
- Ming, A. Y., et al. Dynamics and Distribution of Klothobeta (KLB) and fibroblast growth factor receptor-1 (FGFR1) in living cells reveal the fibroblast growth factor-21 (FGF21)-induced receptor complex. Journal of Biological Chemistry. 287 (24), 19997-20006 (2012).
- Ross, J. A., et al. Oligomerization state of dynamin 2 in cell membranes using TIRF and number and brightness analysis. Biophysical Journal. 100 (3), L15-L17 (2011).
- Hellriegel, C., Caiolfa, V. R., Corti, V., Sidenius, N., Zamai, M. Number and brightness image analysis reveals ATF-induced dimerization kinetics of uPAR in the cell membrane. FASEB J. 25 (9), 2883-2897 (2011).
- Zamai, M., et al. Number and brightness analysis reveals that NCAM and FGF2 elicit different assembly and dynamics of FGFR1 in live cells. Journal of Cell Science. 132 (1), (2019).
- Hassler, K., et al. Total internal reflection fluorescence correlation spectroscopy (TIR-FCS) with low background and high count-rate per molecule. Optics Express. 13 (19), 7415-7423 (2005).
- Di Rienzo, C., Gratton, E., Beltram, F., Cardarelli, F. From fast fluorescence imaging to molecular diffusion law on live cell membranes in a commercial microscope. Journal of Visualized Experiments. (92), e51994 (2014).
- Beenken, A., Mohammadi, M. The FGF family: biology, pathophysiology and therapy. Nature Reviews Drug Discovery. 8 (3), 235-253 (2009).
- Joubert, J., Sharma, D. Light microscopy digital imaging. Current Protocols in Cytometry. , (2011).
- Gell, C., Berndt, M., Enderlein, J., Diez, S. TIRF microscopy evanescent field calibration using tilted fluorescent microtubules. Journal of Microscopy. 234 (1), 38-46 (2009).
- Burghardt, T. P. Measuring incidence angle for through-the-objective total internal reflection fluorescence microscopy. Journal of Biomedical Optics. 17 (12), 126007 (2012).
- Trullo, A., Corti, V., Arza, E., Caiolfa, V. R., Zamai, M. Application limits and data correction in number of molecules and brightness analysis. Microscopy Research and Technique. 76 (11), 1135-1146 (2013).
- Caiolfa, V. R., et al. Monomer-dimer dynamics and distribution of GPI-anchored uPAR are determined by cell surface protein assemblies. Journal of Cell Biology. 179 (5), 1067-1082 (2007).
- Campbell, R. E., et al. A monomeric red fluorescent protein. Proceedings of the National Academy of Sciences of the United States of America. 99 (12), 7877-7882 (2002).
- Cutrale, F., et al. Using enhanced number and brightness to measure protein oligomerization dynamics in live cells. Nature Protocols. 14 (2), 616-638 (2019).
- Dunsing, V., Chiantia, S. A Fluorescence Fluctuation Spectroscopy Assay of Protein-Protein Interactions at Cell-Cell Contacts. Journal of Visualized Experiments. (142), (2018).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados