Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Dynamique d'oligomerisation des récepteurs de surface cellulaire dans les cellules vivantes par microscopie de fluorescence interne totale combinée à l'analyse du nombre et de la luminosité
Dans cet article
Résumé
Nous décrivons une approche de formation image pour la détermination de l'état oligomeric moyen des oligomères mEGFP-marqué-récepteurs induits par la liaison de ligand dans la membrane de plasma des cellules vivantes. Le protocole est basé sur la microscopie de la fluorescence de la réflexion interne totale (TIRF) combinée à l'analyse du nombre et de la luminosité (N-B).
Résumé
Malgré l'importance et l'omniprésence de l'oligomerisation des récepteurs, peu de méthodes sont applicables pour détecter les événements de regroupement et mesurer le degré de regroupement. Ici, nous décrivons une approche de formation image pour déterminer l'état oligomeric moyen des homocomplexes mEGFP-marqué-récepteur dans la membrane des cellules vivantes. Le protocole est basé sur la microscopie de la fluorescence de la réflexion interne totale (TIRF) combinée à l'analyse du nombre et de la luminosité (N-B). La méthode est une méthode semblable à la spectroscopie de corrélation fluorescence (FCS) et à l'histogramme de comptage de photons (PCH), qui sont basées sur l'analyse statistique des fluctuations de l'intensité de fluorescence des fluorophores diffusant dans et hors d'un éclairage volume pendant un temps d'observation. En particulier, n'est une simplification de PCH pour obtenir des informations sur le nombre moyen de protéines dans les mélanges d'oligomériques. Les amplitudes de fluctuation d'intensité sont décrites par la luminosité moléculaire du fluorophore et le nombre moyen de fluorophores dans le volume d'illumination. Ainsi, N-B ne considère que les premier et deuxième moments de la distribution de l'amplitude, à savoir l'intensité moyenne et la variance. C'est, en même temps, la force et la faiblesse de la méthode. Étant donné que seulement deux moments sont pris en considération, n'est pas en passe de déterminer la fraction molaire des oligomères inconnus dans un mélange, mais elle n'estime que l'état moyen d'oligomerisation du mélange. Néanmoins, il peut être appliqué à des séries chronologiques relativement petites (par rapport à d'autres méthodes de moment) d'images de cellules vivantes sur une base pixel par pixel, simplement en surveillant les fluctuations temporelles de l'intensité de la fluorescence. Il réduit le temps efficace par pixel à quelques microsecondes, permettant l'acquisition dans la plage de temps de secondes à millisecondes, ce qui est nécessaire pour la cinétique d'oligomerisation rapide. Enfin, de grandes zones cellulaires ainsi que des compartiments sous-cellulaires peuvent être explorés.
Introduction
Nous décrivons une approche d'imagerie de fluorescence et de luminosité de réflexion interne totale (TIRF-N-B) pour déterminer l'état oligomeric moyen des molécules de récepteur à la membrane de plasma des cellules vivantes, visant à relier l'assemblage de récepteur dynamique à la fonction biologique des protéines (Figure 1).
Sur la liaison extracellulaire de ligand, les récepteurs initient la transduction intracellulaire de signal selon leur conformation, oligomerization, co-récepteurs potentiels et composition de membrane. Malgré l'importance et l'omniprésence de l'oligomerisation des récepteurs, reconnu comme un événement clé dans la signalisation cellulaire1,2,3,4,5,6, 7, peu de méthodes peuvent détecter les événements de clustering et mesurer le degré de clustering expérimentalement8,9. Le volume confocal (x,y 300 nm, z '900 nm) est insuffisamment résolu pour prouver l'interaction moléculaire et la stoichiométrie, même après optimisation par des algorithmes de restauration d'image10. La composition sous-unité des oligomères protéiques ne peut pas être résolue sur une base purement spatiale, même par des méthodes de super-résolution à x, y résolution de 20-70 nm tels que PALM11, STORM12, et STED13. En outre, leur résolution temporelle (de l'ordre de minutes par image) ne peut pas suivre la cinétique dans la gamme de secondes. Le blanchiment par étapes d'une seule molécule ne résout la stoichiométrie des oligomères protéiques que s'ils sont immobiles14.
L'une des méthodes les plus polyvalentes pour mesurer la densité et l'oligomerisation des protéines étiquetées fluorescentes dans des images uniques est l'analyse de distribution de l'intensité spatiale (SpIDA), qui repose sur l'échantillonnage spatial. Il s'applique à la fois aux cellules fixes et vivantes chimiquement, et permet l'analyse de plusieurs régions d'intérêt de la cellule simultanément en utilisant la microscopie de fluorescence standard15. Alternativement, les méthodes de moment, telles que la spectroscopie de fluorescence-corrélation (FCS)16,l'histogramme de comptage de photon (PCH)17,et le nombre et la luminosité (N et B)18,19,sont appropriés pour l'oligomère quantitatif Mesures. Ces méthodes analysent les fluctuations d'intensité de fluorescence qui peuvent être observées à temps lorsque les fluorophores se diffusent dans et hors d'un volume d'éclairage. Les amplitudes des fluctuations d'intensité peuvent être décrites de façon unique par la luminosité moléculaire du fluorophore (mD) et le nombre moyen de fluorophores (n) dans le volume d'éclairage17 (figure 2). Typiquement, le coefficient de diffusion des fluorophores et le nombre moyen de molécules (inversement liés à la valeur G(0) dans le volume d'éclairage peuvent être obtenus par FCS20. Cependant, puisque le temps de diffusion ne s'écaille qu'avec la racine cubique de la masse, le FCS n'est pas suffisamment sensible pour détecter les changements de masse moléculaire21. Dans la pratique, FCS de couleur unique ne peut pas détecter la dimerisation des récepteurs membranaires. PCH résout avec précision les mélanges de différents oligomères. À l'aide de plus de deux moments de la distribution de l'amplitude, il détecte des molécules d'une luminosité différente qui occupent le même volume d'éclairage. Balayage FCS22 et les développements, tels que l'intéressante paire-corrélation de la luminosité moléculaire (pCOMB) approche23, introduit pour étendre la gamme d'applicabilité des méthodes de corrélation de fluorescence dans les systèmes biologiques24 , restent des méthodes à point unique dépourvues de la capacité de mesures rapides dans une grande zone d'une cellule, nécessitant de nombreuses observations consécutives à chaque pixel et acquisition de données dans l'ordre de secondes.
N-B est une version simplifiée de PCH qui ne tient compte que des premiers et deuxièmes moments de l'amplitude de la distribution de fluorescence, à savoir l'intensité moyenne, lt;I-gt;, et la variance,'2 (Figure 2)18,19 et, pour cette raison, il ne peut pas déterminer la fraction molaire des oligomères inconnus dans un mélange, mais seulement estime l'état moyen d'oligomerisation du mélange. Néanmoins, n'a l'avantage de travailler avec des séries temporelles d'images de cellules vivantes relativement plus petites que PCH sur une base pixel par pixel, simplement en surveillant les fluctuations à l'heure de l'intensité de la fluorescence. Étant donné que le temps par pixel réduit le temps par pixel à quelques microsecondes, il peut suivre la cinétique d'oligomerisation rapide sur de grandes zones cellulaires, permettant l'acquisition d'images sur une échelle de temps de secondes dans la microscopie de balayage de raster (par exemple, confocal, 2-photon) et millisecondes microscopie à la caméra (p. ex. TIRFM).
Plusieurs rapports ont démontré la capacité de N-B à quantifier le nombre de sous-unités dans les grappes de protéines par imagerie des régions cellulaires étendues. Des amas de Paxillin-EGFP ont été détectés aux emplacements d'adhérence dans les cellules de CHO-K125,et l'agrégation intracellulaire du peptide pathogène de Httex1p a été décrite dans les cellules coS-726. Le n et B a été appliqué pour suivre l'oligomerisation ligand-conduite du récepteur d'ErbB27,et l'effet du ligand FGF21 sur Klothob (KLB) et FGFR1c dans les cellules de HeLa28. La combinaison de l'imagerie TIRF et de l'analyse N-B a été utilisée pour montrer que le dynamin-2 est principalement tétracumé dans toute la membrane cellulaire29. Nous avons appliqué n'et B à la fois raster balayage et images TIRF pour prouver ligand-conduit dimerization des récepteurs de membrane cellulaire uPAR et FGFR130,31.
Les méthodes de corrélation de fluorescence, telles que le N-B, le FCS et le PCH, sont basées sur l'idée que dans un volume ouvert le nombre d'occupation des particules suit une distribution de Poisson. Parce que seuls les photons que les fluorophores émettent peuvent être détectés, la valeur moyenne 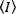 d'une intensité de fluorescence mesurée par rapport au temps dans un pixel de l'image, est le produit du nombre moyen de fluorophores dans le volume d'éclairage, n, et leur luminosité moléculaire,17euros :
d'une intensité de fluorescence mesurée par rapport au temps dans un pixel de l'image, est le produit du nombre moyen de fluorophores dans le volume d'éclairage, n, et leur luminosité moléculaire,17euros :
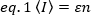
où le nombre de photons émis par unité de temps (conventionnellement par seconde) par molécule lorsque la molécule est au centre du volume d'éclairage.
La luminosité est une propriété de chaque fluorophore dans une acquisition donnée mise en place, tandis que l'intensité est la somme de toutes les contributions de tous les fluorophores. Dans les concours biologiques, la luminosité augmentera avec l'augmentation du nombre de fluorophores qui fluctuent ensemble, donnant des informations sur l'état d'oligomerisation de la protéine fluorescente. Les amplitudes de fluctuation à un pixel donné sont mesurées à partir de la variance du signal de fluorescence,no 2:
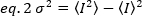
Lorsque la moyenne du carré 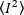 d'intensité, et le
d'intensité, et le 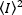 carré de la moyenne de l'intensité, , sont calculer à partir des valeurs d'intensité individuelle dans chaque pixel de chaque image:
carré de la moyenne de l'intensité, , sont calculer à partir des valeurs d'intensité individuelle dans chaque pixel de chaque image:
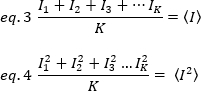
où K est le nombre total d'images dans la série chronologique. Expérimentalement, il est nécessaire de calculer pour l'ensemble de la série d'images la variance qui décrit la diffusion des valeurs d'intensité individuelles à chaque pixel d'une seule image autour de la valeur moyenne de l'intensité. La variance comprend toutes les fluctuations d'origines différentes. Dans une première approximation, la variance due aux particules de diffusion dans le volume d'éclairage,20, peut être séparée de la variance en raison du bruit de tir du détecteur,2d. Les deux écarts sont indépendants; ainsi, l'écart total est donné par leur somme :
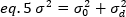
La variance, due aux fluctuations moléculaires dans et hors du volume de détection, dépend linéairement de la luminosité et de l'intensité moléculaires :
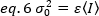
Réorganisation eq. 6 selon eq. 1:
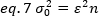
Selon le concept typique de la spectroscopie de corrélation de fluorescence, l'équation 7 indique que la variance due au nombre de fluctuations dépend du carré de la luminosité des particules.
Ensuite, la variance due aux fluctuations du détecteur est une fonction linéaire de l'intensité détectée, en supposant que le détecteur est exploité en dessous de sa limite de saturation19:
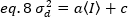
Dans le cas des détecteurs de comptage de photons de1 et de 0,la variance du détecteur est donc égale à l'intensité moyenne :
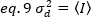
Pour appliquer ces concepts à des mesures réelles dans des cellules vivantes, Gratton et ses collègues18 définissent la luminosité apparente, B, pour chaque pixel comme le rapport de la variance par rapport à l'intensité moyenne :
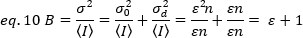
B est le paramètre qui est mesuré expérimentalement. Dans ce travail, les images de séries chronologiques des récepteurs FGFR1 à la membrane plasmatique des cellules HeLa sont capturées par microscopie TIRF et la luminosité apparente moyenne, B, est déterminée par l'analyse N et B. Puis, après l'ajout de FGF2, des séries chronologiques consécutives sont capturées pour suivre les changements dans l'auto-assemblage des molécules de récepteur dans la surface de membrane après stimulation du récepteur avec le ligand canonique.
Cependant, puisque le détecteur du microscope TIRF est une caméra EMCCD, l'expression de la luminosité apparente doit être modifiée comme19:
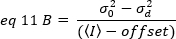
lorsque le décalage est le décalage d'intensité de l'électronique de détection qui est une caractéristique des paramètres du détecteur. La variance et l'intensité moyenne d'un détecteur analogique sont données respectivement par :
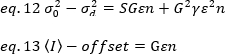
où G est le gain analogique dans les niveaux numériques (DL/photons), S, les niveaux numériques par photon19, est donné par la pente d'une intensité par rapport à l'intrigue de variance pour une source lumineuse avec une intensité constante (pas de fluctuations temporelles). Le facteur de l'aétée est lié à la forme du volume de détection des pixels. Selon Hassler et coll.32, le facteur d'aété est égal à 0,3 pour l'imagerie TIRF travaillant au gain maximum de la caméra de détection19. Les paramètres de décalage, S et G sont des caractéristiques de la caméra et du microscope. La luminosité apparente, B, est obtenue en réorganisant eq. 11 selon eq. 12 et 13:
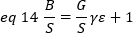
Expérimentalement, est une fonction complexe de l'intensité laser et de l'efficacité de détection du système. Néanmoins, étant donné que le B/S dépend linéairement de l'activité, il est seulement important de déterminer la valeur relative de l'application pour un mode de détection donné :
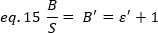
où est proportionnelle à la proportion de ' Pourtant, un étalonnage est effectué à l'aide d'une référence interne.
Protocole
1. Préparation de l'échantillon
- Jour 1. Cellules HeLa de graine dans le milieu complet à une concentration de 100.000-200.000 cellules/mL dans les plats en verre-fond. Graines 6-8 reproduire les plats.
REMARQUE: Dans cet exemple, le milieu est complété par 10% de chaleur inactivée sérum bovin fœtal (FBS), 1 mM de pyruvate de sodium, 100 U/100 g de pénicilline/streptomycine. Plusieurs plats reproduits sont préparés. - Jour 2-3. Lorsque les cellules sont sous-confluences, transfect la moitié des plats avec le plasmide protéique et la seconde moitié avec des plasmides de référence (monomère et dimère), dans un milieu sans sérum.
REMARQUE: La transfection est faite dans le milieu sans sérum complété avec des antibiotiques, 0.1% Albumin de sérum bovin et 25 mM hePES tampon, sans phenol rouge. - Jour 3-4. Vérifiez que les cellules transfectées sont adhérentes au fond de la vaisselle et que la membrane cellulaire est fluorescente. Jeter la vaisselle avec des cellules envahies ou avec une fluorescence très faible.
REMARQUE: Ne laissez pas les cellules surcroître. Les cellules doivent être bien réparties et être adhérées à la surface vitrée du plat (Figure 1A). Les plats en verre précouché peuvent être utilisés pour favoriser l'adhérence cellulaire. La culture cellulaire est testée pour la contamination par le mycoplasme avant toute expérience. Dans cet exemple, les cellules sont transfectées avec un plasmide (A207K)mEGFP-FGFR1 et les cellules de référence sont transfectées avec un GPI-(A207K)mEGFP et un GPI-(A207K)mEGFP-(A207K)mEGFP plasmides utilisant des protocoles standard. Pour la microscopie cellulaire vivante, un milieu sans indicateur est recommandé; Un tampon HEPES de 25 mM est ajouté pour prévenir les changements de pH pendant l'imagerie.
2. Imagerie TIRF — Alignement de la ligne laser et optimisation de l'éclairage TIRF
- Quatre heures avant l'expérience, activez l'incubateur de température du microscope à 37 oC.
- Allumez le microscope, les ordinateurs et les caméras et attendez que les caméras atteignent la température de travail appropriée.
REMARQUE: La température de travail de la caméra utilisée dans cette étude est de -75 oC. - Placez une petite goutte d'huile sur l'objectif. Mettre un plat d'échantillon en place. Fermez les portes de l'incubateur et laissez la température du plat s'aquilper (10 min).
- Allumez la lampe d'épifluorescence et le laser de 488 nm.
- Sélectionnez le mode de contraste d'épifluorescence pour explorer l'échantillon, en cherchant une cellule pour se concentrer à partir de l'oculaire.
REMARQUE: L'utilisation d'une lampe fluorescente pour la recherche des cellules à travers l'oculaire n'est pas obligatoire. Une ligne laser appropriée peut être utilisée à la place. - Sélectionnez le filtre approprié pour recueillir l'émission verte à l'intermédiaire de la caméra microscope (Band Pass Ex 490/20 (500) Band Pass Em 525/50, ou similaire.
- Passez de l'oculaire au port de la caméra (la caméra #1 en mode 1) en mode épifluorescence, affinez la mise au point et passez au mode TIRF. Les modes Epifluorescence et TIRF peuvent être nommés avec une nomenclature différente selon la marque du microscope.
REMARQUE: Il peut y avoir des problèmes de focalisation ou d'alignement du laser s'il n'y a pas de marqueurs fluorescents à l'interface de couverture. Pour aligner le laser correctement (essentiel pour un bon TIRF), concentrez-vous sur le bordereau. Il est souvent très difficile de déterminer si la couverture est au point. Comme une suggestion, se concentrer sur les bords des cellules. - Activez l'auto-alignement en suivant les instructions du microscope TIRF.
REMARQUE: En bref, pour les étapes de 2.4 à 2.8, d'abord trouver les cellules à travers l'oculaire et se concentrer sur eux, puis envoyer l'émission au port de la caméra du microscope TIRF, recentrer les cellules sur l'écran de l'ordinateur microscope et activer la procédure pour l'alignement laser. L'alignement consiste à trouver l'angle critique auquel l'illumination devient évanescente (figure 3). Les microscopes commerciaux peuvent avoir des protocoles d'alignement légèrement différents et être entièrement automatisés; d'autres pourraient avoir une petite caméra pour faciliter la visualisation des conditions d'angle critique. - Choisissez une profondeur d'éclairage appropriée et optimisez la direction du champ évanescent (Figure 3).
REMARQUE: La profondeur de pénétration est constante pour tous les contrôles et échantillons.
3. Imagerie TIRF: Capture de la série chronologique
- Définir une région d'intérêt (ROI) d'au moins 256 x 256 pixels.
REMARQUE: Dans cette configuration, la capture se fait avec la caméra #2 sous un logiciel qui ne contrôle directement que la caméra (voir la légende de la figure 1). - Définir l'exposition à 1 ms et le gain EM à 1000 (c'est le facteur G dans eq. 12 et 13). À une telle vitesse, il pourrait être nécessaire d'ajuster ou d'augmenter la puissance laser. Ici, la puissance laser est de 0,5 mW.
REMARQUE: Selon le type de caméra et les limites imposées par le coefficient de diffusion de la protéine, l'intensité de la fluorescence et le fond, les critères généraux pour la mise en place de la puissance laser ne sont pas de saturer le détecteur, de minimiser le photoblanchiment et de capturer plus vite que possible à un S / N raisonnable. Le gain EM est toujours réglé au maximum de la caméra (voir Introduction). - Exécuter une première séquence d'essai dans des conditions initiales et estimer grossièrement la valeur S/N. Les conditions sont acceptables à S/N 2-3 ou plus, tel que mesuré dans le premier cadre de la première série chronologique.
- Utilisez le curseur du système de fractionnement des émissions reliant la caméra #2 au microscope pour masquer un côté de l'image (Figure 1B, Figure 4A-B)
REMARQUE: Dans cette mise en place, un connecteur d'imagerie multicanal est installé sur la caméra #2 pour permettre l'acquisition de deux images spatialement identiques simultanément. Le système est équipé de glissières pour le montage de différents filtres d'émission. L'un des curseurs monte un masque noir pour couvrir un côté de l'image. La zone masquée est utilisée pour l'étalonnage interne de chaque série chronologique, afin de déterminer les paramètres de la caméra (eq. 12 et eq. 13). De cette façon, il n'est pas nécessaire d'effectuer une étape d'étalonnage indépendante et, surtout, l'étalonnage est effectué parallèlement à la capture de chaque série chronologique. En l'absence de ce système, la caméra peut être calibré en appliquant les protocoles publiés33. - Sélectionnez l'option d'enregistrement automatique de fichiers de l'appareil photo.
- Commencez l'acquisition de la série d'images. Acquérir un minimum de 700 images à un rapport S/N minimum de 2.
REMARQUE: Le nombre d'images nécessaires à l'analyse dépend de la stabilité de l'échantillon au photoblanchiment et de la dispersion des données. Par conséquent, la qualité de chaque série chronologique est évaluée au cours de l'analyse n et B. - Sans sortir le plat du microscope, ajouter le ligand.
- Sélectionnez une cellule avec une membrane de fluorescence brillante et commencez rapidement la première série de temps de la course cinétique.
REMARQUE: Si l'ajout du ligand se fait rapidement, cette première capture fixe le point 0 temps de la cinétique ligand. Le logiciel enregistre l'heure exacte de la capture. - Rechercher une deuxième cellule et acquérir le deuxième point de temps de la cinétique.
REMARQUE: Les routines de visite ponctuelle sont disponibles dans certains microscopes équipés d'étapes motorisées x,y,z. Ceux-ci permettent la mémorisation de positions multiples sur le plat cellulaire, et peuvent aider à garder un intervalle de temps plus constant entre les séries d'images sur différentes cellules. - Capturez une nouvelle cellule pour chaque point de la course cinétique.
REMARQUE: Après la capture, une cellule est partiellement photobleached et il ne peut pas être re-imaged. Pour cette raison, la cinétique est obtenue en combinant des séries chronologiques de nombreuses cellules, chacune capturée à un moment différent. - Pour chaque nouveau plat, répétez le protocole de l'étape 2.3 à 3.9.
REMARQUE: Pour les plats de référence, ajoutez un volume du véhicule (PBS complété avec 0,01% d'albumine de sérum bovin) équivalent à celui utilisé pour le ligand.
4. Nombre et luminosité (N et B) : Vérification de la qualité de la série chronologique
- Convertissez et économisez comme . TIFF les fichiers acquis avec le logiciel de la caméra (.sif fichiers dans cet exemple).
- Importation. TIFF fichiers dans la routine logicielle d'analyse en activant l'interface utilisateur graphique N 'amp; B (GUI) MATLAB.
REMARQUE: Une routine Matlab exécutable Matlab est utilisée ici (analyse N et B à https://www.cnic.es/en/investigacion/2/1187/tecnologia). En ouvrant un importé . Fichier TIFF, la routine génère l'image d'intensité moyenne, le profil d'intensité moyenne et il permet d'inspecter la série image par image (Figure supplémentaire 1). D'autres logiciels sont disponibles pour l'analyse n et B (par exemple, le logiciel SimFCS). - Série de rejet pour laquelle le profil d'intensité moyenne montre plus de 10% de photoblanchiment, et série dans laquelle il y a eu une distorsion évidente de membrane cellulaire ou de traduction pendant l'acquisition.
- Cadres de culture qui sont évidemment flous.
REMARQUE: Un outil de recadrage est implémenté dans la routine pour écarter les images simples ou multiples dans la série d'images. Cette opération est autorisée parce que le temps d'image à l'image n'est pas critique alors que le pixel d'habiter le temps (temps d'exposition) est (voir Discussion). - Conserver pour l'analyse seulement la série avec au moins 500 délais.
5. Nombre et luminosité (N et B) : Détermination des paramètres de la caméra (Offset, et S)
- Activer la caméra deroutine Calibrate .
- Sélectionnez une zone d'au moins 20 x 50 pixels dans la région du bruit du détecteur (figure 4).
REMARQUE: La routine provient d'un histogramme des valeurs (également défini Digital Level, DL) et il renvoie une parcelle de logarithme de la fréquence par rapport aux niveaux numériques. - Dans l'intrigue de niveau Fréquence par rapport à l'intrigue numérique, déplacez le curseur rouge linéaire pour délimiter le Gaussian et la partie linéaire de la courbe.
REMARQUE: Le curseur rouge divise les deux sections de la courbe, et active la routine de retour de la compensation, qui est le centre de la fonction gaussienne de la réponse de la caméra, le ' de l'ajustement Gaussian, et le facteur S, qui est la pente de la partie linéaire de la caméra respons e (Figure 4C-D).
6. Nombre et luminosité (N et B) : Calcul des valeurs B dans la région d'intérêt sélectionnée (ROI)
- Activez la clé B.
REMARQUE: Cette action génère l'image d'intensité moyenne(figure 5, première colonne) et l'image B dans laquelle chaque valeur B individuelle est associée au pixel connexe de l'image (Figure supplémentaire 1). - Appliquer un binning minimum (2 2) pour réduire la dispersion des données et générer l'histogramme B-I(figure 5, deuxième colonne).
REMARQUE: L'histogramme B-I représente la distribution des valeurs B de tous les pixels de l'image par rapport à l'intensité du pixel. Y et B/S; X (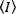 - offset)/S (Figure 1 et eq. 11 et 15).
- offset)/S (Figure 1 et eq. 11 et 15). - Inspectez l'histogramme B-I à l'aide du curseur carré interactif.
- Sélectionnez un retour sur investissement carré pour l'analyse (Figure 5, troisième colonne).
REMARQUE: Le curseur synchronise un masque mobile sur l'image d'intensité moyenne, en soulignant les pixels qui sont sélectionnés à l'intérieur de la zone du curseur carré (Figure supplémentaire 1). Par cette inspection, il est possible d'exclure de l'analyse l'arrière-plan et les zones à très faible intensité. - Générer la carte B du ROI sélectionné (Figure 5, quatrième colonne).
- Enregistrer le fichier ASCII des valeurs B associées à la sélection.
- Importer le fichier ASCII dans un logiciel graphique pour calculer la distribution de fréquence des données et obtenir la valeur moyenne B -S.E (Figure 5, cinquième colonne).
REMARQUE: Si les données sont homogènes, la distribution de fréquence des valeurs B se rapproche d'une distribution gaussienne. - Appliquer l'eq. 15 pour
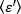
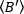 obtenir la luminosité moyenne - 1 [(compte/molécule) par temps d'habitant] pour chaque cellule à chaque moment de la course cinétique. Normaliser les données en fonction de :
obtenir la luminosité moyenne - 1 [(compte/molécule) par temps d'habitant] pour chaque cellule à chaque moment de la course cinétique. Normaliser les données en fonction de :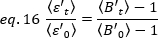
où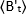 est la valeur Moyenne B mesurée au moment
est la valeur Moyenne B mesurée au moment 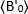 "t" après l'ajout de ligand, et est la valeur Moyenne B mesurée à l'époque t -0 (10-20 s après l'ajout de ligand).
"t" après l'ajout de ligand, et est la valeur Moyenne B mesurée à l'époque t -0 (10-20 s après l'ajout de ligand).
REMARQUE: La normalisation des résultats permet de comparer directement les expériences qui sont menées à différents jours. Il compense les différences dans la luminosité mesurée due à la puissance laser et les fluctuations techniques. - Tracer la luminosité moyenne normalisée par rapport au temps d'acquisition pour construire la course cinétique (Figure 6).
Résultats
Les résultats de deux cellules représentatives HeLa-mEGFP-FGFR1 ensemées dans le même plat de culture sont présentés dans la figure 5 et le tableau supplémentaire 1. Les deux cellules ont été capturées à l'époque 0 min (Figure 5A, top) et 7 min (Figure 5A, bottom) après l'ajout du ligand FGF2.
Discussion
La N-B exige plusieurs précautions dans le choix du modèle cellulaire et de la stratégie d'étiquetage. Il ne peut être appliqué qu'aux cellules vivantes qui restent stablement respectées pendant le temps de capture d'image. Les fluctuations supplémentaires dues à l'ensemble du déplacement rigide de la cellule peuvent être traitées avec des approches appropriées de restauration d'image38. Cependant, généralement quand une cellule se déplace, la membrane cellulaire se déforme égale...
Déclarations de divulgation
Les auteurs n'ont rien à révéler.
Remerciements
Le CNIC est soutenu par le Ministère de Ciencia, Innovacion y Universidades et la Fondation Pro CNIC, et est un Centre d'Excellence Severo Ochoa (SEV-2015-0505). Nous sommes également soutenus par le Fonds européen de développement régional (FEDER) "Una manera de hacer Europa". UC reconnaît le soutien de l'Associazione Italiana Ricerca sul Cancro, de l'Association for International Cancer Research (maintenant connue sous le nom de Worldwide Cancer Research) et du ministère italien de la Santé. A.T. reconnaît la "Fondazione Banca del Monte di Lombardia" pour avoir en partie soutenu son travail avec la bourse PV "Progetto Professionalità Ivano Becchi" 2011-2012.
matériels
| Name | Company | Catalog Number | Comments |
| 3-Colour Fast TIRF Leica AM TIRF MC inverted microscope, with smi-automatic TIRF alignment. The microscope is equipped with a diode 488 nm laser, a 100x 1.46 oil TIRF objective, Ex/Em Bandpass filters at 490/20 and 525/50, temperature/CO2 incubator and Andor DU 8285 VP EMCCD camera. The microscope is operated by Leica LIF software. | Leica Microsystems, Wetzlar, Germany | ||
| Albumin from Bovine Serum 98% minimun | Sigma-Aldrich, St. Louis, MI, USA | A7906-100G | |
| DMEM without Phenol Red with 25 mM HEPES | GIBCO Thermo Fisher Scientific,Waltham, MA, USA | 21063029 | Used serum free for microscopy |
| DMEM high-glucose GlutaMAX I | GIBCO Thermo Fisher Scientific,Waltham, MA, USA | 10566-016 | Used for complete medium |
| Dulbecco's Phosphate Buffered Saline 10x (PBS) | Biowest, Nuaillé, France | X0515-500 | |
| Emission splitting system Photometrics DV2 | TeledynePhotometrics, Tucson, AZ, USA | ||
| Fetal Bovine Serum, qualified, Brazil | GIBCO Thermo Fisher Scientific,Waltham, MA, USA | 10270106 | 10% inactivated supplement for complete medium |
| Glass bottom 35 mm sterile 1.5 dishes | MatTek, Ashland, MA, USA | P35G-0.170-14-C | uncoated, glass thickness 0.17 microns |
| GraphPad Prism | GraphPad Software Inc., San Diego, CA, USA | ||
| Human cervical carcinoma (HeLa), serum-free animal component (AC) cells | Millipore-Sigma ECACC, Darmstadt, Germany | CB_08011102 | |
| iXonEM+ 897 EMCCD (back-illuminated) ANDOR camera controlled by ANDOR Solis software | Oxford Instruments, Andor TM Technology, Abingdon-on-Thames, UK | This camera, installed in an additional port of the microscope, is used for acquiring the N&B time series | |
| Matlab Executable N&B routine | Unit of Microscopy and Dynamic Imaging, CNIC, Madrid, Spain | download at https://www.cnic.es/en/investigacion/2/1187/tecnologia | |
| MatLab v.2018b | The MathWorks, Inc. Natick, MA, USA | https://www.mathworks.com/products/matlab.html | |
| Penicillin:Streptomycin for tissue culture 100x | Biowhittaker Inc. Walkersville, MD, USA | LONZA 17-602E | supplement for medium at Penicillin/Streptomycin 100 U/100µg. |
| pN1-mEGFP-FGFR1 expression vector | Unit of Gynecological Oncology Research, European Institute of Oncology IRCCS, Milan, Italy | Zamai et al., 2019 | |
| pN1-N-Gly-mEGFP-GPI expression vector | Unit of Microscopy and Dynamic Imaging, CNIC, Madrid, Spain | Hellriegel et al., 2011 | |
| pN1-N-Gly-mEGFP-mEGFP-GPI expression vector | Unit of Microscopy and Dynamic Imaging, CNIC, Madrid, Spain | Hellriegel et al., 2011 | |
| Recombinant FGF2 | PeproTech EC, Ltd., London, UK | Ligand solution: 20 ng/mL of FGF2 in PBS supplemented with 0.01%BSA. | |
| Sodium pyruvate GIBCO | ThermoFisher Scientific | 11360070 | 1 mM supplement for medium |
| TransIt-LT1 Transfection Reagent | MirusBio LLC, Madison, WI, USA | MIR 2300 | |
| Trypsin-EDTA (0.25%), phenol red | GIBCO Thermo Fisher Scientific,Waltham, MA, USA | 25200056 | |
| Type F Immersion liquid 10 mL | Leica Microsystems, Wetzlar, Germany | 11513 859 | |
| UltraPure BSA (50 mg/mL) | ThermoFisher Scientific | AM2618 | 0.1% supplement for medium without phenol red used for transfections |
Références
- Agwuegbo, U. C., Jonas, K. C. Molecular and functional insights into gonadotropin hormone receptor dimerization and oligomerization. Minerva Ginecologica. 70 (5), 539-548 (2018).
- Ferre, S., et al. G protein-coupled receptor oligomerization revisited: functional and pharmacological perspectives. Pharmacological Reviews. 66 (2), 413-434 (2014).
- Marsango, S., Ward, R. J., Alvarez-Curto, E., Milligan, G. Muscarinic receptor oligomerization. Neuropharmacology. 136 (Pt C), 401-410 (2018).
- Oishi, A., Cecon, E., Jockers, R. Melatonin Receptor Signaling: Impact of Receptor Oligomerization on Receptor Function. International Review of Cell and Molecular Biology. 338, 59-77 (2018).
- Thelen, M., Munoz, L. M., Rodriguez-Frade, J. M., Mellado, M. Chemokine receptor oligomerization: functional considerations. Current Opinion in Pharmacology. 10 (1), 38-43 (2010).
- Van Craenenbroeck, K. GPCR oligomerization: contribution to receptor biogenesis. Subcellular Biochemistry. 63, 43-65 (2012).
- Wnorowski, A., Jozwiak, K. Homo- and hetero-oligomerization of beta2-adrenergic receptor in receptor trafficking, signaling pathways and receptor pharmacology. Cell Signaling Technology. 26 (10), 2259-2265 (2014).
- Fricke, F., Dietz, M. S., Heilemann, M. Single-molecule methods to study membrane receptor oligomerization. Chemphyschem. 16 (4), 713-721 (2015).
- Vidi, P. A., Ejendal, K. F., Przybyla, J. A., Watts, V. J. Fluorescent protein complementation assays: new tools to study G protein-coupled receptor oligomerization and GPCR-mediated signaling. Molecular and Cellular Endocrinology. 331 (2), 185-193 (2011).
- Trussell, H. J., et al., Trussell, J., et al. . Academic Press Library in Signal Processing. 4, 3-9 (2014).
- Betzig, E., et al. Imaging intracellular fluorescent proteins at nanometer resolution. Science. 313 (5793), 1642-1645 (2006).
- Rust, M. J., Bates, M., Zhuang, X. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM). Nature Methods. 3 (10), 793-795 (2006).
- Nagerl, U. V., Willig, K. I., Hein, B., Hell, S. W., Bonhoeffer, T. Live-cell imaging of dendritic spines by STED microscopy. Proceedings of the National Academy of Sciences of the United States of America. 105 (48), 18982-18987 (2008).
- Tsekouras, K., Custer, T. C., Jashnsaz, H., Walter, N. G., Presse, S. A novel method to accurately locate and count large numbers of steps by photobleaching. Molecular Biology of the Cell. 27 (22), 3601-3615 (2016).
- Godin, A. G., et al. Revealing protein oligomerization and densities in situ using spatial intensity distribution analysis. Proceedings of the National Academy of Sciences of the United States of America. 108 (17), 7010-7015 (2011).
- Qian, H., Elson, E. L. Distribution of molecular aggregation by analysis of fluctuation moments. Proceedings of the National Academy of Sciences of the United States of America. 87 (14), 5479-5483 (1990).
- Chen, Y., Muller, J. D., So, P. T., Gratton, E. The photon counting histogram in fluorescence fluctuation spectroscopy. Biophysical Journal. 77 (1), 553-567 (1999).
- Dalal, R. B., Digman, M. A., Horwitz, A. F., Vetri, V., Gratton, E. Determination of particle number and brightness using a laser scanning confocal microscope operating in the analog mode. Microscopy Research and Technique. 71 (1), 69-81 (2008).
- Unruh, J. R., Gratton, E. Analysis of molecular concentration and brightness from fluorescence fluctuation data with an electron multiplied CCD camera. Biophysical Journal. 95 (11), 5385-5398 (2008).
- Hess, S. T., Huang, S., Heikal, A. A., Webb, W. W. Biological and chemical applications of fluorescence correlation spectroscopy: a review. Biochemistry. 41 (3), 697-705 (2002).
- Muller, J. D., Chen, Y., Gratton, E. Fluorescence correlation spectroscopy. Methods in Enzymology. 361, 69-92 (2003).
- Levi, V., Ruan, Q., Kis-Petikova, K., Gratton, E. Scanning FCS, a novel method for three-dimensional particle tracking. Biochemical Society Transactions. 31 (Pt 5), 997-1000 (2003).
- Hinde, E., et al. Quantifying the dynamics of the oligomeric transcription factor STAT3 by pair correlation of molecular brightness. Nature Communications. 7, 11047 (2016).
- Waithe, D., et al. Optimized processing and analysis of conventional confocal microscopy generated scanning FCS data. Methods. 140-141, 62-73 (2018).
- Digman, M. A., Dalal, R., Horwitz, A. F., Gratton, E. Mapping the number of molecules and brightness in the laser scanning microscope. Biophysical Journal. 94 (6), 2320-2332 (2008).
- Ossato, G., et al. A two-step path to inclusion formation of huntingtin peptides revealed by number and brightness analysis. Biophysical Journal. 98 (12), 3078-3085 (2010).
- Nagy, P., Claus, J., Jovin, T. M., Arndt-Jovin, D. J. Distribution of resting and ligand-bound ErbB1 and ErbB2 receptor tyrosine kinases in living cells using number and brightness analysis. Proceedings of the National Academy of Sciences of the United States of America. 107 (38), 16524-16529 (2010).
- Ming, A. Y., et al. Dynamics and Distribution of Klothobeta (KLB) and fibroblast growth factor receptor-1 (FGFR1) in living cells reveal the fibroblast growth factor-21 (FGF21)-induced receptor complex. Journal of Biological Chemistry. 287 (24), 19997-20006 (2012).
- Ross, J. A., et al. Oligomerization state of dynamin 2 in cell membranes using TIRF and number and brightness analysis. Biophysical Journal. 100 (3), L15-L17 (2011).
- Hellriegel, C., Caiolfa, V. R., Corti, V., Sidenius, N., Zamai, M. Number and brightness image analysis reveals ATF-induced dimerization kinetics of uPAR in the cell membrane. FASEB J. 25 (9), 2883-2897 (2011).
- Zamai, M., et al. Number and brightness analysis reveals that NCAM and FGF2 elicit different assembly and dynamics of FGFR1 in live cells. Journal of Cell Science. 132 (1), (2019).
- Hassler, K., et al. Total internal reflection fluorescence correlation spectroscopy (TIR-FCS) with low background and high count-rate per molecule. Optics Express. 13 (19), 7415-7423 (2005).
- Di Rienzo, C., Gratton, E., Beltram, F., Cardarelli, F. From fast fluorescence imaging to molecular diffusion law on live cell membranes in a commercial microscope. Journal of Visualized Experiments. (92), e51994 (2014).
- Beenken, A., Mohammadi, M. The FGF family: biology, pathophysiology and therapy. Nature Reviews Drug Discovery. 8 (3), 235-253 (2009).
- Joubert, J., Sharma, D. Light microscopy digital imaging. Current Protocols in Cytometry. , (2011).
- Gell, C., Berndt, M., Enderlein, J., Diez, S. TIRF microscopy evanescent field calibration using tilted fluorescent microtubules. Journal of Microscopy. 234 (1), 38-46 (2009).
- Burghardt, T. P. Measuring incidence angle for through-the-objective total internal reflection fluorescence microscopy. Journal of Biomedical Optics. 17 (12), 126007 (2012).
- Trullo, A., Corti, V., Arza, E., Caiolfa, V. R., Zamai, M. Application limits and data correction in number of molecules and brightness analysis. Microscopy Research and Technique. 76 (11), 1135-1146 (2013).
- Caiolfa, V. R., et al. Monomer-dimer dynamics and distribution of GPI-anchored uPAR are determined by cell surface protein assemblies. Journal of Cell Biology. 179 (5), 1067-1082 (2007).
- Campbell, R. E., et al. A monomeric red fluorescent protein. Proceedings of the National Academy of Sciences of the United States of America. 99 (12), 7877-7882 (2002).
- Cutrale, F., et al. Using enhanced number and brightness to measure protein oligomerization dynamics in live cells. Nature Protocols. 14 (2), 616-638 (2019).
- Dunsing, V., Chiantia, S. A Fluorescence Fluctuation Spectroscopy Assay of Protein-Protein Interactions at Cell-Cell Contacts. Journal of Visualized Experiments. (142), (2018).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon