Method Article
Cristalización y determinación estructural de una enzima: complejo de sustratos por cristalografía serie en un chip microfluídico versátil
En este artículo
Resumen
Se describe un versátil dispositivo microfluídico que permite la cristalización de una enzima utilizando el método de contradifusión, la introducción de un sustrato en los cristales por remojo y la determinación de la estructura 3D de la enzima: complejo de sustratos mediante un análisis en serie de cristales dentro del chip a temperatura ambiente.
Resumen
La preparación de cristales de difracción de pozos y su manejo antes de su análisis de rayos X son dos pasos críticos de los estudios biocristalostallográficos. Describimos un chip microfluídico versátil que permite la producción de cristales mediante el método eficiente de contradifusión. El entorno libre de convección proporcionado por los canales microfluídicos es ideal para el crecimiento de cristales y útil para difundir un sustrato en el sitio activo de la enzima cristalina. Aquí aplicamos este enfoque a la enzima CCA-adding de la bacteria psicofólica Planococcus halocryophilus en el ejemplo presentado. Después de la cristalización y la difusión/remojo del sustrato, la estructura cristalina del complejo enzyme:substrate se determinó a temperatura ambiente mediante cristalografía en serie y el análisis de múltiples cristales directamente dentro del chip. Todo el procedimiento conserva las propiedades de difracción genuinas de las muestras porque no requiere manipulación de cristales.
Introducción
La cristalografía es un método para descifrar la arquitectura 3D de las macromoléculas biológicas. Este último es importante para entender cómo una enzima selecciona y procesa sus sustratos. La determinación de una estructura cristalina requiere la cristalización de la macromolécula objetivo y el acondicionamiento de los cristales para su análisis por difracción de rayos X1. Tanto la preparación de cristales como el manejo son pasos cruciales pero delicados que pueden afectar a la calidad del cristal y las propiedades de difracción, y, por lo tanto, la resolución (es decir, la precisión) de la estructura 3D resultante. Para facilitar la preparación de cristales de alta calidad y eliminar el manejo innecesario para preservar sus propiedades de difracción, diseñamos un dispositivo microfluídico fácil de usar y versátil llamado ChipX2,3,4.
En este artículo, demostraremos cómo cargar la solución proteica en canales ChipX utilizando material de laboratorio convencional para preparar cristales mediante contra-difusión. Este método de cristalización proporciona un cribado eficiente de la supersaturación y de las condiciones potenciales de nucleación a lo largo de los canales microfluídicos que contienen la solución enzimática debido al gradiente de concentración generado por la difusión del agente cristalizador5,6.
La configuración del chip es simple, utiliza sólo pipetas de laboratorio estándar y no requiere ningún equipo costoso. Cuando los cristales han crecido en ChipX, los ligandos de la enzima se pueden introducir mediante difusión. Los datos de difracción se recopilan a temperatura ambiente en una serie de cristales contenidos en los canales del chip utilizando una fuente de rayos X sincrotrón. El estudio estructural descrito aquí condujo a la determinación de estructuras de una enzima de maduración de TRNA en su forma de apo y en complejo con un análogo de su sustrato CTP introducido por remojo. Esta proteína llamada enzima que añade CCA polimeriza la cola de trinucleótido CCA en el extremo de 3' de los TRNAs. La comparación de las dos imágenes 3D obtenidas por cristalografía en serie revela los cambios de conformación locales relacionados con la unión del ligando en condiciones más fisiológicas que las utilizadas en la crio-cristalografía. El protocolo descrito en este video es generalmente aplicable a cualquier biomolécula, ya sea una proteína, un ácido nucleico o un complejo multicompetitivo.
Protocolo
1. Configuración de ensayos de cristalización en ChipX
NOTA: El dispositivo microfluídico ChipX se puede obtener de los autores. Una descripción del chip se da en la Figura 1. Las soluciones que contengan el cristalizador (o agente cristalizador) utilizado para desencadenar la cristalización pueden ser de origen comercial o preparadas por el experimentador.
- Carga de la muestra de biomolécula
NOTA: El volumen de muestra realmente necesario para realizar un ensayo individual de contradifusión en la sección recta de cada canal de ChipX es de 300 nL. Sin embargo, para mayor comodidad sugerimos cargar 5 μL para llenar completamente los ocho canales teniendo en cuenta la longitud variable de su sección curva y los volúmenes muertos de entrada.- Pipete 5 μL de soluciones enzimáticas utilizando pipete y punta estándar de 10 μL.
- Introduzca la punta verticalmente en la entrada de la muestra e inyecte la solución hasta que los ocho canales se llenen hasta su extremo opuesto (entrada del depósito cristalante).
- Inyecte 1 μL de aceite de parafina en la entrada de muestra para desconectar los canales entre sí.
- Recupere la solución adicional en el depósito cristalante en la extremidad de cada canal utilizando un pipete estándar de 10 μL.
- Selle la entrada de muestra con un trozo de cinta de 1 cm x 1 cm.
- Carga de las soluciones de cristalización
- Pipete 5 μL de solución de cristalización utilizando pipete y punta estándar de 10 μL. El volumen del depósito es de 10 μL, pero cargar sólo la mitad evita el desbordamiento al sellar con cinta adhesiva y facilita la adición de ligando para los experimentos de remojo. Si las condiciones iniciales de cristalización se obtuvieron mediante difusión de vapor, aumente la concentración cristalante en un factor de 1,5 - 2. Las soluciones pueden ser diferentes en cada depósito (en el caso presentado, fosfato de hidrógeno diammonio de 1 M, acetato de sodio de 100 mM, pH 4.5 se utilizó en todo).
- Oriente la punta del pipete hacia la entrada del canal en la parte en forma de embudo del depósito para evitar la formación de una burbuja de aire tras la deposición de la solución. Evitaría el contacto entre las dos soluciones y la difusión cristalante en el canal.
- Inyecte la solución cristalante en el depósito.
- Selle los depósitos con una cinta adhesiva de 2,5 cm x 1 cm.
- Incubar el chip a 20 °C (la temperatura se puede ajustar dependiendo del objetivo, normalmente entre 4 y 37 °C 4).
2. Etiquetado de proteínas con carboxodamina para la detección de fluorescencia
NOTA: Este paso es opcional. Debe realizarse antes de la carga de la muestra para facilitar la detección de cristales en el chip mediante fluorescencia. El método detallado de rastreo de etiquetado fluorescente fue descrito por Pusey y compañeros de trabajo7. Todos los pasos se llevan a cabo a temperatura ambiente.
- Disolver 5 mg de polvo de éster de carboxrhodamina en 1 ml de dimetil-formamida anhidra, dividir la solución en 0,6 μL aliquots que se almacenarán a -20 °C.
- Prepare una solución de stock de 1 M Na-borate pH 8.75.
- Diluir el stock para preparar el amortiguador de reacción a 0,05 M Na-borate pH 8,75.
- Enjuague una columna de desalación (7 kDa MWCO, 0,5 ml) con 800 μL de búfer de reacción.
- Centrífuga la columna durante 1 min a 1400 x g, retire el filtrado.
- Repita esta operación dos veces (pasos 2.4-2.5) para lavar la columna.
- Deposite 80 μL de proteína en su búfer de almacenamiento en la columna (la proteína se puede diluir hasta 1 mg/ml para aumentar el volumen si es necesario).
- Centrífuga la columna durante 1 min a 1400 x g. Este paso está destinado a transferir la proteína de su búfer de almacenamiento al búfer de reacción.
- Recuperar el flujo a través (que contiene la proteína en el búfer de reacción) y mezclarlo con 0,6 μL de solución de carboxodamina.
- Incubar 5 min a temperatura ambiente.
- Mientras tanto, enjuague la columna 3 x con el búfer de almacenamiento, centrífuga la columna durante 1 min a 1400 x g y deseche el filtrado.
- Deposite la solución de reacción en la columna.
- Centrífuga la columna durante 1 min a 1400 x g y recupere el flujo (es decir, solución de proteína etiquetada en su búfer de almacenamiento).
- Complementar la solución proteica de stock con 0,1-1 % (w/w) de proteína etiquetada.
- Configure los ensayos de cristalización de ChipX como se describe en la sección 1.
- Compruebe la presencia de cristales proteicos en los ensayos excitando la sonda fluorescente con una fuente de luz de longitud de onda de 520 nm.
3. Observación de cristal
NOTA: El dispositivo ChipX se puede manejar sin especial cuidado, incluso con cristales en su interior, excepto si la temperatura necesita ser controlada.
- Utilice cualquier estereomicroscopio para comprobar el resultado de los ensayos de cristalización en ChipX. Su huella tiene las dimensiones estándar de las diapositivas de microscopio y es compatible con cualquier sistema y portaobjetos.
- Compruebe el contenido de los canales microfluídicos a partir del depósito donde la concentración cristalante es la más alta a la entrada de muestra donde la concentración cristalante es la más baja. El material ChipX es transparente a la luz visible, compatible con el uso de polarizadores, así como con iluminación UV para la identificación de cristales proteicos por fluorescencia ttófana intrínsica8.
- Grabe posiciones de cristal utilizando las etiquetas grabadas a lo largo de los canales o marque ubicaciones de cristal con un marcador permanente dibujando puntos de color junto a ellos en la superficie del chip.
4. Cristal empapado con ligandos
NOTA: Este procedimiento es opcional. Se utiliza para introducir ligandos, sustratos enzimáticos o átomos pesados en los cristales y debe llevarse a cabo al menos 24-48 h antes del análisis de rayos X para permitir la difusión compuesta a lo largo de los canales y en los cristales.
- Retire suavemente la cinta de sellado de los depósitos.
- Añadir hasta 5 μL de solución de ligando en uno o varios reservorios utilizando una solución de micropipeto de 10 μL (por ejemplo, 3 μL de 10 mM de citidina-5'-[α,β)-metilno] para lograr una concentración final de 3,75 mM). CMPcPP es un análogo no hidrolizable de CTP, un sustrato natural de la enzima.
- Selle los depósitos con una cinta adhesiva de 2,5 cm x 1 cm.
- Incubar el chip a temperatura controlada durante 24-48 h para permitir la difusión del ligando a lo largo de los canales del chip.
5. Análisis de cristal por cristalografía en serie
NOTA: Esta parte del protocolo debe adaptarse en función de la configuración de la línea de haz y las propiedades de difracción de los cristales. Sólo se dan indicaciones generales para el análisis cristalográfico basado en experimentos realizados en la línea de haz X06DA (SLS, Villigen, Suiza).
- Montaje ChipX en el goniómetro de la línea de haz
NOTA: El archivo para la impresión 3D del soporte ChipX se proporciona en la referencia4.- Apague el crio-chorro de la línea de haz. El análisis aquí se lleva a cabo a temperatura ambiente.
- Monte el ChipX en un soporte dedicado con el canal que contiene los cristales que se analizarán situados en el centro del soporte. El soporte ChipX4 no requiere ningún tornillo o pieza adicional, ya que fue diseñado para proporcionar un ajuste perfecto para ChipX.
- Fije el soporte al goniómetro.
- Recopilación de datos
- Oriente la capa más gruesa (capa superior, Figura 1)de ChipX (en esta orientación las etiquetas a lo largo de los canales son directamente legibles utilizando la cámara de centrado de la línea de haz), hacia el haz directo y la cara más delgada detrás del cristal para minimizar la atenuación de la señal difractada como se describe en la referencia3.
- Para evitar la colisión de ChipX con el material circundante (vigas, colimador), restrinja los movimientos del goniómetro en el rango ±30° (0° correspondiente a que los canales sean perpendiculares al haz de rayos X).
- Encuentra la posición de cristales con la ayuda de las etiquetas grabadas a lo largo de los canales.
- Seleccione una posición de cristal.
- Centrar el cristal mediante la proyección estándar de cuadrícula/ráster de dosis bajas o un procedimiento de 1 clic (el vídeo muestra un ejemplo de cribado de cuadrícula).
- Recopile datos de difracción dentro del rango -30° / +30°.
- Reinicie el procedimiento en los pasos 5.2.4-5.2.6 en otro cristal en el mismo canal después de la traducción del chip.
- Realinee manualmente otro canal ChipX en el centro del soporte y continúe la recopilación de datos en los cristales presentes en este canal.
- Utilice paquetes y procedimientos cristalográficos estándar para procesar y combinar los datos y, a continuación, para resolver y refinar la estructura.
Resultados
El chip microfluídico descrito aquí fue diseñado para permitir una fácil configuración de ensayos de cristalización y análisis de cristal a temperatura ambiente. El procedimiento descrito anteriormente y en el video se aplicó en el marco de la caracterización estructural de la enzima de adición de CCA de la bacteria adaptada en frío Planococcus halocryophilus. Esta enzima pertenece a una familia de polimerasa esencial que cataliza la adición secuencial de la secuencia CCA de 3' en tRNAs utilizando CTP y ATP9,10.
El chip se utilizó por primera vez para preparar cristales de la enzima para el análisis estructural mediante el método de contra-difusión. Con este fin, la solución enzimática se cargó en los ocho canales microfluídicos (cámaras de cristalización) mediante una sola inyección en la entrada de muestra del chip (véase la Figura 1). La enzima se utilizó a 5,5 mg/ml en su búfer de almacenamiento que contenía 20 mM Tris/HCl pH 7,5, 200 mM NaCl y 5 mM MgCl2. Este paso se realizó manualmente con un micropipet estándar de 10 μL. Las soluciones de cristalización (acetato de sodio de 100 mM pH 4,5,1 M de fosfato de hidrógeno diammonium) se depositaron en los depósitos en el otro extremo de los canales.
El procedimiento de carga es sencillo y no tarda más de cinco minutos(figura 2). El cristalante luego se difunde en los canales, crea un gradiente de concentración que desencadena la nucleación de cristal y el crecimiento. Este gradiente evoluciona dinámicamente y explora un continuo de estados de supersaturación5,6 hasta alcanzar un equilibrio de concentración cristalante entre los canales y el depósito. Los ensayos de cristalización normalmente se comprueban bajo el micoscopio durante un período de 2 a 4 semanas para realizar un seguimiento del crecimiento de los cristales. Cristales bipyramidales de enzimas que añaden CCA aparecieron en todos los canales después de unos días de incubación a 20 °C (Figura 3). El etiquetado fluorescente opcional7 de la proteína facilita en gran medida la identificación de cristales proteicos y su discriminación por los cristales de sal(Figura 4).
Explotamos el entorno difusivo en los canales de chip para entregar un sustrato a la enzima que construye los cristales. En el presente caso, CMPcPP, un análogo CTP, se añadió a las soluciones de depósito a una concentración final de 3,75 mM (Figura 5). Esta adición se realizó dos días antes del análisis cristalográfico para permitir que CMPcPP llegara y ocupara el sitio catalítico de la enzima, como más tarde se confirmó por la estructura cristalina (ver más abajo).
Fabricamos un soporte de chip(Figura 6)en ácido poliláctico utilizando una impresora 3D. El soporte permite el montaje en viruta en goniómetros utilizando cabezales magnéticos estándar. Por lo tanto, el chip se puede colocar y traducir fácilmente en el haz de rayos X para llevar los cristales en posición de difracción. La estrategia de recopilación de datos debe adaptarse en función de las características de la línea de haz y de las propiedades cristalinas. En el caso de la enzima de adición de CCA, se recopilaron datos en líneas de haz X06DA y X10SA, Swiss Light Source (SLS), con una longitud de onda de rayos X de detectores de píxeles 1.0 Å y Pilatus 2M-F y 6M, respectivamente. Se recogieron 30-60° de rotación en cada cristal a temperatura ambiente con imágenes de exposición de 0,1° o 0,2° y 0,1 s (ver Tabla 1). Los conjuntos de datos parciales se procesaron individualmente y se cortaron cuando la resolución de los patrones de difracción comenzó a decaer debido a daños por radiación (detectados por la disminución de la relación señal-ruido 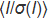 y CC1/2,y un aumento de Rmeas en la carcasa de alta resolución). Los conjuntos de datos completos se reconstituyeron mediante la combinación de datos de 5 cristales (Tabla 1). Las estructuras de cristal se derivaron mediante reemplazo molecular utilizando paquetes y procedimientos cristalográficos estándar para el procesamiento de datos11 y el refinamiento12. La comparación de las estructuras de la enzima y de su complejo con CMPcPP revela la adaptación de conformación local que acompaña a la unión del sustrato en el sitio activo de la enzima que añade CCA(Figura 7).
y CC1/2,y un aumento de Rmeas en la carcasa de alta resolución). Los conjuntos de datos completos se reconstituyeron mediante la combinación de datos de 5 cristales (Tabla 1). Las estructuras de cristal se derivaron mediante reemplazo molecular utilizando paquetes y procedimientos cristalográficos estándar para el procesamiento de datos11 y el refinamiento12. La comparación de las estructuras de la enzima y de su complejo con CMPcPP revela la adaptación de conformación local que acompaña a la unión del sustrato en el sitio activo de la enzima que añade CCA(Figura 7).
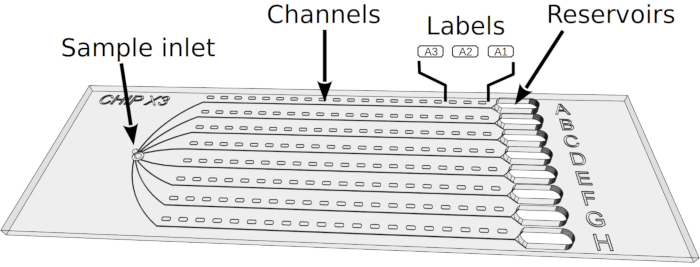
Figura 1: Diseño chipX. El chip consiste en una capa superior hecha de COC (espesor: 1 mm) en la que se imprimen ocho canales microfluídicos y depósitos. Todo el chip está sellado con una capa de COC (espesor: 0,1 mm). Todos los canales están conectados a una sola entrada en el lado izquierdo para la inyección simultánea de muestras y a depósitos individuales en el lado derecho en los que se depositan soluciones de cristalización. Los canales, que constituyen las cámaras de cristalización reales del chip, tienen 4 cm de largo y tienen una sección transversal de 80 μm x 80 μm. Etiquetas (A1, A2, A3, etc.) grabadas a lo largo de los canales facilitan el posicionamiento cristalino bajo el microscopio y la preparación de una lista de muestras para la recopilación de datos. ChipX tiene el tamaño de una diapositiva de microscopio estándar (7,5 cm x 2,5 cm). Haga clic aquí para ver una versión más grande de esta figura.
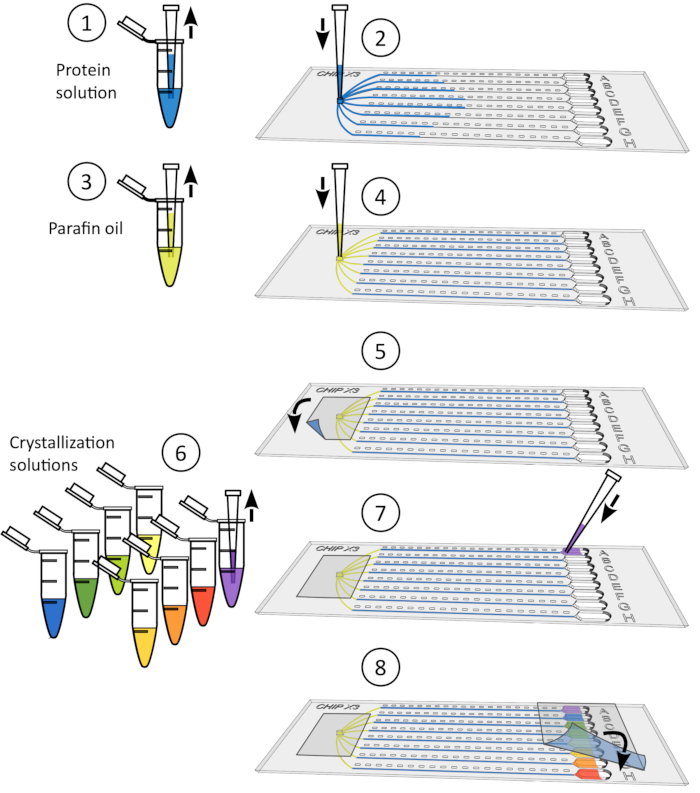
Figura 2: Configuración de ensayos de cristalización en ChipX. 1) Depósito 5-6 μL de soluciones enzimáticas utilizando pipete y punta estándar de 10 μL. 2) Introduzca la punta verticalmente en la entrada de muestra e inyecte la solución en los ocho canales. 3) Pipete 1 μL de aceite de parafina. 4) Introduzca la punta verticalmente en la entrada de muestra e inyecte el aceite para desconectar los canales entre sí. 5) Selle la entrada con un trozo de cinta. 6) Pipete 5 μL de solución de cristalización utilizando pipete y punta estándar de 10 μL. Las soluciones pueden ser diferentes en cada depósito (por ejemplo, a partir de un kit de cribado). 7) Oriente la punta del pipete hacia la entrada del canal en la parte en forma de embudo del depósito (para evitar la formación de una burbuja de aire tras la deposición de la solución) e inyecte la solución cristalante en el depósito. 8) Selle los depósitos con un trozo de cinta e incuba el chip a temperatura controlada. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: Cristales de enzimas que añaden CCA cultivadas por contradifusión en los canales microfluídicos de ChipX. La barra de escala es de 0,1 mm. Haga clic aquí para ver una versión más grande de esta figura.
Figura 4: Procedimiento de remojo de cristal. 1) Retire suavemente la cinta de los depósitos. 2) Deposite hasta 5 μL de solución de ligando utilizando un micropipet de 10 μL. 3) Añadir el ligando a uno o varios depósitos. 4) Selle de nuevo los depósitos con un trozo de cinta e incubar el chip bajo temperatura controlada durante 24-48 h antes de la recolección de datos. Haga clic aquí para ver una versión más grande de esta figura.

Figura 5: El etiquetado fluorescente traza discrimina la proteína (izquierda) de los cristales de sal (derecha). La solución enzimática que añade CCA contenía el 0,4 % (p/p) de proteína etiquetada con carboxodamina. A la derecha, los cristales se iluminan con una fuente de luz de longitud de onda de 520 nm y la imagen se toma con un filtro de paso bajo a 550 nm (LP550); (inserto) estructura del éster carboxrhodamina-succinimidyl. Haga clic aquí para ver una versión más grande de esta figura.

Figura 6: (Izquierda) Dibujo del soporte ChipX y (Derecha) ChipX montado en el goniómetro de la línea de haz X06DA en SLS (Villigen, Suiza) para el análisis de cristal serie. Haga clic aquí para ver una versión más grande de esta figura.
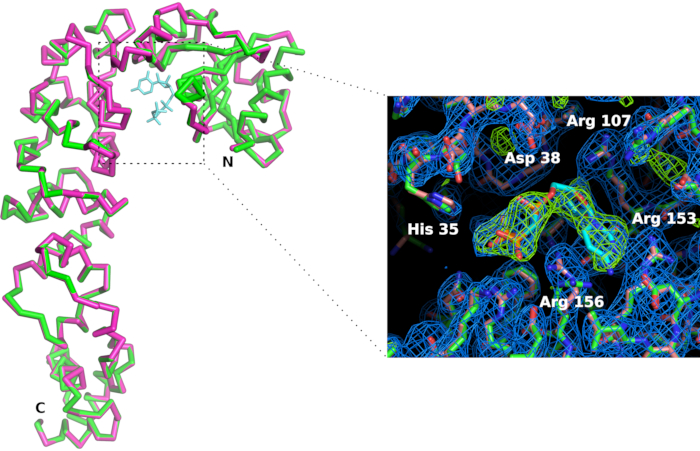
Figura 7: Comparación del sitio activo enzimático de adición de CCA en forma de apo (en rosa) y en el complejo con un análogo CTP (en verde). Aunque la conformación general de la enzima no se ve afectada, la unión del ligando CMPcPP va acompañada de una ligera reorganización de las cadenas laterales en el sitio activo. El mapa de densidad de electrones 2Fo-Fc (en azul) está contorneado a 1,2 sigma. El mapa de densidad de electrones diferenciado a 4 sigma (en verde) confirma la presencia del ligando en el sitio activo. Haga clic aquí para ver una versión más grande de esta figura.
| Muestra cristalizada | Enzima que añade CCA | Enzima de adición de CCA + CMPcPP |
| Análisis de cristales | ||
| Línea de haz de rayos X | SLS – X06DA | SLS – X10SA |
| Longitud de onda (Å) | 1.000 | 1.000 |
| Temperatura (K) | 293 | 293 |
| Detector | Pilatus 2M-F | Pilatus 6M |
| Distancia del detector de cristal (mm) | 300 | 400 |
| Cristales recogidos | 6 | 14 |
| Cristales seleccionados | 5 | 5 |
| Rango de rotación por imagen (°) | 0.1 | 0.2 |
| Tiempo de exposición por imagen (s) | 0.1 | 0.1 |
| No. de las imágenes seleccionadas | 1000 | 540 |
| Rango de rotación total (°) | 100 | 108 |
| Grupo espacial | P43212 | P43212 |
| a, c (Å) | 71.5, 293.8 | 71.4, 293.6 |
| Mosaicidad media (°) | 0.04 | 0.04 |
| Rango de resolución (Å) | 46 – 2.54 (2.6 – 2.54) | 48 – 2.3 (2.4 – 2.3) |
| Total No. de reflexiones | 176105 (9374) | 232642 (32937) |
| No. de reflexiones únicas | 23922 (1598) | 34862 (4066) |
| Integridad (%) | 90.6 (84.6) | 99.5 (100.0) |
| Redundancia | 7.5 (6.0) | 6.7 (8.1) |
 | 8.1 (1.3) | 6.9 (0.7) |
| Rmeas (%) § | 18.6 (126.0) | 18.0 (231.2) |
| CC1/2 (%) £ | 98.7 (55.0) | 98.7 (46.9) |
| Factor B general de la trama Wilson (Å2) | 57.4 | 60.6 |
| Refinamiento cristalográfico | ||
| No. de reflexiones, conjunto de trabajo / conjunto de pruebas | 23583 / 1180 | 34840 / 3405 |
| Rcryst final (%) /R libre (%) | 18.8 / 21.4 | 20.0 / 22.9 |
| No. de átomos no H: en general / proteína / ligando / disolvente | 2998 / 2989 / 0 / 9 | 3057 / 2989 / 29 / 10 |
| R.m.s. desviaciones para enlaces (Å) / ángulos (°) | 0.009 / 1.23 | 0.010 / 1.22 |
| Factores B promedio (Å2):general / proteína / ligando / disolvente | 60.1 / 60.1 / 0 / 52.7 | 62.5 / 62.6 / 60.1 / 55.5 |
| Parcela ramachandran: más favorecida (%) / permitida (%) | 98.1 / 1.9 | 97.2 / 2.8 |
| Id de PDB | 6IBP | 6T52 |
Tabla 1: Estadísticas de recopilación y refinamiento de datos
§ Rmeas independientes de redundancia = Σhkl(N/N-1)1/2Σi | Ii(hkl)- (hkl)>| / ΣhklΣi i i(hkl),donde N es la multiplicidad de datos 17.
£ Los datos con baja en el caparazón exterior (<2.0) se incluyeron en función del criterio CC1/2 (correlación entre dos mitades aleatorias del conjunto de datos > 50%) como propone Karplus & Diederichs 18.
Discusión
Los protocolos actuales en biocristallografía implican la preparación de cristales utilizando métodos como la difusión de vapor o el lote13,14,y su transferencia a un microloop para crioenfriamiento15,16 antes de realizar el análisis de difracción en un chorro de nitrógeno en condiciones criogénicas. Por el contrario, la crioenfriamiento directa de cristal no es posible en ChipX3 y los cristales no se pueden extraer de su canal microfluídico, que se puede ver como limitaciones de esta configuración. Sin embargo, el protocolo descrito en el artículo proporciona una tubería totalmente integrada para la determinación de estructuras cristalinas a temperatura ambiente (es decir, en condiciones más fisiológicas). A pesar de que la recopilación de datos a temperatura ambiente causa un aumento del daño por radiación19,este efecto se contrarresta por un tiempo de adquisición de datos rápido (se recopila un máximo de rotación de 60° en cada cristal) y mediante la fusión de varios conjuntos de datos parciales. Tanto el diseño chipX como el material fueron optimizados para reducir la dispersión de fondo y la atenuación de la señal de difracción3,y la recopilación de datos se puede realizar en cristales con dimensiones equivalentes a la mitad del tamaño de los canales (40 μm)4.
En resumen, las principales ventajas del protocolo son las siguientes. Los cristales se producen en un entorno libre de convección (canales microfluídicos), que es muy favorable al crecimiento de cristales de alta calidad. El método de contra-difusión implementado en ChipX es muy eficiente en la detección del paisaje de supersaturación; la difusión de cristallantes en el canal de chip crea una onda de concentración y sobresaturación que ayuda a determinar las condiciones de nucleación y crecimiento adecuadas5. Los cristales nunca se manejan directamente, pero se analizan in situ, dentro del chip, que conserva sus propiedades de difracción genuinas (es decir, no altera la mosaicidad cristalina por interacción física o criometración)20. El análisis de difracción se realiza en una serie de cristales distribuidos a lo largo de los canales de chip con baja exposición a dosis para minimizar el daño por radiación, y un conjunto de datos completo se ensambla mediante la combinación de datos parciales de la serie. La huella estándar y el diseño simple de ChipX permitirán en el futuro una automatización completa de la recopilación de datos in situ utilizando instalaciones sincrotrón o XFEL. Todos los pasos del protocolo se llevan a cabo en ChipX. Desde el punto de vista del experimentador, la configuración del chip es simple y fácil de realizar con pipetas estándar y no requiere ningún equipo adicional. La conexión de canal similar a un árbol en la entrada de muestra minimiza los volúmenes muertos en el sistema, lo que es importante cuando se trabaja con muestras que son difíciles de purificar o que solo están disponibles en cantidad limitada.
En conclusión, el enfoque de laboratorio en chip implementado en ChipX simplifica y miniaturiza eficientemente el proceso de cristalización mediante la contra-difusión y la determinación de la estructura de cristal, permitiendo pasar de la muestra a su estructura 3D en un solo dispositivo. Es ampliamente aplicable y ofrece una solución rentable y fácil de usar para investigaciones rutinarias de biocristalografía en serie a temperatura ambiente.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Los autores reconocen a swiss light source (Villigen, Suiza) por su contribución al proyecto en las líneas X10SA (PXII) y X06DA (PXIII), Alexandra Bluhm por su contribución al refinamiento de la estructura, Clarissa Worsdale para la grabación de la voz en off y François Schnell (Université de Strasbourg) por su asistencia en la edición de vídeo y SFX. Este trabajo fue apoyado por el Centro Nacional de la Recherche Scientifique (CNRS), la Universidad de Estrasburgo, el consorcio LabEx "NetRNA" (ANR-10-LABX-0036_NETRNA), una financiación doctoral a R.dW de la iniciativa Excellence (IdEx) de la Universidad de Estrasburgo en el marco del Programa Nacional francés "Investissements d'Avenir", una financiación de doctorado a K.R. de la Universidad Franco-Alemana (UFA-DFH, subvención no. CT-30-19), la Deutsche Forschungsgemeinschaft (concesión no. Mo 634/10-1). Los autores se beneficiaron del programa de cooperación PROCOPE Hubert Curien (Ministerio francés de Asuntos Exteriores y Deutscher Akademischer Austauschdienst).
Materiales
| Name | Company | Catalog Number | Comments |
| Axioscope A1 stereomicroscope | Zeiss | Crystal observation (step 3) | |
| Carboxyrhodamine succinimidyl ester | Invitrogen | C-6157 | Protein labeling (step 2) |
| CMPcPP | Jena Bioscience | NU-438 | Crystal soaking (step 4) |
| Crystal clear sealing tape | Hampton research | HR3-511 | ChipX sealing (step 1) |
| Parafin oil | Hampton research | HR3-411 | ChipX loading (step 1) |
| Ultimaker 2 extended+ | Ultimaker | 3D printer - Representative results | |
| UV light source | Xtal Concepts Gmbh | XtalLight100c | Crystal observation (step 3) |
| Zeba spin desalting column 7K MWCO | ThermoFisher Scientific | 89882 | Protein labeling (step 2) |
Referencias
- Giegé, R., Sauter, C. Biocrystallography: past, present, future. HFSP Journal. 4 (3-4), 109-121 (2010).
- Dhouib, K., et al. Microfluidic chips for the crystallization of biomacromolecules by counter-diffusion and on-chip crystal X-ray analysis. Lab on a Chip. 9 (10), 1412-1421 (2009).
- Pinker, F., et al. ChipX: A Novel Microfluidic Chip for Counter-Diffusion Crystallization of Biomolecules and in Situ Crystal Analysis at Room Temperature. Crystal Growth & Design. 13 (8), 3333-3340 (2013).
- de Wijn, R., et al. A simple and versatile microfluidic device for efficient biomacromolecule crystallization and structural analysis by serial crystallography. IUCrJ. 6 (3), 454-464 (2019).
- García-Ruiz, J. M. A supersaturation wave of protein crystallization. Journal of Crystal Growth. 232 (1-4), 149-155 (2001).
- Otálora, F., Gavira, J. A., Ng, J. D., García-Ruiz, J. M. Counterdiffusion methods applied to protein crystallization. Progress in Biophysics and Molecular Biology. 101 (1-3), 26-37 (2009).
- Pusey, M., Barcena, J., Morris, M., Singhal, A., Yuan, Q., Ng, J. Trace fluorescent labeling for protein crystallization. Acta Crystallographica Section F Structural Biology Communications. 71 (7), 806-814 (2015).
- Meyer, A., Betzel, C., Pusey, M. Latest methods of fluorescence-based protein crystal identification. Acta Crystallographica Section F Structural Biology Communications. 71 (2), 121-131 (2015).
- Betat, H., Rammelt, C., Mörl, M. tRNA nucleotidyltransferases: ancient catalysts with an unusual mechanism of polymerization. Cellular and Molecular Life Sciences. 67 (9), 1447-1463 (2010).
- Ernst, F. G. M., Erber, L., Sammler, J., Jühling, F., Betat, H., Mörl, M. Cold adaptation of tRNA nucleotidyltransferases: A tradeoff in activity, stability and fidelity. RNA Biology. 15 (1), 144-155 (2018).
- Kabsch, W. XDS. Acta Crystallographica. Section D, Biological Crystallography. 66 (2), 125-132 (2010).
- Adams, P. D., et al. PHENIX: a comprehensive Python-based system for macromolecular structure solution. Acta Crystallographica. Section D, Biological Crystallography. 66 (2), 213-221 (2010).
- Dessau, M. A., Modis, Y. Protein Crystallization for X-ray Crystallography. Journal of Visualized Experiments. (47), e2285 (2011).
- Sauter, C., Lorber, B., McPherson, A., Giegé, R., Arnold, E., Himmel, D. M., Rossmann, M. G. Crystallization - General Methods. International Tables of Crystallography, Vol. F, Crystallography of Biological Macromolecules. , 99-120 (2012).
- Garman, E. "Cool" crystals: macromolecular cryocrystallography and radiation damage. Current Opinion in Structural Biology. 13 (5), 545-551 (2003).
- Li, D., Boland, C., Aragao, D., Walsh, K., Caffrey, M. Harvesting and Cryo-cooling Crystals of Membrane Proteins Grown in Lipidic Mesophases for Structure Determination by Macromolecular Crystallography. Journal of Visualized Experiments. (67), e4001 (2012).
- Diederichs, K., Karplus, P. A. Improved R-factors for diffraction data analysis in macromolecular crystallography. Nature Structural Biology. 4 (4), 269-275 (1997).
- Karplus, P. A., Diederichs, K. Linking Crystallographic Model and Data Quality. Science. 336 (6084), 1030-1033 (2012).
- de la Mora, E., Coquelle, N., Bury, C. S., Rosenthal, M., Holton, J. M., Carmichael, I., Garman, E. F., Burghammer, M., Colletier, J. -. P., Weik, M. Radiation damage and dose limits in serial synchrotron crystallography at cryo- and room temperatures. Proceedings of the National Academy of Sciences of the United States of America. 117 (8), 4142-4151 (2020).
- Nave, C. A. Description of Imperfections in Protein Crystals. Acta Crystallographica. Section D, Biological Crystallography. 54 (5), 848-853 (1998).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados