Method Article
酵素の結晶化と構造決定:多用途マイクロ流体チップにおける連続結晶学による基質複合体
要約
多目的なマイクロ流体デバイスは、カウンター拡散法を用いた酵素の結晶化、浸漬による結晶中の基質の導入、および室温でチップ内部の結晶の逐次分析による酵素:基質複合体の3D構造決定を可能にする記載されている。
要約
X線分析前の十分に拡散結晶の調製とその取り扱いは、生体結晶学研究の2つの重要なステップです。逆拡散の効率的な方法により結晶の製造を可能にする多目的なマイクロ流体チップについて述べる。マイクロ流体チャネルによって提供される対流のない環境は、結晶成長に最適であり、結晶酵素の活性部位に基質を拡散するのに有用である。ここでは、このアプローチを、本例においてサイクロ球菌 プラノコッカス・ハロクリオフィルス のCCA付加酵素に適用した。結晶化および基質拡散/浸漬後、酵素:基質複合体の結晶構造を、シリアル結晶学とチップ内部の複数結晶の解析により室温で決定した。全体のプロシージャは、結晶の取り扱いを必要としないため、サンプルの本物の回折特性を保持します。
概要
結晶学は、生体高分子の3Dアーキテクチャを解読する方法である。後者は、酵素がその基質をどのように選択して処理するかを理解するために重要です。結晶構造の決定には、X線回折1によるそれらの分析のために、標的高分子の結晶化と結晶のコンディショニングが必要である。結晶の準備と取り扱いは、結晶の品質と回折特性に影響を与える可能性がありますが、繊細な手順であり、結果として得られる3D構造の分解能(精度)です。高品質の結晶の調製を容易にし、その回折特性を維持するために不要な取り扱いを排除するために、我々はChipX2、3、4と呼ばれるユーザーフレンドリーで汎用性の高いマイクロ流体デバイスを設計しました。
本稿では、従来の実験室材料を用いて、反拡散による結晶の調製を行う方法を、ChipXチャネルにタンパク質溶液をロードする方法を紹介します。この結晶化法は、結晶化剤5,6の拡散によって発生する濃度勾配による酵素溶液を含むマイクロ流体チャネルに沿った過飽和及び潜在的核生成条件の効率的なスクリーニングを提供する。
チップのセットアップは簡単で、標準的な実験室用ピペットのみを使用し、高価な機器を必要としません。結晶がChipXで成長した場合、拡散によって酵素のリガンドを導入することができます。次に、シンクロトロンX線源を用いて、チップのチャネルに含まれる一連の結晶の回折データを室温で収集します。ここで説明した構造研究は、そのアポ形態および浸漬によって導入されたCTP基質の類似体との複合体におけるtRNA成熟酵素の構造の決定につながった。CCA付加酵素と呼ばれるこのタンパク質は、tRNAの3'末端でCCAトリヌクレオチドテールを重合します。連続結晶学によって得られた2つの3D画像を比較すると、クライオ結晶学で使用されるものよりも生理学的な条件でリガンドの結合に関連する局所的な立体構造変化を明らかにする。本ビデオに記載されたプロトコルは、タンパク質、核酸、または多成分複合体であるあらゆる生体分子に一般に適用可能である。
プロトコル
1. ChipXでの結晶化アッセイの設定
注: ChipX マイクロ流体デバイスは著者から入手できます。チップの説明は 図 1に示されています。結晶化を引き起こすために使用される結晶剤(または結晶化剤)を含有する溶液は、市販の起源の、または実験者によって調製され得る。
- 生体分子サンプルのロード
注:チップXの各チャンネルのストレートセクションで個々の対拡散アッセイを実行するために実際に必要なサンプル量は300 nLです。しかし、利便性のために、曲面部と流入死量の可変長を考慮して、8つのチャンネルを完全に埋めるために5μLをロードすることをお勧めします。- 標準的な10 μLのピペットとチップを使用した酵素ソリューションのパイプ5 μL。
- サンプルインレットにチップを垂直に導入し、8つのチャネルが反対側の端まで満杯になるまで溶液を注入します(結晶貯蔵所のエントリ)。
- サンプルインレットに1μLのパラフィンオイルを注入し、チャネルを互いに切り離します。
- 標準の10 μLピペットを使用して、各チャンネルの先端部にある結晶タンク内の余分な溶液を回収します。
- サンプルの入口を1cm x 1 cmのテープで密封します。
- 結晶化溶液のロード
- 標準の10 μLピペットとチップを使用した結晶化液のピペット5 μL。リザーバの体積は10μLですが、テープで密封する際にオーバーフローを回避し、浸漬実験のためにリガンドの追加を容易にします。初期結晶化条件が蒸気拡散によって得られた場合、結晶傾斜濃度を1.5~2倍にする。溶液は、すべての貯留槽で異なることができ(提示された場合には、1Mのリン酸水素水酸ダイウム、100 mM酢酸ナトリウム、pH4.5を全体に使用した)。
- ピペットチップを、液状のリザーバの一部の流路に向けて、溶液の堆積時に気泡が形成されないようにします。それはチャネルに2つの溶液および結晶拡散の間の接触を防ぐだろう。
- 結晶剤溶液をリザーバーに注入します。
- 2.5 cm x 1 cmのテープで貯留槽を密封します。
- チップを20°Cでインキュベートする(温度は目標に応じて調整することができる、通常4〜37°C4)。
2. 蛍光検出用カルボキシローダミンによるタンパク質標識
注: この手順はオプションです。蛍光を使用してチップ内の結晶の検出を容易にするために、サンプルローディングの前に実行する必要があります。蛍光標識の微量化方法は、Puseyと同僚によって7.すべてのステップは室温で行われる。
- 5 mgのカルボキシロダミンエステル粉末を1 mL無水ジメチルホルムアミドに溶解し、-20°Cで保存される0.6 μLアリコートに溶液を分割する。
- 1 M Na-ホレート pH 8.75 ストック溶液を準備します。
- ストックを希釈して0.05 M Na-ホレート pH 8.75 で反応バッファーを調製します。
- 800 μL の反応バッファーを使用して脱塩カラム(7 kDa MWCO、0.5 mL)をリンスします。
- 1400 x gで1分間の列を遠心分離し、濾液を取り除きます。
- この操作を2回繰り返し(ステップ2.4~2.5)、カラムを洗浄します。
- カラムの貯蔵バッファーに80 μLのタンパク質を堆積します(必要に応じてタンパク質を1 mg/mLに希釈して体積を増やすことができます)。
- 1400 x g で 1 分間の列を遠心分離します。このステップは、タンパク質をその貯蔵バッファーから反応バッファーに移すことを意図している。
- フロースルー(反応バッファーにタンパク質を含む)を回収し、0.6 μLのカルボキシロダミン溶液と混合します。
- 室温で5分インキュベートします。
- 一方、カラム3xを記憶バッファーでリンスし、1400 x gでカラムを1分間遠心し、濾液を捨てます。
- 反応液をカラムに付着する。
- 1400 x gでカラムを1分間遠心分離し、フロースルー(すなわち、その貯蔵バッファー内の標識タンパク質の溶液)を回収する。
- 標識タンパク質の0.1-1%(w/w)でストックタンパク質溶液を補います。
- セクション1に記載されているようにChipX結晶化アッセイをセットアップします。
- 520 nm波長の光源で蛍光プローブを励起することにより、アッセイ中のタンパク質結晶の存在を確認します。
3. 結晶観察
注意:温度を制御する必要がある場合を除き、ChipXデバイスは、内部の結晶であっても、特別な注意を払わずに取り扱うことができます。
- 任意の実体顕微鏡を使用して、ChipXの結晶化アッセイの結果を確認します。その足跡は顕微鏡のスライドの標準的な次元を有し、あらゆるシステムおよびスライドのホールダーと互換性がある。
- 結晶傾斜濃度が最も高いリザーバから始まるマイクロ流体チャネルの含有量を、結晶傾斜濃度が最も低いサンプル入口に確認します。ChipX材料は、可視光に対して透明であり、偏光子の使用と互換性があるとともに、本質的なトリプトファン蛍光8によるタンパク質結晶同定のためのUV照明と同様である。
- チャネルに沿ってエンボス加工されたラベルを使用して結晶位置を記録したり、チップ表面上の色点を描いて永久的なマーカーでクリスタルの位置をマークしたりします。
4. リガンドで水晶を浸す
注: この手順はオプションです。リガンド、酵素基質または重原子を結晶に導入するために使用され、チャネルに沿って、結晶に化合物の拡散を可能にするために、X線分析の前に少なくとも24〜48時間を行う必要があります。
- 貯留部からシールテープをそっと取り出します。
- 10 μL マイクロピッドを使用して、1つまたは複数のリザーバに最大 5 μL のリガンド溶液を加える(例えば、3 μLの10 mM シチジン-5'-[(α,β)-メチレーノ]三リン酸(CMPcPP)溶液を3.75 mMの最終濃度を達成した。CMPcPPは、CTPの非水素化可能な類似体であり、酵素の天然基質である。
- 2.5 cm x 1 cmのテープで貯留槽を密封します。
- チップのチャネルに沿ったリガンド拡散を可能にするために、24-48時間の制御された温度でチップをインキュベートします。
5. 連続結晶学による結晶分析
注: このプロトコルの部分は、ビームラインの設定と結晶の回折特性に応じて調整する必要があります。X06DAビームライン(SLS、ヴィリゲン、スイス)で行われた実験に基づいて、結晶学的分析のための一般的な適応症のみが与えられる。
- ビームラインゴニオメータ上のChipXマウント
メモ: ChipX ホルダーを 3D 印刷するためのファイルは ref4にあります。- ビームラインのクライオジェットをオフにします。ここでの分析は室温で行われます。
- チップXを専用ホルダーに取り付け、分析する結晶を含むチャネルをホルダーの中央に配置します。ChipX ホルダ4は、チップX に最適なフィット感を提供するように設計されていたため、スクリューや追加パーツは必要ありません。
- ホルダーをゴニオメーターに取り付けます。
- データ収集
- ChipXの最も厚い層(上層、 図1)を向き(この方向では、チャネルに沿ったラベルはビームラインのセンタリングカメラを使用して直接読み取り可能)、直結ビームと結晶の後ろの最も薄い面に向けて、ref3に記載されているように回折信号の減衰を最小限に抑える。
- 周囲の材料(ビームストップ、コリメータ)とのChipXの衝突を避けるために、X線ビームに垂直なチャネルに対応する±30°の範囲でゴニオメーターの動きを制限します。
- チャネルに沿って浮き彫りにされたラベルの助けを借りて、結晶の位置を見つけます。
- 結晶位置を選択します。
- 標準の低線量グリッド/ラスタースクリーニングまたは1クリック手順(ビデオはグリッドスクリーニングの例を示しています)のいずれかで結晶を中心に配置します。
- -30°/+30°の範囲内の回折データを収集します。
- チップの変換後、同じチャンネル内の別の結晶で手順5.2.4~5.2.6で手順を再開します。
- ホルダーの中央にある別のChipXチャンネルを手動で再調整し、このチャンネルに存在する結晶上でデータ収集を実行します。
- 標準的な結晶構造パッケージと手順を使用してデータを処理およびマージし、構造を解析し、改良します。
結果
ここで説明するマイクロ流体チップは、結晶化アッセイの設定と結晶分析を室温で容易に行えるように設計されています。上記の手順およびビデオでは、低温適合細菌プラノコッカスハロクリオフィルスからのCCA付加酵素の構造的特徴付けのフレームに適用した。この酵素は、CTPおよびATP9,10を用いてtRNA上に3'CCA配列の順次添加を触媒する必須ポリメラーゼファミリーに属する。
このチップは、まず、逆拡散法による構造解析用酵素の結晶を調製するために使用された。この結果、酵素溶液は、チップのサンプル入口に1回の注入によって8つのマイクロ流体チャネル(結晶化チャンバー)にロードされた( 図1参照)。この酵素は、20 mMトリス/HCl pH 7.5、200 mM NaClおよび5 mM MgCl2を含む貯蔵バッファー内の5.5 mg/mLで使用された。このステップは標準の10 μLマイクロピペットを使用して手動で行った。結晶化液(100 mM酢酸ナトリウムpH 4.5,1 Mリン酸ナトリウム)を、チャネルの他の極端の貯留槽に沈着させた。
読み込み手順は簡単で、5 分以上かかるわけではありません (図 2)。その後、結晶傾斜はチャネルに拡散し、結晶核化および成長を引き起こす濃度の勾配を作り出す。この勾配は動的に進化し、チャネルとリザーバの間の結晶傾斜濃度の平衡に達するまで、過飽和状態5、6の連続を探る。結晶化アッセイは、通常、結晶の成長を追跡するために2〜4週間の期間にわたってミコスコープの下でチェックされます。CCA付加酵素の双錐体結晶は、20°Cでのインキュベーションの数日後にチャネル全体に現れた(図3)。このタンパク質の任意蛍光標識7は、タンパク質結晶の同定及び塩結晶からのそれらの識別を大いに促進する(図4)。
私たちは、結晶を構築する酵素に基質を提供するために、チップチャネルの拡散環境を利用しました。本例では、CTPアナログのCMPcPPを、3.75mMの最終濃度でリザーバ溶液に添加した(図5)。この添加は、結晶構造によって後で確認されるように、CMPcPPが酵素の触媒部位に到達し、占有することを可能にする結晶学的分析の2日前に行われた(下記参照)。
3Dプリンタを用いて、ポリ乳酸中のチップホルダー(図6)を製造しました。ホルダーは標準的な磁気ヘッドを使用してゴニオメータにチップマウントを可能にする。したがって、チップはX線ビームに簡単に配置および変換して、結晶を回折位置に持ち込むことができます。データ収集戦略は、ビームラインの特性や結晶特性に応じて適応する必要があります。CCA付加酵素の場合、X06DAとX10SAビームライン、スイス光源(SLS)、X線波長1.0Å、ピラトゥス2M-Fおよび6Mピクセル検出器でデータを収集した。30〜60°の回転は、0.1°または0.2°および0.1 s露光の画像を用いて室温で各結晶に集めた(表1を参照)。部分的なデータセットは、放射損傷(信号対雑音比と 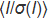 CC1/2の減少、高解像度シェルでのRmeasの増加によって検出された)による回折パターンの分解能が減衰し始めたときに個別に処理され、切断された。5 つの結晶のデータをマージして、データセット全体を再構成しました (表 1) 。結晶構造は、標準的な結晶パッケージとデータ処理11および精製12のための手順を用いて分子置換によって導出された。酵素の構造とCMPcPPとの複合体の構造を比較することで、CCA付加酵素の活性部位における基質結合に伴う局所的な立体構造の適応が明らかになった(図7)。
CC1/2の減少、高解像度シェルでのRmeasの増加によって検出された)による回折パターンの分解能が減衰し始めたときに個別に処理され、切断された。5 つの結晶のデータをマージして、データセット全体を再構成しました (表 1) 。結晶構造は、標準的な結晶パッケージとデータ処理11および精製12のための手順を用いて分子置換によって導出された。酵素の構造とCMPcPPとの複合体の構造を比較することで、CCA付加酵素の活性部位における基質結合に伴う局所的な立体構造の適応が明らかになった(図7)。
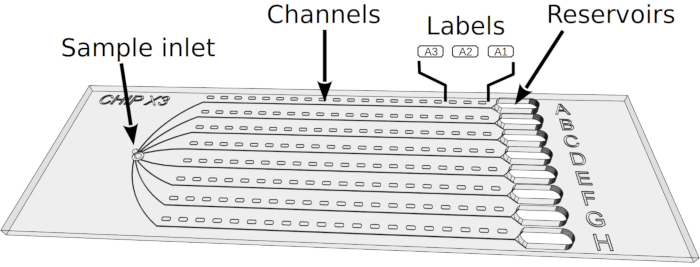
図1:ChipXデザイン チップはCOC(厚さ:1mm)で構成され、8つのマイクロ流体チャネルと貯留層がインプリントされます。チップ全体はCOC(厚さ:0.1mm)の層で密封されています。すべてのチャネルは同時サンプル注入のための左側の単一の入口および結晶化解決が堆積する右側の個々の貯留器に接続される。チップの実際の結晶化チャンバを構成するチャネルは、長さ4cmで、80μm x 80 μmの断面を有する。チャネルに沿ってエンボス加工されたラベル(A1、A2、A3など)は、顕微鏡下での結晶配置とデータ収集用サンプルリストの作成を容易にする。ChipXは標準的な顕微鏡のスライド(7.5 cm x 2.5 cm)のサイズを有する。 この図の大きなバージョンを表示するには、ここをクリックしてください。
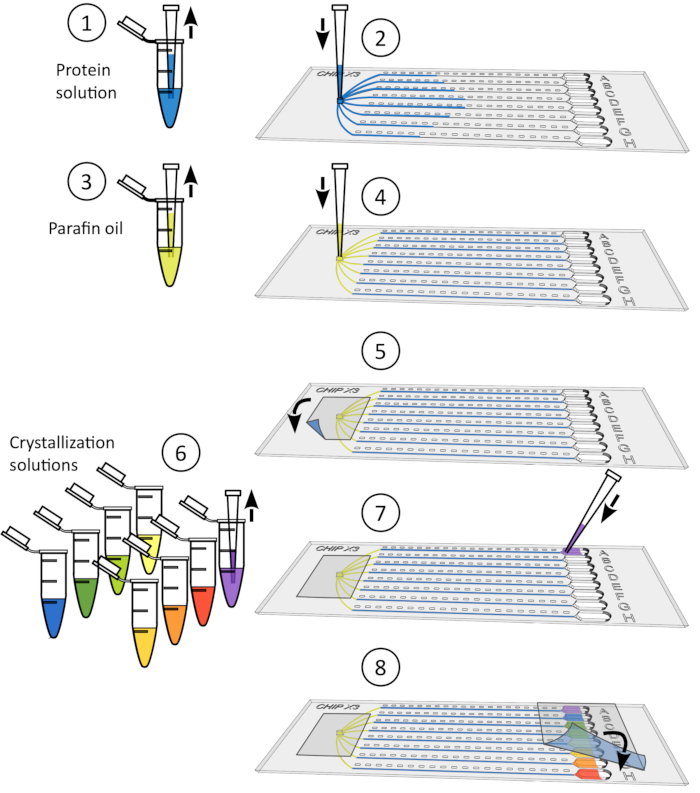
図2:ChipXで結晶化アッセイを設定する 1)標準の10 μLピペレットとチップを使用して5~6 μLの酵素溶液を堆積させる。2) サンプル入口にチップを縦に入れ、8つのチャネルに溶液を注入します。3) パラフィン油1μLをピペット。4)チップをサンプルの入口に縦に入れ、油を注入して、チャネルを互いに切り離します。5) 入口をテープで密封します。6) 標準の10 μLのピペットとチップを使用した結晶化液の5 μLをピペット。ソリューションは、すべての貯水池で異なることができます(例えば、スクリーニングキットから)。7)ピペット先端を、貯留層の漏斗状部分のチャネルの入り口に向け(溶液堆積時に気泡が形成されないようにするため)、水晶溶液をリザーバーに注入する。8)テープで貯水池を密封し、制御された温度でチップをインキュベートします。 この図の大きなバージョンを表示するには、ここをクリックしてください。

図3: ChipXのマイクロ流体チャネルにおける対拡散によって成長したCCA付加酵素の結晶スケールバーは0.1 mmです。この図の大きなバージョンを表示するには、ここをクリックしてください。
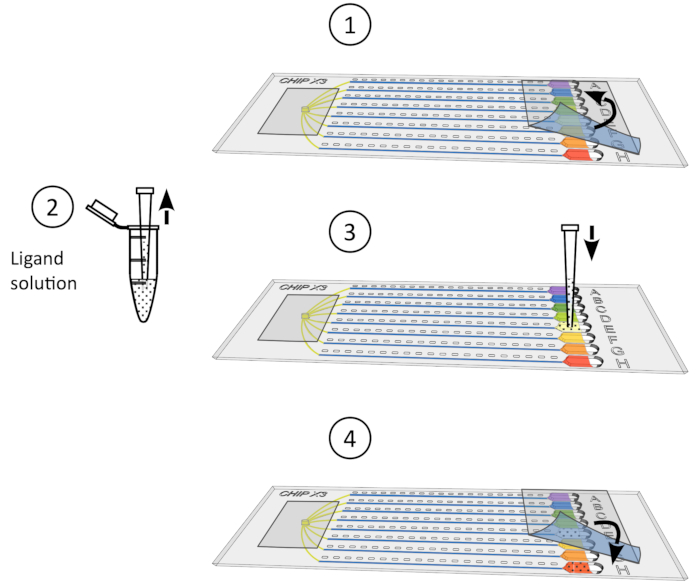
図4:水晶浸漬手順1)は、静かにリザーバからテープを取り出します。2) 10 μL マイクロピペットを用いて最大5 μLのリガンド溶液を堆積する。3)リガンドを1つまたは複数の貯留層に加える。4)テープで再びリザーバを密封し、データ収集の前に24〜48時間制御された温度でチップをインキュベートします。 この図の大きなバージョンを表示するには、ここをクリックしてください。

図5:微量蛍光標識は、タンパク質(左)と塩(右)結晶を区別する。 CCA添加酵素溶液には、カルボキシローダミンで標識されたタンパク質が0.4%(w/w)含まれていました。右側では、結晶は520 nm波長の光源で照らされ、画像は550 nm(LP550)のローパスフィルターで撮影されます。カルボキシローダミン-スクシニミジルエステルの(インセット)構造。 この図の大きなバージョンを表示するには、ここをクリックしてください。

図6:(左)シリアル結晶分析用のSLS(スイス・ヴィリゲン)でビームラインX06DAのゴニオメーターに装着されたChipXホルダーと(右)ChipXの描画。
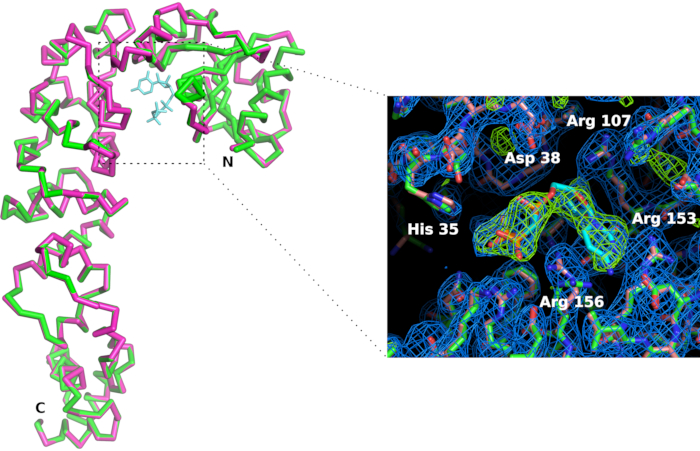
図7: アポ形(ピンク)とCTPアナログ(緑色)と複合体中のCCA付加酵素活性部位の比較酵素の全体的な立体構造は影響を受けませんが、CMPcPPリガンドの結合は、活性部位における側鎖のわずかな再編成を伴います。2Fo-Fc電子密度マップ(青色)は1.2シグマで輪郭を描く。4シグマ(緑色)で輪郭を描いた差電子密度マップは、活性部位におけるリガンドの存在を確認する。この図の大きなバージョンを表示するには、ここをクリックしてください。
| 結晶化サンプル | CCA付加酵素 | CCA添加酵素 + CMPcPP |
| 結晶分析 | ||
| X線ビームライン | SLS – X06DA | SLS – X10SA |
| 波長(Å) | 1.000 | 1.000 |
| 温度 (K) | 293 | 293 |
| 検出 器 | ピラタス2M-F | ピラタス 6M |
| 結晶検出器距離 (mm) | 300 | 400 |
| 収集された結晶 | 6 | 14 |
| 選択された結晶 | 5 | 5 |
| 画像あたりの回転範囲 (°) | 0.1 | 0.2 |
| 画像あたりの露出時間(複数可) | 0.1 | 0.1 |
| いいえ。選択した画像の | 1000 | 540 |
| 総回転範囲 (°) | 100 | 108 |
| スペースグループ | P43212 | P43212 |
| a, c (Å) | 71.5, 293.8 | 71.4, 293.6 |
| 平均モザイク(°) | 0.04 | 0.04 |
| 解像度の範囲 (Å) | 46 – 2.54 (2.6 – 2.54) | 48 – 2.3 (2.4 – 2.3) |
| 合計 No.反射の | 176105 (9374) | 232642 (32937) |
| いいえ。独自の反射の | 23922 (1598) | 34862 (4066) |
| 完全性 (%) | 90.6 (84.6) | 99.5 (100.0) |
| 冗長性 | 7.5 (6.0) | 6.7 (8.1) |
 | 8.1 (1.3) | 6.9 (0.7) |
| ルメス (%) § | 18.6 (126.0) | 18.0 (231.2) |
| CC1/2 (%) | 98.7 (55.0) | 98.7 (46.9) |
| ウィルソンプロットからの全体的なB因子(Å2) | 57.4 | 60.6 |
| 結晶学的改良 | ||
| いいえ。反射の,ワーキングセット/テストセット | 23583 / 1180 | 34840 / 3405 |
| ファイナル・ルクリスト (%) / Rフリー (%) | 18.8 / 21.4 | 20.0 / 22.9 |
| いいえ。非H原子の:全体/タンパク質/リガンド/溶媒 | 2998 / 2989 / 0 / 9 | 3057 / 2989 / 29 / 10 |
| 結合のR.m.s. 偏差 (Å) / 角度 (°) | 0.009 / 1.23 | 0.010 / 1.22 |
| 平均B因子(Å2):全体/タンパク質/リガンド/溶媒 | 60.1 / 60.1 / 0 / 52.7 | 62.5 / 62.6 / 60.1 / 55.5 |
| ラマチャンドランプロット:最も好まれる(%)/許可された(%) | 98.1 / 1.9 | 97.2 / 2.8 |
| PDB ID | 6IBP | 6Q52 |
表 1:データ収集および絞り込み統計
§ 冗長性非依存Rmeas = Σhkl(N/N-1)1/2Σi |I I (hkl)- (hkl) >|/ ΣhklΣi i(hkl)は、ここで N はデータ多重度17 である。
外部シェル (<2.0) の低い を持つデータは、CC1/2 基準 (データセットの 2 つのランダムな半分の相関 > 50%)カルプラス&ディーデリッヒス 18によって提案されたように.
ディスカッション
生体結晶学における電流プロトコルは、凍結原色の条件下で窒素ジェットで回折解析を行う前に、蒸気拡散またはバッチ13,14などの方法を用いて結晶を調製し、その結晶を凍結冷却用マイクロループ15,16に移すことを含む。これに対して、直接結晶の凍結冷却はChipX3では不可能であり、結晶はマイクロ流体チャネルから抽出することができず、このセットアップの限界と見なすことができます。しかしながら、本稿に記載されたプロトコルは、室温での結晶構造の決定(すなわち、より生理的な条件下)を完全に統合したパイプラインを提供する。室温でのデータ収集は放射線損傷の増加を引き起こしますが、この効果は高速データ取得時間(各結晶で最大60°回転が収集される)と複数の部分データセットのマージによって相殺されます。ChipXの設計と材料の両方がバックグラウンド散乱と回折信号減衰3を低減するように最適化され、データ収集は、チャネル(40 μm)4の半分のサイズに相当する寸法の結晶に対して行うことができる。
要約すると、プロトコルの主な利点は以下の通りです。結晶は対流のない環境(マイクロ流体チャネル)で作製され、高品質の結晶の成長に非常に有利です。ChipXで実装されているカウンター拡散法は、過飽和風景のスクリーニングに非常に効率的です。結晶剤のチップチャネルへの拡散は、適切な核生成および成長条件5を決定するのに役立つ濃度と過飽和波を作成します。結晶は、直接扱われることはないが、その場で分析され、チップ内部で分析され、その真の回折特性を保持する(すなわち、物理的相互作用や低温冷却によって結晶モザイク性を変化させない)20。回折解析は、放射の損傷を最小限に抑えるために、チップチャネルに沿って分布する一連の結晶に対して行われ、完全なデータセットはシリーズの部分的なデータをマージすることによって組み立てられます。ChipXの標準的な足跡および簡単な設計は同期またはXFELの設備を使用して、将来的 にsitu データ収集の完全な自動化を可能にする。プロトコルのすべてのステップはChipXで行われます。実験者の視点から、チップのセットアップは、標準のピペットで簡単かつ簡単に実行でき、余分な機器を必要としません。サンプルインレットの木のようなチャネル接続は、システム内のデッドボリュームを最小限に抑え、浄化が困難なサンプルや限られた量でのみ利用可能なサンプルを扱う場合に重要です。
結論として、ChipXに実装されているラボ・オン・チップ・アプローチは、逆拡散と結晶構造の決定によって結晶化のプロセスを簡素化し、効率的に小型化し、サンプルから3D構造に単一のデバイスで行くことを可能にします。広く適用可能であり、室温での定期的な連続生体結晶学の調査のためのユーザーフレンドリーで、費用効果の高い解決を提供する。
開示事項
著者らは開示するものは何もない。
謝辞
著者らは、ビームラインX10SA(PXII)とX06DA(PXIII)、アレクサンドラ・ブルームの構造改良への貢献、ナレーションの録音のためのクラリッサ・ワースデール、ビデオ編集とSFXでの彼の支援のためのフランソワ・シュネル(ストラスブール大学)に関するプロジェクトへのビームタイム割り当てのためのスイス光源(ヴィリゲン、スイス)を認める。この作品は、ストラスブール大学フランス国立国立レシェルシュ・サイエンティフィック(CNRS)によって支援されました。 フランス国立プログラム「投資ダベニール」の枠組みの中でストラスブール大学のエクセレンス・イニシアチブ(IdEx)からR.dWへの博士号を調達したLabExコンソーシアム「NetRNA」(ANR-10-LABX-0036_NETRNA)は、フランス・ドイツ大学(UFA-DFH、助成金なし)からK.R.への博士号を取得しました。CT-30-19)、ドイツ・フォルシュングスゲマイインシャフト(グラント・ノー。Mo 634/10-1)。著者らはPROCOPEヒューバート・キュリアン協力プログラム(フランス外務省とドイツ・アカデミスチャー・アウシュディエンスト)の恩恵を受けた。
資料
| Name | Company | Catalog Number | Comments |
| Axioscope A1 stereomicroscope | Zeiss | Crystal observation (step 3) | |
| Carboxyrhodamine succinimidyl ester | Invitrogen | C-6157 | Protein labeling (step 2) |
| CMPcPP | Jena Bioscience | NU-438 | Crystal soaking (step 4) |
| Crystal clear sealing tape | Hampton research | HR3-511 | ChipX sealing (step 1) |
| Parafin oil | Hampton research | HR3-411 | ChipX loading (step 1) |
| Ultimaker 2 extended+ | Ultimaker | 3D printer - Representative results | |
| UV light source | Xtal Concepts Gmbh | XtalLight100c | Crystal observation (step 3) |
| Zeba spin desalting column 7K MWCO | ThermoFisher Scientific | 89882 | Protein labeling (step 2) |
参考文献
- Giegé, R., Sauter, C. Biocrystallography: past, present, future. HFSP Journal. 4 (3-4), 109-121 (2010).
- Dhouib, K., et al. Microfluidic chips for the crystallization of biomacromolecules by counter-diffusion and on-chip crystal X-ray analysis. Lab on a Chip. 9 (10), 1412-1421 (2009).
- Pinker, F., et al. ChipX: A Novel Microfluidic Chip for Counter-Diffusion Crystallization of Biomolecules and in Situ Crystal Analysis at Room Temperature. Crystal Growth & Design. 13 (8), 3333-3340 (2013).
- de Wijn, R., et al. A simple and versatile microfluidic device for efficient biomacromolecule crystallization and structural analysis by serial crystallography. IUCrJ. 6 (3), 454-464 (2019).
- García-Ruiz, J. M. A supersaturation wave of protein crystallization. Journal of Crystal Growth. 232 (1-4), 149-155 (2001).
- Otálora, F., Gavira, J. A., Ng, J. D., García-Ruiz, J. M. Counterdiffusion methods applied to protein crystallization. Progress in Biophysics and Molecular Biology. 101 (1-3), 26-37 (2009).
- Pusey, M., Barcena, J., Morris, M., Singhal, A., Yuan, Q., Ng, J. Trace fluorescent labeling for protein crystallization. Acta Crystallographica Section F Structural Biology Communications. 71 (7), 806-814 (2015).
- Meyer, A., Betzel, C., Pusey, M. Latest methods of fluorescence-based protein crystal identification. Acta Crystallographica Section F Structural Biology Communications. 71 (2), 121-131 (2015).
- Betat, H., Rammelt, C., Mörl, M. tRNA nucleotidyltransferases: ancient catalysts with an unusual mechanism of polymerization. Cellular and Molecular Life Sciences. 67 (9), 1447-1463 (2010).
- Ernst, F. G. M., Erber, L., Sammler, J., Jühling, F., Betat, H., Mörl, M. Cold adaptation of tRNA nucleotidyltransferases: A tradeoff in activity, stability and fidelity. RNA Biology. 15 (1), 144-155 (2018).
- Kabsch, W. XDS. Acta Crystallographica. Section D, Biological Crystallography. 66 (2), 125-132 (2010).
- Adams, P. D., et al. PHENIX: a comprehensive Python-based system for macromolecular structure solution. Acta Crystallographica. Section D, Biological Crystallography. 66 (2), 213-221 (2010).
- Dessau, M. A., Modis, Y. Protein Crystallization for X-ray Crystallography. Journal of Visualized Experiments. (47), e2285 (2011).
- Sauter, C., Lorber, B., McPherson, A., Giegé, R., Arnold, E., Himmel, D. M., Rossmann, M. G. Crystallization - General Methods. International Tables of Crystallography, Vol. F, Crystallography of Biological Macromolecules. , 99-120 (2012).
- Garman, E. "Cool" crystals: macromolecular cryocrystallography and radiation damage. Current Opinion in Structural Biology. 13 (5), 545-551 (2003).
- Li, D., Boland, C., Aragao, D., Walsh, K., Caffrey, M. Harvesting and Cryo-cooling Crystals of Membrane Proteins Grown in Lipidic Mesophases for Structure Determination by Macromolecular Crystallography. Journal of Visualized Experiments. (67), e4001 (2012).
- Diederichs, K., Karplus, P. A. Improved R-factors for diffraction data analysis in macromolecular crystallography. Nature Structural Biology. 4 (4), 269-275 (1997).
- Karplus, P. A., Diederichs, K. Linking Crystallographic Model and Data Quality. Science. 336 (6084), 1030-1033 (2012).
- de la Mora, E., Coquelle, N., Bury, C. S., Rosenthal, M., Holton, J. M., Carmichael, I., Garman, E. F., Burghammer, M., Colletier, J. -. P., Weik, M. Radiation damage and dose limits in serial synchrotron crystallography at cryo- and room temperatures. Proceedings of the National Academy of Sciences of the United States of America. 117 (8), 4142-4151 (2020).
- Nave, C. A. Description of Imperfections in Protein Crystals. Acta Crystallographica. Section D, Biological Crystallography. 54 (5), 848-853 (1998).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved