Method Article
Realización de mediciones FRET precisas y precisas de una sola molécula utilizando el smfBox de código abierto
* Estos autores han contribuido por igual
En este artículo
Resumen
Este artículo proporciona instrucciones paso a paso para realizar mediciones FRET precisas y totalmente corregidas en biomoléculas individuales y de difusión libre utilizando el smfBox de código abierto y económico, desde el encendido, pasando por la alineación y el enfoque, hasta la recopilación y el análisis de datos.
Resumen
El smfBox es un instrumento de código abierto y rentable recientemente desarrollado para la transferencia de energía de resonancia Förster de molécula única (smFRET), que hace que las mediciones en biomoléculas de difusión libre sean más accesibles. Esta descripción general incluye un protocolo paso a paso para usar este instrumento para realizar mediciones de eficiencias FRET precisas en muestras de ADN dúplex, incluidos detalles de la preparación de la muestra, configuración y alineación del instrumento, adquisición de datos y rutinas de análisis completas. El enfoque presentado, que incluye cómo determinar todos los factores de corrección necesarios para las mediciones precisas de distancia derivadas de FRET, se basa en un gran cuerpo de trabajo colaborativo reciente en toda la Comunidad FRET, que tiene como objetivo establecer protocolos estándar y enfoques de análisis. Este protocolo, que es fácilmente adaptable a una gama de sistemas biomoleculares, se suma a los crecientes esfuerzos en la democratización de smFRET para la comunidad científica en general.
Introducción
La transferencia de energía de resonancia de Förster de molécula única (smFRET) es una técnica que mide la eficiencia FRET entre dos colorantes, un donante y un aceptor, a nivel de moléculas individuales. FRET es un proceso fotofísico que surge de la superposición de espectros de energía de dos colorantes: el donante es excitado por la luz de una longitud de onda específica y transfiere energía no radiativamente al aceptor, lo que resulta en la emisión del aceptor. La eficiencia de esta transferencia es inversamente proporcional a la sexta potencia de la distancia entre los dos tintes, por lo que la eficiencia de transferencia varía con la distancia1. Por lo tanto, esta eficiencia FRET se puede utilizar para determinar la información espacial sobre la(s) molécula(s)2 a la que están unidos los colorantes, dentro de un rango de 3-10 nm. Esta escala, y el hecho de que los cambios en la eficiencia de FRET son sensibles a los movimientos moleculares de Angstrom3, hace que la técnica sea muy adecuada para investigar información estructural sobre biomoléculas, como ácidos nucleicos y proteínas, sin las complicaciones del promedio de conjuntos4,5,6. Si bien los cambios en las eficiencias relativas de FRET se pueden usar para monitorear las interacciones biomoleculares y la dinámica conformacional, arrojando luz sobre procesos celulares clave como el (des)plegamiento de proteínas, la transcripción y la replicación y reparación del ADN, las eficiencias absolutas de FRET se han utilizado para determinar distancias precisas para la determinación de la estructura biomolecular7,8,9,10,11 , superando la necesidad de cristalización o congelación como se requiere para algunos otros métodos estructurales4,12.
Los experimentos smFRET comúnmente toman dos formas, microscopía de fluorescencia de reflexión interna total o confocal (TIRF). Entre ambos enfoques, la dinámica molecular de las biomoléculas se puede investigar típicamente en escalas de tiempo de pico- a milisegundos (moléculas confocales, de difusión libre) hasta milisegundos a horas (TIRF, moléculas inmovilizadas de superficie). Esto se debe a las diferentes configuraciones involucradas en cada técnica. En la microscopía TIRF, las moléculas se inmovilizan en la superficie de un portaobjetos y se excitan mediante una onda evanescente (Figura 1A). Aquí, sin embargo, la atención se centra en la microscopía confocal, ya que este es el formato del smfBox. En la microscopía confocal, las moléculas no se inmovilizan y, en cambio, se difunden libremente a través del movimiento browniano a través del volumen confocal (~ 1 fL), formado por el enfoque de un rayo láser a través de una lente de alta apertura numérica en un punto a cierta profundidad designada dentro de la solución (Figura 1B). La emisión resultante se enfoca de nuevo a través de la misma apertura y se filtra a través de un espejo dicroico (Figura 1C para esquema completo). Luego se enfoca a través de un agujero de alfiler para eliminar cualquier luz desenfocada y en un fotodiodo de avalancha (APD). Cuando el APD detecta un fotón, emite un pulso TTL, cuya sincronización se puede grabar con una resolución de hasta picosegundos. El tiempo de observación de estas moléculas que se difunden libremente en las proximidades del volumen confocal es comúnmente del orden de milisegundos.
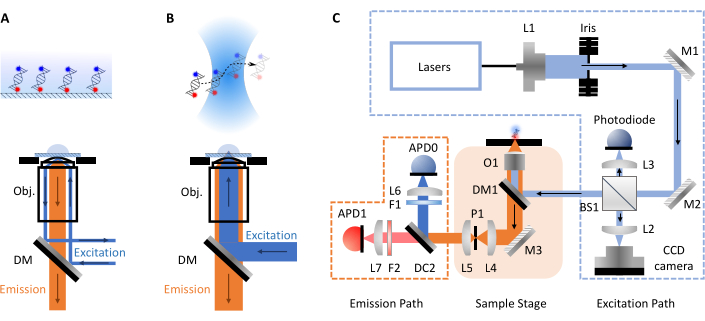
Figura 1: Esquemas que muestran los principios de la microscopía y la configuración de smfBox. (A) Fluorescencia de reflexión interna total (TIRF) Principio de microscopía: la luz de excitación se dirige hacia el borde del objetivo (Obj.) y se somete a una reflexión interna total en la interfaz coverslip-buffer generando un campo de evanescencia en descomposición exponencial para excitar las moléculas unidas a la superficie. (B) Microscopía confocal: Las moléculas que se difunden libremente son excitadas por un punto casi limitado por difracción enfocado en la muestra. (C) La configuración de smfBox utilizada en este protocolo, que muestra todos los componentes clave: fotodiodos de avalancha (APD), divisor de haz (BS), espejos dicroicos (DM), filtros (F), espejos (M), objetivo (O) y agujero de alfiler (P). Haga clic aquí para ver una versión más grande de esta figura.
Más recientemente, las técnicas smFRET incorporaron dos excitaciones de color, donde se alternan láseres que coinciden con las longitudes de onda de excitación del donante y del aceptor5. Esto se puede hacer de una de dos maneras, la primera modulando láseres de onda continua en la escala de tiempo KHz, lo que se conoce como excitación láser alterna (ALEX)13,14. El segundo método intercala pulsos rápidos en la escala de tiempo MHz; se trata de nanosegundos-ALEX15 o excitación intercalada pulsada (PIE)16. En todos estos enfoques, la información del láser aceptor conduce al cálculo de la llamada estequiometría, que puede discriminar entre moléculas con una baja eficiencia FRET y aquellas que carecen de un aceptor (ya sea a través de un etiquetado incompleto o fotoblanqueo). El uso de PIE/ns-ALEX también da acceso a la vida útil fluorescente a nivel de molécula única, y las anisotropías se pueden medir cuando se combinan con ópticas polarizadoras. Esta combinación de mediciones se conoce como detección de fluorescencia multiparamétrica (MFD)9.
A pesar de las muchas ventajas de smFRET, no se usa ampliamente fuera de los laboratorios especializados debido a los altos costos de los instrumentos comerciales y la falta de alternativas simples y de autoconstrucción. Se está produciendo una tendencia creciente hacia el desarrollo de microscopía de código abierto de bajo costo y recientemente han surgido otras plataformas, como Planktonscope17, OpenFlexure Microscope18, Flexiscope19, miCube20, liteTIRF21 y Squid22. Aquí el estudio describe el protocolo para el uso de smfBox, una configuración confocal rentable recientemente desarrollada capaz de medir la eficiencia FRET entre dos colorantes en moléculas individuales de difusión libre. Las instrucciones de construcción detalladas y todo el software operativo necesario están disponibles gratuitamente en: https://craggslab.github.io/smfBox/ 23. La disposición óptica del smfBox se ensambla a partir de componentes fácilmente disponibles comprados a fabricantes asequibles y ampliamente accesibles, mientras que el cuerpo del microscopio (responsable de la mayor parte del gasto en una configuración confocal estándar) ha sido reemplazado por una caja de aluminio anodizado hermética a la luz personalizada (que permite realizar mediciones en condiciones de luz ambiental). Esta caja alberga componentes ópticos clave, incluyendo la excitación dicroica, objetiva y estenopeica, y un enclavamiento láser mecánico, lo que permite su funcionamiento seguro como un producto láser de Clase I (consulte la Figura 1C para un esquema completo). El smfBox utiliza ALEX para validar la estequiometría del tinte y determinar los factores de corrección FRET precisos. Se opera utilizando software de código abierto escrito a medida (smOTTER), que controla todos los aspectos de la adquisición de datos y emite los datos en el formato de código abierto photon-HDF524, compatible con muchas herramientas de análisis de terceros. El smfBox y los protocolos de adquisición y análisis de datos fueron probados recientemente contra otros >20 instrumentos (tanto confocales como TIRF) en un estudio ciego multilaboratorio25. Las eficiencias FRET obtenidas estaban en excelente acuerdo con todos los demás instrumentos, a pesar de que el smfBox costaba solo una fracción del precio de las configuraciones disponibles comercialmente.
Aquí, se describe un protocolo paso a paso para adquirir y analizar eficiencias FRET precisas y absolutas en dúplex de ADN de difusión libre utilizando smfBox, desde el encendido, pasando por la alineación y el enfoque, hasta la recopilación y el análisis de datos. Las muestras utilizadas aquí son tres ADN dúplex (que exhiben eficiencias fret altas, medias y bajas, ver Tabla 1) que fueron evaluadas en el estudio ciego mundial25; sin embargo, el método es adaptable a muchos sistemas moleculares, incluyendo proteínas y otros ácidos nucleicos. La esperanza es que un protocolo tan detallado, junto con las instrucciones de construcción ya existentes para el smfBox23, ayude a que esta poderosa técnica sea aún más accesible para una amplia gama de laboratorios.
Protocolo
1. Componentes de encendido
- Encienda los seis enchufes (sin orden particular): Piezoconcept (z-focus), APD0, APD1, láser verde, láser rojo y fotodetector.
2. Configuración de software 1-Experimental
- Inicie el centro de control láser.
- Asegurar onda continua: se selecciona el modo de corriente constante alterna (CW-ACC ).
- Encendido en ambos láseres.
- Compruebe/ajuste las potencias del láser.
NOTA: La potencia del láser deberá ajustarse como se mide justo antes del dicroico de excitación, ya que los filtros ND y los divisores de haz en la ruta de excitación reducirán la potencia del láser del número dado en el panel de control del láser. Los números dados aquí son potencia en la excitación dicroica, pero la potencia en el control láser que corresponde a esto tendrá que ser resuelta.- Láser verde de 220 μW (515 nm, 40 mW)
- μ70 W para láser rojo (638 nm, 10 mW)
- Software de adquisición smOTTER
- Conecte los láseres, los detectores, la etapa z y la cámara. Configúrelos correctamente (puede variar según la configuración de la tarjeta NI en particular).
3. Alineación de la ruta de emisión (no se requiere de forma rutinaria)
- Configuración de la alineación
- Pipetear 10 μL de colorante Cy3B libre (~100 nM) en el microscopio y enfocar como se describe en los pasos 4.3-4.5 a continuación.
NOTA: Otro tinte donante con alguna fuga en el canal aceptor también funcionaría. - Abra la pestaña Alineación en smOTTER, reduzca la potencia del láser y modifique la escala del eje Y hasta que se vea la lectura desde los detectores.
NOTA: El objetivo aquí es aumentar la señal, por lo que es posible que sea necesario cambiar la escala nuevamente después de que la señal aumente. - Desenrosque los cuatro tornillos en la parte delantera del smfBox y retire el panel frontal.
- Pipetear 10 μL de colorante Cy3B libre (~100 nM) en el microscopio y enfocar como se describe en los pasos 4.3-4.5 a continuación.
- Alineación estenopeica
- Gire la perilla x en el posicionador estenopeico mientras observa la señal en la pestaña Alineación, tratando de aumentar la señal en verde y rojo.
- Ahora gire la perilla y para alinear el agujero de alfiler en la otra dirección.
- Vuelva a la perilla x para comprobar si hay un aumento adicional en la señal.
- Alineación estenopeica-lente
- Gire la lente x perilla en una dirección, esto disminuirá la señal.
- Gire la perilla x agujero en la misma dirección para aumentar la señal nuevamente.
- Si la nueva señal máxima es más alta que antes, continúe moviendo iterativamente tanto el agujero de alfiler como la lente en esa dirección. Si es más bajo que antes, muévase iterativamente en la dirección opuesta.
- Repita arriba para y.
- Alineación de lentes APD
- Comenzando con el APD verde, mueva la perilla x hasta que la señal verde esté en un máximo.
- Repita para la perilla y.
- Regrese a la perilla x, muévase hacia adelante y hacia atrás para encontrar los puntos umbral donde la señal comienza a caer, y déjela en una posición a medio camino entre estos dos puntos.
- Repita para la perilla x.
- Repita los pasos anteriores para la lente APD roja, observando la señal roja.
- Coloque el panel frontal de nuevo en el smfBox y reemplace los tornillos.
NOTA: Realice la alineación anterior mensualmente, y como medida de precaución si se sospecha que el microscopio ha desarrollado una falla de alineación entre las mediciones.
4. Adquisición de datos de medición
- Para la primera muestra, limpie la etapa con tejido de limpieza de lentes empapado en metanol; limpie suavemente el objetivo de un extremo a otro.
- Aplique 3-4 gotas de aceite de inmersión para microscopía en el centro del objetivo; reponer según sea necesario entre muestras.
- Preparación de muestras
- Pipetear 10 μL de muestra (primer uso de agua ultrapura Tipo I) en el centro de un vaso de cubierta limpia.
- Coloque cuidadosamente el vidrio de la cubierta sobre la lente del objetivo bajándolo en ángulo con el aceite para evitar que queden atrapadas las burbujas de aire entre el vidrio de la cubierta y el objetivo.
NOTA: Si es necesario, deslice el vidrio de la cubierta para empujar las burbujas de aire en el aceite lejos del punto focal.
- Para realizar experimentos de excitación láser alterna (ALEX), configure el láser en el panel de ciclos de trabajo láser de la siguiente manera.
- Donante (láser verde) 0 apagado, 45 encendido, 55 apagado
- Aceptor (láser rojo) 50 apagado, 45 encendido, 5 apagado
- Período ALEX (μs): 100
- Haga clic en la pestaña enfoque Z en la cinta; en el panel de adquisición, cambie los láseres a Live e inicie la cámara; ajustar la exposición para que aparezca un punto brillante rodeado centralmente de negro.
NOTA: La cámara captura la luz reflejada por la interfaz vidrio/agua entre el cobertor y la muestra y el círculo visible de luz está en su diámetro más pequeño cuando el punto focal está en esta interfaz. Por lo tanto, es necesario comenzar por debajo de esta interfaz, aumentar la altura z de la interfaz y luego enfocar un poco más por encima del deslizamiento y en la muestra que se está midiendo.- Comenzando desde una posición Z baja, aumente la altura hasta que el punto brillante alcance su tamaño mínimo, luego eleve la altura aún más hasta 20 μm para enfocar el láser por encima del aceite y cubrir el vidrio y en la muestra.
- Detenga la cámara una vez enfocada.
- Para confirmar que la configuración se ha realizado correctamente, controle el rastro de agua ultrapura tipo I para no ver señales de fluorescencia; repetir la confirmación de pureza para los tampones en los que se preparan las muestras.
- Retire con cuidado el vidrio de la cubierta con pinzas de goma para evitar dañar el objetivo, luego coloque la muestra de interés preparada como se indica arriba en el objetivo. Reponer el aceite de inmersión según sea necesario para mantener un área de contacto uniforme entre el vidrio de la cubierta y el objetivo (especial atención prestada al área de contacto de la muestra en el vidrio de la cubierta y el área focal del objetivo)
- Bingo de concentración
- Para garantizar que se obtengan datos de una sola molécula, las muestras deben estar en una concentración a la que se observen de 1 a 5 ráfagas por segundo en el panel de trazas en vivo de smOTTER (esto minimiza la posibilidad de que más de una molécula esté presente en el volumen de detección a la vez). Tome las medidas cuando se haya determinado la concentración adecuada.
- Para evitar la evaporación durante experimentos largos, prepare una cámara de muestra hermética. Presione los aisladores de silicona (orificio de 8-9 mm de diámetro) hacia abajo en el centro de un vidrio de cubierta (coloreado con una punta de pipeta). Luego, pipetee cuidadosamente una muestra en el centro, evitando cualquier contacto con la silicona. Luego, coloque un segundo vidrio de cubierta en la parte superior y presione para formar un sello.
- Verifique el histograma de estequiometría en vivo frente a la eficiencia FRET (ES) para ver si la muestra se está comportando como se esperaba, es decir, la eficiencia FRET esperada y la estequiometría razonable (~ 0.5).
- Cuando el ejemplo esté listo, introduzca los detalles del experimento en el panel de configuración de guardado. Elija un directorio y un nombre de archivo apropiados haciendo clic en el icono de puntos suspensivos (archivos guardados en formato hdf5 / h5) en el panel de configuración de guardado.
- Ingrese información para el nombre de la muestra, los detalles de la muestra, las etiquetas de donante y aceptor, las longitudes de onda de excitación del búfer, el donante y el aceptor, las longitudes de onda de detección y la potencia del láser, y la afiliación del usuario y el usuario.
- Vuelva a la pestaña de seguimiento en vivo en la cinta de opciones y, en el panel de adquisición, introduzca la duración del experimento (en minutos), seleccione un intervalo de guardado adecuado para mitigar la posible pérdida de datos en caso de error. Seleccione Guardar potencias láser, si es necesario.
- Pulse Inicio para tomar los datos.
NOTA: Para permitir una determinación precisa de FRET, se deberán medir al menos dos muestras que contengan los mismos colorantes donantes y aceptores, pero con eficiencias FRET suficientemente diferentes.
5. Análisis/software 2
- Inicie jupyter notebook con el paquete python FRETBursts (las instrucciones de configuración se pueden encontrar aquí: https://craggslab.github.io/smfBox/anasoftware.html)
- Si no se necesita corrección para eficiencias FRET absolutas (es decir, solo los cambios relativos en FRET son suficientes para la medición), inicie Jupyter notebook FRET Analysis 1.4 Uncorrected.ipynb. Utilice esto para exportar figuras o datos como archivos csv para su posterior análisis o trazado en otro software.
NOTA: Cada cuaderno contiene instrucciones detalladas para guiar al usuario a través de los procedimientos de análisis. Para una discusión más detallada de los procedimientos de análisis, incluidos los algoritmos de búsqueda de ráfagas, la corrección de fondo y todos los parámetros de corrección, consulte 23,25. - Si se necesita una corrección para las eficiencias absolutas de FRET, comience calculando los parámetros de diafonía alfa (fuga de emisión del donante en el canal aceptor) y delta (proporción de excitación directa del aceptor bajo excitación del donante).
- Primero inicie Jupyter notebook Correction Factor Finder Alpha-Delta.ipynb y determine los parámetros alfa y delta.
NOTA: Si estos son inconsistentes entre muestras, el microscopio puede haber desarrollado un problema de alineación entre las mediciones o los espectros de los colorantes difieren entre las muestras. - Si los parámetros alfa y delta son consistentes, inicie Jupyter notebook Correction Factor Finder Gamma-Beta.ipynb para determinar los parámetros gamma y beta (que tienen en cuenta las diferencias en la excitación y las eficiencias de detección entre los colorantes).
NOTA: Si la gráfica gamma y beta no encaja bien, el microscopio puede haber desarrollado un problema de alineación entre las mediciones, o los rendimientos cuánticos, o los coeficientes de extinción de los colorantes difieren entre las muestras. - Con los cuatro parámetros de diafonía determinados, estos factores se pueden utilizar en el cuaderno FRET Analysis 1.4 Corrected.ipynb Jupyter para determinar las eficiencias absolutas de FRET.
- Primero inicie Jupyter notebook Correction Factor Finder Alpha-Delta.ipynb y determine los parámetros alfa y delta.
6. Solución de problemas
- Si todas las señales son bajas o los recuentos por ráfaga son más bajos de lo esperado (esto depende del colorante, pero para ATTO-550 y ATTO-647N en el smfBox los valores típicos están entre 50 y 100 recuentos por ms durante una explosión de una sola molécula), realinee el smfBox.
- Puente entre poblaciones doblemente y individualmente etiquetadas en el histograma ES.
NOTA: Esto puede ser causado por trabajar a una concentración demasiado alta (remediar esto diluyendo la muestra), o por fotoblanqueo, que puede ser causado por el uso de una potencia láser demasiado alta (remediar esto reduciendo la potencia del láser). - Si la velocidad de estallido disminuye a lo largo del experimento (es probable que las moléculas marcadas con fluorescencia se adhieran a la cubierta de vidrio), use una mayor concentración de BSA para rectificar.
- Si la velocidad de ráfaga aumenta a lo largo del experimento (es probable que la muestra se evapore), prepare una cámara de muestra hermética como se describió anteriormente.
- Si la señal se bloquea a cero durante la alineación (es probable que el software se vea abrumado por la señal de los detectores), reduzca la potencia del láser o use una muestra de alineación más diluida.
- Si el enfoque Z muestra anillos concéntricos (puede ocurrir si se han colocado varias cubiertas en el objetivo), verifique si hay varias cubiertas en el objetivo.
- Si la muestra contiene ráfagas brillantes y largas de estequiometrías intermedias (causadas por moléculas agregadas), use detergentes o modifique los protocolos de purificación de la muestra.
NOTA: Obtener un búfer limpio puede ser un problema, ya que algunos componentes del búfer a menudo contendrán cantidades muy pequeñas de contaminantes moderadamente fluorescentes que son suficientes para presentarse como ráfagas de un solo color en el rastro de tiempo. Si hay demasiado de esto en el búfer, entonces puede coincidir con ráfagas de muestra y cambiar la eficiencia FRET o la estequiometría que se está midiendo. BSA en particular a menudo puede ser problemático en este sentido, por lo que es útil exponer una solución BSA de stock a una fuente de luz fuerte para fotoblanquear los contaminantes.
Resultados
El protocolo requiere una evaluación crítica de las condiciones experimentales durante la instalación (consulte el paso 4.8 del protocolo). Los primeros resultados adquiridos que determinan el éxito o el fracaso del experimento se logran en esta etapa. Un resultado positivo sería tener entre cinco y una ráfagas por segundo (ver Figura 2B,C). Un resultado negativo sería tener demasiadas (Figura 2A) o muy pocas ráfagas (Figura 2D) dentro de ese marco de tiempo. Sigue siendo posible en esta etapa rectificar estos errores: una muestra con una concentración demasiado alta simplemente debe diluirse; sin embargo, si la concentración es demasiado baja, puede ser necesario preparar una nueva muestra (el determinante es si sigue siendo posible recopilar datos en este marco de tiempo razonable en esta baja concentración).
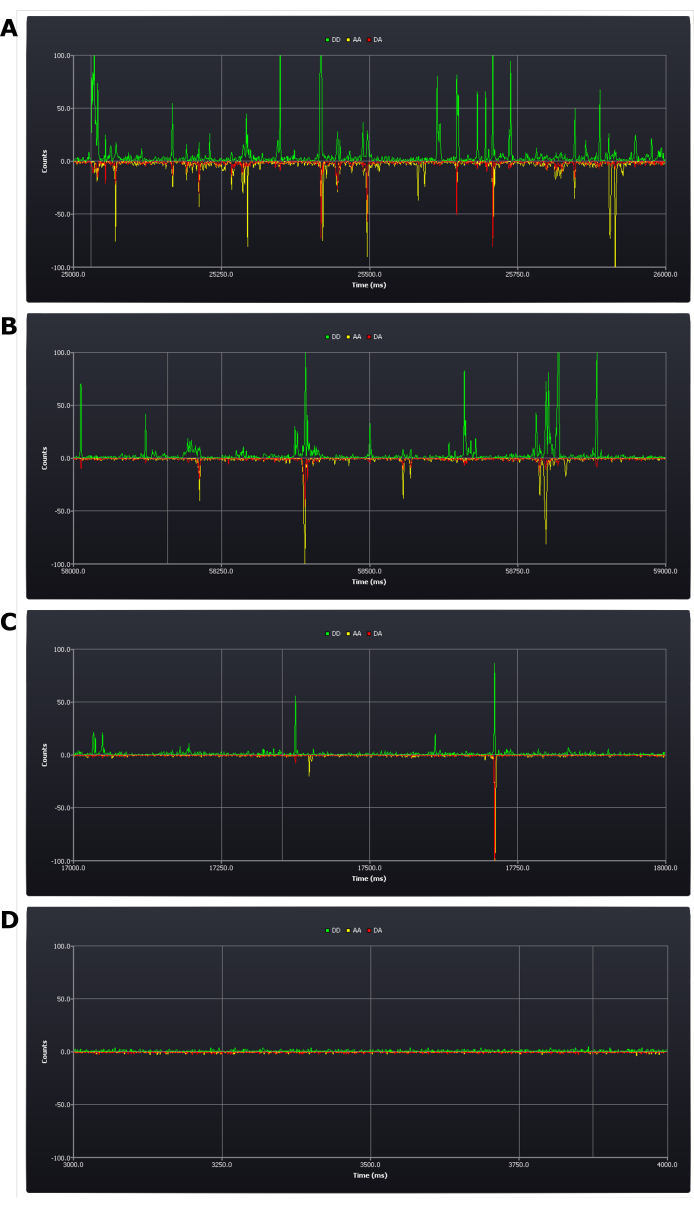
Figura 2: Capturas de pantalla de trazas en vivo durante la configuración experimental que muestran diferentes concentraciones de muestras de ADN dúplex doblemente etiquetadas. (A) demasiado alto, (B) límite superior aceptable, (C) concentración objetivo, (D) demasiado bajo. Los recuentos de fotones (contenedores de 1 ms) se muestran en los tres canales de detección; emisión del donante después de la excitación del donante (DD), emisión del aceptor después de la excitación del donante (DA) y emisión del aceptor después de la excitación del aceptor (AA). Haga clic aquí para ver una versión más grande de esta figura.
Un sistema estático de una sola especie normalmente requeriría de 30 a 60 minutos de medición para obtener las ~ 1,000 ráfagas necesarias para un análisis de datos robusto. La duración del tiempo y el número de ráfagas requeridas aumentarán con múltiples especies o sistemas dinámicos. Después de la recopilación y el análisis de datos utilizando el protocolo, las figuras se exportan desde los cuadernos de Jupyter. La gráfica de alternancia (Figura 3A) debe coincidir con el período ALEX de la configuración experimental. El rastro de tiempo (Figura 3B) se utiliza para evaluar cualitativamente que la concentración de la muestra es razonable. El gráfico de fondo (Figura 3C) muestra la distribución de los períodos de retardo entre fotones con un ajuste lineal a los tiempos más largos para estimar la tasa de fondo26. El rastro de fondo (Figura 3D) puede identificar si hubo cambios en la muestra durante la duración del experimento; principalmente esto se debería a la evaporación durante tiempos de adquisición más largos. Los histogramas ES se generan para todos los fotones (Figura 3E) y especies doblemente etiquetadas (Figura 3F). Finalmente, se genera un histograma 1D E (Figura 3G) con el ajuste gaussiano de los datos de ráfaga.
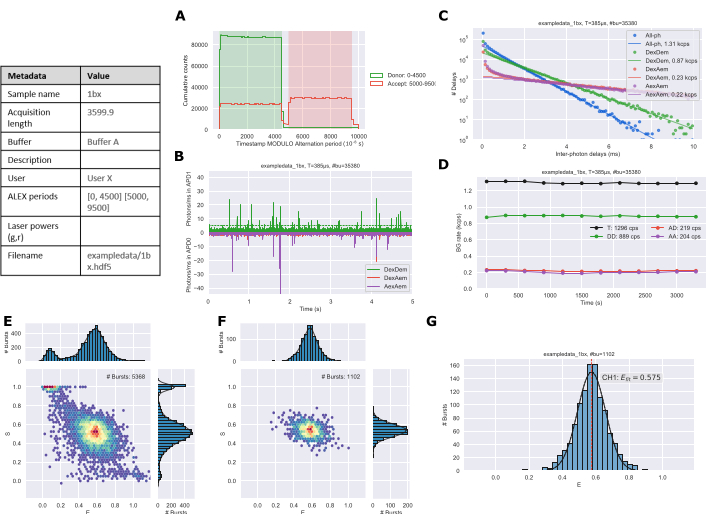
Figura 3: Ejemplo de salida de figuras de datos analizados generados por los Jupyter Notebooks. (A) Diagrama de alternancia, (B) Trazado de tiempo, (C) Determinación de fondo, (D) Tasas de fondo, (E) Histograma ES de todos los fotones, (F) Histograma ES de doble canal y (G) Histograma 1D E. Haga clic aquí para ver una versión más grande de esta figura.
| Nombre | Secuencia |
| 1a | 5'- GAG CTG AAA GTG TCG AGT TTG TTT GAG TGT TTG TCT GG - 3' |
| 3'- CTC GAC TTT CAC AGC TCA AAC AAA CTC ACA AAC AGA CC - 5' | |
| 1b | 5'- GAG CTG AAA GTG TCG AGT TTG TTT GAG TGT TTG TCT GG - 3' |
| 3'- CTC GAC TTT CAC AGC TCA AAC AAA CTC ACA AAC AGA CC - 5' | |
| 1c | 5'- GAG CTG AAA GTG TCG AGT TTG TTT GAG TGT TTG TCT GG - 3' |
| 3'- CTC GAC TTT CAC AGC TCA AAC AAA CTC ACA AAC AGA CC - 5' |
Tabla 1: Secuencias de ADN utilizadas en el protocolo. Los nucleótidos están resaltados en azul y rojo representando los residuos de timina modificados con C2 amino etiquetados con Atto-550 y Atto-647N, respectivamente.
Buscador de factores de corrección Alpha-Delta: Haga clic aquí para descargar este archivo.
Correction Factor Finder Gamma-Beta: Haga clic aquí para descargar este archivo.
Análisis FRET 1.4 Corregido: Haga clic aquí para descargar este archivo.
Análisis FRET 1.4 Sin corregir: Haga clic aquí para descargar este archivo.
Discusión
Los pasos más críticos en el protocolo son la alineación del microscopio y el ajuste de la concentración de la muestra a la dilución correcta. Si la alineación está desactivada, entonces podría haber una señal insuficiente para identificar ráfagas y trazar histogramas, y si se produce una desalineación entre las muestras, la corrección fret precisa puede fallar debido a cambios en las fugas y las eficiencias de detección / excitación. El uso de una concentración apropiada también es importante, una concentración demasiado alta dará ráfagas coincidentes, que contienen múltiples moléculas con eficiencias FRET potencialmente diferentes o estequiometrías de etiquetado. Una concentración demasiado baja dará muy pocas ráfagas para un análisis de datos robusto.
El protocolo descrito aquí es para medir distancias en especies estáticas de FRET individuales. Si la muestra tiene más de un pico en el histograma de eficiencia FRET, o los picos parecen amplios (lo que puede suceder con especies dinámicas), entonces se pueden necesitar más ráfagas para ajustar los histogramas al mismo grado de precisión. Para dos picos bien separados, se necesitará aproximadamente el doble de datos, pero si las poblaciones se superponen ligeramente, se requieren aún más datos.
Si las dos poblaciones se interconvierten en la escala de tiempo del experimento, la dinámica y la cinética del sistema pueden determinarse potencialmente. Pruebas como BVA27 y 2CDE28 pueden confirmar que las ráfagas intermedias son de naturaleza dinámica, mientras que los análisis que incluyen dPDA29,30 o H2MM31 pueden determinar las tasas de interconversión. Los cuadernos Jupyter para BVA y 2CDE están disponibles en el sitio web de FRETBursts26, y el software basado en MATLab PAM32 puede ejecutar análisis BVA, 2CDE y PDA.
El FRET confocal de una sola molécula puede observar fácilmente estados mucho más breves (~1 ms) que TIRF; sin embargo, los tiempos de observación cortos, limitados por la difusión, no dan historia molecular, y por lo tanto no pueden determinar tiempos de permanencia más largos, o redes de transición complejas de la manera en que los experimentos inmovilizados en la superficie pueden.
Como el protocolo mide moléculas que se difunden libremente a una concentración muy baja, funciona mejor cuando se miden distancias intramoleculares en la misma molécula. Las distancias intermoleculares entre moléculas unidas transitoriamente se pueden medir siempre que el Kd de las dos moléculas sea lo suficientemente bajo como para que el complejo exista en una cantidad significativa a la baja concentración de trabajo requerida por el experimento (~ 100 pM). Si el Kd es mucho más alto que esto, entonces solo se verán moléculas etiquetadas individualmente. Este problema puede superarse mediante el uso de microfluídica para mezclar los dos componentes etiquetados a una alta concentración y luego diluir y fluir rápidamente sobre el objetivo antes de que el complejo se disocie33,34.
La medición de las eficiencias de FRET a nivel de molécula única tiene una ventaja significativa sobre las técnicas de conjunto, ya que informa sobre subpoblaciones heterogéneas, que en un experimento de conjunto se promediarían. Además, fret de una sola molécula con ALEX da acceso a eficiencias FRET precisas, que se pueden convertir en distancias precisas. Esto permite la determinación de información estructural más detallada en lugar de simplemente sondear los cambios de distancia relativa. El smfBox tiene todos estos beneficios y capacidades, pero se puede construir con un presupuesto mucho más bajo que los microscopios comparables disponibles comercialmente capaces de smFRET23 confocal.
El smfBox representa una barrera de entrada mucho menor para las técnicas smFRET, lo que permite a los investigadores medir los cambios conformacionales y las distancias precisas dentro y entre las proteínas y los ácidos nucleicos7,8,9,10,11,35.
Divulgaciones
Los autores no declaran intereses contrapuestos.
Agradecimientos
Los autores agradecen las siguientes fuentes de financiación: BBSRC (BB/T008032/1); EPSRC (Studentship to B.A.) y MRC (Studentship to A. R.-T.).
Materiales
| Name | Company | Catalog Number | Comments |
| Amino modified oligonucleotide | Eurogentec | N/A | May be ordered from various suppliers or synthesised; amino modification enables labeling with NHS-ester modified dyes |
| Avalanche photodiode (APD) | Excelitas | SPCM-AQRH-14 | Two APDs are required for the smfBox setup |
| Bovine Serum Albumin (BSA) | Merck | A2153 | System dependant; imaging buffer component (0.1 mg/mL in buffer) |
| Compact Laser Combiner | OMICRON | LightHUB-2 | 515 nm (80 mW) and 638 nm (100 mW) lasers |
| Coverglass | VWR | 630-2742 | Thickness: 0.17 ± 0.01 mm, LxW: 22x22 mm |
| Cy3B | Cytiva | PA63101 | 1 mg, PA63100 (5 mg), PA96106 (25 mg) |
| FRETBursts Python Package | N/A | N/A | Open-source python package for burst analysis of freely-diffusing single-molecule FRET data: https://fretbursts.readthedocs.io |
| Imaging Buffer | N/A | System dependant; 5 mM NaCl, 20 mM MgCl2, 5 mM Tris pH 7.5 and 0.1 mg/mL BSA | |
| Immersion Oil | Olympus | IMMOIL-F30CC | |
| Jupyter notebooks | Project Jupyter | N/A | Open-source web application to create and share documents that contain live code, equations, visualizations and text; data analysis notebooks for smfBox can be found in the SI |
| Lens Tissue | ThorLabs | MC-5 | MC-50E is same item in bulk |
| Magnesium Chloride | Merck | M2670 | System dependant; imaging buffer component (20 mM in buffer) |
| MilliQ/Ultrapure water | N/A | ||
| Nanopoistioner | Piezoconcept | FOC300 | Nanopositioner for accurate positioning of microscope objective |
| NHS-ester modified ATTO-550 | ATTO-TEC | AD 550-31 | 1 mg, AD 550-35 (5 mg) |
| NHS-ester modified ATTO-647N | ATTO-TEC | AD 647N-31 | 1 mg, AD 647N-35 (5 mg) |
| Objective lens | Olympus | N1480700 | Olympus objective series from orignal smfBox discontinued; replaced by N5702300 |
| OMICRON Control Center (OCC)- laser control center | OMICRON | N/A | v3.5.34 - OMICRON laser driver software |
| Press-To-Seal silicone isolator | Grace Bio-Labs | 664201 | 8-9 mm Diameter x 1.7 mm Depth |
| smOTTER | N/A | N/A | Open-source acquisition software for the Craggs Lab smfBox: https://github.com/craggslab/smOTTER |
| Sodium Chloride | Merck | S7653 | System dependant; imaging buffer component (5 mM in buffer) |
| Tris base | Merck | 93362 | System dependant; imaging buffer component (5 mM, pH 7.5 in buffer) |
| Type I ultrapure water | Merck | ZIQ7000T0 | Milli-Q® IQ 7000 Ultrapure Water System |
Referencias
- Forster, T. Intermolecular energy migration and fluorescence. Annals of Physics. 437 (1-2), 55-75 (1948).
- Stryer, L., Haugland, R. P. Energy transfer: a spectroscopic ruler. Proceedings of the National Academy of Sciences. 58 (2), 719-726 (1967).
- Hohlbein, J., et al. Conformational landscapes of DNA polymerase I and mutator derivatives establish fidelity checkpoints for nucleotide insertion. Nature Communications. 4 (1), 2131 (2013).
- Lerner, E., et al. Toward dynamic structural biology: Two decades of single-molecule Förster resonance energy transfer. Science. 359 (6373), (2018).
- Hohlbein, J., Craggs, T. D., Cordes, T. Alternating-laser excitation: single-molecule FRET and beyond. Chemical Society Reviews. 43 (4), 1156-1171 (2014).
- Lerner, E., et al. The FRET-based structural dynamics challenge -- community contributions to consistent and open science practices. arXiv. , (2020).
- Hellenkamp, B., Wortmann, P., Kandzia, F., Zacharias, M., Hugel, T. Multidomain structure and correlated dynamics determined by self-consistent FRET networks. Nature Methods. 14 (2), 174-180 (2017).
- Craggs, T. D., et al. Substrate conformational dynamics facilitate structure-specific recognition of gapped DNA by DNA polymerase. Nucleic Acids Research. 47 (20), 10788-10800 (2019).
- Tsytlonok, M., et al. Dynamic anticipation by Cdk2/Cyclin A-bound p27 mediates signal integration in cell cycle regulation. Nature Communications. 10 (1), 1676 (2019).
- Nagy, J., et al. Complete architecture of the archaeal RNA polymerase open complex from single-molecule FRET and NPS. Nature Communications. 6 (1), 6161 (2015).
- LeBlanc, S. J., et al. Coordinated protein and DNA conformational changes govern mismatch repair initiation by MutS. Nucleic Acids Research. 46 (20), 10782-10795 (2018).
- Segal, M., et al. High-throughput smFRET analysis of freely diffusing nucleic acid molecules and associated proteins. Methods. 169, 21-45 (2019).
- Kapanidis, A. N., et al. Fluorescence-aided molecule sorting: Analysis of structure and interactions by alternating-laser excitation of single molecules. Proceedings of the National Academy of Sciences. 101 (24), 8936-8941 (2004).
- Kapanidis, A. N., et al. Alternating-laser excitation of single molecules. Accounts of Chemical Research. 38 (7), 523-533 (2005).
- Müller, B. K., Zaychikov, E., Brauchle, C., Lamb, D. C. Pulsed interleaved excitation. Biophysical Journal. 89 (5), 3508-3522 (2005).
- Laurence, T. A., Kong, X., Jager, M., Weiss, S. Probing structural heterogeneities and fluctuations of nucleic acids and denatured proteins. Proceedings of the National Academy of Sciences of the United States of America. 102 (48), 17348-17353 (2005).
- Pollina, T., et al. PlanktonScope: Affordable modular imaging platform for citizen oceanography. bioRxiv. , 056978 (2020).
- Collins, J. T., et al. Robotic microscopy for everyone: the OpenFlexure microscope. Biomedical Optics Express. 11 (5), 2447-2460 (2020).
- Courtney, A., Alvey, L. M., Merces, G. O. T., Burke, N., Pickering, M. The Flexiscope: a low cost, flexible, convertible and modular microscope with automated scanning and micromanipulation. Royal Society Open Science. 7 (3), 191949 (2020).
- Martens, K. J. A., et al. Visualisation of dCas9 target search in vivo using an open-microscopy framework. Nature Communications. 10 (1), 3552 (2019).
- Auer, A., et al. Nanometer-scale multiplexed super-resolution imaging with an economic 3D-DNA-PAINT microscope. ChemPhysChem. 19 (22), 3024-3034 (2018).
- Li, H., et al. Squid: Simplifying quantitative imaging platform development and deployment. bioRxiv. , 424613 (2020).
- Ambrose, B., et al. The smfBox is an open-source platform for single-molecule FRET. Nature Communications. 11 (1), 5641 (2020).
- Ingargiola, A., Laurence, T., Boutelle, R., Weiss, S., Michalet, X. Photon-HDF5: An open file format for timestamp-based single-molecule fluorescence experiments. Biophysical Journal. 110 (1), 26-33 (2016).
- Hellenkamp, B., et al. Precision and accuracy of single-molecule FRET measurements-a multi-laboratory benchmark study. Nature Methods. 15 (9), 669-676 (2018).
- Ingargiola, A., Lerner, E., Chung, S., Weiss, S., Michalet, X. FRETBursts: An open source toolkit for analysis of freely-diffusing single-molecule FRET. PLOS One. 11 (8), 0160716 (2016).
- Torella, J. P., Holden, S. J., Santoso, Y., Hohlbein, J., Kapanidis, A. N. Identifying molecular dynamics in single-molecule FRET experiments with burst variance analysis. Biophysical Journal. 100 (6), 1568-1577 (2011).
- Tomov, T. E., et al. Disentangling subpopulations in single-molecule FRET and ALEX experiments with photon distribution analysis. Biophysical Journal. 102 (5), 1163-1173 (2012).
- Santoso, Y., Torella, J. P., Kapanidis, A. N. Characterizing single-molecule FRET dynamics with probability distribution analysis. ChemPhysChem. 11 (10), 2209-2219 (2010).
- Kalinin, S., Valeri, A., Antonik, M., Felekyan, S., Seidel, C. A. M. Detection of structural dynamics by FRET: A photon distribution and fluorescence lifetime analysis of systems with multiple states. The Journal of Physical Chemistry B. 114 (23), 7983-7995 (2010).
- Pirchi, M., et al. Photon-by-photon hidden Markov model analysis for microsecond single-molecule FRET kinetics. The Journal of Physical Chemistry B. 120 (51), 13065-13075 (2016).
- Schrimpf, W., Barth, A., Hendrix, J., Lamb, D. C. PAM: A framework for integrated analysis of imaging, single-molecule, and ensemble fluorescence data. Biophysical Journal. 114 (7), 1518-1528 (2018).
- Zijlstra, N., et al. Rapid microfluidic dilution for single-molecule spectroscopy of low-affinity biomolecular complexes. Angewandte Chemie International Edition. 56 (25), 7126-7129 (2017).
- Hellenkamp, B., Thurn, J., Stadlmeier, M., Hugel, T. Kinetics of transient protein complexes determined via diffusion-independent microfluidic mixing and fluorescence stoichiometry. The Journal of Physical Chemistry B. 122 (49), 11554-11560 (2018).
- Bennet, I. A., et al. Regional conformational flexibility couples substrate specificity and scissile phosphate diester selectivity in human flap endonuclease 1. Nucleic Acids Research. 46 (11), 5618-5633 (2018).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados