Method Article
オープンソースsmfBoxを使用して、正確で正確な単一分子フレット測定を行う
要約
この記事では、オープンソースの安価なsmfBoxを使用して、スイッチオンからアライメント、フォーカス、データ収集と分析まで、個々の生体分子を自由に拡散して、個々の正確なFRET測定を行うためのステップバイステップの手順を説明します。
要約
smfBoxは、単分子フェルスター共鳴エネルギー伝達(smFRET)用の最近開発された費用対効果の高いオープンソース機器で、自由に拡散する生体分子の測定をよりアクセスしやすくします。この概要には、サンプル調製、装置のセットアップと位置合わせ、データ収集、および完全な分析ルーチンの詳細を含む、二重DNAサンプルにおける正確なFRET効率の測定を行うためのステップバイステップのプロトコルが含まれています。提示されたアプローチは、正確なFRET導出距離測定に必要なすべての補正因子を決定する方法を含み、標準的なプロトコルと分析アプローチを確立することを目的としたFRETコミュニティ全体の最近の共同作業の大きな体に基づいています。このプロトコルは、様々な生体分子システムに容易に適応可能であり、より広範な科学界のためのsmFRETの民主化における取り組みの増大を増大させる。
概要
単一分子フェルスター共鳴エネルギー移動(smFRET)は、ドナーと個々の分子のレベルでの2つの色素とアクセプタの間のFRET効率を測定する技術です。FRETは、2つの色素の重なり合うエネルギースペクトルから生じる光物理学的プロセスである:ドナーは特定の波長の光によって励起され、アクセクサに非放射エネルギーを伝達し、アクセクサからの放出をもたらす。この転写の効率は、2つの色素間の距離の第6乗数に反比例するため、転送効率は距離1によって変化する。このように、このFRET効率は、3〜10nmの範囲内で、色素が結合している分子(s)2に関する空間情報を決定するために使用することができる。このスケールは、FRET効率の変化がAngstrom分子運動3に敏感であるという事実と、アンサンブル平均4,5,6の合併症なしに核酸やタンパク質などの生体分子に関する構造情報を調査するのに適しています。相対的なFRET効率の変化は、生体分子相互作用と立体構造ダイナミクスを監視するために使用することができますが、タンパク質(un)フォールディング、転写、DNA複製および修復などの主要な細胞プロセスに光を当て、絶対的なFRET効率は生体分子構造決定のための正確な距離を決定するために使用されています7,8,9,10,11 、結晶化または凍結の必要性を克服する、いくつかの他の構造方法のために必要とされるように4,12。
smFRET実験は、最も一般的に2つの形態、共焦点または全内部反射蛍光(TIRF)顕微鏡を取る。両方のアプローチの間に生体分子の分子動力学は、通常、ピコからミリ秒(共焦点、自由に拡散分子)までのタイムスケールで、ミリ秒から時間(TIRF、表面固定化分子)で調べることができます。これは、各手法に関連するさまざまなセットアップが原因です。TIRF顕微鏡では、分子はスライドの表面に固定化され、エバネッセント波によって励起されます(図1A)。ただし、ここでは、smfBox の形式であるため、共焦点顕微鏡に焦点を当てています。共焦点顕微鏡では、分子は固定化されず、代わりにブラウン運動を介して共焦点体積(〜1fL)を介して自由に拡散し、高い数値開口レンズを通してレーザー光を溶液内の特定の深さのスポットに集積することによって形成される(図1B)。結果として得られる放出は、同じ絞りを通して焦点を合わせ、二色性ミラーを通してフィルタリングされます(図1C は完全な回路図です)。その後、ピンホールを通して焦点を当て、焦点外の光を取り除き、雪崩フォトダイオード(APD)に焦点を当てます。APDがフォトンを検出すると、TTLパルスを出力し、そのタイミングをピコ秒の分解能まで記録できます。これらの分子の観察時間は、共焦点体積付近の近傍で一般的にミリ秒単位で観察される。
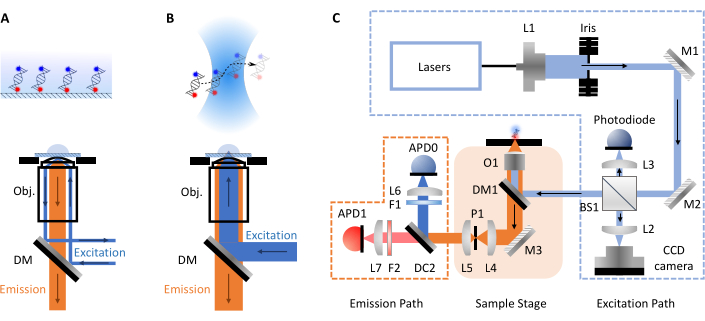
図1:顕微鏡とsmfBoxの設定の原理を示す回路図 (A)全内部反射蛍光(TIRF)顕微鏡原理:励起光は目的の端(Obj.)に向けられ、表面に付着した分子を励起する指数関数的な崩壊エバンジェンスフィールドを生成するカバースリップバッファ界で全内部反射を受ける。(B)共焦点顕微鏡:自由に拡散分子は、サンプルに焦点を当てた近い回折限のスポットによって励起されます。(C) このプロトコルで使用される smfBox セットアップは、すべての主要なコンポーネントを示しています: アバランシェフォトダイオード (APD),ビームスプリッター(BS),二色性鏡(DM)、フィルタ(F)、ミラー(M)、目的(O)およびピンホール(P)。 この図の大きなバージョンを表示するには、ここをクリックしてください。
さらに最近では、smFRET技術は、ドナーとアクセクター励起波長に一致するレーザーが交互に行われ、2つの色励起を組み込んだ。これは、2つの方法のいずれかで行うことができます, 最初のKHzタイムスケールで連続波レーザーを調節することによって, これは、交互レーザー励起(ALEX)13,14として知られています.2番目の方法は、MHzタイムスケールで高速パルスをインターリーブします。これはナノ秒-ALEX15またはパルスインターリーブ励起(PIE)16です。これらすべてのアプローチにおいて、アクセプターレーザーからの情報は、FRET効率の低い分子とアクセプターを欠いている分子(不完全な標識またはフォトブラチリングを通じて)を区別することができる、いわゆるストイチオメトリーの計算につながります。PIE/ns-ALEXを使用すると、単一分子レベルで蛍光寿命にアクセスでき、偏光光学と組み合わせたときに異方性を測定することができます。この測定の組み合わせは、多パラメータ蛍光検出(MFD)9として知られています。
smFRETの多くの利点にもかかわらず、それは商業用具の高いコストと簡単な、自己構築の選択肢の欠如のために専門の実験室の外で広く使用されていません。低コストのオープンソース顕微鏡の開発傾向が高まっており、プランクトンスコープ17、OpenFlexure顕微鏡18、Flexiscope19、miCube20、liteTIRF21、Squid22など、最近、他のプラットフォームが登場しています。本研究では、自由に拡散する単一分子上の2つの色素間のFRET効率を測定することができる最近開発された費用対効果の高い共焦点セットアップであるsmfBoxを使用するためのプロトコルについて説明する。詳細なビルド手順と必要なすべての運用ソフトウェアは、https://craggslab.github.io/smfBox/ 23で自由に入手できます。smfBoxの光学配置は、手頃な価格で広くアクセス可能なメーカーから購入した容易に入手可能なコンポーネントから組み立てられ、顕微鏡本体(標準的な共焦点セットアップの費用の大半を担当)は、カスタムライトタイト陽極酸化アルミニウムボックスに置き換えられました(周囲の光条件下で測定することができます)。このボックスには、励起二色性、目的、ピンホール、および機械的レーザーインターロックを含む主要な光学部品が収容され、クラスIレーザー製品としての安全な動作を可能にします(完全な概略図については図1Cを参照)。smfBoxは、ALEXを使用して色素の測定を検証し、正確なFRET補正因子を決定します。カスタムで書かれたオープンソースソフトウェア(smOTTER)を使用して操作され、データ収集のあらゆる側面を制御し、オープンソースのPhoton-HDF5 format24にデータを出力し、多くのサードパーティ製の分析ツールと互換性があります。smfBoxと取得およびデータ分析プロトコルは、最近、マルチラボブラインドスタディ25で>20の他の機器(共焦点とTIRFの両方)に対してテストされました。得られたFRET効率は、市販のセットアップの価格のほんの一部しかコストがかかるsmfBoxにもかかわらず、他のすべての機器と優れた一致していました。
ここでは、smfBoxを使用してDNA二重を自由に拡散する正確で絶対的なFRET効率を取得し、分析するためのステップバイステッププロトコルを概説し、スイッチオンからアライメントとフォーカス、データ収集および分析までを行います。ここで使用されるサンプルは、世界規模のブラインドスタディ25で評価された3つの二重DNA(高、中、低のFRET効率を示す表1を参照)です。しかし、この方法は、タンパク質および他の核酸を含む多くの分子系に適応可能である。smfBox23 の既存のビルド手順と共に、このような詳細なプロトコルが、この強力な技術を幅広いラボで利用しやすくすることを期待しています。
プロトコル
1. 電源オンコンポーネント
- 6つのプラグ(特に順序なし):ピエゾコンセプト(zフォーカス)、APD0、APD1、グリーンレーザー、赤レーザー、光検出器。
2. ソフトウェア1-実験セットアップ
- レーザーコントロールセンターを起動します。
- 連続波 - 変電電流(CW-ACC)モードが選択されていることを確認します。
- 両方のレーザーの電源を入れ
- レーザーパワーを確認/設定します。
注: 励起経路の ND フィルターとビームスプリッターはレーザー コントロール パネルで指定された数からレーザーパワーを減らすため、レーザーパワーは励起二色音の直前に測定されるように調整する必要があります。ここで示す数字は励起二色性でのパワーですが、これに対応するレーザー制御の電源を取り出す必要があります。- 220 μW グリーンレーザー(515 nm、40 mW)
- μ70 W 赤色レーザー用(638 nm、10 mW)
- smOTTER取得ソフトウェア
- レーザー、検出器、Zステージ、カメラを接続します。正しく設定します(NIカードの設定によって異なる場合があります)。
3. 排出経路のアライメント(日常的に必要とされない)
- 配置の設定
- ピペット10 μLの遊離Cy3B色素(約100 nM)を顕微鏡に、下記のステップ4.3-4.5に記載されているように焦点を合わせます。
注:アクセクサチャネルへの漏れのある別のドナー染料も機能します。 - smOTTER の [位置合わせ ] タブを開き、レーザーパワーを下げ、検出器から読み出しが見えるまで y 軸スケールを変更します。
注:ここでの目標は、信号を増やすために、信号が増加した後にスケールを再び変更する必要があります。 - smfBox の前面にある 4 本のネジを外し、前面パネルを取り外します。
- ピペット10 μLの遊離Cy3B色素(約100 nM)を顕微鏡に、下記のステップ4.3-4.5に記載されているように焦点を合わせます。
- ピンホールの位置合わせ
- 「アライメント」タブで信号を見ながらピンホールポジショナーのxノブを回し、信号を緑色と赤で増やします。
- 今度はyノブを回して、ピンホールをもう一方の方向に合わせます。
- xノブに戻り、信号のさらなる増加を確認します。
- ピンホールレンズのアライメント
- レンズxノブを一方向に回すと、信号が減少します。
- ピンホールのxノブを同じ方向に回して、信号をもう一度増やします。
- 新しい最大信号が以前より高い場合は、ピンホールとレンズの両方をその方向に繰り越し移動します。以前よりも低い場合は、逆方向に繰り方向に移動します。
- 上記の y を繰り返します。
- APDレンズアライメント
- 緑色の APD から始めて、緑色の信号が最大になるまで x ノブを動かします。
- yノブについて繰り返します。
- xノブに戻り、前後に移動して信号が落ち始めるしきい値ポイントを見つけ、この2つのポイントの中間の位置に置きます。
- xノブに対して繰り返します。
- 赤い APD レンズに対して上記の手順を繰り返し、赤信号を見ます。
- フロントパネルをsmfBoxに戻し、ネジを交換します。
注:上記のアライメントmonthylを実行し、顕微鏡が測定間のアライメント欠陥を開発したと疑われる場合は予防措置として行ってください。
4. 測定データ取得
- 最初のサンプルでは、メタノールに浸したレンズ洗浄組織でステージを清掃します。一方の端から端まで、目的を横切ってそっと拭きます。
- 目的の中心に顕微鏡のための浸漬油の3〜4滴を適用します。サンプル間で必要に応じて補充します。
- サンプル準備
- ピペット10 μLのサンプル(最初の使用タイプI超純水)を、クリーンカバーガラスの中心に配置。
- カバーガラスと目的の間に気泡がトラップされないように、油に対して角度で下げる目的レンズの上にカバーガラスを慎重に置きます。
注:必要に応じて、スライドカバーガラスは、焦点から離れて油の気泡を押し出すために周り。
- レーザー励起(ALEX)実験を行うために、レーザーデューティサイクルパネルでレーザーを次のように構成する。
- ドナー(グリーンレーザー)0オフ、45オン、55オフ
- アクサイン(赤レーザー)50オフ、45オン、5オフ
- アレックス期間(μs):100
- リボンの [Z] フォーカス タブをクリックします。取得パネルで、レーザーを Live に切り替えてカメラを起動します。明るいスポットが中央に黒で囲まれているように露出を調整します。
注: カメラは、カバースリップとサンプルの間のガラス/水界面で反射された光をキャプチャし、焦点がこのインターフェイスにあるときに可視光線の円が最小の直径になります。したがって、このインターフェイスの下から始め、インターフェイスに対して Z 高さを大きくし、さらにカバースリップの上と測定されるサンプルに焦点を合わせる必要があります。- 低いZ位置から始めて、明るいスポットが最小サイズになるまで高さを増やし、高さを最大20μm上げて、レーザーを油とカバーガラスの上およびサンプルに集中させます。
- カメラを一度停止します。
- セットアップが適切に行われたことを確認するには、I型超純水の痕跡を監視して蛍光信号を見ない。サンプルが調製されるバッファーの純度の確認を繰り返します。
- ゴム製ピンセットでカバーガラスを慎重に取り外して、損傷を防ぐため、目的に上のように準備した目的のサンプルを置きます。必要に応じて浸漬油を補充し、カバーガラスと目的との間の接触領域を均一に保つ(カバーガラスと目的の焦点領域上のサンプルの接触領域に特別な注意を払う)
- 濃度ビンゴ
- 1分子データを確実に得るためには、smOTTERのライブトレースパネルで毎秒1〜5バーストが観察される濃度でサンプルが必要です(これは、一度に複数の分子が検出体積に存在する可能性を最小限に抑えます)。適切な濃度が決定された時点で測定を行います。
- 長時間の実験で蒸発を防ぐには、気密性の高いサンプルチャンバーを用意してください。カバーガラスの中央にシリコーンアイソレータ(直径8~9mmの穴)を押し下げます(ピペットチップで着色)。次いで、試料を中心に慎重にピペットし、シリコーンとの接触を避ける。その後、2番目のカバーグラスを上に置き、押してシールを形成します。
- ライブストイヒオメトリー対FRET効率(ES)ヒストグラムを調べて、サンプルが期待どおりに動作しているかどうか、すなわち、期待されるFRET効率と合理的なストイチオメトリー(〜0.5)を確認してください。
- サンプルの準備ができたら、テストの詳細を [保存設定] パネルに入力します。保存設定パネルで省略記号アイコン(hdf5/h5形式で保存されたファイル)をクリックして、適切なディレクトリとファイル名を選択します。
- サンプル名、サンプルの詳細、ドナーおよびアクセクサラベル、バッファー、ドナーおよびアクセクサ励起波長、検出波長とレーザーパワー、およびユーザーとユーザーの所属に関する情報を入力します。
- リボンのライブ トレース タブに戻り、取得パネルで実験の長さ (分単位) を入力し、適切な保存間隔を選択して、エラーが発生した場合のデータ損失の可能性を軽減します。必要に応じて 、[レーザーパワーを保存]を選択します。
- [開始] を押してデータを取得します。
注: 正確な FRET の決定を可能にするには、同じドナーとアクセクサの色素を含む少なくとも 2 つのサンプルを測定する必要がありますが、FRET の効率は十分に異なります。
5. 分析/ソフトウェア 2
- FRETBursts Pythonパッケージを使用してJupyterノートブックを起動します(セットアップ手順はこちらから見つけることができます:https://craggslab.github.io/smfBox/anasoftware.html)
- 絶対FRET効率の補正が必要でない場合(つまり、測定にはFRETの相対的な変化だけで十分です)、Jupyterノートブック FRET分析1.4 Uncorrected.ipynbを起動します。他のソフトウェアでの分析やプロットを行うために、図やデータをCSVファイルとしてエクスポートするために使用します。
注: 各ノートブックには、ユーザーが分析手順を案内するための詳細な手順が含まれています。バースト検索アルゴリズム、バックグラウンド補正、およびすべての補正パラメータを含む分析手順のより詳細な説明については、23,25を参照してください。 - 絶対FRET効率の補正が必要な場合は、クロストークパラメータα(アクソクサチャネルへのドナー放出の漏洩)とデルタ(ドナー励起下のアクソクサの直接励起の割合)を計算することから始めます。
- 最初にJupyterノートブック 補正ファクターファインダーアルファデルタ.ipynb を起動し、アルファとデルタのパラメータを決定します。
注: これらがサンプル間で矛盾している場合、測定間の位置合わせの問題が発生した場合や、サンプル間で色素のスペクトルが異なる場合があります。 - アルファとデルタのパラメータが一貫している場合は、Jupyter ノートブック 補正因子ファインダー Gamma-Beta.ipynb を起動して、ガンマおよびベータパラメータ(染料間の励起と検出効率の違いを考慮します)を決定します。
注:ガンマプロットとベータプロットがうまく収まらない場合、顕微鏡は測定間の位置合わせの問題、量子収率、または色素の消光係数がサンプルによって異なる可能性があります。 - 4 つのクロストーク パラメータが決定されると、これらの因子は 、FRET 分析 1.4 補正.ipynb Jupyter ノートブックで使用され、絶対 FRET 効率を決定できます。
- 最初にJupyterノートブック 補正ファクターファインダーアルファデルタ.ipynb を起動し、アルファとデルタのパラメータを決定します。
6. トラブルシューティング
- すべてのシグナルが低いか、またはバーストあたりのカウントが予想よりも低い場合(これは仕分け元ですが、smfBoxのATTO-550およびATTO-647Nの場合は、一分子バースト中に1msあたり50〜100カウントです)、smfBoxを再調整します。
- ESヒストグラム上の二重と1つのラベルの集団の間の橋渡し。
注:これは、あまりにも高い濃度で作業(サンプルを希釈することによってこれを改善する)、またはあまりにも高いレーザーパワーを使用することによって引き起こされる可能性のあるフォトブリーチ(レーザーパワーを減らすことによってこれを改善する)によって引き起こされる可能性があります。 - 破裂率が実験全体を通して低下する場合(蛍光標識された分子がガラスカバースリップに付着している可能性が高い)、BSAの濃度を増加させて修正します。
- 実験中にバースト速度が上昇する場合(サンプルが蒸発する可能性が高い)、前述のように気密性の高いサンプルチャンバを用意する。
- アライメント中に信号がゼロにクラッシュする(ソフトウェアが検出器からの信号に圧倒される可能性がある)場合は、レーザーパワーを下げるか、より希薄なアライメントサンプルを使用します。
- Z-focus が同心円状のリングを示している場合(複数のカバーリップが目的に配置されている場合に起こり得る)、目的の複数のカバーリップをチェックします。
- サンプルに中間ストイチオメトリー(凝集分子によって引き起こされる)の明るく長いバーストが含まれている場合は、洗剤を使用するか、サンプル精製プロトコルを変更します。
注:一部のバッファーコンポーネントは、時間トレース上の単一の色のバーストとして表示するのに十分な非常に少量の適度な蛍光汚染物質を含むことが多いため、クリーンバッファを取得することは問題になる可能性があります。バッファーにこの多くが存在する場合、サンプルバーストと一致し、測定されるFRET効率またはストイチオメトリーを変更することができます。特にBSAはこの点で問題となることが多いため、ストックBSA溶液を強い光源に露出させて汚染物質をフォトブリュチにすることは有用です。
結果
このプロトコルは、セットアップ中の実験条件の重要な評価を必要とします(プロトコルステップ4.8を参照)。この段階で、実験の成功または失敗を決定する最初の結果が得られる。肯定的な結果は、1 秒あたり 5 ~ 1 回のバーストを持つ場合です (図 2B,C を参照)。負の結果として、その時間枠内に多すぎる(図2A)、またはバースト数が少なすぎる(図2D)が発生します。この段階では、これらの誤差を修正することは可能なままです:濃度が高すぎるサンプルは単に希釈する必要があります。しかし、濃度が低すぎる場合は、新しいサンプルを準備する必要があります(決定要因は、この低濃度で合理的な時間枠でデータを収集することが可能かどうかである)。
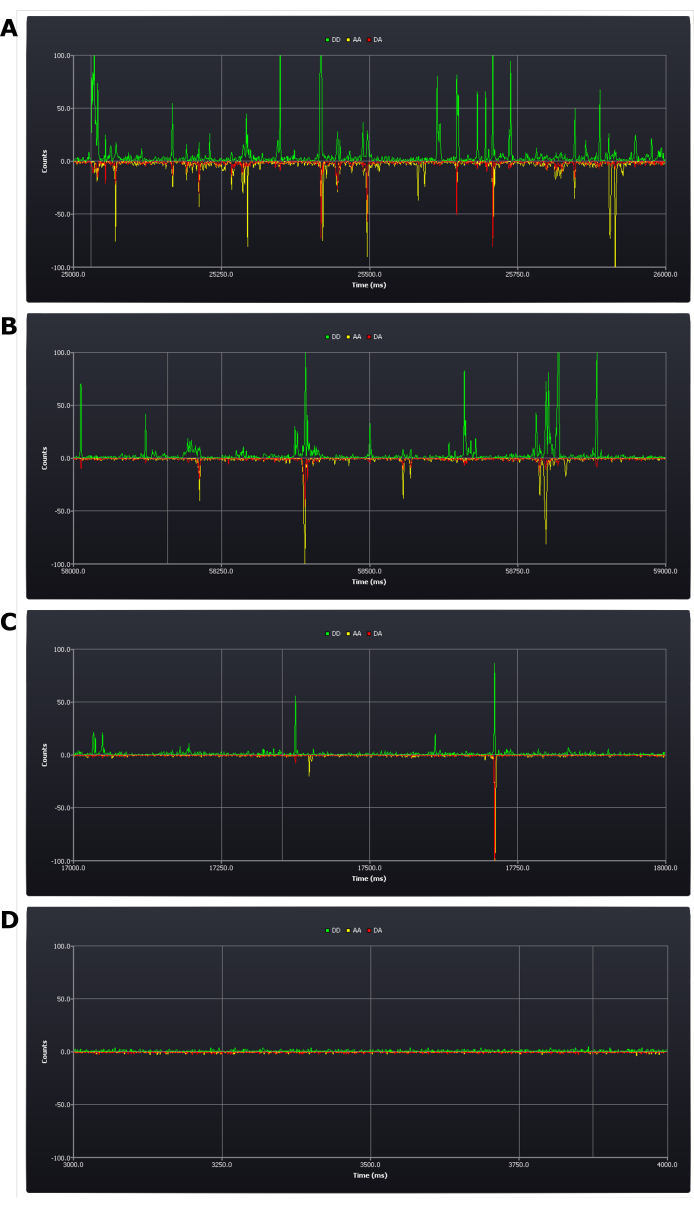
図2:実験中のライブトレースからのスクリーンショットは、二重標識された二重DNAサンプルの異なる濃度を示す。 (A)が高すぎる、(B)上限許容限界、(C)標的濃度、(D)が低すぎる。フォトン数(1 msビン)は3つの検出チャネルに表示されます。ドナー励起後のドナー発光(DD)、ドナー励起後のアクソクサ放出(DA)、およびアクソクサ励起後のアクソクサ放出(AA)。。 この図の大きなバージョンを表示するには、ここをクリックしてください。
静的な単一種システムは、通常、堅牢なデータ分析に必要な約1,000バーストを得るために30〜60分の測定を必要とする。必要なバーストの時間と数の長さは、複数の種や動的システムで増加します。プロトコルの数字を使用したデータ収集と分析に続いてJupyterノートブックからエクスポートします。交互プロット(図3A)は、実験的セットアップのALEX期間と一致する必要があります。時間トレース(図3B)は、サンプル濃度が妥当であることを定性的に評価するために使用される。背景プロット(図3C)は、背景レート26を推定する長い時間に線形フィットしたフォトン間遅延期間の分布を示しています。バックグラウンド トレース (図 3D) は、実験中にサンプルに変化があったかどうかを識別できます。これは主に、取得時間が長い間の蒸発によるものです。ESヒストグラムは、すべてのフォトン(図3E)および二重標識された種(図3F)に対して生成されます。最後に、1D E ヒストグラム(図 3G)は、バースト データのガウスフィッティングで生成されます。
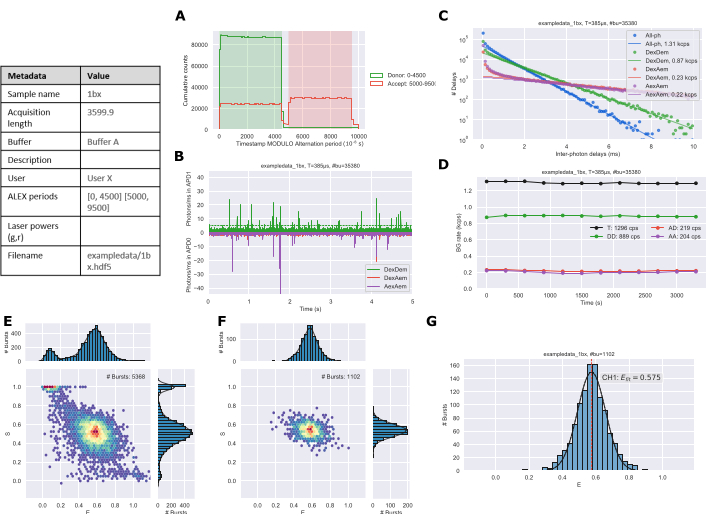
図3:Jupyter Notebooksで生成された分析データの例図出力(A)変動プロット、(B)時間トレース、(C)バックグラウンド判定、(D)バックグラウンドレート、(E)すべてのフォトンESヒストグラム、(F)デュアルチャンネルESヒストグラム、および(G)1D Eヒストグラム。この図の大きなバージョンを表示するには、ここをクリックしてください。
| 名前 | 順序 |
| 1a | 5'- ギャグ CTG AAA GTG TCG AGT TTG TTT ギャグ TGT TTG TCT GG - 3' |
| 3'- CTC GAC TTT CAC AGC TCA AAC AAAA CTC ACA AAC AGA CC - 5' | |
| 1b | 5'- ギャグ CTG AAA GTG TCG AGT TTG TTT ギャグ TGT TTG TCT GG - 3' |
| 3'- CTC GAC TTT CAC AGC TCA AAC AAAA CTC ACA AAC AGA CC - 5' | |
| 1c | 5'- ギャグ CTG AAA GTG TCG AGT TTG TTT ギャグ TGT TTG TCT GG - 3' |
| 3'- CTC GAC TTT CAC AGC TCA AAC AAAA CTC ACA AAC AGA CC - 5' |
表1:プロトコルで用いられるDNA配列。ヌクレオチドは、それぞれAtto-550およびAtto-647Nで標識されたC2アミノ修飾チミン残基を表す青色および赤色で強調される。
補正ファクターファインダーアルファデルタ:このファイルをダウンロードするには、ここをクリックしてください。
補正ファクターファインダーガンマベータ: このファイルをダウンロードするには、ここをクリックしてください。
FRET分析1.4修正: このファイルをダウンロードするには、ここをクリックしてください。
FRET分析 1.4 未修正: このファイルをダウンロードするには、ここをクリックしてください。
ディスカッション
プロトコルの最も重要なステップは、顕微鏡のアライメントと正しい希釈にサンプル濃度を調整することです。アライメントがオフの場合、バーストを識別してヒストグラムをプロットするための信号が不十分である可能性があり、サンプル間でずれが生じた場合、リークおよび検出/励起効率の変化により、正確なFRET補正が失敗する可能性があります。適切な濃度の使用も重要であり、濃度が高すぎると、潜在的に異なるFRET効率または標識ストイチオメトリーを有する複数の分子を含む一致バーストを与える。濃度が低すぎると、堅牢なデータ分析のためにバーストが少なすぎます。
ここで説明するプロトコルは、静的な単一のFRET種の距離を測定するためのものです。サンプルが FRET 効率ヒストグラムに複数のピークを持つ場合、または(動的種で発生する可能性がある)ピークが広く見える場合は、ヒストグラムを同じ精度に適合させるために、より多くのバーストが必要になる場合があります。2 つの十分に分離されたピークでは、約 2 倍のデータが必要になりますが、母集団がわずかに重なっている場合は、さらに多くのデータが必要になります。
2つの集団が実験の時間スケールで相互変換する場合、システムのダイナミクスと運動学を潜在的に決定することができます。BVA27や2CDE28などのテストでは、中間バーストが本質的に動的であることを確認できますが、dPDA29,30またはH2MM31を含む分析では相互変換速度を決定できます。BVAおよび2CDE用のジュピターノートブックはFRETBursts26ウェブサイトで入手でき、MATLabベースのソフトウェアPAM32はBVA、2CDE、およびPDA分析を実行できます。
共焦点単一分子FRETは、TIRFよりもはるかに短く(約1ミリ秒)の状態を容易に観察することができます。しかし、拡散によって制限される短い観察時間は、分子の歴史を与えないので、より長い住まい時間、または表面固定化実験が可能な方法で複雑な遷移ネットワークを決定することができない。
プロトコルは、非常に低い濃度で分子を自由に拡散測定するので、同じ分子上の分子内距離を測定する場合に最適です。一過性結合分子間の分子間距離は、2つの分子のKdが十分に低く、実験で必要とされる低い作業濃度(〜100pM)でかなりの量で複合体が存在することを測定することができる。Kdがこれよりはるかに高い場合は、単一標識された分子のみが見られます。この問題は、マイクロ流体を使用して2つの標識された成分を高濃度で混合し、複雑な解離液33,34の前に目的を迅速に希釈して流すことで克服できます。
単一分子レベルでのFRET効率を測定することは、アンサンブル実験で平均化される不均一な亜集団に関して知らせるので、アンサンブル技術よりも大きな利点がある。さらに、ALEXを使用した単一分子FRETは、正確なFRET効率にアクセスでき、正確な距離に変換できます。これにより、単に相対的な距離の変化を調査するのではなく、より詳細な構造情報を決定できます。smfBoxはこれらすべての利点および機能を運ぶが、共焦点smFRET23が可能な同等の市販の顕微鏡よりもはるかに低い予算で組み立てることができる。
smfBoxはsmFRET技術の参入障壁がはるかに低く、研究者は立体構造の変化とタンパク質と核酸間の正確な距離を測定することを可能にします。
開示事項
著者らは競合する利益を宣言しない。
謝辞
著者らは感謝して、次の資金調達ソースを認める: BBSRC (BB/T008032/1);EPSRC(B.A.への学生シップ)とMRC(A.R.-T.への学生シップ)。
資料
| Name | Company | Catalog Number | Comments |
| Amino modified oligonucleotide | Eurogentec | N/A | May be ordered from various suppliers or synthesised; amino modification enables labeling with NHS-ester modified dyes |
| Avalanche photodiode (APD) | Excelitas | SPCM-AQRH-14 | Two APDs are required for the smfBox setup |
| Bovine Serum Albumin (BSA) | Merck | A2153 | System dependant; imaging buffer component (0.1 mg/mL in buffer) |
| Compact Laser Combiner | OMICRON | LightHUB-2 | 515 nm (80 mW) and 638 nm (100 mW) lasers |
| Coverglass | VWR | 630-2742 | Thickness: 0.17 ± 0.01 mm, LxW: 22x22 mm |
| Cy3B | Cytiva | PA63101 | 1 mg, PA63100 (5 mg), PA96106 (25 mg) |
| FRETBursts Python Package | N/A | N/A | Open-source python package for burst analysis of freely-diffusing single-molecule FRET data: https://fretbursts.readthedocs.io |
| Imaging Buffer | N/A | System dependant; 5 mM NaCl, 20 mM MgCl2, 5 mM Tris pH 7.5 and 0.1 mg/mL BSA | |
| Immersion Oil | Olympus | IMMOIL-F30CC | |
| Jupyter notebooks | Project Jupyter | N/A | Open-source web application to create and share documents that contain live code, equations, visualizations and text; data analysis notebooks for smfBox can be found in the SI |
| Lens Tissue | ThorLabs | MC-5 | MC-50E is same item in bulk |
| Magnesium Chloride | Merck | M2670 | System dependant; imaging buffer component (20 mM in buffer) |
| MilliQ/Ultrapure water | N/A | ||
| Nanopoistioner | Piezoconcept | FOC300 | Nanopositioner for accurate positioning of microscope objective |
| NHS-ester modified ATTO-550 | ATTO-TEC | AD 550-31 | 1 mg, AD 550-35 (5 mg) |
| NHS-ester modified ATTO-647N | ATTO-TEC | AD 647N-31 | 1 mg, AD 647N-35 (5 mg) |
| Objective lens | Olympus | N1480700 | Olympus objective series from orignal smfBox discontinued; replaced by N5702300 |
| OMICRON Control Center (OCC)- laser control center | OMICRON | N/A | v3.5.34 - OMICRON laser driver software |
| Press-To-Seal silicone isolator | Grace Bio-Labs | 664201 | 8-9 mm Diameter x 1.7 mm Depth |
| smOTTER | N/A | N/A | Open-source acquisition software for the Craggs Lab smfBox: https://github.com/craggslab/smOTTER |
| Sodium Chloride | Merck | S7653 | System dependant; imaging buffer component (5 mM in buffer) |
| Tris base | Merck | 93362 | System dependant; imaging buffer component (5 mM, pH 7.5 in buffer) |
| Type I ultrapure water | Merck | ZIQ7000T0 | Milli-Q® IQ 7000 Ultrapure Water System |
参考文献
- Forster, T. Intermolecular energy migration and fluorescence. Annals of Physics. 437 (1-2), 55-75 (1948).
- Stryer, L., Haugland, R. P. Energy transfer: a spectroscopic ruler. Proceedings of the National Academy of Sciences. 58 (2), 719-726 (1967).
- Hohlbein, J., et al. Conformational landscapes of DNA polymerase I and mutator derivatives establish fidelity checkpoints for nucleotide insertion. Nature Communications. 4 (1), 2131(2013).
- Lerner, E., et al. Toward dynamic structural biology: Two decades of single-molecule Förster resonance energy transfer. Science. 359 (6373), (2018).
- Hohlbein, J., Craggs, T. D., Cordes, T. Alternating-laser excitation: single-molecule FRET and beyond. Chemical Society Reviews. 43 (4), 1156-1171 (2014).
- Lerner, E., et al. The FRET-based structural dynamics challenge -- community contributions to consistent and open science practices. arXiv. , (2020).
- Hellenkamp, B., Wortmann, P., Kandzia, F., Zacharias, M., Hugel, T. Multidomain structure and correlated dynamics determined by self-consistent FRET networks. Nature Methods. 14 (2), 174-180 (2017).
- Craggs, T. D., et al. Substrate conformational dynamics facilitate structure-specific recognition of gapped DNA by DNA polymerase. Nucleic Acids Research. 47 (20), 10788-10800 (2019).
- Tsytlonok, M., et al. Dynamic anticipation by Cdk2/Cyclin A-bound p27 mediates signal integration in cell cycle regulation. Nature Communications. 10 (1), 1676(2019).
- Nagy, J., et al. Complete architecture of the archaeal RNA polymerase open complex from single-molecule FRET and NPS. Nature Communications. 6 (1), 6161(2015).
- LeBlanc, S. J., et al. Coordinated protein and DNA conformational changes govern mismatch repair initiation by MutS. Nucleic Acids Research. 46 (20), 10782-10795 (2018).
- Segal, M., et al. High-throughput smFRET analysis of freely diffusing nucleic acid molecules and associated proteins. Methods. 169, 21-45 (2019).
- Kapanidis, A. N., et al. Fluorescence-aided molecule sorting: Analysis of structure and interactions by alternating-laser excitation of single molecules. Proceedings of the National Academy of Sciences. 101 (24), 8936-8941 (2004).
- Kapanidis, A. N., et al. Alternating-laser excitation of single molecules. Accounts of Chemical Research. 38 (7), 523-533 (2005).
- Müller, B. K., Zaychikov, E., Brauchle, C., Lamb, D. C. Pulsed interleaved excitation. Biophysical Journal. 89 (5), 3508-3522 (2005).
- Laurence, T. A., Kong, X., Jager, M., Weiss, S. Probing structural heterogeneities and fluctuations of nucleic acids and denatured proteins. Proceedings of the National Academy of Sciences of the United States of America. 102 (48), 17348-17353 (2005).
- Pollina, T., et al. PlanktonScope: Affordable modular imaging platform for citizen oceanography. bioRxiv. , 056978(2020).
- Collins, J. T., et al. Robotic microscopy for everyone: the OpenFlexure microscope. Biomedical Optics Express. 11 (5), 2447-2460 (2020).
- Courtney, A., Alvey, L. M., Merces, G. O. T., Burke, N., Pickering, M. The Flexiscope: a low cost, flexible, convertible and modular microscope with automated scanning and micromanipulation. Royal Society Open Science. 7 (3), 191949(2020).
- Martens, K. J. A., et al. Visualisation of dCas9 target search in vivo using an open-microscopy framework. Nature Communications. 10 (1), 3552(2019).
- Auer, A., et al. Nanometer-scale multiplexed super-resolution imaging with an economic 3D-DNA-PAINT microscope. ChemPhysChem. 19 (22), 3024-3034 (2018).
- Li, H., et al. Squid: Simplifying quantitative imaging platform development and deployment. bioRxiv. , 424613(2020).
- Ambrose, B., et al. The smfBox is an open-source platform for single-molecule FRET. Nature Communications. 11 (1), 5641(2020).
- Ingargiola, A., Laurence, T., Boutelle, R., Weiss, S., Michalet, X. Photon-HDF5: An open file format for timestamp-based single-molecule fluorescence experiments. Biophysical Journal. 110 (1), 26-33 (2016).
- Hellenkamp, B., et al. Precision and accuracy of single-molecule FRET measurements-a multi-laboratory benchmark study. Nature Methods. 15 (9), 669-676 (2018).
- Ingargiola, A., Lerner, E., Chung, S., Weiss, S., Michalet, X. FRETBursts: An open source toolkit for analysis of freely-diffusing single-molecule FRET. PLOS One. 11 (8), 0160716(2016).
- Torella, J. P., Holden, S. J., Santoso, Y., Hohlbein, J., Kapanidis, A. N. Identifying molecular dynamics in single-molecule FRET experiments with burst variance analysis. Biophysical Journal. 100 (6), 1568-1577 (2011).
- Tomov, T. E., et al. Disentangling subpopulations in single-molecule FRET and ALEX experiments with photon distribution analysis. Biophysical Journal. 102 (5), 1163-1173 (2012).
- Santoso, Y., Torella, J. P., Kapanidis, A. N. Characterizing single-molecule FRET dynamics with probability distribution analysis. ChemPhysChem. 11 (10), 2209-2219 (2010).
- Kalinin, S., Valeri, A., Antonik, M., Felekyan, S., Seidel, C. A. M. Detection of structural dynamics by FRET: A photon distribution and fluorescence lifetime analysis of systems with multiple states. The Journal of Physical Chemistry B. 114 (23), 7983-7995 (2010).
- Pirchi, M., et al. Photon-by-photon hidden Markov model analysis for microsecond single-molecule FRET kinetics. The Journal of Physical Chemistry B. 120 (51), 13065-13075 (2016).
- Schrimpf, W., Barth, A., Hendrix, J., Lamb, D. C. PAM: A framework for integrated analysis of imaging, single-molecule, and ensemble fluorescence data. Biophysical Journal. 114 (7), 1518-1528 (2018).
- Zijlstra, N., et al. Rapid microfluidic dilution for single-molecule spectroscopy of low-affinity biomolecular complexes. Angewandte Chemie International Edition. 56 (25), 7126-7129 (2017).
- Hellenkamp, B., Thurn, J., Stadlmeier, M., Hugel, T. Kinetics of transient protein complexes determined via diffusion-independent microfluidic mixing and fluorescence stoichiometry. The Journal of Physical Chemistry B. 122 (49), 11554-11560 (2018).
- Bennet, I. A., et al. Regional conformational flexibility couples substrate specificity and scissile phosphate diester selectivity in human flap endonuclease 1. Nucleic Acids Research. 46 (11), 5618-5633 (2018).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved