Method Article
Effectuer des mesures FRET à molécule unique précises et précises à l’aide de la smfBox open source
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Cet article fournit des instructions étape par étape pour effectuer des mesures FRET précises entièrement corrigées sur des biomolécules individuelles à diffusion libre à l’aide de la smfBox open source et peu coûteuse, de l’allumage à la collecte et à l’analyse de données, en passant par l’alignement et la mise au point.
Résumé
La smfBox est un instrument open source rentable récemment développé pour le transfert d’énergie de résonance de Förster à molécule unique (smFRET), qui rend les mesures sur les biomolécules à diffusion libre plus accessibles. Cette vue d’ensemble comprend un protocole étape par étape pour l’utilisation de cet instrument afin d’effectuer des mesures de l’efficacité FRET précise dans des échantillons d’ADN duplex, y compris des détails sur la préparation de l’échantillon, la configuration et l’alignement de l’instrument, l’acquisition de données et des routines d’analyse complètes. L’approche présentée, qui comprend la façon de déterminer tous les facteurs de correction requis pour des mesures de distance précises dérivées de FRET, s’appuie sur un grand nombre de travaux collaboratifs récents dans l’ensemble de la communauté FRET, qui visent à établir des protocoles et des approches d’analyse standard. Ce protocole, qui s’adapte facilement à une gamme de systèmes biomoléculaires, s’ajoute aux efforts croissants de démocratisation du smFRET pour la communauté scientifique au sens large.
Introduction
Le transfert d’énergie par résonance de Förster à molécule unique (smFRET) est une technique qui mesure l’efficacité FRET entre deux colorants - un donneur et un accepteur - au niveau de molécules individuelles. FRET est un processus photophysique résultant du chevauchement des spectres d’énergie de deux colorants: le donneur est excité par la lumière d’une longueur d’onde spécifique et transfère l’énergie de manière non radiative à l’accepteur, ce qui entraîne l’émission de l’accepteur. L’efficacité de ce transfert est inversement proportionnelle à la sixième puissance de la distance entre les deux colorants, de sorte que l’efficacité du transfert varie avec la distance1. Ainsi, cette efficacité FRET peut être utilisée pour déterminer des informations spatiales sur la ou les molécules 2 auxquelles les colorants sont attachés, dans une plage de 3 à 10 nm. Cette échelle, et le fait que les changements dans l’efficacité du FRET sont sensibles aux mouvements moléculaires d’Angstrom3, rend la technique bien adaptée à l’étude des informations structurelles sur les biomolécules - telles que les acides nucléiques et les protéines - sans les complications de la moyenne d’ensemble4,5,6. Alors que les changements dans l’efficacité relative du FRET peuvent être utilisés pour surveiller les interactions biomoléculaires et la dynamique conformationnelle, mettant en lumière les processus cellulaires clés tels que le (dé)repliement des protéines, la transcription et la réplication et la réparation de l’ADN, les rendements FRET absolus ont été utilisés pour déterminer des distances précises pour la détermination de la structure biomoléculaire7,8,9,10,11 , en surmontant le besoin de cristallisation ou de congélation comme c’est le cas pour d’autres méthodes structurelles4,12.
Les expériences smFRET prennent le plus souvent deux formes, la microscopie confocale ou la microscopie à fluorescence par réflexion interne totale (TIRF). Entre les deux approches, la dynamique moléculaire des biomolécules peut généralement être étudiée sur des échelles de temps allant du pico- à la milliseconde (molécules confocales, diffusant librement) jusqu’à la milliseconde à l’heure (TIRF, molécules immobilisées de surface). Cela est dû aux différentes configurations impliquées dans chaque technique. En microscopie TIRF, les molécules sont immobilisées à la surface d’une lame et excitées par une onde évanescente (Figure 1A). Ici, cependant, l’accent est mis sur la microscopie confocale car c’est le format de la smfBox. En microscopie confocale, les molécules ne sont pas immobilisées et diffusent librement par mouvement brownien à travers le volume confocal (~1 fL), formé en focalisant un faisceau laser à travers une lentille à haute ouverture numérique dans un endroit à une certaine profondeur désignée dans la solution (Figure 1B). L’émission résultante est focalisée à travers la même ouverture et filtrée à travers un miroir dichroïque (Figure 1C pour le schéma complet). Il est ensuite focalisé à travers un sténopé afin d’éliminer toute lumière floue et sur une photodiode d’avalanche (APD). Lorsque l’APD détecte un photon, il émet une impulsion TTL, dont la synchronisation peut être enregistrée avec une résolution allant jusqu’à la picoseconde. Le temps d’observation de ces molécules à diffusion libre au voisinage du volume confocal est généralement de l’ordre de quelques millisecondes.
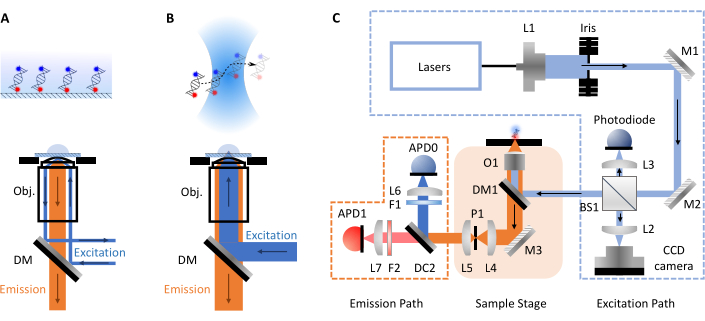
Figure 1: Schémas montrant les principes de la microscopie et la configuration smfBox. (A) Fluorescence par réflexion interne totale (TIRF) Principe de microscopie: la lumière d’excitation est dirigée vers le bord de l’objectif (Obj.) et subit une réflexion interne totale à l’interface coverslip-buffer générant un champ d’évanescence en décomposition exponentielle pour exciter les molécules attachées à la surface. (B) Microscopie confocale: Les molécules à diffusion libre sont excitées par un point proche de la diffraction limitée focalisé dans l’échantillon. (C) La configuration smfBox utilisée dans ce protocole, montrant tous les composants clés: photodiodes d’avalanche (APD), séparateur de faisceau (BS), miroirs dichroïques (DM), filtres (F), miroirs (M), objectif (O) et sténopé (P). Veuillez cliquer ici pour voir une version agrandie de cette figure.
Plus récemment, les techniques smFRET ont incorporé deux excitations de couleur, où des lasers correspondant aux longueurs d’onde d’excitation du donneur et de l’accepteur sont alternés5. Cela peut être fait de deux manières, la première en modulant les lasers à ondes continues sur l’échelle de temps KHz, connue sous le nom d’excitation laser alternée (ALEX)13,14. La deuxième méthode entrelace des impulsions rapides sur l’échelle de temps MHz; il s’agit de la nanoseconde-ALEX15 ou de l’excitation entrelacée pulsée (PIE)16. Dans toutes ces approches, les informations du laser accepteur conduisent au calcul de la stœchiométrie, qui peut discriminer entre les molécules à faible efficacité FRET et celles dépourvues d’accepteur (soit par marquage incomplet, soit par photoblanchiment). L’utilisation de PIE/ns-ALEX donne en outre accès à des durées de vie fluorescentes au niveau d’une seule molécule, et les anisotropies peuvent être mesurées lorsqu’elles sont couplées à une optique polarisante. Cette combinaison de mesures est connue sous le nom de détection de fluorescence multiparamètre (MFD)9.
Malgré les nombreux avantages de smFRET, il n’est pas largement utilisé en dehors des laboratoires spécialisés en raison des coûts élevés des instruments commerciaux et du manque d’alternatives simples et auto-construites. Une tendance croissante vers le développement de la microscopie opensource à faible coût a lieu et d’autres plates-formes ont récemment émergé, notamment Planktonscope17, OpenFlexure Microscope18, Flexiscope19, miCube20, liteTIRF21 et Squid22. L’étude décrit ici le protocole d’utilisation de la smfBox, une configuration confocale rentable récemment développée capable de mesurer l’efficacité FRET entre deux colorants sur des molécules uniques à diffusion libre. Des instructions de construction détaillées et tous les logiciels opérationnels nécessaires sont disponibles gratuitement à l’adresse suivante: https://craggslab.github.io/smfBox/ 23. La disposition optique de la smfBox est assemblée à partir de composants facilement disponibles achetés auprès de fabricants abordables et largement accessibles, tandis que le corps du microscope (responsable de la majorité des dépenses dans une configuration confocale standard) a été remplacé par une boîte en aluminium anodisé étanche à la lumière personnalisée (permettant d’effectuer des mesures dans des conditions de lumière ambiante). Cette boîte contient des composants optiques clés, y compris le dichroïque d’excitation, l’objectif et le sténopé, ainsi qu’un verrouillage laser mécanique, permettant son fonctionnement en toute sécurité en tant que produit laser de classe I (voir la figure 1C pour un schéma complet). La smfBox utilise ALEX pour valider la stœchiométrie du colorant et déterminer des facteurs de correction FRET précis. Il est exploité à l’aide d’un logiciel open source écrit sur mesure (smOTTER), qui contrôle tous les aspects de l’acquisition de données et les génère au format open source photon-HDF524, compatible avec de nombreux outils d’analyse tiers. La smfBox et les protocoles d’acquisition et d’analyse de données ont récemment été testés contre >20 autres instruments (confocaux et TIRF) dans le cadre d’une étude à l’aveugle multi-laboratoire25. Les gains d’efficacité FRET obtenus étaient en excellent accord avec tous les autres instruments, bien que la smfBox ne coûte qu’une fraction du prix des configurations disponibles dans le commerce.
Ici, un protocole étape par étape est décrit pour acquérir et analyser des efficacités FRET précises et absolues sur des duplex d’ADN diffusant librement à l’aide de la smfBox, de l’allumage à la collecte et à l’analyse des données, en passant par l’alignement et la mise au point. Les échantillons utilisés ici sont trois ADN duplex (présentant des rendements FRET élevés, moyens et faibles, voir le tableau 1) qui ont été évalués dans l’étude mondiale à l’aveugle25; cependant, la méthode est adaptable à de nombreux systèmes moléculaires, y compris les protéines et autres acides nucléiques. L’espoir est qu’un protocole aussi détaillé, ainsi que les instructions de construction déjà existantes pour la smfBox23, contribueront à rendre cette technique puissante encore plus accessible à un large éventail de laboratoires.
Protocole
1. Composants sous tension
- Mise sous tension des six prises (sans ordre particulier) : Piezoconcept (z-focus), APD0, APD1, laser vert, laser rouge et photodétecteur.
2. Logiciel 1-Configuration expérimentale
- Lancez le centre de contrôle laser.
- Assurez-vous que le mode Onde continue - courant constant alternatif (CW-ACC) est sélectionné.
- Allumez les deux lasers.
- Vérifiez/réglez les puissances du laser.
REMARQUE: La puissance du laser devra être ajustée comme mesurée juste avant le dichroïque d’excitation, car les filtres ND et les séparateurs de faisceau dans le chemin d’excitation réduiront la puissance du laser par rapport au nombre indiqué sur le panneau de commande laser. Les chiffres donnés ici sont la puissance à l’excitation dichroïque, mais la puissance sur la commande laser qui correspond à cela devra être déterminée.- Laser vert 220 μW (515 nm, 40 mW)
- μ70 W pour laser rouge (638 nm, 10 mW)
- Logiciel d’acquisition smOTTER
- Connectez les lasers, les détecteurs, l’étage Z et la caméra. Configurez-les correctement (peut varier en fonction des paramètres de configuration de la carte NI).
3. Alignement de la trajectoire d’émission (non requis systématiquement)
- Configuration de l’alignement
- Pipette 10 μL de colorant Cy3B libre (~100 nM) sur le microscope et mise au point comme décrit aux étapes 4.3-4.5 ci-dessous.
REMARQUE: Un autre colorant donneur avec une certaine fuite dans le canal accepteur fonctionnerait également. - Ouvrez l’onglet Alignement dans smOTTER, réduisez la puissance du laser et modifiez l’échelle de l’axe Y jusqu’à ce que la lecture soit vue par les détecteurs.
REMARQUE: L’objectif ici est d’augmenter le signal, de sorte que l’échelle peut devoir être modifiée à nouveau après l’augmentation du signal. - Dévissez les quatre vis à l’avant de la smfBox et retirez le panneau avant.
- Pipette 10 μL de colorant Cy3B libre (~100 nM) sur le microscope et mise au point comme décrit aux étapes 4.3-4.5 ci-dessous.
- Alignement des sténopés
- Tournez le bouton x sur le positionneur sténopé tout en regardant le signal sur l’onglet Alignement, en essayant d’augmenter le signal en vert et en rouge.
- Maintenant, tournez le bouton y pour aligner le sténopé dans l’autre sens.
- Revenez au bouton x pour vérifier s’il n’y a pas d’augmentation supplémentaire du signal.
- Alignement sténopé-lentille
- Tournez le bouton x de l’objectif dans une direction, cela diminuera le signal.
- Tournez le bouton pinhole x dans la même direction pour augmenter à nouveau le signal.
- Si le nouveau signal max est plus élevé qu’auparavant, continuez à déplacer de manière itérative le sténopé et l’objectif dans cette direction. S’il est plus bas qu’auparavant, déplacez-vous de manière itérative dans la direction opposée.
- Répétez ci-dessus pour y.
- Alignement de la lentille APD
- En commençant par l’APD vert, déplacez le bouton x jusqu’à ce que le signal vert soit au maximum.
- Répétez l’opération pour le bouton y.
- Revenez au bouton x, déplacez-vous d’avant en arrière pour trouver les points de seuil où le signal commence à tomber, et laissez-le à une position à mi-chemin entre ces deux points.
- Répétez l’opération pour le bouton x.
- Répétez les étapes ci-dessus pour l’objectif APD rouge, en regardant le signal rouge.
- Replacez le panneau avant sur la smfBox et remplacez les vis.
REMARQUE: Effectuez l’alignement mensuel ci-dessus, et par mesure de précaution s’il est soupçonné que le microscope a développé un défaut d’alignement entre les mesures.
4. Acquisition de données de mesure
- Pour le premier échantillon, nettoyez la scène avec du papier hygiénique imbibé de méthanol; essuyez doucement l’objectif d’un bout à l’autre.
- Appliquer 3-4 gouttes d’huile d’immersion pour la microscopie sur le centre de l’objectif; reconstituer au besoin entre les échantillons.
- Préparation des échantillons
- Pipette 10 μL d’échantillon (première utilisation d’eau ultrapure de type I) sur le centre d’un verre de couverture propre.
- Placez soigneusement le verre de couverture sur la lentille de l’objectif en l’abaissant à un angle par rapport à l’huile pour éviter de piéger les bulles d’air entre le verre de couverture et l’objectif.
REMARQUE: Si nécessaire, faites glisser le verre de couverture autour pour pousser les bulles d’air dans l’huile loin du point focal.
- Pour effectuer des expériences d’excitation laser alternée (ALEX), configurez le laser dans le panneau des cycles de service laser comme suit.
- Donneur (laser vert) 0 off, 45 on, 55 off
- Accepteur (laser rouge) 50 off, 45 on, 5 off
- Période ALEX (μs): 100
- Cliquez sur l’onglet Z focus dans le ruban ; dans le panneau d’acquisition, basculez les lasers sur Live et démarrez la caméra; ajustez l’exposition de sorte qu’un point lumineux apparaisse entouré de noir au centre.
REMARQUE: La caméra capture la lumière réfléchie par l’interface verre/eau entre le couvercle et l’échantillon et le cercle visible de la lumière est à son plus petit diamètre lorsque le point focal est à cette interface. Il est donc nécessaire de commencer en dessous de cette interface, d’augmenter la hauteur z jusqu’à l’interface, puis de se concentrer légèrement plus loin au-dessus de la bascule et dans l’échantillon mesuré.- En partant d’une position Z basse, augmentez la hauteur jusqu’à ce que le point lumineux atteigne sa taille minimale, puis augmentez la hauteur jusqu’à 20 μm pour focaliser le laser au-dessus de l’huile et du verre de couverture et dans l’échantillon.
- Arrêtez l’appareil photo une fois au point.
- Pour confirmer que la configuration a été effectuée de manière appropriée, surveillez la trace d’eau ultrapure de type I pour ne voir aucun signal de fluorescence; répéter la confirmation de la pureté des tampons dans lesquels les échantillons sont préparés.
- Retirez soigneusement le verre de couverture avec une pince à épiler en caoutchouc pour éviter d’endommager l’objectif, puis placez l’échantillon d’intérêt préparé comme ci-dessus sur l’objectif. Reconstituer l’huile d’immersion au besoin pour maintenir une zone de contact uniforme entre le verre de couverture et l’objectif (une attention particulière est accordée à la zone de contact de l’échantillon sur le verre de couverture et à la zone focale de l’objectif)
- Bingo de concentration
- Pour s’assurer que les données sur une seule molécule sont obtenues, les échantillons doivent être à une concentration à laquelle 1 à 5 rafales par seconde sont observées dans le panneau de traces vivantes de smOTTER (cela minimise le risque que plus d’une molécule soit présente dans le volume de détection à la fois). Prenez les mesures lorsque la concentration appropriée a été déterminée.
- Pour éviter l’évaporation lors de longues expériences, préparez une chambre d’échantillonnage étanche à l’air. Appuyez sur les isolateurs en silicone (trou de 8-9 mm de diamètre) sur le centre d’un verre de couverture (coloré avec une pointe de pipette). Ensuite, pipetez soigneusement un échantillon au centre, en évitant tout contact avec le silicone. Ensuite, placez un deuxième verre de couverture sur le dessus et appuyez pour former un joint.
- Vérifiez l’histogramme stoichiométrie en direct vs efficacité FRET (ES) pour voir si l’échantillon se comporte comme prévu, c’est-à-dire l’efficacité FRET attendue et la stœchiométrie raisonnable (~0,5).
- Lorsque l’exemple est prêt, entrez les détails de l’expérience dans le panneau des paramètres d’enregistrement. Choisissez un répertoire et un nom de fichier appropriés en cliquant sur l’icône de sélection (fichiers enregistrés au format hdf5/h5) dans le panneau des paramètres de sauvegarde.
- Entrez des informations pour le nom de l’échantillon, les détails de l’échantillon, les étiquettes du donneur et de l’accepteur, le tampon, les longueurs d’onde d’excitation du donneur et de l’accepteur, les longueurs d’onde de détection et la puissance laser, ainsi que l’affiliation de l’utilisateur et de l’utilisateur.
- Revenez à l’onglet trace dynamique du ruban et, dans le panneau d’acquisition, entrez la durée de l’expérience (en minutes), sélectionnez un intervalle d’enregistrement approprié pour atténuer la perte potentielle de données en cas d’erreur. Sélectionnez Enregistrer les puissances laser, si nécessaire.
- Appuyez sur Démarrer pour prendre les données.
REMARQUE: Pour permettre une détermination précise de la FRET, au moins deux échantillons devront être mesurés contenant les mêmes colorants donneurs et accepteurs, mais avec des rendements FRET suffisamment différents.
5. Analyse/logiciel 2
- Lancez le bloc-notes Jupyter avec le package python FRETBursts (les instructions de configuration peuvent être trouvées ici: https://craggslab.github.io/smfBox/anasoftware.html)
- Si la correction de l’efficacité absolue du FRET n’est pas nécessaire (c’est-à-dire que seuls les changements relatifs du FRET sont suffisants pour la mesure), lancez l’ordinateur portable Jupyter FRET Analysis 1.4 Uncorrected.ipynb. Utilisez-le pour exporter des figures ou des données sous forme de fichiers csv pour une analyse plus approfondie ou un traçage dans d’autres logiciels.
REMARQUE : Chaque bloc-notes contient des instructions détaillées pour guider l’utilisateur à travers les procédures d’analyse. Pour une discussion plus détaillée des procédures d’analyse, y compris les algorithmes de recherche en rafale, la correction en arrière-plan et tous les paramètres de correction, voir23,25. - Si une correction pour les rendements FRET absolus est nécessaire, commencez par calculer les paramètres de diaphonie alpha (fuite de l’émission du donneur dans le canal accepteur) et delta (proportion d’excitation directe de l’accepteur sous excitation du donneur).
- Lancez d’abord Jupyter notebook Correction Factor Finder Alpha-Delta.ipynb et déterminez les paramètres alpha et delta.
REMARQUE: Si ceux-ci sont incohérents entre les échantillons, le microscope peut avoir développé un problème d’alignement entre les mesures ou les spectres des colorants diffèrent entre les échantillons. - Si les paramètres alpha et delta sont cohérents, lancez Jupyter notebook Correction Factor Finder Gamma-Beta.ipynb pour déterminer les paramètres gamma et bêta (qui tiennent compte des différences d’excitation et d’efficacité de détection entre les colorants).
REMARQUE: Si le diagramme gamma et bêta ne s’adapte pas bien, le microscope peut avoir développé un problème d’alignement entre les mesures, ou les rendements quantiques, ou les coefficients d’extinction des colorants diffèrent entre les échantillons. - Une fois les quatre paramètres de diaphonie déterminés, ces facteurs peuvent être utilisés dans l’analyse FRET 1.4 Corrected.ipynb Jupyter notebook pour déterminer l’efficacité FRET absolue.
- Lancez d’abord Jupyter notebook Correction Factor Finder Alpha-Delta.ipynb et déterminez les paramètres alpha et delta.
6. Dépannage
- Si tous les signaux sont faibles ou si le nombre par rafale est inférieur à celui prévu (cela dépend du colorant, mais pour ATTO-550 et ATTO-647N sur la smfBox, les valeurs typiques sont comprises entre 50 et 100 comptes par ms lors d’une rafale à molécule unique), réalignez la smfBox.
- Pont entre les populations doublement et individuellement étiquetées sur l’histogramme ES.
REMARQUE: Cela peut être causé soit par le travail à une concentration trop élevée (remédier à cela en diluant l’échantillon), soit par le photoblanchiment, qui peut être causé par l’utilisation d’une puissance laser trop élevée (remédier à cela en réduisant la puissance laser). - Si le taux d’éclatement diminue tout au long de l’expérience (des molécules marquées par fluorescence adhèrent probablement au couvercle en verre), utilisez une concentration accrue de BSA pour rectifier.
- Si le taux d’éclatement augmente tout au long de l’expérience (l’échantillon est susceptible de s’évaporer), préparez une chambre d’échantillonnage étanche à l’air comme décrit ci-dessus.
- Si le signal tombe à zéro pendant l’alignement (le logiciel est probablement submergé par le signal des détecteurs), réduisez la puissance du laser ou utilisez un échantillon d’alignement plus dilué.
- Si Z-focus montre des anneaux concentriques (cela peut se produire si plusieurs couvercles ont été placés sur l’objectif), vérifiez s’il y a plusieurs glissements de couverture sur l’objectif.
- Si l’échantillon contient de longues et brillantes rafales de stœchiométries intermédiaires (causées par des molécules agrégées), utilisez des détergents ou modifiez les protocoles de purification des échantillons.
REMARQUE: Obtenir un tampon propre peut être un problème, car certains composants du tampon contiennent souvent de très petites quantités de contaminants modérément fluorescents qui sont suffisants pour se présenter sous forme de rafales de couleur unique sur la trace temporelle. S’il y en a trop dans le tampon, cela peut coïncider avec les rafales d’échantillon et modifier l’efficacité FRET ou la stœchiométrie mesurée. Le BSA en particulier peut souvent être problématique à cet égard, il est donc utile d’exposer une solution de BSA de base à une source de lumière forte pour photoblanchiment les contaminants.
Résultats
Le protocole nécessite une évaluation critique des conditions expérimentales lors de la configuration (voir l’étape 4.8 du protocole). Les premiers résultats obtenus qui déterminent le succès ou l’échec de l’expérience sont obtenus à ce stade. Un résultat positif serait d’avoir entre cinq et une rafales par seconde (voir Figure 2B,C). Un résultat négatif serait d’avoir trop de rafales (Figure 2A) ou trop peu (Figure 2D) dans ce laps de temps. Il reste possible à ce stade de rectifier ces erreurs: un échantillon avec une concentration trop élevée doit simplement être dilué; toutefois, si la concentration est trop faible, il peut être nécessaire de préparer un nouvel échantillon (le déterminant étant de savoir s’il reste possible à cette faible concentration de recueillir des données dans un délai raisonnable).
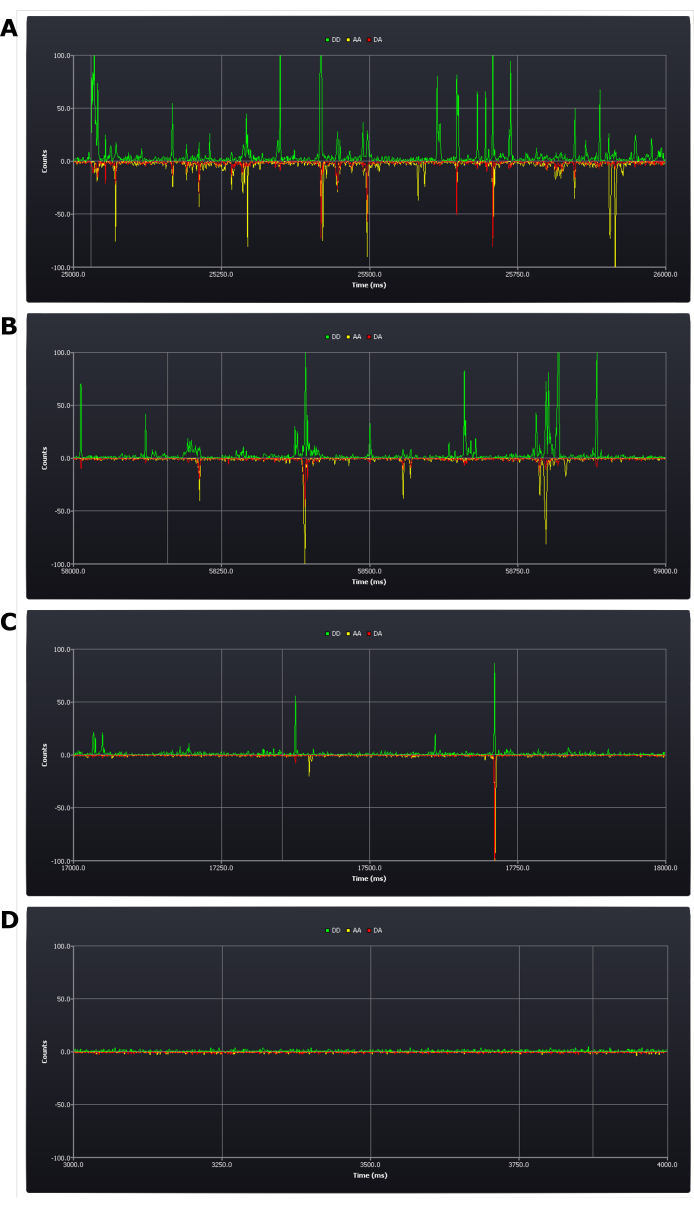
Figure 2 : Captures d’écran de traces vivantes lors de la configuration expérimentale montrant différentes concentrations d’échantillons d’ADN duplex doublement marqués. (A) trop élevé, (B) limite supérieure acceptable, (C) concentration cible, (D) trop faible. Le nombre de photons (bacs de 1 ms) est indiqué dans les trois canaux de détection; émission du donneur après l’excitation du donneur (DD), émission de l’accepteur après l’excitation du donneur (DA) et émission de l’accepteur après l’excitation de l’accepteur (AA). Veuillez cliquer ici pour voir une version agrandie de cette figure.
Un système statique mono-espèce nécessiterait généralement 30 à 60 minutes de mesure pour obtenir les ~ 1 000 rafales nécessaires à une analyse robuste des données. La durée et le nombre de rafales requises augmenteront avec plusieurs espèces ou systèmes dynamiques. Après la collecte et l’analyse des données à l’aide du protocole, les figures sont exportées à partir des blocs-notes Jupyter. Le diagramme d’alternance (figure 3A) doit correspondre à la période ALEX de la configuration expérimentale. La trace temporelle (figure 3B) est utilisée pour évaluer qualitativement que la concentration de l’échantillon est raisonnable. Le diagramme d’arrière-plan (figure 3C) montre la distribution des périodes de retard interphotoniques avec un ajustement linéaire aux temps plus longs pour estimer le taux de fond26. La trace de fond (figure 3D) peut identifier s’il y a eu des changements dans l’échantillon au cours de la durée de l’expérience; cela serait principalement dû à l’évaporation pendant des périodes d’acquisition plus longues. Des histogrammes ES sont générés pour tous les photons (Figure 3E) et les espèces doublement marquées (Figure 3F). Enfin, un histogramme 1D E (Figure 3G) est généré avec un ajustement gaussien des données de rafale.
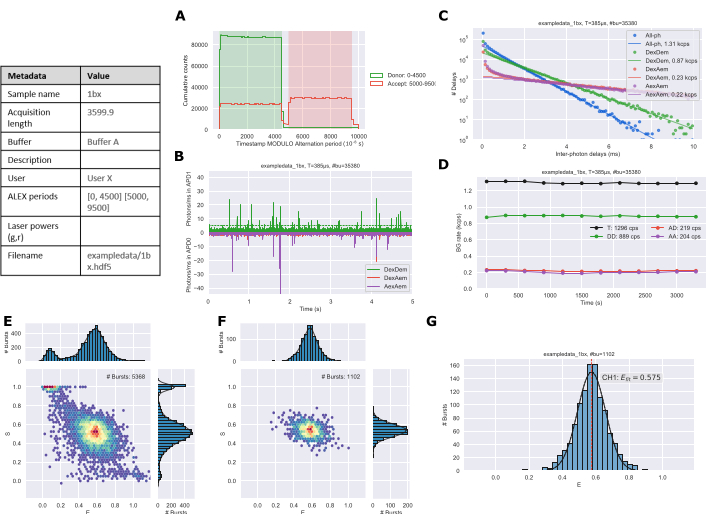
Figure 3 : Exemple de sortie de figure de données analysées générées par les blocs-notes Jupyter. (A) Diagramme d’alternance, (B) Trace temporelle, (C) Détermination de l’arrière-plan, (D) Taux d’arrière-plan, (E) Histogramme ES à tous les photons, (F) Histogramme ES double canal et (G) Histogramme 1D E. Veuillez cliquer ici pour voir une version agrandie de cette figure.
| Nom | Séquence |
| 1a | 5'- GAG CTG AAA GTG TCG AGT TTG TTT GAG TGT TTG TCT GG - 3' |
| 3'- CTC GAC TTT CAC AGC TCA AAC AAA CTC ACA AAC AGA CC - 5' | |
| 1b | 5'- GAG CTG AAA GTG TCG AGT TTG TTT GAG TGT TTG TCT GG - 3' |
| 3'- CTC GAC TTT CAC AGC TCA AAC AAA CTC ACA AAC AGA CC - 5' | |
| 1c | 5'- GAG CTG AAA GTG TCG AGT TTG TTT GAG TGT TTG TCT GG - 3' |
| 3'- CTC GAC TTT CAC AGC TCA AAC AAA CTC ACA AAC AGA CC - 5' |
Tableau 1 : Séquences d’ADN utilisées dans le protocole. Les nucléotides sont surlignés en bleu et en rouge représentant les résidus de thymine modifiés par l’aminé C2 marqués avec Atto-550 et Atto-647N, respectivement.
Correction Factor Finder Alpha-Delta: Veuillez cliquer ici pour télécharger ce fichier.
Correction Factor Finder Gamma-Beta: Veuillez cliquer ici pour télécharger ce fichier.
Analyse FRET 1.4 Corrigé : Veuillez cliquer ici pour télécharger ce fichier.
Analyse FRET 1.4 Non corrigé : Veuillez cliquer ici pour télécharger ce fichier.
Discussion
Les étapes les plus critiques du protocole sont l’alignement du microscope et l’ajustement de la concentration de l’échantillon à la dilution correcte. Si l’alignement est désactivé, il se peut que le signal soit insuffisant pour identifier les rafales et tracer les histogrammes, et si un désalignement se produit entre les échantillons, la correction FRET précise peut échouer en raison de changements dans les rendements de fuite et de détection / excitation. L’utilisation d’une concentration appropriée est également importante, une concentration trop élevée donnera des rafales coïncidentes, contenant plusieurs molécules avec des efficacités FRET potentiellement différentes ou marquant des stœchiométries. Une concentration trop faible donnera trop peu de rafales pour une analyse robuste des données.
Le protocole décrit ici est destiné à mesurer les distances chez les espèces FRET uniques statiques. Si l’échantillon a plus d’un pic dans l’histogramme d’efficacité FRET, ou si les pics semblent larges (ce qui peut arriver avec des espèces dynamiques), alors plus de rafales peuvent être nécessaires pour adapter les histogrammes au même degré de précision. Pour deux pics bien séparés, environ deux fois plus de données seront nécessaires, mais si les populations se chevauchent légèrement, il faut encore plus de données.
Si les deux populations s’interconvertissent sur l’échelle de temps de l’expérience, la dynamique et la cinétique du système peuvent potentiellement être déterminées. Des tests tels que BVA27 et 2CDE28 peuvent confirmer que les sursauts intermédiaires sont de nature dynamique, tandis que des analyses incluant dPDA29,30 ou H2MM31 peuvent déterminer les taux d’interconversion. Les blocs-notes Jupyter pour BVA et 2CDE sont disponibles sur le site Web FRETBursts26, et le logiciel PAM32 basé sur MATLab peut exécuter des analyses BVA, 2CDE et PDA.
La FRET confocale à molécule unique peut facilement observer des états de durée de vie beaucoup plus courte (~1 ms) que le TIRF; cependant, les temps d’observation courts, limités par la diffusion, ne donnent pas d’histoire moléculaire, et ne peuvent donc pas déterminer des temps de séjour plus longs, ou des réseaux de transition complexes de la même manière que les expériences immobilisées en surface.
Comme le protocole mesure des molécules à diffusion libre à une très faible concentration, il fonctionne mieux lors de la mesure de distances intramoléculaires sur la même molécule. Les distances intermoléculaires entre les molécules liées transitoirement peuvent être mesurées à condition que le Kd des deux molécules soit suffisamment faible pour que le complexe existe à une quantité significative à la faible concentration de travail requise par l’expérience (~100 pM). Si le Kd est beaucoup plus élevé que cela, alors seules les molécules marquées individuellement seront vues. Ce problème peut être surmonté en utilisant la microfluidique pour mélanger les deux composants marqués ensemble à une concentration élevée, puis en diluant et en s’écoulant rapidement sur l’objectif avant que le complexe ne se dissocie33,34.
La mesure de l’efficacité fret au niveau de la molécule unique présente un avantage significatif par rapport aux techniques d’ensemble, car elle informe sur des sous-populations hétérogènes qui, dans une expérience d’ensemble, seraient moyennées. De plus, FRET à molécule unique avec ALEX donne accès à des rendements FRET précis, qui peuvent être convertis en distances précises. Cela permet de déterminer des informations structurelles plus détaillées plutôt que de simplement sonder les changements de distance relative. Le smfBox offre tous ces avantages et capacités, mais peut être construit avec un budget beaucoup plus faible que les microscopes comparables disponibles dans le commerce capables de smFRET23 confocale.
La smfBox représente une barrière à l’entrée beaucoup plus faible pour les techniques smFRET, permettant aux chercheurs de mesurer les changements conformationnels et les distances précises à l’intérieur et entre les protéines et les acides nucléiques7,8,9,10,11,35.
Déclarations de divulgation
Les auteurs ne déclarent aucun intérêt concurrent.
Remerciements
Les auteurs remercient les sources de financement suivantes : BBSRC (BB/T008032/1); EPSRC (Studentship to B.A.) et MRC (Studentship to A. R.-T.).
matériels
| Name | Company | Catalog Number | Comments |
| Amino modified oligonucleotide | Eurogentec | N/A | May be ordered from various suppliers or synthesised; amino modification enables labeling with NHS-ester modified dyes |
| Avalanche photodiode (APD) | Excelitas | SPCM-AQRH-14 | Two APDs are required for the smfBox setup |
| Bovine Serum Albumin (BSA) | Merck | A2153 | System dependant; imaging buffer component (0.1 mg/mL in buffer) |
| Compact Laser Combiner | OMICRON | LightHUB-2 | 515 nm (80 mW) and 638 nm (100 mW) lasers |
| Coverglass | VWR | 630-2742 | Thickness: 0.17 ± 0.01 mm, LxW: 22x22 mm |
| Cy3B | Cytiva | PA63101 | 1 mg, PA63100 (5 mg), PA96106 (25 mg) |
| FRETBursts Python Package | N/A | N/A | Open-source python package for burst analysis of freely-diffusing single-molecule FRET data: https://fretbursts.readthedocs.io |
| Imaging Buffer | N/A | System dependant; 5 mM NaCl, 20 mM MgCl2, 5 mM Tris pH 7.5 and 0.1 mg/mL BSA | |
| Immersion Oil | Olympus | IMMOIL-F30CC | |
| Jupyter notebooks | Project Jupyter | N/A | Open-source web application to create and share documents that contain live code, equations, visualizations and text; data analysis notebooks for smfBox can be found in the SI |
| Lens Tissue | ThorLabs | MC-5 | MC-50E is same item in bulk |
| Magnesium Chloride | Merck | M2670 | System dependant; imaging buffer component (20 mM in buffer) |
| MilliQ/Ultrapure water | N/A | ||
| Nanopoistioner | Piezoconcept | FOC300 | Nanopositioner for accurate positioning of microscope objective |
| NHS-ester modified ATTO-550 | ATTO-TEC | AD 550-31 | 1 mg, AD 550-35 (5 mg) |
| NHS-ester modified ATTO-647N | ATTO-TEC | AD 647N-31 | 1 mg, AD 647N-35 (5 mg) |
| Objective lens | Olympus | N1480700 | Olympus objective series from orignal smfBox discontinued; replaced by N5702300 |
| OMICRON Control Center (OCC)- laser control center | OMICRON | N/A | v3.5.34 - OMICRON laser driver software |
| Press-To-Seal silicone isolator | Grace Bio-Labs | 664201 | 8-9 mm Diameter x 1.7 mm Depth |
| smOTTER | N/A | N/A | Open-source acquisition software for the Craggs Lab smfBox: https://github.com/craggslab/smOTTER |
| Sodium Chloride | Merck | S7653 | System dependant; imaging buffer component (5 mM in buffer) |
| Tris base | Merck | 93362 | System dependant; imaging buffer component (5 mM, pH 7.5 in buffer) |
| Type I ultrapure water | Merck | ZIQ7000T0 | Milli-Q® IQ 7000 Ultrapure Water System |
Références
- Forster, T. Intermolecular energy migration and fluorescence. Annals of Physics. 437 (1-2), 55-75 (1948).
- Stryer, L., Haugland, R. P. Energy transfer: a spectroscopic ruler. Proceedings of the National Academy of Sciences. 58 (2), 719-726 (1967).
- Hohlbein, J., et al. Conformational landscapes of DNA polymerase I and mutator derivatives establish fidelity checkpoints for nucleotide insertion. Nature Communications. 4 (1), 2131(2013).
- Lerner, E., et al. Toward dynamic structural biology: Two decades of single-molecule Förster resonance energy transfer. Science. 359 (6373), (2018).
- Hohlbein, J., Craggs, T. D., Cordes, T. Alternating-laser excitation: single-molecule FRET and beyond. Chemical Society Reviews. 43 (4), 1156-1171 (2014).
- Lerner, E., et al. The FRET-based structural dynamics challenge -- community contributions to consistent and open science practices. arXiv. , (2020).
- Hellenkamp, B., Wortmann, P., Kandzia, F., Zacharias, M., Hugel, T. Multidomain structure and correlated dynamics determined by self-consistent FRET networks. Nature Methods. 14 (2), 174-180 (2017).
- Craggs, T. D., et al. Substrate conformational dynamics facilitate structure-specific recognition of gapped DNA by DNA polymerase. Nucleic Acids Research. 47 (20), 10788-10800 (2019).
- Tsytlonok, M., et al. Dynamic anticipation by Cdk2/Cyclin A-bound p27 mediates signal integration in cell cycle regulation. Nature Communications. 10 (1), 1676(2019).
- Nagy, J., et al. Complete architecture of the archaeal RNA polymerase open complex from single-molecule FRET and NPS. Nature Communications. 6 (1), 6161(2015).
- LeBlanc, S. J., et al. Coordinated protein and DNA conformational changes govern mismatch repair initiation by MutS. Nucleic Acids Research. 46 (20), 10782-10795 (2018).
- Segal, M., et al. High-throughput smFRET analysis of freely diffusing nucleic acid molecules and associated proteins. Methods. 169, 21-45 (2019).
- Kapanidis, A. N., et al. Fluorescence-aided molecule sorting: Analysis of structure and interactions by alternating-laser excitation of single molecules. Proceedings of the National Academy of Sciences. 101 (24), 8936-8941 (2004).
- Kapanidis, A. N., et al. Alternating-laser excitation of single molecules. Accounts of Chemical Research. 38 (7), 523-533 (2005).
- Müller, B. K., Zaychikov, E., Brauchle, C., Lamb, D. C. Pulsed interleaved excitation. Biophysical Journal. 89 (5), 3508-3522 (2005).
- Laurence, T. A., Kong, X., Jager, M., Weiss, S. Probing structural heterogeneities and fluctuations of nucleic acids and denatured proteins. Proceedings of the National Academy of Sciences of the United States of America. 102 (48), 17348-17353 (2005).
- Pollina, T., et al. PlanktonScope: Affordable modular imaging platform for citizen oceanography. bioRxiv. , 056978(2020).
- Collins, J. T., et al. Robotic microscopy for everyone: the OpenFlexure microscope. Biomedical Optics Express. 11 (5), 2447-2460 (2020).
- Courtney, A., Alvey, L. M., Merces, G. O. T., Burke, N., Pickering, M. The Flexiscope: a low cost, flexible, convertible and modular microscope with automated scanning and micromanipulation. Royal Society Open Science. 7 (3), 191949(2020).
- Martens, K. J. A., et al. Visualisation of dCas9 target search in vivo using an open-microscopy framework. Nature Communications. 10 (1), 3552(2019).
- Auer, A., et al. Nanometer-scale multiplexed super-resolution imaging with an economic 3D-DNA-PAINT microscope. ChemPhysChem. 19 (22), 3024-3034 (2018).
- Li, H., et al. Squid: Simplifying quantitative imaging platform development and deployment. bioRxiv. , 424613(2020).
- Ambrose, B., et al. The smfBox is an open-source platform for single-molecule FRET. Nature Communications. 11 (1), 5641(2020).
- Ingargiola, A., Laurence, T., Boutelle, R., Weiss, S., Michalet, X. Photon-HDF5: An open file format for timestamp-based single-molecule fluorescence experiments. Biophysical Journal. 110 (1), 26-33 (2016).
- Hellenkamp, B., et al. Precision and accuracy of single-molecule FRET measurements-a multi-laboratory benchmark study. Nature Methods. 15 (9), 669-676 (2018).
- Ingargiola, A., Lerner, E., Chung, S., Weiss, S., Michalet, X. FRETBursts: An open source toolkit for analysis of freely-diffusing single-molecule FRET. PLOS One. 11 (8), 0160716(2016).
- Torella, J. P., Holden, S. J., Santoso, Y., Hohlbein, J., Kapanidis, A. N. Identifying molecular dynamics in single-molecule FRET experiments with burst variance analysis. Biophysical Journal. 100 (6), 1568-1577 (2011).
- Tomov, T. E., et al. Disentangling subpopulations in single-molecule FRET and ALEX experiments with photon distribution analysis. Biophysical Journal. 102 (5), 1163-1173 (2012).
- Santoso, Y., Torella, J. P., Kapanidis, A. N. Characterizing single-molecule FRET dynamics with probability distribution analysis. ChemPhysChem. 11 (10), 2209-2219 (2010).
- Kalinin, S., Valeri, A., Antonik, M., Felekyan, S., Seidel, C. A. M. Detection of structural dynamics by FRET: A photon distribution and fluorescence lifetime analysis of systems with multiple states. The Journal of Physical Chemistry B. 114 (23), 7983-7995 (2010).
- Pirchi, M., et al. Photon-by-photon hidden Markov model analysis for microsecond single-molecule FRET kinetics. The Journal of Physical Chemistry B. 120 (51), 13065-13075 (2016).
- Schrimpf, W., Barth, A., Hendrix, J., Lamb, D. C. PAM: A framework for integrated analysis of imaging, single-molecule, and ensemble fluorescence data. Biophysical Journal. 114 (7), 1518-1528 (2018).
- Zijlstra, N., et al. Rapid microfluidic dilution for single-molecule spectroscopy of low-affinity biomolecular complexes. Angewandte Chemie International Edition. 56 (25), 7126-7129 (2017).
- Hellenkamp, B., Thurn, J., Stadlmeier, M., Hugel, T. Kinetics of transient protein complexes determined via diffusion-independent microfluidic mixing and fluorescence stoichiometry. The Journal of Physical Chemistry B. 122 (49), 11554-11560 (2018).
- Bennet, I. A., et al. Regional conformational flexibility couples substrate specificity and scissile phosphate diester selectivity in human flap endonuclease 1. Nucleic Acids Research. 46 (11), 5618-5633 (2018).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon