Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Methods Article
Intercambio de hidrógeno/deuterio basado en electroforesis capilar para la caracterización conformacional de proteínas con espectrometría de masas de arriba hacia abajo
En este artículo
Resumen
Aquí se presenta un protocolo para un enfoque de intercambio de hidrógeno / deuterio (HDX) basado en electroforesis capilar junto con espectrometría de masas de arriba hacia abajo. Este enfoque caracteriza la diferencia en las estructuras de orden superior entre diferentes especies de proteínas, incluidas proteínas en diferentes estados y diferentes proteoformas, mediante la realización de HDX diferencial concurrente y separación electroforética.
Resumen
Resolver la heterogeneidad conformacional de múltiples estados proteicos que coexisten en solución sigue siendo uno de los principales obstáculos en la caracterización de la terapéutica proteica y la determinación de las vías de transición conformacional críticas para las funciones biológicas, que van desde el reconocimiento molecular hasta la catálisis enzimática. La reacción de intercambio de hidrógeno/deuterio (HDX) junto con el análisis espectrométrico de masas (MS) de arriba hacia abajo proporciona un medio para caracterizar las estructuras y dinámicas de orden superior de proteínas de una manera específica del conformador. El poder de resolución conformacional de esta técnica depende en gran medida de la eficiencia de separar los estados de proteína a nivel de proteína intacta y minimizar el contenido protico residual no deuterado durante las reacciones HDX.
Aquí describimos una variante basada en electroforesis capilar (CE) del enfoque HDX MS que tiene como objetivo mejorar la resolución conformacional. En este enfoque, las proteínas experimentan reacciones HDX mientras migran a través de una solución de electrolito de fondo deuterado (BGE) durante la separación electroforética capilar. Los diferentes estados de proteínas o proteoformas que coexisten en solución se pueden separar de manera eficiente en función de sus diferentes proporciones de carga a tamaño. La diferencia en la movilidad electroforética entre las proteínas y las moléculas de disolvente prótico minimiza el disolvente residual no deuterado, lo que resulta en un entorno de deuteratización casi completo durante el proceso HDX. La interfaz CE-MS microvial de flujo continuo permite una ionización electrospray eficiente de las especies de proteínas eluidas después de una rápida mezcla con la solución modificadora de enfriamiento y desnaturalización en la salida del pulverizador. El análisis de EM de arriba hacia abajo en línea mide el nivel de deuteración global de las especies de proteínas intactas eluidas y, posteriormente, la deuteración de sus fragmentos en fase gaseosa. Este artículo demuestra este enfoque en HDX diferencial para sistemas, incluidas las variantes de proteínas naturales que coexisten en la leche.
Introducción
Distinguir las especies de proteínas en diferentes estados conformacionales, de unión o modificación y caracterizar sus diferencias estructurales es importante para monitorear las vías de transición entre estas especies involucradas en eventos biológicos, que van desde el reconocimiento molecular hasta la catálisis enzimática, y comprender los mecanismos subyacentes a estos eventos. Las técnicas biofísicas convencionales no proporcionan una solución completa debido a las limitaciones como la resolución insuficiente y la pérdida de información dinámica en la solución. El intercambio hidrógeno/deuterio junto con la espectrometría de masas (HDX MS) es una técnica que etiqueta las características estructurales y conformacionales de las proteínas con deuterio (2H) a través del intercambio entre átomos de hidrógeno lábil de proteínas y 2H de la solución 2H2O introducida deliberadamente. Los protones involucrados en el enlace de hidrógeno o que están secuestrados del disolvente en el interior de la proteína no se intercambian fácilmente1. Por lo tanto, como el tipo de cambio en un sitio intercambiable depende en gran medida de su participación en estructuras de orden superior, las estructuras de proteínas pueden ser reveladas a alta resolución espacial por MS que sondea la extensión y la tasa de absorción de 2H en función de las diferentes masas atómicas entre 1H y 2H. En las últimas décadas, HDX MS se ha convertido en una técnica extraordinariamente exitosa para estudiar conformaciones y dinámicas de proteínas2.
En el enfoque clásico de abajo hacia arriba de HDX MS, el conjunto de especies de proteínas en diferentes estados conformacionales, de unión o modificación se proteoliza sin separación a nivel de proteína intacta, lo que hace inviable caracterizar especies individuales mediante el análisis de los fragmentos proteolíticos resultantes con contenido de deuterio enrevesado. En contraste, en el enfoque de arriba hacia abajo, diferentes estados de proteínas o proteoformas que han incorporado diferentes contenidos de deuterio dan lugar a múltiples distribuciones de masas de proteínas intactas en una exploración de EM. Esto permite separar especies individuales mediante la selección masiva de iones correspondientes a cada distribución de masa utilizando un filtro de masa adecuado (como un cuadrupolo) y la caracterización de sus diferencias conformacionales en el posterior análisis de EM en tándem 3,4,5,6. Sin embargo, la eficiencia de separar los estados proteicos o proteoformas en esta estrategia está limitada por el grado de diferencia en sus correspondientes distribuciones de masa.
La electroforesis capilar (CE) proporciona un medio para separar las especies de proteínas en función de sus diferentes cargas y tamaños hidrodinámicos en la fase de solución con alta eficiencia7. La combinación de CE con HDX ofrece una separación adicional de los estados proteicos o proteoformas en la fase de solución. Además, el pequeño volumen del capilar CE permite la utilización de una solución totalmente deuterada como solución electrolítica de fondo (BGE), es decir, el tampón en funcionamiento, lo que hace que el capilar sea un reactor HDX para muestras de proteínas. Debido a la diferencia en la movilidad electroforética entre las proteínas y los reactivos próticos en el proceso de electroforesis, la realización de HDX durante la CE da como resultado un entorno de deuteratización casi completo para los analitos de proteínas con un contenido residual no deuterado mínimo, mejorando así la sensibilidad del análisis estructural utilizando datos hdx. Como tal, desarrollamos un enfoque HDX diferencial basado en CE junto con MS de arriba hacia abajo para caracterizar las estructuras de orden superior de proteínas de una manera específica de estado o proteoforma8.
Este documento describe los protocolos para este enfoque detallando los pasos de preparación de materiales, procedimientos experimentales y análisis de datos. Los factores que pueden afectar el rendimiento del método o la calidad de los datos se enumeran en notas cortas. Los resultados representativos aquí presentados incluyen datos diferenciales de HDX de mezclas de diferentes proteínas y variantes naturales de β-lactoglobulina bovina (β-lg), la principal proteína de suero presente en la leche9. Demostramos eficiencia de separación, reproducibilidad y rendimiento de etiquetado 2H de las dos variantes abundantes de β-lg, es decir, A y B10,11 durante el HDX basado en CE y la caracterización específica de variantes de sus conformaciones.
Protocolo
NOTA: Utilice reactivos de grado de cromatografía líquida de alto rendimiento (HPLC) o grado MS siempre que sea posible para minimizar los contaminantes que pueden interferir con el análisis de EM. No toque la interfaz CE-MS con las manos desnudas durante la medición para evitar la posibilidad de una descarga eléctrica causada por el voltaje electroforético o el voltaje electrospray.
1. Preparación del material
- Modificación del capilar de sílice fundida para CE
- Preparar una solución de hidroxipropilcelulosa (HPC) al 5% (p/p) disolviendo polvo de HPC (peso molecular [MW]: 100 kDa) en agua con agitación continua a temperatura ambiente en un agitador magnético durante ~12 h o hasta la desaparición completa de partículas sólidas12. Elimine cualquier burbuja de aire visible con un ultrasonido.
- Monte un capilar de vidrio de sílice fundido (diámetro interior [ID]: 50 μm, diámetro exterior [OD]: 360 μm) de aproximadamente 85 cm de longitud en un instrumento CE. Enjuague el capilar infundiendo continuamente un disolvente orgánico, como la acetona13, utilizando el muestreador automático de CE a una presión de infusión de 40 psi durante 10-15 min.
- Llene el capilar limpio con solución de HPC con el muestreador automático a una presión de infusión de 40 psi (que a menudo toma ~ 40 min). Infundir aire en el capilar lleno de HPC a 40 psi para garantizar el flujo de aire libre en el capilar, indicado por las burbujas de aire expulsadas del capilar al sumergirse en agua.
- Hornea el capilar recubierto de HPC en un horno programable a temperatura (idealmente el horno de columna con temperatura controlada de un cromatógrafo de gases programado a temperatura) con gas nitrógeno (25 psi) que fluye a través del capilar, siguiendo el programa de temperatura que se muestra en la Figura 1.
- Enfríe el horno a temperatura ambiente antes de sacar el capilar. Utilice este capilar modificado por HPC para la separación CE.
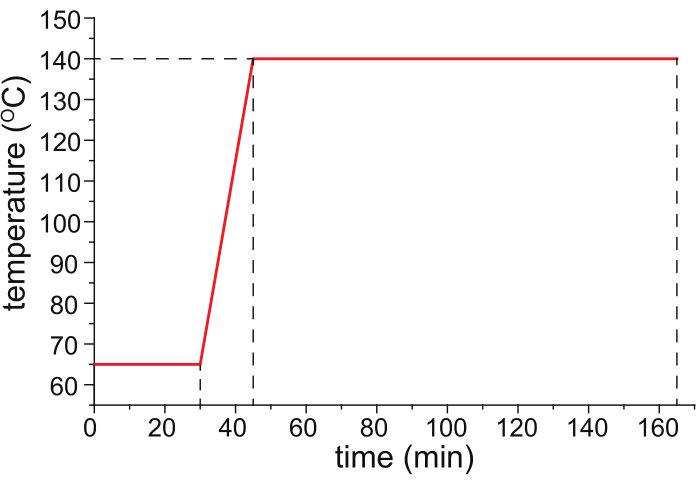
Figura 1: Un programa de temperatura recomendado para la cocción capilar. Haga clic aquí para ver una versión más grande de esta figura.
- Solución de electrolito de fondo (BGE) y solución modificadora8
- Preparar 1-10 ml de BGE a la concentración deseada (por ejemplo, 10 mM) disolviendo la cantidad adecuada de acetato de amonio en 2 H2O. Coloque 200μL-alícuotas de BGE en viales de BGE separados y selle los viales con parafilm para minimizar la reacción HDX entre BGE y el vapor de agua en el aire.
- Preparar 10 ml de una solución modificadora con 75% (v/v) de metanol y 25% (v/v) de agua, con pH ajustado a 2,5 utilizando ácido fórmico.
NOTA: Utilice 2H2O y metanol deuterado para preparar la solución modificadora si los átomos de deuterio en las cadenas laterales y las amidas de la columna vertebral no protegidas deben conservarse para su detección por EM.
- Desalinización de muestras de proteínas
- Prepare una solución de acetato de amonio en agua no deuterada a la concentración deseada.
NOTA: Se recomienda una concentración inferior a 100 mM para evitar una corriente eléctrica alta durante la electroforesis y el efecto de calentamiento de Joule resultante. - Cuando sea necesario, ajuste el pH de la solución de acetato de amonio al nivel deseado utilizando ácido fórmico (para pH < 6.8) o hidróxido de amonio (para pH > 6.8).
- Sustituya los tampones originales de la solución proteica por una solución de acetato de amonio (preparada en agua no deuterada a la concentración deseada; pH ajustado a 7,5 con hidróxido de amonio) a través de al menos cinco concentraciones secuenciales y pasos de dilución a 4 °C utilizando un filtro centrífugo con un corte de MW adecuado.
NOTA: Las muestras de proteínas que se van a desalinizar pueden ser de procedimientos de producción previos (por ejemplo, purificación o formulación) o preparadas disolviendo el polvo liofilizado de proteína. Las "sales" que se eliminarán de las soluciones de muestra en este paso se refieren en general a todos los iones o moléculas pequeñas que no son volátiles. Aunque estas especies pueden separarse eficientemente de las proteínas durante el proceso de electroforesis, se recomienda este paso para evitar comprometer la resolución electroforética y minimizar así la contaminación del espectrómetro de masas. Cuando los analitos de proteínas deban estabilizarse con sales o aditivos específicos, inclúyalos en el BGE. - Determine la concentración de proteínas utilizando un espectrofotómetro UV-Vis de microvolumen.
- Prepare una solución de acetato de amonio en agua no deuterada a la concentración deseada.
2. Funcionamiento del análisis HDX MS basado en CE
NOTA: El espectrómetro de masas utilizado en este enfoque debe estar equipado con un analizador de masas con resolución ultra alta, como una resonancia de ciclotrón de iones de transformada de Fourier (FTICR) u orbitrap, un filtro de masas, como un cuadrupolo que permite la selección de masa de iones precursores para la fragmentación, y disociación por transferencia de electrones (ETD) o disociación de captura de electrones (ECD) funciones para realizar análisis de arriba hacia abajo con datos confiables de MS en tándem (idealmente señales resueltas isotópicamente de iones de fragmentos).
- Optimización de la configuración de CE y MS
- Realice una medición piloto de MS utilizando una fuente estándar de ionización por electropulverización (ESI) rociando la muestra precargada de un capilar de vidrio de borosilicato recubierto de metal (el esquema nanoESI "estático") o la muestra infundida continuamente de un emisor de metal para optimizar la configuración de MS para la medición de proteínas intactas (MS1) y sus fragmentos de fase gaseosa (MS2). Fragmentar las especies proteicas de interés mediante la selección masiva del conjunto de sus iones en un solo estado de carga, seguido de ETD o ECD de los iones precursores.
NOTA: Los ajustes esenciales incluyen los parámetros que afectan la desolvación, la selección masiva de iones precursores (para evitar la interferencia de otras especies) y la eficiencia de fragmentación. Tanto el centro como el ancho de la ventana de selección de masa deben aumentarse para que coincidan con la distribución de masa resultante de los iones de analito después de HDX. Debido a que la ventana de elución de una especie de proteína en CE generalmente varía de 0,5 min a 2 min, evalúe la eficiencia de fragmentación basada en escaneos MS2 acumulados durante una ventana de tiempo comparable. Los valores óptimos de estos parámetros son específicos de la proteína; se remite a los lectores a informes publicados anteriormente para configuraciones ejemplares 8,14. - Realice una medición CE piloto utilizando un instrumento CE equipado con un detector óptico, es decir, un detector de matriz de fotodiodos (PDA) o un detector UV para optimizar la configuración CE para la separación de las especies de proteínas y los tiempos de migración, lo que equivale a los tiempos de reacción HDX.
NOTA: Este paso es opcional dependiendo de la disponibilidad del detector óptico de CE. En ausencia de un detector óptico, la configuración CE se puede optimizar utilizando CE-MS al completar la sección 2.2, siguiendo las instrucciones descritas en la sección 2.3. Los ajustes esenciales incluyen parámetros que afectan la eficiencia de separación, formas de pico que se muestran en electroferogramas y tiempos de elución.
- Realice una medición piloto de MS utilizando una fuente estándar de ionización por electropulverización (ESI) rociando la muestra precargada de un capilar de vidrio de borosilicato recubierto de metal (el esquema nanoESI "estático") o la muestra infundida continuamente de un emisor de metal para optimizar la configuración de MS para la medición de proteínas intactas (MS1) y sus fragmentos de fase gaseosa (MS2). Fragmentar las especies proteicas de interés mediante la selección masiva del conjunto de sus iones en un solo estado de carga, seguido de ETD o ECD de los iones precursores.
- Preacondicionamiento de la configuración CE-HDX
- Limpie la interfaz CE-MS microvial de flujo continuo con una mezcla de 50% de metanol, 49% de agua y 1% de ácido fórmico (v / v) utilizando ultrasonido durante al menos 30 minutos a temperatura ambiente.
- Tras el montaje del capilar modificado con HPC en un instrumento CE, enjuague el capilar con BGE con el muestreador automático durante 10 minutos y deje el capilar lleno de BGE.
- Obtener una longitud adecuada de tubo capilar de sílice fundido sin modificar (ID: 50 μm, OD: 360 μm) como tubo de infusión para la solución modificadora. Conecte el tubo modificador a una jeringa de vidrio hermética al gas con una punta roma con una unión y una funda adecuada, y enjuague el tubo con la solución modificadora con una bomba de infusión durante al menos 10 minutos.
- Inserte las salidas de los tubos capilares y modificadores CE recubiertos de HPC, que se han cargado con las soluciones correspondientes, en la interfaz CE-MS limpia, como se ilustra en la Figura 2.
- Avance la jeringa para la infusión modificadora manualmente o con la bomba de infusión para asegurarse de que la solución modificadora llegue a la punta de la interfaz. Monte la interfaz CE-MS ensamblada en una carcasa de fuente nanoESI de un espectrómetro de masas.
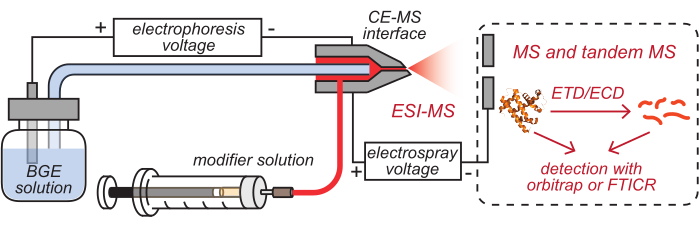
Figura 2: Ilustración esquemática de la configuración de HDX MS basada en CE. Esta cifra ha sido modificada de8. Abreviaturas: BGE = solución de electrolito de fondo; CE = electroforesis capilar; MS = espectrometría de masas; HDX = intercambio hidrógeno/deuterio; ESI = ionización por electropulverización; FTICR = resonancia ciclotrón iónica de transformada de Fourier; ETD = disociación por transferencia de electrones; ECD = disociación por captura de electrones. Haga clic aquí para ver una versión más grande de esta figura.
- Separación CE simultánea, reacción HDX y análisis MS
NOTA: Se recomienda usar BGE deuterado dentro de 1 día después de desprecintar.- Aplique una tensión de pulverización de 3-5 kV a la interfaz CE-MS.
- Comience a infundir la solución modificadora con la bomba de infusión a un caudal que oscila entre 0,1 y 10 μL/min, y asegure un electropulverizador estable en la punta de la interfaz CE-MS.
- Coloque el vial de muestra que contiene el BGE en el muestreador automático y utilícelo en el paso 2.3.4 para adquirir electroferogramas en blanco y espectros de masas en blanco.
- Inyecte la solución de muestra utilizando el muestreador automático a 2 psi y durante una duración adecuada para permitir la inyección de una cantidad deseada de la muestra. Estimar el volumen de inyección utilizando la relación entre el volumen de inyección y los parámetros de inyección15 definidos por la ecuación (1).
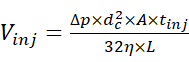 (1)
(1)
Donde Vinj es el volumen de inyección, Δp es la presión de inyección, dc es el diámetro interno del capilar, A es la sección transversal del capilar, tinj es la duración de la inyección, η es la viscosidad del líquido en el capilar y L es la longitud del capilar. - Iniciar la separación CE aplicando una tensión electroforética de 30 kV y una presión de infusión que oscila entre 0 y 2 psi, y adquirir el electroferograma. Mientras tanto, comience la adquisición de los datos de MS en el modo cromatográfico donde el gráfico de corriente iónica se adquiere en función del tiempo, y los escaneos de MS correspondientes no se combinan automáticamente en un solo espectro.
NOTA: Las proteínas experimentan una reacción espontánea de HDX en el punto de contacto de 2moléculas de H2O en BGE durante su migración electroforética en este paso. La detección óptica para CE se puede utilizar además de la detección de EM. Como la detección en columna requiere la eliminación de una cierta longitud de recubrimiento de poliimida en el extremo de salida del capilar de sílice fundido, se debe tener cuidado adicional para evitar daños capilares durante el montaje de la interfaz CE-MS. - Guarde el electroferograma en blanco y los espectros de masas como referencias.
NOTA: Los datos en blanco se deben utilizar para la solución de problemas en lugar de la resta de línea base. - Coloque los viales de muestra que contienen las concentraciones deseadas de las soluciones de muestra de proteína en el muestreador automático. Adquirir los electroferogramas y espectros de masas para las muestras de proteínas siguiendo los pasos 2.3.4-2.3.5. Recolectar un número adecuado de exploraciones de EM para obtener espectros MS1 de las especies de proteínas separadas electroforéticamente y marcadas con H.
- Realice mediciones de MS en tándem para las especies de interés, ya sea después de adquirir los espectros MS1 dentro de la misma ejecución o en una ejecución posterior y separada.
- Cuando sea necesario, ajuste los tiempos de migración/tiempos de reacción HDX cambiando la presión de infusión o la longitud del capilar CE. Si el tiempo de reacción de HDX debe ser más corto que el tiempo de migración, utilice el enfoque descrito anteriormente8, que emplea BGE deuterado y no deuterado en el capilar durante el proceso CE.
- Enjuague el capilar CE con BGE a una presión de 20 psi durante al menos 10 minutos después de cada medición.
- Una vez finalizados los experimentos, limpie la interfaz CE-MS y todos los tubos para el almacenamiento.
- Adquirir un conjunto de datos de la muestra HDX "endpoint" (que se puede preparar utilizando los enfoques descritos anteriormente 6,16) con EM en modo de infusión directa.
NOTA: Este paso solo es necesario cuando se utiliza una solución modificadora deuterada para HDX basado en CE.
3. Análisis de datos
- Análisis de datos de CE
- Utilice una de las siguientes gráficas como electroferograma para determinar las características electroforéticas, incluido el número de picos, los tiempos de migración y la eficiencia de separación: (a) absorbancia UV frente al tiempo de migración, adquirido por el detector óptico del instrumento CE (cuando esté disponible); b) el gráfico de corriente iónica total (TIC) adquirido por MS; c) el gráfico de corriente iónica extraído (EIC/XIC) adquirido por MS.
NOTA: EIC/XIC proporciona la óptima relación señal/ruido (S/N), en general, entre los formatos de electroferogramas antes mencionados. Cabe destacar que incluso en ausencia de sesgos instrumentales, mientras que la absorbancia UV es proporcional a la concentración de masa de la proteína, la señal MS es proporcional a la concentración molar. Por lo tanto, es razonable observar diferencias en los patrones de pico entre los electroferogramas derivados de CE y MS. - Utilice el área bajo la curva (AUC) de los picos mostrados en los electroferogramas para la semicuantización. Para muestras que involucran complejos de proteínas, utilice el enfoque descrito anteriormente17 para deducir los datos de concentración de masa de los electroferogramas TIC/EIC.
- Utilice una de las siguientes gráficas como electroferograma para determinar las características electroforéticas, incluido el número de picos, los tiempos de migración y la eficiencia de separación: (a) absorbancia UV frente al tiempo de migración, adquirido por el detector óptico del instrumento CE (cuando esté disponible); b) el gráfico de corriente iónica total (TIC) adquirido por MS; c) el gráfico de corriente iónica extraído (EIC/XIC) adquirido por MS.
- Análisis de datos de EM
- Obtenga los espectros MS1 y MS2 combinando los escaneos MS1 y MS2 adquiridos dentro de las ventanas de elución correspondientes, respectivamente.
- Determinar las masas de la proteína intacta (M (proteína intacta)) y los fragmentos por cualquiera de los dos métodos siguientes.
- Calcular las masas medias de los iones que dan lugar a los cúmulos de señales resueltos isotópicamente.
- Utilice el centro de las curvas de tipo gaussiano resultantes del ajuste de las envolventes isotópicas correspondientes6.
- Utilice software como Biopharma Finder, ProSight18 o MASH Suite19 para generar la lista de masas de los iones fragmentos e identificarlos.
- Análisis de datos HDX
- Determinar el nivel general de deuteración de una especie de proteína intacta utilizando la ecuación (2).
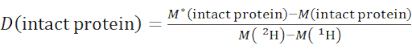 (2)
(2)
donde M (2H) o M (1H) son pesos atómicos de 2H o 1H. El asterisco indica los datos de la muestra con 2H-labeled. - Determinar la protección acumulativa o la deuteración acumulativa de las amidas troncales de un segmento específico.
- Para los datos adquiridos con una solución modificadora deuterada, utilice las ecuaciones (3) y (4) para determinar el nivel de protección acumulativo.
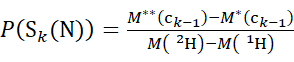 (3)
(3)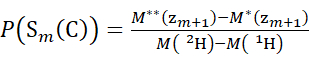 (4)
(4)
Donde P(Sk(N)) es la protección total del segmento N-terminal que abarca los residuos 1 a k, P(Sm(C)) es la protección total del segmento C-terminal que comprende m residuos, M (2H) o M (1H) son pesos atómicos de 2H o 1H, y M (ci) o M (zi) son los pesos moleculares de los iones ci o zi .
NOTA: El doble asterisco indica los datos de la muestra de "punto final" de HDX. - Para los datos adquiridos con una solución modificadora no deuterada, utilice las ecuaciones (5) y (6) para determinar el nivel de deuteración acumulativa.
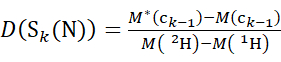 (5)
(5)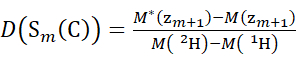 (6)
(6)
Donde D(Sk(N)) es la absorción acumulativa de deuterio del segmento N-terminal que abarca los residuos 1 a k; D(Sm(C)) es la absorción acumulativa de deuterio del segmento C-terminal que comprende m residuos.
- Para los datos adquiridos con una solución modificadora deuterada, utilice las ecuaciones (3) y (4) para determinar el nivel de protección acumulativo.
- Determinar el nivel de deuteración en un grupo amida de la columna vertebral local
- Para los datos adquiridos con una solución modificadora deuterada, utilice las ecuaciones (7), (8), (9), (10) y (11) para determinar el nivel de protección local.
para los datos deducidos a partir de iones C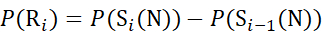 (7)
(7)
para los datos deducidos a partir de iones Z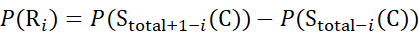 (8)
(8)
Donde P(Ri) es la protección de una amida de la columna vertebral en el residuo i, y el subíndice "total" denota el número total de residuos de la proteína.
Para los sitios de residuos donde faltaban iones de fragmentos posteriores, asigne P(Ri) utilizando las ecuaciones (9) y (10).
para los datos deducidos a partir de iones C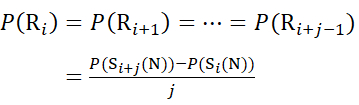 (9)
(9)
para los datos deducidos a partir de iones Z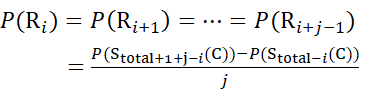 (10)
(10)
Luego, determine el nivel de deuteración D(Ri) en un grupo amida de la columna vertebral local usando la ecuación (11).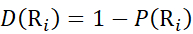 (11)
(11) - Para los datos adquiridos con una solución modificadora no deuterada, utilice las ecuaciones (12), (13), (14) y (15) para determinar el nivel de protección local.
para los datos deducidos a partir de iones C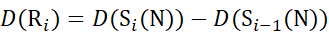 (12)
(12)
para los datos deducidos a partir de iones Z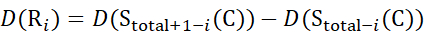 (13)
(13)
Donde D(Ri) es la protección de una amida de la columna vertebral en el residuo i, y el subíndice "total" denota el número total de residuos de la proteína.
Para los sitios de residuos donde faltaban iones de fragmentos posteriores, asigne D(Ri) usando las ecuaciones (14) y (15).
para los datos deducidos a partir de iones C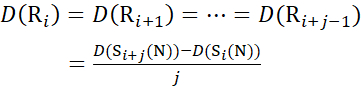 (14)
(14)
para los datos deducidos a partir de iones Z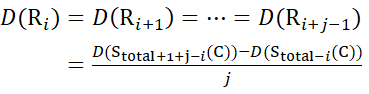 (15)
(15)
- Para los datos adquiridos con una solución modificadora deuterada, utilice las ecuaciones (7), (8), (9), (10) y (11) para determinar el nivel de protección local.
- Determinar el nivel general de deuteración de una especie de proteína intacta utilizando la ecuación (2).
Resultados
El cambio de la presión de infusión de BGE permite ajustar tanto la eficiencia de separación como el tiempo de migración, lo que equivale al tiempo de reacción HDX de las proteínas a separar (Figura 3). Una presión de infusión más baja da como resultado una mejor separación de los picos de CE a costa de la duración del experimento (Figura 3A). Un tiempo de reacción de migración/HDX más largo da como resultado un mayor nivel de deuteración de los a...
Discusión
Los objetivos del recubrimiento de la pared interna del capilar CE incluyen la minimización del flujo electroosmótico y la absorción de proteínas durante el proceso CE13. Aunque el flujo electroosmótico es beneficioso para el análisis CE convencional de moléculas pequeñas debido a su capacidad de conducir especies neutras o con carga opuesta al detector, compromete la eficiencia de separación de especies de proteínas con tamaños similares y cargas netas en solución. El recubrimiento de...
Divulgaciones
D. D. Y. Chen es uno de los fundadores del Instituto de Biomoléculas Knowledge for Health, que está comercializando la interfaz CE-MS microvial de flujo continuo. Otros autores no tienen nada que revelar.
Agradecimientos
Este trabajo fue apoyado por subvenciones de la Fundación Nacional de Ciencias Naturales de China (NSFC 21974069). Los autores también recibieron apoyo del Instituto de Análisis Celular del Laboratorio de la Bahía de Shenzhen, China; Jiangsu Collaborative Innovation Center of Biomedical Functional Materials; y jiangsu Key Laboratory of Biomedical Materials en la Universidad Normal de Nanjing, China.
Materiales
| Name | Company | Catalog Number | Comments |
| ammonium acetate | Fisher Chemical | A/3446/50 | ≥99% |
| CESI 8000 plus capillary electrophoresis system | Sciex, USA | ||
| centrifuge | Eppendorf | 5406000097 | |
| centrifugal filter | Merck | UFC201024 | 10 kDa cutoff |
| deuterium oxide | Energy Chemical | E090001 | 99.9 % D |
| formic acid | Acros Organics | 270480250 | |
| fused silica glass capillary | Polymicro Technologies | 1068150017 | ID 50μm, OD 360μm |
| gas chromatography | Agilent | GC6890N | |
| hydrochloric acid | Sigma Aldrich | 258148 | |
| hydroxypropyl cellulose | Aladdin | H113415 | MW 100000 |
| magnetic stirrers | DLAB | 8030101212 | |
| methanol | Fisher Chemical | A456-4 | MS grade |
| microvolume UV-Vis spectrophotometer | DeNovix | 84677JK7731 | |
| myoglobin | Sigma Aldrich | M1882 | |
| Orbitrap Fusion Lumos mass spectrometer | Thermo Fisher Scientific, USA | ||
| PA 800 Plus Pharmaceutical Analysis CE System | Beckman Coulter, USA | ||
| Q Exactive UHMR mass Spectrometer | Thermo Fisher Scientific, Germany | ||
| sodium hydroxide | Sigma Aldrich | S5881 | |
| ubiquitin | Sigma Aldrich | U6253 | |
| ultrasonicator | SCIENTZ | SB-5200 | |
| β-lactoglobulin | Sigma Aldrich | L0130 | |
Referencias
- Kaltashov, I. A., Bobst, C. E., Pawlowski, J., Wang, G. Mass spectrometry-based methods in characterization of the higher order structure of protein therapeutics. Journal of Pharmaceutical and Biomedical Analysis. 184, 113169 (2020).
- Engen, J. R., Botzanowski, T., Peterle, D., Georgescauld, F., Wales, T. E. Developments in hydrogen/deuterium exchange mass spectrometry. Analytical Chemistry. 93 (1), 567-582 (2021).
- Pan, J., Han, J., Borchers, C. H., Konermann, L. Conformer-specific hydrogen exchange analysis of Abeta(1-42) oligomers by top-down electron capture dissociation mass spectrometry. Analytical Chemistry. 83 (13), 5386-5393 (2011).
- Pan, J., Han, J., Borchers, C. H., Konermann, L. Structure and dynamics of small soluble Abeta(1-40) oligomers studied by top-down hydrogen exchange mass spectrometry. Biochemistry. 51 (17), 3694-3703 (2012).
- Pan, J., Borchers, C. H. Top-down structural analysis of posttranslationally modified proteins by Fourier transform ion cyclotron resonance-MS with hydrogen/deuterium exchange and electron capture dissociation. Proteomics. 13 (6), 974-981 (2013).
- Wang, G., Abzalimov, R. R., Bobst, C. E., Kaltashov, I. A. Conformer-specific characterization of nonnative protein states using hydrogen exchange and top-down mass spectrometry. Proceedings of the National Academy of Sciences of the United. States of America. 110 (50), 20087-20092 (2013).
- Mironov, G. G., Clouthier, C. M., Akbar, A., Keillor, J. W., Berezovski, M. V. Simultaneous analysis of enzyme structure and activity by kinetic capillary electrophoresis-MS. Nature Chemical Biology. 12 (11), 918-922 (2016).
- Shen, Y., Zhao, X., Wang, G., Chen, D. D. Y. Differential hydrogen/deuterium exchange during proteoform separation enables characterization of conformational differences between coexisting protein states. Analytical Chemistry. 91 (6), 3805-3809 (2019).
- Kontopidis, G., Holt, C., Sawyer, L. Invited review: β-lactoglobulin: binding properties, structure, and function. Journal of Dairy Science. 87 (4), 785-796 (2004).
- Qin, B. Y., et al. Structural basis of the Tanford transition of bovine β-lactoglobulin. Biochemistry. 37 (40), 14014-14023 (1998).
- Qin, B. Y., Bewley, M. C., Creamer, L. K., Baker, E. N., Jameson, G. B. Functional implications of structural differences between variants A and B of bovine beta-lactoglobulin. Protein Science. 8 (1), 75-83 (1999).
- Wang, L., et al. High resolution capillary isoelectric focusing mass spectrometry analysis of peptides, proteins, and monoclonal antibodies with a flow-through microvial interface. Analytical Chemistry. 90 (15), 9495-9503 (2018).
- Busch, M. H. A., Kraak, J. C., Poppe, H. Cellulose acetate-coated fused-silica capillaries for the separation of proteins by capillary zone electrophoresis. Journal of Chromatography A. 1695 (2), 287-296 (1995).
- Zhao, X., Shen, Y., Tong, W., Wang, G., Chen, D. D. Y. Deducing disulfide patterns of cysteine-rich proteins using signature fragments produced by top-down mass spectrometry. Analyst. 143 (4), 817-823 (2018).
- Sutera, S. P., Skalak, R. The history of Poiseuille's law. Annual Review of Fluid Mechanics. 25 (1), 1-20 (1993).
- Wang, G., Kaltashov, I. A. Approach to characterization of the higher order structure of disulfide-containing proteins using hydrogen/deuterium exchange and top-down mass spectrometry. Analytical Chemistry. 86 (15), 7293-7298 (2014).
- Wang, G., Johnson, A. J., Kaltashov, I. A. Evaluation of electrospray ionization mass spectrometry as a tool for characterization of small soluble protein aggregates. Analytical Chemistry. 84 (3), 1718-1724 (2012).
- Fellers, R. T., et al. ProSight Lite: graphical software to analyze top-down mass spectrometry data. Proteomics. 15 (7), 1235-1238 (2015).
- Cai, W., et al. MASH Suite Pro: A comprehensive software tool for top-down proteomics. Molecular & Cellular Proteomics. 15 (2), 703-714 (2016).
- Paterson, G. R., Hill, J. P., Otter, D. E. Separation of β-lactoglobulin A, B and C variants of bovine whey using capillary zone electrophoresis. Journal of Chromatography A. 700 (1), 105-110 (1995).
- Wang, G., Kaltashov, I. A. Approach to characterization of the higher order structure of disulfide-containing proteins using hydrogen/deuterium exchange and top-down mass spectrometry. Analytical Chemistry. 86 (15), 7293-7298 (2014).
- Nicolardi, S., et al. On-line electrochemical reduction of disulfide bonds: improved FTICR-CID and -ETD coverage of oxytocin and hepcidin. Journal of the American Society for Mass Spectrometry. 24 (12), 1980-1987 (2013).
- Adhikari, S., Xia, Y., McLuckey, S. A. Top-down analysis of disulfide-linked proteins using photoinduced radical reactions and ET-DDC. International Journal of Mass Spectrometry. 444, 116173 (2019).
- Rush, M. J. P., Riley, N. M., Westphall, M. S., Coon, J. J. Top-down characterization of proteins with intact disulfide bonds using activated-ion electron transfer dissociation. Analytical Chemistry. 90 (15), 8946-8953 (2018).
- Zhong, X., Maxwell, E. J., Chen, D. D. Y. Mass transport in a micro flow-through vial of a junction-at-the-tip capillary electrophoresis-mass spectrometry interface. Analytical Chemistry. 83 (12), 4916-4923 (2011).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados