Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Methods Article
Échange hydrogène/deutérium basé sur l’électrophorèse capillaire pour la caractérisation conformationnelle des protéines avec spectrométrie de masse descendante
Dans cet article
Résumé
Présenté ici est un protocole pour une approche d’échange hydrogène/deutérium (HDX) basée sur l’électrophorèse capillaire couplée à une spectrométrie de masse descendante. Cette approche caractérise la différence dans les structures d’ordre supérieur entre différentes espèces de protéines, y compris les protéines dans différents états et différentes protéoformes, en effectuant une séparation différentielle simultanée HDX et électrophorétique.
Résumé
La résolution de l’hétérogénéité conformationnelle de plusieurs états protéiques qui coexistent en solution reste l’un des principaux obstacles à la caractérisation des thérapies protéiques et à la détermination des voies de transition conformationnelle essentielles aux fonctions biologiques, allant de la reconnaissance moléculaire à la catalyse enzymatique. La réaction d’échange hydrogène/deutérium (HDX) couplée à une analyse par spectrométrie de masse (MS) descendante fournit un moyen de caractériser les structures et la dynamique d’ordre supérieur des protéines d’une manière spécifique au conformateur. Le pouvoir de résolution conformationnelle de cette technique dépend fortement de l’efficacité de la séparation des états protéiques au niveau des protéines intactes et de la minimisation de la teneur protique résiduelle non deutérée pendant les réactions HDX.
Nous décrivons ici une variante basée sur l’électrophorèse capillaire (CE) de l’approche HDX MS qui vise à améliorer la résolution conformationnelle. Dans cette approche, les protéines subissent des réactions HDX tout en migrant à travers une solution d’électrolyte de fond deutérée (BGE) pendant la séparation électrophorétique capillaire. Différents états protéiques ou protéoformes qui coexistent en solution peuvent être séparés efficacement en fonction de leurs différents rapports charge/taille. La différence de mobilité électrophorétique entre les protéines et les molécules de solvant protique minimise le solvant résiduel non deutéré, ce qui entraîne un environnement de deutération presque complet pendant le processus HDX. L’interface microviale CE-MS à écoulement permet une ionisation par électropulvérisation efficace des espèces protéiques éluées après un mélange rapide avec la solution modificateur de trempe et de dénaturation à la sortie du pulvérisateur. L’analyse descendante en ligne de la SEP mesure le niveau global de deutération des espèces protéiques intactes éluées et, par la suite, la deutération de leurs fragments en phase gazeuse. Cet article démontre cette approche dans le HDX différentiel pour les systèmes, y compris les variantes protéiques naturelles coexistant dans le lait.
Introduction
Il est important de distinguer les espèces protéiques dans différents états conformationnels, de liaison ou de modification et de caractériser leurs différences structurelles pour surveiller les voies de transition entre ces espèces impliquées dans des événements biologiques, allant de la reconnaissance moléculaire à la catalyse enzymatique, et comprendre les mécanismes sous-jacents à ces événements. Les techniques biophysiques conventionnelles ne fournissent pas une solution complète en raison des limites telles que la résolution insuffisante et la perte d’informations dynamiques dans la solution. L’échange hydrogène/deutérium couplé à la spectrométrie de masse (HDX MS) est une technique qui marque les caractéristiques structurelles et conformationnelles des protéines avec le deutérium (2H) via l’échange entre les atomes d’hydrogène labile des protéines et 2H de la solution 2H2Odélibérément introduite. Les protons impliqués dans la liaison hydrogène ou qui sont séquestrés du solvant à l’intérieur de la protéine ne s’échangent pas facilement1. Ainsi, comme le taux de change sur un site échangeable dépend fortement de son implication dans des structures d’ordre supérieur, les structures protéiques peuvent être révélées à haute résolution spatiale par MS qui sonde l’étendue et le taux d’absorption de 2H en fonction des différentes masses atomiques entre 1H et 2H. Au cours des dernières décennies, HDX MS est devenu une technique remarquablement réussie pour étudier les conformations et la dynamique des protéines2.
Dans l’approche ascendante classique de HDX MS, l’ensemble des espèces protéiques dans différents états de conformation, de liaison ou de modification est protéolysé sans séparation au niveau des protéines intactes, ce qui rend impossible de caractériser des espèces individuelles en analysant les fragments protéolytiques résultants avec des teneurs en deutérium alambiquées. En revanche, dans l’approche descendante, différents états protéiques ou protéoformes qui ont incorporé différentes teneurs en deutérium donnent lieu à de multiples distributions de masses protéiques intactes dans un scan MS. Cela permet de séparer les espèces individuelles par la sélection de masse d’ions correspondant à chaque distribution de masse à l’aide d’un filtre de masse approprié (tel qu’un quadripolaire) et la caractérisation de leurs différences conformationnelles dans l’analyse ultérieure en tandem MS 3,4,5,6. Cependant, l’efficacité de la séparation des états protéiques ou des protéoformes dans cette stratégie est limitée par l’ampleur de la différence dans leurs distributions de masse correspondantes.
L’électrophorèse capillaire (EC) fournit un moyen de séparer les espèces de protéines en fonction de leurs différentes charges et tailles hydrodynamiques dans la phase de solution avec une efficacité élevée7. La combinaison de CE avec HDX offre une séparation supplémentaire des états protéiques ou protéoformes dans la phase de solution. De plus, le petit volume du capillaire CE permet l’utilisation d’une solution entièrement deutérée comme solution d’électrolyte de fond (BGE), c’est-à-dire le tampon de fonctionnement, rendant le capillaire comme un réacteur HDX pour les échantillons de protéines. En raison de la différence de mobilité électrophorétique entre les protéines et les réactifs protiques dans le processus d’électrophorèse, la conduite de HDX pendant l’EC entraîne un environnement de deutération presque complet pour les analytes protéiques avec un contenu résiduel non deutéré minimal, améliorant ainsi la sensibilité de l’analyse structurelle à l’aide de données HDX. En tant que tel, nous avons développé une approche HDX différentielle basée sur CE couplée à une SEP descendante pour caractériser les structures d’ordre supérieur des protéines d’une manière spécifique à l’état ou à la protéoforme8.
Cet article décrit les protocoles de cette approche en détaillant les étapes de la préparation du matériel, de la procédure expérimentale et de l’analyse des données. Les facteurs susceptibles d’affecter les performances de la méthode ou la qualité des données sont énumérés dans de brèves notes. Les résultats représentatifs présentés ici comprennent des données HDX différentielles sur les mélanges de différentes protéines et variantes naturelles de la β-lactoglobuline bovine (β-lg), la principale protéine de lactosérum présente dans le lait9. Nous démontrons l’efficacité de séparation, la reproductibilité et les performances d’étiquetage 2H des deux variantes abondantes de β-lg, c’est-à-dire A et B10,11 lors du HDX basé sur CE et de la caractérisation spécifique à la variante de leurs conformations.
Protocole
REMARQUE : Utilisez des réactifs de qualité chromatographique liquide à haute performance (CLHP) ou de qualité MS dans la mesure du possible afin de minimiser les contaminants susceptibles d’interférer avec l’analyse de la SEP. Ne touchez pas l’interface CE-MS à mains nues pendant la mesure pour éviter la possibilité d’un choc électrique causé par la tension électrophorétique ou la tension d’électropulvérisation.
1. Préparation du matériel
- Modification du capillaire de silice fondue pour CE
- Préparer une solution d’hydroxypropylcellulose (HPC) à 5 % (p/p) en dissolvant la poudre de HPC (poids moléculaire [MW] : 100 kDa) dans de l’eau en remuant continuellement à température ambiante sur un agitateur magnétique pendant ~12 h ou jusqu’à disparition complète des particules solides12. Retirez toutes les bulles d’air visibles avec un ultrasonateur.
- Monter un capillaire en verre de silice fondu (diamètre intérieur [ID]: 50 μm, diamètre extérieur [OD]: 360 μm) d’environ 85 cm de longueur dans un instrument CE. Rincez le capillaire en infusant continuellement un solvant organique, tel que l’acétone13, à l’aide de l’échantillonneur automatique de CE à une pression de perfusion de 40 psi pendant 10-15 min.
- Remplissez le capillaire nettoyé avec une solution HPC à l’aide de l’échantillonneur automatique à une pression de perfusion de 40 psi (ce qui prend souvent environ 40 minutes). Infuser de l’air dans le capillaire rempli de HPC à 40 psi pour assurer une circulation d’air libre dans le capillaire, indiquée par les bulles d’air éjectées du capillaire lors de l’immersion dans l’eau.
- Cuire le capillaire revêtu de HPC dans un four programmable en température (idéalement le four à colonne à température contrôlée d’un chromatographe en phase gazeuse programmé en température) avec de l’azote gazeux (25 psi) circulant à travers le capillaire, en suivant le programme de température illustré à la figure 1.
- Refroidir le four à température ambiante avant de retirer le capillaire. Utilisez ce capillaire modifié par HPC pour la séparation CE.
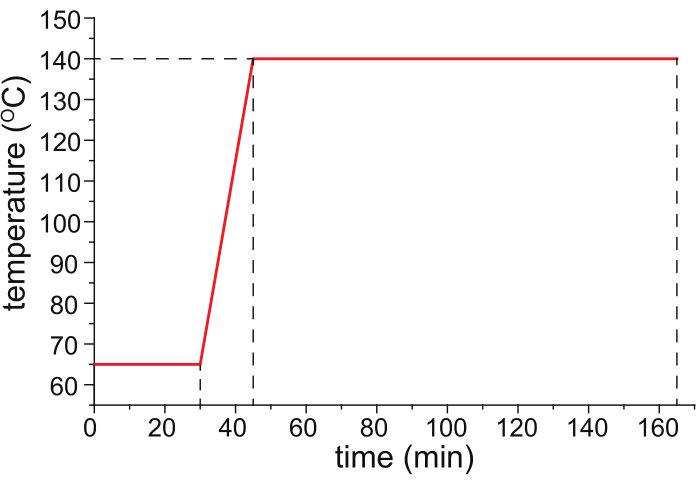
Figure 1 : Programme de température recommandée pour la cuisson capillaire. Veuillez cliquer ici pour l’agrandir.
- Solution d’électrolyte de fond (BGE) et solution modificateur8
- Préparer 1 à 10 mL de BGE à la concentration souhaitée (p. ex., 10 mM) en dissolvant la quantité appropriée d’acétate d’ammonium dans 2H2O. Placer 200 aliquotes de BGE dans des flacons de BGE séparés et sceller les flacons avec un parafilm pour minimiser la réaction HDX entre le BGE et la vapeur d’eau dans l’air.
- Préparer 10 mL d’une solution modificateur avec 75 % (v/v) de méthanol et 25 % (v/v) d’eau, avec un pH ajusté à 2,5 % à l’aide d’acide formique.
REMARQUE: Utilisez 2H2O et du méthanol deutéré pour préparer la solution modificateur si les atomes de deutérium dans les chaînes latérales et les amides dorsaux non protégés doivent être conservés pour la détection par MS.
- Dessalage d’échantillons de protéines
- Préparer une solution d’acétate d’ammonium dans de l’eau non deutérée à la concentration souhaitée.
REMARQUE: Une concentration inférieure à 100 mM est recommandée pour éviter un courant électrique élevé pendant l’électrophorèse et l’effet de chauffage Joule qui en résulte. - Si nécessaire, ajuster le pH de la solution d’acétate d’ammonium au niveau souhaité en utilisant de l’acide formique (pour un pH < 6,8) ou de l’hydroxyde d’ammonium (pour un pH > 6,8).
- Remplacer les tampons d’origine de la solution protéique par une solution d’acétate d’ammonium (préparée dans de l’eau non deutérée à la concentration souhaitée; pH ajusté à 7,5 avec de l’hydroxyde d’ammonium) par au moins cinq concentrations séquentielles et étapes de dilution à 4 °C à l’aide d’un filtre centrifuge avec une coupure de MW appropriée.
REMARQUE : Les échantillons de protéines à dessaler peuvent provenir de procédés de production antérieurs (p. ex., purification ou formulation) ou être préparés en dissolvant la poudre de protéine lyophilisée. Les « sels » à éliminer des solutions d’échantillons dans cette étape se réfèrent en général à tous les petits ions ou molécules qui ne sont pas volatils. Bien que ces espèces puissent être séparées efficacement des protéines pendant le processus d’électrophorèse, cette étape est recommandée pour éviter de compromettre la résolution électrophorétique et ainsi minimiser la contamination du spectromètre de masse. Lorsque les analytes protéiques doivent être stabilisés par des sels ou des additifs spécifiques, incluez-les dans le BGE. - Déterminez la concentration en protéines à l’aide d’un spectrophotomètre UV-Vis en microvolume.
- Préparer une solution d’acétate d’ammonium dans de l’eau non deutérée à la concentration souhaitée.
2. Fonctionnement de l’analyse HDX MS basée sur CE
REMARQUE: Le spectromètre de masse utilisé dans cette approche doit être équipé d’un analyseur de masse à ultra-haute résolution, tel qu’une résonance cyclotronionnelle à transformée de Fourier (FTICR) ou orbitrap, un filtre de masse, tel qu’un quadripolaire qui permet la sélection en masse d’ions précurseurs pour la fragmentation, et des fonctions de dissociation par transfert d’électrons (ETD) ou de dissociation par capture d’électrons (ECD) pour effectuer une analyse descendante avec des données MS tandem fiables (idéalement des signaux isotopiquement résolus d’ions fragments).
- Optimisation des paramètres CE et MS
- Effectuer une mesure pilote de la SEP à l’aide d’une source d’ionisation par électropulvérisation standard (ESI) en pulvérisant soit l’échantillon préchargé à partir d’un capillaire en verre borosilicate revêtu de métal (le schéma nanoESI « statique ») soit l’échantillon infusé en continu à partir d’un émetteur métallique afin d’optimiser les paramètres MS pour la mesure des protéines intactes (MS1) et de leurs fragments en phase gazeuse (MS2). Fragmenter les espèces protéiques d’intérêt par sélection en masse de l’ensemble de ses ions dans un seul état de charge, suivi de l’ETD ou de l’ECD des ions précurseurs.
REMARQUE: Les paramètres essentiels comprennent les paramètres qui affectent la désolvation, la sélection en masse des ions précurseurs (pour éviter les interférences d’autres espèces) et l’efficacité de la fragmentation. Le centre et la largeur de la fenêtre de sélection de masse doivent être augmentés pour correspondre à la distribution de masse résultante des ions analyte après HDX. Étant donné que la fenêtre d’élution d’une espèce protéique dans CE varie généralement de 0,5 min à 2 min, évaluez l’efficacité de la fragmentation en fonction des scans MS2 accumulés sur une fenêtre de temps comparable. Les valeurs optimales de ces paramètres sont spécifiques aux protéines; les lecteurs sont renvoyés aux rapports publiés précédemment pour des paramètres exemplaires 8,14. - Effectuer une mesure CE pilote à l’aide d’un instrument CE équipé d’un détecteur optique, c’est-à-dire un détecteur à réseau de photodiodes (PDA) ou un détecteur UV pour optimiser les paramètres CE pour la séparation des espèces de protéines et les temps de migration, ce qui équivaut aux temps de réaction HDX.
REMARQUE: Cette étape est facultative en fonction de la disponibilité du détecteur optique de CE. En l’absence d’un détecteur optique, les réglages CE peuvent être optimisés à l’aide de CE-MS à la fin de la section 2.2, en suivant les instructions décrites à la section 2.3. Les paramètres essentiels comprennent les paramètres qui affectent l’efficacité de séparation, les formes de crête indiquées dans les électrophérogrammes et les temps d’élution.
- Effectuer une mesure pilote de la SEP à l’aide d’une source d’ionisation par électropulvérisation standard (ESI) en pulvérisant soit l’échantillon préchargé à partir d’un capillaire en verre borosilicate revêtu de métal (le schéma nanoESI « statique ») soit l’échantillon infusé en continu à partir d’un émetteur métallique afin d’optimiser les paramètres MS pour la mesure des protéines intactes (MS1) et de leurs fragments en phase gazeuse (MS2). Fragmenter les espèces protéiques d’intérêt par sélection en masse de l’ensemble de ses ions dans un seul état de charge, suivi de l’ETD ou de l’ECD des ions précurseurs.
- Pré-conditionnement de la configuration CE-HDX
- Nettoyez l’interface CE-MS microviale à écoulement avec un mélange de 50% de méthanol, 49% d’eau et 1% d’acide formique (v / v) en utilisant les ultrasons pendant au moins 30 minutes à température ambiante.
- Lors du montage du capillaire modifié par HPC sur un instrument CE, rincez le capillaire avec BGE à l’aide de l’échantillonneur automatique pendant 10 minutes et laissez le capillaire rempli de BGE.
- Obtenir une longueur appropriée de tube capillaire en silice fondue non modifiée (ID: 50 μm, OD: 360 μm) comme tuyau de perfusion pour la solution modificateur. Connectez le tuyau modificateur à une seringue en verre étanche aux gaz avec une pointe émoussée à l’aide d’une union et d’un manchon approprié, et rincez le tube avec la solution modificateur à l’aide d’une pompe à perfusion pendant au moins 10 minutes.
- Insérez les sorties des tubes capillaires et modificateurs CE revêtus de HPC, qui ont été chargées avec les solutions correspondantes, dans l’interface CE-MS nettoyée, comme illustré à la figure 2.
- Avancez la seringue pour la perfusion de modificateur manuellement ou avec la pompe à perfusion pour vous assurer que la solution modificateur atteint l’extrémité de l’interface. Montez l’interface CE-MS assemblée sur un boîtier de source nanoESI d’un spectromètre de masse.
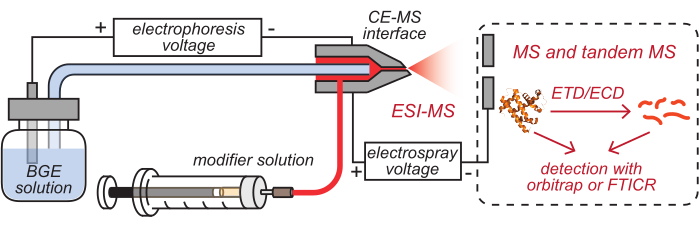
Figure 2 : Illustration schématique de la configuration HDX MS basée sur CE. Ce chiffre a été modifié par rapport à8. Abréviations: BGE = solution d’électrolyte de fond; CE = électrophorèse capillaire; MS = spectrométrie de masse; HDX = échange hydrogène/deutérium; ESI = ionisation par électropulvérisation; FTICR = résonance cyclotronionionnelle à transformée de Fourier; ETD = dissociation par transfert d’électrons; ECD = dissociation par capture d’électrons. Veuillez cliquer ici pour voir une version agrandie de cette figure.
- Séparation CE simultanée, réaction HDX et analyse MS
REMARQUE: Il est recommandé d’utiliser bgE deutéré dans les 1 jour suivant la descellement.- Appliquez une tension de pulvérisation de 3 à 5 kV sur l’interface CE-MS.
- Commencez à infuser la solution modificateur avec la pompe à perfusion à un débit allant de 0,1 à 10 μL/min et assurez une électropulvérisation stable à l’extrémité de l’interface CE-MS.
- Placez le flacon d’échantillon contenant le BGE dans l’échantillonneur automatique et utilisez-le à l’étape 2.3.4 pour acquérir des électrophérogrammes vierges et des spectres de masse vierges.
- Injecter la solution d’échantillon à l’aide de l’échantillonneur automatique à 2 psi et pendant une durée appropriée pour permettre l’injection d’une quantité souhaitée de l’échantillon. Estimer le volume d’injection en utilisant la relation entre le volume d’injection et les paramètresd’injection 15 définie par l’équation (1).
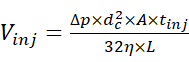 (1)
(1)
Où Vinj est le volume d’injection, Δp est la pression d’injection, dc est le diamètre intérieur du capillaire, A est la section transversale du capillaire, tinj est la durée de l’injection, η est la viscosité du liquide dans le capillaire et L est la longueur du capillaire. - Commencez la séparation CE en appliquant une tension électrophorétique de 30 kV et une pression d’infusion allant de 0 à 2 psi, et acquérez l’électrophérogramme. Pendant ce temps, commencez l’acquisition des données MS en mode chromatographique où le graphique de courant ionique est acquis en fonction du temps, et les scans MS correspondants ne sont pas automatiquement combinés en un seul spectre.
REMARQUE: Les protéines subissent une réaction spontanée HDX au point de contact des molécules 2H2O dans BGE au cours de leur migration électrophorétique à cette étape. La détection optique pour CE peut être utilisée en plus de la détection MS. Comme la détection sur colonne nécessite l’enlèvement d’une certaine longueur de revêtement en polyimide à l’extrémité de sortie du capillaire de silice fondue, des précautions supplémentaires doivent être prises pour éviter les dommages capillaires lors de l’assemblage de l’interface CE-MS. - Enregistrez l’électrophérogramme vierge et les spectres de masse comme références.
REMARQUE : Les données vides doivent être utilisées pour le dépannage plutôt que pour la soustraction de base. - Placez les flacons d’échantillon contenant les concentrations souhaitées des solutions d’échantillons de protéines dans l’échantillonneur automatique. Acquérir les électrophérogrammes et les spectres de masse pour les échantillons de protéines en suivant les étapes 2.3.4-2.3.5. Recueillir un nombre adéquat de scintigraphies de SP pour obtenir des spectres MS1 des espèces de protéines séparées électrophorétiquement et marquées 2H.
- Effectuer des mesures MS en tandem pour les espèces d’intérêt soit après avoir acquis les spectres MS1 au cours de la même série, soit lors d’une exécution ultérieure distincte.
- Si nécessaire, ajustez les temps de migration/temps de réaction HDX en modifiant la pression de perfusion ou la longueur du capillaire CE. Si le temps de réaction HDX doit être plus court que le temps de migration, utilisez l’approche décrite précédemment8, qui utilise à la fois le BGE deutéré et non deutéré dans le capillaire pendant le processus CE.
- Rincer le capillaire CE avec BGE à une pression de 20 psi pendant au moins 10 min après chaque mesure.
- Une fois les expériences terminées, nettoyez l’interface CE-MS et tous les tubes pour le stockage.
- Acquérir un ensemble de données de l’échantillon « critère d’évaluation » HDX (qui peut être préparé à l’aide des approches décrites précédemment 6,16) avec la SEP en mode perfusion directe.
REMARQUE : cette étape n’est requise que lorsqu’une solution modificateur deutérée est utilisée pour hdX basé sur CE.
3. Analyse des données
- Analyse des données CE
- Utilisez l’un des diagrammes suivants comme électrophérogramme pour déterminer les caractéristiques électrophorétiques, y compris le nombre de pics, les temps de migration et l’efficacité de séparation : (a) absorbance UV par rapport au temps de migration, acquise par le détecteur optique de l’instrument CE (le cas échéant); b) le graphique du courant ionique total (TIC) acquis par les États membres; c) le graphique du courant ionique extrait (EIC/XIC) acquis par les États membres.
REMARQUE: EIC / XIC fournit le rapport signal / bruit optimal (S / N), en général, parmi les formats d’électrophérogrammes susmentionnés. Il est à noter que même en l’absence de biais instrumentaux, alors que l’absorbance UV est proportionnelle à la concentration massique de protéines, le signal MS est proportionnel à la concentration molaire. Par conséquent, il est raisonnable d’observer des différences dans les modèles de pic entre les électrophérogrammes dérivés de l’EC et de la SEP. - Utilisez l’aire sous la courbe (ASC) des pics indiqués dans les électrophérogrammes pour la semi-quantification. Pour les échantillons impliquant des complexes protéiques, utilisez l’approche décrite précédemment17 pour déduire les données de concentration massique des électrophérogrammes TIC/EIC.
- Utilisez l’un des diagrammes suivants comme électrophérogramme pour déterminer les caractéristiques électrophorétiques, y compris le nombre de pics, les temps de migration et l’efficacité de séparation : (a) absorbance UV par rapport au temps de migration, acquise par le détecteur optique de l’instrument CE (le cas échéant); b) le graphique du courant ionique total (TIC) acquis par les États membres; c) le graphique du courant ionique extrait (EIC/XIC) acquis par les États membres.
- Analyse des données sur la SEP
- Obtenez les spectres MS1 et MS2 en combinant les balayages MS1 et MS2 acquis dans les fenêtres d’élution correspondantes, respectivement.
- Déterminez les masses de la protéine intacte (M (protéine intacte)) et des fragments par l’une des deux méthodes suivantes.
- Calculer les masses moyennes des ions donnant naissance aux amas de signaux à résolution isotopique.
- Utilisez le centre des courbes de type gaussien résultant de l’ajustement des enveloppes isotopiques correspondantes6.
- Utilisez des logiciels tels que Biopharma Finder, ProSight18 ou MASH Suite19 pour générer la liste de masse des ions fragments et les identifier.
- Analyse des données HDX
- Déterminer le niveau global de deutération d’une espèce protéique intacte à l’aide de l’équation (2).
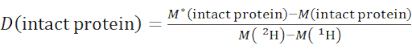 (2)
(2)
où M (2H) ou M (1H) sont des masses atomiques de 2H ou 1H. L’astérisque indique les données de l’échantillon marqué 2H. - Déterminer la protection cumulative ou la deutération cumulative des amides dorsaux d’un segment spécifique.
- Pour les données acquises avec une solution modificateur deutérée, utilisez les équations (3) et (4) pour déterminer le niveau de protection cumulatif.
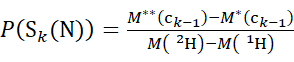 (3)
(3)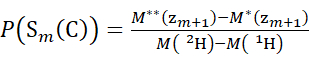 (4)
(4)
Où P(Sk(N)) est la protection totale du segment N-terminal couvrant les résidus 1 à k, P(Sm(C)) est la protection totale du segment C-terminal comprenant m résidus, M (2H) ou M (1H) sont des poids atomiques de 2H ou 1H, et M (ci) ou M (zi) sont les poids moléculaires des ions ci ou zi .
REMARQUE : Le double astérisque indique les données de l’exemple hdx « endpoint ». - Pour les données acquises avec une solution modificateur non deutérée, utilisez les équations (5) et (6) pour déterminer le niveau de deutération cumulatif.
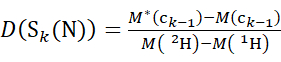 (5)
(5)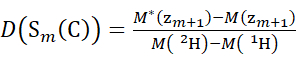 (6)
(6)
Où D(Sk(N)) est l’absorption cumulative de deutérium du segment N-terminal couvrant les résidus 1 à k; D(Sm(C)) est l’absorption cumulative de deutérium du segment C-terminal comprenant m résidus.
- Pour les données acquises avec une solution modificateur deutérée, utilisez les équations (3) et (4) pour déterminer le niveau de protection cumulatif.
- Déterminer le niveau de deutération au niveau d’un groupe amide dorsal local
- Pour les données acquises avec une solution modificateur deutérée, utilisez les équations (7), (8), (9), (10) et (11) pour déterminer le niveau de protection local.
pour les données déduites des ions c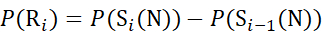 (7)
(7)
pour les données déduites des z-ions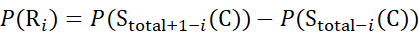 (8)
(8)
Où P(Ri) est la protection d’un amide dorsal au résidu i, et l’indice « total » désigne le nombre total de résidus de la protéine.
Pour les sites de résidus où des ions fragments ultérieurs étaient manquants, attribuez P(Ri) à l’aide des équations (9) et (10).
pour les données déduites des ions c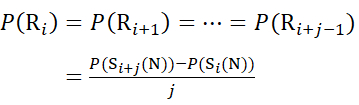 (9)
(9)
pour les données déduites des z-ions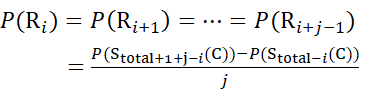 (10)
(10)
Ensuite, déterminez le niveau de deutération D(Ri) à un groupe amide dorsal local à l’aide de l’équation (11).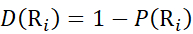 (11)
(11) - Pour les données acquises avec une solution modificateur non deutérée, utilisez les équations (12), (13), (14) et (15) pour déterminer le niveau de protection local.
pour les données déduites des ions c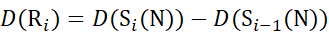 (12)
(12)
pour les données déduites des z-ions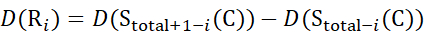 (13)
(13)
Où D(Ri) est la protection d’un amide dorsal au résidu i, et l’indice « total » désigne le nombre total de résidus de la protéine.
Pour les sites de résidus où des ions fragments ultérieurs étaient manquants, assignez D(Ri) à l’aide des équations (14) et (15).
pour les données déduites des ions c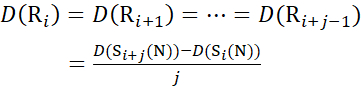 (14)
(14)
pour les données déduites des z-ions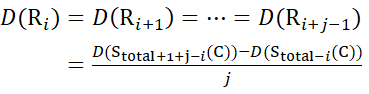 (15)
(15)
- Pour les données acquises avec une solution modificateur deutérée, utilisez les équations (7), (8), (9), (10) et (11) pour déterminer le niveau de protection local.
- Déterminer le niveau global de deutération d’une espèce protéique intacte à l’aide de l’équation (2).
Résultats
La modification de la pression de perfusion de BGE permet d’ajuster à la fois l’efficacité de séparation et le temps de migration, ce qui équivaut au temps de réaction HDX des protéines à séparer (Figure 3). Une pression de perfusion plus faible se traduit par une meilleure séparation des pics CE au détriment de la durée de l’expérience (Figure 3A). Un temps de migration/réaction HDX plus long entraîne un niveau plus élevé de deutération d...
Discussion
Les objectifs du revêtement de la paroi interne du capillaire CE comprennent la minimisation du flux électroosmotique et l’absorption des protéines au cours du processus CE13. Bien que l’écoulement électroosmotique soit bénéfique pour l’analyse CE conventionnelle de petites molécules en raison de sa capacité à conduire des espèces neutres ou chargées de manière opposée au détecteur, il compromet l’efficacité de séparation des espèces de protéines de tailles et de charges...
Déclarations de divulgation
D. D. Y. Chen est l’un des fondateurs du Knowledge for Health Institute for Biomolecules, qui commercialise l’interface CE-MS microviale à flux continu. D’autres auteurs n’ont rien à divulguer.
Remerciements
Ce travail a été soutenu par des subventions de la National Natural Science Foundation of China (NSFC 21974069). Les auteurs ont également reçu le soutien de l’Institute for Cell Analysis, Shenzhen Bay Laboratory, Chine; Centre d’innovation collaborative du Jiangsu pour les matériaux fonctionnels biomédicaux; et Jiangsu Key Laboratory of Biomedical Materials à l’Université normale de Nanjing, en Chine.
matériels
| Name | Company | Catalog Number | Comments |
| ammonium acetate | Fisher Chemical | A/3446/50 | ≥99% |
| CESI 8000 plus capillary electrophoresis system | Sciex, USA | ||
| centrifuge | Eppendorf | 5406000097 | |
| centrifugal filter | Merck | UFC201024 | 10 kDa cutoff |
| deuterium oxide | Energy Chemical | E090001 | 99.9 % D |
| formic acid | Acros Organics | 270480250 | |
| fused silica glass capillary | Polymicro Technologies | 1068150017 | ID 50μm, OD 360μm |
| gas chromatography | Agilent | GC6890N | |
| hydrochloric acid | Sigma Aldrich | 258148 | |
| hydroxypropyl cellulose | Aladdin | H113415 | MW 100000 |
| magnetic stirrers | DLAB | 8030101212 | |
| methanol | Fisher Chemical | A456-4 | MS grade |
| microvolume UV-Vis spectrophotometer | DeNovix | 84677JK7731 | |
| myoglobin | Sigma Aldrich | M1882 | |
| Orbitrap Fusion Lumos mass spectrometer | Thermo Fisher Scientific, USA | ||
| PA 800 Plus Pharmaceutical Analysis CE System | Beckman Coulter, USA | ||
| Q Exactive UHMR mass Spectrometer | Thermo Fisher Scientific, Germany | ||
| sodium hydroxide | Sigma Aldrich | S5881 | |
| ubiquitin | Sigma Aldrich | U6253 | |
| ultrasonicator | SCIENTZ | SB-5200 | |
| β-lactoglobulin | Sigma Aldrich | L0130 | |
Références
- Kaltashov, I. A., Bobst, C. E., Pawlowski, J., Wang, G. Mass spectrometry-based methods in characterization of the higher order structure of protein therapeutics. Journal of Pharmaceutical and Biomedical Analysis. 184, 113169 (2020).
- Engen, J. R., Botzanowski, T., Peterle, D., Georgescauld, F., Wales, T. E. Developments in hydrogen/deuterium exchange mass spectrometry. Analytical Chemistry. 93 (1), 567-582 (2021).
- Pan, J., Han, J., Borchers, C. H., Konermann, L. Conformer-specific hydrogen exchange analysis of Abeta(1-42) oligomers by top-down electron capture dissociation mass spectrometry. Analytical Chemistry. 83 (13), 5386-5393 (2011).
- Pan, J., Han, J., Borchers, C. H., Konermann, L. Structure and dynamics of small soluble Abeta(1-40) oligomers studied by top-down hydrogen exchange mass spectrometry. Biochemistry. 51 (17), 3694-3703 (2012).
- Pan, J., Borchers, C. H. Top-down structural analysis of posttranslationally modified proteins by Fourier transform ion cyclotron resonance-MS with hydrogen/deuterium exchange and electron capture dissociation. Proteomics. 13 (6), 974-981 (2013).
- Wang, G., Abzalimov, R. R., Bobst, C. E., Kaltashov, I. A. Conformer-specific characterization of nonnative protein states using hydrogen exchange and top-down mass spectrometry. Proceedings of the National Academy of Sciences of the United. States of America. 110 (50), 20087-20092 (2013).
- Mironov, G. G., Clouthier, C. M., Akbar, A., Keillor, J. W., Berezovski, M. V. Simultaneous analysis of enzyme structure and activity by kinetic capillary electrophoresis-MS. Nature Chemical Biology. 12 (11), 918-922 (2016).
- Shen, Y., Zhao, X., Wang, G., Chen, D. D. Y. Differential hydrogen/deuterium exchange during proteoform separation enables characterization of conformational differences between coexisting protein states. Analytical Chemistry. 91 (6), 3805-3809 (2019).
- Kontopidis, G., Holt, C., Sawyer, L. Invited review: β-lactoglobulin: binding properties, structure, and function. Journal of Dairy Science. 87 (4), 785-796 (2004).
- Qin, B. Y., et al. Structural basis of the Tanford transition of bovine β-lactoglobulin. Biochemistry. 37 (40), 14014-14023 (1998).
- Qin, B. Y., Bewley, M. C., Creamer, L. K., Baker, E. N., Jameson, G. B. Functional implications of structural differences between variants A and B of bovine beta-lactoglobulin. Protein Science. 8 (1), 75-83 (1999).
- Wang, L., et al. High resolution capillary isoelectric focusing mass spectrometry analysis of peptides, proteins, and monoclonal antibodies with a flow-through microvial interface. Analytical Chemistry. 90 (15), 9495-9503 (2018).
- Busch, M. H. A., Kraak, J. C., Poppe, H. Cellulose acetate-coated fused-silica capillaries for the separation of proteins by capillary zone electrophoresis. Journal of Chromatography A. 1695 (2), 287-296 (1995).
- Zhao, X., Shen, Y., Tong, W., Wang, G., Chen, D. D. Y. Deducing disulfide patterns of cysteine-rich proteins using signature fragments produced by top-down mass spectrometry. Analyst. 143 (4), 817-823 (2018).
- Sutera, S. P., Skalak, R. The history of Poiseuille's law. Annual Review of Fluid Mechanics. 25 (1), 1-20 (1993).
- Wang, G., Kaltashov, I. A. Approach to characterization of the higher order structure of disulfide-containing proteins using hydrogen/deuterium exchange and top-down mass spectrometry. Analytical Chemistry. 86 (15), 7293-7298 (2014).
- Wang, G., Johnson, A. J., Kaltashov, I. A. Evaluation of electrospray ionization mass spectrometry as a tool for characterization of small soluble protein aggregates. Analytical Chemistry. 84 (3), 1718-1724 (2012).
- Fellers, R. T., et al. ProSight Lite: graphical software to analyze top-down mass spectrometry data. Proteomics. 15 (7), 1235-1238 (2015).
- Cai, W., et al. MASH Suite Pro: A comprehensive software tool for top-down proteomics. Molecular & Cellular Proteomics. 15 (2), 703-714 (2016).
- Paterson, G. R., Hill, J. P., Otter, D. E. Separation of β-lactoglobulin A, B and C variants of bovine whey using capillary zone electrophoresis. Journal of Chromatography A. 700 (1), 105-110 (1995).
- Wang, G., Kaltashov, I. A. Approach to characterization of the higher order structure of disulfide-containing proteins using hydrogen/deuterium exchange and top-down mass spectrometry. Analytical Chemistry. 86 (15), 7293-7298 (2014).
- Nicolardi, S., et al. On-line electrochemical reduction of disulfide bonds: improved FTICR-CID and -ETD coverage of oxytocin and hepcidin. Journal of the American Society for Mass Spectrometry. 24 (12), 1980-1987 (2013).
- Adhikari, S., Xia, Y., McLuckey, S. A. Top-down analysis of disulfide-linked proteins using photoinduced radical reactions and ET-DDC. International Journal of Mass Spectrometry. 444, 116173 (2019).
- Rush, M. J. P., Riley, N. M., Westphall, M. S., Coon, J. J. Top-down characterization of proteins with intact disulfide bonds using activated-ion electron transfer dissociation. Analytical Chemistry. 90 (15), 8946-8953 (2018).
- Zhong, X., Maxwell, E. J., Chen, D. D. Y. Mass transport in a micro flow-through vial of a junction-at-the-tip capillary electrophoresis-mass spectrometry interface. Analytical Chemistry. 83 (12), 4916-4923 (2011).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon