このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Methods Article
トップダウン質量分析によるタンパク質の立体構造特性評価のためのキャピラリー電気泳動ベースの水素/重水素交換
要約
ここでは、キャピラリー電気泳動ベースの水素/重水素交換(HDX)アプローチとトップダウン質量分析を組み合わせたプロトコルを紹介します。このアプローチは、同時差動HDXおよび電気泳動分離を行うことによって、異なる状態および異なるプロテオ形態のタンパク質を含む、異なるタンパク質種間の高次構造の違いを特徴付ける。
要約
溶液中で共存する複数のタンパク質状態の立体構造不均一性の解決は、分子認識から酵素触媒作用に至るまで、タンパク質治療薬の特性評価および生物学的機能に重要な立体構造遷移経路の決定における主要な障害の1つであり続けている。水素/重水素交換(HDX)反応とトップダウン質量分析(MS)分析を組み合わせることで、タンパク質の高次構造とダイナミクスをコンフォーマー特異的な方法で特徴付ける手段を提供します。この技術の立体構造分解能は、インタクトなタンパク質レベルでタンパク質状態を分離し、HDX反応中に残留する非重水素化プロトン含有量を最小限に抑える効率に大きく依存します。
ここでは、立体配座分解能の向上を目的としたHDX MSアプローチのキャピラリー電気泳動(CE)ベースのバリアントについて説明します。このアプローチでは、タンパク質は、キャピラリー電気泳動分離中に重水素化バックグラウンド電解質溶液(BGE)を通って移動しながらHDX反応を受ける。溶液中に共存する異なるタンパク質状態またはプロテオフォームは、それらの異なる電荷対サイズ比に基づいて効率的に分離することができる。タンパク質とプロトン性溶媒分子との間の電気泳動移動度の差は、残留非重水素化溶媒を最小限に抑え、HDXプロセス中にほぼ完全な重水素化環境をもたらす。フロースルーマイクロバイアルCE-MSインターフェースは、噴霧器の出口で急冷および変性修飾剤溶液と迅速に混合した後、溶出タンパク質種の効率的なエレクトロスプレーイオン化を可能にする。オンラインのトップダウンMS分析は、溶出された無傷のタンパク質種の全球重水素化レベルを測定し、続いてそれらの気相断片の重水素化を測定する。このホワイトペーパーでは、牛乳中に共存する天然のタンパク質バリアントを含む、システム用の差動HDXにおけるこのアプローチを実証します。
概要
異なる立体構造、結合、または修飾状態のタンパク質種を区別し、それらの構造的差異を特徴付けることは、分子認識から酵素触媒作用に至るまで、生物学的事象に関与するこれらの種間の遷移経路をモニターし、これらの事象の根底にあるメカニズムを理解するために重要である。従来の生物物理学的技術は、不十分な分解能および溶液中の動的情報の損失などの制限のために、完全な解決策を提供しない。質量分析(HDX MS)と組み合わせた水素/重水素交換は、意図的に導入された2H2O溶液からのタンパク質の不安定な水素原子と2Hとの間の交換を介して、タンパク質の構造的および立体構造的特徴を重水素(2H)で標識する技術である。水素結合に関与するプロトンは、タンパク質内部で溶媒から隔離されているものや、容易に交換しない1。このように、交換可能な部位における交換速度は、その高次構造への関与に大きく依存するため、1Hと2Hの異なる原子質量に基づいて2H取り込みの程度と速度をプローブするMSによって、タンパク質構造を高い空間分解能で明らかにすることができる。ここ数十年にわたり、HDX MSはタンパク質の立体構造とダイナミクス2を研究するための卓越した技術となっています。
HDX MSの古典的なボトムアップアプローチでは、異なる立体構造、結合、または修飾状態のタンパク質種のアンサンブルは、無傷のタンパク質レベルで分離することなくタンパク質分解されるため、複雑な重水素含有量で得られたタンパク質分解断片を分析することによって個々の種を特徴付けることは不可能です。対照的に、トップダウンアプローチでは、異なる重水素含有量を組み込んだ異なるタンパク質状態またはプロテオフォームは、MSスキャンにおいて無傷のタンパク質塊の複数の分布を生じる。これにより、適切な質量フィルタ(四重極など)を用いた各質量分布に対応するイオンの質量選択と、その後のタンデムMS分析3、4、5、6におけるそれらの立体構造の違いの特性評価によって、個々の種を分離することができる。しかしながら、この戦略におけるタンパク質状態またはプロテオフォームの分離効率は、それらの対応する質量分布の違いの程度によって制限される。
キャピラリー電気泳動(CE)は、溶液相におけるそれらの異なる電荷および流体力学的サイズに基づいてタンパク質種を高効率で分離する手段を提供する7。CEとHDXを組み合わせることで、溶液相におけるタンパク質状態またはプロテオフォームのさらなる分離が可能になります。さらに、CEキャピラリーの容量が小さいため、完全に重水素化された溶液をバックグラウンド電解質溶液(BGE)、すなわちランニングバッファーとして利用でき、キャピラリーをタンパク質サンプルのHDXリアクターとしてレンダリングできます。電気泳動プロセスにおけるタンパク質とプロトン性試薬間の電気泳動移動度の違いにより、CE中にHDXを実施すると、残留非重水素化含有量が最小限に抑えられたタンパク質分析物の重水素化環境がほぼ完全になり、HDXデータを使用した構造解析の感度が向上します。そのため、CEベースの差動HDXアプローチをトップダウンMSと組み合わせて開発し、タンパク質の高次構造を状態またはプロテオフォーム特異的な方法で特徴付けました8。
このホワイトペーパーでは、材料調製、実験手順、およびデータ分析の手順を詳述することによって、このアプローチのプロトコルについて説明します。メソッドのパフォーマンスまたはデータ品質に影響を与える可能性のある要因は、短いメモにリストされています。ここで提示される代表的な結果には、乳中に存在する主要な乳清タンパク質であるウシβ-ラクトグロブリン(β-lg)の異なるタンパク質および天然変異体の混合物の差動HDXデータが含まれる9。我々は、CEベースのHDXおよびそれらの立体構造の変異体特異的特性評価中に、β-lgの2つの豊富な変異体、すなわちAおよびB10,11の分離効率、再現性、および2H標識性能を実証する。
プロトコル
注: MS 分析を妨げる可能性のある汚染物質を最小限に抑えるために、可能な限り高速液体クロマトグラフィー (HPLC) グレードまたは MS グレードの試薬を使用してください。電気泳動電圧またはエレクトロスプレー電圧のいずれかによって引き起こされる感電の可能性を避けるために、測定中は素手でCE-MSインターフェースに触れないでください。
1. 材料準備
- CE用溶融シリカキャピラリーの改質
- HPC粉末(分子量[MW]:100kDa)を水に溶解して5%(w/w)ヒドロキシプロピルセルロース(HPC)溶液を調製し、マグネチックスターラー上で室温で約12時間または固体粒子12が完全に消失するまで連続的に撹拌する。超音波装置で目に見える気泡を取り除きます。
- 長さ約85cmの溶融シリカガラスキャピラリー(内径[ID]:50μm、外径[OD]:360μm)をCE機器に取り付けます。CEのオートサンプラーを使用して、アセトン13などの有機溶媒を連続的に注入してキャピラリーをすすぎ、注入圧力40psiで10〜15分間処理します。
- オートサンプラーを使用して、洗浄したキャピラリーをHPC溶液で40psiの注入圧力(多くの場合、約40分かかります)で満たします。HPC充填キャピラリーに40psiで空気を注入して、キャピラリー内の自由な空気の流れを確保します(水に浸漬するとキャピラリーから噴出する気泡によって示されます)。
- 図1に示す温度プログラムに従って、HPCコーティングされたキャピラリーを温度プログラム可能なオーブン(理想的には温度プログラムされたガスクロマトグラフの温度制御カラムオーブン)で、キャピラリー内を窒素ガス(25psi)で焼きます。
- キャピラリーを取り出す前にオーブンを室温まで冷やしてください。このHPC修飾キャピラリーはCE分離に使用してください。
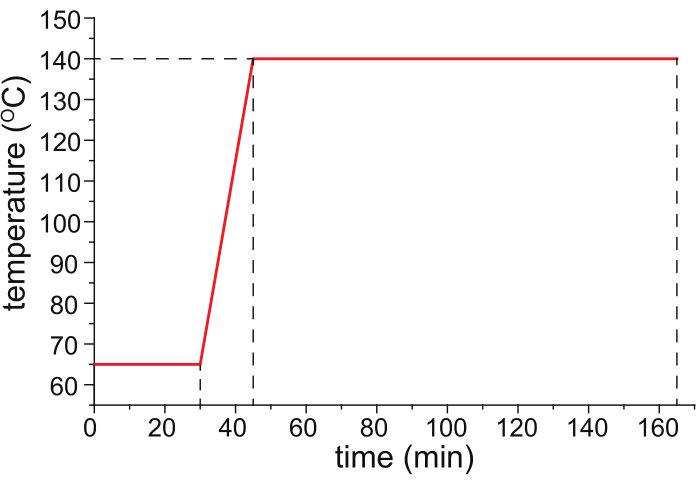
図 1: 毛細血管のベーキングに推奨される温度プログラム。 この図の拡大版を表示するには、ここをクリックしてください。
- バックグラウンド電解質(BGE)溶液及び改質剤溶液8
- 適切な量の酢酸アンモニウムを 2H2Oに溶解して、所望の濃度(例えば、10mM)で1〜10mLのBGEを調製し、200μLアリコートのBGEを別々のBGEバイアルに入れ、バイアルをパラフィルムで密封して、空気中のBGEと水蒸気との間のHDX反応を最小限に抑える。
- 75%(v/v)メタノールと25%(v/v)水を含む改質剤溶液10mLを調製し、ギ酸を使用してpHを2.5に調整する。
注:側鎖の重水素原子および保護されていない主鎖アミドをMSによる検出のために保持する必要がある場合は、2H2Oおよび重水素化メタノールを使用して改質剤溶液を調製する。
- タンパク質サンプルの脱塩
- 所望の濃度で非重水素化水中に酢酸アンモニウム溶液を調製する。
注: 電気泳動中の大電流とその結果生じるジュール加熱効果を避けるために、100 mM 未満の濃度をお勧めします。 - 必要に応じて、酢酸アンモニウム溶液のpHを、ギ酸(pH<6.8の場合)または水酸化アンモニウム(pH>6.8の場合)を用いて所望のレベルに調整する。
- タンパク質溶液の元の緩衝液を酢酸アンモニウム溶液(所望の濃度の非重水素水で調製し、pHを水酸化アンモニウムで7.5に調整)で置換し、適切なMWカットオフを有する遠心フィルターを使用して、少なくとも5つの連続した濃度および4°Cでの希釈ステップを行う。
注:脱塩されるタンパク質試料は、事前の製造手順(例えば、精製または製剤化)から、またはタンパク質の凍結乾燥粉末を溶解することによって調製され得る。この工程で試料溶液から除去される「塩」とは、一般に、不揮発性であるすべての小イオンまたは分子を指す。これらの種は電気泳動プロセス中にタンパク質から効率的に分離することができますが、電気泳動分解能を損なうことを避け、質量分析計の汚染を最小限に抑えるために、このステップが推奨されます。タンパク質分析物を特定の塩または添加剤によって安定化する必要がある場合は、BGEにそれらを含めます。 - マイクロボリュームUV-Vis分光光度計を用いてタンパク質濃度を決定する。
- 所望の濃度で非重水素化水中に酢酸アンモニウム溶液を調製する。
2. CEベースのHDX MS解析の運用
注:このアプローチで使用される質量分析計には、フーリエ変換イオンサイクロトロン共鳴(FTICR)やorbitrapなどの超高解像度の質量分析器、フラグメンテーションのための前駆体イオンの質量選択を可能にする四重極などの質量フィルタ、および信頼性の高いタンデムMSデータ(理想的にはフラグメントイオンの同位体分解信号)でトップダウン分析を実行するための電子移動解離(ETD)または電子捕獲解離(ECD)機能が装備されている必要があります。
- CE および MS 設定の最適化
- 金属被覆ホウケイ酸ガラスキャピラリーからプリロードされたサンプル(「静的」ナノESIスキーム)または金属エミッタから連続的に注入されたサンプルのいずれかを噴霧して、標準的なエレクトロスプレーイオン化(ESI)ソースを使用してパイロットMS測定を実行し、インタクトタンパク質(MS1)およびその気相フラグメント(MS2)の測定のためのMS設定を最適化します。単一の電荷状態におけるそのイオンのアンサンブルの質量選択によって目的のタンパク質種を断片化し、続いて前駆体イオンのETDまたはECDを断片化する。
注: 重要な設定には、脱溶媒和に影響を与えるパラメータ、前駆体イオンの質量選択 (他の種からの干渉を避けるため)、フラグメンテーション効率が含まれます。質量選択ウィンドウの中心と幅の両方を、HDX後の分析物イオンの結果生じる質量分布に合わせて大きくする必要があります。CE中のタンパク質種の溶出ウィンドウは通常0.5分から2分の範囲であるため、同等の時間ウィンドウにわたって蓄積されたMS2スキャンに基づいて断片化効率を評価します。これらのパラメータの最適値はタンパク質特異的です。読者は、例示的な設定8、14について以前に公開されたレポートを参照される。 - 光検出器、すなわちフォトダイオードアレイ(PDA)検出器またはUV検出器を備えたCE機器を使用してパイロットCE測定を実行し、タンパク質種の分離のためのCE設定を最適化し、HDX反応時間に相当する移動時間。
メモ: この手順は、CE の光学検出器の可用性に応じてオプションです。光学検出器がない場合、CE 設定は、セクション 2.2 の完了時に、セクション 2.3 で説明されている指示に従って、CE-MS を使用して最適化できます。重要な設定には、分離効率に影響を与えるパラメータ、電気泳動図に表示されるピーク形状、溶出時間が含まれます。
- 金属被覆ホウケイ酸ガラスキャピラリーからプリロードされたサンプル(「静的」ナノESIスキーム)または金属エミッタから連続的に注入されたサンプルのいずれかを噴霧して、標準的なエレクトロスプレーイオン化(ESI)ソースを使用してパイロットMS測定を実行し、インタクトタンパク質(MS1)およびその気相フラグメント(MS2)の測定のためのMS設定を最適化します。単一の電荷状態におけるそのイオンのアンサンブルの質量選択によって目的のタンパク質種を断片化し、続いて前駆体イオンのETDまたはECDを断片化する。
- CE-HDXセットアップの事前調整
- 50%メタノール、49%水、および1%ギ酸(v / v)の混合物でフロースルーマイクロバイアルCE-MS界面を室温で少なくとも30分間超音波処理を使用して洗浄する。
- HPC修飾キャピラリーをCE機器に取り付けたら、オートサンプラーを使用してキャピラリーをBGEで10分間すすぎ、キャピラリーをBGEで満たしたままにします。
- 改質剤溶液用の注入チューブとして、適切な長さの未改質溶融シリカキャピラリーチューブ(ID:50μm、OD:360μm)を得る。ユニオンと適切なスリーブを使用して、改質剤チューブを鈍い先端で気密ガラスシリンジに接続し、注入ポンプを使用して改質剤溶液でチューブを少なくとも10分間すすいでください。
- 図2に示すように、対応する溶液をロードしたHPCコーティングされたCEキャピラリーおよびモディファイアチューブの出口を、洗浄されたCE-MSインターフェースに挿入します。
- モディファイア注入用のシリンジを手動または注入ポンプで進めて、モディファイヤ溶液がインターフェースの先端に到達するようにします。組み立てられたCE-MSインターフェースを質量分析計のnanoESIソースハウジングに取り付けます。
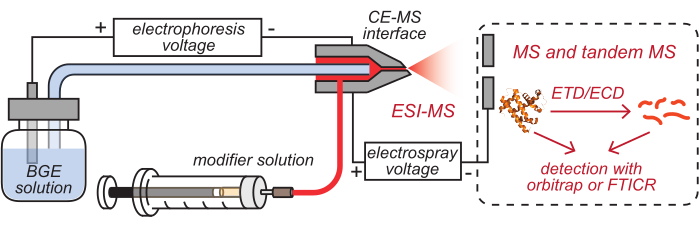
図2:CEベースのHDX MSセットアップの概略図 この図は8から修正されています。略語:BGE =バックグラウンド電解質溶液;CE = キャピラリー電気泳動;MS = 質量分析;HDX = 水素/重水素交換;ESI = エレクトロスプレーイオン化;FTICR = フーリエ変換イオンサイクロトロン共鳴;ETD = 電子移動解離;ECD = 電子捕獲解離。 この図の拡大版を表示するには、ここをクリックしてください。
- CE分離、HDX反応、MS分析の同時実施
注:重水素化BGEは、密封解除後1日以内に使用することをお勧めします。- CE-MSインターフェースに3~5kVのスプレー電圧を印加します。
- 0.1 ~ 10 μL/minの流量で輸液ポンプでモディファイア溶液の注入を開始し、CE-MSインターフェースの先端で安定したエレクトロスプレーを確保します。
- BGEを含むサンプルバイアルをオートサンプラーに入れ、ステップ2.3.4でそれを使用してブランク電気泳動図およびブランク質量スペクトルを取得します。
- オートサンプラーを使用してサンプル溶液を2psiで適切な期間注入し、所望の量のサンプルを注入できるようにします。式(1)で定義される噴射量と噴射パラメータ15 との関係を用いて噴射量を推定する。
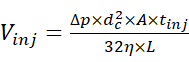 (1)
(1)
ここで、Vinjは注入容積、Δpは注入圧力、dcはキャピラリーの内径、Aはキャピラリーの断面、tinjは注入期間、ηはキャピラリー内の液体の粘度、Lはキャピラリーの長さである。 - 30kVの電気泳動電圧と0〜2psiの範囲の注入圧力を印加することによってCE分離を開始し、電気泳動図を取得する。一方、イオン電流グラフが時間の関数として取得されるクロマトグラフィーモードでMSデータの取得を開始し、対応するMSスキャンが自動的に1つのスペクトルに結合されない。
注:タンパク質は、このステップにおける電気泳動移行中にBGE中の2H2O分子と接触する時点で自発的なHDX反応を受ける。CEの光学検出は、MS検出に加えて使用することができる。オンカラム検出では、溶融シリカキャピラリーの出口端にある一定の長さのポリイミドコーティングを除去する必要があるため、CE-MS界面の組み立て中にキャピラリーの損傷を避けるために追加の注意を払う必要があります。 - 空白の電気泳動図と質量スペクトルを基準として保存します。
メモ: 空白のデータは、ベースラインの減算ではなくトラブルシューティングに使用されます。 - 所望の濃度のタンパク質試料溶液を含むサンプルバイアルをオートサンプラーに入れる。手順2.3.4-2.3.5に従ってタンパク質サンプルの電気泳動図と質量スペクトルを取得します。適切な数のMSスキャンを収集して、電気泳動的に分離された2つのH標識タンパク質種の MS1スペクトルを取得します。
- 目的の種について、同じ実行内でMS1スペクトルを取得した後、または後続の別々の実行でタンデムMS測定を実行します。
- 必要に応じて、注入圧力またはCEキャピラリーの長さを変更して、移行時間/HDX反応時間を調整します。HDX反応時間をマイグレーション時間よりも短くする必要がある場合は、CEプロセス中にキャピラリー内の重水素化BGEと非重水素化BGEの両方を使用する前述のアプローチ8を使用します。
- 各測定後、CEキャピラリーをBGEで20psiの圧力で少なくとも10分間フラッシュします。
- 実験が完了したら、CE-MSインターフェースとすべてのチューブを清掃して保管してください。
- HDXの「エンドポイント」サンプル(前述のアプローチ6,16を使用して調製可能)のデータセットを、MSを直接注入モードで取得します。
メモ: この手順は、CE ベースの HDX に重水素化修飾子ソリューションを使用する場合にのみ必要です。
3. データ解析
- CEデータの分析
- 以下のプロットのいずれかを電気泳動図として使用して、ピーク数、移動時間、および分離効率を含む電気泳動特性を決定する:(a)CE装置の光学検出器によって取得されたUV吸光度対移動時間(利用可能な場合)。(b)MSによって取得された総イオン電流(TIC)グラフ;(c)MSが取得した抽出イオン電流(EIC/XIC)グラフ。
注: EIC/XICは、一般に、前述の電球図フォーマットの中で最適な信号/ノイズ比(S/N)を提供します。器械的バイアスがなくても、UV吸光度はタンパク質の質量濃度に比例するが、MSシグナルはモル濃度に比例することが注目に値する。したがって、CEとMS由来の電気泳動図の間でピークパターンの違いを観察することは合理的です。 - 半定量には、電気泳動図に示されているピークの曲線下面積(AUC)を使用します。タンパク質複合体を含むサンプルの場合、前述のアプローチ17 を使用して、TIC/EIC電気泳動図から質量濃度データを推定します。
- 以下のプロットのいずれかを電気泳動図として使用して、ピーク数、移動時間、および分離効率を含む電気泳動特性を決定する:(a)CE装置の光学検出器によって取得されたUV吸光度対移動時間(利用可能な場合)。(b)MSによって取得された総イオン電流(TIC)グラフ;(c)MSが取得した抽出イオン電流(EIC/XIC)グラフ。
- MSデータの分析
- 対応する溶出ウィンドウ内で取得したMS1およびMS2スキャンをそれぞれ組み合わせて、MS1およびMS2スペクトルを取得します。
- インタクトタンパク質(M (インタクトタンパク質))およびフラグメントの質量を、以下の2つの方法のいずれかによって決定する。
- 同位体分解シグナルクラスターを生じるイオンの平均質量を計算します。
- 対応する同位体エンベロープ6のフィッティングから生じるガウス様曲線の中心を使用する。
- Biopharma Finder、ProSight18、MASH Suite19 などのソフトウェアを使用して、フラグメントイオンの質量リストを生成し、それらを同定します。
- HDXデータの分析
- 式(2)を用いて、無傷のタンパク質種の全体的な重水素化レベルを決定する。
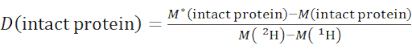 (2)
(2)
ここで、M (2 H) または M (1 H) は 2H または 1H の原子量です。アスタリスクは、2H標識サンプルのデータを示す。 - 特定のセグメントの骨格アミドの累積保護または累積重水素化を決定する。
- 重水素化修飾子解で取得したデータの場合、式(3)と式(4)を使用して累積保護レベルを求めます。
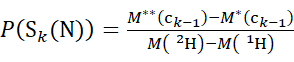 (3)
(3)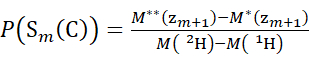 (4)
(4)
ここで、P(Sk(N))は残基1〜kにまたがるN末端セグメントの総保護であり、P(Sm(C))はm残基を含むC末端セグメントの総保護であり、M(2H)またはM(1H)は2Hまたは1Hの原子量であり、M(ci)または M(zi)はciまたはziイオンの分子量である。
メモ:二重アスタリスクは、HDXの「エンドポイント」サンプルのデータを示します。 - 非重水素化修飾子解で取得したデータの場合、式(5)および式(6)を使用して累積重水素化レベルを決定します。
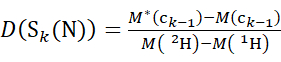 (5)
(5)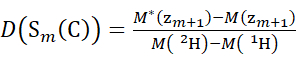 (6)
(6)
ここで、D(S k(N))は、残基1〜kにまたがるN末端セグメントの累積重水素取り込みである。D(Sm(C))は、m残基を含むC末端セグメントの累積重水素取り込みである。
- 重水素化修飾子解で取得したデータの場合、式(3)と式(4)を使用して累積保護レベルを求めます。
- 局所骨格アミド基における重水素化レベルを決定する
- 重水素化修飾子解で取得したデータの場合、式(7)、(8)、(9)、(10)、および(11)を使用して局所保護レベルを決定します。
Cイオンから推定されるデータの場合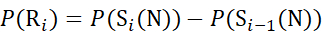 (7)
(7)
zイオンから推定されるデータの場合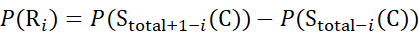 (8)
(8)
ここで、P(Ri)は残基iにおける主鎖アミドの保護であり、添え字「total」はタンパク質の総残基数を示す。
後続のフラグメントイオンが欠落している残基部位については、式(9)および式(10)を使用してP(Ri)を割り当てる。
Cイオンから推定されるデータの場合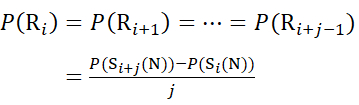 (9)
(9)
zイオンから推定されるデータの場合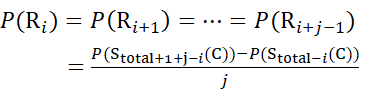 (10)
(10)
次に、式(11)を用いて局所骨格アミド基における重水素化レベルD(Ri)を求める。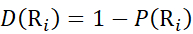 (11)
(11) - 非重水素化修飾子解で集録されたデータの場合、式(12)、(13)、(14)、および(15)を使用して局所保護レベルを決定します。
Cイオンから推定されるデータの場合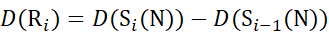 (12)
(12)
zイオンから推定されるデータの場合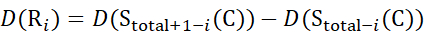 (13)
(13)
ここで、D(Ri)は残基iにおける主鎖アミドの保護であり、添え字「total」はタンパク質の総残基数を示す。
後続のフラグメントイオンが欠落していた残基部位については、式(14)および式(15)を用いてD(Ri)を割り当てる。
Cイオンから推定されるデータの場合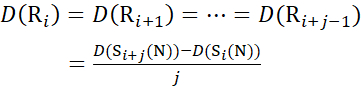 (14)
(14)
zイオンから推定されるデータの場合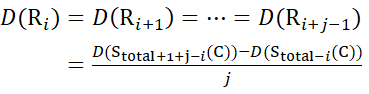 (15)
(15)
- 重水素化修飾子解で取得したデータの場合、式(7)、(8)、(9)、(10)、および(11)を使用して局所保護レベルを決定します。
- 式(2)を用いて、無傷のタンパク質種の全体的な重水素化レベルを決定する。
結果
BGEの注入圧力を変えることで、分離効率と遊走時間の両方を調整でき、分離するタンパク質のHDX反応時間に相当します(図3)。注入圧力が低いほど、実験期間を犠牲にしてCEピークの分離が良好になります(図3A)。マイグレーション/HDX反応時間が長くなると、タンパク質分析物の重水素化レベルが高くなります(図3B-D
ディスカッション
CEキャピラリーの内壁を被覆する目的には、CEプロセス13中の電気浸透流およびタンパク質吸収の最小化が含まれる。電気浸透流は、中性または反対に荷電した種を検出器に駆動する能力のために、小分子の従来のCE分析に有益であるが、溶液中で同様のサイズおよび正味電荷を有するタンパク質種の分離効率を損なう。キャピラリーをHPCでコーティングすると、キャピラリ?...
開示事項
D. D. Y. Chenは、フロースルーマイクロバイアルCE-MSインターフェースを商業化しているKnowledge for Health Institute for Biomoleculesの創設者の一人です。他の著者は開示するものは何もありません。
謝辞
この研究は、中国国家自然科学財団(NSFC 21974069)からの助成金によって支援されました。著者らはまた、中国の深セン湾研究所の細胞分析研究所からも支援を受けた。生物医学的機能材料の江蘇省共同イノベーションセンター;南京師範大学(中国)の江蘇省生物医学材料主要研究所。
資料
| Name | Company | Catalog Number | Comments |
| ammonium acetate | Fisher Chemical | A/3446/50 | ≥99% |
| CESI 8000 plus capillary electrophoresis system | Sciex, USA | ||
| centrifuge | Eppendorf | 5406000097 | |
| centrifugal filter | Merck | UFC201024 | 10 kDa cutoff |
| deuterium oxide | Energy Chemical | E090001 | 99.9 % D |
| formic acid | Acros Organics | 270480250 | |
| fused silica glass capillary | Polymicro Technologies | 1068150017 | ID 50μm, OD 360μm |
| gas chromatography | Agilent | GC6890N | |
| hydrochloric acid | Sigma Aldrich | 258148 | |
| hydroxypropyl cellulose | Aladdin | H113415 | MW 100000 |
| magnetic stirrers | DLAB | 8030101212 | |
| methanol | Fisher Chemical | A456-4 | MS grade |
| microvolume UV-Vis spectrophotometer | DeNovix | 84677JK7731 | |
| myoglobin | Sigma Aldrich | M1882 | |
| Orbitrap Fusion Lumos mass spectrometer | Thermo Fisher Scientific, USA | ||
| PA 800 Plus Pharmaceutical Analysis CE System | Beckman Coulter, USA | ||
| Q Exactive UHMR mass Spectrometer | Thermo Fisher Scientific, Germany | ||
| sodium hydroxide | Sigma Aldrich | S5881 | |
| ubiquitin | Sigma Aldrich | U6253 | |
| ultrasonicator | SCIENTZ | SB-5200 | |
| β-lactoglobulin | Sigma Aldrich | L0130 | |
参考文献
- Kaltashov, I. A., Bobst, C. E., Pawlowski, J., Wang, G. Mass spectrometry-based methods in characterization of the higher order structure of protein therapeutics. Journal of Pharmaceutical and Biomedical Analysis. 184, 113169 (2020).
- Engen, J. R., Botzanowski, T., Peterle, D., Georgescauld, F., Wales, T. E. Developments in hydrogen/deuterium exchange mass spectrometry. Analytical Chemistry. 93 (1), 567-582 (2021).
- Pan, J., Han, J., Borchers, C. H., Konermann, L. Conformer-specific hydrogen exchange analysis of Abeta(1-42) oligomers by top-down electron capture dissociation mass spectrometry. Analytical Chemistry. 83 (13), 5386-5393 (2011).
- Pan, J., Han, J., Borchers, C. H., Konermann, L. Structure and dynamics of small soluble Abeta(1-40) oligomers studied by top-down hydrogen exchange mass spectrometry. Biochemistry. 51 (17), 3694-3703 (2012).
- Pan, J., Borchers, C. H. Top-down structural analysis of posttranslationally modified proteins by Fourier transform ion cyclotron resonance-MS with hydrogen/deuterium exchange and electron capture dissociation. Proteomics. 13 (6), 974-981 (2013).
- Wang, G., Abzalimov, R. R., Bobst, C. E., Kaltashov, I. A. Conformer-specific characterization of nonnative protein states using hydrogen exchange and top-down mass spectrometry. Proceedings of the National Academy of Sciences of the United. States of America. 110 (50), 20087-20092 (2013).
- Mironov, G. G., Clouthier, C. M., Akbar, A., Keillor, J. W., Berezovski, M. V. Simultaneous analysis of enzyme structure and activity by kinetic capillary electrophoresis-MS. Nature Chemical Biology. 12 (11), 918-922 (2016).
- Shen, Y., Zhao, X., Wang, G., Chen, D. D. Y. Differential hydrogen/deuterium exchange during proteoform separation enables characterization of conformational differences between coexisting protein states. Analytical Chemistry. 91 (6), 3805-3809 (2019).
- Kontopidis, G., Holt, C., Sawyer, L. Invited review: β-lactoglobulin: binding properties, structure, and function. Journal of Dairy Science. 87 (4), 785-796 (2004).
- Qin, B. Y., et al. Structural basis of the Tanford transition of bovine β-lactoglobulin. Biochemistry. 37 (40), 14014-14023 (1998).
- Qin, B. Y., Bewley, M. C., Creamer, L. K., Baker, E. N., Jameson, G. B. Functional implications of structural differences between variants A and B of bovine beta-lactoglobulin. Protein Science. 8 (1), 75-83 (1999).
- Wang, L., et al. High resolution capillary isoelectric focusing mass spectrometry analysis of peptides, proteins, and monoclonal antibodies with a flow-through microvial interface. Analytical Chemistry. 90 (15), 9495-9503 (2018).
- Busch, M. H. A., Kraak, J. C., Poppe, H. Cellulose acetate-coated fused-silica capillaries for the separation of proteins by capillary zone electrophoresis. Journal of Chromatography A. 1695 (2), 287-296 (1995).
- Zhao, X., Shen, Y., Tong, W., Wang, G., Chen, D. D. Y. Deducing disulfide patterns of cysteine-rich proteins using signature fragments produced by top-down mass spectrometry. Analyst. 143 (4), 817-823 (2018).
- Sutera, S. P., Skalak, R. The history of Poiseuille's law. Annual Review of Fluid Mechanics. 25 (1), 1-20 (1993).
- Wang, G., Kaltashov, I. A. Approach to characterization of the higher order structure of disulfide-containing proteins using hydrogen/deuterium exchange and top-down mass spectrometry. Analytical Chemistry. 86 (15), 7293-7298 (2014).
- Wang, G., Johnson, A. J., Kaltashov, I. A. Evaluation of electrospray ionization mass spectrometry as a tool for characterization of small soluble protein aggregates. Analytical Chemistry. 84 (3), 1718-1724 (2012).
- Fellers, R. T., et al. ProSight Lite: graphical software to analyze top-down mass spectrometry data. Proteomics. 15 (7), 1235-1238 (2015).
- Cai, W., et al. MASH Suite Pro: A comprehensive software tool for top-down proteomics. Molecular & Cellular Proteomics. 15 (2), 703-714 (2016).
- Paterson, G. R., Hill, J. P., Otter, D. E. Separation of β-lactoglobulin A, B and C variants of bovine whey using capillary zone electrophoresis. Journal of Chromatography A. 700 (1), 105-110 (1995).
- Wang, G., Kaltashov, I. A. Approach to characterization of the higher order structure of disulfide-containing proteins using hydrogen/deuterium exchange and top-down mass spectrometry. Analytical Chemistry. 86 (15), 7293-7298 (2014).
- Nicolardi, S., et al. On-line electrochemical reduction of disulfide bonds: improved FTICR-CID and -ETD coverage of oxytocin and hepcidin. Journal of the American Society for Mass Spectrometry. 24 (12), 1980-1987 (2013).
- Adhikari, S., Xia, Y., McLuckey, S. A. Top-down analysis of disulfide-linked proteins using photoinduced radical reactions and ET-DDC. International Journal of Mass Spectrometry. 444, 116173 (2019).
- Rush, M. J. P., Riley, N. M., Westphall, M. S., Coon, J. J. Top-down characterization of proteins with intact disulfide bonds using activated-ion electron transfer dissociation. Analytical Chemistry. 90 (15), 8946-8953 (2018).
- Zhong, X., Maxwell, E. J., Chen, D. D. Y. Mass transport in a micro flow-through vial of a junction-at-the-tip capillary electrophoresis-mass spectrometry interface. Analytical Chemistry. 83 (12), 4916-4923 (2011).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved