Method Article
Adaptación de organoides gastrointestinales para la infección por patógenos y la secuenciación de células individuales en condiciones de nivel de bioseguridad 3 (BSL-3)
En este artículo
Resumen
Este protocolo describe cómo infectar organoides intestinales humanos desde su lado apical o basolateral para caracterizar las interacciones huésped/patógeno a nivel de una sola célula utilizando la tecnología de secuenciación de ARN de una sola célula (scRNAseq).
Resumen
Los organoides intestinales humanos constituyen el mejor modelo celular para estudiar las infecciones patógenas del tracto gastrointestinal. Estos organoides se pueden derivar de todas las secciones del tracto gastrointestinal (gástrico, yeyuno, duodeno, íleon, colon, recto) y, tras la diferenciación, contienen la mayoría de los tipos de células que se encuentran naturalmente en cada sección individual. Por ejemplo, los organoides intestinales contienen enterocitos que absorben nutrientes, células secretoras (Cáliz, Paneth y enteroendocrino), células madre, así como todos los intermediarios de diferenciación específicos del linaje (por ejemplo, tipos de células tempranas o inmaduras). La mayor ventaja en el uso de organoides derivados del tracto gastrointestinal para estudiar enfermedades infecciosas es la posibilidad de identificar con precisión qué tipo de célula es el objetivo del patógeno entérico y abordar si las diferentes secciones del tracto gastrointestinal y sus tipos de células específicas responden de manera similar a los desafíos de los patógenos. En los últimos años, se han empleado modelos gastrointestinales, así como organoides de otros tejidos, para estudiar el tropismo viral y los mecanismos de patogénesis. Sin embargo, utilizar todas las ventajas del uso de organoides cuando se emplean virus altamente patógenos representa un desafío técnico y requiere estrictas consideraciones de bioseguridad. Además, como los organoides a menudo se cultivan en tres dimensiones, el lado basolateral de las células está orientado hacia el exterior del organoide, mientras que su lado apical está orientado hacia el interior (lumen) de los organoides. Esta organización plantea un desafío para los patógenos entéricos, ya que muchas infecciones entéricas se inician desde el lado apical / luminal de las células después de la ingestión. El siguiente manuscrito proporcionará un protocolo completo para preparar organoides intestinales humanos para la infección con patógenos entéricos al considerar el lado de la infección (apical vs. basolateral) para realizar la secuenciación de ARN de una sola célula para caracterizar las interacciones huésped/patógeno específicas del tipo celular. Este método detalla la preparación de los organoides, así como las consideraciones necesarias para realizar este trabajo bajo condiciones de contención de nivel de bioseguridad 3 (BSL-3).
Introducción
El estudio del tropismo específico del tipo celular y la respuesta inmune específica del tipo celular a los virus entéricos humanos ha sido históricamente un desafío debido a la falta de modelos celulares humanos primarios. Esta limitación ha sido parcialmente erradicada con el desarrollo de organoides1. En el caso del tracto gastrointestinal, se han desarrollado modelos organoides gástricos e intestinales para humanos y varias otras especies (porejemplo, murinos, bovinos, felinos, murciélagos)2,3,4,5,6. Los organoides intestinales reproducen la arquitectura estructural del epitelio intestinal humano y contienen estructuras similares a criptas y vellosidades, linajes intestinales funcionales e incluso se han utilizado para identificar linajes celulares previamente desconocidos. Se pueden utilizar dos enfoques diferentes para cultivar organoides intestinales. Primero, las células madre intestinales que contienen criptas se aíslan de resecciones tisulares o biopsias y se cultivan bajo condiciones de cultivo específicas (por ejemplo, Wnt3A, R-spondin, Noggin y EGF) para expandirse y luego diferenciar las células madre a la mayoría de los linajes de células intestinales (porejemplo, enterocitos, células de Paneth, células caliciformes, células enteroendocrinas)7. Este método permite el aislamiento de organoides de todas las secciones del tracto gastrointestinal(porejemplo, estómago, duodeno, yeyuno, íleon y colon). El segundo método se basa en células madre pluripotentes o embrionarias inducidas por humanos, que luego se diferencian en un proceso gradual en células epiteliales intestinales8. Estos organoides inducidos basados en células madre a menudo se describen como de naturaleza más embrionaria en comparación con los organoides derivados del paciente. Si bien todos estos modelos organoides han sido críticos para desentrañar las señales de desarrollo necesarias para formar el tracto intestinal, su uso en la investigación de enfermedades infecciosas aún está en su infancia.
Virus entérico es un término amplio que abarca todos los virus que infectan a través del tracto gastrointestinal, como los picornavirus(porejemplo, EV-71), los reovirus (por ejemplo, rotavirus) y los calicivirus (por ejemplo, norovirus)9. Los virus entéricos inician su ciclo de vida infeccioso a través de la ingestión de alimentos y agua contaminados, lo que deja a las personas en los países en desarrollo en alto riesgo debido a la descarga de desechos no tratados en el medio ambiente y la falta de atención médica después del inicio de la infección10. Dependiendo del tipo de patógeno, la infección puede provocar gastroenteritis, vómitos y / o diarrea acuosa debido a la fuga del revestimiento intestinal. Los norovirus humanos son un patógeno entérico altamente prevalente y altamente infeccioso, que conduce a más de 600 millones de infecciones y 15 millones de hospitalizaciones en todo el mundo11. Los organoides han sido clave para la investigación del norovirus, ya que apoyan la infección y replicación del norovirus humano, que anteriormente no podía cultivarse en modelos de cultivo celular estándar12.
En las últimas dos décadas, los coronavirus han surgido como patógenos humanos clave13. Esta familia incluye el mersma altamente patógeno, el SARS-CoV-1 y el SARS-CoV-2, que requieren estrictas contenciones de nivel de seguridad al realizar investigaciones sobre estos virus. Curiosamente, si bien estos tres patógenos son reconocidos principalmente por sus síntomas respiratorios inducidos y angustia, ahora es evidente que estos virus no solo infectan el tracto respiratorio, sino también otros órganos. Una patología importante inducida en pacientes infectados por SARS-CoV-2 además de la dificultad respiratoria es la presencia de síntomas gastrointestinales14. Una fracción de los pacientes infectados por SARS-CoV-2 muestra tales síntomas, que van desde diarrea muy leve a severa. Además, los genomas del SARS-CoV-2 se pueden detectar en biopsias de heces y del tracto gastrointestinal de pacientes infectados15. Es importante destacar que la presencia de síntomas gastrointestinales no se limita al SARS-CoV-2, ya que también se observaron en pacientes infectados por MERS y SARS-CoV-1. Para comprender cómo el SARS-CoV-2 induce malestar gastrointestinal e identificar con precisión el tropismo del SARS-CoV-2 en el tracto gastrointestinal, los organoides intestinales humanos han sido una herramienta clave y ahora se explotan para desentrañar las respuestas específicas del tipo celular a este patógeno16,17.
El perfil transcripcional de una población celular (secuenciación masiva de ARN) ha sido una práctica estándar al evaluar infecciones por patógenos tanto de líneas celulares inmortalizadas como de organoides. Si bien esto nos permite determinar los cambios globales en respuesta a los patógenos (por ejemplo, la regulación al alza de las citoquinas), RNAseq a granel no nos permite determinar por qué las células específicas de una población son más propensas a la infección que otras. La secuenciación de ARN unicelular (scRNAseq) se ha convertido en una poderosa herramienta para desentrañar los programas transcripcionales específicos del linaje celular y se puede utilizar para determinar cómo estos programas apoyan o reprimen la infección por virus18,19. La primera descripción de scRNAseq fue en 2009 y se utilizó para evaluar los perfiles de transcripción de las diferentes células encontradas en un blastómerode ratón 20. Estas tecnologías ahora se han expandido y se pueden implementar a través de varias plataformas diferentes. Las primeras versiones de esta tecnología aplicaban el clasificador celular activado por fluorescencia (FACS) para separar células individuales para su secuenciación, que a menudo se limitaba a placas de 96 o 384 pocillos, dando así 300 células individuales para analizar por muestra21. Estos métodos ahora han sido avanzados por las plataformas de secuenciación de una sola célula, que utilizan un dispositivo microfluídico para encapsular células individuales en gotas individuales con códigos de barras que contienen perlas. Esta tecnología permite capturar hasta 10.000 células por condición de muestra.
La combinación de la tecnología de organoides con scRNAseq nos permite estudiar cómo los patógenos entéricos afectan el tracto gastrointestinal de una manera específica del tipo de célula. Sin embargo, es necesario tener en cuenta varias consideraciones técnicas y de bioseguridad. En primer lugar, los métodos clásicos de cultivo de organoides (organoides tridimensionales (3D), incrustados en una matriz extracelular (ECM)) exponen el lado basolateral de las células epiteliales al exterior del organoide. A medida que los patógenos entéricos inician su infección a través de la ingestión de alimentos / agua contaminados, la infección se inicia con mayor frecuencia desde el lado apical de las células, que no es accesible en estos organoides intestinales 3D. Por lo tanto, los organoides deben estar preparados para hacer que el lado apical sea accesible para la infección por patógenos, ya sea a través de la siembra 2D, exponiendo así directamente el lado apical de las células, o a través de la microinyección22,23. En segundo lugar, para realizar scRNAseq de muestras biológicas infectadas, es importante considerar su naturaleza infecciosa. Si bien se han propuesto métodos para fijar células e inactivar patógenos antes del aislamiento de una sola célula para el RNAseq posterior, estos métodos a menudo conducen a una disminución en la calidad de la secuenciación18. El siguiente protocolo describirá varios enfoques para infectar organoides intestinales con virus entéricos considerando el lado de la infección (infección apical vs. basolateral) (Figura 1). Además, el protocolo incluirá un flujo de trabajo para disociar y aislar células individuales de organoides infectados con virus altamente patógenos para scRNAseq. El protocolo destacará los pasos clave que deben implementarse cuando se trabaja bajo condiciones de contención de nivel de bioseguridad 3 (BSL-3) para evitar la generación de aerosoles y la posible contaminación.
Protocolo
Se recibió tejido humano de resección de colon o biopsias de íleon del Hospital Universitario de Heidelberg para el siguiente protocolo. Este estudio se llevó a cabo bajo las recomendaciones del Hospital Universitario de Heidelberg con el consentimiento informado por escrito de todos los sujetos de acuerdo con la Declaración de Helsinki. Todas las muestras fueron recibidas y mantenidas de manera anónima. El protocolo fue aprobado por la comisión de Ética del Hospital Universitario de Heidelberg bajo el protocolo S-443/2017.
1. Mantenimiento y paso a través de organoides intestinales y de colon
PRECAUCIÓN: Los organoides intestinales humanos se derivan de tejido humano o de células madre pluripotentes / embrionarias inducidas, y como tales, se requiere aprobación ética. Es necesario seguir las regulaciones específicas de cada país. El material humano generalmente no se prueba y, por lo tanto, a menudo se considera material BSL-2. Es necesario confirmar las condiciones de contención adecuadas en el país en el que se lleva a cabo el experimento.
- Preparar organoides intestinales y de colon a partir de tejidos aislados y/o biopsias utilizando métodos previamente descritos2. Además, se pueden encontrar más detalles técnicos sobre cómo preparar organoides a partir de material derivado del paciente o de iPSC en Lees et al. y Mahe et al.24,25.
- Una vez que se establecen los cultivos de organoides, siga la rutina de división descrita a continuación para preparar organoides para realizar infecciones con patógenos entéricos.
- Sembrar 20-100 organoides en 50 μL de solución de ECM al 100% en placas tratadas con cultivo no tisular de 24 pocillos. Añadir 500 μL de medios de crecimiento (Tabla 1) por pozo.
- Cambie el medio cada 2 días eliminando 250 μL de los medios antiguos y agregando 250 μL de los medios de crecimiento frescos a cada pozo. Caliente el medio a temperatura ambiente antes de cambiarlo.
PRECAUCIÓN: Los medios fríos licuarán la solución de ECM que contiene organoides. - Pasa los organoides una vez por semana cuando el interior comienza a oscurecerse debido a la acumulación de células muertas. Un ejemplo de esto se puede encontrar en la Figura 2.
- El día de la división, retire la solución de ECM de -20 °C y descongele en hielo.
- Coloque la nueva placa de cultivo celular vacía que se utilizará para sembrar los organoides después de dividirlos a 37 ° C (esto requiere un mínimo de 1 h para estar caliente y podría calentarse durante la noche). Calentar los medios de cultivo a temperatura ambiente.
- Retire el medio de crecimiento con una pipeta P1000 y agregue 500 μL de solución salina tamponada con fosfato (PBS) en frío a cada pozo durante 3 minutos para licuar parcialmente la solución de ECM y disociar de la placa.
- Para garantizar la interrupción completa de la solución ECM, utilice una pipeta P1000 (configuración a 450 μL). Pipetee hacia arriba y hacia abajo 10 veces para resuspendir el PBS, la solución de ECM y los organoides, transfiera los organoides resuspendidos a un tubo cónico de 15 ml y colóquelos en hielo.
- Recolectar múltiples pocillos de los mismos organoides en el mismo tubo cónico.
- Si se dividen múltiples organoides diferentes (diferentes donantes, diferentes secciones, diferentes pretratamientos, etc.) al mismo tiempo, recójalos en diferentes tubos cónicos. Mientras recolecta, mantenga los tubos cónicos en hielo.
- Girar las muestras a 500 x g durante 5 min a 4 °C. Retire cuidadosamente el PBS con una pipeta para mantener el gránulo organoide en la parte inferior.
- Evite usar un sistema de residuos de vacío con una pipeta ajustada, ya que el pellet de organoides está muy suelto y se puede aspirar fácilmente al quitar el PBS demasiado rápido.
- Agregue 1 ml de tripsina al 0.05% al tubo cónico y resuspenda los organoides mediante pipeteo hacia arriba y hacia abajo 10 veces con una pipeta P1000. Incubar el tubo cónico que contiene organoides a 37 °C durante 3 min.
- Agregue 2 ml de medios DMEM/F12 que contengan 10% de suero fetal bovino (FBS) y 1% de penicilina/estreptomicina para detener la digestión y resuspend para interrumpir los organoides mediante pipeteo hacia arriba y hacia abajo 10 veces con una pipeta P1000.
- Girar las muestras a 500 x g durante 5 min a 4 °C. Retire los tubos cónicos de la centrífuga y guárdelos en hielo.
- Retire cuidadosamente el medio / tripsina con una pipeta desechable de 5 ml para mantener el gránulo organoide en la parte inferior. Deje alrededor de 500 μL de medio y retírelo con una pipeta P1000.
- Una vez que se haya extraído el medio/tripsina de todos los tubos cónicos, retire la placa tratada con cultivo no celular de 24 pocillos de la incubadora de 37 °C y colóquela debajo de la campana de cultivo celular.
- Agregue una solución de ECM al 100% (mantenida en hielo) al tubo cónico con los organoides divididos en una proporción de 1: 3 a 1: 5, dependiendo de los hábitos de crecimiento de los organoides donantes individuales. (Por ejemplo, si se pasó un pocillo que contiene 20-100 organoides en una gota de 50 μL de solución de ECM, vuelva a suspender el gránulo de organoides en 150-250 μL de solución de ECM helada).
- Semilla 50 μL de solución de ECM/mezcla de organoides por pocillo de la placa tratada con cultivo no celular de 24 pocillos precalentada a 37 °C.
NOTA: La solución de ECM se polimerizará muy rápidamente una vez que se haya calentado. Mantenga la solución de ECM fría en todo momento (almacenada en hielo) durante el uso. Cuando los organoides han sido resuspendidos, sembrar inmediatamente en una nueva placa. Para los principiantes, se recomienda almacenar una caja de puntas de pipeta a -20 ° C para permitir tiempo adicional para la siembra. - Incubar la placa de 24 pocillos a 37 °C durante 10-15 min para permitir que la solución de ECM se polimerice.
- Después de la polimerización, añadir 500 μL de medios de crecimiento (Tabla 1)26 a cada pocillo e incubar los organoides a 37 °C. Revise los organoides diariamente por microscopía. Cambie el medio cada 2 días como en el paso 1.4.
2. Preparación de organoides en dos dimensiones (2D) para la infección apical
NOTA: El siguiente protocolo describirá cómo sembrar organoides intestinales como una monocapa de células en una placa de cultivo celular para infectar las células del epitelio intestinal desde su lado apical. Use la placa de 48 pocillos para los experimentos de secuenciación y el portaobjetos de la cámara de fondo de vidrio de 8 pocillos para controlar la infección utilizando enfoques de inmunofluorescencia.
- Cultivar y mantener los organoides como se describe en la sección 1.
- Antes de sembrar organoides en 2D, cubra las placas de 48 pocillos y el portaobjetos de la cámara de fondo de vidrio de 8 pocillos con 200 μL de colágeno humano al 2,5% en agua por pozo durante 1 h a 37 °C.
NOTA: Los organoides intestinales se siembran mejor en placas de 48 pocillos y en diapositivas de cámara de fondo de vidrio de 8 pocillos. La experiencia ha demostrado que no infectan bien en placas de 96 pocillos. Los insertos Transwell también se pueden usar para permitir la infección apical y basolateral en 2D. Si se utilizan transpocillos, controle la resistencia eléctrica transepitelial (TEER) antes de la infección para confirmar una monocapa confluente. Normalmente, los organoides intestinales tienen una barrera estrecha con un TEER de 450-600 Ohm/cm2. - Separe 100-150 organoides en cada pozo de una placa de 48 pocillos o para un pozo de un portaobjetos de cámara de fondo de vidrio de 8 pocillos.
- Para estimar el número de organoides, cuente el número de organoides presentes en la gota de solución de ECM de 50 μL de la placa de 24 pocillos preparada en la sección 1. En promedio, se necesitan 1-2 pozos, lo que dará aproximadamente 15,000-30,000 células.
- Para interrumpir la solución de ECM y tripsinizar los organoides, siga los pasos 1.8-1.17.
- Retire el colágeno humano de las placas de 48 pocillos y de la corredera de la cámara de fondo de vidrio de 8 pocillos.
- Resuspender el pellet organoide en el tubo cónico en 250 μL de medio de crecimiento/pozo y añadir la mezcla a los pocillos recubiertos de colágeno. Coloque la placa en una incubadora de 37 °C.
NOTA: Al realizar los experimentos, se comparan múltiples condiciones (simulacro vs. infectado +/- tratamiento de interés). Para minimizar la variabilidad entre cada pocillo de organoides sembrados en 2D, recoja el número total de organoides necesarios en el mismo tubo cónico. La recolección de organoides de hasta 12 pocillos de una placa de 24 pocillos puede usar 1 ml de tripsina y 2 ml de medios neutralizantes. Cuando use 12-24 pocillos, aumente esto a 2 ml de tripsina y 4 ml de medios neutralizantes. - Después de 48 h, retire la placa de la incubadora y colóquela en la campana de cultivo celular. Retire el medio de crecimiento y reemplácelo por 250 μL por pocillo de medio de diferenciación (Tabla 1). Repita este cambio de medios 48 h más tarde.
- Después de 48 h, confirme la diferenciación (cuatro días después del cambio a medios de diferenciación) extrayendo ARN, haciendo ADNc con 250 ng de ARN y realizando qPCR basada en verde SYBR utilizando cebadores para células madre (OLFM4 y / o SMOC2), células caliciformes (MUC2), células enteroendocrinas (CHGA) y enterocitos (SI y / o CYP3A4) (Figura 3).
NOTA: Al cambiar de medios de crecimiento a medios de diferenciación, los organoides disminuirán su expresión de marcadores de células madre (OLFM4 y / o SMOC2) y aumentarán la expresión de células caliciformes (MUC2), células enteroendocrinas (CHGA) y enterocitos (SI y / o CYP3A4) marcadores por qPCR. Prepare un pozo adicional y manténgalo con medios de crecimiento para comparar el nivel de expresión de cada marcador específico del tipo de célula en los medios de crecimiento frente a los medios de diferenciación. - Tras la confirmación de la diferenciación, los organoides están listos para la infección apical. Mover los organoides al nivel de contención de bioseguridad requerido para el patógeno de elección.
PRECAUCIÓN: Consulte las regulaciones locales del instituto y los procedimientos operativos estándar cuando manipule patógenos bajo contención BSL-2 o BSL-3. - Para infectar los organoides intestinales cultivados en 2D desde su lado apical, retire los medios con una pipeta P1000 y agregue el patógeno diluido a la multiplicidad de infección requerida para el experimento en medios de diferenciación (el volumen mínimo de mezcla de patógeno / medio es de 50 μL por pozo y el volumen máximo es de 250 μL por pozo).
- Permita que la infección proceda durante 2 h a 37 ° C con una mesa basculante ubicada en la incubadora de cultivo celular o meciendo manualmente la placa cada 15-20 min. Optimice este tiempo en función del patógeno de su elección.
- Después de 2 h, retire el medio con una pipeta P1000 y reemplácelo con 250 μL por pocillo de medio de diferenciación fresco. Incubar células a 37 °C hasta el punto temporal de secuenciación unicelular.
- Para preparar las células para la secuenciación unicelular, continúe con la sección 4.
3. Preparación de organoides en tres dimensiones (3D) para la infección apical y basolateral
- Cultivar organoides como se describe en la sección 1 en una placa tratada con cultivo no celular de 24 pocillos.
- Dos días después de la transmisión, retire la placa de la incubadora y colóquela en una campana de cultivo celular.
- Retire el medio de crecimiento con una pipeta P1000, reemplácelo por 500 μL/pocillo de medio de diferenciación precalentado a temperatura ambiente y coloque la placa en la incubadora de 37 °C.
- Después de 48 h, reemplazar con medios de diferenciación frescos (500 μL/pozo).
NOTA: Los organoides se mantienen en medios de diferenciación durante un total de 4 días antes de la infección. - Confirme la diferenciación como se describe en el paso 2.9.
- Tras la confirmación de que se ha producido la diferenciación, los organoides están listos para la infección.
- Descongelar ECM sobre hielo. Caliente el medio de diferenciación a temperatura ambiente y caliente una placa de 24 pocillos a 37 °C.
- Para realizar infecciones, retire los organoides de la solución de ECM.
NOTA: Elimine la mayor cantidad posible de solución de ECM, ya que los virus prefieren adherirse a la solución de ECM en lugar de a las células. Si queda demasiada solución de ECM alrededor de los organoides, la infectividad se verá gravemente afectada. - Interrumpir la ECM agregando 500 μL de PBS frío 1x e incubando durante 3 min. Use un P1000 para pipetear hacia arriba y hacia abajo 10x. Combine los organoides en un tubo y centrífuga a 500 x g durante 5 min. Retire PBS con un P1000.
NOTA: Para minimizar la variabilidad entre cada condición de infección, combine los organoides provenientes de varios pocillos de una placa de 24 pocillos en el mismo tubo cónico. Por ejemplo, si se requieren ocho condiciones para la infección, se combinarán ocho pocillos de una placa de 24 pocillos (que contienen ~ 100 organoides cada uno) y posteriormente se dividirán en ocho pocillos de una nueva placa de 24 pocillos después de la interrupción de la aguja (consulte el paso 3.12). - Resuspendir los organoides en 1 mL de medios de diferenciación. Para la infección apical y basolateral siga el paso 3.10.1 y para la infección basolateral sólo siga el paso 3.10.2.
- Conecte una aguja de 27 G a una jeringa de 1 ml y vuelva a suspender el gránulo seis veces para permitir la interrupción de los organoides y que se produzca una infección tanto en el lado apical como en el basolateral. Continúe con el paso 3.11.
NOTA: Los organoides deben ser los organoides clásicos de aspecto de quiste y brotes de pulpo con un centro oscuro cuando se observan antes de la interrupción de la aguja. Después de la interrupción, estos organoides parecerán más pequeños con menos o ningún interior oscuro. Al realizar la infección, siempre habrá un mínimo de dos condiciones (simulacro e infección), un mínimo de dos pocillos de una placa de 24 pocillos se combinará inicialmente en un tubo cónico de 15 ml para la interrupción basada en agujas (lo que explica por qué se usa un mínimo de 1 ml para la resuspensión). Cuando realice más de dos condiciones, combine hasta 10 pocillos de una placa de 24 pocillos en un solo tubo cónico de 15 ml y resuspenda en 1 ml de medios de diferenciación para la interrupción de la aguja. Después de la interrupción, agregue 500 μL de medios de diferenciación por n + 1 (por ejemplo, si usa 10 pozos, luego recargue con 4.5 mL para tener un total de 5.5 mL). Si se necesitan más de 10 condiciones, use varios tubos cónicos. Se recomienda una jeringa de 1 ml ya que los organoides se interrumpen mejor con este volumen de la jeringa. Una alternativa a una jeringa es emplear una punta P1000 bien aplanada. - Resuspendir los organoides en 500 μL de medios de diferenciación por pozo. Tenga cuidado de no interrumpir los organoides y asegúrese de que el lado apical no sea accesible. Continúe con el paso 3.11.
- Conecte una aguja de 27 G a una jeringa de 1 ml y vuelva a suspender el gránulo seis veces para permitir la interrupción de los organoides y que se produzca una infección tanto en el lado apical como en el basolateral. Continúe con el paso 3.11.
- Transfiera 500 μL de suspensiones organoides por pozo a una placa de cultivo celular de 24 pocillos utilizando una pipeta P1000 y mueva la placa al nivel de contención de bioseguridad requerido para el patógeno de elección.
PRECAUCIÓN: Consulte las regulaciones locales y los procedimientos operativos estándar del instituto al manipular patógenos bajo contención BSL-2 o BSL-3. Siempre realice la interrupción de la aguja de los organoides fuera del BSL-3 para evitar posibles accidentes con la aguja. Las regulaciones locales también pueden impedir el uso de agujas en el BSL-3. - Agregue el patógeno diluido en medios de diferenciación para alcanzar la multiplicidad de infección requerida para el experimento. No exceda un volumen total de 500 μL (para tener 1 ml en total en la placa de 24 pocillos).
- Permita que la infección continúe durante 2 h (esta vez deberá adaptarse al patógeno de elección) a 37 ° C con una mesa basculante ubicada en la incubadora de cultivo celular o meciendo manualmente la placa cada 15-20 min.
- Después de la incubación de 2 h, recoja los organoides en un tubo de 1,5 ml por condición y gire a 500 x g durante 5 minutos a 4 °C.
- Retire el medio que contiene el patógeno con una pipeta P1000 y guárdelo en hielo. Lave el pellet de organoides una vez con PBS.
- Resuspendir los organoides en 50 μL de solución de ECM al 100% (que previamente se descongeló en hielo), placa en una placa tratada con cultivo no celular de 24 pocillos precalentada a 37 °C e incubar la placa de 24 pocillos a 37 °C durante 10-15 min para permitir que la solución de ECM se polimerice.
- Después de la polimerización, agregue 500 μL de medios de diferenciación a temperatura ambiente a cada pozo e incube a 37 ° C hasta la cosecha para la secuenciación de una sola célula.
NOTA: Si la cosecha debe ocurrir más de 48 h después de la siembra, cambie el medio cada 2 días retirando el medio viejo y reemplazándolo con 500 μL de medios de diferenciación frescos. Sin embargo, la experiencia ha demostrado que dado que los organoides se cultivan bajo medios de diferenciación, no sobrevivirán mucho más de 48 h.
4. Preparación de solución unicelular y preparación de perlas de gel en emulsión (GEM) en condiciones de nivel 3 de bioseguridad (BSL-3)
NOTA: La finalización de los siguientes pasos requiere que el equipo de secuenciación de una sola célula(Tabla de Materiales)y una máquina de PCR capaz de manejar reacciones de 100 μL estén presentes dentro de una instalación BSL-3. Los organoides se cultivan como se describe en la sección 1 y se infectan como se describe en las secciones 2-3 dependiendo del patógeno y la ruta de entrada. En un momento predeterminado después de la infección, los organoides se cosechan. A continuación se describe el método utilizado para cosechar organoides intestinales.
- Antes de extraer organoides infectados, retire las perlas de gel unicelular(Tabla de materiales)de -80 ° C y caliente a temperatura ambiente (al menos 30 minutos antes).
- Además, equilibre el reactivo RT, el agente reductor B y la enzima RT C (todos almacenados a -20 °C) a temperatura ambiente. Vuelva a suspender el oligo del interruptor de plantilla como se describe en las instrucciones del fabricante.
NOTA: Todos los siguientes pasos deben realizarse con respecto a las regulaciones locales de bioseguridad siguiendo el procedimiento operativo estándar establecido para el patógeno considerado. Como regla general para el trabajo BSL-3, se debe garantizar que el exterior de todos los recipientes (placas, tubos, etc.) que salen de una campana de cultivo celular se desinfecten adecuadamente. Lo mismo se aplica a las manos / guantes de la persona que realiza los experimentos. - Para realizar infecciones en organoides sembrados en 2D (sección 2) se procede al paso 4.4. Para realizar infecciones en organoides 3D (sección 3) se procede a las secciones 4.5.
- Para las infecciones de organoides 2D, lleve la placa de cultivo celular a la campana y retire el medio de diferenciación con una pipeta P1000.
- Agregue 250 μL de 1x PBS a temperatura ambiente a cada pozo.
- Retire el PBS con una pipeta P1000 y agregue 250 μL de enzima de disociación a temperatura ambiente (por ejemplo, TrypLE Express) a cada pocillo de una placa de 48 pocillos. Cámbiese los guantes, limpie la placa y colóquela con la enzima de disociación en la incubadora de 37 °C.
- Observar la disociación celular por microscopía cada 5 min.
NOTA: Aproximadamente, se tarda 15 minutos en disociar los organoides intestinales cultivados en 2D en células individuales. Este tiempo deberá ajustarse, ya que dependerá de cuántos organoides se sembraron e infectaron inicialmente, así como del patógeno, ya que algunos patógenos son más citopáticos y hacen que los organoides se disocien mucho más rápido que otros. - Después de la confirmación de una suspensión aparente de una sola célula, lleve la placa de vuelta a la campana de cultivo celular y detenga la digestión agregando 250 μL de medios DMEM / F12 que contengan 10% de FBS por pocillo de una placa de 48 pocillos.
- Recoge las células en un tubo cónico de 15 ml usando una pipeta P1000.
- Cambie los guantes, limpie el tubo y haga girar las muestras a 500 x g durante 5 minutos a 4 °C.
- Retire cuidadosamente el medio / enzima de disociación con un P1000 para mantener el gránulo celular en la parte inferior. Resuspend células individuales en un volumen mínimo de PBS que contiene 0.1% BSA. Para los organoides intestinales, resuspend en 250 μL por cada pocillo de la placa de 48 pocillos.
- Pase las suspensiones celulares en un tubo FACS con un filtro para eliminar cualquier grupo grande y coloque el tubo FACS que contiene células en hielo.
- Continúe con el paso 4.6 para continuar con el conteo de celdas y la preparación de la mezcla maestra.
- Para las infecciones de organoides 3D, retire la placa de la incubadora y colóquela en la campana de cultivo celular.
- Retire el medio de diferenciación de cada pocillo de la placa de 24 pocillos con una pipeta P1000. Agregue 500 μL de PBS helado 1x a cada pozo e incube durante 3 minutos en hielo.
- Para garantizar la interrupción total de la solución ECM, utilice una pipeta P1000 (establecida en 450 μL). Pipetear hacia arriba y hacia abajo 10 veces para resuspendir el PBS, la solución de ECM y los organoides; transfiera los organoides resuspendidos a un tubo cónico de 15 ml y colóquelos en hielo. Recoger cada condición de infección en su propio tubo cónico de 15 ml.
NOTA: El uso de un tubo cónico de 15 ml proporciona un gránulo celular mejor y más distinto que un tubo cónico de 50 ml o un tubo de 1,5 ml. - Cámbiese los guantes y retire el tubo de la campana de cultivo celular. Limpie el exterior del tubo.
- Girar las muestras a 500 x g durante 5 min a 4 °C.
- Mueva el tubo de nuevo a la campana de cultivo celular y retire el PBS con una pipeta P1000 para evitar la resuspción del pellet de organoides de la parte inferior del tubo.
- Resuspend el pellet en 1 ml de enzima de disociación (por ejemplo, TrypLE Express). Cámbiese los guantes, limpie el tubo e incube las muestras a 37 °C.
- Cada 10 minutos durante 30 minutos, mueva el tubo hacia atrás en la campana de cultivo celular y resuspenda los organoides mediante la pipeteación hacia arriba y hacia abajo con una pipeta P1000 10 veces.
NOTA: Normalmente, los organoides intestinales requieren alrededor de 30 minutos para formar una suspensión de una sola célula cuando se infectan en 3D. - Para determinar cuándo los organoides se disocian a células individuales, tome 10 μL de la suspensión organoide con una pipeta p10.
- Coloque la suspensión en un portaobjetos de plástico desechable. Selle el puerto de entrada de muestra con cinta transparente.
- Cámbiese los guantes y limpie el exterior del mostrador de la celda. Use un microscopio de campo brillante para determinar si se fabrica una suspensión de una sola célula.
- Después de la confirmación de una suspensión de una sola célula, detenga la digestión agregando 1 ml de medios DMEM / F12 que contengan 10% de FBS y pipetee hacia arriba y hacia abajo 10 veces con una pipeta P1000.
- Cambie los guantes, limpie el tubo y haga girar las muestras a 500 x g durante 5 minutos a 4 °C.
- Retire cuidadosamente el medio / enzima de disociación con una pipeta P1000 para evitar la resuspción del pellet celular desde la parte inferior del tubo.
- Resuspend las células individuales en un volumen mínimo de PBS que contenga 0.1% de BSA. Para organoides intestinales resuspend en 250 μL.
- Pase las suspensiones celulares en un tubo FACS con un filtro para eliminar cualquier grupo grande y coloque el tubo FACS que contiene células en hielo.
- Continúe con el paso 4.6 para continuar con el conteo de celdas y la preparación de la mezcla maestra.
- Determine el número de células por μL agregando 10 μL de la suspensión celular a una cámara de conteo de células de plástico desechable.
- Selle el puerto de entrada de muestra con cinta transparente antes de retirar la muestra de la campana de cultivo celular, ya que en esta etapa, la suspensión celular sigue siendo infecciosa.
- Cambie los guantes, limpie la cámara de conteo de células y cuente el número de células con un microscopio de campo brillante.
- Dentro de la campana de cultivo celular, prepare una mezcla maestra de reactivo RT, oligo interruptor de plantilla, agente reductor B y enzima RT C en un tubo de 1,5 ml según las instrucciones del fabricante, dependiendo del número de muestras en el experimento. Para cada muestra, alícuota 33,4 μL de mezcla maestra en un tubo de PCR y guárdela en hielo.
- Agregue las celdas y el agua a la mezcla maestra de acuerdo con el número de celda objetivo como se describe en las instrucciones del fabricante.
NOTA: el 50%-60% de las células se recuperan normalmente (es decir, cuando se cargan 10.000 células en el chip, se utilizan 5.000-6.000 células para el análisis). Por lo tanto, siempre cargue 10,000 celdas. Si la densidad celular no lo permite, centrífuga las células y vuelva a suspenderlas en un volumen menor. Tenga cuidado de no sobrecargar el chip, ya que esto resultará en la obstrucción del chip. Además, si las células no se disocian adecuadamente, habrá un mayor riesgo de obtener múltiples células por cuenta, que deberán eliminarse en el procesamiento bioinformático posterior. - Cambie los guantes, mueva el controlador de una sola celda a la campana de cultivo celular y prepare el chip ya que el chip solo está cubierto con una junta que no está sellada.
NOTA: La máquina debe estar dentro de la campana para evitar la exposición a suspensiones celulares infectadas y posibles aerosoles. - Agregue el chip de una sola celda al soporte del chip y llene los carriles no utilizados con 50% de glicerol.
- Agregue la mezcla maestra, las perlas y el aceite de partición a los carriles utilizados para las muestras según las instrucciones del fabricante.
- Cubra el chip con la junta, cargue el chip en el controlador e inicie el programa.
NOTA: Se recomienda cargar solo seis de los ocho carriles. Los problemas con emulsiones inadecuadas a menudo ocurren en los carriles uno y ocho. La empresa dice que todos los carriles son independientes, y esto no debería ocurrir; sin embargo, si es posible, evite estos dos carriles y ejecute dos chips si se necesitan ocho muestras. - Al finalizar el programa, retire el chip y la junta.
- Utilice una pipeta multicanal y transfiera 100 μL de las emulsiones a un tubo de PCR limpio. Asegúrese de que cada pozo tenga un color blanco uniforme que indique que se ha producido una emulsión completa.
- Cambie los guantes, limpie los tubos y transfiera el tubo de PCR a una máquina de PCR que pueda soportar reacciones de 100 μL. Ejecute el programa: 53 °C durante 45 min; 85 °C durante 5 min.
- Al finalizar, conservar las muestras a 4 °C. En este punto, procese la reacción de acuerdo con las instrucciones del fabricante o guárdela a 4 °C durante 3 días o -20 °C durante 1 semana.
- Después de 5 min a 85 °C, la mayoría de los virus envueltos se inactivarán, eliminarán el ADNc del BSL-3 de acuerdo con el procedimiento operativo normal y realizarán las preparaciones de la biblioteca en un laboratorio BSL-1.
NOTA: Este experimento debe realizarse como tres réplicas biológicas (por ejemplo, en tres días diferentes) porque el alcance de la diferenciación de cada tipo de célula puede variar ligeramente entre cada experimento. Las bibliotecas de secuenciación resultantes podrían indexarse de manera diferente y secuenciarse juntas en una ejecución de secuenciación.
Resultados
Preparación de organoides para la secuenciación unicelular
Los resultados de la secuenciación de una sola célula dependen en gran medida del uso de células de buena calidad. Para garantizar que los organoides sean de buena calidad, deben mantenerse y observarse adecuadamente a diario para determinar cuándo están listos para ser divididos(Figura 2). El momento de la división de los organoides depende del donante; algunos donantes crecen más rápidamente y necesitan ser divididos cada 5 días, mientras que otros son más lentos y necesitan ser divididos cada 10 días. En promedio, los organoides se dividen una vez por semana cuando los centros se oscurecen(Figura 2B). Si se permite que los organoides se vuelvan demasiado grandes y acumulen demasiadas células muertas en el centro, el organoide morirá.
Los organoides se mantienen en un medio que contiene altas cantidades de Wnt3A. Esto apoya el nicho de células madre y promueve que los organoides continúen creciendo y proliferando. Bajo estas condiciones de crecimiento, los organoides contienen altas cantidades de células madre y células amplificadoras de tránsito y una menor cantidad de poblaciones celulares diferenciadas como enterocitos maduros, células caliciformes y células enteroendocrinas. Sin embargo, para imitar la complejidad celular que se encuentra dentro del intestino humano, es importante impulsar la diferenciación celular y producir más de estas células. Esto se logra cambiando las condiciones de los medios y eliminando Wnt3A, y reduciendo R-Spondin y Noggin (Tabla 1). Normalmente, la diferenciación celular hacia enterocitos, células caliciformes y células enteroendocrinas requiere 4 días de medios de diferenciación(Figura 3). Es clave para obtener una buena diferenciación; de lo contrario, la evaluación del tropismo del patógeno y las respuestas específicas del tipo celular se volverá difícil.
Confirmación de la inactivación del patógeno BSL-3
La inactivación completa debe confirmarse con el patógeno de elección y validarse que es seguro eliminar el ADNc del BSL-3. Para el SARS-CoV-2, la inactivación completa del virus se validó tomando 100 μL de SARS-CoV-2 e incubándolo en una máquina de PCR durante 5 min a 85 °C. El virus se agregó de nuevo a las células Vero naïve, y la infección por el virus se comparó con el virus no tratado térmicamente mediante inmunofluorescencia y ensayos de placa a las 24 h, 48 h y 72 h después de la infección para garantizar que todas las partículas ya no fueran infecciosas. Estos resultados se enviaron a la agencia reguladora local, y tras su aprobación se realizó el experimento de una sola célula y el procesamiento de ADNc.
Resultados de la secuenciación unicelular
Para evaluar cómo el SARS-CoV-2 infecta los organoides humanos del colon y el íleon, se realizó la secuenciación unicelular. Los organoides se prepararon como se describió anteriormente y se infectaron en un formato 2D para permitir la infección apical por SARS-CoV-2. Las células infectadas se recolectaron a las 12 h y 24 h después de la infección y se procesaron para la secuenciación de una sola célula como se describió anteriormente. El análisis de los datos de secuenciación unicelular nos permitió determinar que solo una subpoblación (enterocito inmaduro 2) de células epiteliales intestinales humanas soportaba la infección por SARS-CoV-2(Figura 4). Además, como no todas las células de una población estaban infectadas, se analizaron tanto las células infectadas como las células transeúntes no infectadas(Figura 5). Estos resultados mostraron que el SARS-CoV-2 indujo una cascada de señales proinflamatorias en las células infectadas, mientras que las células espectadoras no infectadas mostraron una respuesta inmune mediada por interferón. Además, scRNA-Seq mostró que las células infectadas no podían detectar interferones debido al bloqueo de la vía mediado por virus(Figura 5). No fue posible obtener esta información cuando se utilizó la secuenciación masiva de ARN.

Figura 1:Esquema que representa los tres métodos diferentes para preparar organoides intestinales humanos para la infección con patógenos entéricos. La infección apical se puede lograr a través de la siembra de organoides intestinales en 2D. Una infección apical y basolateral se puede realizar mediante la interrupción del organoide 3D. Finalmente, una infección basolateral solo se puede realizar infectando organoides intestinales 3D intactos. Cada uno de estos métodos se puede utilizar para generar muestras para la secuenciación de una sola célula. Haga clic aquí para ver una versión más grande de esta figura.
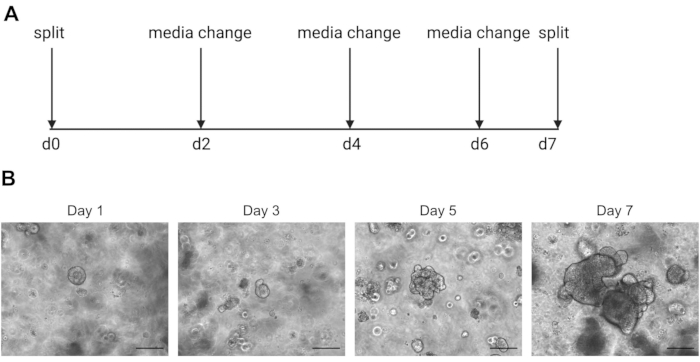
Figura 2: Esquema de mantenimiento de organoides e imágenes representativas de campo brillante. (A) Esquema para el mantenimiento y el paso de organoides intestinales humanos. (B). Imágenes representativas de campo brillante de los días 1, 3, 5 y 7 después de la división. Para el día 7 los organoides se vuelven grandes y oscuros debido a la acumulación de células muertas y están listos para ser divididos. La barra de escala indica 25 μm. Haga clic aquí para ver una versión más grande de esta figura.
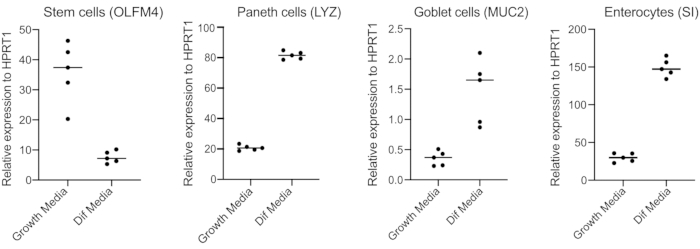
Figura 3: qPCR representativa de organoides intestinales humanos 4 días después del cambio a medios de diferenciación. Los organoides intestinales se mantuvieron en medios de crecimiento o se cambiaron a medios de diferenciación durante 4 días. Se recolectó ARN y se realizó qPCR para marcadores de células madre (OLFM4), células de Paneth (LYZ), células caliciformes (MUC2) y enterocitos (SI). N = 5. Haga clic aquí para ver una versión más grande de esta figura.
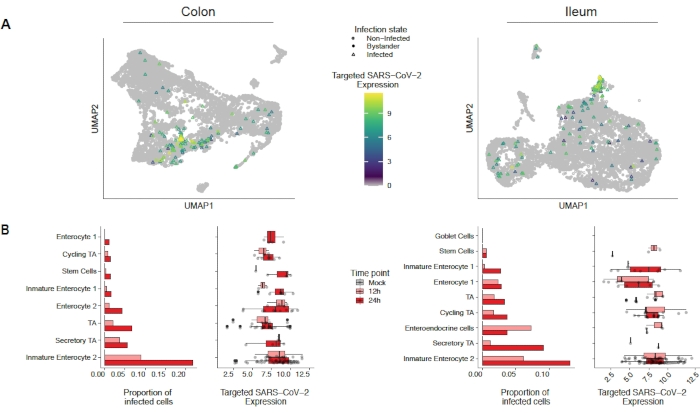
Figura 4: Identificación de la población celular infectada por sars-CoV-2. Los organoides derivados del colon humano y del íleon fueron infectados con SARS-CoV-2. Después de 12 y 24 h después de la infección, las células fueron recolectadas y sometidas a secuenciación de ARN unicelular para identificar qué poblaciones celulares soportaban la infección por SARS-CoV-2. Se encontró que la infección por el virus aumenta con el tiempo e infecta principalmente enterocitos inmaduros 2. Esta cifra ha sido modificada a partir de Triana et al.19. Haga clic aquí para ver una versión más grande de esta figura.
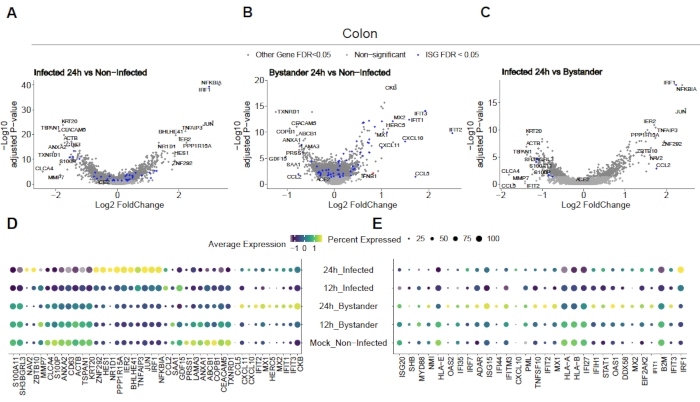
Figura 5: Determinación de la respuesta inmune innata intrínseca. Los organoides derivados del colon humano fueron infectados con SARS-CoV-2. Después de 12 y 24 h, las células posteriores a la infección se recolectaron y se sometieron a una secuenciación de ARN unicelular para determinar la respuesta inmune innata intrínseca tanto en células transeúntes infectadas viralmente como no infectadas. Las células infectadas por SARS-CoV-2 mostraron una fuerte respuesta proinflamatoria, mientras que las células transeúntes no infectadas mostraron una respuesta mediada por interferón. Figura modificada de Triana et al.19. Haga clic aquí para ver una versión más grande de esta figura.
| Medios de crecimiento | |
| Compuesto | Concentración final |
| Anuncio DMEM/F12 | GlutaMAX (1X) |
| +GlutaMAX | HEPES 1 mM |
| +HEPES | Pluma 10 U/mL |
| +P/S | Estreptococo 10 μg/mL |
| L-WRN | 50% en volumen |
| B27 | 01:50 |
| N-acetil-cisteína | 1 mM |
| FEAG | 50 ng/ml |
| A83-01 | 500 nM |
| IGF-1 | 100 ng/ml |
| FGF básico | 50 ng/ml |
| Gastrina | 10 mM |
| Medios de diferenciación | |
| Compuesto | Concentración final |
| Anuncio DMEM/F12 | GlutaMAX (1x) |
| +GlutaMAX | HEPES 1 mM |
| +HEPES | Pluma 10 U/mL |
| +P/S | Estreptococo 10 μg/mL |
| B27 | 01:50 |
| N-acetil-cisteína | 1 mM |
| R-espondina | 5% en volumen |
| Cabeza | 50 ng/ml |
| FEAG | 50 ng/ml |
| Gastrina | 10 mM |
| A83-01 | 500 nM |
Tabla 1: Composición de medios para medios de crecimiento y diferenciación.
Discusión
Los patógenos entéricos con mayor frecuencia inician su ciclo de vida infectando las células epiteliales intestinales desde su lado apical frente a la luz del intestino. Si bien los organoides son bien reconocidos como un buen modelo para reproducir la complejidad celular y la organización del epitelio intestinal, su organización como estructuras tridimensionales y cerradas hace que su membrana apical sea inaccesible para el patógeno. Este protocolo describió métodos para infectar organoides intestinales desde su lado apical, su lado basolateral o ambos con patógenos BLS-3. Estos protocolos se pueden adaptar fácilmente para estudiar cualquier patógeno entérico bajo contención de BSL-2 o BLS-3 o cualquier otro modelo organoide siguiendo algunos pasos críticos que se destacan a continuación. El método descrito anteriormente es para el aislamiento y la preparación de gotas unicelulares de acuerdo con las regulaciones en Alemania. Como descargo de responsabilidad, este protocolo no describe las medidas de manejo de bioseguridad (procedimientos operativos estándar) que deben tomarse mientras se trabaja en condiciones BSL-3. También es importante insistir en que las regulaciones pueden variar en otros países y que se debe contactar a las autoridades locales para asegurarse de que se respeten todas las regulaciones locales.
Uno de los pasos críticos en la siembra de organoides en dos dimensiones para la infección apical es controlar que las células se diferencien de manera similar en comparación con cuando se cultivan como organoides tridimensionales clásicos. Dependiendo del patógeno entérico, el tropismo podría restringirse a células muy raras o a células que necesitan ser altamente diferenciadas. En este caso, el uso de un organoide bidimensional que no se diferenció completamente podría resultar en la conclusión errónea de que este patógeno entérico no puede infectar los organoides intestinales. Se sugiere, si es posible, realizar infecciones utilizando las tres configuraciones de este protocolo: organoide 2D solo para infección apical (sección 2), organoides 3D abiertos para infección apical y basolateral (sección 3) y organoides 3D completos solo para infección basolateral (sección 3). Este enfoque ayudará a discernir la ruta de entrada del patógeno (apical vs. basolateral) y también controlará que se haya logrado un nivel similar de diferenciación. Una alternativa para la infección apical 2D es la microinyección, que utilizará un organoide 3D pero entregará el patógeno directamente en el lado apical (ver Bartfeld et al.27 para más detalles). Este método requiere un inyector experto para garantizar que el patógeno se coloque correctamente y que los organoides permanezcan intactos. La microinyección se usa comúnmente en la contención de BSL-2 y puede no ser adecuada para la contención de BSL-3.
Una consideración importante adicional al realizar experimentos de infección en organoides sembrados en 2D es la densidad celular final. Como se mencionó en el paso 2.3, se sembrarán 100-150 organoides en un pozo de una placa de 48 pocillos o un pozo de un tobogán de cámara de fondo de vidrio de 8 pocillos. Dependiendo de la línea organoide y de la persona que maneja los organoides, el tamaño de estos organoides puede ser significativamente diferente. Esto podría resultar en densidades celulares muy diferentes en la placa de 48 pocillos o en el tobogán de la cámara de fondo de vidrio de 8 pocillos. Dependiendo del virus entérico, algunos virus prefieren células más escasas, mientras que otros también podrán infectar células confluentes. El origen molecular de tales diferencias en la infectividad para diferentes confluencias celulares no está claro; por lo tanto, los experimentos piloto destinados a encontrar la mejor densidad celular para el patógeno entérico de elección deben realizarse antes de realizar una mayor caracterización aguas abajo.
A menudo, la clasificación FACS se realiza antes de realizar la emulsión de gotas de una sola célula. Este paso se utiliza a menudo para separar las células muertas de las células vivas y las células individuales de los dobletes. Cuando se trabaja con patógenos BSL-3, se requiere que la instalación esté equipada con un clasificador FACS apropiado, lo que no suele ser el caso. Además, no todas las células de un organoide tienen el mismo tamaño, y a menudo es difícil discriminar entre un doblete o una célula más grande, lo que causa un riesgo de selección negativa contra un tipo de célula específico. Además, todavía hay discusión en el campo sobre si el tiempo necesario para clasificar entre 5.000-10.000 para cada muestra podría resultar en una modificación significativa del perfil de transcripción de las células individuales. Si bien se han descrito métodos de fijación celular compatibles con la secuenciación unicelular (por ejemplo, metanol y RNAassist), se observó que esto conduce a una disminución en la calidad de la secuenciación18. Finalmente, se sospecha que la clasificación de células utilizando marcadores de muerte celular también puede conducir a un sesgo. Dada la proliferación direccional y la diferenciación de las células a través del eje cripta-vellosidad, las células más diferenciadas, que van a ser desprendidas y liberadas, se encuentran en la punta de las vellosidades. Estas células a menudo son positivas para diferentes marcadores de vías de muerte celular (porejemplo, apoptosis, necrosis y necroptosis); sin embargo, cuando se observa la infección por rotavirus del intestino del ratón, la punta de las vellosidades es el área más infectada28. Por lo tanto, filtrar las células que pueden parecer positivas para los marcadores de muerte resultaría en una selección negativa de las células infectadas que pueden representar la infección fisiológica. Actualmente, no existe una buena solución para clasificar y fijar organoides antes de la secuenciación de una sola célula. Se recomienda el uso de células vivas y sin clasificar, ya que se necesitan más estudios para encontrar protocolos alternativos adecuados.
La secuenciación de una sola célula ha revolucionado la forma en que se pueden evaluar las respuestas celulares. Esta técnica permite la identificación de respuestas específicas del linaje celular tanto en condiciones basales como bajo infecciones por patógenos. Este método ha abierto puertas en muchos campos que antes estaban limitados por lecturas masivas. Si bien este método es muy poderoso, tiene sus limitaciones. Una limitación clave es el extenso análisis bioinformático que se requiere aguas abajo de la secuenciación. Esto es especialmente clave cuando se analizan tejidos y se asignan tipos de células donde actualmente no hay anotación. Se requiere tener un bioinformático capacitado para apoyar todos los estudios de una sola célula.
Este protocolo describe cómo sembrar y manejar organoides intestinales humanos, infectarlos con patógenos entéricos y realizar scRNAseq. Ahora es posible adaptar este enfoque a otros órganos, ya que se han desarrollado sistemas modelo de organoides para la mayoría de los órganos. Los organoides pulmonares e hepáticos están organizados de manera similar en comparación con los organoides intestinales, y como tales, utilizando un enfoque análogo podrían transponerse a estos organoides. El control crítico será validar que cuando se cultivan en dos dimensiones o se abren, estos organoides logran una diferenciación similar a la de sus contrapartes organoides 3D. Las características específicas y los genes que definen un estado diferenciado son específicos para cada modelo de órgano. Otros modelos de organoides, como los organoides renales y vasculares, grandes estructuras densas, necesitarán métodos adicionales para disociar en serie estas estructuras en células individuales.
Divulgaciones
Los autores no declaran intereses financieros contrapuestos.
Agradecimientos
Megan Stanifer y Steeve Boulant fueron apoyadas por becas de investigación de la Deutsche Forschungsgemeinschaft (DFG): (Proyecto número 240245660, 278001972, 415089553 y 272983813 a Steeve Boulant y 416072091 a Megan Stanifer), el estado de Baden-Wuerttemberg y el Bundesministerium für Bildung und Forschung BMBF 01KI20239B a MS y 01KI20198A y (NUM-COVID 19, Organo-Strat 01KX2021) a SB. Los esquemas se crearon en BioRender.
Materiales
| Name | Company | Catalog Number | Comments |
| Recombinant mouse noggin | Peprotech | Cat#250-38 | |
| [Leu15]-Gastrin I | Sigma-Aldrich | Cat# G9145 | |
| 0.05% Trypsin-EDTA | Thermo Fischer Scientific | Cat#25300054 | |
| 24-well non-cell culture treated plate | Corning | Cat#3738 | |
| 8-well glass bottom chamber slide | iBIDI | Cart#80827 | |
| A83-01 | Tocris | Cat#2939 | |
| Advanced DMEM/F12 | Thermo Fischer Scientific | Cat# 12634010 | |
| B27 | Thermo Fischer Scientific | Cat#17504-044 | |
| Chromium Controller & Next GEM Accessory Kit | 10X Genomics | Cat#1000202 | Used in the preparation of single cell solution and preparation of Gel beads-in-emulsion (GEM) |
| Chromium Next GEM Chip G Single Cell Kit | 10X Genomics | Cat #1000127 | Used in the preparation of single cell solution and preparation of Gel beads-in-emulsion (GEM) |
| Chromium Next GEM Single Cell 3′ Kit v3.1 | 10X Genomics | Cat#1000268 | Used in the preparation of single cell solution and preparation of Gel beads-in-emulsion (GEM) |
| Collagen from human placenta | Sigma Aldrich | Cat#C5533-5MG | |
| CYP34A forward | Eurofins | GATGGCTCTCATCCCAGACTT | Primers used to check for differentiation |
| CYP3A4 reverse | Eurofins | AGTCCATGTGAATGGGTTCC | Primers used to check for differentiation |
| DMEM/F12 | Thermo Fischer Scientific | Cat#11320074 | |
| EDTA | Sigma Aldrich | Car#E9884 | |
| Fast Read 102 counting slides | Biosigma | Cat# BVS100 | |
| Fetal Bovein Serum (FBS) | Capricorn | Cat#FBS-12A | |
| GlutaMAX | Thermo Fischer Scientific | Cat# 35050061 | |
| HEPES | Thermo Fischer Scientific | Cat3 15630080 | |
| L-WRN cells | ATCC | CRL-3276 | This cell line is used to make the conditioned media containg Wnt 3A, R-Spondin and Noggin. The protocol for the production of the conditioned media can be found on the manufacterures site. |
| MatriGel. GFR, LDEV free | Corning | Cat#354230 | |
| MUC-2 forward | Eurofins | TGTAGGCATCGCTCTTCTCA | Primers used to check for differentiation |
| MUC-2 reverse | Eurofins | GACACCATCTACCTCACCCG | Primers used to check for differentiation |
| N-acetyl-cysteine | Sigma Aldrich | Cat# A9165 | |
| OLMF4 forward | Eurofins | ACCTTTCCCGTGGACAGAGT | Primers used to check for differentiation |
| OLMF4 reverse | Eurofins | TGGACATATTCCCTCACTTTGGA | Primers used to check for differentiation |
| Penicillin/Streptomycin | Thermo Fischer Scientific | Cat#15140122 | |
| Recombinant human FGF basic | Peprotech | Cat#100-18B | |
| Recombinant human IGF-1 | BioLegend | Cat#590904 | |
| Recombinant mouse EGF | Thermo Fischer Scientific | Cat# PMG8043 | |
| SI forward | Eurofins | AATCCTTTTGGCATCCAGATT | Primers used to check for differentiation |
| SI reverse | Eurofins | GCAGCCAAGAATCCCAAT | Primers used to check for differentiation |
| TrypLE Express | Thermo Fischer Scientific | Cat#12605036 | |
| Y-27632 | Caymann Chemicals | Cat#10005583 |
Referencias
- Kretzschmar, K., Clevers, H. Organoids: Modeling development and the stem cell niche in a dish. Developmental Cell. 38, 590-600 (2016).
- Sato, T., et al. Long-term expansion of epithelial organoids from human colon, adenoma, adenocarcinoma, and Barrett's epithelium. Gastroenterology. 141, 1762-1772 (2011).
- Sato, T., et al. Single Lgr5 stem cells build crypt-villus structures in vitro without a mesenchymal niche. Nature. 459, 262-265 (2009).
- Tekes, G., Ehmann, R., Boulant, S., Stanifer, M. L. Development of feline ileum- and colon-derived organoids and their potential use to support feline coronavirus infection. Cells. 9, (2020).
- Derricott, H., et al. Developing a 3D intestinal epithelium model for livestock species. Cell and Tissue Research. 375, 409-424 (2019).
- Zhou, J., et al. Infection of bat and human intestinal organoids by SARS-CoV-2. Nature Medicine. 26, 1077-1083 (2020).
- Sato, T., Clevers, H. Growing self-organizing mini-guts from a single intestinal stem cell: mechanism and applications. Science. 340, 1190-1194 (2013).
- Spence, J. R., et al. Directed differentiation of human pluripotent stem cells into intestinal tissue in vitro. Nature. 470, 105-109 (2011).
- de Graaf, M., van Beek, J., Koopmans, M. P. Human norovirus transmission and evolution in a changing world. Nature Reviews Microbiology. 14, 421-433 (2016).
- Leshem, E., et al. Real-world effectiveness of pentavalent rotavirus vaccine among bedouin and jewish children in southern Israel. Clinical Infectious Diseases : An Official Publication of the Infectious Diseases Society Of America. 62, 155-160 (2016).
- Karst, S. M. The influence of commensal bacteria on infection with enteric viruses. Nature Reviews Microbiology. 14, 197-204 (2016).
- Ettayebi, K., et al. Replication of human noroviruses in stem cell-derived human enteroids. Science. 353, 1387-1393 (2016).
- de Wit, E., van Doremalen, N., Falzarano, D., Munster, V. J. SARS and MERS: recent insights into emerging coronaviruses. Nature Reviews Microbiology. 14, 523-534 (2016).
- Scaldaferri, F., et al. The thrilling journey of SARS-CoV-2 into the intestine: From pathogenesis to future clinical implications. Inflammatory Bowel Diseases. 26, 1306-1314 (2020).
- Kipkorir, V., Cheruiyot, I., Ngure, B., Misiani, M., Munguti, J. Prolonged SARS-CoV-2 RNA detection in anal/rectal swabs and stool specimens in COVID-19 patients after negative conversion in nasopharyngeal RT-PCR test. Journal of Medical Virology. 92, 2328-2331 (2020).
- Lamers, M. M., et al. SARS-CoV-2 productively infects human gut enterocytes. Science. 369, 50-54 (2020).
- Stanifer, M. L., et al. Critical role of type III interferon in controlling SARS-CoV-2 Infection in human intestinal epithelial cells. Cell Reports. 32, 107863 (2020).
- Triana, S., et al. Single-cell transcriptomics reveals immune response of intestinal cell types to viral infection. bioRxiv. , (2020).
- Triana, S., et al. Single-cell analyses reveal SARS-CoV-2 interference with intrinsic immune response in the human gut. Molecular Systems Biology. 17, 10232 (2021).
- Tang, F., et al. mRNA-Seq whole-transcriptome analysis of a single cell. Nature Methods. 6, 377-382 (2009).
- Hwang, B., Lee, J. H., Bang, D. Single-cell RNA sequencing technologies and bioinformatics pipelines. Experimental & Molecular Medicine. 50, 1-14 (2018).
- García-Rodríguez, I., Sridhar, A., Pajkrt, D., Wolthers, K. C. Put some guts into it: Intestinal organoid models to study viral infection. Viruses. 12, 1288 (2020).
- Stanifer, M. L., et al. Asymmetric distribution of TLR3 leads to a polarized immune response in human intestinal epithelial cells. Nature Microbiology. 5, 181-191 (2020).
- Lees, E. A., et al. Using human induced pluripotent stem cell-derived intestinal organoids to study and modify epithelial cell protection against salmonella and other pathogens. Journal of Visualized Experiments: JoVE. (147), e59478 (2019).
- Mahe, M. M., Sundaram, N., Watson, C. L., Shroyer, N. F., Helmrath, M. A. Establishment of human epithelial enteroids and colonoids from whole tissue and biopsy. Journal of Visualized Experiments: JoVE. (97), e52483 (2015).
- Fujii, M., et al. Human Intestinal organoids maintain self-renewal capacity and cellular diversity in niche-inspired culture condition. Cell Stem Cell. 23 (6), 787-793 (2018).
- Bartfeld, S., Clevers, H. Organoids as model for infectious diseases: Culture of human and murine stomach organoids and microinjection of Helicobacter Pylori. Journal of Visualized Experiments: JoVE. (105), e53359 (2015).
- Hernandez, P. P., et al. Interferon-lambda and interleukin 22 act synergistically for the induction of interferon-stimulated genes and control of rotavirus infection. Nature Immunology. 16, 698-707 (2015).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados