Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Ensayos de transferencia de energía de resonancia de Förster resueltos en el tiempo para la medición de proteínas STAT fosforiladas endógenas en células humanas
En este artículo
Resumen
Los protocolos de ensayo basados en células de transferencia de energía de resonancia de Förster resueltos en el tiempo se describen para la cuantificación simple, específica, sensible y robusta del transductor de señal fosforilada endógena y el activador de la transcripción (STAT) 1/3/4/5/6 proteínas en lisados celulares en un formato de 384 pocillos.
Resumen
La vía de señalización de la quinasa Janus (JAK)/transductor de señal y activador de la transcripción (STAT) desempeña un papel crucial en la mediación de las respuestas celulares a las citoquinas y los factores de crecimiento. Las proteínas STAT son activadas por fosforilación de tirosina mediada principalmente por JAK. La activación anormal de las vías de señalización STAT está implicada en muchas enfermedades humanas, especialmente el cáncer y las afecciones relacionadas con el sistema inmunológico. Por lo tanto, la capacidad de monitorear la fosforilación de la proteína STAT dentro del entorno de señalización celular nativa es importante tanto para la investigación académica como para la investigación de descubrimiento de fármacos. Los formatos de ensayo tradicionales disponibles para cuantificar las proteínas STAT fosforiladas incluyen western blotting y el ensayo de inmunoabsorción ligado a enzimas (ELISA). Estos métodos heterogéneos son intensivos en mano de obra, de bajo rendimiento y, a menudo, no confiables (específicos) en el caso de Western Blotting. Los métodos homogéneos (sin lavado) están disponibles, pero siguen siendo caros.
Aquí, se proporcionan protocolos detallados para la medición sensible, robusta y rentable en un formato de 384 pocillos de niveles endógenos de STAT1 fosforilado (Y701), STAT3 (Y705), STAT4 (Y693), STAT5 (Y694 / Y699) y STAT6 (Y641) en lisados celulares de células adherentes o de suspensión utilizando la nueva plataforma de transferencia de energía de resonancia de Förster resuelta en el tiempo (TR-FRET) THUNDER. El flujo de trabajo para el ensayo celular es simple, rápido y está diseñado para el cribado de alto rendimiento (HTS). El protocolo de ensayo es flexible, utiliza una muestra de bajo volumen (15 μL), requiere solo un paso de adición de reactivos y se puede adaptar a aplicaciones de bajo rendimiento y alto rendimiento. Cada inmunoensayo sándwich fosfo-STAT se valida en condiciones optimizadas con agonistas e inhibidores conocidos y genera los valores esperados de farmacología y factor Z'. Como los ensayos TR-FRET son ratiométricos y no requieren pasos de lavado, proporcionan una reproducibilidad mucho mejor que los enfoques tradicionales. Juntos, este conjunto de ensayos proporciona nuevas herramientas rentables para un análisis más completo de proteínas STAT fosforiladas específicas después del tratamiento celular y la detección y caracterización de moduladores específicos y selectivos de la vía de señalización JAK / STAT.
Introducción
La vía de señalización JAK/STAT juega un papel clave en la mediación de las respuestas celulares a diversas citoquinas, interferones, factores de crecimiento y moléculas relacionadas1,2. La unión de estos ligandos a receptores específicos de la superficie celular da como resultado la activación de los JAK, que a su vez activan las proteínas STAT por fosforilación de residuos específicos de tirosina. La fosforilación STAT da como resultado su dimerización y translocación en el núcleo, donde ejercen su efecto sobre la transcripción de genes diana regulados. La familia STAT consta de siete miembros: STAT1, STAT2, STAT3, STAT4, STAT5a, STAT5b y STAT6. Los miembros desempeñan un papel complejo y esencial en la regulación de los procesos celulares fisiológicos, incluida la proliferación, la diferenciación, la apoptosis, la angiogénesis y la regulación del sistema inmunológico. La activación anormal de las vías de señalización STAT está implicada en muchas enfermedades humanas, especialmente el cáncer y las afecciones relacionadas con el sistema inmunológico3,4. Por lo tanto, la capacidad de evaluar la fosforilación de la proteína STAT dentro del entorno de señalización celular nativa es importante tanto para la investigación académica como para la investigación de descubrimiento de fármacos.
Hasta la fecha, los métodos convencionales utilizados para medir los niveles de proteínas fosforiladas intracelulares, incluidos los STAT, se basan en anticuerpos e incluyen western blotting, ELISA y citometría de flujo de fósforo. Estos métodos heterogéneos requieren mucha mano de obra, consumen mucho tiempo, son propensos a errores, de bajo rendimiento y, a menudo, poco confiables (por ejemplo, problemas de especificidad) en el caso de Western Blotting5. Por el contrario, los ensayos homogéneos requieren menos pasos experimentales, utilizan volúmenes de muestra más pequeños y son susceptibles a HTS. Hay cinco plataformas homogéneas de inmunoensayo basadas en células disponibles comercialmente que se pueden usar para monitorear cuantitativamente la fosforilación dependiente de JAK de STATs en lisados celulares: SureFire, HTRF, LANCE, LanthaScreen y Lumit. Cada una de estas plataformas tiene sus ventajas y desventajas.
SureFire se basa en la tecnología de canalización de oxígeno luminiscente, que utiliza perlas de donantes y aceptores recubiertas para capturar específicamente un par de anticuerpos, uno de los cuales es biotinilado. En presencia de proteína fosforilada, los dos anticuerpos acercan las perlas donante y aceptora, lo que permite la generación de una señal quimioluminiscente6. Si bien es versátil y sensible, esta tecnología es costosa, se ve afectada por la biotina en el medio de cultivo, es muy sensible a la temperatura ambiente y la luz, y requiere un lector especial para la detección. HTRF y LANCE se basan en la tecnología TR-FRET que utiliza complejos de iones lantánidos luminiscentes de larga duración (quelatos de europio o terbio, o criptoto de europio) como moléculas donantes y fluoróforos de color rojo lejano como moléculas aceptoras7. Cuando dos anticuerpos específicos de proteínas marcados con moléculas donantes o aceptoras se acercan mucho, se produce FRET, causando un aumento en la fluorescencia del aceptor y una disminución en la fluorescencia del donante. Estas señales fluorescentes de larga duración se pueden medir de una manera termal y resuelta en el tiempo para reducir la interferencia del ensayo y aumentar la calidad de los datos. Otras ventajas de TR-FRET son que no es sensible a la luz, permite lecturas repetidas y exhibe una estabilidad de señal larga. Si bien TR-FRET está ampliamente implementado en HTS debido a su versatilidad, sensibilidad y alta robustez, todas las plataformas comerciales de ensayo basadas en TR-FRET son costosas, lo que impide su amplia adopción en laboratorios académicos y pequeños laboratorios industriales. El ensayo LanthaScreen también utiliza una lectura basada en TR-FRET, pero depende de una línea celular U2OS diseñada que expresa de manera estable la proteína de fusión verde fluorescente (GFP)-STAT1 combinada con un anticuerpo STAT1 fosfoespecífico marcado con terbio8. Además de ser limitado en términos de elección de proteínas de señalización, este método requiere la compra de costosas líneas celulares transfectadas, reduciendo su aplicabilidad y aumentando la posibilidad de artefactos experimentales. Lumit es una plataforma genérica de inmunoensayo bioluminiscente que utiliza anticuerpos secundarios (anti-ratón y anti-conejo) marcados químicamente con las subunidades nanobit pequeñas y grandes de NanoLuc Luciferase9. La unión de dos anticuerpos primarios a la proteína diana acerca a los anticuerpos secundarios para formar una enzima activa que genera una señal de luminiscencia. Si bien la luminiscencia es generalmente una lectura sensible y robusta, el requisito de anticuerpos primarios criados en dos especies diferentes limita las opciones para el diseño del ensayo. Además, el uso de anticuerpos secundarios en matrices de muestras complejas puede ser propenso a la interferencia del ensayo.
Por lo tanto, todavía existe la necesidad de una plataforma de ensayo basada en células confiable, rápida pero asequible para medir proteínas STAT fosforiladas y totales individuales de una manera compatible con HTS. Para abordar esta necesidad, se desarrolló una nueva plataforma de inmunoensayo basado en células de alto rendimiento basada en una tecnología TR-FRET mejorada (THUNDER) y diseñada para permitir una medición simple, sensible, robusta y rentable de proteínas intracelulares expresadas endógenamente (fosforiladas o totales) en lisados celulares. Las ventajas de esta tecnología se derivan de la combinación de un par FRET donante/aceptor que exhibe una compatibilidad espectral excepcional y una señal TR-FRET, anticuerpos rigurosamente validados y tampones de lisis optimizados. Estos ensayos se formatean como inmunoensayos sándwich y utilizan un flujo de trabajo sencillo de tres pasos (Figura 1). Las células se tratan primero para modular la fosforilación de proteínas y luego se lisan con el tampón de lisis específico proporcionado en el kit. La proteína STAT fosforilada o total objetivo en el lisado celular se detecta en una sola etapa de adición e incubación de reactivos con un par de anticuerpos marcados con fluoróforos que reconocen epítopos distintos en la proteína objetivo (Figura 2). Un anticuerpo está marcado con un donante de quelato de europio (Eu-Ab1), mientras que el segundo anticuerpo está marcado con un fluoróforo aceptor de color rojo lejano (FR-Ab2). Los dos anticuerpos marcados se unen a la proteína en solución, acercando las dos etiquetas. La excitación del quelato de europio del donante a 320 o 340 nm desencadena un FRET al aceptor, que emite una señal TR-FRET de larga duración a 665 nm proporcional a la concentración de proteína objetivo (fosforilada o total) en el lisado celular.
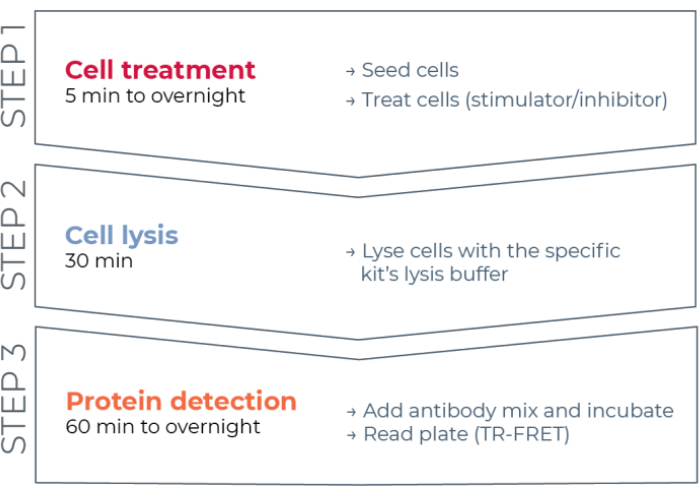
Figura 1: Flujo de trabajo de ensayo TR-FRET. El flujo de trabajo consta de tres pasos: tratamiento celular, lisis celular y detección de proteínas mediante TR-FRET. En el protocolo de ensayo de dos placas, los lisados se transfieren a una placa de detección blanca de 384 pocillos, mientras que en el protocolo de una placa, todos los pasos se llevan a cabo en la misma placa de detección blanca de 384 pocillos (protocolo todo en un pozo). Independientemente del protocolo de ensayo utilizado, la detección de proteínas se realiza en el mismo volumen total (20 μL por pocillo). Abreviatura: TR-FRET = transferencia de energía de resonancia de Förster resuelta en el tiempo. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Principio de inmunoensayo sándwich TR-FRET. Un anticuerpo está marcado con el donante de quelato de europio (Eu-Ab1) y el segundo con el aceptor de fluoróforo pequeño de color rojo lejano (FR-Ab2). Los dos anticuerpos marcados se unen específicamente a epítopos distintos en la proteína objetivo (fosforilada o total) en el lisado celular, acercando los dos fluoróforos. La excitación del quelato de europio del donante a 320 o 340 nm desencadena un FRET del donante a las moléculas aceptoras, que a su vez emiten una señal a 665 nm. Esta señal es proporcional a la concentración de proteína en el lisado celular. En ausencia de la proteína diana específica, los fluoróforos donante y aceptor están demasiado distantes entre sí para que ocurra FRET. Abreviaturas: FRET = transferencia de energía de resonancia de Förster; TR-FRET = FRET resuelto en el tiempo; Ab = anticuerpo; FR = rojo lejano; Eu - quelato de europio; P = fosforilación. Haga clic aquí para ver una versión más grande de esta figura.
Aquí, se proporcionan protocolos detallados para medir, en un formato de 384 pocillos, los niveles intracelulares de STAT1 fosforilado (Y701), STAT3 (Y705), STAT4 (Y693), STAT5 (Y694 / Y699) y STAT6 (Y641), junto con STAT1, STAT3, STAT5 y STAT6 totales, en lisados celulares de celdas adherentes o de suspensión utilizando la plataforma THUNDER TR-FRET. Estos protocolos definen los pasos para el tratamiento celular, la lisis y la detección de proteínas diana basadas en TR-FRET utilizando un protocolo de transferencia de dos placas o un protocolo todo en un pocillo de una placa. Estos ensayos basados en células se aplican para determinar el perfil farmacológico de los activadores e inhibidores conocidos de la vía JAK/STAT. Se demuestra la robustez e idoneidad de los ensayos seleccionados para HTS. Por último, se discuten los experimentos clave para la optimización de ensayos, junto con recomendaciones para la solución de problemas de ensayos.
Protocolo
1. Cultivo celular
- Mantener las células en una incubadora humidificada de 37 °C/5% de CO2 y cultivarlas con DMEM suplementado con suero fetal bovino (FBS) al 10% (células HeLa y A431) o RPMI suplementado con 15% de FBS (células U266B1). Cultive las células hasta que alcancen el 70-80% de confluencia, luego tripsinícelas y páselas o utilícelas para los ensayos.
NOTA: Los medios de cultivo contenían rojo fenol. No se realizó inanición sérica para ninguna línea celular antes de realizar los ensayos.
2. Titulación del estimulador o inhibidor utilizando el protocolo de ensayo de dos placas con células adherentes
NOTA: Este procedimiento describe cómo determinar las potencias del estimulador o inhibidor mediante la generación de una curva concentración-respuesta a partir de una serie de dilución del compuesto de prueba.
- Siembra celular
- Dispensar 50 μL de células a la densidad preoptimizada (40.000 células HeLa/pozo para STAT3 y STAT6; 75.000 células A431/pozo para STAT5) en una placa tratada con cultivo de tejidos de 96 pocillos en el medio de cultivo apropiado. Incubar durante la noche a 37 °C/5% de CO2.
NOTA: Es necesario determinar la densidad celular óptima y el tiempo de incubación del cultivo.
- Dispensar 50 μL de células a la densidad preoptimizada (40.000 células HeLa/pozo para STAT3 y STAT6; 75.000 células A431/pozo para STAT5) en una placa tratada con cultivo de tejidos de 96 pocillos en el medio de cultivo apropiado. Incubar durante la noche a 37 °C/5% de CO2.
- Diluciones de compuestos de ensayo
- Prepare series intermedias de dilución 2x de compuestos de prueba diluyendo en serie compuestos (diluciones de intervalo medio logarítmico) en 12 pocillos de una placa de polipropileno de 96 pocillos en un medio libre de suero.
NOTA: Se recomienda realizar una curva de concentración-respuesta de intervalo medio logarítmico de 12 puntos en al menos duplicado para una estimación precisa del EC50 o IC50. - Alternativamente, para los compuestos de prueba hidrofóbicos solubles en dimetilsulfóxido (DMSO), realice las diluciones iniciales en 100% DMSO y luego diluya la serie de dilución del compuesto en un medio libre de suero.
NOTA: La tolerancia del ensayo a DMSO debe establecerse antes de realizar una titulación del compuesto de prueba en el vehículo DMSO. Es importante mantener concentraciones iguales de disolvente entre las células tratadas y no tratadas. Además, al probar diluciones seriadas de compuestos, las concentraciones de disolventes siempre deben permanecer constantes en toda la serie de dilución.
- Prepare series intermedias de dilución 2x de compuestos de prueba diluyendo en serie compuestos (diluciones de intervalo medio logarítmico) en 12 pocillos de una placa de polipropileno de 96 pocillos en un medio libre de suero.
- Tratamiento celular
- Para la estimulación celular, agregue 50 μL de medio libre de suero solo (células no tratadas) o que contenga el estimulador (2x).
- Incubar durante el tiempo preoptimizado a temperatura ambiente (RT) o 37 °C (interferón (IFN) α2b/20 min a RT para STAT3; factor de crecimiento epidérmico (EGF)/10 min a RT para STAT5; interleucina (IL)-4/20 min a RT para STAT6). Continúe a continuación con la sección 2.4.
NOTA: Es necesario determinar la temperatura óptima de incubación. - Para la inhibición celular, agregue 25 μL de medio libre de suero solo (células no tratadas) o que contenga el inhibidor (4x).
- Incubar para el tiempo preoptimizado a RT o 37 °C (inhibidor de JAK 1/30 min a RT para STAT3 y STAT6; Erlotinib/15 min en RT para STAT5).
- Añadir 25 μL de medio libre de suero solo (células no tratadas) o que contenga el estimulador (4x) en su EC80.
- Incubar para el tiempo preoptimizado a RT o 37 °C (mismas condiciones que para el paso 2.3.2).
- Lisis celular
- Prepare el tampón de lisis suplementado 1x específico del kit según lo indicado por el fabricante.
NOTA: Es obligatorio complementar el 1x Lysis Buffer con el 100x Phosphatase Inhibitor Cocktail diluido a una concentración final de 1x. El tampón de lisis suplementado 1x contiene 1 mM de fluoruro de sodio, 2 mM de ortovanadato de sodio y 2 mM de beta-glicerofosfato. Otros inhibidores de la fosfatasa no son necesarios y deben evitarse. No se recomiendan los tampones de lisis y los inhibidores de la fosfatasa distintos de los incluidos en el kit, ya que pueden contener ingredientes que podrían interferir con la medición. - Retire y deseche cuidadosamente el medio de cultivo celular aspirando el sobrenadante.
- Agregue inmediatamente 50 μL de 1x tampón de lisis suplementado.
NOTA: El volumen del búfer de lisis (25-50 μL) puede optimizarse. - Incubar durante 30 min a RT bajo agitación (agitador de placa orbital establecido a 400 rpm; agitación moderada).
NOTA: El tiempo de incubación de lisis (30-60 min) puede optimizarse. Los lisados se pueden utilizar inmediatamente para la detección de proteínas diana o congelarse a -80 °C.
- Prepare el tampón de lisis suplementado 1x específico del kit según lo indicado por el fabricante.
- Detección TR-FRET
- Prepare la mezcla de detección de anticuerpos 4x en el búfer de detección 1x según lo indicado por el fabricante.
- En este paso de transferencia, pipetee cuidadosamente 15 μL de lisado celular de la placa de cultivo de 96 pocillos a un pocillo de una microplaca blanca de bajo volumen de 384 pocillos.
- Agregue 15 μL del lisado de control positivo y 15 μL de 1x Lysis Buffer (control negativo) a los pozos de ensayo separados.
- Agregue 5 μL de 4x Antibody Detection Mix (ya sea Eu-Ab1 / FR-Ab2 para la detección de la fosfoproteína o Eu-Ab3 / FR-Ab4 para la detección de la proteína total) a cada uno de los pozos de ensayo.
- Cubra la placa con un sellador de placas e incube durante 1 h hasta durante la noche en RT, dependiendo del ensayo (consulte la Hoja de Datos Técnicos correspondiente).
NOTA: El tiempo de lectura óptimo debe optimizarse para cada ensayo y línea celular. La placa se puede leer varias veces sin un efecto negativo en el rendimiento del ensayo. - Retire el sellador de placa adhesiva y lea la placa en un lector de microplacas compatible con TR-FRET.
NOTA: Se recomiendan fluorómetros basados en filtros, aunque se pueden usar algunos instrumentos monocromadores. Verifique que el módulo óptico apropiado (filtros y espejo) para TR-FRET esté instalado. Utilice una longitud de onda de excitación de 320 o 340 nm para excitar el quelato de europio. Lea ensayos a 615 nm (o 620 nm) y 665 nm para detectar tanto la emisión del europio donante como el fluoróforo aceptor, respectivamente. La configuración del instrumento dependerá del lector en particular. Los datos presentados aquí se obtuvieron utilizando excitación basada en lámpara, retraso de 90 μs, tiempo de integración de 300 μs y 100 destellos por pozo. El ensayo fosfo-STAT4, sin embargo, se leyó utilizando excitación láser para generar relaciones señal-fondo (S/B) más altas.
3. Valoración del estimulador o inhibidor utilizando el protocolo de ensayo de dos placas con células de suspensión
- Dilución de compuestos de ensayo
- Preparar series intermedias de dilución 2x de compuestos de ensayo como se describe en los pasos 2.2.1 y 2.2.2.
- Siembra y tratamiento celular
- Dispensar 20 μL de células a la densidad preoptimizada (200.000 células U266B1/pozo para STAT1; 400.000 células U266B1/pozo para STAT4) en una placa tratada con cultivo de tejidos de 96 pocillos en el medio de cultivo apropiado. Proceder directamente al tratamiento celular o incubar 2-4 h a 37 °C, 5% CO2.
NOTA: Este paso debe optimizarse para diferentes tipos de celdas. - Para la estimulación celular, agregue 20 μL de medio libre de suero solo (células no tratadas) o que contenga el estimulador (2x).
- Incubar para el tiempo preoptimizado a RT o 37 °C (IFNα2b/15 min a RT para STAT1; IFNα2b/25 min a 37 °C para STAT4). Continúe a continuación con la sección 3.3.
NOTA: Es necesario determinar la temperatura óptima de incubación. - Para la inhibición celular, agregue 10 μL de medio libre de suero solo (células no tratadas) o que contenga el inhibidor (4x).
- Incubar durante el tiempo preoptimizado a RT o 37 °C (inhibidor de JAK 1/30 min a RT para STAT1 y STAT4).
- Añadir 10 μL de medio libre de suero solo (células no tratadas) o que contenga el estimulador (4x) en su EC80.
- Incubar para el tiempo preoptimizado a RT o 37 °C (mismas condiciones que para el paso 3.2.3).
- Dispensar 20 μL de células a la densidad preoptimizada (200.000 células U266B1/pozo para STAT1; 400.000 células U266B1/pozo para STAT4) en una placa tratada con cultivo de tejidos de 96 pocillos en el medio de cultivo apropiado. Proceder directamente al tratamiento celular o incubar 2-4 h a 37 °C, 5% CO2.
- Lisis celular
- Prepare el tampón de lisis suplementado 5x específico del kit según lo indicado por el fabricante.
NOTA: Es obligatorio complementar el tampón de lisis 5x con el cóctel inhibidor de fosfatasa 100x diluido a una concentración final de 5x. El tampón de lisis suplementado 5x contiene 5 mM de fluoruro de sodio, ortovanadato de sodio de 10 mM y 10 mM de beta-glicerofosfato. Otros inhibidores de la fosfatasa no son necesarios y deben evitarse. - Agregue 10 μL de 5x tampón de lisis suplementado.
- Incubar durante 30 min a RT bajo agitación (agitador de placa orbital establecido a 400 rpm; agitación moderada).
NOTA: El tiempo de incubación de lisis (30-60 min) puede optimizarse. Los lisados se pueden utilizar inmediatamente para la detección de proteínas diana o congelarse a -80 °C.
- Prepare el tampón de lisis suplementado 5x específico del kit según lo indicado por el fabricante.
- Detección TR-FRET
- Después de la lisis celular, realice la etapa de detección TR-FRET como se describe en la sección 2.5 para el protocolo de ensayo de 2 placas para células adherentes.
4. Titulación del estimulador utilizando el protocolo de ensayo de una placa con células adherentes o de suspensión
- Dilución de compuestos de ensayo
- Prepare series de dilución intermedia de compuestos de prueba a 3x diluyendo en serie los compuestos (diluciones de intervalo de medio registro) en 12 pocillos de una placa de polipropileno de 96 pocillos en un medio libre de suero.
- Siembra y tratamiento celular
- Dispensar 8 μL de células a la densidad preoptimizada (160.000 células U266B1/pozo para STAT4; 80.000 células HeLa/pozo para STAT6), en el medio de cultivo libre de suero apropiado, en una placa de ensayo blanca de bajo volumen de 384 pocillos. Proceder directamente al tratamiento celular o incubar 2-4 h a 37 °C, 5% CO2.
NOTA: El requisito de un período de incubación de cultivo celular antes del tratamiento debe determinarse para diferentes tipos de células. - Para la estimulación celular, agregue 4 μL de medio libre de suero solo (células no tratadas) o que contenga el estimulador (3x).
- Incubar durante el tiempo preoptimizado a temperatura ambiente o a 37 °C (IFNα2b/25 min a 37 °C para STAT4; IL-4/20 min a 37°C para STAT6). Continúe a continuación con la sección 4.3.
- Dispensar 8 μL de células a la densidad preoptimizada (160.000 células U266B1/pozo para STAT4; 80.000 células HeLa/pozo para STAT6), en el medio de cultivo libre de suero apropiado, en una placa de ensayo blanca de bajo volumen de 384 pocillos. Proceder directamente al tratamiento celular o incubar 2-4 h a 37 °C, 5% CO2.
- Lisis celular
- Prepare el tampón de lisis suplementado 5x específico del kit según lo indicado por el fabricante.
NOTA: Es obligatorio complementar el tampón de lisis 5x con el cóctel inhibidor de fosfatasa 100x diluido a una concentración final de 5x. - Agregue 3 μL de 5x tampón de lisis suplementado.
- Incubar durante 30 min a RT bajo agitación (agitador de placa orbital a 400 rpm).
NOTA: El tiempo de incubación de lisis (30-60 min) puede optimizarse. Los lisados se pueden utilizar inmediatamente o congelar a 80 °C.
- Prepare el tampón de lisis suplementado 5x específico del kit según lo indicado por el fabricante.
- Detección TR-FRET
- Agregue 15 μL de lisado de control positivo (sin diluir) y 15 μL de tampón de lisis suplementado 1x (control negativo) a los pozos de ensayo separados.
- Agregue 5 μL de 4x Antibody Detection Mix (ya sea Eu-Ab1/FR-Ab2 para la fosfoproteína o Eu-Ab3-/FR-Ab4 para la proteína total) preparado en 1x Detection Buffer a cada uno de los pozos de ensayo.
- Cubra la placa con un sellador de placas e incube durante 1 h hasta durante la noche en RT, dependiendo del ensayo.
NOTA: El tiempo de lectura óptimo debe optimizarse para cada ensayo y línea celular. La placa se puede leer varias veces sin un efecto negativo en el rendimiento del ensayo. - Retire el sellador de placa adhesiva. Lea la placa en un lector de microplacas compatible con TR-FRET.
5. Análisis de datos
- Calcule la relación TR-FRET para cada pozo utilizando la siguiente fórmula (1):
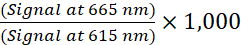 (1)
(1)
NOTA: Debido a que la señal TR-FRET se lee en un modo de resolución temporal, la resta de fondo generalmente no es necesaria. Si se realiza la resta de fondo, use los pocillos libres de células que contienen el tampón de lisis suplementado 1x (control negativo) para la resta de fondo. Determine la relación TR-FRET promedio de los pozos libres de celdas y luego reste este valor de la relación TR-FRET de cada pozo. - Para las curvas de concentración-respuesta, analice los datos de acuerdo con una regresión no lineal utilizando la ecuación logística de 4 parámetros (curva dosis-respuesta sigmoidal con pendiente variable) y una ponderación de datos 1/Y2 para generar valores EC50 o IC50 .
- Para el experimento del factor Z', analice los datos de acuerdo con la siguiente fórmula (2)10
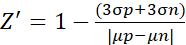 (2)
(2)
Cuando μ y σ son los valores medios y las desviaciones estándar para el control positivo (p; células estimuladas) y el control negativo (n; células no tratadas), respectivamente.
Resultados
Cada ensayo THUNDER TR-FRET se validó farmacológicamente mediante el tratamiento de células adherentes (HeLa o A431) o de suspensión (U266B1) con activadores o inhibidores específicos de la vía JAK/STAT y luego midiendo los niveles de STATs fosforilados y totales específicos, cuando corresponda. Los ensayos se realizaron en formato de 384 pocillos utilizando el protocolo de transferencia de dos placas y las condiciones de ensayo preoptimizadas. La Figura 3, la Fig...
Discusión
En comparación con los métodos convencionales para el análisis de fosfoproteínas, como el western blotting y los métodos basados en ELISA, el flujo de trabajo para un ensayo celular THUNDER TR-FRET es simple y rápido, utiliza una muestra de bajo volumen (15 μL), está diseñado para HTS en un formato de 384 pocillos y es altamente susceptible de automatización. El protocolo de ensayo es flexible y se puede adaptar fácilmente a aplicaciones de rendimiento medio y alto. Los ensayos se pueden ejecutar utilizando un...
Divulgaciones
Intereses contrapuestos: Jaime Padros, Mireille Caron y Geneviève Chatel son empleados de BioAuxilium Research, que fabrica los kits de ensayo THUNDER TR-FRET utilizados en este estudio. Además, Jaime Padros y Mireille Caron son accionistas de BioAuxilium Research. Esto no altera la adhesión de los autores a todas las políticas de JoVE sobre el intercambio de datos y materiales.
Agradecimientos
Ninguno.
Materiales
| Name | Company | Catalog Number | Comments |
| 96-well microplate, clear, flat bottom, polystyrene, tissue culture-treated, sterile | Corning | 3595 | This is the plate for culturing cells when using the two-plate assay protocol. Other cell culture 96-well plates can be used |
| 384-well microplate, white, low-volume | PerkinElmer | 6007290 | This is the plate for TR-FRET detection when using the two- or one-plate assay protocols. Other low-volume, white 384-well plates can be used |
| A431 cell line | ATCC | CRL-1555 | |
| Adhesive microplate seal | PerkinElmer | 6050185 | |
| DMSO | Fisher | D159-4 | |
| Dulbecco’s modified Eagle medium (DMEM) | Wisent | 319-005-CL | THUNDER TR-FRET is compatible with culture medium containing phenol red |
| EnVision Xcite Multilabel plate reader | PerkinElmer | 2104-0020A | The assays can be performed on a variety of plate readers equipped with the TR-FRET option |
| Erlotinib hydrochloride | Sigma | CDS022564 | |
| Falcon tissue culture treated flasks | Fisher | 13-680-65 | |
| Fetal bovine serum (FBS) | Wisent | 098-150 | |
| HeLa cell line | ATCC | CCL-2 | |
| JAK Inhibitor 1 | Cayman Chemical | 15146 | |
| Orbital plate shaker | Many options available | Not applicable | |
| Recombinant human EGF | PeproTech | AF-100-15 | |
| Recombinant human IFNα2b | ProSpec | CYT-460 | |
| Recombinant human IL-4 | R&D Systems | 204-IL | |
| Roswell Park Memorial Institute 1640 medium (RPMI) | Wisent | 350-007-CL | THUNDER TR-FRET is compatible with culture medium containing phenol red |
| THUNDER Phospho-STAT1 (Y701) + Total STAT1 TR-FRET Cell Signaling Assay Kit | BioAuxilium Research | KIT-STAT1PT-500 | |
| THUNDER Phospho-STAT3 (Y705) + Total STAT3 Cell Signaling Assay Kit | BioAuxilium Research | KIT-STAT3PT-500 | |
| THUNDER Phospho-STAT4 (Y693) TR-FRET Cell Signaling Assay Kit | BioAuxilium Research | KIT-STAT4P-500 | |
| THUNDER Phospho-STAT5 (Y694/Y699) + Total STAT5 TR-FRET Cell Signaling Assay Kit | BioAuxilium Research | KIT-STAT5PT-500 | |
| THUNDER Phospho-STAT6 (Y641) + Total STAT6 TR-FRET Cell Signaling Assay Kit | BioAuxilium Research | KIT-STAT6PT-500 | |
| Trypsin/EDTA 0.05% | Wisent | 325-542-CL | |
| U266B1 cell line | ATCC | TIB-196 | |
| Ultrapure water | NA | NA | Use Milli-Q grade water (18 MΩ.cm) to dilute Lysis Buffer and Detection Buffer |
Referencias
- Villarino, A., Kanno, Y., O'Shea, J. Mechanisms and consequences of Jak-STAT signaling in the immune system. Nature Immunology. 18 (4), 374-384 (2017).
- Hammarén, H. M., Virtanen, A. T., Raivola, J., Silvennoinen, O. The regulation of JAKs in cytokine signaling and its breakdown in disease. Cytokine. 118, 48-63 (2019).
- O'Shea, J. J., et al. The JAK-STAT pathway: impact on human disease and therapeutic intervention. Annual Review of Medicine. 66, 311-328 (2015).
- Verhoeven, Y., et al. et al. potential and controversy of targeting STAT family members in cancer. Seminars in Cancer Biology. 60 (2), 41-56 (2020).
- Gilda, J. E., et al. et al. blotting inaccuracies with unverified antibodies: need for a Western blotting minimal reporting standard (WBMRS). PLoS One. 10 (8), 0135392 (2015).
- Binder, C., et al. et al. and utilization of the SureFire phospho-STAT5 assay for a cell-based screening campaign. Assay and Drug Development Technologies. 6 (1), 27-37 (2008).
- Ayoub, M. A., et al. Homogeneous time-resolved fluorescence-based assay to monitor extracellular signal-regulated kinase signaling in a high-throughput format. Frontiers in Endocrinology. 5, 94 (2014).
- Robers, M. B., Machleidt, T., Carlson, C. B., Bi, K. Cellular LanthaScreen and beta-lactamase reporter assays for high-throughput screening of JAK2 inhibitors. Assay and Drug Development Technologies. 6 (4), 519-529 (2008).
- Hwang, B., Engel, L., Goueli, S. A., Zegzouti, H. A. Homogeneous bioluminescent immunoassay to probe cellular signaling pathway regulation. Communications Biology. 3 (8), 1-12 (2020).
- Zhang, J. H., Chung, T. D., Oldenburg, K. R. A simple statistical parameter for use in evaluation and validation of high-throughput screening assays. Journal of Biomolecular Screening. 4 (2), 67-73 (1999).
- Osmond, R. I. W., Das, S., Crouch, M. F. Development of cell-based assays for cytokine receptor signaling, using an AlphaScreen SureFire assay format. Analytical Biochemistry. 403 (1-2), 94-101 (2010).
- Haan, C., et al. Jak1 has a dominant role over Jak3 in signal transduction through γc-containing cytokine receptors. Chemistry & Biology. 18 (3), 314-323 (2011).
- Kim, Y., Apetri, M., Luo, B., Settleman, J. E., Anderson, K. S. Differential effects of tyrosine kinase inhibitors on normal and oncogenic EGFR signaling and downstream effectors. Molecular Cancer Research. 13 (4), 765-774 (2015).
- Qian, J., et al. Comparison of two homogeneous cell-based kinase assays for JAK2 V617F: SureFire pSTAT5 and GeneBLAzer fluorescence resonance energy transfer assays. ASSAY and Drug Development Technologies. 10 (2), 212-217 (2012).
- Iversen, P. W., et al., Markossian, S., et al. HTS assay validation. Assay Guidance Manual. , (2012).
- Muelas, M. W., Ortega, F., Breitling, R., Bendtsen, C., Westerhoff, H. V. Rational cell culture optimization enhances experimental reproducibility in cancer cells. Scientific Reports. 8, 3029 (2018).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados