Method Article
Kit de herramientas integradoras para analizar señales celulares: fuerzas, movimiento, morfología y fluorescencia
* Estos autores han contribuido por igual
En este artículo
Resumen
La plataforma Integrative Toolkit to Analyze Cellular Signals (iTACS) automatiza el proceso de medición simultánea de una amplia variedad de señales químicas y mecánicas en células adherentes. iTACS está diseñado para facilitar el desarrollo impulsado por la comunidad y permitir a los investigadores utilizar todas las características de la plataforma, independientemente de su formación académica.
Resumen
La evaluación cuantitativa de las fuerzas celulares y el movimiento avanzó considerablemente en las últimas cuatro décadas. Estos avances proporcionaron el marco para examinar los perspicaces procesos de señalización mecánica en los sistemas de cultivo celular. Sin embargo, el campo se enfrenta actualmente a tres problemas: la falta de estandarización de la calidad de los datos adquiridos, los errores técnicos en el análisis y la visualización de datos, y quizás lo más importante, la tecnología sigue estando en gran medida fuera del alcance de los laboratorios comunes de biología celular. Para superar estas limitaciones, desarrollamos una nueva plataforma experimental: Integrative Toolkit to Analyze Cellular Signals (iTACS). iTACS consta de dos componentes: Módulo de Adquisición y Capacitación (AcTrM) y Módulo de Análisis y Visualización (AnViM). AcTrM se basa en μManager, un software de control de microscopio basado en NIH-ImageJ, y facilita el autoentrenamiento del usuario y la automatización de los protocolos comunes de adquisición de imágenes. AnViM se basa en NIH-ImageJ y facilita la automatización fácil de usar del análisis de datos y la visualización perspicaz de los resultados. Estos experimentos implican el cultivo de células adherentes en hidrogeles, la obtención de imágenes de marcadores fiduciarios incrustados en el hidrogel y, finalmente, la extracción de estas imágenes de una caracterización mecánica integral de las células. Actualmente, iTACS permite al usuario analizar y rastrear una amplia gama de propiedades, incluida la morfología, el movimiento, las fuerzas citoesqueléticas y la fluorescencia de células individuales y su región vecina. El problema de la estandarización de la calidad se abordó en AcTrM con una técnica de reenfoque guiada por imágenes de referencia. Los problemas técnicos en el análisis de datos se abordaron en AnViM con un procedimiento de segmentación de imágenes de múltiples frentes, un enfoque fácil de usar para identificar las condiciones de contorno y una nueva visualización de datos basada en propiedades celulares. AcTrM está diseñado para facilitar la transformación directa de microscopios de fluorescencia básicos en equipos experimentales de mecánica celular, y AnViM está equipado para permitir a los usuarios medir señales mecánicas celulares sin requerir una formación en ingeniería. iTACS estará disponible para la comunidad de investigación como una suite de código abierto con capacidades de desarrollo impulsadas por la comunidad.
Introducción
Las herramientas de análisis de datos e imágenes ópticas de uso común emplean tecnologías de hardware y software que son casi anticuadas. El retraso en la traducción e implementación de los avances en dispositivos electrónicos, enfoques computacionales y análisis matemático en herramientas comunes de biología celular experimental es una restricción importante en el ritmo de crecimiento en nuestro conocimiento de la fisiología celular. Actualmente, los investigadores de biología celular encuentran herramientas de biología molecular al alcance, pero herramientas basadas en principios de ingeniería están fuera de su alcance. Una de estas herramientas basadas en principios de ingeniería es la microscopía de esfuerzo monocapa (MSM)1,2. Si bien el MSM ha sido adaptado y estudiado en varios laboratorios de todo el mundo, su uso se limita principalmente a laboratorios con experiencia en ingeniería3,4,5,6,7,8,9.
NIH-ImageJ es una de las herramientas de código abierto más populares entre los investigadores de biología celular10. Los avances impulsados por la contribución de la comunidad de usuarios han sido fundamentales para su popularidad11,12. ImageJ tiene características que permiten a los usuarios desarrollar aplicaciones con una combinación de un lenguaje de programación avanzado y enfoques de scripting simplificados. Estas características facilitan a los usuarios con conocimientos básicos de programación para implementar, adaptar y avanzar en cualquier nueva contribución al software. Sobre la base de estas cualidades de NIH-ImageJ, hemos desarrollado el Kit de herramientas integrativas para analizar señales celulares (iTACS), que permite una integración de bajo costo de las herramientas de hardware y software deseadas para automatizar la medición de una amplia variedad de señales químicas y mecánicas a través de células adherentes11,12.
iTACS consta de dos componentes: Módulo de Adquisición y Capacitación (AcTrM) y Módulo de Análisis y Visualización (AnViM). AcTrM se basa en μManager, una aplicación de adquisición de imágenes basada en NIH-ImageJ, para permitir a los usuarios configurar mediciones de lapso de tiempo de propiedades ópticas tradicionales y una variedad de propiedades físicas de células adherentes en múltiples muestras12. AcTrM facilita la formación del usuario a través de instrucciones concisas incluidas en la interfaz gráfica. Además, tiene una característica novedosa de enfoque automático basado en imágenes de referencia que está diseñado para facilitar las mediciones en tiempo real de las fuerzas físicas y permitir la estandarización de la calidad de los datos adquiridos.
AnViM se basa en complementos de ImageJ, software acelerado y scripts de manejo de archivos que permiten a los usuarios evaluar cuantitativamente más de 50 propiedades, incluida la forma celular, el tamaño, la orientación, la velocidad y la dirección del movimiento, las tracciones ejercidas sobre la matriz extracelular (ECM) y en las células vecinas, los momentos contráctiles y de cizallamiento de las células adherentes individuales y su región vecina. AnViM facilita a los usuarios cuantificar las propiedades físicas celulares sin dominar los antecedentes técnicos subyacentes11. Además, permite el análisis de datos en modo interactivo o de procesamiento por lotes. Genera mapas de calor que revelan la variación espacial y gráficos que muestran la variación temporal de las propiedades de las células individuales.
En un experimento típico, el usuario cultiva células en un hidrogel elástico con proteínas de matriz extracelular apropiadas en la superficie superior y dos tipos de marcadores fluorescentes incrustados. Esencialmente, las imágenes de estos marcadores fluorescentes antes y después del cultivo de las células son suficientes para cuantificar las fuerzas dentro y alrededor de las células individuales2,13. AnViM mapea estos resultados en celdas individuales del clúster adherente y genera imágenes y gráficos perspicaces.
Protocolo
NOTA: Las muestras examinadas utilizando la plataforma iTACS son células adheridas a un sustrato blando. El protocolo para evaluar señales mecánicas y químicas se divide en dos partes secuenciales: Módulo de Adquisición y Capacitación (AcTrM) y Módulo de Análisis y Visualización (AnViM).
1. Módulo de Adquisición y Capacitación (AcTrM)
NOTA: AcTrM automatiza el proceso de adquisición de datos y la autoformación del usuario. Antes de cualquier adquisición de datos, preparar un sustrato blando capaz de proporcionar la información necesaria para cuantificar las fuerzas que las células ejercen sobre él.
- Preparación de hidrogel
NOTA: El objetivo aquí es preparar un módulo de cizallamiento de 1.250 Pa, un espesor aproximado de 100 μm y un hidrogel de poliacrilamida (PAA) de 22 mm de diámetro.- Preparar la solución de poliacrilamida siguiendo el método de Yeung et al. y fundir los hidrogeles siguiendo los pasos descritos por Trepat et al.14,15. Una excepción al procedimiento es que las perlas incrustadas inmediatamente debajo de las células tienen 0,5 μm de diámetro y emiten fluorescencia amarilla.
- Omita el paso de unir cuentas de 2 μm a la cubierta si se espera que el área de visualización tenga una gran región libre de células.
NOTA: El protocolo de preparación de hidrogel está ahora bastante establecido en el campo16. A lo largo de la descripción posterior, las cuentas de 0,5 μm se denominan «cuentas superiores», y las cuentas de 2 μm se denominan «cuentas inferiores». Sin embargo, las cuentas inferiores son opcionales cuando la región fotografiada contiene una gran área libre de celdas. El patrón de cuentas superior de tal región libre de células servirá para el propósito del patrón de cuentas inferior. - Monte el hidrogel con perlas fluorescentes incrustadas en la etapa del microscopio.
- Permita 15-20 minutos para que la temperatura de la placa alcance un estado estacionario.
NOTA: La superficie superior de la placa está funcionalizada con proteínas de la matriz extracelular, pero las células aún no están sembradas en el hidrogel.
- Adquisición de imágenes de referencia
- Parte 1: Creación de una lista de posiciones
NOTA: La entrada manual en este paso incluye seguir las indicaciones de AcTrM para elegir ubicaciones con la mejor distribución de cuentas. En este paso no se utilizan archivos creados previamente. Las nuevas carpetas clave producidas en este paso incluyen las carpetas 't0imgs', 'tnimgs' y 'textfiles', y el archivo producido incluye el archivo de lista de posiciones. Consulte la Figura 2 para obtener la descripción visual de los siguientes pasos que guían la creación de una lista de posiciones mediante AcTrM. El ejemplo demostrado adquiere datos a un aumento de 20x.- Inicie μManager 2.0 - Beta.
- Ejecute AcTrM haciendo clic en el botón AcTrM.bsh en la ventana Recursos de IMBL .
- En la ventana titulada Definir propiedades de adquisición, seleccione el objetivo apropiado, la tolerancia para la precisión de la recuperación de la posición lateral (es decir, XY), el tamaño del paso de la operación de reenfoque aproximado y refinado (es decir, z) y el valor del coeficiente de correlación de imagen aceptable para la recuperación del enfoque.
NOTA: Una tolerancia más fina para la recuperación de la posición lateral ralentizará la recuperación de la posición, pero una tolerancia sustancial necesitará corrección de posición durante el análisis de datos y puede causar dificultades en la recuperación del enfoque. Un tamaño de paso más pequeño ralentizará la operación de reenfoque, pero un tamaño de paso muy alto puede causar un movimiento rápido con oportunidades de perder el enfoque. - En la ventana AcTrM - Paso 0 , seleccione Nueva adquisición para crear una lista de posiciones.
NOTA: La ventana titulada AcTrM - Paso 1 enumera todos los pasos siguientes clave. Estos pasos implican mover el escenario, ajustar el enfoque y hacer clic en los botones del μManager. Estos pasos guían en la construcción de la lista de posiciones adecuadas para la adquisición de datos. - Haga clic en Live in Micromanager para visualizar las muestras de ajustes manuales enumeradas en la ventana AcTrM - Paso 1 .
- Siga los pasos enumerados en esta ventana.
- Ahora adquiere la imagen de las cuentas. Seleccione el canal apropiado para las cuentas superiores.
- Asegúrese de mirar las cuentas inferiores también. Para hacerlo, seleccione los canales para las cuentas inferiores. Se ve una imagen borrosa de cuentas inferiores.
NOTA: Los canales se pueden cambiar desde el menú desplegable preestablecido en la ventana principal de μManager. Si se utilizan las cuentas inferiores en el experimento, asegúrese de que estén presentes en la posición seleccionada. Estas cuentas aparecerán borrosas. - Al seleccionar la posición adecuada, haga clic en Marcar en la ventana Lista de posiciones del escenario para guardar la posición.
NOTA: La mejor posición se define por una distribución densa y uniforme de las cuentas superiores incrustadas inmediatamente debajo de la superficie superior (es decir, cuentas superiores) y al menos dos cuentas unidas al vidrio de la cubierta (es decir, cuentas inferiores) (Figura suplementaria S1). La recuperación del enfoque es más rápida si las cuentas inferiores aparecen como anillos grandes y desenfocados. Sin embargo, si los experimentos se realizan en una posición sin necesidad de enfoque o recuperación de posición lateral, ignore las instrucciones relacionadas con la imagen de las cuentas inferiores. - Siga los pasos 6 a 9 descritos en la Figura 2 para incluir posiciones adicionales en la lista.
NOTA: Elija algunas posiciones adicionales para que una ronda de eliminación pueda permitir las mejores posiciones elegidas en el plato.
- Parte 2: Adquisición de imágenes de referencia
NOTA: Este paso no implica ninguna entrada manual. Los archivos clave creados anteriormente utilizados en este paso incluyen 'textfiles/*.pos'. Los nuevos archivos y carpetas clave producidos en este paso incluyen aquellos dentro de las carpetas 't0imgs' y 'tnimgs'. Consulte la Figura 3 y la Figura suplementaria S2 para obtener la descripción visual de los siguientes pasos que guían la adquisición de imágenes de referencia mediante AcTrM. La ventana AcTrM - Paso 2 también enumera todos los pasos clave.- Siguiendo los pasos enumerados en la ventana AcTrM - Paso 2 , tome las decisiones en la ventana Adquisición multidimensional . Por ejemplo, para realizar un lapso de tiempo prolongado, indique el número de imágenes que se tomarán, seleccione el primer canal como canal de fase y, a continuación, seleccione el siguiente para las cuentas superiores y el siguiente para las cuentas inferiores. Haga clic en Cerrar en la ventana Adquisición multidimensional y luego haga clic en Aceptar en la ventana ActrM - Paso 2 .
- En la ventana AcTrM - Paso 3 , elija activar la recuperación de posición XYZ basada en imágenes de referencia. La elección es generalmente sí. Sin embargo, si las imágenes se adquieren en una posición sin espacio para el enfoque o la deriva del escenario, la elección en la ventana AcTrM - Paso 3 será No.
- En la ventana AcTrM - Opciones de recuperación XYZ , establezca el canal para la recuperación XYZ aproximada, si desea realizar la recuperación XYZ refinada, la región y el canal para la recuperación XYZ refinada y la dirección para comenzar el reenfoque (Figura suplementaria S4). Cuando se complete, AcTrM adquirirá imágenes de referencia.
NOTA: Las opciones típicas para el canal serán el canal de cuentas inferior. La recuperación XYZ funciona mejor cuando se lleva a cabo con la imagen completa en la implementación actual. Las imágenes de referencia generalmente incluirán una imagen de luz transmitida del hidrogel, una imagen fluorescente de las cuentas superiores y una imagen fluorescente de las cuentas inferiores. El contenido de cada conjunto de imágenes puede variar en función de las elecciones realizadas en la ventana Adquisición multidimensional . Aún así, el software adquirirá el conjunto de imágenes de referencia en cada posición determinada en la Figura 2. Al final de este paso, se crean tres carpetas en el directorio elegido: 't0imgs', 'tnimgs' y 'textfiles'. La carpeta 't0imgs' contiene imágenes de referencia en el formato reconocido por μManager y utilizado en pasos posteriores; La carpeta 'tnimgs' contiene imágenes TIFF separadas con nombres de archivo 'c0_p0_t0.tif', 'c1_p0_t0.tif', etc. Aquí, 'c' representa el canal, 'p' representa la posición y 't' representa el tiempo. Después de estas letras están el número de canal, el número de posición y el número de marco, respectivamente. La carpeta 'textfiles' contiene la lista de posiciones en formato XML y las opciones del usuario para la adquisición de imágenes y la recuperación de posiciones.
- Parte 1: Creación de una lista de posiciones
- Siembra y crecimiento celular
NOTA: La plataforma iTACS tiene la flexibilidad de acomodar los protocolos de preparación de muestras utilizados en la evaluación in vitro común del comportamiento mecánico de las células adherentes, incluidas las células dispersas, la monocapa totalmente confluente, el ensayo de formación de redes y las monocapas con agujeros o brechas significativas.- Cultivo de células endoteliales microvasculares pulmonares de rata para confluir en un matraz17,18.
- Separe las células usando tripsina. Resuspendir las células en un medio de cultivo que contenga 10% de suero fetal bovino a una concentración de 1 x 106 células/ml.
- Coloque una gota de 5 μL de las células resuspendidas sobre una superficie de hidrogel parcialmente seca y colóquela en la incubadora de cultivo celular.
NOTA: Después de 2 días en el medio de cultivo que contiene 10% de suero fetal bovino, las células en esa gota forman una isla abarrotada de células1.
- Adquisición automatizada de imágenes para el experimento restante
NOTA: La entrada manual para este paso incluye seguir las indicaciones de AcTrM para reanudar la adquisición de imágenes. Los archivos clave creados previamente utilizados por este paso incluyen 'textfiles/*.pos' y archivos de imagen de cuentas inferiores en la carpeta 't0imgs'. Los nuevos archivos clave producidos en este paso incluyen archivos de lista de posición actualizados e imágenes 'tnimgs / * _t * .tif'.- Asegúrese de que el sistema de control ambiental del microscopio alcance condiciones estables de cultivo de tejidos.
- Monte suavemente la placa que contiene las células cultivadas en la etapa del microscopio.
- Permita 15-20 min para que la temperatura y la humedad se estabilicen.
Nota : consulte la figura 4 para obtener la descripción visual de los siguientes pasos que guían la adquisición de imágenes para el experimento restante mediante AcTrM. - Inicie μManager 2.0 - Beta.
- Ejecute AcTrM haciendo clic en el botón AcTrM.bsh en la ventana Recursos de IMBL .
Nota : omita las opciones realizadas en la ventana Definir propiedades de adquisición , pero utilice las opciones realizadas en el paso anterior. - En la ventana AcTrM - Paso 0 , seleccione Continuar adquisición en pausa.
- Seleccione la ampliación y el directorio identificados en la ventana Adquisición multidimensional en el paso anterior donde se guardaron las carpetas de datos ('t0imags', 'tnimgs' y 'textfiles').
Nota : el título de la ventana guía al usuario para elegir el directorio apropiado. - En la ventana Reposicionamiento de la placa , seleccione opciones para reposicionar la placa (tres posiciones no colineales) junto con el canal utilizado para el reposicionamiento.
NOTA: Las imágenes guardadas se verán en la cámara. Si las imágenes superpuestas se muestran como una imagen en color rojo, verde y negro, realice un ajuste manual. - Haga clic en Aceptar para continuar con la adquisición.
NOTA: Este paso supera los ligeros cambios de la irreproducibilidad de la alineación de la placa cuando la placa se vuelve a montar en la etapa del microscopio. Siga las indicaciones de AcTrM para acelerar la alineación en cada una de las tres posiciones mostrando una imagen compuesta de la imagen de referencia que se muestra en rojo (generalmente de las cuentas inferiores) y la imagen observada actualmente a través del objetivo que se muestra en verde. Obtener el verde lo suficientemente cerca del rojo deja menos trabajo para el software. Cuando la superposición es perfecta, la imagen compuesta aparece amarilla y negra. Lo suficientemente cerca es típicamente cuando las mismas cuentas inferiores son visibles tanto en imágenes rojas como verdes, y los correspondientes anillos rojos y verdes desenfocados se tocan entre sí. Una vez completado el reposicionamiento de la placa, AcTrM lleva el escenario a cada posición seleccionada y recupera la posición XYZ haciendo coincidir la imagen de referencia con lo que se ve actualmente a través de la cámara. La primera posición lateral (XY) coincide, y luego el enfoque (Z) se hace coincidir. La recuperación aproximada es seguida por una recuperación refinada de la posición y el enfoque. Las actualizaciones del estado actual del experimento se muestran en la pantalla a través de una ventana de cuatro partes que se muestra en la Figura suplementaria S3. Las imágenes adquiridas se guardan en la carpeta 'tnimgs' después de '*_t1.tif', '*_t2.tif', indicando el recuento de tiempo para el conjunto de imágenes. Si la recuperación XYZ identifica una posición actualizada, la nueva lista de posiciones se genera y se guarda en la carpeta 'textfiles'.
2. Módulo de Análisis y Visualización (AnViM)
- Configuración del análisis automatizado de datos
NOTA: La entrada manual en este paso incluye la identificación de la ubicación de la carpeta 'tnimgs'. Los archivos clave creados previamente utilizados en este paso incluyen 'tnimgs/*.tif'. Los nuevos archivos y carpetas clave producidos en este paso incluyen la carpeta 'análisis' en la misma carpeta principal que 'tnimgs' y carpetas para cada posición 'analysis/p*'. Dentro de cada 'análisis/p*', este paso crea nuevos archivos de imagen '*.tif' dentro de las carpetas 'phs' y 'tny'. Estas imágenes son imágenes recortadas y desviadas de células y cuentas superiores. Los otros archivos creados incluyen 'analysis/analysisChoices.txt' que enumera las opciones de análisis, 'analysis/p*/skipAnalysis.txt', que enumera los casos en los que se omite el análisis debido a una deriva preexistente significativa, y 'analysis/p*/part_shift_values_pixel_degree.dat' que enumera los valores estimados de deriva. AnViM no altera los datos sin procesar en la carpeta 'tnimgs'. Consulte la Figura 5 para obtener la descripción visual de los siguientes pasos que guían la configuración del análisis automatizado de datos mediante AnViM.- Inicie el software de Fiji y seleccione la primera opción en el menú desplegable MSM etiquetado como MSM - preprocesamiento.
- Utilizando el cuadro de diálogo del explorador de archivos, seleccione la carpeta que contiene la carpeta 'tnimgs', que contiene los datos analizados.
- Defina el canal para las imágenes. Siguiendo las directrices de la Figura Suplementaria S5, identifique los números de canal de la imagen de luz transmitida de las celdas (imagen de contraste de fase de las celdas), imagen de cuentas inferiores e imagen de cuentas superiores. Las siguientes tres casillas de verificación ('Mover archivos a la carpeta de posición', 'Hacer corrección adicional para el movimiento rígido' y 'Recortar datos y guardar en la carpeta de posición') suelen estar marcadas. Finalmente, defina la tolerancia para rechazar datos muy desviados, tamaño de píxel y lados de la imagen donde se cruzan las celdas.
- Responda al mensaje para mejorar el brillo y el contraste de las imágenes de cuentas inferiores para que las cuentas aparezcan de manera prominente. Ajuste esto usando la barra deslizante en el menú y haga clic en Aceptar.
- Ahora realice la corrección de posición para deshacerse de los turnos, si los hay. Una vez hecho esto, se crea una carpeta de análisis.
NOTA: La mejora del contraste normalmente no es necesaria, pero esta disposición está disponible para experimentos en los que es necesario minimizar la exposición a los láseres. Después de esa elección, AnViM copia los archivos de la carpeta 'tnimgs' a la carpeta 'análisis'. Los archivos correspondientes a cada posición se guardan en las carpetas 'analysis/p0, analysis/p1', etc. Dentro de cada una de estas carpetas de posición, AnViM crea carpetas 'cels', 'defs' y 'refs' que contienen la imagen de luz transmitida original, las imágenes de cuentas superiores y las imágenes de cuentas inferiores, respectivamente (Figura suplementaria S6). AnViM luego analiza el movimiento rígido en las imágenes de las cuentas inferiores y crea una carpeta 'phs' que contiene una imagen de luz transmitida corregida de las celdas y una carpeta 'tny' que contiene imágenes actualizadas de cuentas superiores. Finalmente, las opciones del usuario de los canales, las operaciones, la tolerancia, el tamaño de píxel y el cruce de límites se guardan en el archivo 'analysisChoices.txt' (Figura suplementaria S6).
- Cuantificación de la deformación de hidrogel y monocapa
NOTA: Es importante tener en cuenta que la cuantificación del movimiento a partir de una secuencia de imágenes es un campo en rápida evolución19. La tecnología se optimiza constantemente para las características, incluida la velocidad, la precisión, las características específicas dentro de las imágenes en bruto y los patrones de deformación específicos. Por lo tanto, es probable que algunos usuarios puedan utilizar un enfoque de cuantificación de deformación diferente al que se presenta aquí.-
Parte 1: Cuantificación de la deformación del hidrogel
NOTA: Las entradas manuales en este paso incluyen la identificación del directorio de datos y la selección de la resolución de la cuadrícula. Los archivos clave creados previamente utilizados en este paso incluyen 'p*/tny/*.tif'. Los nuevos archivos clave producidos en esto incluyen 'p * / desplazamiento / * _disp.dat' que enumera los vectores de desplazamiento de la superficie superior del hidrogel. Consulte la Figura 6 para obtener la descripción visual de los siguientes pasos para participar, a través de AnViM, en el análisis de velocimetría de imágenes de partículas en las imágenes de cuentas superiores20.- Inicie el software de Fiji y, en el menú desplegable de MSM , seleccione MSM - Deformación en gel.
- Desde aquí, seleccione la opción que sea adecuada para el experimento.
- Utilizando el cuadro de diálogo del explorador de archivos, seleccione el directorio principal de la carpeta 'análisis' que contiene los datos analizados.
- En la ventana Parámetros para calcular la deformación del gel , seleccione el tamaño de ventana de correlación cruzada, el nivel de ruido y el umbral adecuados (Figura suplementaria S7)20.
NOTA: Los resultados se guardan en un directorio de 'desplazamiento' recién creado dentro de cada carpeta de posición 'análisis/p0, análisis/p1', etc. Aquí es donde se almacenan todos los archivos de salida.
-
Parte 2: Cuantificación de la deformación monocapa
NOTA: Las entradas manuales en este paso incluyen la identificación del directorio de datos y la selección de la resolución de la cuadrícula. Los archivos clave creados previamente utilizados en este paso incluyen 'p*/tny/*.tif'. Los nuevos archivos clave producidos en esto incluyen 'p * / velocidad / * _vel.dat' que enumera los vectores de movimiento de las células. Consulte la Figura 7 para obtener la descripción visual de los pasos para activar, a través de AnViM, el análisis de velocimetría de imagen de partículas en las imágenes de cuentas superiores20. El procedimiento es similar al seguido para cuantificar la deformación del hidrogel y selecciona MSM - Cellular Motion en el menú desplegable MSM . Los resultados se guardan en un directorio de 'velocidad' recién creado dentro de cada carpeta de posición 'analysis/p0', 'analysis/p1', etc.
-
Parte 1: Cuantificación de la deformación del hidrogel
- Cuantificación de las fuerzas célula-ECM y célula-célula
NOTA: La entrada manual en este paso incluye la identificación del directorio de datos, la indicación de la rigidez del hidrogel y la respuesta a las solicitudes de segmentación de clústeres celulares para detectar células de la región sin células. Los archivos clave creados previamente utilizados en este paso incluyen 'p*/displacement/*_disp.dat'. Los nuevos archivos clave producidos en este paso incluyen: 'p*/traction/*_trac.dat', que enumera las fuerzas ejercidas por las células sobre el hidrogel; «p*/traction/*_domain.dat», que enumera la ubicación de los puntos de la cuadrícula que contienen celdas; y los archivos de entrada 'p*/traction.in, p*/clusterInput.txt' y 'p*/prestress.in' que registran las opciones del usuario para este paso. Tras la primera evaluación cuantitativa de la ECM celular y de las fuerzas célula-célula, se han desarrollado varias variaciones de la técnica1,15. Las variaciones se centran en casos particulares de sustratos, células, condiciones experimentales o herramientas numéricas7,8,21,22. Consulte la Figura 8 para la descripción visual que se realizará a través de AnViM, la microscopía de tracción por transformada de Fourier y el análisis de microscopía de esfuerzo monocapa en los datos de deformación del hidrogel1,2,15.- Inicie el software de Fiji y, en el menú desplegable MSM , seleccione MSM - Cell-ECM y Cell-Cell Forces (tercera opción en el menú desplegable).
- Utilizando el cuadro de diálogo del explorador de archivos, seleccione el directorio principal de la carpeta 'análisis' que contiene las carpetas 'tnimgs' y 'análisis' que contienen los datos analizados. Haga clic en Seleccionar.
- En la ventana Parámetros para calcular fuerzas celulares , introduzca el módulo de cizallamiento, el grosor del hidrogel y el nivel de ruido previsto. Seleccione Aceptar. Esto le permite ejecutar la tracción a través de la función MATLAB.
NOTA: Después de estas entradas, AnViM calcula las fuerzas de celda-ECM. - Después de eso, indique si la monocapa es confluente. Si toda la imagen de la celda está cubierta de células, entonces la respuesta es Sí. En ese caso, las fuerzas célula-célula se calcularán en todo el marco. Por otro lado, si una parte de la imagen no tiene celdas, entonces la respuesta es No. En ese caso, siga las indicaciones de AnViM para facilitar la segmentación de la región de la imagen que contiene las celdas.
- Si la respuesta es no, dibuje manualmente un polígono alrededor del objeto que no es de celda cuando el software se lo solicite y, a continuación, seleccione un método o métodos apropiados para la segmentación. El software le pedirá el color (blanco o negro) de las celdas.
- Marque la opción Rellenar puntos automáticamente en el software y haga clic en Aceptar.
NOTA: Los pasos para segmentar una monocapa no confluente se tratarán en la primera parte de la sección 2.4. Las fuerzas de Cell-ECM se almacenan en archivos '*_trac.dat' en un directorio recién creado 'traction' dentro de cada carpeta de posición, y la entrada al software de cálculo de fuerza cell-ECM se guarda en el archivo 'traction.in' (Figura suplementaria S8). Las fuerzas celda-celda se almacenan en archivos '*_prestress.dat' en un directorio recién creado 'prestress' dentro de cada carpeta de posición, y la entrada al software de cálculo de fuerza celda-celda se guarda en el archivo 'prestress.in' (Figura suplementaria S9).
- Asignación de valores de puntos de cuadrícula en celdas individuales
NOTA: Uno de los enfoques actuales de iTACS es introducir un enfoque directo para interpretar las señales mecánicas medidas en el campo. Este enfoque es beneficioso para examinar las interacciones mecánicas entre las células vecinas de un grupo.18. En general, las propiedades cuantificadas hasta ahora se encuentran en puntos de cuadrícula espaciados regularmente a través del clúster celular. Estos datos identifican la mediana, la media y la desviación estándar de las propiedades seleccionadas dentro del límite morfológico de las células individuales y las asignan como propiedades/ señales físicas celulares. A partir de estos, la desviación estándar se utiliza para indicar la variabilidad, la diferencia entre la media y la mediana se utiliza para indicar la naturaleza de la distribución, y el valor medio se utiliza para indicar el estado general de las celdas para las propiedades seleccionadas.- Parte 1: Segmentación de la región de la imagen que contiene celdas
NOTA: La entrada manual a este paso incluye la identificación del directorio de datos y la respuesta a las solicitudes de segmentación para detectar celdas de la región sin celdas. Los archivos clave creados previamente utilizados en este paso incluyen 'p*/phs/*.tif'. Los nuevos archivos clave producidos en este paso incluyen 'p*/phs/bwImages/*.tif', que son imágenes binarias que separan las regiones celulares de las regiones sin células, 'p*/phs/textResults/frameNum_percentHealed.txt', que enumera el porcentaje de área de imagen cubierta por celdas en cada instancia, y 'p*/clusterInput.txt', que registra las elecciones de usuario ingresadas en la interfaz AcTrM. Consulte la figura 9 para obtener la descripción visual de los pasos siguientes para segmentar la región de la imagen que contiene celdas. Esta parte debe completarse antes de calcular las fuerzas célula-célula. En ese caso, no es necesario repetir estos pasos. Además, si estos pasos se llevan a cabo antes de los cálculos de fuerza celda-celda, no se solicitan al principio de los cálculos de fuerza.- Inicie el software de Fiji y, en el menú desplegable de MSM , seleccione Segmentación - Clúster celular.
- Utilizando el cuadro de diálogo del explorador de archivos, seleccione el directorio de posición 'analysis/p0', 'analysis/p1', etc., que contiene propiedades cuantificadas en puntos de cuadrícula espaciados regularmente.
- Indique si la monocapa es confluente.
NOTA: Los pasos siguientes se invocan solo cuando la monocapa no es confluente. Siga las indicaciones de AnViM para aplicar el novedoso enfoque multifacético para identificar las regiones de imagen que contienen celdas. El proceso involucra cuatro métodos, cada uno de los cuales aborda la segmentación de una manera diferente. Uno o varios de estos métodos utilizados en combinación cubren una amplia variedad de imágenes de las células. Por lo tanto, pruebe diferentes enfoques para encontrar qué combinación funciona mejor para sus datos. - Ajuste el 'Factor de conexión de dominio' y el 'Factor de frotis' para obtener una segmentación óptima.
NOTA: El 'Factor de frotis' hace que las regiones que contienen células sean más grandes. - Indique si las celdas aparecen en blanco o negro en la imagen binaria.
- En la ventana Instrucciones para limpiar la imagen de límite , elija si desea limpiar la imagen de forma automática o manual.
NOTA: Las características no deseadas de las imágenes son puntos negros en los píxeles ocupados por las celdas y manchas blancas en los píxeles desconectados del grupo de celdas que se va a analizar. El análisis solo se puede hacer en las regiones que están conectadas. Por lo tanto, varias regiones desconectadas deben analizarse por separado.
- Parte 2: Segmentación de las células individuales en las imágenes
NOTA: Las entradas manuales a este paso incluyen la identificación del directorio de datos y la respuesta a las solicitudes de segmentación para detectar celdas individuales dentro de la imagen. Los archivos clave creados previamente utilizados en este paso incluyen 'p*/phs/*.tif' y 'p*/phs/bwImages/*.tif'. Los nuevos archivos clave producidos incluyen 'p*/phs/textResults/*.csv', que contienen propiedades morfológicas celulares. Consulte la Figura 10 para obtener la descripción visual de los siguientes pasos para segmentar celdas individuales de la monocapa.- Inicie el software de Fiji y, en el menú desplegable de MSM , seleccione Segmentación - Celdas individuales.
- Utilizando el cuadro de diálogo del explorador de archivos, seleccione el directorio de posición 'analysis/p0', 'analysis/p1', etc., que contiene propiedades cuantificadas en puntos de cuadrícula espaciados regularmente.
- En la ventana Elegir los parámetros de entrada , indique la relación de aspecto máxima, la prominencia de cuánto se destaca el centro de celdas en comparación con el límite de la celda y dos parámetros de desenfoque para guiar la detección de las celdas (Figura suplementaria S10).
- En la pila de imágenes para cada posición, dibuje un polígono en la celda normal más pequeña. Esto se utiliza para calcular el área. Cualquier cosa más pequeña que esto no será considerada como una celda por el software.
- A continuación, dibuje un polígono alrededor de la celda normal más grande e indique si el centro celular o la interfaz celda-celda es más brillante.
NOTA: AnViM crea archivos 'phs/textResults/0001.csv', 'phs/textResults/0002.csv', etc., para cada fotograma que contiene información sobre las celdas dentro de ese marco. En esta etapa, este archivo incluye información morfológica sobre las células, incluyendo área, centroide, perímetro, orientación, circularidad, relación de aspecto, redondez, solidez, distancia de la región sin células. La unidad de longitud en estas propiedades es píxeles y el ángulo está en grados. Finalmente, este archivo se actualiza para contener la intensidad, el movimiento y las fuerzas de los píxeles celulares en los pasos siguientes.
- Parte 3: Mapeo de las intensidades de píxeles en las celdas
NOTA: Las entradas manuales en este paso incluyen la identificación del directorio de datos y la selección de propiedades y parámetros de asignación. Los archivos clave creados previamente utilizados en este paso incluyen 'p*/phs/*.tif' (o 'p*/fluo/*.tif ') y 'p*/phs/bwImages/*.tif'. Los nuevos archivos clave producidos en este paso incluyen 'p*/phs/textResults/*.csv', que enumera las propiedades morfológicas celulares. Consulte la Figura 11 para obtener la descripción visual de los siguientes pasos para evaluar las intensidades de píxeles en la región abarcada por celdas individuales y su región vecina y definirlas como propiedades de las celdas.- Inicie el software fiji y, en el menú desplegable MSM , seleccione Mapa en celdas - Intensidades.
- Utilizando el cuadro de diálogo del explorador de archivos, seleccione el directorio de posición 'analysis/p0', 'analysis/p1', etc., que contiene propiedades cuantificadas en puntos de cuadrícula espaciados regularmente.
- Seleccione Detectar división celular o Cuantificar fluorescencia celular cuando el software se lo solicite. Defina el tamaño de la región vecina. Marque las casillas Recopilar propiedades de la región vecina y Recopilar propiedades de las celdas. Haga clic en Aceptar.
- En la ventana Propósito del registro de la intensidad celular, indique qué tipo de imagen se utilizará para el mapeo de intensidad, el tamaño de la región vecina y si se recopilan datos para celdas individuales o ambas celdas y sus regiones vecinas (Figura suplementaria S11).
NOTA: El mapeo de las intensidades de píxeles de una imagen de contraste de fase permite la detección de eventos de división celular. El mapeo de intensidades de imagen fluorescente permitirá la detección de fluctuaciones en las moléculas citoplasmáticas marcadas fluorescentemente. La salida de los pasos anteriores es la media, la mediana, la desviación estándar, el mínimo y el máximo de los valores de intensidad de píxeles para celdas individuales. Estos números se introducen como nuevas columnas en los archivos 'phs/textResults/0001.csv', 'phs/textResults/0002.csv', etc.
- Parte 4: Mapeo de las fuerzas y el movimiento en las células
NOTA: Las entradas manuales a este paso incluyen la identificación del directorio de datos y la selección de propiedades y parámetros de asignación. Los archivos clave creados previamente utilizados en este paso incluyen 'p*/phs/textResults/*.csv', 'p*/velocity/*_vel.dat', 'p*/displacement/*_disp.dat', 'p*/traction/*_trac.dat', 'p*/prestress/*_prestress.dat' y 'p*/phs/bwImages/*.tif '. No se crean nuevos archivos durante este paso. En su lugar, la nueva información (fuerza celular y propiedades de movimiento) se agrega a los archivos 'p*/phs/textResults/*.csv '. Consulte la Figura 12 para la descripción visual de los pasos para evaluar las fuerzas y el movimiento en la región abarcada por las células individuales y su región vecina y definirlos como propiedades de las células.- Elija en el menú desplegable MSM Mapa de celdas - Datos de movimiento/fuerza (Figura suplementaria S12).
- A continuación, siga los pasos enumerados en el paso 2.4.3.
NOTA: Se añaden nuevas columnas a los archivos 'phs/textResults/0001.csv', 'phs/textResults/0002.csv', etc. e incluyen la media, mediana y desviación estándar de la velocidad, la orientación de la velocidad, la tensión citoesquelética media, la anisotropía de tensión, la energía de deformación en el hidrogel, la orientación de la tensión más alta y la magnitud de la tracción célula-ECM (Figura suplementaria S13).
- Parte 1: Segmentación de la región de la imagen que contiene celdas
- Visualización de resultados
- Parte 1: Seguimiento de las identidades celulares a través del experimento
NOTA: Las entradas manuales a este paso incluyen la identificación del directorio de datos y la selección de propiedades asignadas para visualizar. Los archivos clave creados previamente utilizados en este paso incluyen 'p*/phs/textResults/*.csv'. Los nuevos archivos clave producidos durante este paso incluyen 'p*/phs/textResults/cellTimeTrace_*.csv', que contiene un rastro de tiempo de los datos celulares. Consulte la Figura 13 para obtener la descripción visual de los siguientes pasos para realizar un seguimiento de las propiedades de las celdas individuales durante toda la duración del experimento.- Inicie el software de Fiji y, en el menú desplegable de MSM , seleccione Resultados - Seguimiento de datos.
- Usando el cuadro de diálogo del navegador de archivos, seleccione el directorio de posición 'analysis/p0', 'analysis/p1', etc., que contiene las propiedades celulares que se van a rastrear. Cuando haya terminado, haga clic en Seleccionar.
- En la ventana Elegir punto de partida para el seguimiento , elija el marco de inicio para la supervisión y el número de fotogramas de seguimiento simultáneos (Figura suplementaria S14). Siempre comience desde el fotograma número 2, ya que la velocidad no se puede determinar para el fotograma número 1. Cuando haya terminado, haga clic en Aceptar.
- En la ventana Comprobar las variables para realizar seguimientos , elija las variables escribiendo los nombres de las variables o seleccionando los términos de las casillas de verificación (Figura suplementaria S15). Luego, haga clic en Aceptar.
NOTA: El seguimiento genera archivos 'phs/textResults/cellTimeTrace_*.csv' con cada columna que contiene un número de celda único y cada fila sucesiva que representa la instancia de tiempo consecutiva.
- Parte 2: Generación de pistas de tiempo de las propiedades celulares evaluadas
NOTA: Las entradas manuales a este paso incluyen la identificación del directorio de datos y la selección de propiedades asignadas para visualizar. Los archivos clave creados previamente utilizados en este paso incluyen 'p*/phs/textResults/cellTimeTrace_*.csv'. Los nuevos archivos clave producidos durante este paso incluyen 'p*/phs/trackedDataPlots/*.tif', que contienen gráficos de seguimiento de tiempo y 'p*/phs/trackedDataPlots/*.csv', que contienen datos de gráficos. Consulte la Figura 14 para obtener la descripción visual de los siguientes pasos para generar seguimientos de tiempo de las propiedades celulares evaluadas.- Inicie el software de Fiji y, en el menú desplegable de MSM , seleccione Resultados - gráfico.
- Usando el cuadro de diálogo del navegador de archivos, seleccione el directorio de posición 'analysis/p0', 'analysis/p1', etc., que contiene las propiedades celulares rastreadas que se van a trazar.
- Elija si desea limitar el trazado a celdas con pistas ininterrumpidas haciendo clic en el botón Sí o No .
Nota : esta opción omite las celdas, que no se pudieron detectar en una o más instancias de tiempo. - Elija el número de variables que se trazarán simultáneamente y haga clic en Aceptar.
NOTA: Actualmente, AnViM permite mostrar un máximo de tres variables en la misma gráfica. - Elija variables individuales para trazar mediante un menú desplegable.
NOTA: Estos pasos crean un archivo de formato de archivo de imagen etiquetado (TIFF) y un archivo de datos separado por comas dentro de la carpeta 'phs/trackedDataPlots/'. El nombre de archivo se compone de nombres de variables separados por un guión bajo y termina con un número de celda.
- Parte 3: Generación de mapas de calor de las propiedades celulares evaluadas
NOTA: Las entradas manuales en este paso incluyen la identificación del directorio de datos y la selección de propiedades asignadas para visualizar. Los archivos clave creados previamente utilizados en este paso incluyen 'p*/phs/textResults/cellTimeTrace_*.csv'. Los nuevos archivos clave generados en este paso incluyen 'p*/phs/segmentedImages/cellPropMaps/*.tif', que son los mapas de calor de las propiedades celulares. Consulte la Figura 15 para obtener la descripción visual de los pasos para generar mapas de calor de las propiedades celulares evaluadas.- Elija en el menú desplegable de MSM Resultados - Imagen.
- Siga los pasos descritos en el paso 2.5.2.
NOTA: La salida se almacena como archivos de archivo TIFF en la carpeta 'phs/segmentedImages/cellPropMaps/'. Los nombres de archivo son el número de instancia de tiempo y el nombre de variable separados por un guión bajo.
- Parte 1: Seguimiento de las identidades celulares a través del experimento
Resultados
Presentamos aquí dos de los resultados clave para el ejemplo demostrado. La primera salida es el rastro de tiempo de la velocidad celular y la tensión citoesquelética para la célula número 1 (Figura 16). Las propiedades se muestran en un eje vertical compartido para facilitar la asociación visual entre las propiedades, y el eje horizontal indica el número de instancia de tiempo. En este experimento, se adquirieron fotogramas sucesivos en un intervalo de 15 minutos. La segunda salida es una matriz de mapas de calor 1 h en el experimento (Figura 17). Las propiedades que se muestran aquí incluyen área de propagación, orientación, circularidad, velocidad, dirección de movimiento, orientación de tensión máxima, tensión citoesquelética, tracciones de sustrato y anisotropía de tensión de células individuales.

Figura 1: Estructura del kit de herramientas integradoras para analizar señales celulares (iTACS). Dos componentes clave de iTACS son el Módulo de Adquisición y Capacitación (AcTrM) y el Módulo de Análisis y Visualización (AnViM). AcTrM puede utilizar varias técnicas de preparación de hidrogel que existen actualmente para preparar hidrogeles que se pueden mantener firmemente en una etapa de microscopio, cualquier siembra celular y un protocolo de crecimiento que retiene las células en un plano focal. AnViM puede utilizar varias técnicas para cuantificar la deformación del hidrogel y la monocapa, las fuerzas de ECM de célula y las fuerzas de célula-célula. Todos estos componentes preferidos por el usuario del protocolo de mediciones de fuerza se pueden acomodar en iTACS, y se han identificado con cajas discontinuas. Los componentes identificados con cajas sólidas son contribuciones novedosas a la tecnología de medición de fuerza celular. La visualización en AnViM se centra en el valor medio y la variabilidad de las propiedades entre celdas individuales. Haga clic aquí para ver una versión más grande de esta figura.
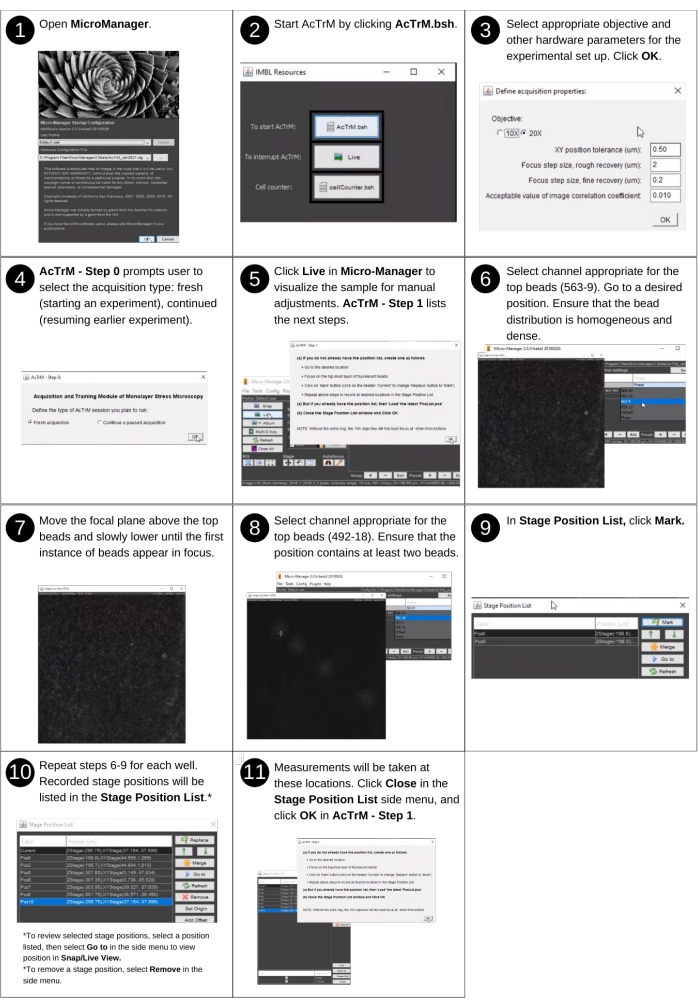
Figura 2: Adquisición de imágenes de referencia - parte 1. Pasos para crear una lista de posiciones usando AcTrM. Haga clic aquí para ver una versión más grande de esta figura.
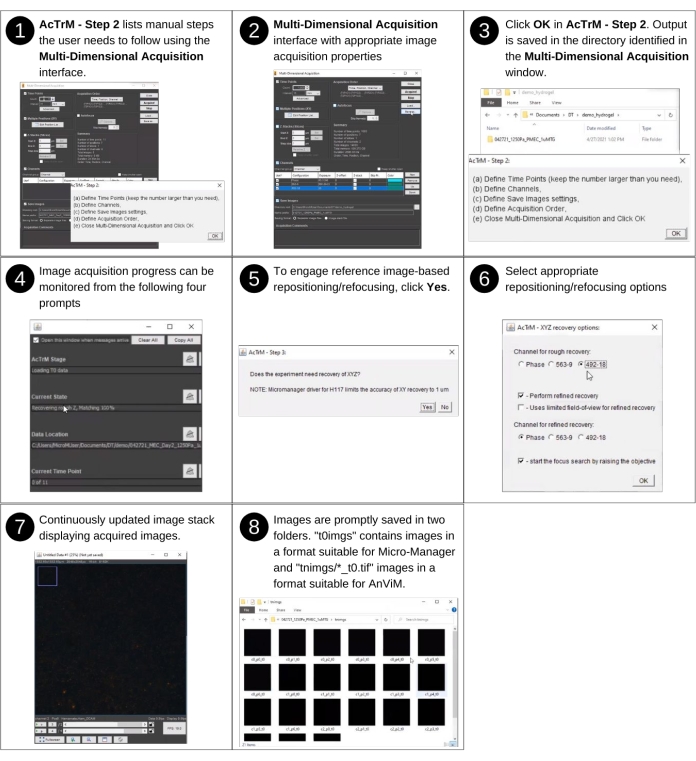
Figura 3: Adquisición de imágenes de referencia - parte 2. Pasos para adquirir imágenes de referencia utilizando AcTrM. Las vistas detalladas de los pasos 2, 4 y 6 se presentan en las figuras suplementarias S2, S3 y S4, respectivamente. Haga clic aquí para ver una versión más grande de esta figura.
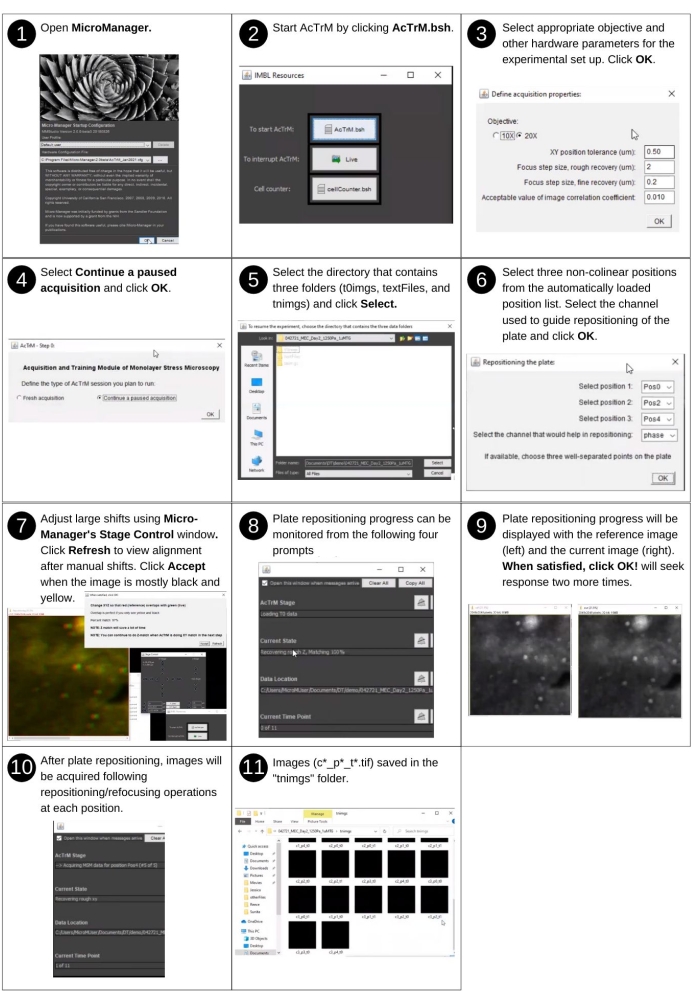
Figura 4: Adquisición automatizada de imágenes para el experimento restante. Pasos para reanudar la adquisición de imágenes para evaluar el comportamiento celular utilizando AcTrM. Haga clic aquí para ver una versión más grande de esta figura.

Figura 5: Configuración del análisis automatizado de datos. Pasos para comenzar el análisis automatizado de imágenes utilizando AnViM. El software reconoce el formato de imagen utilizado por AcTrM. En la figura suplementaria S5 y en la figura suplementaria S6 , respectivamente, se presenta una vista detallada de los paneles de las etapas 3 y 5. Haga clic aquí para ver una versión más grande de esta figura.
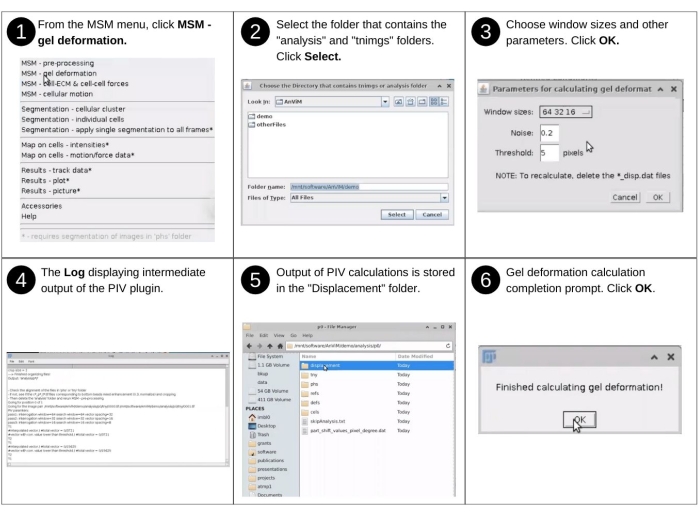
Figura 6: Cuantificación de la deformación de hidrogel y monocapa - parte 1. Pasos para involucrar, a través de AnViM, la implementación de La Velocimetría de Imagen de Partículas de Tseng, Q. et al., PNAS (2012)20 para cuantificar la deformación de la superficie superior del hidrogel. Los usuarios también pueden implementar dentro de AnViM otros enfoques para cuantificar la deformación del hidrogel. En la figura suplementaria S7 se presenta una vista detallada del paso 3. Haga clic aquí para ver una versión más grande de esta figura.
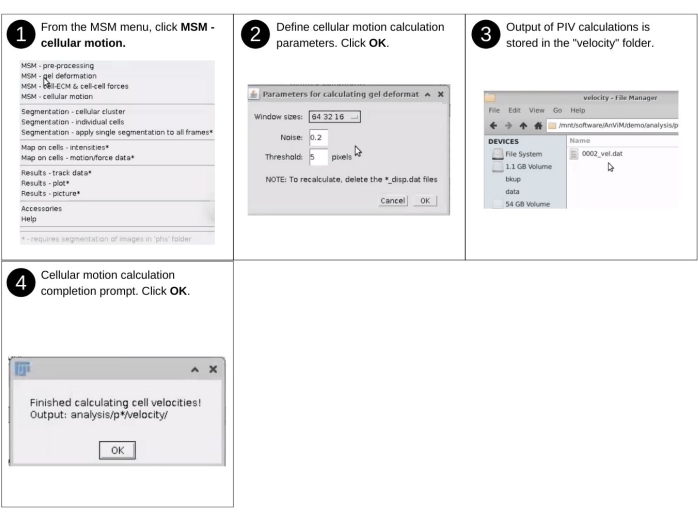
Figura 7: Cuantificación de la deformación de hidrogel y monocapa - parte 2. Pasos para involucrar, a través de AnViM, la implementación de la Velocimetría de Imagen de Partículas de Tseng, Q. et al., PNAS (2012)20 para cuantificar el movimiento local de células individuales. Los usuarios también pueden implementar dentro de AnViM otros enfoques para cuantificar el movimiento celular. Haga clic aquí para ver una versión más grande de esta figura.
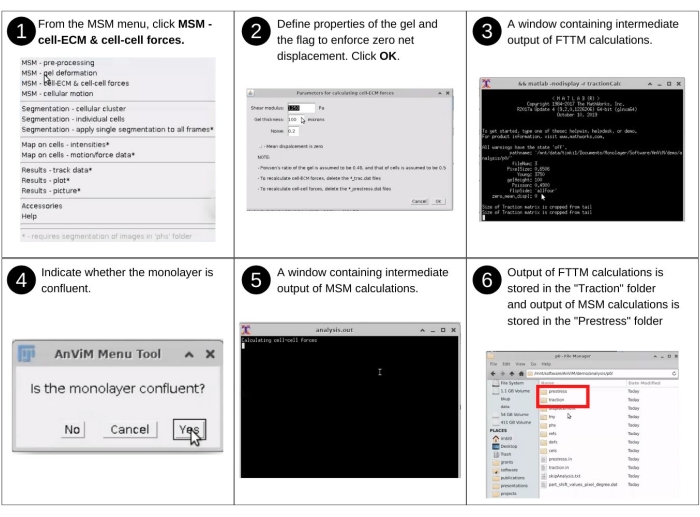
Figura 8: Cuantificación de las fuerzas célula-ECM y célula-célula. Pasos para realizar análisis de imágenes para involucrar, a través de AnViM, la implementación de la Microscopía de Tracción de Transformada de Fourier de Trepat et al., Nature Physics (2009)15 para cuantificar las fuerzas ejercidas por las células sobre el hidrogel, y la implementación de microscopía de estrés monocapa de Tambe et al., Nature Materials (2011)1 para cuantificar las fuerzas dentro de las células individuales y entre las células vecinas. Los usuarios también pueden implementar dentro de AnViM otros enfoques para cuantificar las fuerzas célula-ECM y célula-célula. Una vista detallada del paso 6 se presenta en la Figura suplementaria S8 y la Figura suplementaria S9. Haga clic aquí para ver una versión más grande de esta figura.
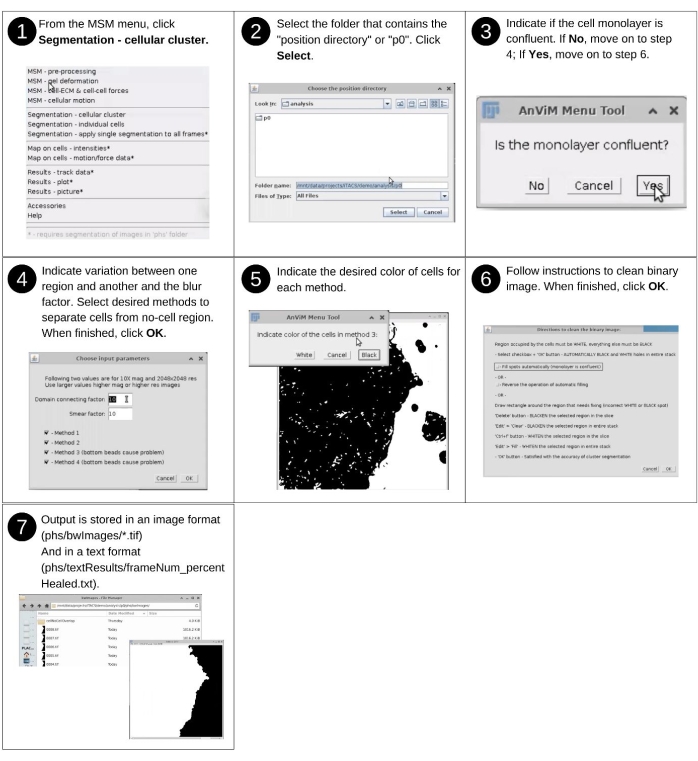
Figura 9: Asignación de valores de puntos de cuadrícula en celdas individuales - parte 1. Pasos para segmentar las regiones de imagen que contienen células utilizando un nuevo enfoque múltiple. Este enfoque se puede utilizar para segmentar el contraste de fase, el campo brillante o las imágenes de fluorescencia de las células. Haga clic aquí para ver una versión más grande de esta figura.
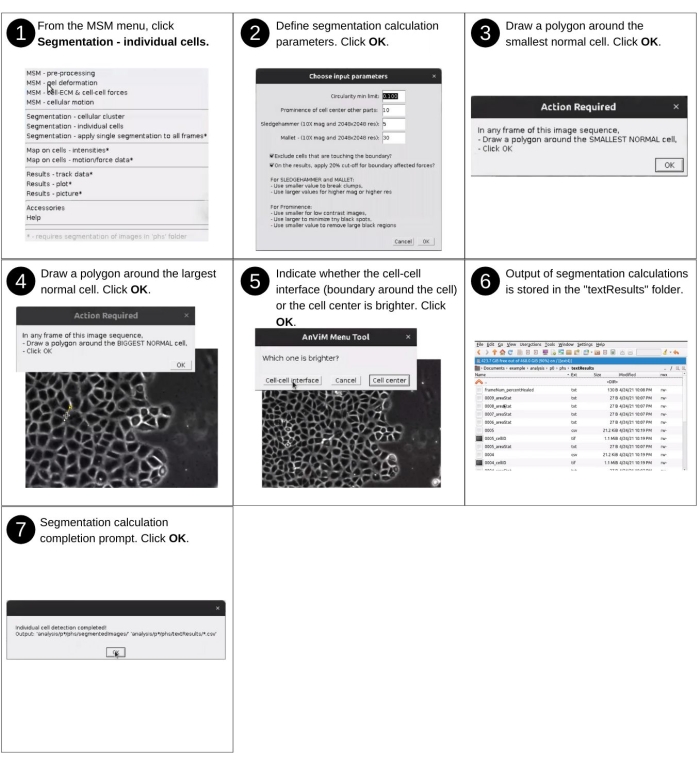
Figura 10: Asignación de valores de puntos de cuadrícula en celdas individuales - parte 2. Pasos para segmentar células individuales de una monocapa utilizando un nuevo enfoque multifacético desarrollado en AnViM. Este enfoque se puede utilizar para segmentar el contraste de fase, el campo brillante o las imágenes de fluorescencia de las células. En la figura suplementaria S10 se presenta una vista detallada del paso 2. Haga clic aquí para ver una versión más grande de esta figura.
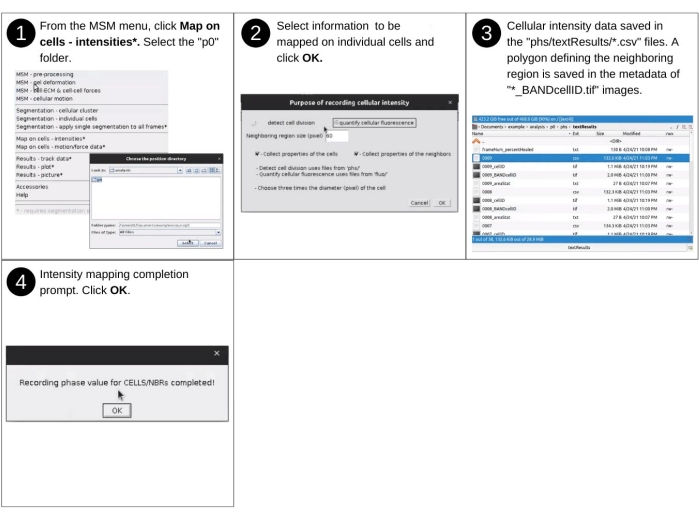
Figura 11: Asignación de valores de puntos de cuadrícula en celdas individuales - parte 3. Pasos para evaluar las intensidades de píxeles en la región dentro de las celdas individuales y dentro de la región vecina de las celdas individuales utilizando AnViM. Las intensidades evaluadas incluyen la intensidad de la luz transmitida y la intensidad de la fluorescencia. Esta parte mapea el valor medio y la desviación estándar de las intensidades de píxeles dentro de celdas individuales y dentro de una región vecina de celdas individuales. En la figura suplementaria S11 se presenta una vista detallada del paso 2. Haga clic aquí para ver una versión más grande de esta figura.
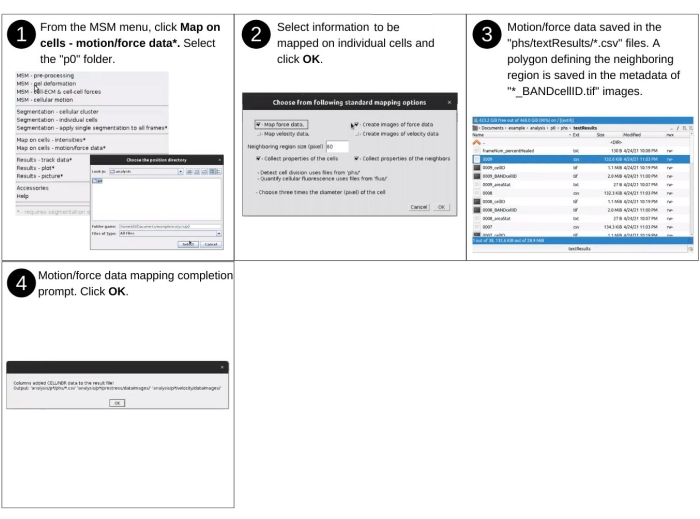
Figura 12: Asignación de valores de puntos de cuadrícula en celdas individuales - parte 4. Pasos para evaluar las fuerzas y las propiedades de movimiento de los puntos de la cuadrícula dentro de las celdas individuales y dentro de la región vecina de las celdas individuales utilizando AnViM. Esta parte mapea el valor medio y la desviación estándar de las propiedades dentro de celdas individuales y dentro de una región vecina de celdas individuales. Una vista detallada de los pasos 2 y 3 se presenta en la Figura Suplementaria S12 y la Figura Suplementaria S13. Haga clic aquí para ver una versión más grande de esta figura.
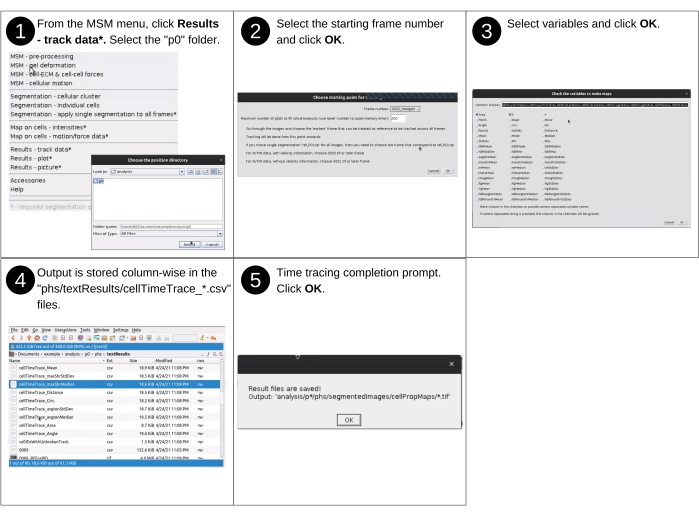
Figura 13: Visualización de resultados - parte 1. Pasos para realizar un seguimiento de las propiedades de las celdas individuales durante toda la duración del experimento utilizando AnViM. Una vista detallada de los pasos 2 y 3 se presenta en la Figura Suplementaria S14 y la Figura Suplementaria S15. Haga clic aquí para ver una versión más grande de esta figura.
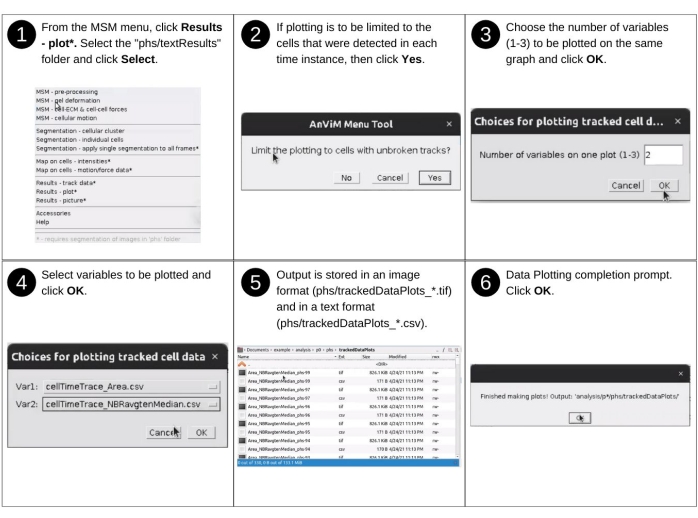
Figura 14: Visualización de resultados - parte 2. Pasos para generar trazas de tiempo de las propiedades evaluadas mediante AnViM. El usuario tiene la opción de trazar hasta tres propiedades en un gráfico. Se generan rastros de tiempo para todas las células o solo para aquellas células para las que el seguimiento fue exitoso en todo el experimento. Una vista detallada del paso 5 se presenta en la Figura suplementaria S16 y la Figura suplementaria S17. Haga clic aquí para ver una versión más grande de esta figura.
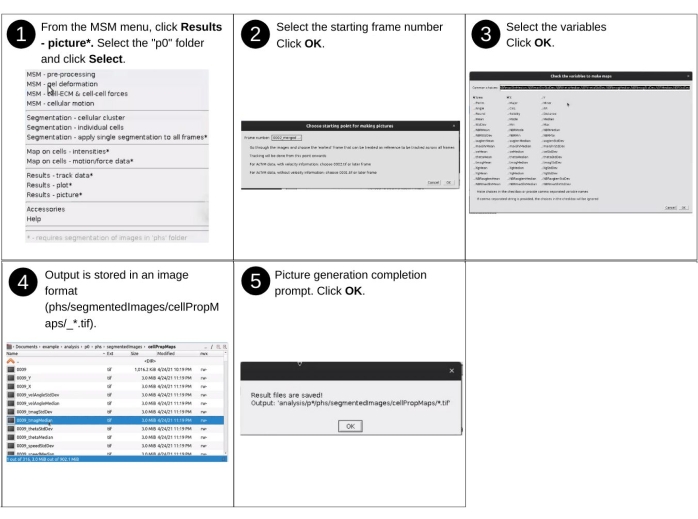
Figura 15: Visualización de resultados - parte 3. Pasos para generar mapas de calor de las propiedades evaluadas utilizando AnViM. Se generan mapas de calor para todos los fotogramas que siguen al fotograma inicial y todas las propiedades seleccionadas. En la figura complementaria S18 se presenta una vista detallada del paso 3. Haga clic aquí para ver una versión más grande de esta figura.
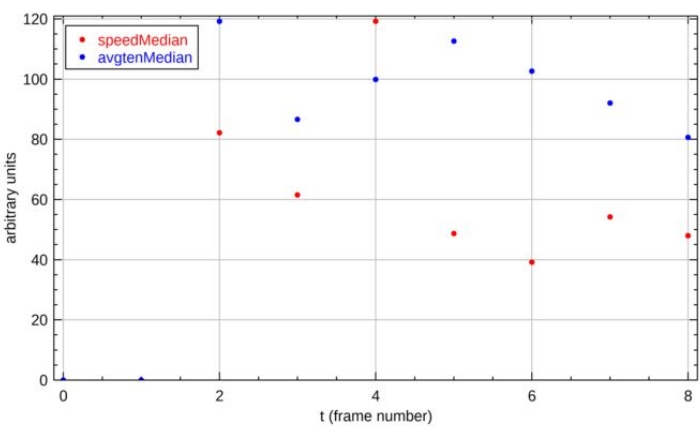
Figura 16: Trazas de tiempo para el ID de celda 1. Dos propiedades mostradas son la tensión citoesquelética celular ("avgtenMedian") y la velocidad celular ("speedMedian"). Tanto la tensión citoesquelética celular como la velocidad celular se cuantifican como el valor medio a través de los puntos de la cuadrícula dentro de las células. Las dos propiedades se trazan en el mismo eje con unidades arbitrarias para visualizar las relaciones entre las propiedades evaluadas. Los nombres de variables adicionales se enumeran en la Tabla suplementaria S1. Haga clic aquí para ver una versión más grande de esta figura.
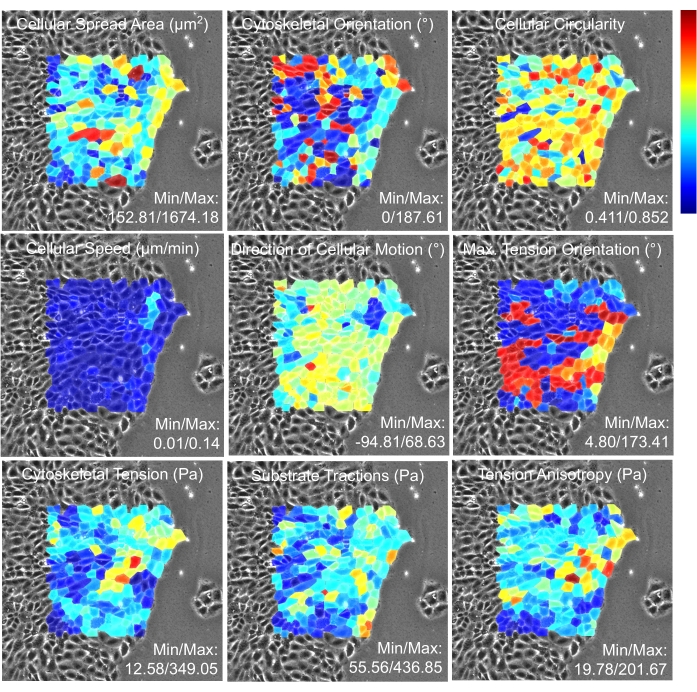
Figura 17: Mapas de calor de las propiedades de las células individuales a través de la monocapa analizada. Cada celda se colorea con el valor medio de la propiedad indicada en el panel. Por lo tanto, el rojo profundo indica el valor celular máximo en el espectro de color, y el azul profundo indica el valor celular mínimo en toda la monocapa analizada. Como se describe en Tambe et al., PLoS One (2013)2, las células ubicadas más cerca del límite tienen fuerzas mecánicas afectadas por propiedades desconocidas de las células fuera de la imagen. Por lo tanto, el mapa de calor se genera para las células lejos del límite. Haga clic aquí para ver una versión más grande de esta figura.
Figura S1: Imágenes de muestra de la cuenta fluorescente superior e inferior. Haga clic aquí para descargar este archivo.
Figura S2: Una vista detallada del paso 2 de la Figura 3. Haga clic aquí para descargar este archivo.
Figura S3: Una vista detallada del paso 4 de la Figura 3. Haga clic aquí para descargar este archivo.
Figura S4 A: vista detallada del paso 6 de la Figura 3. Haga clic aquí para descargar este archivo.
Figura S5: Una vista detallada del paso 3 de la Figura 5. Haga clic aquí para descargar este archivo.
Figura S6: Una vista detallada del paso 5 de la Figura 5. Haga clic aquí para descargar este archivo.
Figura S7: Una vista detallada del paso 3 de la Figura 6. Haga clic aquí para descargar este archivo.
Figura S8: Una vista detallada de la salida de fuerza celda-ECM del paso 6 de la Figura 8. Haga clic aquí para descargar este archivo.
Figura S9: Una vista detallada de la salida de fuerza celda-celda del paso 6 de la Figura 8. Haga clic aquí para descargar este archivo.
Figura S10: Una vista detallada del paso 2 de la Figura 10. Haga clic aquí para descargar este archivo.
Figura S11: Una vista detallada del paso 2 de la Figura 11. Haga clic aquí para descargar este archivo.
Figura S12: Una vista detallada del paso 2 de la Figura 12. Haga clic aquí para descargar este archivo.
Figura S13: Una vista detallada de la salida del paso 3 de la Figura 12. Haga clic aquí para descargar este archivo.
Figura S14: Una vista detallada del paso 2 de la figura 13. Haga clic aquí para descargar este archivo.
Figura S15: Una vista detallada del paso 3 de la Figura 13. Haga clic aquí para descargar este archivo.
Figura S16: Una vista detallada de los archivos de datos generados en el paso 5 de la Figura 14. Haga clic aquí para descargar este archivo.
Figura S17: Una vista detallada de un gráfico generado en el paso 5 de la Figura 14. Haga clic aquí para descargar este archivo.
Figura S18: Una vista detallada de un mapa de calor y el archivo que contiene el rango del espectro de color generado en el paso 4 de la Figura 15. Haga clic aquí para descargar este archivo.
Tabla S1: Una lista de propiedades seleccionadas cuantificadas por iTACS. Haga clic aquí para descargar esta tabla.
Discusión
Las células adherentes utilizan señales mecánicas y químicas para sobrevivir, crecer y funcionar. Una amplia variedad de software de microscopía optimiza la experiencia del usuario en la evaluación de las señales químicas a través de imágenes basadas en fluorescencia. Sin embargo, la evaluación de las señales mecánicas implica capacidades que no están disponibles en el software de microscopía estándar. Además, la evaluación de señales mecánicas es más eficiente cuando la adquisición de datos se integra con el análisis de datos. La falta de una plataforma unificada que satisfaga las necesidades únicas de la evaluación mecánica de señales ha sido una brecha tecnológica importante en la biología celular experimental. El Kit de herramientas integradoras para analizar señales celulares (iTACS) está diseñado para satisfacer esta brecha. Los dos componentes de iTACS, AnViM y AcTrM, equipan a los usuarios con las capacidades necesarias para cuantificar las propiedades celulares de cuatro grandes categorías: fuerzas, movimiento, morfología y fluorescencia / brillo. A través de estas categorías, iTACS es actualmente capaz de revelar más de 50 aspectos únicos de células adherentes individuales. Estos aspectos comprenden propiedades específicas de cada categoría amplia, incluyendo su valor representativo y variabilidad a través de la celda (Tabla suplementaria S1). Por ejemplo, dentro de las fuerzas, hay fuerzas de tracción a través del citoesqueleto, anisotropía de esta tensión, la orientación de la tensión máxima y tensión de corte a través de la interfaz célula-ECM que tiene una profunda influencia en el comportamiento de las células adherentes1,3,6.
Un enfoque novedoso para examinar el comportamiento mecánico de células individuales de una monocapa
Las células individuales de una monocapa participan en un intercambio de señales de naturaleza química y mecánica3. Estos dos tipos de señales se transmiten a través de la monocapa celular de una manera diferente23. Sin embargo, el conocimiento de la transmisión de señales mecánicas va a la zaga del de la transmisión de señales químicas. Esta brecha de conocimiento coincide con una falta sostenida de enfoques simples e intuitivos para evaluar las señales mecánicas celulares. El novedoso enfoque de mapeo de datos descrito aquí está equipado para llenar este vacío. Dicho mapeo revela que la fluctuación de la tensión citoesquelética intrínseca en la región vecina de una célula sirve como señales de relajación, fluidización y anclaje que regulan los cambios en la forma, el tamaño y la velocidad celular de la célula18. Los mapas de las propiedades de las regiones vecinas exhiben patrones de "subdivisión multicelular" donde las células dentro de la subdivisión están expuestas a un microambiente relativamente uniforme y las células en el límite de la subdivisión están expuestas a un microambiente notablemente no uniforme18.
Accesibilidad de la tecnología de medición de fuerza
Existe una variedad de protocolos para fabricar hidrogeles PAA, analizar la deformación del hidrogel y el movimiento celular, y cuantificar las fuerzas célula-ECM y célula-célula1,2,7,8,9,13,14,15,18,20,21,24,25,26,27 ,28,29,30,31,32. Sin embargo, estos desarrollos permanecen fuera del alcance de los laboratorios comunes de biología celular y confinados a laboratorios con experiencia en ingeniería. Al automatizar los aspectos técnicos de estos enfoques e integrarlos bajo una plataforma unificada y fácil de usar, el objetivo de iTACS es hacer de la evaluación de señales mecánicas una actividad rutinaria en la investigación y educación en biología celular experimental.
ImageJ permite a los usuarios desarrollar aplicaciones utilizando enfoques que requerirían poca o ninguna capacitación11. iTACS se construye en gran medida utilizando enfoques de scripting simples para facilitar el desarrollo continuo impulsado por la comunidad. Una gran parte de AcTrM se programa utilizando scripts de BeanShell, y la mayor parte de AnViM se programa utilizando imageJ Macros. Estos scripts y la guía para implementar estas capacidades en el microscopio del usuario están disponibles a través de GitHub (https://github.com/IntegrativeMechanobiologyLaboratory/iTACS).
Estandarización de la calidad de las imágenes adquiridas
Aunque las técnicas basadas en sustrato elástico para cuantificar las fuerzas físicas en las células adherentes se han desarrollado e implementado en varios laboratorios, el protocolo aún carece de estandarización. Un área que más necesita estandarización es la calidad de las imágenes de cuentas superiores adquiridas (Figura suplementaria S1). Problemas significativos surgen de la deriva en el enfoque a lo largo del experimento. Nuestro novedoso enfoque de reenfoque basado en imágenes de referencia hace que dicho enfoque sea un proceso objetivo. Los parámetros definidos en el primer paso de AcTrM imponen los límites de calidad objetivos necesarios. Otras medidas de estandarización pueden programarse en futuras versiones de AcTrM.
La amplia aplicabilidad de iTACS
Además de cuantificar numerosos aspectos de las células adherentes, la estructura iTACS facilita su uso para diversos protocolos y necesidades experimentales. AcTrM permite la autoformación del usuario guiada por software. Las imágenes de alta velocidad requeridas, por ejemplo, por la evaluación simultánea de las fluctuaciones de calcio citoplasmático están actualmente limitadas por la velocidad del hardware de reposicionamiento y reenfoque y se realizan mejor en un lugar a la vez. Sin embargo, la implementación actual está bien equipada para imágenes a largo plazo, imágenes interrumpidas, donde la muestra no se puede retener en la etapa del microscopio durante toda la duración del experimento. Dado que las imágenes de referencia se adquieren al comienzo del experimento, iTACS permite la obtención de imágenes en tiempo real de señales mecánicas, abriendo nuevas vías en aplicaciones de detección de drogas. AnViM permite a los usuarios proporcionar información altamente técnica en términos sencillos. La capacidad de cuantificar un amplio espectro de propiedades celulares y rastrearlas a lo largo del experimento constituye capacidades críticas necesarias para descubrir nuevos mecanismos de comunicación intercelular.
Para el desarrollo futuro de iTACS, hemos identificado cuatro áreas de enfoque: (1) mejora de la adquisición de datos y la velocidad de análisis de datos, (2) implementación de enfoques para evaluar nuevas señales celulares13, (3) desarrollo de talleres y módulos educativos sobre evaluación de señales celulares basada en iTACS, (4) desarrollo de soluciones de automatización de bajo costo.
Divulgaciones
Los autores declaran que no hay conflictos de intereses.
Agradecimientos
D.T.T. agradece al personal afiliado al Centro de Biología Pulmonar de la Universidad del Sur de Alabama por estimular las discusiones sobre las necesidades de investigación de biología celular experimental. Estas discusiones fueron cruciales para iniciar el desarrollo de iTACS.
Este trabajo fue apoyado en parte por subvenciones del Instituto Nacional de Salud / Instituto Nacional de Sangre Cardíaca Pulmonar, P01 HL66299 y R37 HL60024 (Stevens), R01-HL118334 (Álvarez), F32-HL144040-01 (Xu), y de la Universidad del Sur de Alabama a través del Fondo de Investigación del Cáncer Abraham Mitchell (Singh, Palanki, Tambe), Beca de Investigación y Desarrollo Académico (Tambe), Honors College y Summer Undergraduate Research Fellow (Nguyen).
Materiales
| Name | Company | Catalog Number | Comments |
| Reagents and components used in prepare glass surface for hydrogel coating | |||
| (3-Aminopropyl)trimethoxysilane, 97% | Aldrich chemistry | 13822565 | |
| 2% Bis Solution | Bio-rad | 1610142 | |
| 3-(Trimethoxysilyl)propyl methacrylate,98% | Acros organics | 2530850 | |
| 40% Acrylamide Solution | Bio-rad | 1610140 | |
| Glass bottom 35 mm dish/ 6 or 12 or 24 well plates | MatTek or CellVis | ||
| Glutaraldehyde, EM Grade, 25% | Polysciences | 1909100 | |
| Sodium Hydroxide | Sigma-aldrich | 1002074706 | |
| Reagents and components used in preparing suitable hydrogel | |||
| 2% Bis Solution | Bio-rad | 1610142 | |
| 40% Acrylamide Solution | Bio-rad | 1610140 | |
| Ammonium Persulfate | Bio-rad | 1610700 | |
| Cover Slips | Electron Microscopy Sciences | 7222301 | |
| Dulbecco's Phosphate Buffered Saline (1M) | Gibco | 14190136 | |
| FluoSpheres carboxylate 0.2 um, yellow-green(505/515) | Invitrogen | F8811 | |
| FluoSpheres carboxylate 0.5 um, red(580/605) | Invitrogen | F8812 | |
| FluoSpheres carboxylate 2.0 um, red(580/605) | Invitrogen | F8826 | |
| Rain-X | |||
| TEMED | Bio-rad | 1610801 | |
| Reagents used in coating extracellular matrix on the hydrogel | |||
| Collagen Type I Rat Tail | Corning | 354236 | |
| HEPES(1M) | Gibco | 15630080 | |
| Phosphate Buffered Saline (1M) | Gibco | 10010023 | |
| Sulfo-SANPAH | CovaChem | 102568434 | |
| Microscope hardware used in the current study | |||
| Camera | Hamamatsu Flash 4.0 LT sCMOS Camera | C11440-42U | |
| H117 ProScanTM Stages | Prior Scientific | ||
| Light source- Lambda DG4 and Lambda DG5 | Sutter instrument company | ||
| Microscope | Nikon eclipse TE2000-S | 550372 | |
| ProScan III Universal Microscope Automation Controller | Prior Scientific | ||
| Stagetop incubator | ibidi | 11922 | |
| Stepper Motor Focus Drive | Prior Scientific |
Referencias
- Tambe, D. T., et al. Collective cell guidance by cooperative intercellular forces. Nature Materials. 10 (6), 469-475 (2011).
- Tambe, D. T., et al. Monolayer stress microscopy: limitations, artifacts, and accuracy of recovered intercellular stresses. PloS One. 8 (2), 55172 (2013).
- Das, T., et al. A molecular mechanotransduction pathway regulates collective migration of epithelial cells. Nature Cell Biology. 17 (3), 276-287 (2015).
- Vedula, S. R., et al. Mechanics of epithelial closure over non-adherent environments. Nature Communications. 6, 6111 (2015).
- Lamason, R. L., et al. Rickettsia Sca4 reduces vinculin-mediated intercellular tension to promote spread. Cell. 167 (3), 670-683 (2016).
- Sunyer, R., et al. Collective cell durotaxis emerges from long-range intercellular force transmission. Science. 353 (6304), 1157-1161 (2016).
- Dong, L., Oberai, A. A. Recovery of cellular traction in three-dimensional nonlinear hyperelastic matrices. Computer Methods in Applied Mechanics and Engineering. 314, 296-313 (2017).
- Nier, V., et al. Kalman inversion stress microscopy. Biophysical Journal. 115 (9), 1808-1816 (2018).
- Serrano, R., et al. Three-dimensional Monolayer Stress Microscopy. Biophysical Journal. 117 (1), 111-128 (2019).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ: 25 years of Image Analysis. Nature Methods. 9 (7), 671-675 (2012).
- Schindelin, J., et al. Fiji - an Open Source platform for biological image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Edelstein, A., Amodaj, N., Hoover, K., Vale, R., Stuurman, N. Computer control of microscopes using microManager. Current Protocols in Molecular Biology. , 20 (2010).
- Patel, N. G., et al. Unleashing shear: Role of intercellular traction and cellular moments in collective cell migration. Biochemical and Biophysical Research Communications. 522 (2), 279-285 (2020).
- Yeung, T., et al. Effects of substrate stiffness on cell morphology, cytoskeletal structure, and adhesion. Cell Motility and the Cytoskeleton. 60 (1), 24-34 (2005).
- Trepat, X., et al. Physical forces during collective cell migration. Nature Physics. 5, 426-430 (2009).
- Aratyn-Schaus, Y., Oakes, P. W., Stricker, J., Winter, S. P., Gardel, M. L. Preparation of complaint matrices for quantifying cellular contraction. Journal of Visualized Experiments: JoVE. (46), (2010).
- King, J., et al. Structural and functional characteristics of lung macro- and microvascular endothelial cell phenotypes. Microvascular Research. 67 (2), 139-151 (2004).
- Patel, G., et al. Mechanical signaling in a pulmonary microvascular endothelial cell monolayer. Biochemical and Biophysical Research Communications. 519 (2), 337-343 (2019).
- Kähler, C. J., et al. Main results of the 4th International PIV Challenge. Experiments in Fluids. 57 (6), 97 (2016).
- Tseng, Q., et al. Spatial organization of the extracellular matrix regulates cell-cell junction positioning. Proceedings of the National Academy of Sciences of the United States of America. 109 (5), 1506-1511 (2012).
- Alvarez-Gonzalez, B., et al. Two-layer elastographic 3-D traction force microscopy. Scientific Reports. 7, 39315 (2017).
- Makarchuk, S., Beyer, N., Gaiddon, C., Grange, W., Hebraud, P. Holographic traction force microscopy. Scientific Reports. 8 (1), 3038 (2018).
- Janmey, P. A., Miller, R. T. Mechanisms of mechanical signaling in development and disease. Journal of Cell Science. 124 (1), 9-18 (2011).
- Dembo, M., Oliver, T., Ishihara, A., Jacobson, K. Imaging the traction stresses exerted by locomoting cells with the elastic substratum method. Biophysical Journal. 70 (4), 2008-2022 (1996).
- Dembo, M., Wang, Y. L. Stresses at the cell-to-substrate interface during locomotion of fibroblasts. Biophysical Journal. 76 (4), 2307-2316 (1999).
- Butler, J. P., Tolic-Norrelykke, I. M., Fabry, B., Fredberg, J. J. Traction fields, moments, and strain energy that cells exert on their surroundings. American Journal of Physiology: Cell Physiology. 282 (3), 595-605 (2002).
- Saez, A., et al. Traction forces exerted by epithelial cell sheets. Journal of Physics. Condensed Matter. 22 (19), 194119 (2010).
- Deforet, M., et al. Automated velocity mapping of migrating cell populations (AVeMap). Nature Methods. 9 (11), 1081-1083 (2012).
- Polio, S. R., Smith, M. L. Patterned hydrogels for simplified measurement of cell traction forces. Methods in Cell Biology. 121, 17-31 (2014).
- Toyjanova, J., et al. 3D Viscoelastic traction force microscopy. Soft Matter. 10 (40), 8095-8106 (2014).
- Serra-Picamal, X., Conte, V., Sunyer, R., Munoz, J. J., Trepat, X. Mapping forces and kinematics during collective cell migration. Methods in Cell Biology. 125, 309-330 (2015).
- Charrier, E. E., et al. A novel method to make viscoelastic polyacrylamide gels for cell culture and traction force microscopy. APL Bioengineering. 4 (3), 036104 (2020).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados