Method Article
Интегративный инструментарий для анализа клеточных сигналов: сил, движения, морфологии и флуоресценции
* Эти авторы внесли равный вклад
В этой статье
Резюме
Платформа Integrative Toolkit to Analysis Cellular Signals (iTACS) автоматизирует процесс одновременного измерения широкого спектра химических и механических сигналов в адгезивных ячейках. iTACS предназначен для содействия развитию, ориентированного на сообщество, и позволяет исследователям использовать все функции платформы независимо от их образования.
Аннотация
Количественная оценка клеточных сил и движения значительно продвинулась за последние четыре десятилетия. Эти достижения обеспечили основу для изучения проницательных механических сигнальных процессов в системах клеточных культур. Тем не менее, область в настоящее время сталкивается с тремя проблемами: отсутствие качественной стандартизации полученных данных, технические ошибки в анализе и визуализации данных, и, возможно, самое главное, технология остается в значительной степени недоступной для обычных лабораторий клеточной биологии. Чтобы преодолеть эти ограничения, мы разработали новую экспериментальную платформу - Integrative Toolkit to Analyze Cellular Signals (iTACS). iTACS состоит из двух компонентов: модуля сбора и обучения (AcTrM) и модуля анализа и визуализации (AnViM). AcTrM основан на μManager - программном обеспечении для управления микроскопом на основе NIH-ImageJ - и облегчает самообучение пользователей и автоматизацию общих протоколов получения изображений. AnViM основан на NIH-ImageJ и облегчает удобную автоматизацию анализа данных и проницательную визуализацию результатов. Эти эксперименты включают культивирование адгезивных клеток на гидрогелях, визуализацию фидуциальных маркеров, встроенных в гидрогель, и, наконец, извлечение из этих изображений всесторонней механической характеристики клеток. В настоящее время iTACS позволяет пользователю анализировать и отслеживать широкий спектр свойств, включая морфологию, движение, цитоскелетные силы и флуоресценцию отдельных клеток и их соседней области. Вопрос стандартизации качества был рассмотрен в AcTrM с помощью эталонного метода перефокусировки с визуальным контролем. Технические проблемы в анализе данных были решены в AnViM с помощью многосторонней процедуры сегментации изображений, удобного для пользователя подхода к определению граничных условий и новой визуализации данных на основе клеточных свойств. AcTrM предназначен для облегчения простого преобразования базовых флуоресцентных микроскопов в экспериментальные установки клеточной механики, а AnViM оснащен, чтобы позволить пользователям измерять клеточные механические сигналы, не требуя инженерного опыта. iTACS будет доступен исследовательскому сообществу в виде пакета с открытым исходным кодом с возможностями разработки, управляемыми сообществом.
Введение
Широко используемые оптические инструменты визуализации и анализа данных используют аппаратные и программные технологии, которые почти устарели. Отставание в переводе и внедрении достижений в области электронных устройств, вычислительных подходов и математического анализа в общие экспериментальные инструменты клеточной биологии является основным ограничением темпов роста наших знаний о клеточной физиологии. В настоящее время исследователи клеточной биологии находят инструменты молекулярной биологии в пределах досягаемости, но инструменты, основанные на инженерных принципах, находятся вне досягаемости. Одним из таких инструментов, основанных на инженерных принципах, является монослойная стрессовая микроскопия (МСМ)1,2. В то время как МСМ был адаптирован и изучен в различных лабораториях по всему миру, его использование в основном ограничено лабораториями с инженерным опытом3,4,5,6,7,8,9.
NIH-ImageJ является одним из самых популярных инструментов с открытым исходным кодом среди исследователей клеточной биологии10. Достижения, основанные на вкладе сообщества пользователей, были центральными для его популярности11,12. ImageJ имеет функции, которые позволяют пользователям разрабатывать приложения с сочетанием передового языка программирования и упрощенных подходов к написанию сценариев. Эти функции помогают пользователям с базовыми знаниями в области программирования внедрять, адаптировать и продвигать любой новый вклад в программное обеспечение. Основываясь на этих качествах NIH-ImageJ, мы разработали Интегративный инструментарий для анализа клеточных сигналов (iTACS), который обеспечивает недорогую интеграцию необходимых аппаратных и программных средств для автоматизации измерения широкого спектра химических и механических сигналов в адгезивных ячейках11,12.
iTACS состоит из двух компонентов: модуля сбора и обучения (AcTrM) и модуля анализа и визуализации (AnViM). AcTrM построен на μManager - приложении для сбора изображений на основе NIH-ImageJ - чтобы пользователи могли настраивать покадровые измерения традиционных оптических свойств и различных физических свойств адгезивных ячеек в нескольких образцах12. AcTrM облегчает обучение пользователей с помощью кратких указаний, включенных в графический интерфейс. Кроме того, он имеет новую функцию автофокусировки на основе эталонных изображений, которая предназначена для облегчения измерений физических сил в режиме реального времени и обеспечения качественной стандартизации полученных данных.
AnViM построен на плагинах ImageJ, ускоренном программном обеспечении и скриптах обработки файлов, которые позволяют пользователям количественно оценивать более 50 свойств, включая клеточную форму, размер, ориентацию, скорость и направление движения, тяги, оказываемые на внеклеточный матрикс (ECM), а также на соседние клетки, сократительные и сдвиговые моменты как отдельных адгезивных клеток, так и их соседней области. AnViM облегчает пользователям количественную оценку физических свойств клеток, не осваивая базовый технический фон11. Кроме того, он позволяет анализировать данные в интерактивном или пакетном режиме обработки. Он генерирует тепловые карты, показывающие пространственные вариации, и графики, показывающие временное изменение свойств отдельных клеток.
В типичном эксперименте пользователь культивирует клетки на эластическом гидрогеле с соответствующими белками внеклеточного матрикса на верхней поверхности и двумя типами встроенных флуоресцентных маркеров. По сути, изображения этих флуоресцентных маркеров до и после культивирования клеток достаточны для количественной оценки сил внутри и вокруг отдельных клеток2,13. AnViM сопоставляет эти результаты с отдельными ячейками адгезивного кластера и генерирует проницательные изображения и графики.
протокол
ПРИМЕЧАНИЕ: Образцы, исследованные с использованием платформы iTACS, представляют собой ячейки, прикрепленные к мягкой подложке. Протокол оценки механических и химических сигналов разделен на две последовательные части: Модуль сбора и обучения (AcTrM) и Модуль анализа и визуализации (AnViM).
1. Модуль приобретения и обучения (AcTrM)
ПРИМЕЧАНИЕ: AcTrM автоматизирует процесс сбора данных и самообучения пользователей. Перед любым сбором данных подготовьте мягкий субстрат, способный предоставлять информацию, необходимую для количественной оценки сил, которые клетки оказывают на него.
- Гидрогельный препарат
ПРИМЕЧАНИЕ: Цель состоит в том, чтобы подготовить модуль сдвига 1 250 Па, толщину около 100 мкм и гидрогель полиакриламида (ПАА) диаметром 22 мм.- Готовят раствор полиакриламида, следуя методу Yeung et al., и отливают гидрогели, следуя этапам, описанным Trepat et al.14,15. Одним из исключений из процедуры является то, что шарики, встроенные непосредственно под ячейки, имеют диаметр 0,5 мкм и излучают желтую флуоресценцию.
- Пропустите этап прикрепления бусин размером 2 мкм к покровному стеклу, если ожидается, что область просмотра будет иметь большую бесклеточную область.
ПРИМЕЧАНИЕ: Протокол получения гидрогеля в настоящее время достаточно установлен в этой области16. На протяжении всего последующего описания бусины размером 0,5 мкм называются «верхними бусинами», а бусины 2 мкм называются «нижними бусинами». Однако нижние бусины являются необязательными, если изображенная область содержит большую бесклеточную область. Рисунок верхней бусины из такой бесклеточной области будет служить цели нижнего узора бусины. - Установите гидрогель со встроенными флуоресцентными шариками на ступень микроскопа.
- Подождите 15-20 минут, чтобы температура пластины достигла устойчивого состояния.
ПРИМЕЧАНИЕ: Верхняя поверхность пластины функционализирована белками внеклеточного матрикса, но клетки еще не засеяны гидрогелем.
- Получение эталонных изображений
- Часть 1: Создание списка вакансий
ПРИМЕЧАНИЕ: Ручной ввод на этом шаге включает в себя следование подсказкам AcTrM для выбора мест с наилучшим распределением бусин. На этом шаге не используются ранее созданные файлы. Основные новые папки, созданные на этом шаге, включают папки 't0imgs', 'tnimgs' и 'textfiles', а созданный файл включает файл списка позиций. На рисунке 2 приведено визуальное описание следующих шагов, которые направляют создание списка позиций с помощью AcTrM. Показанный пример получает данные с 20-кратным увеличением.- Запуск μManager 2.0 - Бета.
- Запустите AcTrM, нажав кнопку AcTrM.bsh в окне Ресурсы IMBL .
- В окне Определение свойств захвата выберите соответствующую цель, допуск на точность восстановления бокового положения (т.е. XY), размер шага грубой и уточненной операции перефокусировки (т.е. z) и значение приемлемого коэффициента корреляции изображения для восстановления фокуса.
ПРИМЕЧАНИЕ: Более тонкий допуск для восстановления бокового положения замедлит восстановление позиции, но существенный допуск потребует коррекции положения во время анализа данных и может вызвать трудности с восстановлением фокуса. Меньший размер шага замедлит операцию перефокусировки, но очень высокий размер шага может привести к быстрому движению с возможностью пропустить фокус. - В окне AcTrM - Шаг 0 выберите Новое приобретение , чтобы создать список позиций.
ПРИМЕЧАНИЕ: В окне под названием AcTrM - Step 1 перечислены все ключевые последующие шаги. Эти шаги включают перемещение сцены, настройку фокуса и нажатие кнопок в μManager. Эти шаги служат руководством при построении списка позиций, подходящих для сбора данных. - Нажмите Live in Micromanager , чтобы визуализировать образцы для ручной настройки, перечисленные в окне AcTrM - Шаг 1 .
- Выполните действия, перечисленные в этом окне.
- Теперь приобретите изображение бисера. Выберите подходящий канал для верхних бусин.
- Обязательно посмотрите на нижние бусины. Для этого выберите каналы для нижних бусин. Видно размытое изображение нижних бусин.
ПРИМЕЧАНИЕ: Каналы можно переключать из предустановленного выпадающего меню в главном окне μManager. Если в эксперименте используются нижние бусины, убедитесь, что они присутствуют в выбранном положении. Эти бусины будут выглядеть размытыми. - Выбрав подходящую позицию, нажмите на Mark в окне Список позиций stage , чтобы сохранить позицию.
ПРИМЕЧАНИЕ: Наилучшее положение определяется плотным и равномерным распределением верхних бусин, встроенных непосредственно под верхнюю поверхность (т.е. верхних бусин) и, по крайней мере, двух бусин, прикрепленных к покровному стеклу (т.е. нижним бусинам) (дополнительный рисунок S1). Восстановление фокусировки происходит быстрее, если нижние бусины выглядят как большие, расфокусированные кольца. Однако, если эксперименты проводятся в одном положении без необходимости фокусировки или восстановления бокового положения, игнорируйте инструкции, связанные с изображением нижних бусин. - Выполните шаги 6-9, описанные на рисунке 2 , чтобы включить дополнительные позиции в список.
ПРИМЕЧАНИЕ: Выберите несколько дополнительных позиций, чтобы раунд исключения мог позволить выбрать лучшие позиции на тарелке.
- Часть 2: Получение справочных изображений
ПРИМЕЧАНИЕ: Этот шаг не включает в себя ручной ввод. Ранее созданные ключевые файлы, используемые на этом шаге, включают 'textfiles/*.pos'. Ключевые новые файлы и папки, созданные на этом шаге, включают файлы и папки в папках 't0imgs' и 'tnimgs'. См. рисунок 3 и дополнительный рисунок S2 для визуального описания следующих шагов, которые направляют получение эталонных изображений с помощью AcTrM. В окне AcTrM - Шаг 2 также перечислены все ключевые шаги.- Выполнив действия, перечисленные в окне AcTrM - Шаг 2 , сделайте выбор в окне Многомерное приобретение . Например, чтобы выполнить длительный интервал, укажите количество изображений, которые необходимо сделать, выберите первый канал в качестве фазового канала, а затем выберите следующий для верхних бусин, а следующий после этого - для нижних бусин. Нажмите кнопку Закрыть в окне Многомерное получение , а затем нажмите кнопку ОК в окне ActrM - Шаг 2 .
- В окне AcTrM - Шаг 3 выберите, чтобы включить восстановление позиции XYZ на основе эталонного изображения. Выбор, как правило, да. Однако, если изображения получены в одном положении без места для фокусировки или дрейфа ступени, выбор в окне AcTrM - Шаг 3 будет отрицательным.
- В окне Параметры восстановления AcTrM - XYZ задайте канал для грубого восстановления XYZ, следует ли выполнять уточненное восстановление XYZ, регион и канал для уточненного восстановления XYZ и направление начала перефокусировки (дополнительный рисунок S4). По завершении AcTrM получит эталонные образы.
ПРИМЕЧАНИЕ: Типичным выбором для канала будет нижний канал бусин. Восстановление XYZ работает лучше всего, когда выполняется с полным изображением в текущей реализации. Эталонные изображения обычно включают в себя прошедшее световое изображение гидрогеля, флуоресцентное изображение верхних бусин и флуоресцентное изображение нижних бусин. Содержимое каждого набора изображений может отличаться в зависимости от выбора, сделанного в окне Многомерное получение . Тем не менее, программное обеспечение будет получать эталонное изображение, установленное в каждой позиции, определенной на рисунке 2. В конце этого шага в выбранном каталоге создаются три папки: 't0imgs', 'tnimgs' и 'textfiles'. Папка 't0imgs' содержит эталонные изображения в формате, распознаваемом μManager и используемом на последующих этапах; Папка 'tnimgs' содержит отдельные TIFF изображения с именами файлов 'c0_p0_t0.tif', 'c1_p0_t0.tif' и т.д. Здесь «c» означает канал, «p» означает позицию, а «t» означает время. За этими буквами следуют номер канала, номер позиции и номер кадра соответственно. Папка 'textfiles' содержит список позиций в формате XML и пользовательский выбор для получения изображений и восстановления позиции.
- Часть 1: Создание списка вакансий
- Посев и рост клеток
ПРИМЕЧАНИЕ: Платформа iTACS обладает гибкостью для размещения протоколов подготовки образцов, используемых в общей оценке in vitro механического поведения адгезивных клеток, включая разреженные ячейки, полностью сливающийся монослой, анализ формирования сетей и монослои с отверстиями или значительными зазорами.- Культивирование легочных микрососудистых эндотелиальных клеток крысы до слияния в колбу17,18.
- Отсоедините клетки с помощью трипсина. Повторно суспендировать клетки в культуральной среде, содержащей 10% фетальной бычьей сыворотки до концентрации 1 х 106 клеток/мл.
- Поместите каплю 5 мкл ресуспендированных клеток на частично сухую поверхность гидрогеля и поместите ее в инкубатор клеточной культуры.
ПРИМЕЧАНИЕ: Через 2 дня в культуральной среде, содержащей 10% фетальной бычьей сыворотки, клетки в этой капле образуют переполненный остров клеток1.
- Автоматическое получение изображений для оставшегося эксперимента
ПРИМЕЧАНИЕ: Ручной ввод для этого шага включает в себя следующие запросы AcTrM для возобновления получения изображения. Ключевые ранее созданные файлы, используемые на этом шаге, включают 'textfiles/*.pos' и файлы изображений нижней бусины в папке 't0imgs'. Ключевые новые файлы, созданные на этом шаге, включают обновленные файлы списка позиций и изображения 'tnimgs/*_t*.tif'.- Убедитесь, что система контроля окружающей среды микроскопа достигает стабильных условий культивирования тканей.
- Аккуратно установите пластину, содержащую культивируемые клетки, на ступень микроскопа.
- Дайте 15-20 мин, чтобы температура и влажность стабилизировались.
ПРИМЕЧАНИЕ: Смотрите рисунок 4 для визуального описания следующих шагов, которые направляют получение изображений для оставшегося эксперимента с использованием AcTrM. - Запуск μManager 2.0 - Бета.
- Запустите AcTrM, нажав кнопку AcTrM.bsh в окне Ресурсы IMBL .
ПРИМЕЧАНИЕ: Игнорируйте выбор, сделанный в окне Определение свойств приобретения, но используйте выбор, сделанный на предыдущем шаге. - В окне AcTrM - Шаг 0 выберите Продолжить приостановленное получение.
- Выберите увеличение и каталог, указанные в окне Многомерное получение на предыдущем шаге, где были сохранены папки данных ('t0imags', 'tnimgs' и 'textfiles').
ПРИМЕЧАНИЕ: Заголовок окна направляет пользователя на выбор соответствующего каталога. - В окне Изменение положения пластины выберите варианты перемещения пластины (три неколлинеарных положения) вместе с каналом, используемым для перемещения.
ПРИМЕЧАНИЕ: Сохраненные изображения будут видны в камере. Если перекрывающиеся изображения отображаются в виде изображения красного, зеленого и черного цветов, выполните ручную настройку. - Нажмите « Принять », чтобы продолжить приобретение.
ПРИМЕЧАНИЕ: Этот шаг преодолевает незначительные сдвиги от невоспроизводимости выравнивания пластин при повторном монтаже пластины на ступень микроскопа. Следуйте подсказкам AcTrM, чтобы ускорить выравнивание в каждой из трех позиций, показывая составное изображение эталонного изображения, показанного красным цветом (обычно нижних бусин), и изображения, которое в настоящее время наблюдается через цель, показанную зеленым цветом. Получение зеленого достаточно близкого к красному оставляет меньше работы для программного обеспечения. Когда перекрытие идеальное, составное изображение выглядит желтым и черным. Достаточно близко обычно, когда одни и те же нижние бусины видны как на красных, так и на зеленых изображениях, а соответствующие красные и зеленые кольца вне фокуса касаются друг друга. После завершения перемещения пластины AcTrM переносит сцену в каждое выбранное положение и восстанавливает положение XYZ, сопоставляя эталонное изображение с тем, что в настоящее время видно через камеру. Первая боковая позиция (XY) сопоставляется, а затем фокус (Z) сопоставляется. Грубое восстановление сопровождается уточненным восстановлением положения и фокуса. Обновления текущего состояния эксперимента отображаются на экране через четырехкомпонентное окно, показанное на дополнительном рисунке S3. Полученные изображения сохраняются в папке 'tnimgs' после '*_t1.tif', '*_t2.tif', указывая время для набора изображений. Если восстановление XYZ определяет обновленную позицию, новый список позиций генерируется и сохраняется в папке 'textfiles'.
2. Модуль анализа и визуализации (AnViM)
- Настройка автоматизированного анализа данных
ПРИМЕЧАНИЕ: Ручной ввод на этом шаге включает в себя определение местоположения папки 'tnimgs'. Ключевые ранее созданные файлы, используемые на этом шаге, включают 'tnimgs/*.tif'. Ключевые новые файлы и папки, созданные на этом шаге, включают папку 'analysis' в той же родительской папке, что и 'tnimgs' и папки для каждой позиции 'analysis/p*'. В каждом 'analysis/p*' этот шаг создает новые файлы изображений '*.tif' в папках 'phs' и 'tny'. Эти изображения представляют собой обрезанные и де-дрейфующие изображения ячеек и верхних бусин. Другие созданные файлы включают 'analysis/analysisChoices.txt', в котором перечислены варианты анализа, 'analysis/p*/skipAnalysis.txt', в котором перечислены случаи, когда анализ пропускается из-за значительного ранее существовавшего дрейфа, и 'analysis/p*/part_shift_values_pixel_degree.dat', в котором перечислены оценочные значения дрейфа. AnViM не изменяет необработанные данные в папке 'tnimgs'. На рисунке 5 приведено визуальное описание следующих шагов, которые направляют настройку автоматизированного анализа данных с помощью AnViM.- Запустите программное обеспечение Fiji и выберите первый вариант в выпадающем меню MSM с пометкой MSM - предварительная обработка.
- В диалоговом окне файлового браузера выберите папку, содержащую папку 'tnimgs', в которой содержатся проанализированные данные.
- Определите канал для изображений. Следуя рекомендациям, приведенным на дополнительном рисунке S5, определите номера каналов проходящего светового изображения ячеек (фазово контрастное изображение ячеек), изображения нижних бусин и изображения верхних бусин. Обычно устанавливаются следующие три флажка («Переместить файлы в папку положения», «Выполнить дополнительную коррекцию для жесткого движения» и «Обрезать данные и сохранить в папке положения»). Наконец, определите допуск на отклонение сильно дрейфующих данных, размер пикселя и стороны изображения, где пересекаются ячейки.
- Реагируйте на подсказку для повышения яркости и контрастности изображений нижней бусины, чтобы бусины выглядели заметно. Отрегулируйте это с помощью ползунка в меню и нажмите OK.
- Теперь выполните коррекцию положения, чтобы избавиться от сдвигов, если таковые имеются. После этого создается папка анализа.
ПРИМЕЧАНИЕ: Повышение контрастности, как правило, не требуется, но это положение доступно для экспериментов, где воздействие лазеров должно быть сведено к минимуму. После этого anViM копирует файлы из папки 'tnimgs' в папку 'analysis'. Файлы, соответствующие каждой позиции, сохраняются в папках 'analysis/p0, analysis/p1' и т.д. В каждой из этих позиционных папок AnViM создает папки 'cels', 'defs' и 'refs', содержащие исходное изображение пропускаемого света, изображения верхних бусин и изображения нижних бусин соответственно (дополнительный рисунок S6). Затем AnViM анализирует жесткое движение в изображениях нижних бусин и создает папку «phs», содержащую исправленное прошедшее световое изображение ячеек, и папку «tny», содержащую обновленные изображения верхних бусин. Наконец, пользовательский выбор каналов, операций, допусков, размера пикселя и пересечения границ сохраняется в файле 'analysisChoices.txt' (дополнительный рисунок S6).
- Количественная оценка деформации гидрогеля и монослоя
ПРИМЕЧАНИЕ: Важно отметить, что количественная оценка движения из последовательности изображений является быстро развивающейся областью19. Технология постоянно оптимизируется для функций, включая скорость, точность, конкретные особенности в необработанных изображениях и конкретные деформационные паттерны. Следовательно, вполне вероятно, что некоторые пользователи могут использовать другой подход к количественной оценке деформации, чем тот, который представлен здесь.-
Часть 1: Количественная оценка деформации гидрогеля
ПРИМЕЧАНИЕ: Ручной ввод на этом этапе включает идентификацию каталога данных и выбор разрешения сетки. Ключевые ранее созданные файлы, используемые на этом шаге, включают 'p*/tny/*.tif'. Ключевые новые файлы, созданные в этом, включают 'p*/смещение/*_disp.dat", в котором перечислены векторы смещения верхней поверхности гидрогеля. На рисунке 6 приведено визуальное описание следующих шагов по вовлечению с помощью AnViM анализа велоциметрии изображения частиц на верхних изображениях бусин20.- Запустите программное обеспечение Fiji и в раскрывающемся меню MSM выберите MSM - Gel Deformation.
- Здесь выберите вариант, подходящий для эксперимента.
- Используя диалоговое окно файлового браузера, выберите родительский каталог папки 'analysis', содержащей анализируемые данные.
- В окне Параметры для расчета деформации геля выберите соответствующий размер окна перекрестной корреляции, уровень шума и пороговое значение (дополнительный рисунок S7)20.
ПРИМЕЧАНИЕ: Результаты сохраняются во вновь созданном каталоге 'displacement' в каждой папке позиций 'analysis/p0, analysis/p1' и т.д. Здесь хранятся все выходные файлы.
-
Часть 2: Количественная оценка монослойной деформации
ПРИМЕЧАНИЕ: Ручной ввод на этом шаге включает идентификацию каталога данных и выбор разрешения сетки. Ключевые ранее созданные файлы, используемые на этом шаге, включают 'p*/tny/*.tif'. Ключевые новые файлы, созданные в этом, включают 'p*/velocity/*_vel.dat', в котором перечислены векторы движения ячеек. На рисунке 7 приведено визуальное описание шагов по вовлечению с помощью AnViM анализа велоциметрии изображения частиц на верхних изображениях бусин20. Процедура аналогична той, которая используется для количественной оценки деформации гидрогеля, и выбирает MSM - Cellular Motion из раскрывающегося меню MSM . Результаты сохраняются во вновь созданном каталоге 'velocity' в каждой папке позиций 'analysis/p0', 'analysis/p1' и т.д.
-
Часть 1: Количественная оценка деформации гидрогеля
- Количественная оценка клеточных ECM и клеточно-клеточных сил
ПРИМЕЧАНИЕ: Ручной ввод на этом этапе включает идентификацию каталога данных, указание жесткости гидрогеля и реакцию на подсказки сегментации клеточного кластера для обнаружения клеток из бесклеточной области. Ключевые ранее созданные файлы, используемые на этом шаге, включают 'p*/displacement/*_disp.dat'. Ключевые новые файлы, полученные на этом этапе, включают: 'p*/traction/*_trac.dat', в котором перечислены силы, оказываемые клетками на гидрогель; 'p*/traction/*_domain.dat', в котором указано расположение точек сетки, содержащих ячейки; и входные файлы 'p*/traction.in, p*/clusterInput.txt' и 'p*/prestress.in', которые записывают выбор пользователя для этого шага. После первой количественной оценки клеточно-ECM и клеточно-клеточных сил было разработано несколько вариаций метода1,15. Вариации сосредоточены на конкретных случаях субстратов, клеток, экспериментальных условий или численных инструментов7,8,21,22. См. рисунок 8 для визуального описания для взаимодействия с помощью AnViM, тяговой микроскопии с преобразованием Фурье и монослойной стрессовой микроскопии на данных деформации гидрогеля1,2,15.- Запустите программное обеспечение Fiji и в раскрывающемся меню MSM выберите MSM - Cell-ECM и Cell-Cell Forces (третий вариант в раскрывающемся меню).
- Используя диалоговое окно файлового браузера, выберите родительский каталог папки 'analysis', содержащий папки 'tnimgs' и 'analysis', содержащие анализируемые данные. Нажмите выбрать.
- В окне Параметры расчета клеточных сил введите модуль сдвига, толщину гидрогеля и ожидаемый уровень шума. Нажмите кнопку ОК. Это позволяет ему выполнять тягу через функцию MATLAB.
ПРИМЕЧАНИЕ: Следуя этим входам, AnViM вычисляет силы CELL-ECM. - После этого укажите, является ли монослой сливающимся. Если все изображение ячейки покрыто ячейками, то ответ — Да. В этом случае силы ячейки-ячейки будут вычислены по всему кадру. С другой стороны, если часть изображения не имеет клеток, то ответ отрицательный. В этом случае следуйте подсказкам AnViM, чтобы облегчить сегментацию области изображения, содержащей ячейки.
- Если ответ отрицательный, вручную нарисуйте многоугольник вокруг неячеистого объекта, когда программное обеспечение предложит сделать это, а затем выберите соответствующий метод (методы) для сегментации. Программное обеспечение запросит цвет (черный или белый) ячеек.
- Проверьте опцию Заполнять пятна автоматически в программном обеспечении и нажмите OK.
ПРИМЕЧАНИЕ: Этапы сегментации несмесящего монослоя будут рассмотрены в первой части раздела 2.4. Силы Cell-ECM хранятся в файлах '*_trac.dat' во вновь созданном каталоге 'traction' в каждой папке позиции, а входные данные в программное обеспечение для расчета силы cell-ECM сохраняются в файле 'traction.in' (дополнительный рисунок S8). Силы ячейки-ячейки хранятся в файлах '*_prestress.dat' во вновь созданном каталоге 'prestress' в каждой папке позиции, а входные данные в программное обеспечение для расчета силы ячейки-ячейки сохраняются в файле 'prestress.in' (Дополнительный рисунок S9).
- Сопоставление значений точек сетки на отдельных ячейках
ПРИМЕЧАНИЕ: Одним из текущих направлений деятельности iTACS является внедрение простого подхода к интерпретации измеренных механических сигналов в полевых условиях. Такой подход полезен для изучения механических взаимодействий между соседними клетками кластера.18. В целом, свойства, количественно оцененные до сих пор, находятся на регулярно расположенных точках сетки по всему сотовому кластеру. Эти данные идентифицируют медианное, среднее и стандартное отклонение выбранных свойств в пределах морфологической границы отдельных клеток и присваивают их в качестве клеточных физических свойств / сигналов. Из них стандартное отклонение используется для указания изменчивости, разница между средним и медианой используется для указания характера распределения, а медианное значение используется для указания общего состояния ячеек для выбранных свойств.- Часть 1: Сегментация области изображения, содержащей ячейки
ПРИМЕЧАНИЕ: Ручной ввод на этом шаге включает идентификацию каталога данных и ответ на запросы сегментации для обнаружения ячеек из области без ячеек. Ключевые ранее созданные файлы, используемые на этом шаге, включают 'p*/phs/*.tif'. Ключевые новые файлы, созданные на этом шаге, включают 'p*/phs/bwImages/*.tif', которые представляют собой двоичные изображения, отделяющие области ячеек от бесклеточных областей, 'p*/phs/textResults/frameNum_percentHealed.txt', в котором указана процентная площадь изображения, охватываемая ячейками в каждом экземпляре, и 'p*/clusterInput.txt', который записывает пользовательские выборы, введенные в интерфейсе AcTrM. На рисунке 9 приведено визуальное описание следующих шагов по сегментации области изображения, содержащей ячейки. Эта часть должна быть завершена перед вычислением сил ячейки-клетки. В этом случае эти шаги не нужно повторять. Кроме того, если эти шаги проводятся до расчета силы ячейки-ячейки, они не запрашиваются в начале расчетов силы.- Запустите программное обеспечение Fiji и в раскрывающемся меню MSM выберите Сегментация - сотовый кластер.
- Используя диалоговое окно файлового браузера, выберите каталог позиций 'analysis/p0', 'analysis/p1' и т.д., который содержит свойства, количественно оцениваемые в регулярно расположенных точках сетки.
- Укажите, является ли монослой сливающимся.
ПРИМЕЧАНИЕ: Приведенные ниже шаги вызываются только тогда, когда монослой не сливается. Следуйте подсказкам AnViM, чтобы применить новый многосторонний подход к идентификации областей изображения, содержащих ячейки. Процесс включает в себя четыре метода, каждый из которых подходит к сегментации по-разному. Один или несколько из этих методов, используемых в комбинации, охватывают широкий спектр изображений клеток. Поэтому попробуйте разные подходы, чтобы найти, какая комбинация лучше всего подходит для их данных. - Отрегулируйте «Коэффициент подключения домена» и «Фактор размазывания» для получения оптимальной сегментации.
ПРИМЕЧАНИЕ: «Фактор мазка» делает области, содержащие клетки, больше. - Укажите, отображаются ли ячейки черными или белыми на двоичном изображении.
- В окне Инструкции по очистке пограничного изображения выберите, следует ли очищать изображение автоматически или вручную.
ПРИМЕЧАНИЕ: Нежелательными особенностями изображений являются черные пятна на пикселях, занятых ячейками, и белые пятна на пикселях, отсоединенных от кластера ячеек, подлежащих анализу. Анализ может быть сделан только на регионах, которые подключены. Таким образом, несколько разрозненных областей должны анализироваться отдельно.
- Часть 2: Сегментация отдельных клеток на изображениях
ПРИМЕЧАНИЕ: Ручной ввод на этом шаге включает идентификацию каталога данных и ответ на запросы сегментации для обнаружения отдельных ячеек в изображении. Ключевые ранее созданные файлы, используемые на этом шаге, включают 'p*/phs/*.tif' и 'p*/phs/bwImages/*.tif'. Основные новые созданные файлы включают 'p*/phs/textResults/*.csv', которые содержат клеточные морфологические свойства. Смотрите рисунок 10 для визуального описания следующих шагов для сегментации отдельных ячеек монослоя.- Запустите программное обеспечение Fiji и в раскрывающемся меню MSM выберите Сегментация - Отдельные ячейки.
- Используя диалоговое окно файлового браузера, выберите каталог позиций 'analysis/p0', 'analysis/p1' и т.д., который содержит свойства, количественно оцениваемые в регулярно расположенных точках сетки.
- В окне Выбор входных параметров укажите максимальное соотношение сторон, заметность того, насколько центр ячейки выделяется по сравнению с границей ячейки, и два параметра размытия для определения ячеек (дополнительный рисунок S10).
- В стеке изображений для каждой позиции нарисуйте многоугольник на наименьшей нормальной ячейке. Это используется для расчета площади. Все, что меньше этого, не будет рассматриваться программным обеспечением как клетка.
- Затем нарисуйте многоугольник вокруг самой большой нормальной ячейки и укажите, ярче ли центр ячейки или интерфейс ячейка-ячейка.
ПРИМЕЧАНИЕ: Затем AnViM создает файлы 'phs/textResults/0001.csv', 'phs/textResults/0002.csv' и т.д., для каждого кадра, содержащего информацию о ячейках в этом фрейме. На этом этапе этот файл включает морфологическую информацию о клетках, включая площадь, центроид, периметр, ориентацию, окружность, соотношение сторон, округлость, твердость, расстояние от неклеточной области. Единицей длины в этих свойствах являются пиксели, а угол — в градусах. Наконец, этот файл обновляется, чтобы содержать интенсивность сотовых пикселей, движение и силы на последующих шагах.
- Часть 3: Отображение интенсивности пикселей в ячейках
ПРИМЕЧАНИЕ: Ручной ввод на этом шаге включает идентификацию каталога данных и выбор свойств и параметров сопоставления. Ключевые ранее созданные файлы, используемые на этом шаге, включают 'p*/phs/*.tif' (или 'p*/fluo/*.tif ') и 'p*/phs/bwImages/*.tif'. Ключевые новые файлы, созданные на этом шаге, включают 'p*/phs/textResults/*.csv', в котором перечислены клеточные морфологические свойства. Смотрите рисунок 11 для визуального описания следующих шагов для оценки интенсивности пикселей в области, охватываемой отдельными ячейками и их соседней областью, и определения их как свойств ячеек.- Запустите программное обеспечение Fiji и в раскрывающемся меню MSM выберите Map on Cells - Intensities.
- Используя диалоговое окно файлового браузера, выберите каталог позиций 'analysis/p0', 'analysis/p1' и т.д., который содержит свойства, количественно оцениваемые в регулярно расположенных точках сетки.
- Выберите «Обнаружить деление клеток» или «Количественно оценить клеточную флуоресценцию» при запросе программного обеспечения. Определите размер соседнего региона. Установите флажки Собирать свойства соседней области и Собирать свойства ячеек. Нажмите OK.
- В окне «Цель записи интенсивности сотовой связи» укажите, какой тип изображения следует использовать для отображения интенсивности, размер соседней области и собираются ли данные для отдельных ячеек или для обеих ячеек и их соседних областей (дополнительный рисунок S11).
ПРИМЕЧАНИЕ: Отображение интенсивности пикселей фазово-контрастного изображения позволяет обнаруживать события деления клеток. Картирование интенсивности флуоресцентного изображения позволит обнаруживать флуктуации в флуоресцентно помеченных цитоплазматических молекулах. Результатом вышеуказанных шагов являются средние, медианные, стандартные отклонения, минимальные и максимальные значения интенсивности пикселей для отдельных ячеек. Эти номера вводятся в виде новых столбцов в файлы 'phs/textResults/0001.csv', 'phs/textResults/0002.csv' и т.д.
- Часть 4: Отображение сил и движения на клетках
ПРИМЕЧАНИЕ: Ручной ввод на этом шаге включает идентификацию каталога данных и выбор свойств и параметров сопоставления. Ключевые ранее созданные файлы, используемые на этом шаге, включают 'p*/phs/textResults/*.csv', 'p*/velocity/*_vel.dat', 'p*/displacement/*_disp.dat', 'p*/traction/*_trac.dat', 'p*/prestress/*_prestress.dat' и 'p*/phs/bwImages/*.tif '. На этом шаге новые файлы не создаются. Вместо этого новая информация (свойства клеточной силы и движения) добавляется в файлы 'p*/phs/textResults/*.csv '. Смотрите рисунок 12 для визуального описания шагов по оценке сил и движения в области, охватываемой отдельными клетками и их соседней областью, и определению их как свойств клеток.- Выберите из раскрывающегося меню MSM Map on Cells - Motion/Force Data (Дополнительный рисунок S12).
- Затем выполните действия, перечисленные в шаге 2.4.3.
ПРИМЕЧАНИЕ: Новые столбцы добавляются в файлы 'phs/textResults/0001.csv', 'phs/textResults/0002.csv' и т.д. и включают среднее, медианное и стандартное отклонение скорости, ориентацию скорости, среднее цитоскелетное напряжение, анизотропию напряжения, энергию деформации в гидрогеле, ориентацию наибольшего напряжения и величину тяги CELL-ECM (дополнительный рисунок S13).
- Часть 1: Сегментация области изображения, содержащей ячейки
- Визуализация результатов
- Часть 1: Отслеживание клеточных идентичностей в эксперименте
ПРИМЕЧАНИЕ: Ручной ввод на этом шаге включает идентификацию каталога данных и выбор сопоставленных свойств для визуализации. Ключевые ранее созданные файлы, используемые на этом шаге, включают 'p*/phs/textResults/*.csv'. Ключевые новые файлы, созданные на этом этапе, включают 'p*/phs/textResults/cellTimeTrace_*.csv', который содержит временную трассировку сотовых данных. На рисунке 13 приведено визуальное описание следующих шагов по отслеживанию свойств отдельных ячеек на протяжении всего эксперимента.- Запустите программное обеспечение Fiji и в раскрывающемся меню MSM выберите Результаты - Отслеживание данных.
- Используя диалоговое окно файлового браузера, выберите каталог позиций 'analysis/p0', 'analysis/p1' и т.д., содержащий клеточные свойства для отслеживания. Когда все будет готово, нажмите Выбрать.
- В окне Выбор начальной точки для отслеживания выберите начальный кадр для мониторинга и количество одновременно отслеживаемых кадров (дополнительный рисунок S14). Всегда начинайте с кадра No 2, так как скорость не может быть определена для кадра No 1. Когда все будет готово, нажмите OK.
- В окне Проверка переменных для создания треков выберите переменные, введя имена переменных или выбрав термины из флажков (дополнительный рисунок S15). Затем нажмите OK.
ПРИМЕЧАНИЕ: При отслеживании генерируются файлы 'phs/textResults/cellTimeTrace_*.csv", каждый столбец которых содержит уникальный номер ячейки, а каждая последующая строка представляет последовательный экземпляр времени.
- Часть 2: Генерация временных треков оцениваемых клеточных свойств
ПРИМЕЧАНИЕ: Ручной ввод на этом шаге включает идентификацию каталога данных и выбор сопоставленных свойств для визуализации. Ключевые ранее созданные файлы, используемые на этом шаге, включают 'p*/phs/textResults/cellTimeTrace_*.csv'. Ключевые новые файлы, созданные на этом этапе, включают 'p*/phs/trackedDataPlots/*.tif', которые содержат графики тайм-трека и 'p*/phs/trackedDataPlots/*.csv', которые содержат данные графика. На рисунке 14 приведено визуальное описание следующих шагов для создания временных треков оцениваемых клеточных свойств.- Запустите программное обеспечение Fiji и в раскрывающемся меню MSM выберите Результаты - график.
- Используя диалоговое окно файлового браузера, выберите каталог позиций 'analysis/p0', 'analysis/p1' и т.д., содержащий отслеживаемые клеточные свойства для построения.
- Выберите, следует ли ограничить построение ячейками с неповрежденными дорожками, нажав кнопку «Да » или « Нет ».
ПРИМЕЧАНИЕ: Этот параметр игнорирует ячейки, которые не могут быть обнаружены в одном или нескольких временных экземплярах. - Выберите количество переменных, которые будут построены одновременно, и нажмите OK.
ПРИМЕЧАНИЕ: В настоящее время AnViM позволяет отображать максимум три переменные на одном графике. - Выберите отдельные переменные для построения с помощью раскрывающегося меню.
ПРИМЕЧАНИЕ: Эти шаги создают файл формата изображения с тегами (TIFF) и файл данных с разделителями-запятыми в папке 'phs/trackedDataPlots/'. Имя файла состоит из имен переменных, разделенных символом подчеркивания и заканчивающихся номером ячейки.
- Часть 3: Генерация тепловых карт оцениваемых клеточных свойств
ПРИМЕЧАНИЕ: Ручной ввод на этом шаге включает идентификацию каталога данных и выбор сопоставленных свойств для визуализации. Ключевые ранее созданные файлы, используемые на этом шаге, включают 'p*/phs/textResults/cellTimeTrace_*.csv'. Ключевые новые файлы, сгенерированные на этом шаге, включают 'p*/phs/segmentedImages/cellPropMaps/*.tif', которые являются тепловыми картами свойств сотовой связи. Смотрите рисунок 15 для визуального описания шагов по созданию тепловых карт оцениваемых клеточных свойств.- Выберите из раскрывающегося меню MSM Результаты - Рисунок.
- Выполните действия, описанные в шаге 2.5.2.
ПРИМЕЧАНИЕ: Выходные данные хранятся в виде файлов TIFF в папке 'phs/segmentedImages/cellPropMaps/'. Имена файлов — это номер экземпляра времени и имя переменной, разделенные символом подчеркивания.
- Часть 1: Отслеживание клеточных идентичностей в эксперименте
Результаты
Мы представляем здесь два ключевых выхода для показанного примера. Первым выходом является временной след клеточной скорости и цитоскелетного напряжения для клетки No 1 (рисунок 16). Свойства отображаются на общей вертикальной оси, чтобы облегчить визуальную ассоциацию между свойствами, а горизонтальная ось указывает номер экземпляра времени. В этом эксперименте последовательные кадры были получены с интервалом в 15 минут. Вторым выходом является массив тепловых карт через 1 ч в эксперименте (рисунок 17). Свойства, показанные здесь, включают область распространения, ориентацию, цикличность, скорость, направление движения, максимальную ориентацию напряжения, цитоскелетное напряжение, стяжение субстрата и анизотропию напряжения отдельных клеток.

Рисунок 1: Структура интегративного инструментария для анализа клеточных сигналов (iTACS). Двумя ключевыми компонентами iTACS являются модуль приобретения и обучения (AcTrM) и модуль анализа и визуализации (AnViM). AcTrM может использовать различные методы приготовления гидрогеля, которые в настоящее время существуют для приготовления гидрогелей, которые можно прочно удерживать на стадии микроскопа, любого посева клеток и протокола роста, который удерживает клетки в одной фокальной плоскости. AnViM может использовать различные методы для количественной оценки гидрогелевой и монослойной деформации, сил КЛЕТОЧНОЙ ЭВМ и клеточно-клеточной силы. Все эти предпочтительные пользователем компоненты протокола измерения силы могут быть размещены в iTACS, и они были идентифицированы пунктирными полями. Компоненты, идентифицированные с твердыми коробками, являются новым вкладом в технологию измерения клеточной силы. Визуализация в AnViM фокусируется на медианном значении и изменчивости свойств в отдельных ячейках. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
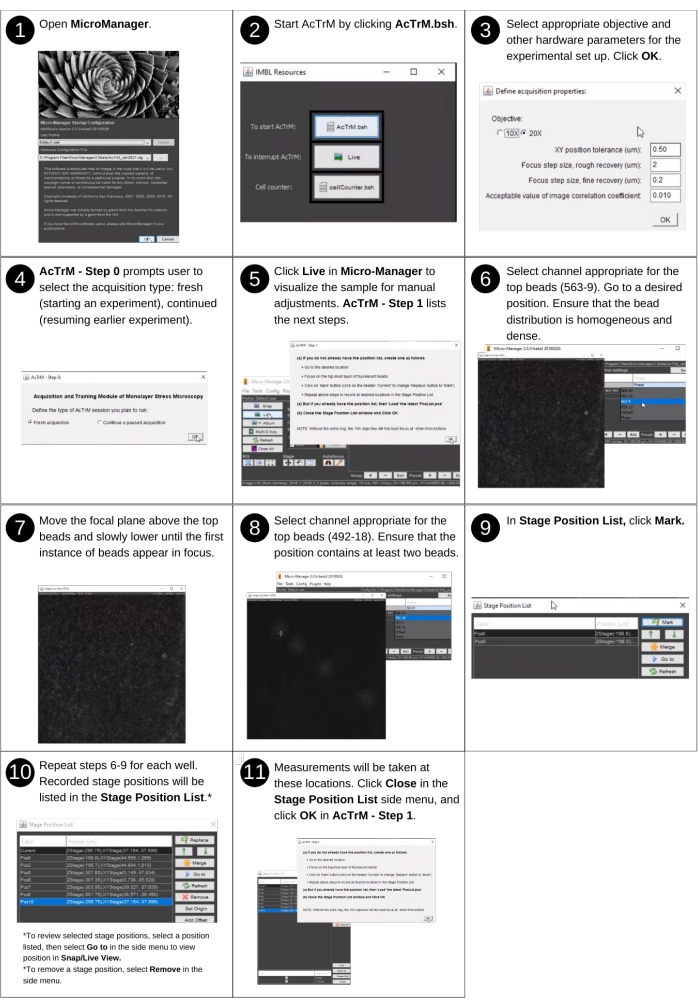
Рисунок 2: Получение эталонного изображения - часть 1. Шаги по созданию списка позиций с помощью AcTrM. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
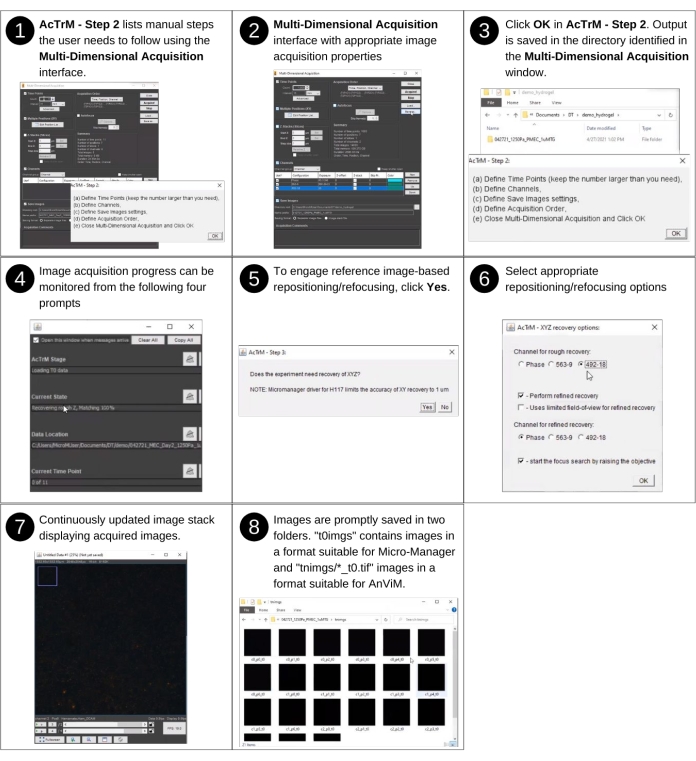
Рисунок 3: Получение эталонного изображения - часть 2. Шаги по получению справочных изображений с помощью AcTrM. Подробные представления шагов 2, 4 и 6 представлены на дополнительных рисунках S2, S3 и S4 соответственно. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
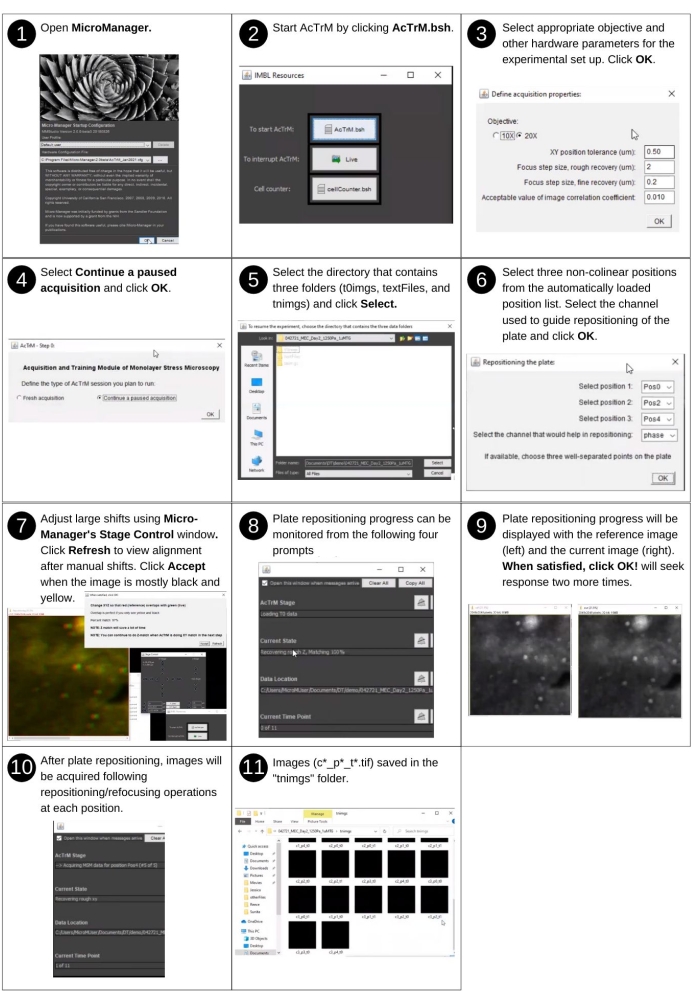
Рисунок 4: Автоматическое получение изображений для оставшегося эксперимента. Шаги для возобновления сбора изображений для оценки поведения клеток с помощью AcTrM. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.

Рисунок 5: Настройка автоматизированного анализа данных. Шаги для начала автоматизированного анализа изображений с помощью AnViM. Программа распознает формат изображения, используемый AcTrM. Подробное представление панелей на этапах 3 и 5 представлено на дополнительном рисунке S5 и дополнительном рисунке S6, соответственно. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
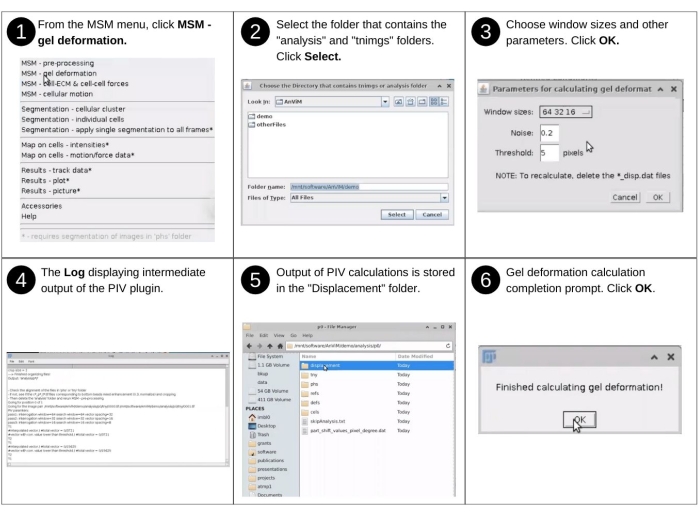
Рисунок 6: Количественная оценка деформации гидрогеля и монослоя - часть 1. Шаги по вовлечению , через AnViM, реализации велоциметрии изображения частиц Tseng, Q. et al., PNAS (2012)20 для количественной оценки деформации верхней поверхности гидрогеля. Пользователи также могут реализовать в AnViM другие подходы для количественной оценки деформации гидрогеля. Подробное представление шага 3 представлено на дополнительном рисунке S7. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
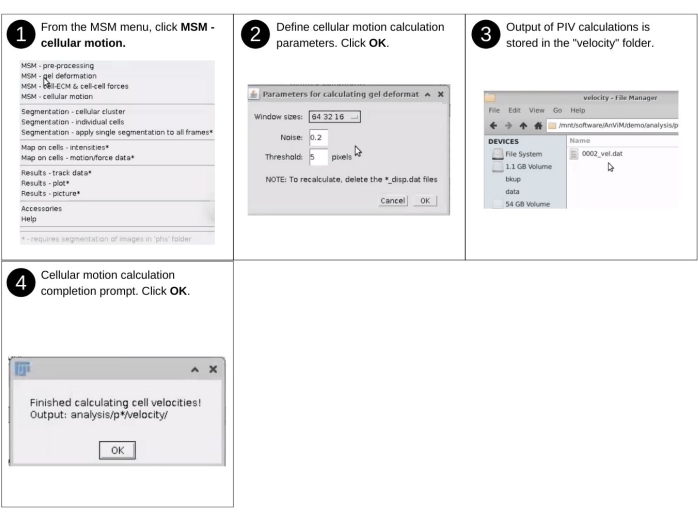
Рисунок 7: Количественная оценка деформации гидрогеля и монослоя - часть 2. Шаги по вовлечению , через AnViM, реализации велоциметрии изображения частиц Tseng, Q. et al., PNAS (2012)20 для количественной оценки локального движения отдельных клеток. Пользователи также могут реализовать в AnViM другие подходы для количественной оценки клеточного движения. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
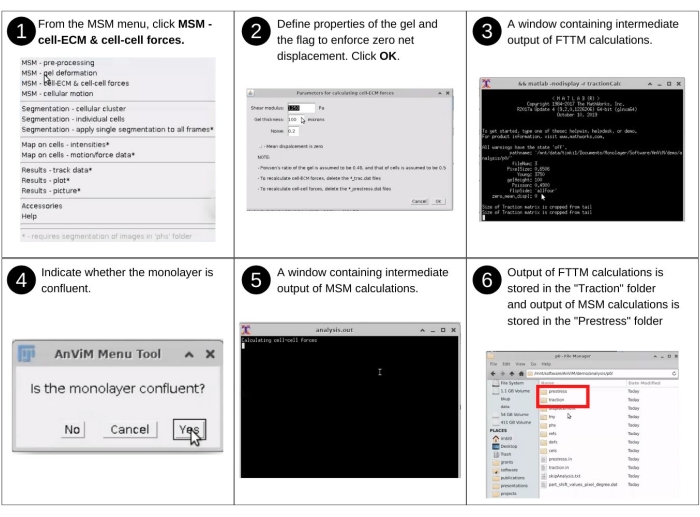
Рисунок 8: Количественная оценка клеточных ECM и клеточно-клеточных сил. Шаги по выполнению анализа изображений для привлечения, с помощью AnViM, реализации Траковой микроскопии преобразования Фурье Trepat et al., Nature Physics (2009)15 для количественной оценки сил, оказываемых клетками на гидрогель, и реализации монослойной стрессовой микроскопии Tambe et al., Nature Materials (2011)1 для количественной оценки сил внутри отдельных клеток и между соседними клетками. Пользователи также могут реализовать в AnViM другие подходы для количественной оценки клеточных ECM и клеточных сил. Подробное представление шага 6 представлено на дополнительном рисунке S8 и дополнительном рисунке S9. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
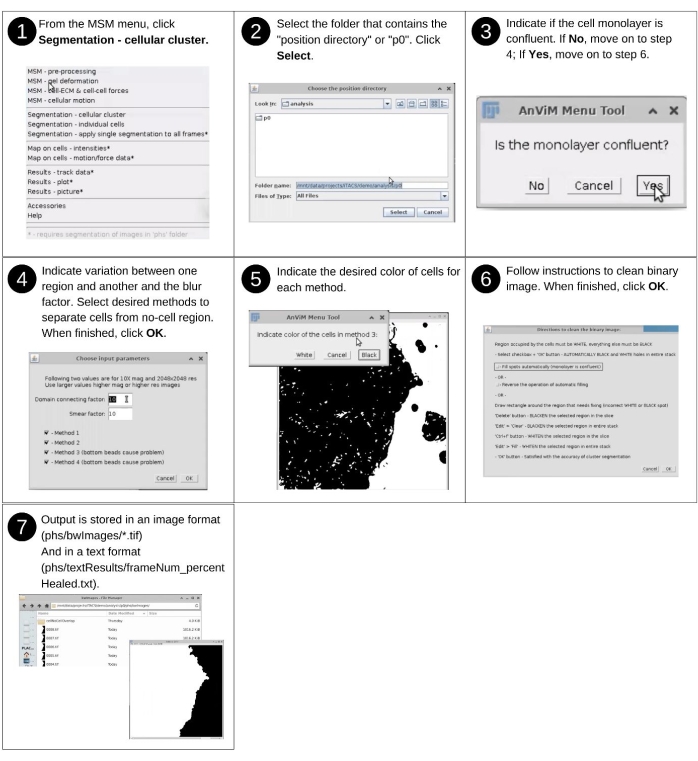
Рисунок 9: Отображение значений точек сетки на отдельных ячейках - часть 1. Шаги по сегментации областей изображения, содержащих ячейки, с использованием нового многостороннего подхода. Этот подход может быть использован для сегментирования фазового контраста, яркого поля или флуоресцентных изображений клеток. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
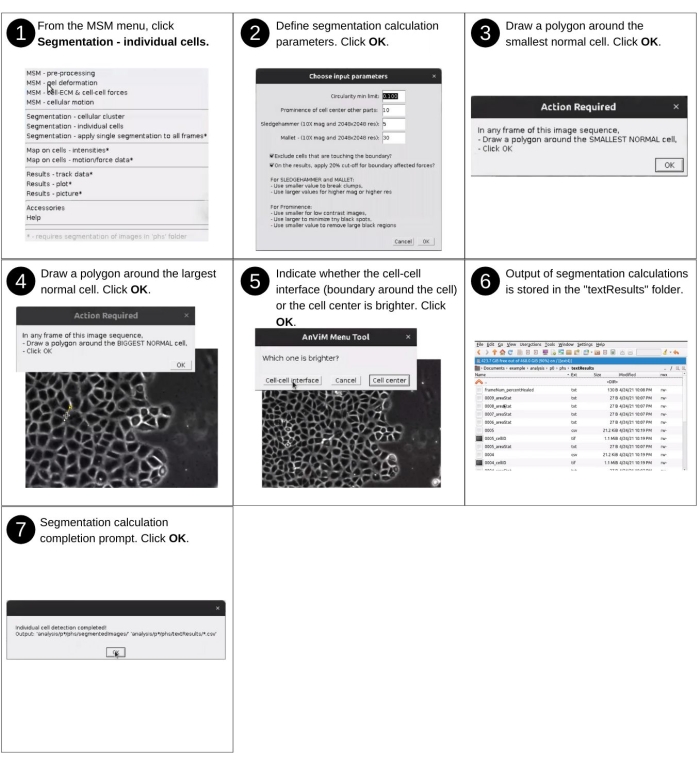
Рисунок 10: Отображение значений точек сетки на отдельных ячейках - часть 2. Шаги по сегментации отдельных ячеек монослоя с использованием нового многостороннего подхода, разработанного в AnViM. Этот подход может быть использован для сегментирования фазового контраста, яркого поля или флуоресцентных изображений клеток. Подробное представление шага 2 представлено на дополнительном рисунке S10. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
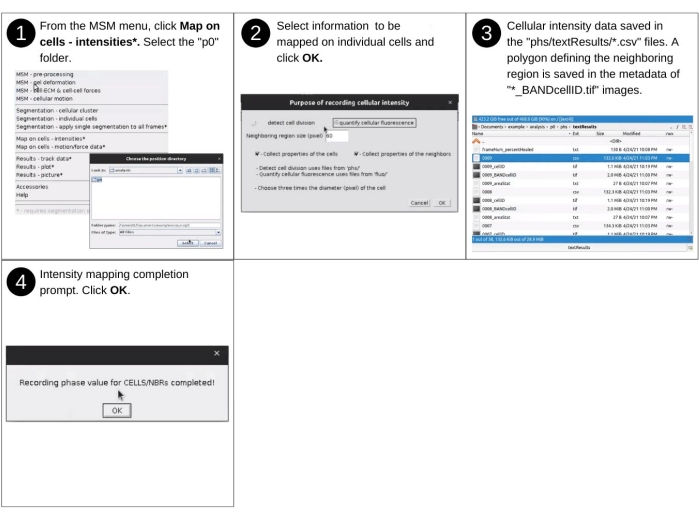
Рисунок 11: Отображение значений точек сетки на отдельных ячейках - часть 3. Шаги для оценки интенсивности пикселей в области внутри отдельных клеток и в соседней области отдельных ячеек с помощью AnViM. Оцениваемые интенсивности включают интенсивность пропускаемого света и интенсивность флуоресценции. Эта часть отображает медианное значение и стандартное отклонение интенсивности пикселей в отдельных ячейках и в соседней области отдельных ячеек. Подробное представление шага 2 представлено на дополнительном рисунке S11. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
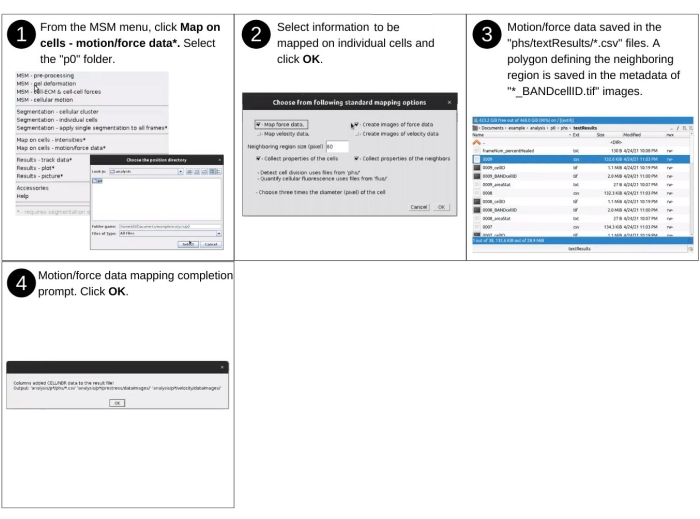
Рисунок 12: Отображение значений точек сетки на отдельных ячейках - часть 4. Шаги для оценки сил и свойств движения точек сетки внутри отдельных ячеек и в соседней области отдельных ячеек с использованием AnViM. Эта часть отображает медианное значение и стандартное отклонение свойств в отдельных ячейках и в соседней области отдельных ячеек. Подробное представление шагов 2 и 3 представлено на дополнительном рисунке S12 и дополнительном рисунке S13. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
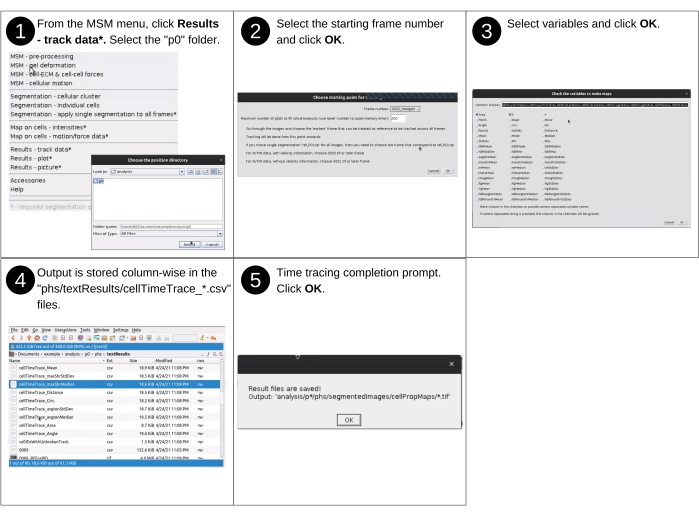
Рисунок 13: Визуализация результатов - часть 1. Шаги для отслеживания свойств отдельных ячеек на протяжении всего эксперимента с помощью AnViM. Подробное представление этапов 2 и 3 представлено на дополнительном рисунке S14 и дополнительном рисунке S15. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
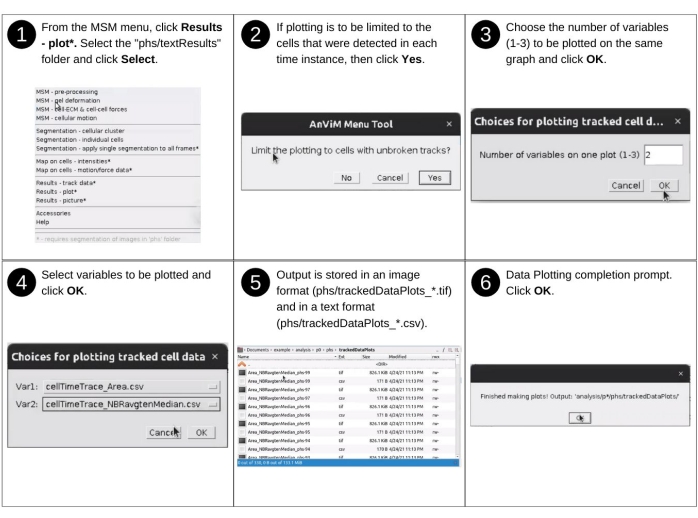
Рисунок 14: Визуализация результатов - часть 2. Шаги для создания временных следов оцениваемых свойств с помощью AnViM. Пользователь имеет возможность построить до трех свойств в одном графике. Временные следы генерируются либо для всех клеток, либо только для тех клеток, для которых отслеживание было успешным на протяжении всего эксперимента. Подробное представление шага 5 представлено на дополнительном рисунке S16 и дополнительном рисунке S17. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
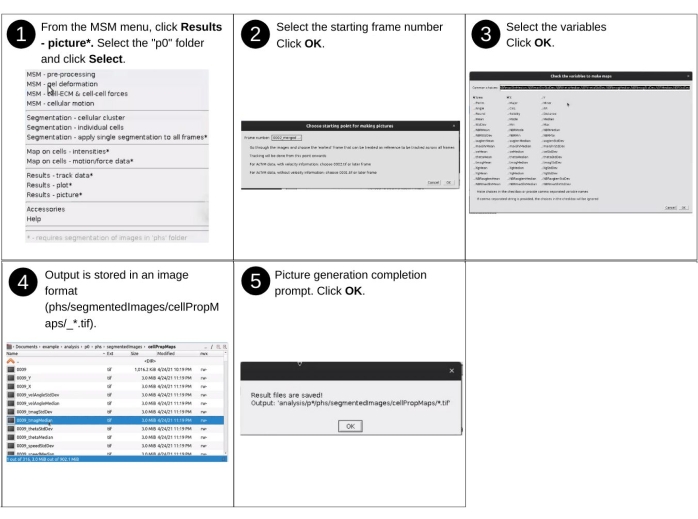
Рисунок 15: Визуализация результатов - часть 3. Шаги по созданию тепловых карт оцениваемых свойств с использованием AnViM. Тепловые карты генерируются для всех кадров, следующих за начальным кадром и всеми выбранными свойствами. Подробное представление шага 3 представлено на дополнительном рисунке S18. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
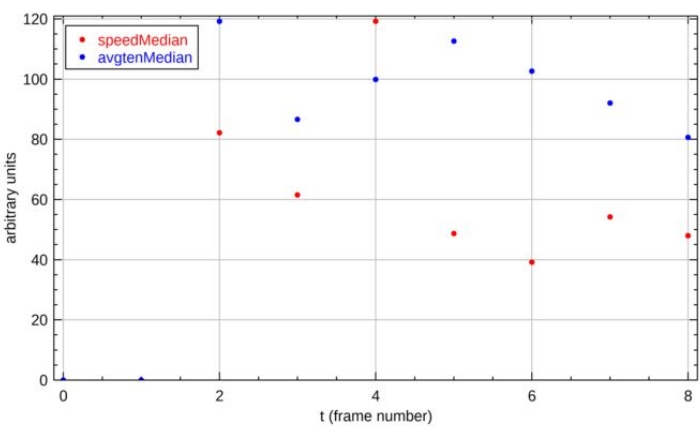
Рисунок 16: Временные трассировки для ячейки ID 1. Двумя отображаемыми свойствами являются клеточное цитоскелетное напряжение («avgtenMedian») и клеточная скорость («speedMedian»). Как клеточное цитоскелетное напряжение, так и клеточная скорость количественно определяются как медианное значение в точках сетки внутри клеток. Эти два свойства строятся на одной оси с произвольными единицами для визуализации отношений между оцениваемыми свойствами. Дополнительные имена переменных перечислены в дополнительной таблице S1. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
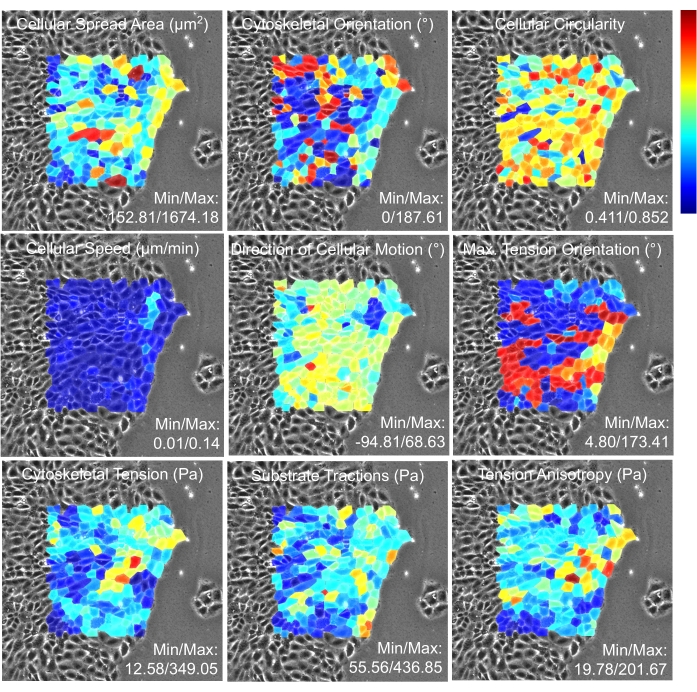
Рисунок 17: Тепловые карты свойств отдельных клеток в анализируемом монослое. Каждая ячейка окрашивается медианным значением свойства, указанного на панели. Таким образом, глубокий красный цвет указывает на максимальное клеточное значение в цветовом спектре, а глубокий синий указывает на минимальное клеточное значение по всему анализируемому монослою. Как описано в Tambe et al., PLoS One (2013)2, клетки, расположенные ближе к границе, имеют механические силы, на которые влияют неизвестные свойства клеток вне изображения. Следовательно, тепловая карта генерируется для ячеек, удаленных от границы. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Рисунок S1: Образцы изображений верхней и нижней флуоресцентной бусины. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Рисунок S2: Подробное представление шага 2 из рисунка 3. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Рисунок S3: Подробное представление шага 4 из рисунка 3. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Рисунок S4 A: подробное представление шага 6 из рисунка 3. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Рисунок S5: Подробное представление шага 3 из рисунка 5. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Рисунок S6: Подробное представление шага 5 из рисунка 5. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Рисунок S7: Подробное представление шага 3 из рисунка 6. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Рисунок S8: Подробное представление выхода силы CELL-ECM шага 6 из рисунка 8. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Рисунок S9: Подробное представление выхода силы ячейки-ячейки на шаге 6 из рисунка 8. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Рисунок S10: Подробное представление шага 2 из рисунка 10. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Рисунок S11: Подробное представление шага 2 из рисунка 11. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Рисунок S12: Подробное представление шага 2 из рисунка 12. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Рисунок S13: Подробное представление выходных данных шага 3 из рисунка 12. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Рисунок S14: Подробное представление шага 2 на рисунке 13. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Рисунок S15: Подробное представление шага 3 из рисунка 13. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Рисунок S16: Подробное представление файлов данных, сгенерированных на шаге 5 из рисунка 14. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Рисунок S17: Подробное представление графика, сгенерированного на шаге 5 из рисунка 14. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Рисунок S18: Подробное представление тепловой карты и файла, содержащего диапазон цветового спектра, сгенерированного на шаге 4 из рисунка 15. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Таблица S1: Перечень выбранных свойств, количественно оцененных iTACS. Пожалуйста, нажмите здесь, чтобы загрузить эту таблицу.
Обсуждение
Адгезивные клетки используют как механические, так и химические сигналы, чтобы выжить, расти и функционировать. Широкий спектр программного обеспечения для микроскопии оптимизирует пользовательский опыт при оценке химических сигналов с помощью флуоресцентной визуализации. Однако оценка механических сигналов включает в себя возможности, которые недоступны в стандартном программном обеспечении для микроскопии. Кроме того, оценка механических сигналов наиболее эффективна, когда сбор данных интегрирован с анализом данных. Отсутствие единой платформы, отвечающей уникальным потребностям оценки механических сигналов, стало серьезным технологическим пробелом в экспериментальной клеточной биологии. Интегративный инструментарий для анализа клеточных сигналов (iTACS) предназначен для устранения этого пробела. Два компонента iTACS, AnViM и AcTrM, предоставляют пользователям необходимые возможности для количественной оценки клеточных свойств четырех широких категорий: силы, движение, морфология и флуоресценция/яркость. В этих категориях iTACS в настоящее время способен выявить более 50 уникальных аспектов отдельных адгезивных клеток. Эти аспекты включают специфические свойства каждой широкой категории, включая их репрезентативное значение и изменчивость по ячейке (Дополнительная таблица S1). Например, внутри сил существуют силы растяжения через цитоскелет, анизотропия этого напряжения, ориентация максимального напряжения и напряжение сдвига через границу раздела клетка-ECM, что оказывает глубокое влияние на поведение адгезивных клеток1,3,6.
Новый подход к исследованию механического поведения отдельных клеток монослоя
Отдельные клетки монослоя занимаются обменом сигналами химической и механической природы3. Эти два типа сигналов передаются по сотовому монослою по-разному23. Однако знания о механической передаче сигнала отстают от знаний о химической передаче сигнала. Этот пробел в знаниях совпадает с устойчивым отсутствием простых и интуитивно понятных подходов к оценке клеточных механических сигналов. Новый подход к картированию данных, описанный здесь, оснащен для заполнения этого пробела. Такое картирование показывает, что флуктуация внутреннего цитоскелетного напряжения в соседней области клетки служит релаксацией, флюидизацией и закреплением сигналов, которые регулируют изменения клеточной формы, размера и скорости клетки18. Карты свойств соседних областей демонстрируют паттерны «многоклеточного деления», где клетки внутри подразделения подвергаются воздействию относительно однородной микросреды, а клетки на границе деления подвергаются воздействию удивительно неоднородной микросреды18.
Доступность технологии измерения силы
Существует множество протоколов для создания гидрогелей PAA, анализа деформации гидрогеля и клеточного движения, а также количественной оценки клеточно-ECM и клеточно-клеточных сил1,2,7,8,9,13,14,15,18,20,21,24,25,26,27 ,28,29,30,31,32. Тем не менее, эти разработки остаются вне досягаемости обычных лабораторий клеточной биологии и ограничены лабораториями с инженерным опытом. Автоматизируя технические аспекты этих подходов и интегрируя их в единую и удобную для пользователя платформу, цель iTACS состоит в том, чтобы сделать оценку механических сигналов рутинной деятельностью в экспериментальных исследованиях и образовании в области клеточной биологии.
ImageJ позволяет пользователям разрабатывать приложения, используя подходы, которые потребуют небольшого обучения или вообще не потребуют его обучения11. iTACS в значительной степени построен с использованием простых подходов к написанию сценариев для содействия непрерывному развитию, ориентированному на сообщество. Большая часть AcTrM программируется с помощью скриптов BeanShell, а основная часть AnViM программируется с помощью макросов ImageJ. Эти скрипты и руководства по реализации этих возможностей на микроскопе пользователя доступны через GitHub (https://github.com/IntegrativeMechanobiologyLaboratory/iTACS).
Стандартизация качества получаемых изображений
Хотя методы количественной оценки физических сил в адгезивных клетках на основе упругого субстрата были разработаны и внедрены в различных лабораториях, протоколу все еще не хватает стандартизации. Одной из областей, которая больше всего нуждается в стандартизации, является качество полученных изображений верхнего бисера (дополнительный рисунок S1). Значительные проблемы возникают из-за дрейфа в фокусе на протяжении всего эксперимента. Наш новый подход к перефокусировке на основе эталонных изображений делает такое сосредоточение объективным процессом. Параметры, определенные на самом первом этапе AcTrM, накладывают необходимые объективные ограничения качества. Другие меры по стандартизации могут быть запрограммированы в будущих версиях AcTrM.
Широкая применимость iTACS
В дополнение к количественной оценке многочисленных аспектов адгезивных клеток, структура iTACS облегчает его использование для различных экспериментальных протоколов и потребностей. AcTrM позволяет программно управлять самообучением пользователей. Высокоскоростная визуализация, необходимая, например, для одновременной оценки колебаний цитоплазматического кальция, в настоящее время ограничена скоростью перепозиционирования и перефокусировки оборудования и лучше всего выполняется в одном месте за раз. Однако текущая реализация хорошо оснащена для долгосрочной визуализации, прерванной визуализации, где образец не может быть сохранен на стадии микроскопа в течение всего периода эксперимента. Поскольку эталонные изображения получены в начале эксперимента, iTACS позволяет визуализировать механические сигналы в режиме реального времени, открывая новые возможности в приложениях для скрининга лекарств. AnViM позволяет пользователям предоставлять высокотехническую информацию в терминах непрофессионала. Способность количественно оценивать широкий спектр клеточных свойств и отслеживать их на протяжении всего эксперимента представляет собой критические возможности, необходимые для открытия новых механизмов межклеточной связи.
Для будущего развития iTACS мы определили четыре основных направления: (1) повышение скорости сбора и анализа данных, (2) внедрение подходов к оценке новых сотовых сигналов13, (3) разработка семинаров и учебных модулей по оценке сотового сигнала на основе iTACS, (4) разработка недорогих решений автоматизации.
Раскрытие информации
Авторы заявляют об отсутствии конфликта интересов.
Благодарности
D.T.T. благодарит сотрудников, связанных с Центром биологии легких в Университете Южной Алабамы, за стимулирование дискуссий о потребностях экспериментальной клеточной биологии в исследованиях. Эти обсуждения сыграли решающую роль в инициировании разработки iTACS.
Эта работа была частично поддержана грантами Национального института здоровья / Национального института крови сердца легких, P01 HL66299 и R37 HL60024 (Стивенс), R01-HL118334 (Альварес), F32-HL144040-01 (Xu), а также Университета Южной Алабамы через Фонд исследований рака Абрахама Митчелла (Сингх, Паланки, Тамбе), Грант на исследования и научные разработки (Тамбе), Колледж с отличием и Летний научный сотрудник бакалавриата (Нгуен).
Материалы
| Name | Company | Catalog Number | Comments |
| Reagents and components used in prepare glass surface for hydrogel coating | |||
| (3-Aminopropyl)trimethoxysilane, 97% | Aldrich chemistry | 13822565 | |
| 2% Bis Solution | Bio-rad | 1610142 | |
| 3-(Trimethoxysilyl)propyl methacrylate,98% | Acros organics | 2530850 | |
| 40% Acrylamide Solution | Bio-rad | 1610140 | |
| Glass bottom 35 mm dish/ 6 or 12 or 24 well plates | MatTek or CellVis | ||
| Glutaraldehyde, EM Grade, 25% | Polysciences | 1909100 | |
| Sodium Hydroxide | Sigma-aldrich | 1002074706 | |
| Reagents and components used in preparing suitable hydrogel | |||
| 2% Bis Solution | Bio-rad | 1610142 | |
| 40% Acrylamide Solution | Bio-rad | 1610140 | |
| Ammonium Persulfate | Bio-rad | 1610700 | |
| Cover Slips | Electron Microscopy Sciences | 7222301 | |
| Dulbecco's Phosphate Buffered Saline (1M) | Gibco | 14190136 | |
| FluoSpheres carboxylate 0.2 um, yellow-green(505/515) | Invitrogen | F8811 | |
| FluoSpheres carboxylate 0.5 um, red(580/605) | Invitrogen | F8812 | |
| FluoSpheres carboxylate 2.0 um, red(580/605) | Invitrogen | F8826 | |
| Rain-X | |||
| TEMED | Bio-rad | 1610801 | |
| Reagents used in coating extracellular matrix on the hydrogel | |||
| Collagen Type I Rat Tail | Corning | 354236 | |
| HEPES(1M) | Gibco | 15630080 | |
| Phosphate Buffered Saline (1M) | Gibco | 10010023 | |
| Sulfo-SANPAH | CovaChem | 102568434 | |
| Microscope hardware used in the current study | |||
| Camera | Hamamatsu Flash 4.0 LT sCMOS Camera | C11440-42U | |
| H117 ProScanTM Stages | Prior Scientific | ||
| Light source- Lambda DG4 and Lambda DG5 | Sutter instrument company | ||
| Microscope | Nikon eclipse TE2000-S | 550372 | |
| ProScan III Universal Microscope Automation Controller | Prior Scientific | ||
| Stagetop incubator | ibidi | 11922 | |
| Stepper Motor Focus Drive | Prior Scientific |
Ссылки
- Tambe, D. T., et al. Collective cell guidance by cooperative intercellular forces. Nature Materials. 10 (6), 469-475 (2011).
- Tambe, D. T., et al. Monolayer stress microscopy: limitations, artifacts, and accuracy of recovered intercellular stresses. PloS One. 8 (2), 55172 (2013).
- Das, T., et al. A molecular mechanotransduction pathway regulates collective migration of epithelial cells. Nature Cell Biology. 17 (3), 276-287 (2015).
- Vedula, S. R., et al. Mechanics of epithelial closure over non-adherent environments. Nature Communications. 6, 6111 (2015).
- Lamason, R. L., et al. Rickettsia Sca4 reduces vinculin-mediated intercellular tension to promote spread. Cell. 167 (3), 670-683 (2016).
- Sunyer, R., et al. Collective cell durotaxis emerges from long-range intercellular force transmission. Science. 353 (6304), 1157-1161 (2016).
- Dong, L., Oberai, A. A. Recovery of cellular traction in three-dimensional nonlinear hyperelastic matrices. Computer Methods in Applied Mechanics and Engineering. 314, 296-313 (2017).
- Nier, V., et al. Kalman inversion stress microscopy. Biophysical Journal. 115 (9), 1808-1816 (2018).
- Serrano, R., et al. Three-dimensional Monolayer Stress Microscopy. Biophysical Journal. 117 (1), 111-128 (2019).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ: 25 years of Image Analysis. Nature Methods. 9 (7), 671-675 (2012).
- Schindelin, J., et al. Fiji - an Open Source platform for biological image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Edelstein, A., Amodaj, N., Hoover, K., Vale, R., Stuurman, N. Computer control of microscopes using microManager. Current Protocols in Molecular Biology. , 20 (2010).
- Patel, N. G., et al. Unleashing shear: Role of intercellular traction and cellular moments in collective cell migration. Biochemical and Biophysical Research Communications. 522 (2), 279-285 (2020).
- Yeung, T., et al. Effects of substrate stiffness on cell morphology, cytoskeletal structure, and adhesion. Cell Motility and the Cytoskeleton. 60 (1), 24-34 (2005).
- Trepat, X., et al. Physical forces during collective cell migration. Nature Physics. 5, 426-430 (2009).
- Aratyn-Schaus, Y., Oakes, P. W., Stricker, J., Winter, S. P., Gardel, M. L. Preparation of complaint matrices for quantifying cellular contraction. Journal of Visualized Experiments: JoVE. (46), (2010).
- King, J., et al. Structural and functional characteristics of lung macro- and microvascular endothelial cell phenotypes. Microvascular Research. 67 (2), 139-151 (2004).
- Patel, G., et al. Mechanical signaling in a pulmonary microvascular endothelial cell monolayer. Biochemical and Biophysical Research Communications. 519 (2), 337-343 (2019).
- Kähler, C. J., et al. Main results of the 4th International PIV Challenge. Experiments in Fluids. 57 (6), 97 (2016).
- Tseng, Q., et al. Spatial organization of the extracellular matrix regulates cell-cell junction positioning. Proceedings of the National Academy of Sciences of the United States of America. 109 (5), 1506-1511 (2012).
- Alvarez-Gonzalez, B., et al. Two-layer elastographic 3-D traction force microscopy. Scientific Reports. 7, 39315 (2017).
- Makarchuk, S., Beyer, N., Gaiddon, C., Grange, W., Hebraud, P. Holographic traction force microscopy. Scientific Reports. 8 (1), 3038 (2018).
- Janmey, P. A., Miller, R. T. Mechanisms of mechanical signaling in development and disease. Journal of Cell Science. 124 (1), 9-18 (2011).
- Dembo, M., Oliver, T., Ishihara, A., Jacobson, K. Imaging the traction stresses exerted by locomoting cells with the elastic substratum method. Biophysical Journal. 70 (4), 2008-2022 (1996).
- Dembo, M., Wang, Y. L. Stresses at the cell-to-substrate interface during locomotion of fibroblasts. Biophysical Journal. 76 (4), 2307-2316 (1999).
- Butler, J. P., Tolic-Norrelykke, I. M., Fabry, B., Fredberg, J. J. Traction fields, moments, and strain energy that cells exert on their surroundings. American Journal of Physiology: Cell Physiology. 282 (3), 595-605 (2002).
- Saez, A., et al. Traction forces exerted by epithelial cell sheets. Journal of Physics. Condensed Matter. 22 (19), 194119 (2010).
- Deforet, M., et al. Automated velocity mapping of migrating cell populations (AVeMap). Nature Methods. 9 (11), 1081-1083 (2012).
- Polio, S. R., Smith, M. L. Patterned hydrogels for simplified measurement of cell traction forces. Methods in Cell Biology. 121, 17-31 (2014).
- Toyjanova, J., et al. 3D Viscoelastic traction force microscopy. Soft Matter. 10 (40), 8095-8106 (2014).
- Serra-Picamal, X., Conte, V., Sunyer, R., Munoz, J. J., Trepat, X. Mapping forces and kinematics during collective cell migration. Methods in Cell Biology. 125, 309-330 (2015).
- Charrier, E. E., et al. A novel method to make viscoelastic polyacrylamide gels for cell culture and traction force microscopy. APL Bioengineering. 4 (3), 036104 (2020).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены