Method Article
Toolkit integrativo per analizzare i segnali cellulari: forze, movimento, morfologia e fluorescenza
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
La piattaforma Integrative Toolkit to Analyze Cellular Signals (iTACS) automatizza il processo di misurazione simultanea di un'ampia varietà di segnali chimici e meccanici nelle cellule aderenti. iTACS è progettato per facilitare lo sviluppo guidato dalla comunità e consentire ai ricercatori di utilizzare tutte le funzionalità della piattaforma indipendentemente dal loro background educativo.
Abstract
La valutazione quantitativa delle forze cellulari e del movimento è avanzata considerevolmente negli ultimi quattro decenni. Questi progressi hanno fornito il quadro per esaminare i processi di segnalazione meccanica approfonditi nei sistemi di coltura cellulare. Tuttavia, il campo deve attualmente affrontare tre problemi: mancanza di standardizzazione della qualità dei dati acquisiti, errori tecnici nell'analisi e nella visualizzazione dei dati e, forse più importante, la tecnologia rimane in gran parte fuori dalla portata dei comuni laboratori di biologia cellulare. Per superare queste limitazioni, abbiamo sviluppato una nuova piattaforma sperimentale - Integrative Toolkit to Analyze Cellular Signals (iTACS). iTACS è costituito da due componenti: Acquisition and Training Module (AcTrM) e Analysis and Visualization Module (AnViM). AcTrM si basa su μManager - un software di controllo del microscopio basato su NIH-ImageJ - e facilita l'autoformazione dell'utente e l'automazione dei comuni protocolli di acquisizione delle immagini. AnViM si basa su NIH-ImageJ e facilita l'automazione user-friendly dell'analisi dei dati e la visualizzazione approfondita dei risultati. Questi esperimenti prevedono la coltura di cellule aderenti su idrogel, l'imaging di marcatori fiduciali incorporati nell'idrogel e, infine, l'estrazione da queste immagini di una caratterizzazione meccanica completa delle cellule. Attualmente, iTACS consente all'utente di analizzare e tracciare una vasta gamma di proprietà, tra cui morfologia, movimento, forze citoscheletriche e fluorescenza delle singole cellule e della loro regione vicina. Il problema della standardizzazione della qualità è stato affrontato in AcTrM con, una tecnica di rifocalizzazione guidata da immagini di riferimento. I problemi tecnici nell'analisi dei dati sono stati affrontati in AnViM con una procedura di segmentazione delle immagini su più fronti, un approccio user-friendly per identificare le condizioni al contorno e una nuova visualizzazione dei dati basata sulla proprietà cellulare. AcTrM è progettato per facilitare la semplice trasformazione dei microscopi a fluorescenza di base in carri sperimentali di meccanica cellulare e AnViM è attrezzato per consentire agli utenti di misurare i segnali meccanici cellulari senza richiedere un background ingegneristico. iTACS sarà disponibile per la comunità di ricerca come suite open source con capacità di sviluppo guidate dalla comunità.
Introduzione
Gli strumenti di imaging ottico e analisi dei dati comunemente usati impiegano tecnologie hardware e software che sono quasi antiquate. Il ritardo nella traduzione e nell'implementazione dei progressi nei dispositivi elettronici, negli approcci computazionali e nell'analisi matematica in comuni strumenti sperimentali di biologia cellulare è un vincolo importante sul ritmo di crescita della nostra conoscenza della fisiologia cellulare. Attualmente, i ricercatori di biologia cellulare trovano strumenti di biologia molecolare a portata di mano, ma strumenti basati su principi ingegneristici sono fuori portata. Uno di questi strumenti basati su principi ingegneristici è la microscopia da sforzo monostrato (MSM)1,2. Mentre MSM è stato adattato e studiato in vari laboratori in tutto il mondo, il suo uso è principalmente limitato ai laboratori con esperienza ingegneristica3,4,5,6,7,8,9.
NIH-ImageJ è uno degli strumenti open source più popolari tra i ricercatori di biologia cellulare10. I progressi guidati dal contributo della comunità di utenti sono stati fondamentali per la sua popolarità11,12. ImageJ ha funzionalità che consentono agli utenti di sviluppare applicazioni con un mix di un linguaggio di programmazione avanzato e approcci di scripting semplificati. Queste funzionalità facilitano gli utenti con conoscenze di programmazione di base per implementare, adattare e far avanzare qualsiasi nuovo contributo al software. Basandoci su queste qualità di NIH-ImageJ, abbiamo sviluppato l'Integrative Toolkit to Analyze Cellular Signals (iTACS), che consente un'integrazione a basso costo degli strumenti hardware e software desiderati per automatizzare la misurazione di un'ampia varietà di segnali chimici e meccanici tra le cellule aderenti11,12.
iTACS comprende due componenti: Acquisition and Training Module (AcTrM) e Analysis and Visualization Module (AnViM). AcTrM è basato su μManager - un'applicazione di acquisizione di immagini basata su NIH-ImageJ - per consentire agli utenti di impostare misurazioni time-lapse delle proprietà ottiche tradizionali e una varietà di proprietà fisiche delle celle aderenti in più campioni12. AcTrM facilita la formazione dell'utente attraverso indicazioni concise incluse nell'interfaccia grafica. Inoltre, ha una nuova funzione di messa a fuoco automatica basata su immagini di riferimento progettata per facilitare le misurazioni in tempo reale delle forze fisiche e consentire la standardizzazione della qualità dei dati acquisiti.
AnViM è costruito su plugin ImageJ, software accelerato e script di gestione dei file che consentono agli utenti di valutare quantitativamente più di 50 proprietà, tra cui forma cellulare, dimensioni, orientamento, velocità e direzione del movimento, trazioni esercitate sulla matrice extracellulare (ECM) e sulle cellule vicine, momenti contrattili e di taglio di entrambe le singole cellule aderenti e della loro regione vicina. AnViM facilita gli utenti a quantificare le proprietà fisiche cellulari senza padroneggiare il background tecnico sottostante11. Inoltre, consente l'analisi dei dati in modalità di elaborazione interattiva o batch. Genera mappe di calore che rivelano la variazione spaziale e grafici che mostrano la variazione temporale delle proprietà delle singole cellule.
In un esperimento tipico, l'utente coltiva cellule su un idrogel elastico con appropriate proteine della matrice extracellulare sulla superficie superiore e due tipi di marcatori fluorescenti incorporati. Essenzialmente, le immagini di questi marcatori fluorescenti prima e dopo la coltura delle cellule sono sufficienti per quantificare le forze all'interno e intorno alle singole cellule2,13. AnViM mappa questi risultati su singole celle del cluster aderente e genera immagini e grafici approfonditi.
Protocollo
NOTA: I campioni esaminati utilizzando la piattaforma iTACS sono cellule aderenti a un substrato morbido. Il protocollo per la valutazione dei segnali meccanici e chimici è diviso in due parti sequenziali: Acquisition and Training Module (AcTrM) e Analysis and Visualization Module (AnViM).
1. Modulo di acquisizione e formazione (AcTrM)
NOTA: AcTrM automatizza il processo di acquisizione dei dati e l'autoformazione dell'utente. Prima di qualsiasi acquisizione di dati, preparare un substrato morbido in grado di fornire le informazioni necessarie per quantificare le forze che le cellule esercitano su di esso.
- Preparazione idrogel
NOTA: L'obiettivo qui è quello di preparare un modulo di taglio di 1.250 Pa, uno spessore di circa 100 μm e un idrogel di poliacrilammide (PAA) di diametro 22 mm.- Preparare la soluzione di poliacrilammide seguendo il metodo di Yeung et al. e gettare gli idrogel seguendo i passaggi descritti da Trepat et al.14,15. Un'eccezione alla procedura è che le perline incorporate immediatamente sotto le cellule hanno un diametro di 0,5 μm ed emettono fluorescenza gialla.
- Saltare il passaggio di fissaggio di perline da 2 μm al vetro di copertura se si prevede che l'area di visualizzazione abbia una grande regione priva di cellule.
NOTA: il protocollo di preparazione dell'idrogel è ora abbastanza consolidato nel campo16. In tutta la descrizione successiva, le perle da 0,5 μm sono indicate come "perline superiori" e le perle da 2 μm sono indicate come "perle inferiori". Tuttavia, le perline inferiori sono facoltative quando la regione con immagini contiene un'ampia area priva di celle. Il modello di perline superiore da una tale regione priva di cellule servirà allo scopo del modello di perline inferiore. - Montare l'idrogel con perline fluorescenti incorporate sul palco del microscopio.
- Lasciare 15-20 minuti affinché la temperatura della piastra raggiunga uno stato stazionario.
NOTA: La superficie superiore della piastra è funzionalizzata con proteine della matrice extracellulare, ma le cellule non sono ancora seminate sull'idrogel.
- Acquisizione di immagini di riferimento
- Parte 1: Creazione di un elenco di posizioni
NOTA: l'input manuale in questo passaggio include la seguente applicazione delle istruzioni AcTrM per scegliere le posizioni con la migliore distribuzione di perline. In questo passaggio non vengono utilizzati file creati in precedenza. Le nuove cartelle chiave prodotte in questo passaggio includono le cartelle 't0imgs', 'tnimgs' e 'textfiles' e il file prodotto include il file dell'elenco delle posizioni. Vedere la Figura 2 per la descrizione visiva dei passaggi seguenti che guidano la creazione di un elenco di posizioni utilizzando AcTrM. L'esempio dimostrato acquisisce dati con ingrandimento 20x.- Avviare μManager 2.0 - Beta.
- Eseguire AcTrM facendo clic sul pulsante AcTrM.bsh nella finestra Risorse IMBL .
- Nella finestra intitolata Definisci proprietà di acquisizione, selezionare l'obiettivo appropriato, la tolleranza per l'accuratezza del recupero della posizione laterale (ad esempio, XY), la dimensione del passo dell'operazione di rimessa a fuoco approssimativa e raffinata (ad esempio, z) e il valore del coefficiente di correlazione dell'immagine accettabile per il recupero della messa a fuoco.
NOTA: una tolleranza più fine per il recupero della posizione laterale rallenterà il recupero della posizione, ma una tolleranza sostanziale richiederà la correzione della posizione durante l'analisi dei dati e potrebbe causare difficoltà nel recupero della messa a fuoco. Una dimensione del passo più piccola rallenterà l'operazione di rifocalizzazione, ma una dimensione del passo molto elevata può causare un movimento rapido con l'opportunità di perdere la messa a fuoco. - Nella finestra AcTrM - Step 0 , selezionare Fresh Acquisition per creare un elenco di posizioni.
NOTA: la finestra intitolata AcTrM - Step 1 elenca tutti i passaggi successivi chiave. Questi passaggi comportano lo spostamento dello stage, la regolazione della messa a fuoco e il clic sui pulsanti nel μManager. Questi passaggi guidano nella costruzione dell'elenco delle posizioni adatte all'acquisizione dei dati. - Fare clic su Live in Micromanager per visualizzare i campioni per le regolazioni manuali elencati nella finestra AcTrM - Step 1 .
- Seguire i passaggi elencati in questa finestra.
- Ora acquisisci l'immagine delle perline. Selezionare il canale appropriato per le perline superiori.
- Assicurati di guardare anche le perline inferiori. Per fare ciò, selezionare i canali per le perline inferiori. Si vede un'immagine sfocata delle perline inferiori.
NOTA: i canali possono essere commutati dal menu a discesa preimpostato nella finestra principale di μManager. Se nell'esperimento vengono utilizzate le perle inferiori, assicurarsi che siano presenti nella posizione selezionata. Queste perline appariranno sfocate. - Dopo aver selezionato la posizione appropriata, fare clic su Contrassegna nella finestra Elenco posizioni stage per salvare la posizione.
NOTA: La posizione migliore è definita da una distribuzione densa e uniforme delle perline superiori incorporate immediatamente sotto la superficie superiore (cioè perline superiori) e da almeno due perline attaccate al vetro di copertura (cioè perline inferiori) (Figura supplementare S1). Il recupero della messa a fuoco è più veloce se le perline inferiori appaiono come grandi ghiere sfocate. Tuttavia, se gli esperimenti vengono eseguiti in una posizione senza necessità di messa a fuoco o recupero della posizione laterale, ignorare le istruzioni relative all'immagine delle perline inferiori. - Seguire i passaggi da 6 a 9 descritti nella Figura 2 per includere posizioni aggiuntive nell'elenco.
NOTA: Scegli alcune posizioni aggiuntive in modo che un round di eliminazione possa consentire le migliori posizioni scelte sul piatto.
- Parte 2: Acquisizione di immagini di riferimento
NOTA: questo passaggio non comporta alcun input manuale. I file chiave creati in precedenza utilizzati in questo passaggio includono 'textfiles/*.pos'. I nuovi file e cartelle chiave prodotti in questo passaggio includono quelli all'interno delle cartelle 't0imgs' e 'tnimgs'. Vedere la Figura 3 e la Figura supplementare S2 per la descrizione visiva dei seguenti passaggi che guidano l'acquisizione di immagini di riferimento utilizzando AcTrM. La finestra AcTrM - Step 2 elenca anche tutti i passaggi chiave.- Seguendo i passaggi elencati nella finestra AcTrM - Step 2 , effettuare le scelte nella finestra Acquisizione multidimensionale . Ad esempio, per eseguire un intervallo di tempo prolungato, indicare un numero di immagini da acquisire, selezionare il primo canale come canale di fase, quindi selezionare il successivo per le perline superiori e quello successivo è per le perline inferiori. Fare clic su Chiudi nella finestra Acquisizione multidimensionale , quindi fare clic su OK nella finestra ActrM - Step 2 .
- Nella finestra AcTrM - Step 3 , scegliere di attivare il ripristino della posizione XYZ basato su immagine di riferimento. La scelta è generalmente sì. Tuttavia, se le immagini vengono acquisite in una posizione senza spazio per la messa a fuoco o la deriva dello stage, la scelta nella finestra AcTrM - Step 3 sarà No.
- Nella finestra Opzioni di ripristino AcTrM - XYZ , impostare il canale per il ripristino XYZ approssimativo, se eseguire il ripristino XYZ raffinato, la regione e il canale per il recupero XYZ raffinato e la direzione per iniziare la rimessa a fuoco (Figura supplementare S4). Una volta completato, AcTrM acquisirà immagini di riferimento.
NOTA: le scelte tipiche per il canale saranno il canale delle perline inferiori. Il recupero XYZ funziona meglio se eseguito con l'immagine completa nell'implementazione corrente. Le immagini di riferimento includono in genere un'immagine di luce trasmessa dell'idrogel, un'immagine fluorescente di perline superiori e un'immagine fluorescente delle perline inferiori. Il contenuto di ogni set di immagini può variare in base alle scelte effettuate nella finestra Acquisizione multidimensionale . Tuttavia, il software acquisirà l'immagine di riferimento impostata in ogni posizione determinata nella Figura 2. Alla fine di questo passaggio, nella directory scelta vengono create tre cartelle: 't0imgs', 'tnimgs' e 'textfiles'. La cartella 't0imgs' contiene immagini di riferimento nel formato riconosciuto da μManager e utilizzato nei passaggi successivi; La cartella 'tnimgs' contiene immagini TIFF separate con nomi di file 'c0_p0_t0.tif', 'c1_p0_t0.tif', ecc. Qui, 'c' sta per il canale, 'p' sta per la posizione e 't' sta per il tempo. Seguono rispettivamente il numero di canale, il numero di posizione e il numero di fotogramma. La cartella 'textfiles' contiene l'elenco delle posizioni in formato XML e le scelte dell'utente per l'acquisizione delle immagini e il recupero della posizione.
- Parte 1: Creazione di un elenco di posizioni
- Semina e crescita cellulare
NOTA: La piattaforma iTACS ha la flessibilità di accogliere i protocolli di preparazione dei campioni utilizzati nella comune valutazione in vitro del comportamento meccanico delle cellule aderenti, comprese le cellule sparse, il monostrato completamente confluente, il test di formazione della rete e i monostrati con fori o lacune significative.- Coltura di cellule endoteliali microvascolari polmonari di ratto alla confluenza in un pallone17,18.
- Staccare le cellule usando la tripsina. Risospese le cellule in un terreno di coltura contenente il 10% di siero bovino fetale ad una concentrazione 1 x 106 cellule/ml.
- Posizionare una goccia da 5 μL delle cellule risospese su una superficie di idrogel parzialmente asciutta e posizionarla nell'incubatore di colture cellulari.
NOTA: Dopo 2 giorni nel terreno di coltura contenente il 10% di siero bovino fetale, le cellule in quella goccia formano un'isola affollata di cellule1.
- Acquisizione automatica delle immagini per l'esperimento rimanente
NOTA: l'input manuale per questo passaggio include la seguente visualizzazione delle richieste AcTrM per riprendere l'acquisizione dell'immagine. I file chiave creati in precedenza utilizzati da questo passaggio includono 'textfiles/*.pos' e file di immagine del tallone inferiore nella cartella 't0imgs'. I nuovi file chiave prodotti in questo passaggio includono i file dell'elenco delle posizioni aggiornati e le immagini 'tnimgs/*_t*.tif'.- Assicurarsi che il sistema di controllo ambientale del microscopio raggiunga condizioni di coltura tissutale stabili.
- Montare delicatamente la piastra contenente cellule coltivate sullo stadio del microscopio.
- Lasciare 15-20 minuti affinché la temperatura e l'umidità si stabilizzino.
NOTA: vedere la Figura 4 per la descrizione visiva dei passaggi seguenti che guidano l'acquisizione di immagini per l'esperimento rimanente utilizzando AcTrM. - Avviare μManager 2.0 - Beta.
- Eseguire AcTrM facendo clic sul pulsante AcTrM.bsh nella finestra Risorse IMBL .
Nota : ignorare le scelte effettuate nella finestra Definisci proprietà di acquisizione , ma utilizzare le scelte effettuate nel passaggio precedente. - Nella finestra AcTrM - Passaggio 0 , selezionare Continua acquisizione in pausa.
- Selezionare l'ingrandimento e la directory identificati nella finestra Acquisizione multidimensionale nel passaggio precedente in cui sono state salvate le cartelle di dati ('t0imags', 'tnimgs' e 'textfiles').
NOTA: il titolo della finestra guida l'utente a scegliere la directory appropriata. - Nella finestra Riposizionamento della piastra , selezionate le opzioni per il riposizionamento della piastra (tre posizioni non collegiali) insieme al canale utilizzato per il riposizionamento.
NOTA: le immagini salvate verranno visualizzate nella fotocamera. Se le immagini sovrapposte vengono visualizzate come immagini a colori rosso, verde e nero, eseguire la regolazione manuale. - Clicca su Accetta per procedere con l'acquisizione.
NOTA: questo passaggio supera i lievi spostamenti dall'irriproducibilità dell'allineamento delle piastre quando la piastra viene rimontata sullo stadio del microscopio. Seguire le istruzioni AcTrM per accelerare l'allineamento in ciascuna delle tre posizioni mostrando un'immagine composita dell'immagine di riferimento mostrata in rosso (tipicamente delle perline inferiori) e l'immagine attualmente osservata attraverso l'obiettivo mostrato in verde. Ottenere il verde sufficientemente vicino al rosso lascia meno lavoro per il software. Quando la sovrapposizione è perfetta, l'immagine composita appare gialla e nera. Sufficientemente vicino è in genere quando le stesse perle inferiori sono visibili sia nelle immagini rosse che in quelle verdi e le corrispondenti ghiere rosse e verdi fuori fuoco si toccano. Al termine del riposizionamento della piastra, AcTrM porta il palco in ogni posizione selezionata e recupera la posizione XYZ abbinando l'immagine di riferimento a ciò che è attualmente visto attraverso la fotocamera. La prima posizione laterale (XY) viene abbinata e quindi viene abbinata la messa a fuoco (Z). Il recupero approssimativo è seguito da un raffinato recupero della posizione e della messa a fuoco. Gli aggiornamenti dello stato corrente dell'esperimento vengono visualizzati sullo schermo attraverso una finestra in quattro parti mostrata nella Figura supplementare S3. Le immagini acquisite vengono salvate nella cartella 'tnimgs' dopo '*_t1.tif', '*_t2.tif', indicando il conteggio temporale per il set di immagini. Se XYZ recovery identifica una posizione aggiornata, il nuovo elenco di posizioni viene generato e salvato nella cartella 'textfiles'.
2. Modulo di analisi e visualizzazione (AnViM)
- Impostazione dell'analisi automatizzata dei dati
NOTA: l'input manuale in questo passaggio include l'identificazione della posizione della cartella 'tnimgs'. I file chiave creati in precedenza utilizzati in questo passaggio includono 'tnimgs/*.tif'. I nuovi file e cartelle chiave prodotti in questo passaggio includono la cartella 'analisi' nella stessa cartella principale di 'tnimgs' e le cartelle per ogni posizione 'analysis/p*'. All'interno di ogni 'analisi/p*', questo passaggio crea un nuovo file immagine '*.tif' all'interno delle cartelle 'phs' e 'tny'. Queste immagini sono immagini ritagliate e de-drifted di cellule e perline superiori. Gli altri file creati includono 'analysis/analysisChoices.txt' che elenca le scelte di analisi, 'analysis/p*/skipAnalysis.txt', che elenca i casi in cui l'analisi viene saltata a causa di una significativa deriva preesistente, e 'analysis/p*/part_shift_values_pixel_degree.dat' che elenca i valori di deriva stimati. AnViM non altera i dati grezzi nella cartella 'tnimgs'. Vedere la Figura 5 per la descrizione visiva dei passaggi seguenti che guidano l'impostazione dell'analisi automatica dei dati tramite AnViM.- Avviare il software Fiji e selezionare la prima opzione nel menu a discesa MSM etichettato come MSM - pre-elaborazione.
- Utilizzando la finestra di dialogo del browser dei file, selezionare la cartella contenente la cartella 'tnimgs', che contiene i dati analizzati.
- Definire il canale per le immagini. Seguendo le linee guida della Figura supplementare S5, identificare i numeri di canale dell'immagine luminosa trasmessa delle celle (immagine a contrasto di fase delle celle), immagine delle perline inferiori e immagine delle perline superiori. Le seguenti tre caselle di controllo ("Sposta i file nella cartella della posizione", "Esegui ulteriori correzioni per il movimento rigido" e "Ritaglia i dati e salva nella cartella della posizione") sono in genere selezionate. Infine, definire la tolleranza per il rifiuto di dati fortemente derivati, dimensioni dei pixel e lati dell'immagine in cui le celle si incrociano.
- Rispondi alla richiesta di migliorare la luminosità e il contrasto delle immagini di perline inferiori in modo che le perline appaiano in modo prominente. Regolalo usando la barra di scorrimento nel menu e fai clic su OK.
- Ora esegui la correzione della posizione per sbarazzarti dei turni, se presenti. Una volta fatto, viene creata una cartella di analisi.
NOTA: in genere non è necessario migliorare il contrasto, ma questa disposizione è disponibile per gli esperimenti in cui l'esposizione ai laser deve essere ridotta al minimo. Dopo tale scelta, AnViM copia i file dalla cartella 'tnimgs' alla cartella 'analysis'. I file corrispondenti a ciascuna posizione vengono salvati nelle cartelle 'analisi/p0, analisi/p1', ecc. All'interno di ciascuna di queste cartelle di posizione, AnViM crea cartelle 'cels', 'defs' e 'refs' contenenti rispettivamente l'immagine luminosa trasmessa originale, le immagini di perline superiori e le immagini di perline inferiori (Figura supplementare S6). AnViM analizza quindi il movimento rigido nelle immagini delle perline inferiori e crea una cartella "phs" contenente un'immagine luminosa trasmessa corretta delle celle e una cartella "tny" contenente immagini di perline superiori aggiornate. Infine, le scelte dell'utente dei canali, delle operazioni, della tolleranza, della dimensione dei pixel e dell'incrocio dei confini vengono salvate nel file 'analysisChoices.txt' (Figura supplementare S6).
- Quantificazione della deformazione di idrogel e monostrato
NOTA: È importante notare che la quantificazione del movimento da una sequenza di immagini è un campo in rapida evoluzione19. La tecnologia è costantemente ottimizzata per le caratteristiche, tra cui velocità, precisione, caratteristiche specifiche all'interno delle immagini raw e modelli di deformazione specifici. Quindi, è probabile che alcuni utenti possano utilizzare un approccio di quantificazione della deformazione diverso da quello qui presentato.-
Parte 1: Quantificazione della deformazione dell'idrogel
NOTA: gli input manuali in questo passaggio includono l'identificazione della directory dei dati e la selezione della risoluzione della griglia. I file chiave creati in precedenza utilizzati in questo passaggio includono "p* / tny / * .tif". I nuovi file chiave prodotti in questo includono "p * / displacement / * _disp.dat" che elenca i vettori di spostamento della superficie superiore dell'idrogel. Vedere la Figura 6 per la descrizione visiva dei seguenti passaggi per eseguire, tramite AnViM, l'analisi di velocimetria delle immagini di particelle sulle immagini delle perline superiori20.- Avviare il software Fiji e dal menu a discesa MSM selezionare MSM - Deformazione gel.
- Da qui, seleziona l'opzione adatta all'esperimento.
- Utilizzando la finestra di dialogo del browser dei file, selezionare la directory principale della cartella "analisi" contenente i dati analizzati.
- Nella finestra Parametri per il calcolo della deformazione del gel , selezionare le dimensioni della finestra di correlazione incrociata, il livello di rumore e la soglia appropriati (Figura supplementare S7)20.
NOTA: i risultati vengono salvati in una directory 'displacement' appena creata all'interno di ogni cartella di posizione 'analysis/p0, analysis/p1', ecc. Qui è dove vengono memorizzati tutti i file di output.
-
Parte 2: Quantificazione della deformazione monostrato
NOTA: gli input manuali in questo passaggio includono l'identificazione della directory dei dati e la selezione della risoluzione della griglia. I file chiave creati in precedenza utilizzati in questo passaggio includono "p* / tny / * .tif". I nuovi file chiave prodotti in questo includono 'p* / velocità / * _vel.dat" che elenca i vettori di movimento delle celle. Vedere la Figura 7 per la descrizione visiva dei passaggi per eseguire, tramite AnViM, l'analisi di velocimetria dell'immagine delle particelle sulle immagini delle perline superiori20. La procedura è simile a quella seguita per quantificare la deformazione dell'idrogel e seleziona MSM - Cellular Motion dal menu a discesa MSM . I risultati vengono salvati in una directory 'velocità' appena creata all'interno di ogni cartella di posizione 'analysis/p0', 'analysis/p1', ecc.
-
Parte 1: Quantificazione della deformazione dell'idrogel
- Quantificazione delle forze cellula-ECM e cellula-cellula
NOTA: l'input manuale in questo passaggio include l'identificazione della directory dei dati, l'indicazione della rigidità dell'idrogel e la risposta alle richieste di segmentazione del cluster cellulare per rilevare le cellule dalla regione senza cellule. I principali file creati in precedenza utilizzati in questo passaggio includono "p* / displacement / * _disp.dat". I nuovi file chiave prodotti in questa fase includono: 'p*/traction/*_trac.dat', che elenca le forze esercitate dalle cellule sull'idrogel; «p*/traction/*_domain.dat», che elenca la posizione dei punti della griglia contenenti celle; e i file di input 'p*/traction.in, p*/clusterInput.txt' e 'p*/prestress.in' che registrano le scelte dell'utente per questo passaggio. A seguito della prima valutazione quantitativa delle forze cellula-ECM e cellula-cellula, sono state sviluppate diverse varianti alla tecnica1,15. Le variazioni si concentrano su casi particolari di substrati, cellule, condizioni sperimentali o strumenti numerici7,8,21,22. Vedere la Figura 8 per la descrizione visiva da eseguire tramite AnViM, la microscopia a trazione a trasformata di Fourier e l'analisi al microscopio da sforzo monostrato sui dati di deformazione dell'idrogel1,2,15.- Avviare il software Fiji e dal menu a discesa MSM selezionare MSM - Cell-ECM e Cell-Cell Forces (terza opzione nel menu a discesa).
- Utilizzando la finestra di dialogo del browser dei file, selezionare la directory principale della cartella 'analisi' contenente le cartelle 'tnimgs' e 'analisi' contenenti i dati analizzati. Fare clic su Seleziona.
- Nella finestra Parametri per il calcolo delle forze cellulari , immettere il modulo di taglio, lo spessore dell'idrogel e il livello di rumore previsto. Selezionare OK. Ciò consente di eseguire la trazione tramite la funzione MATLAB.
NOTA: a seguito di questi input, AnViM calcola le forze ECM delle celle. - Successivamente, indica se il monostrato è confluente. Se l'intera immagine della cella è coperta di celle, la risposta è Sì. In tal caso, le forze cellula-cella verranno calcolate su tutto il fotogramma. D'altra parte, se una parte dell'immagine non ha celle, allora la risposta è No. In tal caso, seguire le istruzioni anViM per facilitare la segmentazione dell'area dell'immagine che contiene le celle.
- Se la risposta è no, disegnare manualmente un poligono attorno all'oggetto non cella quando il software richiede di farlo, quindi selezionare uno o più metodi appropriati per la segmentazione. Il software chiederà il colore (bianco o nero) delle celle.
- Seleziona l'opzione Riempi punti automaticamente nel software e fai clic su OK.
NOTA: I passaggi per la segmentazione di un monostrato non confluente saranno trattati nella prima parte della sezione 2.4. Le forze Cell-ECM sono memorizzate in file '*_trac.dat' in una directory 'traction' appena creata all'interno di ogni cartella di posizione, e l'input al software di calcolo della forza cell-ECM viene salvato nel file 'traction.in' (Figura supplementare S8). Le forze cella-cella sono memorizzate in file '*_prestress.dat' in una directory appena creata 'prestress' all'interno di ogni cartella di posizione, e l'input al software di calcolo della forza cella-cella viene salvato nel file 'prestress.in' (Figura supplementare S9).
- Mappatura dei valori dei punti griglia sulle singole celle
NOTA: Uno degli obiettivi attuali di iTACS è quello di introdurre un approccio diretto per interpretare i segnali meccanici misurati sul campo. Questo approccio è utile per esaminare le interazioni meccaniche tra cellule vicine di un cluster18. Nel complesso, le proprietà quantificate fino ad ora sono su punti griglia regolarmente distanziati attraverso il cluster cellulare. Questi dati identificano la deviazione mediana, media e standard delle proprietà selezionate all'interno del confine morfologico delle singole cellule e le assegnano come proprietà fisiche / segnali cellulari. Da questi, la deviazione standard viene utilizzata per indicare la variabilità, la differenza tra media e mediana viene utilizzata per indicare la natura della distribuzione e il valore mediano viene utilizzato per indicare lo stato complessivo delle celle per le proprietà selezionate.- Parte 1: Segmentazione della regione dell'immagine che contiene le celle
NOTA: l'input manuale per questo passaggio include l'identificazione della directory di dati e la risposta alle richieste di segmentazione per rilevare le celle dall'area senza celle. I file chiave creati in precedenza utilizzati in questo passaggio includono "p* / phs / * .tif". I nuovi file chiave prodotti in questo passaggio includono 'p*/phs/bwImages/*.tif' che sono immagini binarie che separano le regioni delle celle dalle regioni senza celle, 'p*/phs/textResults/frameNum_percentHealed.txt', che elenca la percentuale di area dell'immagine coperta dalle celle in ogni istanza, e 'p*/clusterInput.txt', che registra le scelte dell'utente inserite nell'interfaccia AcTrM. Vedere la Figura 9 per la descrizione visiva dei passaggi seguenti per segmentare l'area dell'immagine che contiene le celle. Questa parte deve essere completata prima di calcolare le forze cellula-cella. In tal caso, questi passaggi non devono essere ripetuti. Inoltre, se questi passaggi vengono eseguiti prima dei calcoli della forza cella-cella, non vengono richiesti all'inizio dei calcoli della forza.- Avviare il software Fiji e dal menu a discesa MSM selezionare Segmentazione - Cluster cellulare.
- Utilizzando la finestra di dialogo del browser dei file, selezionare la directory di posizione 'analysis/p0', 'analysis/p1', ecc., che contiene le proprietà quantificate su punti griglia regolarmente distanziati.
- Indicare se il monostrato è confluente.
NOTA: i passaggi seguenti vengono richiamati solo quando il monostrato non è confluente. Seguire le istruzioni anViM per applicare il nuovo approccio su più fronti per identificare le regioni dell'immagine che contengono le celle. Il processo prevede quattro metodi, ognuno dei quali si avvicina alla segmentazione in modo diverso. Uno o più di questi metodi utilizzati in combinazione coprono un'ampia varietà di immagini delle cellule. Pertanto, prova diversi approcci per trovare quale combinazione funziona meglio per i loro dati. - Regola il "fattore di connessione del dominio" e il "fattore di striscio" per ottenere una segmentazione ottimale.
NOTA: il "fattore di striscio" rende più grandi le regioni che contengono le cellule. - Indicare se le celle appaiono in bianco o nero nell'immagine binaria.
- Nella finestra Istruzioni per pulire l'immagine limite , scegliere se pulire l'immagine automaticamente o manualmente.
NOTA: le caratteristiche indesiderate delle immagini sono macchie nere sui pixel occupati dalle celle e macchie bianche sui pixel disconnessi dal cluster di celle da analizzare. L'analisi può essere eseguita solo sulle regioni collegate. Quindi più regioni disconnesse devono essere analizzate separatamente.
- Parte 2: Segmentazione delle singole celle nelle immagini
NOTA: gli input manuali per questo passaggio includono l'identificazione della directory dei dati e la risposta alle richieste di segmentazione per rilevare singole celle all'interno dell'immagine. I principali file creati in precedenza utilizzati in questo passaggio includono 'p*/phs/*.tif' e 'p*/phs/bwImages/*.tif'. I nuovi file chiave prodotti includono 'p*/phs/textResults/*.csv', che contengono proprietà morfologiche cellulari. Vedere la Figura 10 per la descrizione visiva dei seguenti passaggi per segmentare le singole celle del monostrato.- Avviare il software Fiji e dal menu a discesa MSM selezionare Segmentazione - Singole celle.
- Utilizzando la finestra di dialogo del browser dei file, selezionare la directory di posizione 'analysis/p0', 'analysis/p1', ecc., che contiene le proprietà quantificate su punti griglia regolarmente distanziati.
- Nella finestra Scegli i parametri di input , indicare le proporzioni massime, la prominenza di quanto spicca il centro della cella rispetto al limite della cella e due parametri di sfocatura per guidare il rilevamento delle celle (Figura supplementare S10).
- Sulla pila di immagini per ogni posizione, disegna un poligono sulla cella normale più piccola. Questo viene utilizzato per calcolare l'area. Qualsiasi cosa più piccola di questa non sarà considerata come una cella dal software.
- Quindi disegna un poligono attorno alla cella normale più grande e indica se il centro della cella o l'interfaccia cella-cella è più luminosa.
NOTA: AnViM crea quindi i file 'phs/textResults/0001.csv', 'phs/textResults/0002.csv' , ecc., per ogni fotogramma contenente informazioni sulle celle all'interno di quel fotogramma. In questa fase, questo file include informazioni morfologiche sulle cellule, tra cui area, centroide, perimetro, orientamento, circolarità, proporzioni, arrotondamento, solidità, distanza dalla regione senza cellule. L'unità di lunghezza in queste proprietà è pixel e l'angolo è in gradi. Infine, questo file viene aggiornato per contenere l'intensità dei pixel cellulari, il movimento e le forze nei passaggi successivi.
- Parte 3: Mappatura delle intensità dei pixel sulle celle
NOTA: gli input manuali in questo passaggio includono l'identificazione della directory dei dati e la selezione delle proprietà e dei parametri di mapping. I principali file creati in precedenza utilizzati in questo passaggio includono 'p*/phs/*.tif' (o 'p*/fluo/*.tif ') e 'p*/phs/bwImages/*.tif'. I nuovi file chiave prodotti in questo passaggio includono 'p*/phs/textResults/*.csv', che elenca le proprietà morfologiche cellulari. Vedere la Figura 11 per la descrizione visiva dei passaggi seguenti per valutare le intensità dei pixel nella regione compresa tra le singole celle e la regione limitrofa e definirle come proprietà delle celle.- Avviare il software Fiji e dal menu a discesa MSM selezionare Mappa su celle - Intensità.
- Utilizzando la finestra di dialogo del browser dei file, selezionare la directory di posizione 'analysis/p0', 'analysis/p1', ecc., che contiene le proprietà quantificate su punti griglia regolarmente distanziati.
- Selezionare Rileva divisione cellulare o Quantifica fluorescenza cellulare quando richiesto dal software. Definire le dimensioni dell'area adiacente. Selezionare le caselle Raccogli proprietà dell'area limitrofa e Raccogli proprietà delle celle. Fare clic su OK.
- Nella finestra Scopo della registrazione dell'intensità cellulare, indicare il tipo di immagine da utilizzare per la mappatura dell'intensità, le dimensioni della regione vicina e se vengono raccolti dati per singole celle o per entrambe le celle e le regioni limitrofe (Figura supplementare S11).
NOTA: la mappatura delle intensità dei pixel di un'immagine a contrasto di fase consente di rilevare gli eventi di divisione cellulare. La mappatura delle intensità delle immagini fluorescenti consentirà il rilevamento delle fluttuazioni nelle molecole citoplasmatiche marcate fluorescentemente. L'output dei passaggi precedenti è il valore medio, mediano, deviazione standard, minimo e massimo dell'intensità dei pixel per le singole celle. Questi numeri vengono inseriti come nuove colonne nei file 'phs/textResults/0001.csv', 'phs/textResults/0002.csv' , ecc.
- Parte 4: Mappatura delle forze e del movimento sulle cellule
NOTA: gli input manuali per questo passaggio includono l'identificazione della directory dei dati e la selezione delle proprietà e dei parametri di mapping. I principali file creati in precedenza utilizzati in questo passaggio includono 'p*/phs/textResults/*.csv', 'p*/velocity/*_vel.dat', 'p*/displacement/*_disp.dat', 'p*/traction/*_trac.dat', 'p*/prestress/*_prestress.dat' e 'p*/phs/bwImages/*.tif '. Durante questo passaggio non vengono creati nuovi file. Invece, le nuove informazioni (forza cellulare e proprietà di movimento) vengono aggiunte ai file 'p*/phs/textResults/*.csv'. Vedere la Figura 12 per la descrizione visiva dei passaggi per valutare le forze e il movimento nella regione compresa tra le singole celle e la regione limitrofa e definirli come proprietà delle celle.- Scegliere dal menu a discesa MSM Mappa sulle celle - Dati di movimento/forza (Figura supplementare S12).
- Quindi, seguire i passaggi elencati nel passaggio 2.4.3.
NOTA: nuove colonne vengono aggiunte ai file 'phs/textResults/0001.csv', 'phs/textResults/0002.csv', ecc. e includono la deviazione media, mediana e standard della velocità, l'orientamento della velocità, la tensione citoscheletrica media, l'anisotropia della tensione, l'energia di deformazione nell'idrogel, l'orientamento della tensione più alta e l'entità della trazione cellulare-ECM (Figura supplementare S13).
- Parte 1: Segmentazione della regione dell'immagine che contiene le celle
- Visualizzazione dei risultati
- Parte 1: Monitoraggio delle identità cellulari attraverso l'esperimento
NOTA: gli input manuali a questo passaggio includono l'identificazione della directory dei dati e la selezione delle proprietà mappate da visualizzare. I file chiave creati in precedenza utilizzati in questo passaggio includono "p*/phs/textResults/*.csv". I nuovi file chiave prodotti durante questo passaggio includono "p*/phs/textResults/cellTimeTrace_*.csv", che contiene una traccia temporale dei dati cellulari. Vedere la Figura 13 per la descrizione visiva dei passaggi seguenti per tenere traccia delle proprietà delle singole celle per l'intera durata dell'esperimento.- Avviare il software Fiji e dal menu a discesa MSM selezionare Risultati - Traccia dati.
- Utilizzando la finestra di dialogo del browser dei file, selezionare la directory di posizione 'analysis/p0', 'analysis/p1', ecc., contenente le proprietà cellulari da tracciare. Al termine, fare clic su Seleziona.
- Nella finestra Scegli punto di partenza per il tracciamento , scegliere il fotogramma iniziale per il monitoraggio e il numero di fotogrammi tracciati contemporaneamente (Figura supplementare S14). Inizia sempre dal fotogramma numero 2 poiché la velocità non può essere determinata per il fotogramma numero 1. Al termine, fare clic su OK.
- Nella finestra Controlla le variabili per creare tracce , scegliere le variabili digitando i nomi delle variabili o selezionando i termini dalle caselle di controllo (Figura supplementare S15). Quindi, fare clic su OK.
NOTA: il rilevamento genera file 'phs/textResults/cellTimeTrace_*.csv' con ogni colonna contenente un numero di cella univoco e ogni riga successiva che rappresenta l'istanza temporale consecutiva.
- Parte 2: Generazione di tracce temporali delle proprietà cellulari valutate
NOTA: gli input manuali a questo passaggio includono l'identificazione della directory dei dati e la selezione delle proprietà mappate da visualizzare. I principali file creati in precedenza utilizzati in questo passaggio includono "p*/phs/textResults/cellTimeTrace_*.csv". I nuovi file chiave prodotti durante questo passaggio includono 'p*/phs/trackedDataPlots/*.tif', che contengono grafici di tracce temporali e 'p*/phs/trackedDataPlots/*.csv', che contengono dati di plottaggio. Vedere la Figura 14 per la descrizione visiva dei seguenti passaggi per generare tracce temporali delle proprietà cellulari valutate.- Avviare il software Fiji e dal menu a discesa MSM selezionare Risultati - grafico.
- Utilizzando la finestra di dialogo del browser dei file, selezionare la directory di posizione 'analysis/p0', 'analysis/p1', ecc., contenente le proprietà cellulari tracciate da tracciare.
- Scegliere se limitare il plottaggio alle celle con tracce ininterrotte facendo clic sul pulsante Sì o No .
Nota : questa opzione ignora le celle, che non è stato possibile rilevare in una o più istanze temporali. - Scegliete il numero di variabili da tracciare contemporaneamente e cliccate su OK.
NOTA: attualmente, AnViM consente di visualizzare un massimo di tre variabili nello stesso plottaggio. - Scegliete singole variabili per il plottaggio utilizzando un menu a discesa.
NOTA: questa procedura consente di creare un file TIFF (Tagged Image File Format) e un file di dati separato da virgole all'interno della cartella 'phs/trackedDataPlots/'. Il nome del file è composto da nomi di variabili separati da un carattere di sottolineatura e termina con un numero di cella.
- Parte 3: Generazione di mappe di calore delle proprietà cellulari valutate
NOTA: gli input manuali in questo passaggio includono l'identificazione della directory dei dati e la selezione delle proprietà mappate da visualizzare. I principali file creati in precedenza utilizzati in questo passaggio includono "p*/phs/textResults/cellTimeTrace_*.csv". I nuovi file chiave generati in questo passaggio includono 'p*/phs/segmentedImages/cellPropMaps/*.tif', che sono le mappe di calore delle proprietà cellulari. Vedere la Figura 15 per la descrizione visiva dei passaggi per generare mappe di calore delle proprietà cellulari valutate.- Scegliere dal menu a discesa MSM Risultati - Immagine.
- Seguire i passaggi descritti nel passaggio 2.5.2.
NOTA: l'output viene memorizzato come file TIFF nella cartella 'phs/segmentedImages/cellPropMaps/'. I nomi dei file sono il numero di istanza temporale e il nome della variabile separati da un carattere di sottolineatura.
- Parte 1: Monitoraggio delle identità cellulari attraverso l'esperimento
Risultati
Presentiamo qui due dei principali risultati per l'esempio dimostrato. La prima uscita è la traccia temporale della velocità cellulare e della tensione citoscheletrica per la cella numero 1 (Figura 16). Le proprietà vengono visualizzate su un asse verticale condiviso per facilitare l'associazione visiva tra le proprietà e l'asse orizzontale indica il numero di istanza temporale. In questo esperimento, sono stati acquisiti fotogrammi successivi a un intervallo di 15 minuti. Il secondo output è una serie di mappe di calore a 1 ora dall'inizio dell'esperimento (Figura 17). Le proprietà mostrate qui includono area di diffusione, orientamento, circolarità, velocità, direzione del movimento, orientamento alla massima tensione, tensione citoscheletrica, trazioni del substrato e anisotropia di tensione delle singole cellule.

Figura 1: Struttura del toolkit integrativo per l'analisi dei segnali cellulari (iTACS). Due componenti chiave di iTACS sono acquisition and training Module (AcTrM) e Analysis and Visualization Module (AnViM). AcTrM può utilizzare varie tecniche di preparazione dell'idrogel attualmente esistenti per preparare idrogel che possono essere tenuti saldamente su uno stadio di microscopio, qualsiasi semina cellulare e un protocollo di crescita che mantiene le cellule in un piano focale. AnViM può utilizzare varie tecniche per quantificare la deformazione idrogel e monostrato, le forze cellula-ECM e le forze cellula-cellula. Tutti questi componenti preferiti dall'utente del protocollo di misurazione della forza possono essere ospitati in iTACS e sono stati identificati con caselle tratteggiate. I componenti identificati con scatole solide sono nuovi contributi alla tecnologia di misurazione della forza cellulare. La visualizzazione in AnViM si concentra sul valore mediano e sulla variabilità delle proprietà tra le singole celle. Fare clic qui per visualizzare una versione più grande di questa figura.
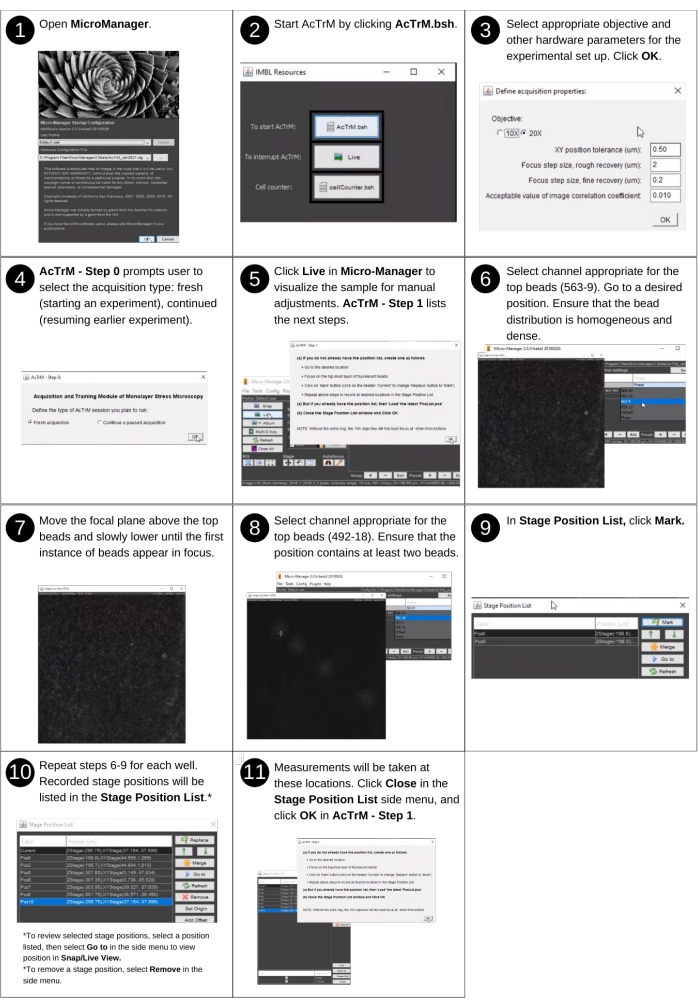
Figura 2: Acquisizione di immagini di riferimento - parte 1. Passaggi per la creazione di un elenco di posizioni utilizzando AcTrM. Fare clic qui per visualizzare una versione più grande di questa figura.
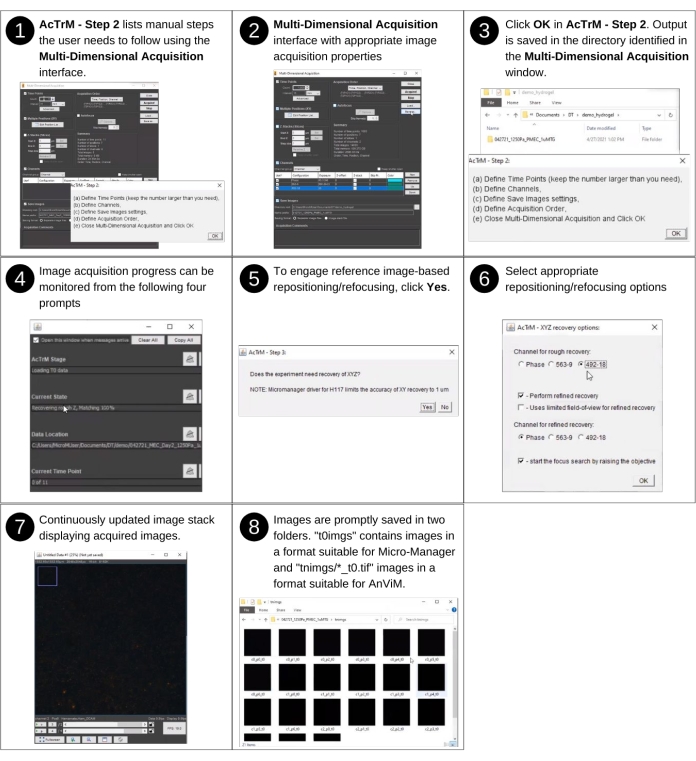
Figura 3: Acquisizione di immagini di riferimento - parte 2. Passaggi per l'acquisizione di immagini di riferimento utilizzando AcTrM. Le viste dettagliate dei passaggi 2, 4 e 6 sono presentate rispettivamente nelle figure supplementari S2, S3 e S4. Fare clic qui per visualizzare una versione più grande di questa figura.
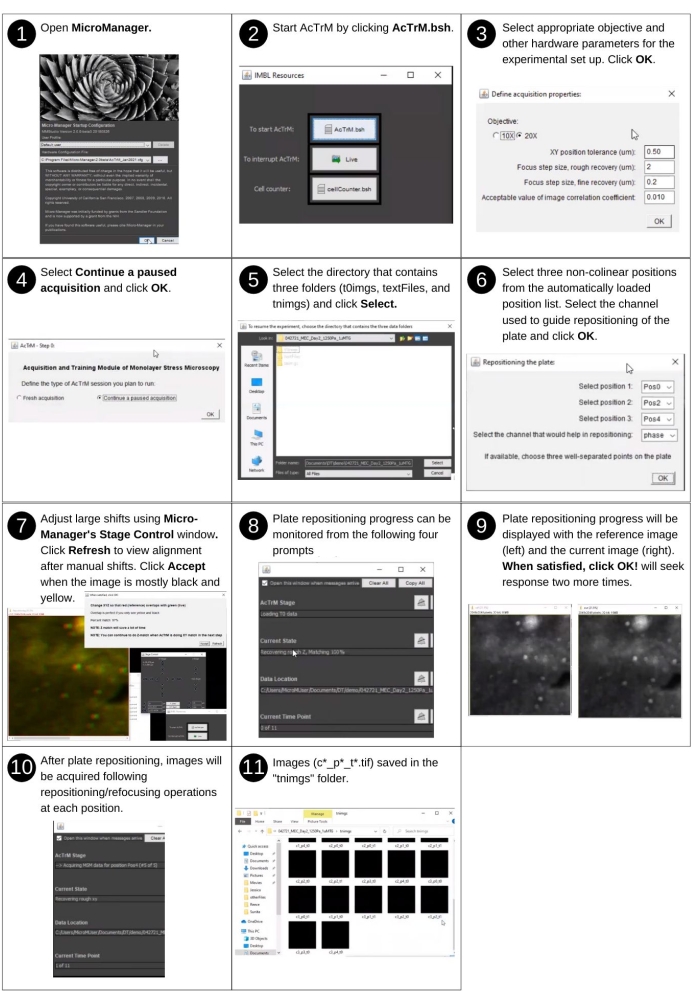
Figura 4: Acquisizione automatica delle immagini per l'esperimento rimanente. Passaggi per riprendere l'acquisizione dell'immagine per valutare il comportamento cellulare utilizzando AcTrM. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 5: Impostazione dell'analisi automatizzata dei dati. Passaggi per iniziare l'analisi automatica delle immagini utilizzando AnViM. Il software riconosce il formato di immagine utilizzato da AcTrM. Una vista dettagliata dei pannelli nei passaggi 3 e 5 è presentata rispettivamente nella figura supplementare S5 e nella figura supplementare S6. Fare clic qui per visualizzare una versione più grande di questa figura.
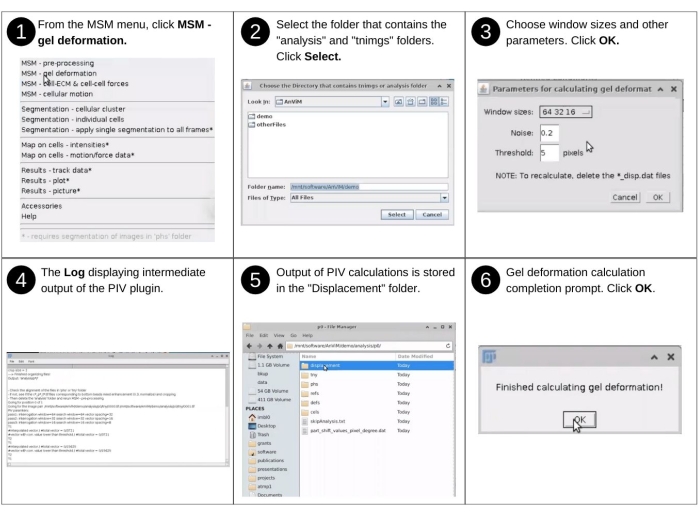
Figura 6: Quantificazione della deformazione di idrogel e monostrato - parte 1. Passaggi per coinvolgere, tramite AnViM, l'implementazione della particle image velocimetry di Tseng, Q. et al., PNAS (2012)20 per quantificare la deformazione della superficie superiore dell'idrogel. Gli utenti possono anche implementare all'interno di AnViM altri approcci per quantificare la deformazione dell'idrogel. Una vista dettagliata del passaggio 3 è presentata nella figura supplementare S7. Fare clic qui per visualizzare una versione più grande di questa figura.
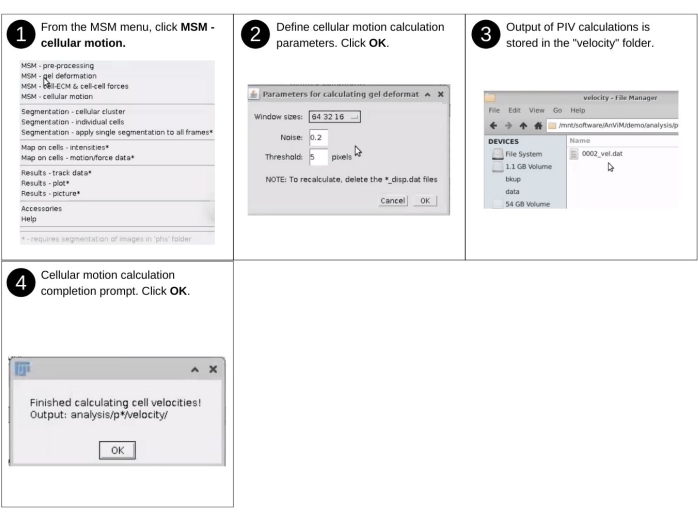
Figura 7: Quantificazione della deformazione di idrogel e monostrato - parte 2. Passi per coinvolgere, tramite AnViM, l'implementazione della particle image velocimetry di Tseng, Q. et al., PNAS (2012)20 per quantificare il movimento locale delle singole cellule. Gli utenti possono anche implementare all'interno di AnViM altri approcci per quantificare il movimento cellulare. Fare clic qui per visualizzare una versione più grande di questa figura.
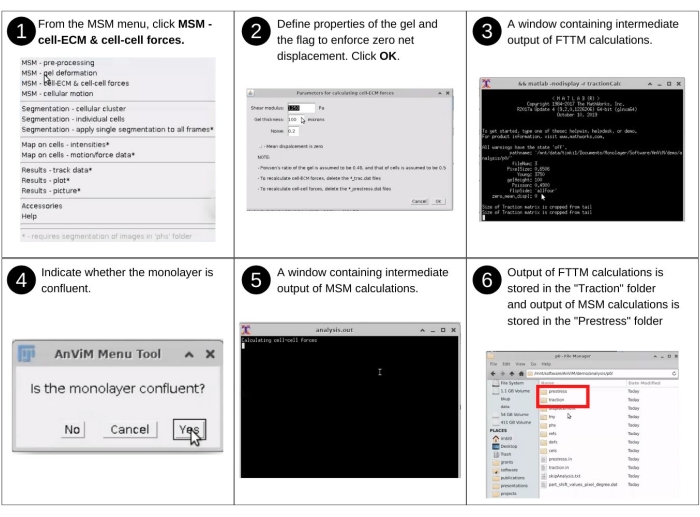
Figura 8: Quantificazione delle forze cellula-ECM e cellula-cellula. Passi per eseguire l'analisi delle immagini per coinvolgere, tramite AnViM, l'implementazione della microscopia di trazione a trasformata di Fourier di Trepat et al., Nature Physics (2009)15 per quantificare le forze esercitate dalle cellule sull'idrogel e l'implementazione della microscopia da sforzo monostrato di Tambe et al., Nature Materials (2011)1 per quantificare le forze all'interno delle singole cellule e tra cellule vicine. Gli utenti possono anche implementare all'interno di AnViM altri approcci per quantificare le forze cellula-ECM e cellula-cellula. Una vista dettagliata del passaggio 6 è presentata nella figura supplementare S8 e nella figura supplementare S9. Fare clic qui per visualizzare una versione più grande di questa figura.
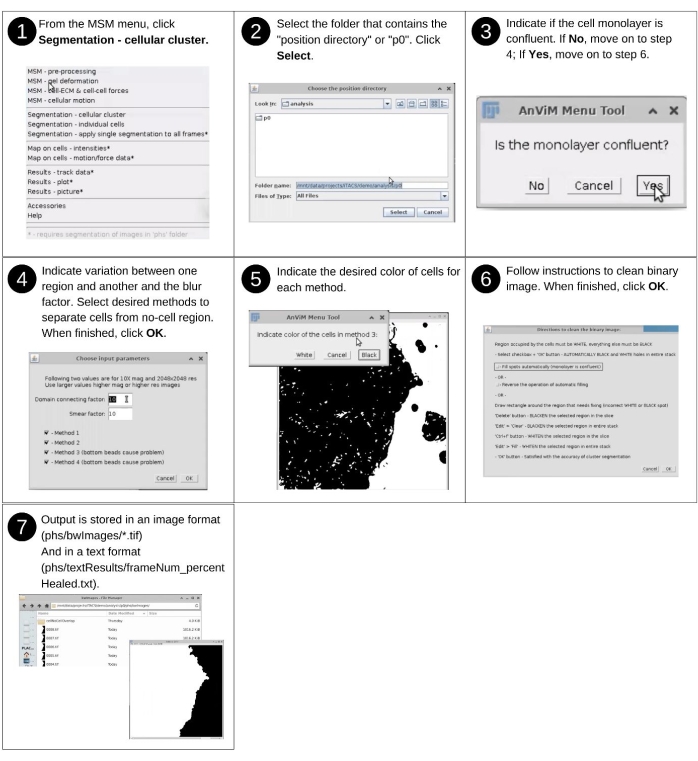
Figura 9: Mappatura dei valori dei punti della griglia su singole celle - parte 1. Passaggi per segmentare le regioni dell'immagine contenenti celle utilizzando un nuovo approccio su più fronti. Questo approccio può essere utilizzato per segmentare il contrasto di fase, il campo luminoso o le immagini a fluorescenza delle cellule. Fare clic qui per visualizzare una versione più grande di questa figura.
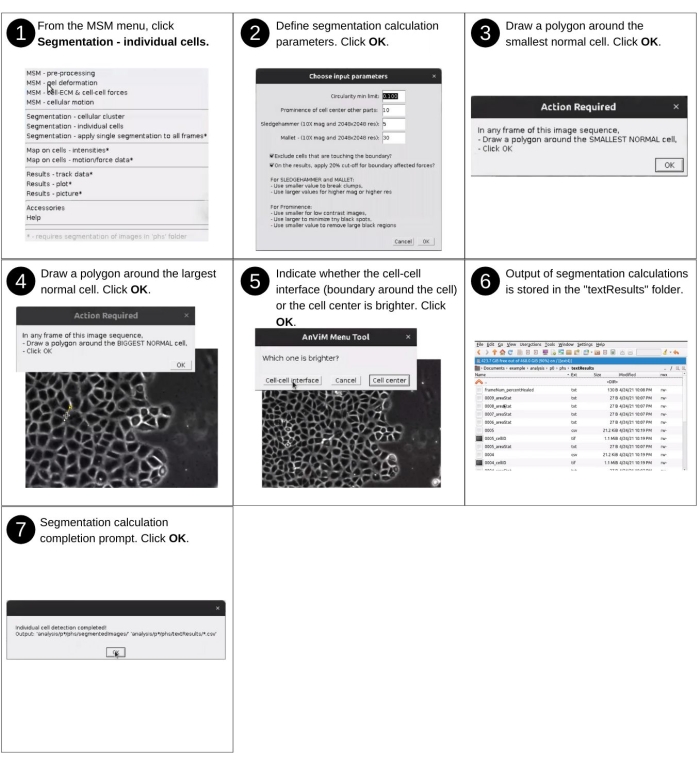
Figura 10: Mappatura dei valori dei punti della griglia su singole celle - parte 2. Passaggi per segmentare singole cellule di un monostrato utilizzando un nuovo approccio su più fronti sviluppato in AnViM. Questo approccio può essere utilizzato per segmentare il contrasto di fase, il campo luminoso o le immagini a fluorescenza delle cellule. Una vista dettagliata del passaggio 2 è presentata nella figura supplementare S10. Fare clic qui per visualizzare una versione più grande di questa figura.
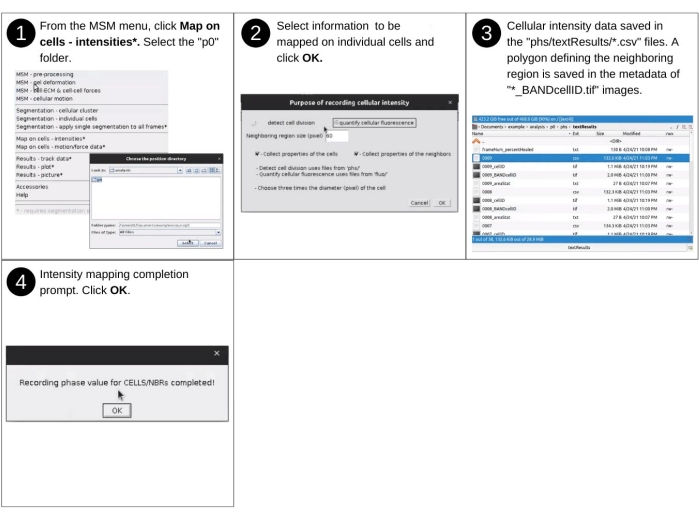
Figura 11: Mappatura dei valori dei punti della griglia su singole celle - parte 3. Passaggi per valutare le intensità dei pixel nella regione all'interno delle singole celle e all'interno della regione vicina delle singole celle utilizzando AnViM. Le intensità valutate includono l'intensità della luce trasmessa e l'intensità della fluorescenza. Questa parte mappa il valore mediano e la deviazione standard delle intensità dei pixel all'interno delle singole celle e all'interno di una regione vicina di singole celle. Una vista dettagliata del passaggio 2 è presentata nella figura supplementare S11. Fare clic qui per visualizzare una versione più grande di questa figura.
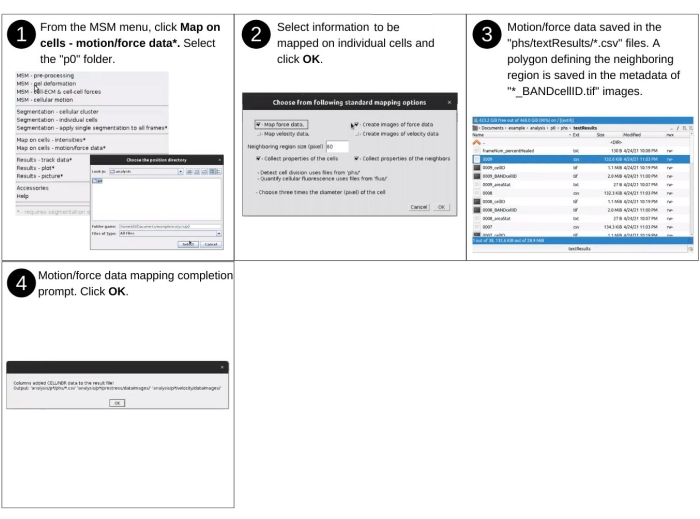
Figura 12: Mappatura dei valori dei punti della griglia su singole celle - parte 4. Passaggi per valutare le forze e le proprietà di movimento dei punti della griglia all'interno delle singole celle e all'interno della regione vicina delle singole celle utilizzando AnViM. Questa parte mappa il valore mediano e la deviazione standard delle proprietà all'interno delle singole celle e all'interno di una regione vicina di singole celle. Una visione dettagliata dei passaggi 2 e 3 è presentata nella figura supplementare S12 e nella figura supplementare S13. Fare clic qui per visualizzare una versione più grande di questa figura.
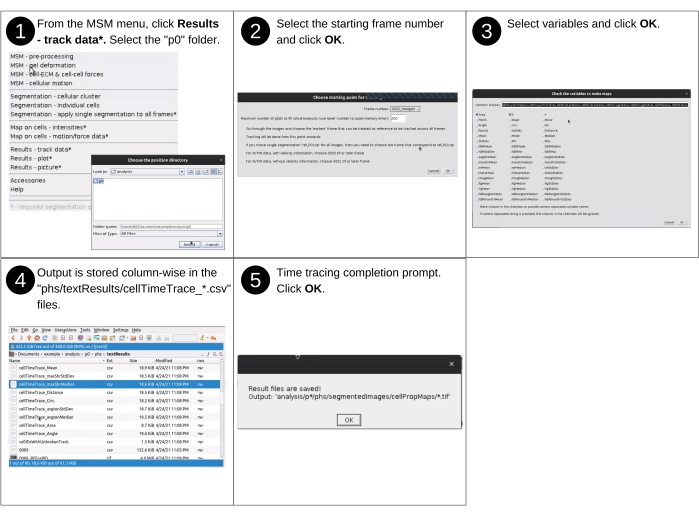
Figura 13: Visualizzazione dei risultati - parte 1. Passaggi per tenere traccia delle proprietà delle singole celle per l'intera durata dell'esperimento utilizzando AnViM. Una vista dettagliata dei passaggi 2 e 3 è presentata nella figura supplementare S14 e nella figura supplementare S15. Fare clic qui per visualizzare una versione più grande di questa figura.
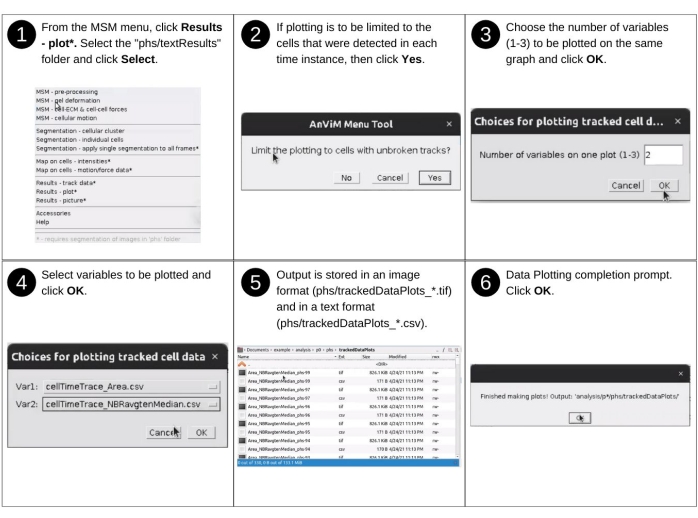
Figura 14: Visualizzazione dei risultati - parte 2. Passaggi per generare tracce temporali delle proprietà valutate utilizzando AnViM. L'utente ha la possibilità di tracciare fino a tre proprietà in un grafico. Le tracce temporali vengono generate per tutte le cellule o solo per quelle per le quali il monitoraggio ha avuto successo nell'intero esperimento. Una vista dettagliata della fase 5 è presentata nella figura supplementare S16 e nella figura supplementare S17. Fare clic qui per visualizzare una versione più grande di questa figura.
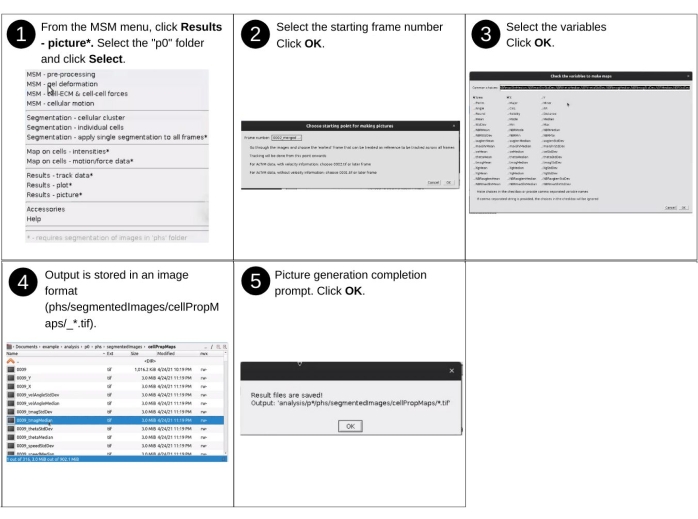
Figura 15: Visualizzazione dei risultati - parte 3. Passaggi per generare mappe di calore delle proprietà valutate utilizzando AnViM. Le mappe di calore vengono generate per tutti i fotogrammi che seguono il fotogramma iniziale e tutte le proprietà selezionate. Una vista dettagliata del passaggio 3 è presentata nella Figura S18 supplementare. Fare clic qui per visualizzare una versione più grande di questa figura.
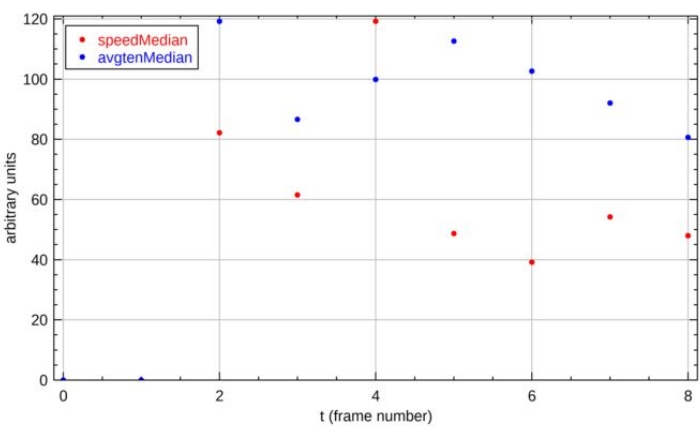
Figura 16: Tracce temporali per l'ID cella 1. Due proprietà visualizzate sono la tensione citoscheletrica cellulare ("avgtenMedian") e la velocità cellulare ("speedMedian"). Sia la tensione citoscheletrica cellulare che la velocità cellulare sono quantificate come valore mediano attraverso i punti della griglia all'interno delle cellule. Le due proprietà vengono tracciate sullo stesso asse con unità arbitrarie per visualizzare le relazioni tra le proprietà valutate. Ulteriori nomi di variabili sono elencati nella tabella supplementare S1. Fare clic qui per visualizzare una versione più grande di questa figura.
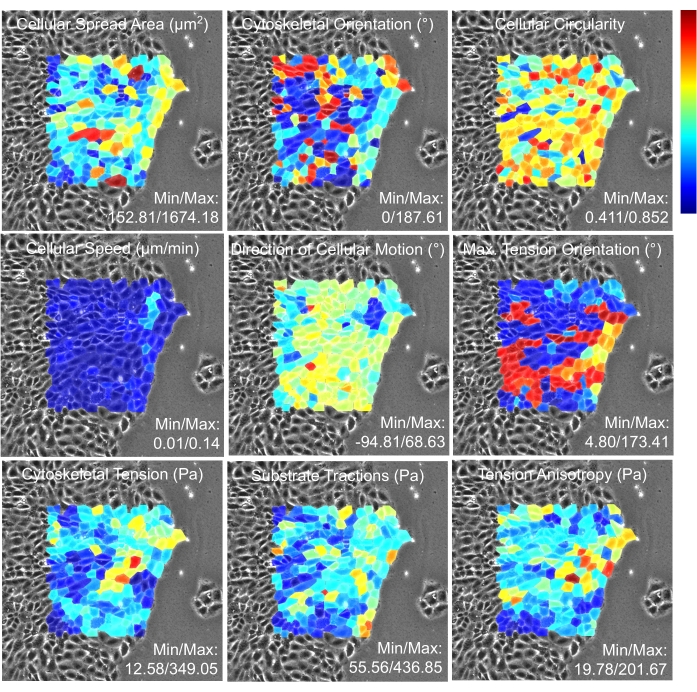
Figura 17: Mappe termiche delle proprietà delle singole celle attraverso il monostrato analizzato. Ogni cella è colorata con il valore mediano della proprietà indicato nel pannello. Pertanto, il rosso intenso indica il valore cellulare massimo nello spettro dei colori e il blu profondo indica il valore cellulare minimo attraverso il monostrato analizzato. Come descritto in Tambe et al., PLoS One (2013)2, le cellule situate più vicino al confine hanno forze meccaniche influenzate da proprietà sconosciute delle cellule al di fuori dell'immagine. Quindi la mappa di calore viene generata per le cellule lontane dal confine. Fare clic qui per visualizzare una versione più grande di questa figura.
Figura S1: Immagini di esempio del tallone fluorescente superiore e inferiore. Fare clic qui per scaricare questo file.
Figura S2: Una vista dettagliata del passaggio 2 dalla Figura 3. Fare clic qui per scaricare questo file.
Figura S3: Una vista dettagliata del passaggio 4 dalla Figura 3. Fare clic qui per scaricare questo file.
Figura S4 A: visualizzazione dettagliata del passaggio 6 dalla Figura 3. Fare clic qui per scaricare questo file.
Figura S5: Una vista dettagliata del passaggio 3 dalla Figura 5. Fare clic qui per scaricare questo file.
Figura S6: Una vista dettagliata del passaggio 5 dalla Figura 5. Fare clic qui per scaricare questo file.
Figura S7: Una vista dettagliata del passaggio 3 dalla Figura 6. Fare clic qui per scaricare questo file.
Figura S8: Una vista dettagliata dell'output di forza della cella-ECM del passaggio 6 dalla Figura 8. Fare clic qui per scaricare questo file.
Figura S9: Una vista dettagliata dell'output di forza cella-cella del passaggio 6 dalla Figura 8. Fare clic qui per scaricare questo file.
Figura S10: Una vista dettagliata del passaggio 2 dalla Figura 10. Fare clic qui per scaricare questo file.
Figura S11: Una vista dettagliata del passaggio 2 dalla Figura 11. Fare clic qui per scaricare questo file.
Figura S12: Una vista dettagliata del passaggio 2 dalla Figura 12. Fare clic qui per scaricare questo file.
Figura S13: Una vista dettagliata dell'output del passaggio 3 dalla Figura 12. Fare clic qui per scaricare questo file.
Figura S14: Una vista dettagliata del passaggio 2 nella Figura 13. Fare clic qui per scaricare questo file.
Figura S15: Una vista dettagliata del passaggio 3 dalla Figura 13. Fare clic qui per scaricare questo file.
Figura S16: Una visualizzazione dettagliata dei file di dati generati nel passaggio 5 della Figura 14. Fare clic qui per scaricare questo file.
Figura S17: Una vista dettagliata di un grafico generato nel passaggio 5 della Figura 14. Fare clic qui per scaricare questo file.
Figura S18: Una vista dettagliata di una mappa termica e del file contenente l'intervallo dello spettro di colori generato nel passaggio 4 della Figura 15. Fare clic qui per scaricare questo file.
Tabella S1: Un elenco di proprietà selezionate quantificate da iTACS. Fare clic qui per scaricare questa tabella.
Discussione
Le cellule aderenti utilizzano segnali sia meccanici che chimici per sopravvivere, crescere e funzionare. Un'ampia varietà di software di microscopia ottimizza l'esperienza dell'utente nella valutazione dei segnali chimici attraverso l'imaging basato sulla fluorescenza. Tuttavia, la valutazione dei segnali meccanici coinvolge funzionalità che non sono disponibili nel software di microscopia standard. Inoltre, la valutazione dei segnali meccanici è più efficiente quando l'acquisizione dei dati è integrata con l'analisi dei dati. La mancanza di una piattaforma unificata che soddisfi le esigenze uniche della valutazione del segnale meccanico è stata una grande lacuna tecnologica nella biologia cellulare sperimentale. L'Integrative Toolkit to Analyze Cellular Signals (iTACS) è progettato per colmare questa lacuna. I due componenti di iTACS, AnViM e AcTrM, forniscono agli utenti le capacità necessarie per quantificare le proprietà cellulari di quattro grandi categorie: forze, movimento, morfologia e fluorescenza / luminosità. In queste categorie, iTACS è attualmente in grado di rivelare più di 50 aspetti unici delle singole cellule aderenti. Questi aspetti comprendono proprietà specifiche di ogni ampia categoria, compreso il loro valore rappresentativo e la variabilità attraverso la cella (Tabella supplementare S1). Ad esempio, all'interno delle forze, ci sono forze di trazione attraverso il citoscheletro, l'anisotropia di questa tensione, l'orientamento della massima tensione e lo stress di taglio attraverso l'interfaccia cellula-ECM che ha una profonda influenza sul comportamento delle cellule aderenti1,3,6.
Un nuovo approccio per esaminare il comportamento meccanico delle singole cellule di un monostrato
Le singole cellule di un monostrato sono impegnate in uno scambio di segnali di natura chimica e meccanica3. Questi due tipi di segnali vengono trasmessi attraverso il monostrato cellulare in modo diverso23. Tuttavia, la conoscenza della trasmissione meccanica del segnale è in ritardo rispetto a quella della trasmissione del segnale chimico. Questa lacuna di conoscenza coincide con una prolungata mancanza di approcci semplici e intuitivi per valutare i segnali meccanici cellulari. Il nuovo approccio di mappatura dei dati qui descritto è attrezzato per colmare questa lacuna. Tale mappatura rivela che la fluttuazione della tensione citoscheletrica intrinseca nella regione vicina di una cellula funge da segnali di rilassamento, fluidizzazione e ancoraggio che regolano i cambiamenti nella forma, nelle dimensioni e nella velocità cellulare della cellula18. Le mappe delle proprietà delle regioni vicine mostrano modelli di "suddivisione multicellulare" in cui le cellule all'interno della suddivisione sono esposte a un microambiente relativamente uniforme e le cellule al confine della suddivisione sono esposte a un microambiente notevolmente non uniforme18.
Accessibilità della tecnologia di misurazione della forza
Esiste una varietà di protocolli per produrre idrogel PAA, analizzare la deformazione dell'idrogel e il movimento cellulare e quantificare le forze cellula-ECM e cellula-cellula1,2,7,8,9,13,14,15,18,20,21,24,25,26,27 ,28,29,30,31,32. Tuttavia, questi sviluppi rimangono fuori dalla portata dei comuni laboratori di biologia cellulare e confinati a laboratori con esperienza ingegneristica. Automatizzando gli aspetti tecnici di questi approcci e integrandoli in una piattaforma unificata e user-friendly, l'obiettivo di iTACS è quello di rendere la valutazione dei segnali meccanici un'attività di routine nella ricerca e nell'educazione sperimentale di biologia cellulare.
ImageJ consente agli utenti di sviluppare applicazioni utilizzando approcci che richiederebbero poca o nessuna formazione11. iTACS è in gran parte costruito utilizzando semplici approcci di scripting per facilitare lo sviluppo continuo guidato dalla comunità. La maggior parte di AcTrM viene programmata utilizzando script BeanShell e la maggior parte di AnViM viene programmata utilizzando macro ImageJ. Questi script e le linee guida per l'implementazione di queste funzionalità sul microscopio dell'utente sono disponibili tramite GitHub (https://github.com/IntegrativeMechanobiologyLaboratory/iTACS).
Standardizzazione qualitativa delle immagini acquisite
Sebbene le tecniche basate sul substrato elastico per quantificare le forze fisiche nelle cellule aderenti siano state sviluppate e implementate in vari laboratori, il protocollo manca ancora di standardizzazione. Un'area che necessita maggiormente di standardizzazione è la qualità delle immagini di perline superiori acquisite (Figura supplementare S1). Problemi significativi sorgono dalla deriva a fuoco durante l'esperimento. Il nostro nuovo approccio di rifocalizzazione basato su immagini di riferimento rende tale messa a fuoco un processo oggettivo. I parametri definiti nella primissima fase di AcTrM impongono i necessari limiti oggettivi di qualità. Altre misure di standardizzazione possono essere programmate nelle versioni future di AcTrM.
L'ampia applicabilità di iTACS
Oltre a quantificare numerosi aspetti delle cellule aderenti, la struttura iTACS ne facilita l'uso per vari protocolli sperimentali e necessità. AcTrM consente l'auto-formazione dell'utente guidata dal software. L'imaging ad alta velocità richiesto, ad esempio, dalla valutazione simultanea delle fluttuazioni citoplasmatiche del calcio è attualmente limitato dalla velocità di riposizionamento e rifocalizzazione dell'hardware ed è meglio farlo in una posizione alla volta. Tuttavia, l'attuale implementazione è ben attrezzata per l'imaging a lungo termine, l'imaging interrotto, in cui il campione non può essere conservato sullo stadio del microscopio per l'intera durata dell'esperimento. Poiché le immagini di riferimento vengono acquisite all'inizio dell'esperimento, iTACS consente l'imaging in tempo reale dei segnali meccanici, aprendo nuove strade nelle applicazioni di screening dei farmaci. AnViM consente agli utenti di fornire informazioni altamente tecniche in termini profani. La capacità di quantificare un ampio spettro di proprietà cellulari e di seguirle durante l'esperimento costituisce capacità critiche necessarie per scoprire nuovi meccanismi di comunicazione intercellulare.
Per lo sviluppo futuro di iTACS, abbiamo identificato quattro aree di interesse: (1) miglioramento della velocità di acquisizione e analisi dei dati, (2) implementazione di approcci per valutare nuovi segnali cellulari13, (3) sviluppo di workshop e moduli educativi sulla valutazione del segnale cellulare basata su iTACS, (4) sviluppo di soluzioni di automazione a basso costo.
Divulgazioni
Gli autori non dichiarano conflitti di interesse.
Riconoscimenti
D.T.T. ringrazia il personale affiliato al Center for Lung Biology della University of South Alabama per aver stimolato le discussioni sulle esigenze di ricerca sperimentale di biologia cellulare. Queste discussioni sono state cruciali per avviare lo sviluppo di iTACS.
Questo lavoro è stato supportato in parte da sovvenzioni del National Institute of Health / National Heart Lung Blood Institute, P01 HL66299 e R37 HL60024 (Stevens), R01-HL118334 (Alvarez), F32-HL144040-01 (Xu), e dalla University of South Alabama attraverso Abraham Mitchell Cancer Research Fund (Singh, Palanki, Tambe), Research and Scholarly Development Grant (Tambe), Honors College e Summer Undergraduate Research Fellow (Nguyen).
Materiali
| Name | Company | Catalog Number | Comments |
| Reagents and components used in prepare glass surface for hydrogel coating | |||
| (3-Aminopropyl)trimethoxysilane, 97% | Aldrich chemistry | 13822565 | |
| 2% Bis Solution | Bio-rad | 1610142 | |
| 3-(Trimethoxysilyl)propyl methacrylate,98% | Acros organics | 2530850 | |
| 40% Acrylamide Solution | Bio-rad | 1610140 | |
| Glass bottom 35 mm dish/ 6 or 12 or 24 well plates | MatTek or CellVis | ||
| Glutaraldehyde, EM Grade, 25% | Polysciences | 1909100 | |
| Sodium Hydroxide | Sigma-aldrich | 1002074706 | |
| Reagents and components used in preparing suitable hydrogel | |||
| 2% Bis Solution | Bio-rad | 1610142 | |
| 40% Acrylamide Solution | Bio-rad | 1610140 | |
| Ammonium Persulfate | Bio-rad | 1610700 | |
| Cover Slips | Electron Microscopy Sciences | 7222301 | |
| Dulbecco's Phosphate Buffered Saline (1M) | Gibco | 14190136 | |
| FluoSpheres carboxylate 0.2 um, yellow-green(505/515) | Invitrogen | F8811 | |
| FluoSpheres carboxylate 0.5 um, red(580/605) | Invitrogen | F8812 | |
| FluoSpheres carboxylate 2.0 um, red(580/605) | Invitrogen | F8826 | |
| Rain-X | |||
| TEMED | Bio-rad | 1610801 | |
| Reagents used in coating extracellular matrix on the hydrogel | |||
| Collagen Type I Rat Tail | Corning | 354236 | |
| HEPES(1M) | Gibco | 15630080 | |
| Phosphate Buffered Saline (1M) | Gibco | 10010023 | |
| Sulfo-SANPAH | CovaChem | 102568434 | |
| Microscope hardware used in the current study | |||
| Camera | Hamamatsu Flash 4.0 LT sCMOS Camera | C11440-42U | |
| H117 ProScanTM Stages | Prior Scientific | ||
| Light source- Lambda DG4 and Lambda DG5 | Sutter instrument company | ||
| Microscope | Nikon eclipse TE2000-S | 550372 | |
| ProScan III Universal Microscope Automation Controller | Prior Scientific | ||
| Stagetop incubator | ibidi | 11922 | |
| Stepper Motor Focus Drive | Prior Scientific |
Riferimenti
- Tambe, D. T., et al. Collective cell guidance by cooperative intercellular forces. Nature Materials. 10 (6), 469-475 (2011).
- Tambe, D. T., et al. Monolayer stress microscopy: limitations, artifacts, and accuracy of recovered intercellular stresses. PloS One. 8 (2), 55172 (2013).
- Das, T., et al. A molecular mechanotransduction pathway regulates collective migration of epithelial cells. Nature Cell Biology. 17 (3), 276-287 (2015).
- Vedula, S. R., et al. Mechanics of epithelial closure over non-adherent environments. Nature Communications. 6, 6111 (2015).
- Lamason, R. L., et al. Rickettsia Sca4 reduces vinculin-mediated intercellular tension to promote spread. Cell. 167 (3), 670-683 (2016).
- Sunyer, R., et al. Collective cell durotaxis emerges from long-range intercellular force transmission. Science. 353 (6304), 1157-1161 (2016).
- Dong, L., Oberai, A. A. Recovery of cellular traction in three-dimensional nonlinear hyperelastic matrices. Computer Methods in Applied Mechanics and Engineering. 314, 296-313 (2017).
- Nier, V., et al. Kalman inversion stress microscopy. Biophysical Journal. 115 (9), 1808-1816 (2018).
- Serrano, R., et al. Three-dimensional Monolayer Stress Microscopy. Biophysical Journal. 117 (1), 111-128 (2019).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ: 25 years of Image Analysis. Nature Methods. 9 (7), 671-675 (2012).
- Schindelin, J., et al. Fiji - an Open Source platform for biological image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Edelstein, A., Amodaj, N., Hoover, K., Vale, R., Stuurman, N. Computer control of microscopes using microManager. Current Protocols in Molecular Biology. , 20 (2010).
- Patel, N. G., et al. Unleashing shear: Role of intercellular traction and cellular moments in collective cell migration. Biochemical and Biophysical Research Communications. 522 (2), 279-285 (2020).
- Yeung, T., et al. Effects of substrate stiffness on cell morphology, cytoskeletal structure, and adhesion. Cell Motility and the Cytoskeleton. 60 (1), 24-34 (2005).
- Trepat, X., et al. Physical forces during collective cell migration. Nature Physics. 5, 426-430 (2009).
- Aratyn-Schaus, Y., Oakes, P. W., Stricker, J., Winter, S. P., Gardel, M. L. Preparation of complaint matrices for quantifying cellular contraction. Journal of Visualized Experiments: JoVE. (46), (2010).
- King, J., et al. Structural and functional characteristics of lung macro- and microvascular endothelial cell phenotypes. Microvascular Research. 67 (2), 139-151 (2004).
- Patel, G., et al. Mechanical signaling in a pulmonary microvascular endothelial cell monolayer. Biochemical and Biophysical Research Communications. 519 (2), 337-343 (2019).
- Kähler, C. J., et al. Main results of the 4th International PIV Challenge. Experiments in Fluids. 57 (6), 97 (2016).
- Tseng, Q., et al. Spatial organization of the extracellular matrix regulates cell-cell junction positioning. Proceedings of the National Academy of Sciences of the United States of America. 109 (5), 1506-1511 (2012).
- Alvarez-Gonzalez, B., et al. Two-layer elastographic 3-D traction force microscopy. Scientific Reports. 7, 39315 (2017).
- Makarchuk, S., Beyer, N., Gaiddon, C., Grange, W., Hebraud, P. Holographic traction force microscopy. Scientific Reports. 8 (1), 3038 (2018).
- Janmey, P. A., Miller, R. T. Mechanisms of mechanical signaling in development and disease. Journal of Cell Science. 124 (1), 9-18 (2011).
- Dembo, M., Oliver, T., Ishihara, A., Jacobson, K. Imaging the traction stresses exerted by locomoting cells with the elastic substratum method. Biophysical Journal. 70 (4), 2008-2022 (1996).
- Dembo, M., Wang, Y. L. Stresses at the cell-to-substrate interface during locomotion of fibroblasts. Biophysical Journal. 76 (4), 2307-2316 (1999).
- Butler, J. P., Tolic-Norrelykke, I. M., Fabry, B., Fredberg, J. J. Traction fields, moments, and strain energy that cells exert on their surroundings. American Journal of Physiology: Cell Physiology. 282 (3), 595-605 (2002).
- Saez, A., et al. Traction forces exerted by epithelial cell sheets. Journal of Physics. Condensed Matter. 22 (19), 194119 (2010).
- Deforet, M., et al. Automated velocity mapping of migrating cell populations (AVeMap). Nature Methods. 9 (11), 1081-1083 (2012).
- Polio, S. R., Smith, M. L. Patterned hydrogels for simplified measurement of cell traction forces. Methods in Cell Biology. 121, 17-31 (2014).
- Toyjanova, J., et al. 3D Viscoelastic traction force microscopy. Soft Matter. 10 (40), 8095-8106 (2014).
- Serra-Picamal, X., Conte, V., Sunyer, R., Munoz, J. J., Trepat, X. Mapping forces and kinematics during collective cell migration. Methods in Cell Biology. 125, 309-330 (2015).
- Charrier, E. E., et al. A novel method to make viscoelastic polyacrylamide gels for cell culture and traction force microscopy. APL Bioengineering. 4 (3), 036104 (2020).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon