Method Article
Kit de ferramentas integrativas para analisar sinais celulares: forças, movimento, morfologia e fluorescência
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
A plataforma Integrative Toolkit to Analyze Cellular Signals (iTACS) automatiza o processo de medição simultânea de uma grande variedade de sinais químicos e mecânicos em células aderentes. O iTACS foi projetado para facilitar o desenvolvimento orientado pela comunidade e permitir que os pesquisadores usem todos os recursos da plataforma, independentemente de sua formação educacional.
Resumo
A avaliação quantitativa das forças celulares e do movimento avançou consideravelmente nas últimas quatro décadas. Esses avanços forneceram a estrutura para examinar processos cricáveis de sinalização mecânica em sistemas de cultura celular. No entanto, o campo enfrenta atualmente três problemas: falta de padronização de qualidade dos dados adquiridos, erros técnicos na análise e visualização de dados, e talvez o mais importante, a tecnologia permanece em grande parte fora do alcance dos laboratórios comuns de biologia celular. Para superar essas limitações, desenvolvemos uma nova plataforma experimental - Kit de Ferramentas Integrativas para Análise de Sinais Celulares (iTACS). O iTACS consiste em dois componentes: Módulo de Aquisição e Treinamento (AcTrM) e Módulo de Análise e Visualização (AnViM). O AcTrM é baseado no μManager - um software de controle de microscópio baseado em NIH-ImageJ - e facilita o auto-treinamento e a automação do usuário de protocolos comuns de aquisição de imagens. O AnViM é baseado no NIH-ImageJ e facilita a automação amigável da análise de dados e visualização criciosa de resultados. Esses experimentos envolvem acultura de células aderentes em hidrogéis, marcadores fiduciais de imagem incorporados no hidrogel, e finalmente extrair dessas imagens uma caracterização mecânica abrangente das células. Atualmente, o iTACS permite que o usuário analise e rastreie uma ampla gama de propriedades, incluindo morfologia, movimento, forças citoesqueletal e fluorescência de células individuais e região vizinha. A questão da padronização da qualidade foi abordada no AcTrM com uma técnica de refoco guiado por imagem de referência. As questões técnicas na análise de dados foram abordadas no AnViM com um procedimento de segmentação de imagens multifacetado, uma abordagem fácil de usar para identificar condições de fronteira e uma nova visualização de dados baseados em propriedades celulares. O AcTrM foi projetado para facilitar a transformação direta de microscópios básicos de fluorescência em plataformas experimentais de mecânica celular, e o AnViM está equipado para permitir que os usuários meçam sinais mecânicos celulares sem exigir um fundo de engenharia. O iTACS estará disponível para a comunidade de pesquisa como um conjunto de código aberto com recursos de desenvolvimento orientados para a comunidade.
Introdução
Ferramentas de análise óptica e análise de dados comumente usadas empregam tecnologias de hardware e software quase antiquadas. A defasagem na tradução e implementação de avanços em dispositivos eletrônicos, abordagens computacionais e análise matemática em ferramentas comuns de biologia celular experimental é uma grande restrição no ritmo de crescimento em nosso conhecimento da fisiologia celular. Atualmente, pesquisadores de biologia celular encontram ferramentas de biologia molecular ao alcance, mas ferramentas baseadas em princípios de engenharia estão fora de alcance. Uma dessas ferramentas baseadas em princípios de engenharia é a Monolayer Stress Microscopy (MSM)1,2. Embora a MSM tenha sido adaptada e estudada em diversos laboratórios em todo o mundo, seu uso está confinado principalmente a laboratórios com expertise em engenharia3,4,5,6,7,8,9.
O NIH-ImageJ é uma das ferramentas de código aberto mais populares entre os pesquisadores de biologia celular10. Os avanços orientados pela contribuição da comunidade de usuários têm sido centrais para sua popularidade11,12. O ImageJ possui recursos que permitem que os usuários desenvolvam aplicativos com uma mistura de uma linguagem de programação avançada e abordagens simplificadas de script. Esses recursos facilitam os usuários com conhecimento básico de programação para implementar, adaptar e avançar qualquer nova contribuição para o software. Com base nessas qualidades do NIH-ImageJ, desenvolvemos o Integrative Toolkit para Analisar sinais celulares (iTACS), que permite uma integração de baixo custo das ferramentas de hardware e software desejadas para automatizar a medição de uma grande variedade de sinais químicos e mecânicos entre células aderentes11,12.
O iTACS é composto por dois componentes: Módulo de Aquisição e Treinamento (AcTrM) e Módulo de Análise e Visualização (AnViM). O AcTrM é construído no μManager - um aplicativo de aquisição de imagem baseado em NIH-ImageJ - para permitir que os usuários configurem medições de lapso de tempo de propriedades ópticas tradicionais e uma variedade de propriedades físicas de células aderentes em várias amostras12. O AcTrM facilita o treinamento do usuário através de direções concisas incluídas na interface gráfica. Além disso, possui um novo recurso de autofoco baseado em imagem de referência que foi projetado para facilitar medições em tempo real de forças físicas e permitir a padronização da qualidade dos dados adquiridos.
O AnViM é construído sobre plugins ImageJ, software acelerado e scripts de manuseio de arquivos que permitem aos usuários avaliar quantitativamente mais de 50 propriedades, incluindo forma celular, tamanho, orientação, velocidade e direção de movimento, trações exercidas na matriz extracelular (ECM) e em células vizinhas, momentos de contractile e tesoura de ambas as células aderentes individuais e sua região vizinha. O AnViM facilita que os usuários quantifiquem as propriedades físicas celulares sem dominar o fundo técnico subjacente11. Além disso, permite a análise de dados em um modo interativo ou de processamento em lote. Gera mapas de calor revelando variação espacial e gráficos mostrando a variação temporal das propriedades das células individuais.
Em um experimento típico, o usuário cultura células em um hidrogel elástico com proteínas de matriz extracelular apropriadas na superfície superior e dois tipos de marcadores fluorescentes incorporados. Essencialmente, imagens desses marcadores fluorescentes antes e depois de culminar as células são suficientes para quantificar as forças dentro e ao redor de células individuais2,13. O AnViM mapeia esses resultados em células individuais do cluster aderente e gera imagens e gráficos perspicazes.
Protocolo
NOTA: As amostras examinadas usando a plataforma iTACS são células aderidas a um substrato macio. O protocolo de avaliação de sinais mecânicos e químicos é dividido em duas partes sequenciais: Módulo de Aquisição e Treinamento (AcTrM) e Módulo de Análise e Visualização (AnViM).
1. Módulo de Aquisição e Treinamento (AcTrM)
NOTA: A AcTrM automatiza o processo de aquisição de dados e auto-treinamento do usuário. Antes de qualquer aquisição de dados, prepare um substrato macio capaz de fornecer informações necessárias para quantificar forças que as células exercem sobre ele.
- Preparação de hidrogel
NOTA: O objetivo aqui é preparar um módulo de cisalhamento de 1.250 Pa, aproximadamente 100 μm de espessura, e um hidrogel de poliacrilamida de 22 mm de diâmetro (PAA).- Prepare a solução de poliacrilamida seguindo o método de Yeung et al. e lance os hidrogéis seguindo as etapas descritas por Trepat et al.14,15. Uma exceção ao procedimento é que as contas incorporadas imediatamente sob as células têm 0,5 μm de diâmetro e emitem fluorescência amarela.
- Pule o passo de anexar contas de 2 μm ao vidro de cobertura se a área de visualização tiver uma região grande e livre de células.
NOTA: O protocolo de preparação de hidrogel está agora bastante estabelecido no campo16. Ao longo da descrição subsequente, as contas de 0,5 μm são referidas como "contas superiores", e as contas de 2 μm são referidas como "contas inferiores". No entanto, as contas inferiores são opcionais onde a região de imagem contém uma grande área livre de células. O padrão de contas superior de uma região livre de células servirá ao propósito do padrão de contas inferior. - Monte o hidrogel com contas fluorescentes incorporadas no estágio do microscópio.
- Deixe 15-20 min para que a temperatura da placa atinja um estado estável.
NOTA: A superfície superior da placa é funcionalizada com proteínas de matriz extracelular, mas as células ainda não estão semeadas no hidrogel.
- Aquisição de imagem de referência
- Parte 1: Criando uma lista de posições
NOTA: A entrada manual nesta etapa inclui seguir as instruções acTrM para escolher locais com a melhor distribuição de contas. Nenhum arquivo criado anteriormente é usado nesta etapa. As principais novas pastas produzidas nesta etapa incluem pastas 't0imgs', 'tnimgs' e 'textfiles', e o arquivo produzido inclui o arquivo de lista de posição. Consulte a Figura 2 para obter a descrição visual das seguintes etapas que orientam a criação de uma lista de posições usando AcTrM. O exemplo demonstrado adquire dados em ampliação de 20x.- Iniciar μManager 2.0 - Beta.
- Execute o AcTrM clicando no botão AcTrM.bsh na janela Recursos IMBL .
- Na janela intitulada Define Propriedades de Aquisição, selecione o objetivo adequado, tolerância para a precisão da recuperação da posição lateral (ou seja, XY), tamanho da etapa da operação de refoco áspero e refinado (ou seja, z) e valor do coeficiente de correlação de imagem aceitável para recuperação do foco.
NOTA: Uma tolerância mais fina para a recuperação da posição lateral retardará a recuperação da posição, mas uma tolerância substancial precisará de correção de posição durante a análise dos dados e poderá causar dificuldade na recuperação do foco. Um tamanho de passo menor diminuirá a operação de refoco, mas um tamanho de passo muito alto pode causar movimento rápido com oportunidades de perder o foco. - Na janela AcTrM - Passo 0 , selecione Nova Aquisição para criar uma lista de posições.
NOTA: A janela intitulada AcTrM - Passo 1 lista todos os principais próximos passos. Essas etapas envolvem mover o palco, ajustar o foco e clicar em botões no μManager. Essas etapas orientam na construção da lista de posições adequadas para aquisição de dados. - Clique em Live in Micromanager para visualizar as amostras para ajustes manuais listados na janela AcTrM - Passo 1 .
- Siga os passos listados nesta janela.
- Agora adquira a imagem das contas. Selecione o canal apropriado para as contas superiores.
- Certifique-se de olhar para as contas inferiores também. Para isso, selecione os canais para as contas inferiores. Uma imagem embaçada de contas inferiores é vista.
NOTA: Os canais podem ser alternados a partir do menu suspenso predefinido na janela principal do μManager. Se as contas inferiores forem usadas no experimento, certifique-se de que elas estejam presentes na posição selecionada. Essas contas vão parecer embaçadas. - Ao selecionar a posição apropriada, clique em Marcar na janela Lista de posição do estágio para salvar a posição.
NOTA: A melhor posição é definida por uma distribuição densa e uniforme das contas superiores embutidas imediatamente sob a superfície superior (ou seja, contas superiores) e pelo menos duas contas presas ao vidro de cobertura (ou seja, contas inferiores) (Figura Suplementar S1). A recuperação do foco é mais rápida se as contas inferiores aparecerem como anéis grandes e fora de foco. No entanto, se os experimentos forem feitos em uma posição sem necessidade de foco ou recuperação de posição lateral, ignore as instruções relacionadas à imagem das contas inferiores. - Siga os passos 6-9 descritos na Figura 2 para incluir posições adicionais na lista.
NOTA: Escolha algumas posições adicionais para que uma rodada de eliminação possa permitir as melhores posições escolhidas na placa.
- Parte 2: Aquisição de imagens de referência
NOTA: Esta etapa não envolve nenhuma entrada manual. Arquivos-chave criados anteriormente usados nesta etapa incluem 'textfiles/*.pos'. Os principais arquivos e pastas produzidos nesta etapa incluem aqueles dentro das pastas 't0imgs' e 'tnimgs'. Consulte Figura 3 e Figura Suplementar S2 para obter a descrição visual das seguintes etapas que orientam a aquisição de imagens de referência usando AcTrM. A janela AcTrM - Passo 2 também lista todos os passos principais.- Seguindo as etapas listadas na janela AcTrM - Passo 2 , faça as escolhas na janela Aquisição Multidimensional . Por exemplo, para executar um lapso de longo tempo, indique uma série de imagens a serem tiradas, selecione o primeiro canal como canal de fase e, em seguida, selecione o próximo para as contas superiores e a seguinte é para contas inferiores. Clique em Fechar na janela Aquisição Multidimensional e, em seguida, clique em OK na janela ActrM - Passo 2 .
- Na janela AcTrM - Passo 3 , escolha engajar a recuperação de posição XYZ baseada em imagem de referência. A escolha é geralmente sim. No entanto, se as imagens forem adquiridas em uma posição sem espaço para foco ou deriva de palco, a escolha na janela AcTrM - Passo 3 será No.
- Na janela Opções de Recuperação AcTrM - XYZ , defina o canal para recuperação áspera do XYZ, quer realize a recuperação, região e canal de XYZ refinados e a direção para começar a refocar (Figura Suplementar S4). Quando concluída, a AcTrM adquirirá imagens de referência.
NOTA: As opções típicas para o canal serão o canal de contas inferiores. A recuperação do XYZ funciona melhor quando realizada com a imagem completa na implementação atual. As imagens de referência normalmente incluem uma imagem de luz transmitida do hidrogel, uma imagem fluorescente de contas superiores e uma imagem fluorescente das contas inferiores. O conteúdo de cada conjunto de imagens pode variar de acordo com as escolhas feitas na janela Aquisição Multidimensional . Ainda assim, o software adquirirá o conjunto de imagens de referência em cada posição determinada na Figura 2. Ao final desta etapa, três pastas são criadas no diretório escolhido: 't0imgs', 'tnimgs' e 'textfiles'. A pasta 't0imgs' contém imagens de referência no formato reconhecido pelo μManager e usado em etapas subsequentes; A pasta 'tnimgs' contém imagens TIFF separadas com nomes de arquivos 'c0_p0_t0.tif', 'c1_p0_t0.tif', etc. Aqui, 'c' significa o canal, 'p' significa a posição, e 't' significa para o tempo. Seguindo essas letras estão o número do canal, número de posição e número do quadro, respectivamente. Os 'arquivos de texto' da pasta contêm a lista de posições no formato XML e as escolhas do usuário para aquisição de imagens e recuperação de posições.
- Parte 1: Criando uma lista de posições
- Semeadura celular e crescimento
NOTA: A plataforma iTACS tem a flexibilidade de acomodar protocolos de preparação de amostras usados em avaliação in vitro comum do comportamento mecânico de células aderentes, incluindo células esparsas, monocamada totalmente confluente, ensaio de formação de rede e monocamadas com buracos ou lacunas significativas.- Cultura rato células microvasculares microvasculares para confluência em um frasco17,18.
- Desprende as células usando trippsina. Resuspend as células em um meio de cultura contendo 10% de soro bovino fetal a uma concentração de 1 x 106 células/mL.
- Coloque uma gota de 5 μL das células resuspended em uma superfície de hidrogel parcialmente seca e coloque-a na incubadora de cultura celular.
NOTA: Após 2 dias no meio cultural contendo 10% de soro bovino fetal, as células dessa gotícula formam uma ilha lotada de células1.
- Aquisição automatizada de imagens para o experimento restante
NOTA: A entrada manual para esta etapa inclui seguir as instruções acTrM para retomar a aquisição da imagem. Os principais arquivos criados anteriormente usados por esta etapa incluem 'textfiles/*.pos' e arquivos de imagem de contas inferiores na pasta 't0imgs'. Os principais novos arquivos produzidos nesta etapa incluem arquivos de lista de posição atualizadas e imagens 'tnimgs/*_t*.tif'.- Certifique-se de que o sistema de controle ambiental do microscópio atinja condições estáveis de cultura tecidual.
- Monte suavemente a placa contendo células cultivadas no estágio do microscópio.
- Deixe 15-20 min para que a temperatura e a umidade se estabilizem.
NOTA: Consulte a Figura 4 para obter a descrição visual das seguintes etapas que orientam a aquisição de imagens para o experimento restante usando AcTrM. - Iniciar μManager 2.0 - Beta.
- Execute o AcTrM clicando no botão AcTrM.bsh na janela Recursos IMBL .
NOTA: Ignore as escolhas feitas na janela Definir Propriedades de Aquisição , mas use as escolhas feitas na etapa anterior. - Na janela AcTrM - Passo 0 , selecione Continuar a aquisição pausada.
- Selecione a ampliação e o diretório identificados na janela Aquisição Multidimensional na etapa anterior, onde as pastas de dados ('t0imags', 'tnimgs' e 'textfiles') foram salvas.
NOTA: O título da janela orienta o usuário a escolher o diretório apropriado. - Na janela Reposicionamento da placa , selecione opções para reposicionar a placa (três posições não-lineares) juntamente com o canal usado para reposicionamento.
NOTA: Imagens salvas serão vistas na câmera. Se as imagens sobrepostas forem exibidas como uma imagem de cor vermelha, verde e preta, realize o ajuste manual. - Clique em Aceitar para prosseguir com a aquisição.
NOTA: Este passo supera as ligeiras mudanças da irreprodutibilidade do alinhamento da placa quando a placa é remontada no estágio do microscópio. Siga o AcTrM solicita agilizar o alinhamento em cada uma das três posições, mostrando uma imagem composta da imagem de referência mostrada em vermelho (tipicamente das contas inferiores) e a imagem atualmente observada através do objetivo mostrado em verde. Obter o verde suficientemente perto do vermelho deixa menos trabalho para o software. Quando a sobreposição é perfeita, a imagem composta aparece amarela e preta. Suficientemente perto é normalmente quando as mesmas contas inferiores são visíveis em imagens vermelhas e verdes, e os anéis vermelho e verde correspondentes estão se tocando. Após o reposicionamento da placa estar concluído, o AcTrM sobe ao palco para cada posição selecionada e recupera a posição XYZ combinando a imagem de referência com o que é visto atualmente através da câmera. A primeira posição lateral (XY) é combinada, e então o foco (Z) é combinado. A recuperação áspera é seguida pela recuperação refinada da posição e do foco. As atualizações do status atual do experimento são exibidas na tela através de uma janela de quatro partes mostrada na Figura Suplementar S3. As imagens adquiridas são salvas na pasta 'tnimgs' após '*_t1.tif', '*_t2.tif', indicando a contagem de tempo para o conjunto de imagens. Se a recuperação do XYZ identificar uma posição atualizada, a nova lista de posições será gerada e salva na pasta 'textfiles'.
2. Módulo de Análise e Visualização (AnViM)
- Configuração automatizada da análise de dados
NOTA: A entrada manual nesta etapa inclui identificar a localização da pasta 'tnimgs'. Os principais arquivos criados anteriormente usados nesta etapa incluem 'tnimgs/*.tif'. Os principais arquivos e pastas produzidos nesta etapa incluem a pasta 'análise' na mesma pasta pai como 'tnimgs' e pastas para cada posição 'análise/p*'. Dentro de cada 'análise/p*', esta etapa cria um novo arquivo de imagem '*.tif' nas pastas 'phs' e 'tny'. Essas imagens são cropped e des drifted imagens de células e contas superiores. Os outros arquivos criados incluem 'análise/análiseChoices.txt' que lista escolhas de análise, 'análise/p*/skipAnalysis.txt', que lista casos em que a análise é ignorada devido à deriva pré-existente significativa, e 'análise/p*/part_shift_values_pixel_degree.dat' que lista valores de deriva estimados. A AnViM não altera os dados brutos na pasta 'tnimgs'. Consulte a Figura 5 para obter a descrição visual das seguintes etapas que orientam a configuração da análise automatizada de dados usando o AnViM.- Inicie o software Fiji e selecione a primeira opção no menu suspenso msm rotulado como MSM - pré-processamento.
- Usando a caixa de diálogo do navegador de arquivos, selecione a pasta contendo a pasta 'tnimgs', que contém os dados analisados.
- Defina o canal para as imagens. Seguindo as diretrizes da Figura Suplementar S5, identifique os números de canais da imagem de luz transmitida das células (imagem de contraste de fase das células), imagem de contas inferiores e imagem superior das contas. As seguintes três caixas de seleção ('Mover arquivos para a pasta de posição', 'Fazer correção adicional para movimento rígido' e 'Cortar dados e salvar na pasta de posição') são normalmente verificadas. Por fim, defina tolerância para rejeitar dados fortemente derivados, tamanho do pixel e lados da imagem onde as células se cruzam.
- Responda ao prompt para melhorar o brilho e o contraste das imagens de contas inferiores para que as contas apareçam com destaque. Ajuste isso usando a barra de controle deslizante no menu e clique em OK.
- Agora execute a correção de posição para se livrar dos turnos, se houver. Uma vez feita, uma pasta de análise é criada.
NOTA: O aprimoramento do contraste normalmente não é necessário, mas esta disposição está disponível para experimentos onde a exposição a lasers precisa ser minimizada. Após essa escolha, o AnViM copia os arquivos da pasta 'tnimgs' para a pasta 'análise'. Os arquivos correspondentes a cada posição são salvos nas pastas 'análise/p0, análise/p1', etc. Dentro de cada uma dessas pastas de posição, a AnViM faz pastas 'cels', 'defs' e 'refs' contendo a imagem de luz transmitida original, imagens de contas superiores e imagens de contas inferiores, respectivamente (Figura Suplementar S6). O AnViM então analisa o movimento rígido nas imagens das contas inferiores e cria uma pasta 'phs' contendo uma imagem de luz transmitida corrigida das células e uma pasta 'tny' contendo imagens de contas superiores atualizadas. Finalmente, as escolhas dos usuários dos canais, operações, tolerância, tamanho do pixel e cruzamento de limites são salvas no arquivo 'analysisChoices.txt' (Figura Suplementar S6).
- Quantificação da deformação de hidrogel e monocamadas
NOTA: É importante notar que a quantificação do movimento a partir de uma sequência de imagens é um campo em rápida evolução19. A tecnologia é constantemente otimizada para recursos, incluindo velocidade, precisão, características específicas dentro das imagens brutas e padrões específicos de deformação. Portanto, é provável que alguns usuários possam usar uma abordagem de quantificação de deformação diferente da apresentada aqui.-
Parte 1: Quantificação da deformação do hidrogel
NOTA: As entradas manuais nesta etapa incluem a identificação do diretório de dados e a seleção da resolução da grade. Os principais arquivos criados anteriormente usados nesta etapa incluem 'p*/tny/*.tif'. Os principais novos arquivos produzidos neste incluem 'p*/displacement/*_disp.dat' que lista vetores de deslocamento da superfície superior do hidrogel. Consulte a Figura 6 para obter a descrição visual das seguintes etapas para engajar, via AnViM, a análise de Velocimetria de Imagem de Partículas nas imagens superiores das contas20.- Inicie o software Fiji e, a partir do menu suspenso do MSM , selecione MSM - Deformação de Gel.
- A partir daqui, selecione a opção que é adequada para o experimento.
- Usando a caixa de diálogo do navegador de arquivos, selecione o diretório pai da pasta 'análise' contendo os dados analisados.
- Na janela Parâmetros para calcular a deformação do gel , selecione o tamanho da janela de correlação cruzada apropriada, o nível de ruído e o limiar (Figura Suplementar S7)20.
NOTA: Os resultados são salvos em um diretório recém-criado de 'deslocamento' dentro de cada pasta de posição 'análise/p0, análise/p1', etc. É aqui que todos os arquivos de saída são armazenados.
-
Parte 2: Quantificação da deformação de monocamadas
NOTA: As entradas manuais nesta etapa incluem a identificação do diretório de dados e a seleção da resolução da grade. Os principais arquivos criados anteriormente usados nesta etapa incluem 'p*/tny/*.tif'. Os principais novos arquivos produzidos neste incluem 'p*/velocity/*_vel.dat' que lista vetores de movimento das células. Consulte a Figura 7 para obter a descrição visual das etapas a serem engatar, via AnViM, a análise de Velocimetria de Imagem de Partículas nas imagens superiores das contas20. O procedimento é semelhante ao seguido para quantificar a deformação do hidrogel e seleciona o MSM - Cellular Motion do menu suspenso MSM . Os resultados são salvos em um diretório 'velocity' recém-criado dentro de cada pasta de posição 'análise/p0', 'análise/p1', etc.
-
Parte 1: Quantificação da deformação do hidrogel
- Quantificação de células-ECM e forças célula-célula
NOTA: A entrada manual nesta etapa inclui identificar o diretório de dados, indicar a rigidez do hidrogel e responder às solicitações de segmentação de clustercelular para detectar células da região sem células. Os principais arquivos criados anteriormente usados nesta etapa incluem 'p*/displacement/*_disp.dat'. Os principais arquivos produzidos nesta etapa incluem: 'p*/traction/*_trac.dat', que lista forças exercidas pelas células do hidrogel; 'p*/traction/*_domain.dat', que lista a localização dos pontos de grade contendo células; e arquivos de entrada 'p*/traction.in, p*/clusterInput.txt' e 'p*/prestress.in' que registram as escolhas do usuário para esta etapa. Após a primeira avaliação quantitativa das forças célula-ECM e células-células, várias variações à técnica foram desenvolvidas1,15. As variações se concentram em casos específicos de substratos, células, condições experimentais ou ferramentas numéricas7, 8,21,22. Consulte a Figura 8 para obter a descrição visual para se engajar via AnViM, a Microscopia de Tração de Quatro Transformações e a análise de Microscopia de Estresse monocamada nos dados de deformação do hidrogel1,2,15.- Inicie o software Fiji e, a partir do menu suspenso do MSM , selecione MSM - Cell-ECM e Cell-Cell Forces (terceira opção no menu suspenso).
- Usando a caixa de diálogo do navegador de arquivos, selecione o diretório pai da pasta 'análise' contendo as pastas 'tnimgs' e 'análise' contendo dados analisados. Clique em Selecionar.
- Na janela Parâmetros para calcular forças celulares , entre no módulo de cisalhamento, espessura do hidrogel e no nível de ruído previsto. Selecione OK. Isso permite que ele execute a tração através da função MATLAB.
NOTA: Seguindo essas entradas, o AnViM calcula as forças do ECM celular. - Depois disso, indique se a monocamada é confluente. Se toda a imagem celular está coberta de células, então a resposta é Sim. Nesse caso, as forças celulares serão computadas em todo o quadro. Por outro lado, se uma parte da imagem não tem células, então a resposta é Não. Nesse caso, siga as instruções do AnViM para facilitar a segmentação da região de imagem que contém células.
- Se a resposta for não, desenhe manualmente um polígono em torno do objeto não-celular quando o software solicitar isso e selecione um método apropriado para segmentação. O software pedirá a cor (preto ou branco) das células.
- Verifique a opção Preencher pontos automaticamente no software e clique em OK.
NOTA: As etapas para segmentar uma monocamada não confluente serão cobertas na primeira parte da 'Seção 2.4. As forças do Cell-ECM são armazenadas em arquivos '*_trac.dat' em uma 'tração' de diretório recém-criada dentro de cada pasta de posição, e a entrada para o software de cálculo de força cell-ECM é salva no arquivo 'traction.in' (Figura Suplementar S8). As forças de células celulares são armazenadas em arquivos '*_prestress.dat' em um diretório recém-criado 'prestress' dentro de cada pasta de posição, e a entrada para o software de cálculo de força celular é salva no arquivo 'prestress.in' (Figura Suplementar S9).
- Mapeamento de valores de pontos de grade em células individuais
NOTA: Um dos focos atuais do iTACS é introduzir uma abordagem simples para interpretar os sinais mecânicos medidos no campo. Esta abordagem é benéfica para examinar interações mecânicas entre células vizinhas de um cluster18. No geral, as propriedades quantificadas até agora estão em pontos de grade regularmente espaçados em todo o aglomerado celular. Esses dados identificam o desvio mediano, médio e padrão das propriedades selecionadas dentro do limite morfológico das células individuais e as atribui como propriedades/sinais físicos celulares. A partir disso, o desvio padrão é utilizado para indicar variabilidade, a diferença entre média e mediana é usada para indicar a natureza da distribuição, e o valor médio é usado para indicar o estado geral das células para as propriedades selecionadas.- Parte 1: Segmentação da região da imagem que contém células
NOTA: A entrada manual nesta etapa inclui identificar o diretório de dados e responder às solicitações de segmentação para detectar células da região sem células. Os principais arquivos criados anteriormente usados nesta etapa incluem 'p*/phs/*.tif'. Os principais novos arquivos produzidos nesta etapa incluem 'p*/phs/bwImages/*.tif' que são imagens binárias que separam regiões celulares de regiões sem células, 'p*/phs/textResults/frameNum_percentHealed.txt', que lista por cento área de imagem coberta por células em cada instância, e 'p*/clusterInput.txt', que registra escolhas de usuário inseridas na interface AcTrM. Consulte a Figura 9 para obter a descrição visual das seguintes etapas para segmentar a região da imagem que contém células. Esta parte deve ser concluída antes de calcular as forças célula-célula. Nesse caso, essas etapas não precisam ser repetidas. Além disso, se essas etapas forem conduzidas antes dos cálculos de força celular-célula, elas não são solicitadas no início dos cálculos da força.- Inicie o software Fiji e, a partir do menu suspenso do MSM , selecione Segmentação - Cluster Celular.
- Usando a caixa de diálogo do navegador de arquivos, selecione o diretório de posição 'analysis/p0', 'analysis/p1', etc., que contém propriedades quantificadas em pontos de grade regularmente espaçados.
- Indique se a monocamada é confluente.
NOTA: As etapas abaixo são invocadas somente quando a monocamada não estiver confluente. Siga o AnViM solicita a aplicação da nova abordagem multifacetado para identificar as regiões de imagem que contêm células. O processo envolve quatro métodos - cada um abordando a segmentação de uma forma diferente. Um ou vários desses métodos usados em combinação cobrem uma grande variedade de imagens das células. Portanto, tente diferentes abordagens para descobrir qual combinação funciona melhor para seus dados. - Ajuste o 'fator de conexão de domínio' e 'Fator de difamação' para obter a segmentação ideal.
NOTA: O 'fator difamação' torna as regiões que contêm células maiores. - Indique se as células aparecem em preto ou branco na imagem binária.
- Nas instruções para limpar a janela Imagem de limite , escolha se deve limpar a imagem automaticamente ou manualmente.
NOTA: Características indesejadas das imagens são manchas pretas nos pixels ocupados por células e manchas brancas nos pixels desconectados do cluster celular a serem analisados. A análise só pode ser feita nas regiões conectadas. Assim, várias regiões desconectadas devem ser analisadas separadamente.
- Parte 2: Segmentação das células individuais nas imagens
NOTA: As entradas manuais para esta etapa incluem identificar o diretório de dados e responder às solicitações de segmentação para detectar células individuais dentro da imagem. Os principais arquivos criados anteriormente usados nesta etapa incluem 'p*/phs/*.tif' e 'p*/phs/bwImages/*.tif'. Os principais arquivos produzidos incluem 'p*/phs/textResults/*.csv', que contêm propriedades morfológicas celulares. Consulte a Figura 10 para obter a descrição visual das seguintes etapas para segmentar células individuais da monocamada.- Inicie o software Fiji e, a partir do menu suspenso do MSM , selecione Segmentação - Células Individuais.
- Usando a caixa de diálogo do navegador de arquivos, selecione o diretório de posição 'analysis/p0', 'analysis/p1', etc., que contém propriedades quantificadas em pontos de grade regularmente espaçados.
- Na janela Escolher os Parâmetros de Entrada , indicar a proporção máxima, destaque de quanto o centro celular se destaca em relação ao limite celular e dois parâmetros de desfoque para orientar a detecção das células (Figura Suplementar S10).
- Na pilha de imagens para cada posição, desenhe um polígono na menor célula normal. Isso é usado para calcular a área. Qualquer coisa menor do que isso não será considerada como uma célula pelo software.
- Em seguida, desenhe um polígono em torno da maior célula normal e indique se o centro celular ou a interface celular é mais brilhante.
NOTA: O AnViM cria então arquivos 'phs/textResults/0001.csv', 'phs/textResults/0002.csv' , etc., para cada quadro contendo informações sobre células dentro desse quadro. Nesta fase, este arquivo inclui informações morfológicas sobre as células, incluindo área, centroide, perímetro, orientação, circularidade, proporção, arredondamento, solidez, distância da região sem células. A unidade de comprimento nessas propriedades são pixels, e o ângulo está em graus. Finalmente, este arquivo é atualizado para conter intensidade, movimento e forças de pixels celulares nas etapas subsequentes.
- Parte 3: Mapeamento das intensidades dos pixels nas células
NOTA: As entradas manuais nesta etapa incluem identificar o diretório de dados e selecionar propriedades e parâmetros de mapeamento. Os principais arquivos criados anteriormente usados nesta etapa incluem 'p*/phs/*.tif' (ou 'p*/fluo/*.tif ') e 'p*/phs/bwImages/*.tif'. Os principais novos arquivos produzidos nesta etapa incluem 'p*/phs/textResults/*.csv', que lista propriedades morfológicas celulares. Consulte a Figura 11 para obter a descrição visual das seguintes etapas para avaliar as intensidades de pixels na região englobadas por células individuais e sua região vizinha e defini-las como propriedades das células.- Inicie o software Fiji e, a partir do menu suspenso do MSM , selecione Mapa em Células - Intensidades.
- Usando a caixa de diálogo do navegador de arquivos, selecione o diretório de posição 'analysis/p0', 'analysis/p1', etc., que contém propriedades quantificadas em pontos de grade regularmente espaçados.
- Selecione Detectar divisão celular ou quantificar fluorescência celular quando solicitado pelo software. Defina o tamanho da região vizinha. Verifique as propriedades de coleta da região vizinha e colete propriedades das caixas de células. Clique em OK.
- No Propósito de Registrar a janela intensidade celular, indique que tipo de imagem deve ser usada para mapeamento de intensidade, o tamanho da região vizinha e se os dados são coletados para células individuais ou ambas as células e suas regiões vizinhas (Figura Suplementar S11).
NOTA: Mapear as intensidades dos pixels de uma imagem de contraste de fase permite a detecção de eventos de divisão celular. Mapear intensidades de imagem fluorescentes permitirá a detecção de flutuações nas moléculas citoplasmáticas marcadas fluorescentemente. A saída das etapas acima é os valores médios, medianos, de desvio padrão, mínimo e máximo de intensidade de pixel para células individuais. Esses números são inseridos como novas colunas nos arquivos 'phs/textResults/0001.csv', 'phs/textResults/0002.csv' , etc.
- Parte 4: Mapeamento das forças e movimento nas células
NOTA: As entradas manuais para esta etapa incluem identificar o diretório de dados e selecionar propriedades e parâmetros de mapeamento. Os principais arquivos criados anteriormente usados nesta etapa incluem 'p*/phs/textResults/*.csv', 'p*/velocity/*_vel.dat', 'p*/displacement/*_disp.dat', 'p*/traction/*_trac.dat', 'p*/prestress/*_prestress.dat', e 'p*/phs/bwImages/*.tif'. Nenhum novo arquivo é criado durante esta etapa. Em vez disso, as novas informações (propriedades de força celular e movimento) são adicionadas aos arquivos 'p*/phs/textResults/*.csv '. Consulte a Figura 12 para a descrição visual das etapas para avaliar as forças e o movimento na região englobadas por células individuais e sua região vizinha e defini-las como propriedades das células.- Escolha entre o mapa do menu suspenso do MSM em células - Dados de movimento/força (Figura Suplementar S12).
- Em seguida, siga os passos listados na etapa 2.4.3.
NOTA: Novas colunas são adicionadas aos arquivos 'phs/textResults/0001.csv', 'phs/textResults/0002.csv', etc. e incluem o desvio médio, mediano e padrão de velocidade, a orientação da velocidade, tensão citoesqueleta média, anisotropia de tensão, energia de tensão no hidrogel, orientação de maior tensão e magnitude da tração célula-ECM (Figura Suplementar S13).
- Parte 1: Segmentação da região da imagem que contém células
- Visualização de resultados
- Parte 1: Rastreamento das identidades celulares através do experimento
NOTA: As entradas manuais para esta etapa incluem identificar o diretório de dados e selecionar propriedades mapeadas para visualizar. Os principais arquivos criados anteriormente usados nesta etapa incluem 'p*/phs/textResults/*.csv'. Os principais arquivos produzidos durante esta etapa incluem 'p*/phs/textResults/cellTimeTrace_*.csv', que contém um rastreamento de tempo dos dados celulares. Consulte a Figura 13 para obter a descrição visual das seguintes etapas para rastrear as propriedades de células individuais durante toda a duração do experimento.- Inicie o software Fiji e, a partir do menu suspenso do MSM , selecione Resultados - Rastrear Dados.
- Usando a caixa de diálogo do navegador de arquivo, selecione o diretório de posição 'análise/p0', 'análise/p1', etc., contendo propriedades celulares a serem rastreadas. Quando terminar clique em Selecionar.
- Na janela Escolher ponto de partida para rastreamento , escolha o quadro de partida para monitoramento e o número de quadros rastreados simultaneamente (Figura Suplementar S14). Comece sempre a partir do quadro número 2, pois a velocidade não pode ser determinada para o quadro número 1. Quando terminar, clique em OK.
- Na janela Verificar as Variáveis para Fazer Trilhas , escolha as variáveis digitando os nomes de variável ou selecionando os termos das caixas de seleção (Figura Suplementar S15). Em seguida, clique em OK.
NOTA: O rastreamento gera arquivos 'phs/textResults/cellTimeTrace_*.csv' com cada coluna contendo um número de celular único e cada linha sucessiva representando a instância temporal consecutiva.
- Parte 2: Geração de faixas temporidas das propriedades celulares avaliadas
NOTA: As entradas manuais para esta etapa incluem identificar o diretório de dados e selecionar propriedades mapeadas para visualizar. Os principais arquivos criados anteriormente usados nesta etapa incluem 'p*/phs/textResults/cellTimeTrace_*.csv'. Os principais arquivos produzidos durante esta etapa incluem 'p*/phs/trackedDataPlots/*.tif', que contêm plots de faixa de tempo e 'p*/phs/trackedDataPlots/*.csv', que contêm dados de plot. Consulte a Figura 14 para obter a descrição visual das seguintes etapas para gerar faixas de tempo das propriedades celulares avaliadas.- Inicie o software Fiji e, a partir do menu suspenso do MSM , selecione Resultados - plot.
- Usando a caixa de diálogo do navegador de arquivos, selecione o diretório de posição 'análise/p0', 'análise/p1', etc., contendo propriedades celulares rastreadas a serem plotadas.
- Escolha se deve limitar a plotagem a células com faixas ininterruptas clicando no botão Sim ou Não .
NOTA: Esta opção ignora as células, que não puderam ser detectadas em uma ou mais instâncias de tempo. - Escolha o número de variáveis a serem plotadas simultaneamente e clique em OK.
NOTA: Atualmente, o AnViM permite que um máximo de três variáveis sejam exibidas no mesmo lote. - Escolha variáveis individuais para plotar usando um menu suspenso.
NOTA: Essas etapas criam um arquivo TIFF (Image file format, formato de arquivo de imagem marcado) e um arquivo de dados separado por círia na pasta 'phs/trackedDaDataPlots/'. O nome do arquivo é composto de nomes de variáveis separados por um sublinhado e termina com um número de celular.
- Parte 3: Geração de mapas de calor das propriedades celulares avaliadas
NOTA: As entradas manuais nesta etapa incluem identificar o diretório de dados e selecionar propriedades mapeadas para visualizar. Os principais arquivos criados anteriormente usados nesta etapa incluem 'p*/phs/textResults/cellTimeTrace_*.csv'. Os principais novos arquivos gerados nesta etapa incluem 'p*/phs/segmentedImages/cellPropMaps/*.tif', que são os mapas de calor das propriedades celulares. Consulte a Figura 15 para obter a descrição visual das etapas para gerar mapas de calor das propriedades celulares avaliadas.- Escolha entre os resultados do menu suspenso do MSM - Imagem.
- Siga os passos descritos na etapa 2.5.2.
NOTA: A saída é armazenada como arquivos TIFF na pasta 'phs/segmentedImages/cellPropMaps/'. Os nomes dos arquivos são o número da instância de tempo e o nome variável separados por um sublinhado.
- Parte 1: Rastreamento das identidades celulares através do experimento
Resultados
Apresentamos aqui duas das principais saídas para o exemplo demonstrado. A primeira saída é o traço de tempo da velocidade celular e da tensão citoesquelético para a célula número 1 (Figura 16). As propriedades são mostradas em um eixo vertical compartilhado para facilitar a associação visual entre as propriedades, e o eixo horizontal indica o número da instância de tempo. Neste experimento, foram adquiridos quadros sucessivos em um intervalo de 15 minutos. A segunda saída é uma matriz de mapas de calor 1 h no experimento (Figura 17). As propriedades aqui mostradas incluem área de disseminação, orientação, circularidade, velocidade, direção de movimento, orientação de tensão máxima, tensão citoesquelletal, traçãos substratos e anisotropia de tensão de células individuais.

Figura 1: Estrutura do Kit de Ferramentas Integrativas para Análise de Sinais Celulares (iTACS). Dois componentes-chave do iTACS são Módulo de Aquisição e Treinamento (AcTrM) e Módulo de Análise e Visualização (AnViM). AcTrM pode usar várias técnicas de preparação de hidrogel que existem atualmente para preparar hidrogéis que podem ser mantidos firmemente em um estágio de microscópio, qualquer semeadura celular, e um protocolo de crescimento que retém células em um plano focal. O AnViM pode usar várias técnicas para quantificar a deformação de hidrogel e monocamada, forças de células ECM e forças células celulares. Todos esses componentes preferidos pelo usuário do protocolo de medição de força podem ser acomodados no iTACS, e foram identificados com caixas tracejadas. Os componentes identificados com caixas sólidas são novas contribuições para a tecnologia de medição da força celular. A visualização no AnViM se concentra no valor mediano e variabilidade das propriedades em células individuais. Clique aqui para ver uma versão maior desta figura.
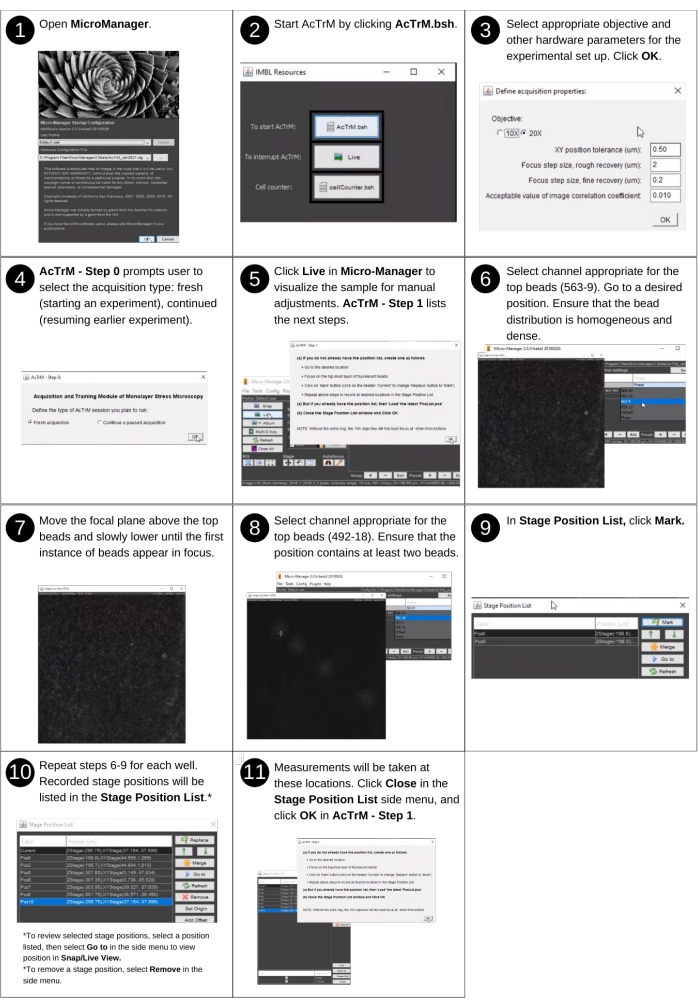
Figura 2: Aquisição de imagem de referência - parte 1. Etapas para criar uma lista de posições usando AcTrM. Clique aqui para ver uma versão maior desta figura.
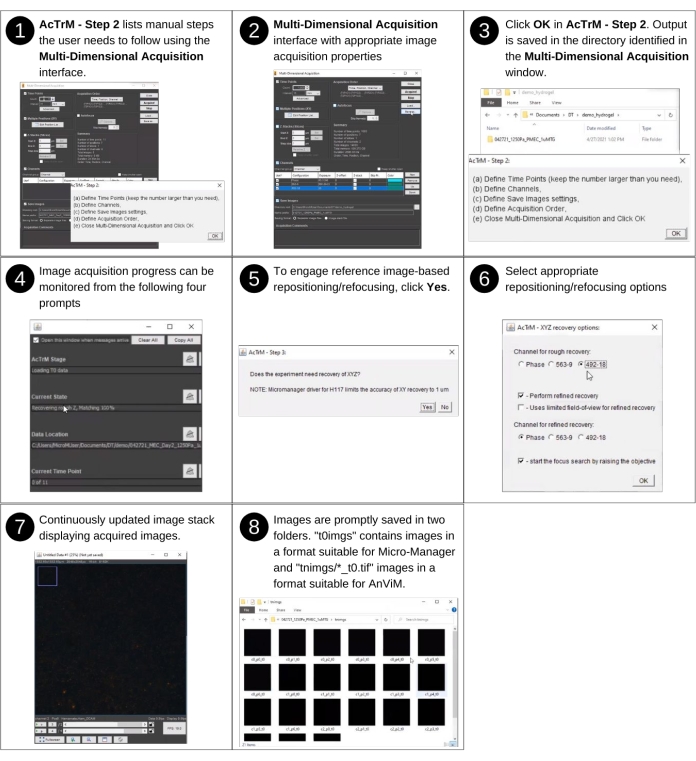
Figura 3: Aquisição de imagem de referência - parte 2. Etapas para a aquisição de imagens de referência usando AcTrM. As visões detalhadas das etapas 2, 4 e 6 são apresentadas nas Figuras Suplementares S2, S3 e S4, respectivamente. Clique aqui para ver uma versão maior desta figura.
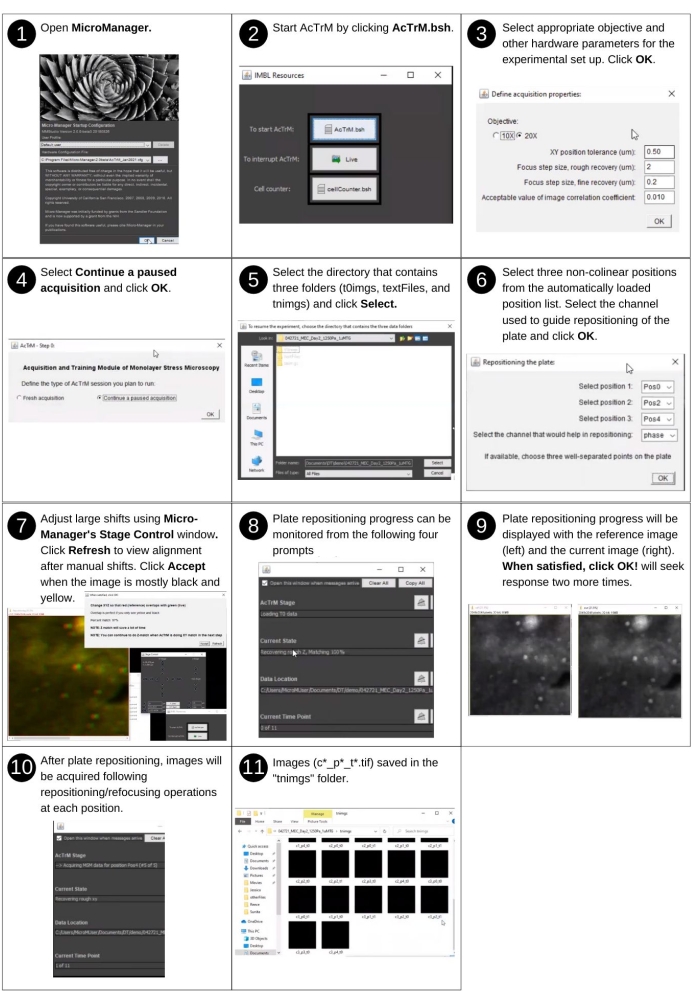
Figura 4: Aquisição automatizada de imagens para o experimento restante. Etapas para retomar a aquisição de imagens para avaliar o comportamento celular usando o AcTrM. Clique aqui para ver uma versão maior desta figura.

Figura 5: Configuração automatizada da análise de dados. Etapas para iniciar a análise automatizada de imagens usando o AnViM. O software reconhece o formato de imagem usado pela AcTrM. Uma visão detalhada dos painéis nas etapas 3 e 5 é apresentada nas Figuras Suplementares S5 e Figura Suplementar S6, respectivamente. Clique aqui para ver uma versão maior desta figura.
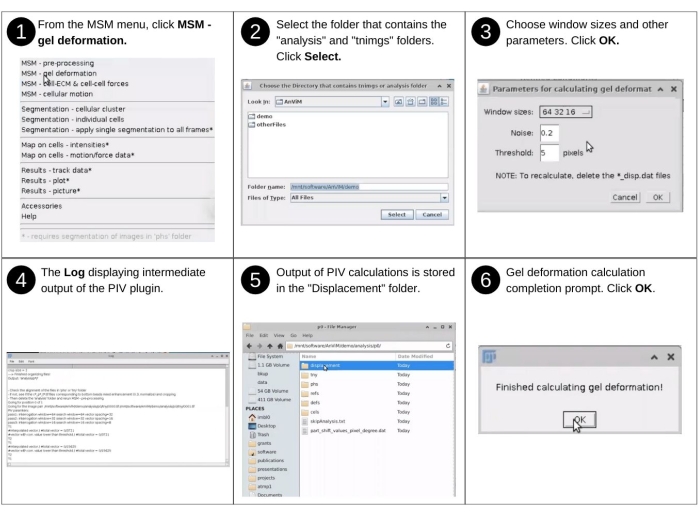
Figura 6: Quantificação da deformação de hidrogel e monocamadoreira - parte 1. Etapas para engajar, via AnViM, a implementação da Velocimetria de Imagem de Partículas de Tseng, Q. et al., PNAS (2012)20 para quantificar a deformação da superfície superior do hidrogel. Os usuários também podem implementar dentro do AnViM outras abordagens para quantificar a deformação do hidrogel. Uma visão detalhada da etapa 3 é apresentada na Figura Suplementar S7. Clique aqui para ver uma versão maior desta figura.
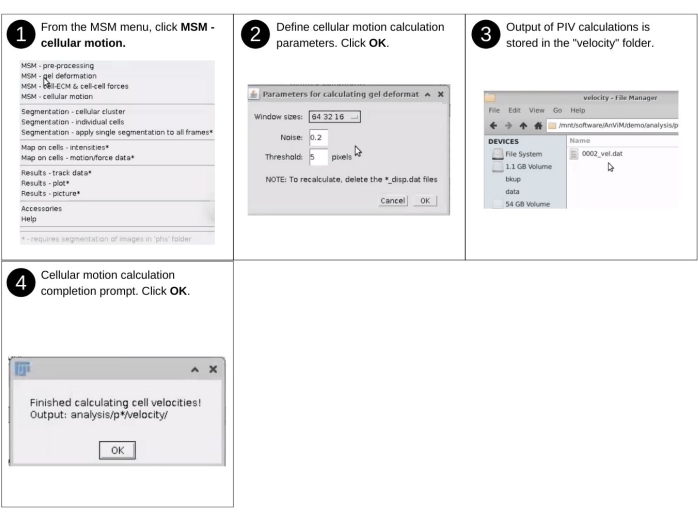
Figura 7: Quantificação da deformação de hidrogel e monocamadora - parte 2. Etapas para engajar, via AnViM, a implementação da Velocimetria de Imagem de Partículas de Tseng, Q. et al., PNAS (2012)20 para quantificar o movimento local de células individuais. Os usuários também podem implementar dentro do AnViM outras abordagens para quantificar o movimento celular. Clique aqui para ver uma versão maior desta figura.
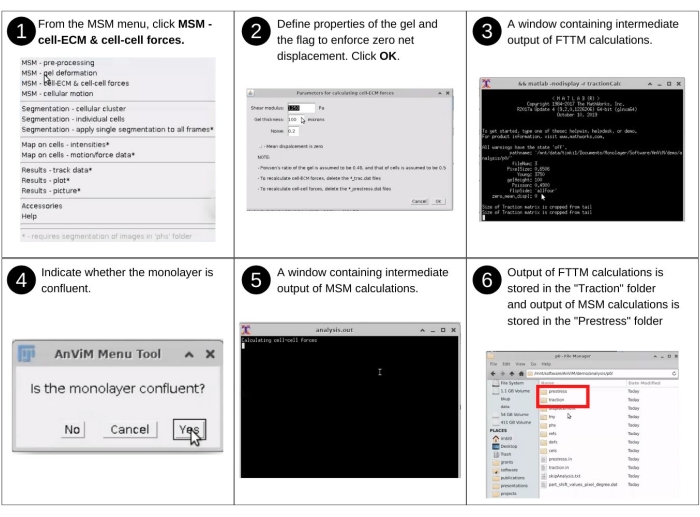
Figura 8: Quantificação das forças célula-ECM e células-células. Etapas para realizar a análise de imagem para engajar, via AnViM, a implementação da Microscopia de Tração de Quatro Transformações de Trepat et al., Nature Physics (2009)15 para quantificar forças exercidas pelas células no hidrogel, e a implementação da Microscopia de Estresse Monocamada de Tambe et al., Nature Materials (2011)1 para quantificar forças dentro de células individuais e entre células vizinhas. Os usuários também podem implementar dentro do AnViM outras abordagens para quantificar as forças célula-ECM e células-células. Uma visão detalhada da etapa 6 é apresentada em Figura Suplementar S8 e Figura Suplementar S9. Clique aqui para ver uma versão maior desta figura.
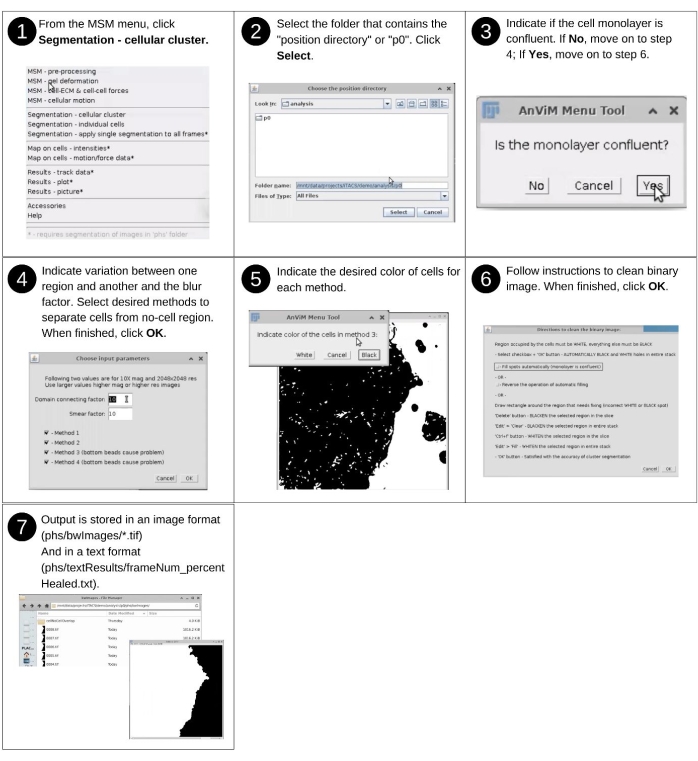
Figura 9: Mapeando valores de pontos de grade em células individuais - parte 1. Passos para segmentar as regiões de imagem contendo células usando uma nova abordagem multifacetado. Essa abordagem pode ser usada para segmentar imagens de contraste de fase, campo brilhante ou fluorescência das células. Clique aqui para ver uma versão maior desta figura.
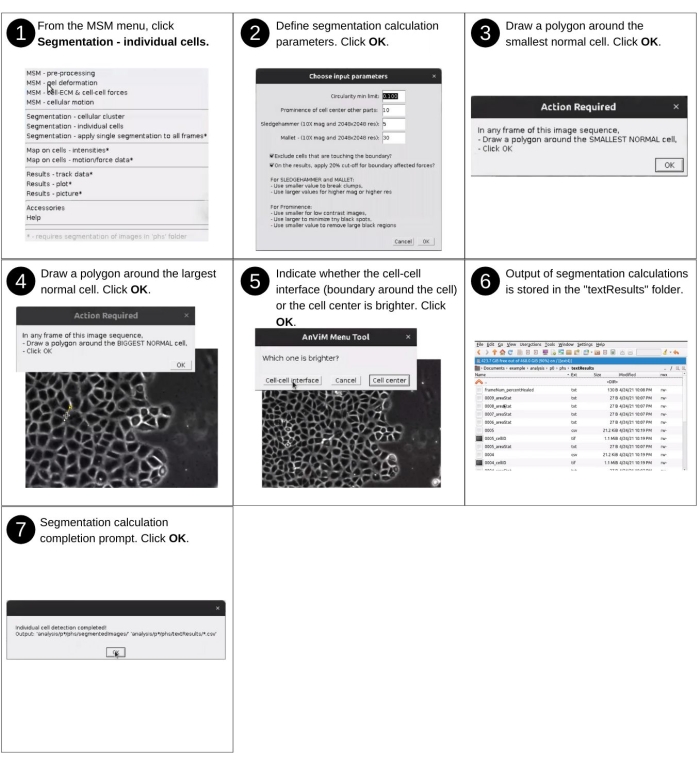
Figura 10: Mapeamento de valores de pontos de grade em células individuais - parte 2. Passos para segmentar células individuais de uma monocamada usando uma nova abordagem multifacetado desenvolvida no AnViM. Essa abordagem pode ser usada para segmentar imagens de contraste de fase, campo brilhante ou fluorescência das células. Uma visão detalhada da etapa 2 é apresentada na Figura Suplementar S10. Clique aqui para ver uma versão maior desta figura.
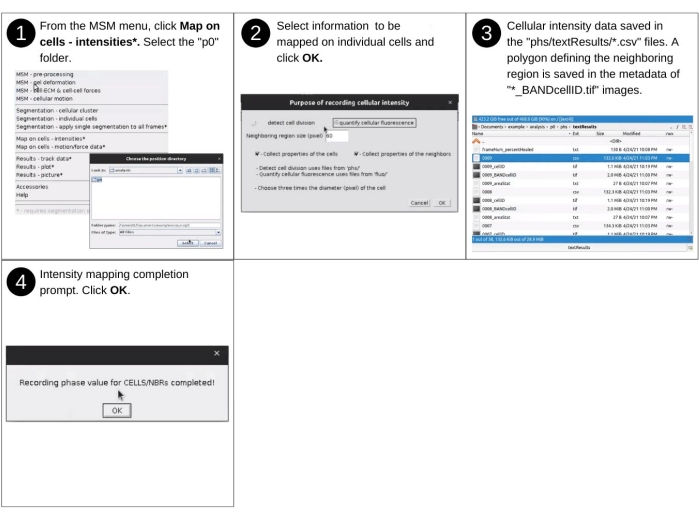
Figura 11: Mapeamento de valores de pontos de grade em células individuais - parte 3. Etapas para avaliar a intensidade dos pixels na região dentro de células individuais e dentro da região vizinha de células individuais usando OViM. As intensidades avaliadas incluem intensidade de luz transmitida e intensidade de fluorescência. Esta parte mapeia o valor mediano e o desvio padrão das intensidades dos pixels dentro de células individuais e dentro de uma região vizinha de células individuais. Uma visão detalhada da etapa 2 é apresentada na Figura Suplementar S11. Clique aqui para ver uma versão maior desta figura.
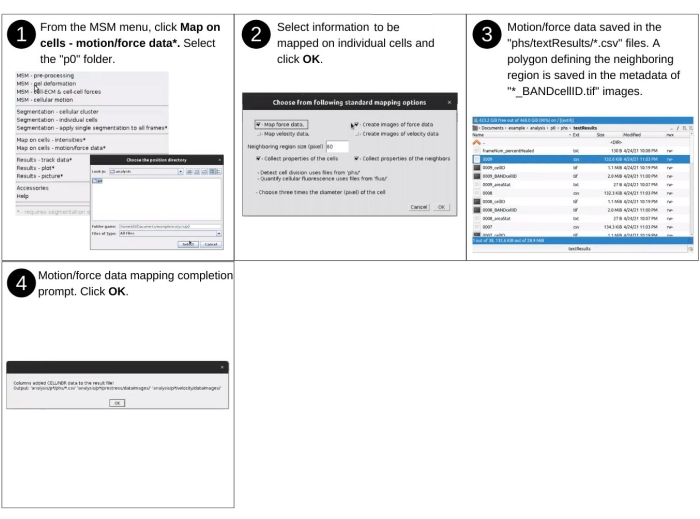
Figura 12: Mapeamento de valores de pontos de grade em células individuais - parte 4. Etapas para avaliar forças e propriedades de movimento dos pontos de grade dentro de células individuais e dentro da região vizinha de células individuais usando OViM. Esta parte mapeia o valor mediano e o desvio padrão das propriedades dentro de células individuais e dentro de uma região vizinha de células individuais. Uma visão detalhada das etapas 2 e 3 são apresentadas nas Figuras Complementares S12 e Figura Suplementar S13. Clique aqui para ver uma versão maior desta figura.
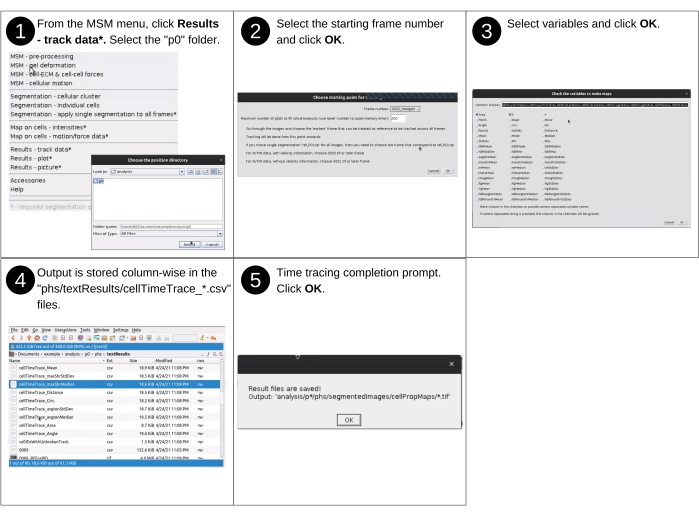
Figura 13: Visualização dos resultados - parte 1. Etapas para rastrear propriedades de células individuais durante toda a duração do experimento usando o AnViM. Uma visão detalhada das etapas 2 e 3 são apresentadas nas Figuras Complementares S14 e Figura Suplementar S15. Clique aqui para ver uma versão maior desta figura.
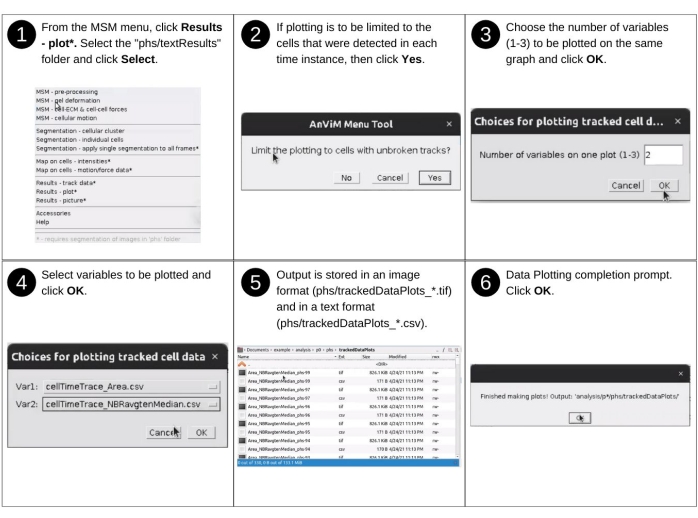
Figura 14: Visualização dos resultados - parte 2. Etapas para gerar traços de tempo das propriedades avaliadas usando o AnViM. O usuário tem a opção de traçar até três propriedades em um gráfico. Os traços de tempo são gerados para todas as células ou apenas para aquelas células para as quais o rastreamento foi bem sucedido em todo o experimento. Uma visão detalhada da etapa 5 é apresentada na Figura Suplementar S16 e Figura Suplementar S17. Clique aqui para ver uma versão maior desta figura.
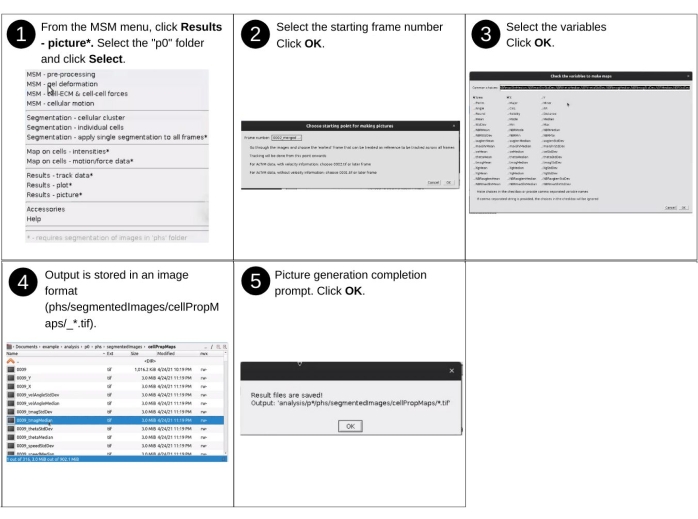
Figura 15: Visualização dos resultados - parte 3. Etapas para gerar mapas de calor das propriedades avaliadas usando o AnViM. Mapas de calor são gerados para todos os quadros após o quadro de partida e todas as propriedades selecionadas. Uma visão detalhada da etapa 3 é apresentada na Figura Suplementar S18. Clique aqui para ver uma versão maior desta figura.
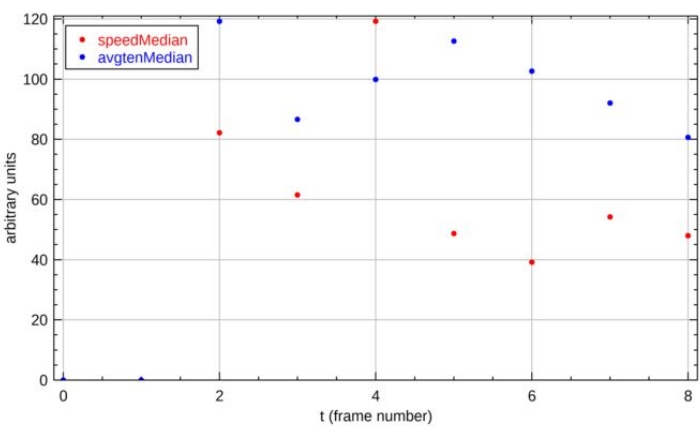
Figura 16: Traços de tempo para iD celular 1. Duas propriedades exibidas são a tensão citoesquelético celular ("avgtenMedian") e a velocidade celular ("speedMedian"). Tanto a tensão citoesquelético celular quanto a velocidade celular são quantificadas como o valor médio através dos pontos de grade dentro das células. As duas propriedades são traçadas no mesmo eixo com unidades arbitrárias para visualizar as relações entre as propriedades avaliadas. Nomes de variáveis adicionais estão listados na Tabela Suplementar S1. Clique aqui para ver uma versão maior desta figura.
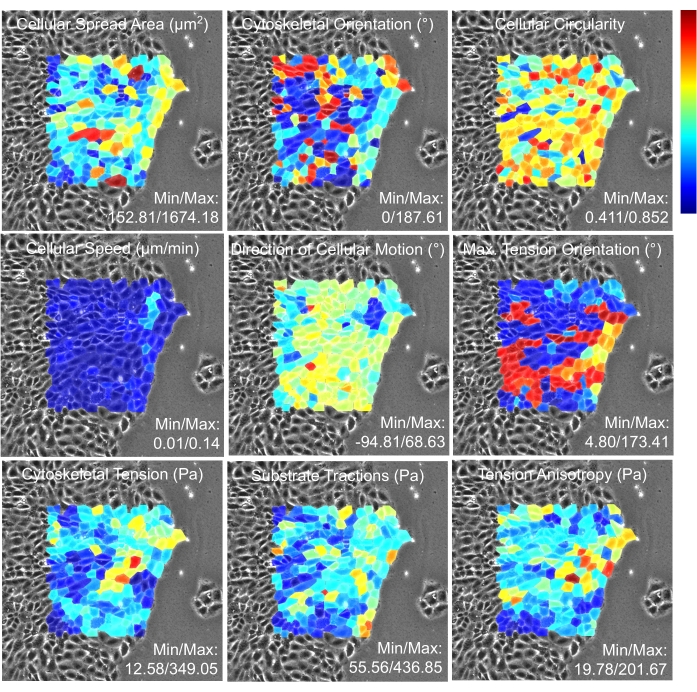
Figura 17: Mapas de calor das propriedades de células individuais através da monocamada analisada. Cada célula é colorida com o valor médio da propriedade indicada no painel. Assim, o vermelho profundo indica o valor celular máximo no espectro de cores, e o azul profundo indica o valor celular mínimo através da monocamada analisada. Como descrito em Tambe et al., PLoS One (2013)2, as células localizadas mais próximas ao limite têm forças mecânicas afetadas por propriedades desconhecidas das células fora da imagem. Assim, o mapa de calor é gerado para células distantes da fronteira. Clique aqui para ver uma versão maior desta figura.
Figura S1: Amostrar imagens da conta fluorescente superior e inferior. Clique aqui para baixar este Arquivo.
Figura S2: Uma visão detalhada do passo 2 da Figura 3. Clique aqui para baixar este Arquivo.
Figura S3: Uma visão detalhada da etapa 4 da Figura 3. Clique aqui para baixar este Arquivo.
Figura S4 A: visão detalhada da etapa 6 da Figura 3. Clique aqui para baixar este Arquivo.
Figura S5: Uma visão detalhada do passo 3 da Figura 5. Clique aqui para baixar este Arquivo.
Figura S6: Uma visão detalhada da etapa 5 da Figura 5. Clique aqui para baixar este Arquivo.
Figura S7: Uma visão detalhada da etapa 3 da Figura 6. Clique aqui para baixar este Arquivo.
Figura S8: Uma visão detalhada da saída de força célula-ECM da etapa 6 da Figura 8. Clique aqui para baixar este Arquivo.
Figura S9: Uma visão detalhada da saída da força celular da etapa 6 da Figura 8. Clique aqui para baixar este Arquivo.
Figura S10: Uma visão detalhada do passo 2 da Figura 10. Clique aqui para baixar este Arquivo.
Figura S11: Uma visão detalhada do passo 2 da Figura 11. Clique aqui para baixar este Arquivo.
Figura S12: Uma visão detalhada do passo 2 da Figura 12. Clique aqui para baixar este Arquivo.
Figura S13: Uma visão detalhada da saída da etapa 3 da Figura 12. Clique aqui para baixar este Arquivo.
Figura S14: Uma visão detalhada do passo 2 na Figura 13. Clique aqui para baixar este Arquivo.
Figura S15: Uma visão detalhada da etapa 3 da Figura 13. Clique aqui para baixar este Arquivo.
Figura S16: Uma visão detalhada dos arquivos de dados gerados na etapa 5 da Figura 14. Clique aqui para baixar este Arquivo.
Figura S17: Uma visão detalhada de uma trama gerada na etapa 5 da Figura 14. Clique aqui para baixar este Arquivo.
Figura S18: Uma visão detalhada de um mapa de calor e do arquivo contendo a faixa do espectro de cores gerada na etapa 4 da Figura 15. Clique aqui para baixar este Arquivo.
Tabela S1: Uma lista de propriedades selecionadas quantificadas pelo iTACS. Clique aqui para baixar esta Tabela.
Discussão
As células aderentes usam sinais mecânicos e químicos para sobreviver, crescer e funcionar. Uma grande variedade de softwares de microscopia otimiza a experiência do usuário na avaliação dos sinais químicos através de imagens baseadas em fluorescência. No entanto, a avaliação dos sinais mecânicos envolve capacidades que não estão disponíveis no software padrão de microscopia. Além disso, a avaliação dos sinais mecânicos é mais eficiente quando a aquisição de dados é integrada à análise de dados. A falta de uma plataforma unificada que atenda às necessidades únicas de avaliação de sinais mecânicos tem sido uma grande lacuna tecnológica na biologia celular experimental. O Kit de Ferramentas Integrativas para Analisar Sinais Celulares (iTACS) foi projetado para atender a essa lacuna. Os dois componentes do iTACS, AnViM e AcTrM, equipam os usuários com as capacidades necessárias para quantificar propriedades celulares de quatro grandes categorias: forças, movimento, morfologia e fluorescência/brilho. Nestas categorias, o iTACS é atualmente capaz de revelar mais de 50 aspectos únicos de células aderentes individuais. Esses aspectos compreendem propriedades específicas de cada categoria ampla, incluindo seu valor representativo e variabilidade em toda a célula (Tabela Suplementar S1). Por exemplo, dentro das forças, há forças de tração em todo o citoesqueleto, anisotropia dessa tensão, orientação de tensão máxima e estresse de cisalhamento em toda a interface cell-ECM que tem uma profunda influência no comportamento das células aderentes1,3,6.
Uma nova abordagem para examinar o comportamento mecânico de células individuais de uma monocamada
Células individuais de uma monocamada estão envolvidas em uma troca de sinais de natureza química e mecânica3. Estes dois tipos de sinais são transmitidos através da monocamada celular de uma maneira diferente23. No entanto, o conhecimento de transmissão de sinal mecânico fica atrás do da transmissão de sinal químico. Essa lacuna de conhecimento coincide com uma falta sustentada de abordagens simples e intuitivas para avaliar sinais mecânicos celulares. A nova abordagem de mapeamento de dados descrita aqui está equipada para preencher essa lacuna. Tal mapeamento revela que a flutuação da tensão citoesquelética intrínseca na região vizinha de uma célula serve como sinais de relaxamento, fluidização e ancoragem que regulam mudanças na forma, tamanho e velocidade da célula18. Mapas das propriedades das regiões vizinhas exibem padrões de "subdivisão multicelular", onde as células dentro da subdivisão são expostas a um microambiente relativamente uniforme e as células no limite da subdivisão são expostas a um microambiente notavelmente não-universitário18.
Acessibilidade da tecnologia de medição de força
Existe uma variedade de protocolos para fazer hidrogéis PAA, analisar a deformação de hidrogel e movimento celular, e quantificar as forças célula-ECM e células-células1,2,7,8,9,13,14,15,18,20,21,24,25,26,27 28,29,30,31,32. No entanto, esses desenvolvimentos permanecem fora do alcance dos laboratórios comuns de biologia celular e confinados a laboratórios com expertise em engenharia. Ao automatizar os aspectos técnicos dessas abordagens e integrá-las sob uma plataforma unificada e fácil de usar, o objetivo do iTACS é tornar a avaliação dos sinais mecânicos uma atividade rotineira na pesquisa e educação em biologia celular experimental.
O ImageJ permite que os usuários desenvolvam aplicativos usando abordagens que exigiriam pouco ou nenhum treinamento11. O iTACS é em grande parte construído usando abordagens simples de scripting para facilitar o desenvolvimento contínuo orientado pela comunidade. Uma grande parte do AcTrM é programado usando scripts BeanShell, e a maior parte do AnViM é programada usando ImageJ Macros. Esses scripts e orientações para implementar esses recursos no microscópio do usuário estão disponíveis através do GitHub (https://github.com/IntegrativeMechanobiologyLaboratory/iTACS).
Padronização de qualidade das imagens adquiridas
Embora as técnicas baseadas em substratos elásticos para quantificar forças físicas em células aderentes tenham sido desenvolvidas e implementadas em vários laboratórios, o protocolo ainda carece de padronização. Uma área que mais precisa ser padronizada é a qualidade das imagens de contas superiores adquiridas (Figura Suplementar S1). Questões significativas surgem da deriva em foco ao longo do experimento. Nossa nova abordagem de refoco baseada em referências de imagem faz de um processo objetivo. Os parâmetros definidos na primeira etapa da AcTrM impõem limites de qualidade objetivo necessários. Outras medidas de padronização podem ser programadas em versões futuras do AcTrM.
A ampla aplicabilidade do iTACS
Além de quantificar inúmeros aspectos das células aderentes, a estrutura do iTACS facilita seu uso para vários protocolos e necessidades experimentais. O AcTrM permite o auto-treinamento do usuário guiado por software. Imagens de alta velocidade exigidas, por exemplo, pela avaliação simultânea das flutuações citoplasmáticas de cálcio são atualmente limitadas pela velocidade de reposicionamento e refoco do hardware e é melhor feita em um local de cada vez. No entanto, a implementação atual está bem equipada para imagens de longo prazo, imagens interrompidas, onde a amostra não pode ser retida no estágio do microscópio durante toda a duração do experimento. Uma vez que as imagens de referência são adquiridas no início do experimento, o iTACS permite imagens em tempo real de sinais mecânicos, abrindo novos caminhos em aplicações de triagem de drogas. O AnViM permite que os usuários forneçam informações altamente técnicas em termos leigos. A capacidade de quantificar um amplo espectro de propriedades celulares e rastreá-las ao longo do experimento constitui capacidades críticas necessárias para descobrir novos mecanismos de comunicação intercelulares.
Para o desenvolvimento futuro do iTACS, identificamos quatro áreas focais: (1) aprimoramento da aquisição de dados e da velocidade de análise de dados, (2) implementação de abordagens para avaliar novos sinais celulares13, (3) desenvolvimento de workshops e módulos de educação na avaliação de sinais celulares baseada em iTACS, (4) desenvolvimento de soluções de automação de baixo custo.
Divulgações
Os autores não declaram conflitos de interesse.
Agradecimentos
D.T.T. agradece equipe afiliada ao Centro de Biologia Pulmonar da Universidade do Sul do Alabama por estimular discussões sobre as necessidades experimentais de pesquisa de biologia celular. Essas discussões foram cruciais para iniciar o desenvolvimento do ITACS.
Este trabalho foi apoiado em parte por doações do Instituto Nacional de Saúde/Instituto Nacional de Sangue do Coração, P01 HL66299 e R37 HL60024 (Stevens), R01-HL118334 (Alvarez), F32-HL144040-01 (Xu), e da Universidade do Sul do Alabama através do Abraham Mitchell Cancer Research Fund (Singh, Palanki, Tambe), Research and Scholarly Development Grant (Tambe), Honors College, Summer Research Fellow (Nguyen).
Materiais
| Name | Company | Catalog Number | Comments |
| Reagents and components used in prepare glass surface for hydrogel coating | |||
| (3-Aminopropyl)trimethoxysilane, 97% | Aldrich chemistry | 13822565 | |
| 2% Bis Solution | Bio-rad | 1610142 | |
| 3-(Trimethoxysilyl)propyl methacrylate,98% | Acros organics | 2530850 | |
| 40% Acrylamide Solution | Bio-rad | 1610140 | |
| Glass bottom 35 mm dish/ 6 or 12 or 24 well plates | MatTek or CellVis | ||
| Glutaraldehyde, EM Grade, 25% | Polysciences | 1909100 | |
| Sodium Hydroxide | Sigma-aldrich | 1002074706 | |
| Reagents and components used in preparing suitable hydrogel | |||
| 2% Bis Solution | Bio-rad | 1610142 | |
| 40% Acrylamide Solution | Bio-rad | 1610140 | |
| Ammonium Persulfate | Bio-rad | 1610700 | |
| Cover Slips | Electron Microscopy Sciences | 7222301 | |
| Dulbecco's Phosphate Buffered Saline (1M) | Gibco | 14190136 | |
| FluoSpheres carboxylate 0.2 um, yellow-green(505/515) | Invitrogen | F8811 | |
| FluoSpheres carboxylate 0.5 um, red(580/605) | Invitrogen | F8812 | |
| FluoSpheres carboxylate 2.0 um, red(580/605) | Invitrogen | F8826 | |
| Rain-X | |||
| TEMED | Bio-rad | 1610801 | |
| Reagents used in coating extracellular matrix on the hydrogel | |||
| Collagen Type I Rat Tail | Corning | 354236 | |
| HEPES(1M) | Gibco | 15630080 | |
| Phosphate Buffered Saline (1M) | Gibco | 10010023 | |
| Sulfo-SANPAH | CovaChem | 102568434 | |
| Microscope hardware used in the current study | |||
| Camera | Hamamatsu Flash 4.0 LT sCMOS Camera | C11440-42U | |
| H117 ProScanTM Stages | Prior Scientific | ||
| Light source- Lambda DG4 and Lambda DG5 | Sutter instrument company | ||
| Microscope | Nikon eclipse TE2000-S | 550372 | |
| ProScan III Universal Microscope Automation Controller | Prior Scientific | ||
| Stagetop incubator | ibidi | 11922 | |
| Stepper Motor Focus Drive | Prior Scientific |
Referências
- Tambe, D. T., et al. Collective cell guidance by cooperative intercellular forces. Nature Materials. 10 (6), 469-475 (2011).
- Tambe, D. T., et al. Monolayer stress microscopy: limitations, artifacts, and accuracy of recovered intercellular stresses. PloS One. 8 (2), 55172 (2013).
- Das, T., et al. A molecular mechanotransduction pathway regulates collective migration of epithelial cells. Nature Cell Biology. 17 (3), 276-287 (2015).
- Vedula, S. R., et al. Mechanics of epithelial closure over non-adherent environments. Nature Communications. 6, 6111 (2015).
- Lamason, R. L., et al. Rickettsia Sca4 reduces vinculin-mediated intercellular tension to promote spread. Cell. 167 (3), 670-683 (2016).
- Sunyer, R., et al. Collective cell durotaxis emerges from long-range intercellular force transmission. Science. 353 (6304), 1157-1161 (2016).
- Dong, L., Oberai, A. A. Recovery of cellular traction in three-dimensional nonlinear hyperelastic matrices. Computer Methods in Applied Mechanics and Engineering. 314, 296-313 (2017).
- Nier, V., et al. Kalman inversion stress microscopy. Biophysical Journal. 115 (9), 1808-1816 (2018).
- Serrano, R., et al. Three-dimensional Monolayer Stress Microscopy. Biophysical Journal. 117 (1), 111-128 (2019).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ: 25 years of Image Analysis. Nature Methods. 9 (7), 671-675 (2012).
- Schindelin, J., et al. Fiji - an Open Source platform for biological image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Edelstein, A., Amodaj, N., Hoover, K., Vale, R., Stuurman, N. Computer control of microscopes using microManager. Current Protocols in Molecular Biology. , 20 (2010).
- Patel, N. G., et al. Unleashing shear: Role of intercellular traction and cellular moments in collective cell migration. Biochemical and Biophysical Research Communications. 522 (2), 279-285 (2020).
- Yeung, T., et al. Effects of substrate stiffness on cell morphology, cytoskeletal structure, and adhesion. Cell Motility and the Cytoskeleton. 60 (1), 24-34 (2005).
- Trepat, X., et al. Physical forces during collective cell migration. Nature Physics. 5, 426-430 (2009).
- Aratyn-Schaus, Y., Oakes, P. W., Stricker, J., Winter, S. P., Gardel, M. L. Preparation of complaint matrices for quantifying cellular contraction. Journal of Visualized Experiments: JoVE. (46), (2010).
- King, J., et al. Structural and functional characteristics of lung macro- and microvascular endothelial cell phenotypes. Microvascular Research. 67 (2), 139-151 (2004).
- Patel, G., et al. Mechanical signaling in a pulmonary microvascular endothelial cell monolayer. Biochemical and Biophysical Research Communications. 519 (2), 337-343 (2019).
- Kähler, C. J., et al. Main results of the 4th International PIV Challenge. Experiments in Fluids. 57 (6), 97 (2016).
- Tseng, Q., et al. Spatial organization of the extracellular matrix regulates cell-cell junction positioning. Proceedings of the National Academy of Sciences of the United States of America. 109 (5), 1506-1511 (2012).
- Alvarez-Gonzalez, B., et al. Two-layer elastographic 3-D traction force microscopy. Scientific Reports. 7, 39315 (2017).
- Makarchuk, S., Beyer, N., Gaiddon, C., Grange, W., Hebraud, P. Holographic traction force microscopy. Scientific Reports. 8 (1), 3038 (2018).
- Janmey, P. A., Miller, R. T. Mechanisms of mechanical signaling in development and disease. Journal of Cell Science. 124 (1), 9-18 (2011).
- Dembo, M., Oliver, T., Ishihara, A., Jacobson, K. Imaging the traction stresses exerted by locomoting cells with the elastic substratum method. Biophysical Journal. 70 (4), 2008-2022 (1996).
- Dembo, M., Wang, Y. L. Stresses at the cell-to-substrate interface during locomotion of fibroblasts. Biophysical Journal. 76 (4), 2307-2316 (1999).
- Butler, J. P., Tolic-Norrelykke, I. M., Fabry, B., Fredberg, J. J. Traction fields, moments, and strain energy that cells exert on their surroundings. American Journal of Physiology: Cell Physiology. 282 (3), 595-605 (2002).
- Saez, A., et al. Traction forces exerted by epithelial cell sheets. Journal of Physics. Condensed Matter. 22 (19), 194119 (2010).
- Deforet, M., et al. Automated velocity mapping of migrating cell populations (AVeMap). Nature Methods. 9 (11), 1081-1083 (2012).
- Polio, S. R., Smith, M. L. Patterned hydrogels for simplified measurement of cell traction forces. Methods in Cell Biology. 121, 17-31 (2014).
- Toyjanova, J., et al. 3D Viscoelastic traction force microscopy. Soft Matter. 10 (40), 8095-8106 (2014).
- Serra-Picamal, X., Conte, V., Sunyer, R., Munoz, J. J., Trepat, X. Mapping forces and kinematics during collective cell migration. Methods in Cell Biology. 125, 309-330 (2015).
- Charrier, E. E., et al. A novel method to make viscoelastic polyacrylamide gels for cell culture and traction force microscopy. APL Bioengineering. 4 (3), 036104 (2020).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados