Method Article
Integratives Toolkit zur Analyse zellulärer Signale: Kräfte, Bewegung, Morphologie und Fluoreszenz
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Die Plattform Integrative Toolkit to Analyze Cellular Signals (iTACS) automatisiert den Prozess der gleichzeitigen Messung einer Vielzahl von chemischen und mechanischen Signalen in adhärente Zellen. iTACS wurde entwickelt, um die Community-getriebene Entwicklung zu erleichtern und es Forschern zu ermöglichen, alle Plattformfunktionen unabhängig von ihrem Bildungshintergrund zu nutzen.
Zusammenfassung
Die quantitative Bewertung zellulärer Kräfte und Bewegungen hat sich in den letzten vier Jahrzehnten erheblich weiterentwickelt. Diese Fortschritte bildeten den Rahmen, um aufschlussreiche mechanische Signalprozesse in Zellkultursystemen zu untersuchen. Derzeit steht das Feld jedoch vor drei Problemen: mangelnde Qualitätsstandardisierung der erfassten Daten, technische Fehler bei der Datenanalyse und -visualisierung, und vielleicht am wichtigsten ist, dass die Technologie für gängige zellbiologische Labors weitgehend unerreichbar bleibt. Um diese Einschränkungen zu überwinden, haben wir eine neue experimentelle Plattform entwickelt - Integrative Toolkit to Analyze Cellular Signals (iTACS). iTACS besteht aus zwei Komponenten: Acquisition and Training Module (AcTrM) und Analysis and Visualization Module (AnViM). AcTrM basiert auf μManager - einer NIH-ImageJ-basierten Mikroskopsteuerungssoftware - und erleichtert das Selbsttraining des Benutzers und die Automatisierung gängiger Bilderfassungsprotokolle. AnViM basiert auf NIH-ImageJ und ermöglicht eine benutzerfreundliche Automatisierung der Datenanalyse und aufschlussreiche Visualisierung der Ergebnisse. Diese Experimente beinhalten die Kultivierung adhärenter Zellen auf Hydrogelen, die Abbildung von in das Hydrogel eingebetteten fiduzialen Markern und schließlich die Extraktion einer umfassenden mechanischen Charakterisierung der Zellen aus diesen Bildern. Derzeit ermöglicht iTACS dem Benutzer, eine Vielzahl von Eigenschaften zu analysieren und zu verfolgen, einschließlich Morphologie, Bewegung, Zytoskelettkräfte und Fluoreszenz einzelner Zellen und ihrer Nachbarregion. Das Problem der Qualitätsstandardisierung wurde in AcTrM mit einer referenzbildgeführten Refokussierungstechnik behandelt. Die technischen Fragestellungen in der Datenanalyse wurden in AnViM mit einem mehrgleisigen Bildsegmentierungsverfahren, einem benutzerfreundlichen Ansatz zur Identifizierung von Randbedingungen und einer neuartigen zellulären eigenschaftsbasierten Datenvisualisierung adressiert. AcTrM wurde entwickelt, um die einfache Umwandlung von grundlegenden Fluoreszenzmikroskopen in experimentelle zellmechanische Rigs zu erleichtern, und AnViM ist so ausgestattet, dass Benutzer zelluläre mechanische Signale messen können, ohne einen technischen Hintergrund zu benötigen. iTACS wird der Forschungsgemeinschaft als Open-Source-Suite mit Community-gesteuerten Entwicklungsfunktionen zur Verfügung stehen.
Einleitung
Häufig verwendete optische Bildgebungs- und Datenanalysetools verwenden Hardware- und Softwaretechnologien, die nahezu veraltet sind. Die Verzögerung bei der Übersetzung und Implementierung von Fortschritten bei elektronischen Geräten, Computeransätzen und mathematischen Analysen in gängige experimentelle zellbiologische Werkzeuge ist eine haupt einschränkung des wachstumstempos unseres wissens über die zellphysiologische physiologie. Derzeit finden zellbiologische Forscher molekularbiologische Werkzeuge in Reichweite, aber Werkzeuge, die auf technischen Prinzipien basieren, sind unerreichbar. Ein solches auf technischen Prinzipien basierendes Werkzeug ist die Monolayer Stress Microscopy (MSM)1,2. Während MSM in verschiedenen Labors weltweit angepasst und untersucht wurde, ist seine Verwendung in erster Linie auf Labore mit technischem Know-how beschränkt3,4,5,6,7,8,9.
NIH-ImageJ ist eines der beliebtesten Open-Source-Tools unter Zellbiologieforschern10. Beitragsgesteuerte Fortschritte in der Benutzergemeinschaft waren von zentraler Bedeutung für seine Popularität11,12. ImageJ verfügt über Funktionen, mit denen Benutzer Anwendungen mit einer Mischung aus einer erweiterten Programmiersprache und vereinfachten Skriptansätzen entwickeln können. Diese Funktionen erleichtern es Benutzern mit grundlegenden Programmierkenntnissen, jeden neuen Beitrag zur Software zu implementieren, anzupassen und voranzutreiben. Aufbauend auf diesen Qualitäten von NIH-ImageJ haben wir das Integrative Toolkit to Analyze Cellular Signals (iTACS) entwickelt, das eine kostengünstige Integration gewünschter Hardware- und Softwarewerkzeuge ermöglicht, um die Messung einer Vielzahl von chemischen und mechanischen Signalen über adhärente Zellen hinweg zu automatisieren11,12.
iTACS besteht aus zwei Komponenten: Acquisition and Training Module (AcTrM) und Analysis and Visualization Module (AnViM). AcTrM basiert auf μManager - einer NIH-ImageJ-basierten Bilderfassungsanwendung -, mit der Benutzer Zeitraffermessungen herkömmlicher optischer Eigenschaften und einer Vielzahl physikalischer Eigenschaften von adhäsiven Zellen in mehreren Proben einrichten können12. AcTrM erleichtert die Benutzerschulung durch prägnante Anweisungen, die in der grafischen Benutzeroberfläche enthalten sind. Darüber hinaus verfügt es über eine neuartige Funktion des Referenzbild-basierten Autofokus, der Echtzeitmessungen physikalischer Kräfte erleichtern und eine Qualitätsstandardisierung der erfassten Daten ermöglichen soll.
AnViM basiert auf ImageJ-Plugins, beschleunigter Software und Dateihandhabungsskripten, mit denen Benutzer mehr als 50 Eigenschaften quantitativ bewerten können, einschließlich zellulärer Form, Größe, Ausrichtung, Geschwindigkeit und Bewegungsrichtung, Traktionen auf der extrazellulären Matrix (ECM) und benachbarten Zellen, kontraktilen und Schermomenten sowohl einzelner adhärenter Zellen als auch ihrer Nachbarregion. AnViM ermöglicht es Anwendern, die zellulären physikalischen Eigenschaften zu quantifizieren, ohne den zugrunde liegenden technischen Hintergrund zu beherrschen11. Darüber hinaus ermöglicht es die Datenanalyse in einem interaktiven oder Batch-Verarbeitungsmodus. Es generiert Heatmaps, die räumliche Variationen aufdecken, und Diagramme, die die zeitliche Variation der Eigenschaften einzelner Zellen zeigen.
In einem typischen Experiment kultiviert der Anwender Zellen auf einem elastischen Hydrogel mit entsprechenden extrazellulären Matrixproteinen auf der Oberseite und zwei Arten von eingebetteten fluoreszierenden Markern. Im Wesentlichen reichen Bilder dieser fluoreszierenden Marker vor und nach der Kultivierung der Zellen aus, um die Kräfte innerhalb und um einzelne Zellen zu quantifizieren2,13. AnViM bildet diese Ergebnisse auf einzelne Zellen des adhärenten Clusters ab und generiert aufschlussreiche Bilder und Graphen.
Protokoll
HINWEIS: Proben, die mit der iTACS-Plattform untersucht werden, sind Zellen, die auf ein weiches Substrat geklebt sind. Das Protokoll zur Bewertung mechanischer und chemischer Signale ist in zwei aufeinanderfolgende Teile unterteilt: Acquisition and Training Module (AcTrM) und Analysis and Visualization Module (AnViM).
1. Akquisitions- und Trainingsmodul (AcTrM)
HINWEIS: AcTrM automatisiert den Prozess der Datenerfassung und des Selbsttrainings der Benutzer. Bereiten Sie vor jeder Datenerfassung ein weiches Substrat vor, das in der Lage ist, Informationen zu liefern, die zur Quantifizierung der Kräfte erforderlich sind, die Zellen darauf ausüben.
- Hydrogel-Vorbereitung
HINWEIS: Das Ziel hier ist die Herstellung eines 1.250 Pa-Schermoduls, einer Dicke von etwa 100 μm und eines Polyacrylamid-Hydrogels (PAA) mit einem Durchmesser von 22 mm.- Polyacrylamidlösung nach der Methode von Yeung et al. herstellen und die Hydrogele nach den von Trepat et al.14,15 beschriebenen Schritten gießen. Eine Ausnahme von dem Verfahren ist, dass die unmittelbar unter den Zellen eingebetteten Kügelchen einen Durchmesser von 0,5 μm haben und gelbe Fluoreszenz emittieren.
- Überspringen Sie den Schritt, 2 μm Perlen am Deckglas anzubringen, wenn erwartet wird, dass der Betrachtungsbereich einen großen zellfreien Bereich aufweist.
HINWEIS: Das Hydrogel-Vorbereitungsprotokoll ist inzwischen in diesem Bereich ziemlich etabliert16. In der nachfolgenden Beschreibung werden die 0,5 μm Perlen als "obere Perlen" und die 2 μm Perlen als "untere Perlen" bezeichnet. Die unteren Perlen sind jedoch optional, wenn der abgebildete Bereich einen großen zellfreien Bereich enthält. Das obere Perlenmuster aus einer solchen zellfreien Region dient dem Zweck des unteren Perlenmusters. - Montieren Sie das Hydrogel mit eingebetteten fluoreszierenden Kügelchen auf dem Mikroskoptisch.
- Warten Sie 15-20 min, bis die Temperatur der Platte einen stationären Zustand erreicht.
HINWEIS: Die Oberseite der Platte ist mit extrazellulären Matrixproteinen funktionalisiert, aber die Zellen sind noch nicht auf dem Hydrogel ausgesät.
- Referenzbilderfassung
- Teil 1: Anlegen einer Positionsliste
HINWEIS: Die manuelle Eingabe in diesem Schritt umfasst das Befolgen der AcTrM-Anweisungen, um Standorte mit der besten Perlenverteilung auszuwählen. In diesem Schritt werden keine zuvor erstellten Dateien verwendet. Zu den wichtigsten neuen Ordnern, die in diesem Schritt erstellt werden, gehören die Ordner "t0imgs", "tnimgs" und "textfiles", und die erzeugte Datei enthält die Positionslistendatei. In Abbildung 2 finden Sie eine visuelle Beschreibung der folgenden Schritte, die die Erstellung einer Positionsliste mit AcTrM leiten. Das demonstrierte Beispiel erfasst Daten bei 20-facher Vergrößerung.- Starten Sie μManager 2.0 - Beta.
- Führen Sie AcTrM aus, indem Sie im Fenster IMBL-Ressourcen auf die Schaltfläche AcTrM.bsh klicken.
- Wählen Sie im Fenster Erfassungseigenschaften definieren das geeignete Ziel, die Toleranz für die Genauigkeit der Wiederherstellung der lateralen Position (d. h. XY), die Schrittgröße des groben und verfeinerten Refokussierungsvorgangs (d. h. z) und den Wert des akzeptablen Bildkorrelationskoeffizienten für die Fokuswiederherstellung aus.
HINWEIS: Eine feinere Toleranz für die Wiederherstellung der lateralen Position verlangsamt die Positionswiederherstellung, aber eine wesentliche Toleranz erfordert eine Positionskorrektur während der Datenanalyse und kann Schwierigkeiten bei der Fokuswiederherstellung verursachen. Eine kleinere Schrittgröße verlangsamt den Refokussierungsvorgang, aber eine sehr hohe Schrittgröße kann zu einer schnellen Bewegung führen und den Fokus verfehlen. - Wählen Sie im Fenster AcTrM - Schritt 0 die Option Frische Akquisition aus, um eine Positionsliste zu erstellen.
HINWEIS: Das Fenster mit dem Titel AcTrM - Schritt 1 listet alle wichtigen nächsten Schritte auf. Diese Schritte umfassen das Verschieben der Bühne, das Anpassen des Fokus und das Klicken auf Schaltflächen im μManager. Diese Schritte führen zur Erstellung der Liste der für die Datenerfassung geeigneten Positionen. - Klicken Sie auf Live in Micromanager , um die Beispiele für manuelle Anpassungen zu visualisieren, die im Fenster AcTrM - Schritt 1 aufgeführt sind.
- Führen Sie die in diesem Fenster aufgeführten Schritte aus.
- Erwerben Sie nun das Bild der Perlen. Wählen Sie den geeigneten Kanal für die oberen Perlen aus.
- Achten Sie darauf, auch die unteren Perlen zu betrachten. Wählen Sie dazu die Kanäle für die unteren Perlen aus. Ein verschwommenes Bild von unteren Perlen ist zu sehen.
HINWEIS: Kanäle können über das voreingestellte Dropdown-Menü im μManager-Hauptfenster umgeschaltet werden. Wenn die unteren Perlen im Experiment verwendet werden, stellen Sie sicher, dass sie an der ausgewählten Position vorhanden sind. Diese Perlen erscheinen verschwommen. - Klicken Sie nach auswahl der entsprechenden Position im Fenster Bühnenpositionsliste auf Markieren, um die Position zu speichern.
HINWEIS: Die beste Position wird durch eine dichte und gleichmäßige Verteilung der oberen Perlen definiert, die unmittelbar unter der oberen Oberfläche eingebettet sind (d. h. obere Perlen) und mindestens zwei Perlen, die am Deckglas befestigt sind (d. h. untere Perlen) (ergänzende Abbildung S1). Die Wiederherstellung des Fokus ist schneller, wenn die unteren Perlen als große, unscharfe Ringe erscheinen. Wenn Experimente jedoch in einer Position durchgeführt werden, ohne dass ein Fokus oder eine seitliche Positionswiederherstellung erforderlich ist, ignorieren Sie die Anweisungen im Zusammenhang mit dem Bild der unteren Perlen. - Führen Sie die in Abbildung 2 beschriebenen Schritte 6 bis 9 aus, um zusätzliche Positionen in die Liste aufzunehmen.
HINWEIS: Wählen Sie ein paar zusätzliche Positionen, damit eine Eliminierungsrunde die besten Positionen auf der Platte ermöglichen kann.
- Teil 2: Aufnahme von Referenzbildern
HINWEIS: Dieser Schritt beinhaltet keine manuelle Eingabe. Zu den zuvor erstellten Schlüsseldateien, die in diesem Schritt verwendet werden, gehören 'textfiles/*.pos'. Zu den wichtigsten neuen Dateien und Ordnern, die in diesem Schritt erstellt werden, gehören die Dateien und Ordner in den Ordnern "t0imgs" und "tnimgs". Siehe Abbildung 3 und ergänzende Abbildung S2 für die visuelle Beschreibung der folgenden Schritte, die die Erfassung von Referenzbildern mit AcTrM steuern. Das Fenster AcTrM - Schritt 2 listet auch alle wichtigen Schritte auf.- Befolgen Sie die im Fenster AcTrM - Schritt 2 aufgeführten Schritte und treffen Sie die Auswahlmöglichkeiten im Fenster Mehrdimensionale Erfassung. Um beispielsweise einen langen Zeitablauf durchzuführen, geben Sie eine Anzahl von Bildern an, die aufgenommen werden sollen, wählen Sie den ersten Kanal als Phasenkanal aus, und wählen Sie dann den nächsten für die oberen Perlen und den übernächsten für die unteren Perlen aus. Klicken Sie im Fenster Mehrdimensionale Erfassung auf Schließen und dann im Fenster ActrM - Schritt 2 auf OK.
- Wählen Sie im Fenster AcTrM - Schritt 3 aus, ob die referenzbildbasierte XYZ-Positionswiederherstellung aktiviert werden soll. Die Wahl ist in der Regel ja. Wenn Bilder jedoch an einer Position ohne Platz für Fokus- oder Bühnendrift aufgenommen werden, ist die Wahl im Fenster AcTrM - Schritt 3 Nein.
- Legen Sie im Fenster AcTrM - XYZ-Wiederherstellungsoptionen den Kanal für die grobe XYZ-Wiederherstellung fest, ob eine verfeinerte XYZ-Wiederherstellung durchgeführt werden soll, die Region und den Kanal für die verfeinerte XYZ-Wiederherstellung sowie die Richtung, mit der Neufokussierung zu beginnen (Ergänzende Abbildung S4). Nach Fertigstellung erfasst AcTrM Referenzbilder.
HINWEIS: Typische Optionen für den Kanal sind der untere Perlenkanal. Die XYZ-Wiederherstellung funktioniert am besten, wenn sie mit dem vollständigen Abbild in der aktuellen Implementierung ausgeführt wird. Referenzbilder enthalten typischerweise ein Durchlichtbild des Hydrogels, ein fluoreszierendes Bild der oberen Perlen und ein fluoreszierendes Bild der unteren Perlen. Der Inhalt jedes Bildsatzes kann je nach auswahl der im Fenster " Mehrdimensionale Erfassung" getroffenen Optionen variieren. Dennoch erfasst die Software den Referenzbildsatz an jeder in Abbildung 2 festgelegten Position. Am Ende dieses Schritts werden drei Ordner im ausgewählten Verzeichnis erstellt: 't0imgs', 'tnimgs' und 'textfiles'. Der Ordner 't0imgs' enthält Referenzbilder in dem format, das von μManager erkannt und in den nachfolgenden Schritten verwendet wird; Der Ordner 'tnimgs' enthält separate TIFF-Bilder mit den Dateinamen 'c0_p0_t0.tif', 'c1_p0_t0.tif' usw. Hier steht 'c' für den Kanal, 'p' für die Position und 't' für die Zeit. Nach diesen Buchstaben folgen die Kanalnummer, die Positionsnummer bzw. die Framenummer. Der Ordner 'textfiles' enthält die Positionsliste im XML-Format und Benutzerauswahlmöglichkeiten für die Bilderfassung und Positionswiederherstellung.
- Teil 1: Anlegen einer Positionsliste
- Zellaussaat und -wachstum
HINWEIS: Die iTACS-Plattform verfügt über die Flexibilität, Probenvorbereitungsprotokolle zu berücksichtigen, die bei der allgemeinen In-vitro-Bewertung des mechanischen Verhaltens von adhärenten Zellen verwendet werden, einschließlich spärlicher Zellen, vollständig konfluenter Monoschicht, Netzwerkbildungsassay und Monoschichten mit Löchern oder signifikanten Lücken.- Kultur pulmonale mikrovaskuläre Endothelzellen der Ratte bis zum Zusammenfluss in einem Kolben17,18.
- Trennen Sie die Zellen mit Trypsin. Resuspendieren Sie die Zellen in einem Kulturmedium, das 10% fötales Rinderserum enthält, auf eine Konzentration von 1 x 106 Zellen/ml.
- Legen Sie ein 5-μL-Tröpfchen der resuspendierten Zellen auf eine teilweise trockene Hydrogeloberfläche und legen Sie es in den Zellkultur-Inkubator.
HINWEIS: Nach 2 Tagen in dem Kulturmedium, das 10% fötales Rinderserum enthält, bilden die Zellen in diesem Tröpfchen eine überfüllte Zellinsel1.
- Automatisierte Bildaufnahme für das verbleibende Experiment
HINWEIS: Die manuelle Eingabe für diesen Schritt umfasst das Befolgen der AcTrM-Eingabeaufforderungen, um die Bildaufnahme fortzusetzen. Zu den wichtigsten zuvor erstellten Dateien, die in diesem Schritt verwendet werden, gehören 'textfiles/*.pos' und die unteren Perlenbilddateien im Ordner 't0imgs'. Zu den wichtigsten neuen Dateien, die in diesem Schritt erstellt werden, gehören aktualisierte Positionslistendateien und Bilder 'tnimgs/*_t*.tif'.- Stellen Sie sicher, dass das Umweltkontrollsystem des Mikroskops stabile Gewebekulturbedingungen erreicht.
- Montieren Sie die Platte mit den kultivierten Zellen vorsichtig auf dem Mikroskoptisch.
- Warten Sie 15-20 min, bis sich Temperatur und Luftfeuchtigkeit stabilisiert haben.
HINWEIS: In Abbildung 4 finden Sie eine visuelle Beschreibung der folgenden Schritte, die die Aufnahme von Bildern für das verbleibende Experiment mit AcTrM leiten. - Starten Sie μManager 2.0 - Beta.
- Führen Sie AcTrM aus, indem Sie im Fenster IMBL-Ressourcen auf die Schaltfläche AcTrM.bsh klicken.
HINWEIS: Ignorieren Sie die im Fenster Akquisitionseigenschaften definieren getroffenen Auswahlmöglichkeiten, verwenden Sie jedoch die im vorherigen Schritt getroffenen Auswahlmöglichkeiten. - Wählen Sie im Fenster AcTrM - Schritt 0 die Option Angehaltene Erfassung fortsetzen aus.
- Wählen Sie im vorherigen Schritt die Vergrößerung und das Verzeichnis aus, die im Fenster Mehrdimensionale Erfassung identifiziert wurden und in dem die Datenordner ('t0imags', 'tnimgs' und 'textfiles') gespeichert wurden.
HINWEIS: Der Titel des Fensters führt den Benutzer zur Auswahl des entsprechenden Verzeichnisses. - Wählen Sie im Fenster Platten neu positionieren (Repositioning the Plate ) die Auswahlmöglichkeiten für die Neupositionierung der Platte (drei nichtkollineare Positionen) zusammen mit dem Kanal aus, der für die Neupositionierung verwendet wird.
HINWEIS: Gespeicherte Bilder werden in der Kamera angezeigt. Wenn überlappende Bilder als rotes, grünes und schwarzes Bild angezeigt werden, führen Sie eine manuelle Anpassung durch. - Klicken Sie auf Akzeptieren , um mit dem Erwerb fortzufahren.
HINWEIS: Dieser Schritt überwindet die leichten Verschiebungen von der Unreproduzierbarkeit der Plattenausrichtung, wenn die Platte wieder auf dem Mikroskoptisch montiert wird. Befolgen Sie die AcTrM-Anweisungen, um die Ausrichtung an jeder der drei Positionen zu beschleunigen, indem Sie ein zusammengesetztes Bild des rot dargestellten Referenzbildes (typischerweise der unteren Perlen) und des Bildes anzeigen, das derzeit durch das grün dargestellte Objektiv beobachtet wird. Das Grün ausreichend nahe an Rot zu bringen, lässt weniger Arbeit für die Software übrig. Wenn die Überlappung perfekt ist, erscheint das zusammengesetzte Bild gelb und schwarz. Ausreichend nah ist typischerweise, wenn die gleichen unteren Perlen sowohl in roten als auch in grünen Bildern sichtbar sind und die entsprechenden roten und grünen unscharfen Ringe berühren. Nachdem die Neupositionierung der Platte abgeschlossen ist, bringt AcTrM die Bühne zu jeder ausgewählten Position und stellt die XYZ-Position wieder her, indem das Referenzbild mit dem abgeglichen wird, was gerade durch die Kamera gesehen wird. Die erste laterale (XY) Position wird abgeglichen, und dann wird der Fokus (Z) abgeglichen. Auf die grobe Erholung folgt eine verfeinerte Wiederherstellung der Position und des Fokus. Aktualisierungen des aktuellen Status des Experiments werden auf dem Bildschirm über ein vierteiliges Fenster angezeigt, das in ergänzender Abbildung S3 dargestellt ist. Die aufgenommenen Bilder werden im Ordner 'tnimgs' nach '*_t1.tif', '*_t2.tif' gespeichert, wobei die Zeitanzahl für das Bildset angegeben wird. Wenn die XYZ-Wiederherstellung eine aktualisierte Position identifiziert, wird die neue Positionsliste generiert und im Ordner "Textdateien" gespeichert.
2. Analyse- und Visualisierungsmodul (AnViM)
- Einrichten der automatisierten Datenanalyse
HINWEIS: Die manuelle Eingabe in diesem Schritt umfasst die Identifizierung des Speicherorts des Ordners "tnimgs". Zu den wichtigsten zuvor erstellten Dateien, die in diesem Schritt verwendet werden, gehören 'tnimgs/*.tif'. Zu den wichtigsten neuen Dateien und Ordnern, die in diesem Schritt erstellt werden, gehören der Ordner "analysis" im selben übergeordneten Ordner wie 'tnimgs' und Ordner für jede Position 'analysis/p*'. Innerhalb jedes 'analysis/p*' erstellt dieser Schritt eine neue '*.tif' Bilddateien in den Ordnern 'phs' und 'tny'. Diese Bilder sind beschnittene und entdriftete Bilder von Zellen und oberen Perlen. Zu den anderen erstellten Dateien gehören 'analysis/analysisChoices.txt', die Analyseoptionen auflistet, 'analysis/p*/skipAnalysis.txt', die Fälle auflistet, in denen die Analyse aufgrund einer signifikanten bereits bestehenden Drift übersprungen wird, und 'analysis/p*/part_shift_values_pixel_degree.dat', die geschätzte Driftwerte auflistet. AnViM ändert die Rohdaten im Ordner 'tnimgs' nicht. In Abbildung 5 finden Sie eine visuelle Beschreibung der folgenden Schritte, die die Einrichtung der automatisierten Datenanalyse mit AnViM leiten.- Starten Sie die Fidschi-Software und wählen Sie die erste Option im MSM-Dropdown-Menü mit der Bezeichnung MSM - Vorverarbeitung.
- Wählen Sie im Dialogfeld des Dateibrowsers den Ordner aus, der den Ordner "tnimgs" enthält, der die analysierten Daten enthält.
- Definieren Sie den Kanal für die Bilder. Identifizieren Sie gemäß den Richtlinien aus der ergänzenden Abbildung S5 die Kanalnummern des Durchlichtbildes der Zellen (Phasenkontrastbild der Zellen), des unteren Perlenbildes und des oberen Perlenbildes. Die folgenden drei Kontrollkästchen ("Dateien in den Positionsordner verschieben", "Zusätzliche Korrektur für starre Bewegungen vornehmen" und "Daten zuschneiden und im Positionsordner speichern") sind in der Regel aktiviert. Definieren Sie schließlich die Toleranz für die Ablehnung stark gedrifteter Daten, die Pixelgröße und die Seiten des Bildes, an denen sich die Zellen kreuzen.
- Reagieren Sie auf die Aufforderung, die Helligkeit und den Kontrast der unteren Perlenbilder zu verbessern, damit die Perlen prominent angezeigt werden. Passen Sie dies über den Schieberegler im Menü an und klicken Sie auf OK.
- Führen Sie nun eine Positionskorrektur durch, um die Verschiebungen, falls vorhanden, loszuwerden. Sobald Sie fertig sind, wird ein Analyseordner erstellt.
HINWEIS: Eine Kontrastverstärkung ist in der Regel nicht erforderlich, aber diese Bestimmung ist für Experimente verfügbar, bei denen die Exposition gegenüber Lasern minimiert werden muss. Nach dieser Auswahl kopiert AnViM die Dateien aus dem Ordner 'tnimgs' in den Ordner 'analysis'. Die Dateien, die jeder Position entsprechen, werden in den Ordnern 'analysis/p0, analysis/p1' usw. gespeichert. Innerhalb jedes dieser Positionsordner erstellt AnViM die Ordner "cels", "defs" und "refs", die das ursprüngliche Durchlichtbild, die Bilder der oberen Perlen und die Bilder der unteren Perlen enthalten (ergänzende Abbildung S6). AnViM analysiert dann starre Bewegungen in den Bildern der unteren Perlen und erstellt einen "phs" -Ordner, der ein korrigiertes Durchlichtbild der Zellen und einen "tny" -Ordner mit aktualisierten oberen Perlenbildern enthält. Schließlich werden die Benutzerauswahl der Kanäle, Operationen, Toleranz, Pixelgröße und Grenzziehung in der Datei "analysisChoices.txt" gespeichert (ergänzende Abbildung S6).
- Quantifizierung der Verformung von Hydrogel und Monoschicht
HINWEIS: Es ist wichtig zu beachten, dass die Quantifizierung der Bewegung aus einer Bildsequenz ein sich schnell entwickelndes Feld ist19. Die Technologie wird ständig für Funktionen optimiert, einschließlich Geschwindigkeit, Genauigkeit, spezifische Merkmale in den Rohbildern und spezifische Verformungsmuster. Daher ist es wahrscheinlich, dass einige Benutzer einen anderen Deformationsquantifizierungsansatz als den hier vorgestellten verwenden.-
Teil 1: Quantifizierung der Hydrogelverformung
HINWEIS: Zu den manuellen Eingaben in diesem Schritt gehören die Identifizierung des Datenverzeichnisses und die Auswahl der Rasterauflösung. Zu den wichtigsten zuvor erstellten Dateien, die in diesem Schritt verwendet werden, gehören 'p*/tny/*.tif'. Zu den wichtigsten neuen Dateien, die dabei erzeugt werden, gehören "p * / displacement / * _disp.dat", das Verschiebungsvektoren der oberen Oberfläche des Hydrogels auflistet. In Abbildung 6 finden Sie eine visuelle Beschreibung der folgenden Schritte, um über AnViM die Particle Image Velocimetry-Analyse auf den oberen Beads-Bildern zu aktivieren20.- Starten Sie die Fiji-Software, und wählen Sie im Dropdown-Menü MSM die Option MSM - Gel Deformation aus.
- Wählen Sie hier die Option aus, die für das Experiment geeignet ist.
- Wählen Sie im Dialogfeld des Dateibrowsers das übergeordnete Verzeichnis des Ordners "Analyse" aus, der die analysierten Daten enthält.
- Wählen Sie im Fenster Parameter für die Berechnung der Gelverformung die entsprechende Querkorrelationsfenstergröße, den Geräuschpegel und den Schwellenwert aus (Ergänzende Abbildung S7)20.
HINWEIS: Die Ergebnisse werden in einem neu erstellten 'Displacement'-Verzeichnis innerhalb jedes Positionsordners 'analysis/p0, analysis/p1' usw. gespeichert. Hier werden alle Ausgabedateien gespeichert.
-
Teil 2: Quantifizierung der Monolayer-Verformung
HINWEIS: Zu den manuellen Eingaben in diesem Schritt gehören die Identifizierung des Datenverzeichnisses und die Auswahl der Rasterauflösung. Zu den wichtigsten zuvor erstellten Dateien, die in diesem Schritt verwendet werden, gehören 'p*/tny/*.tif'. Zu den wichtigsten neuen Dateien, die dabei erzeugt werden, gehören "p * / velocity / * _vel.dat", die Bewegungsvektoren der Zellen auflistet. In Abbildung 7 finden Sie eine visuelle Beschreibung der Schritte, um über AnViM die Particle Image Velocimetry-Analyse auf den oberen Perlenbildern zu aktivieren20. Das Verfahren ähnelt dem zur Quantifizierung der Hydrogelverformung und wählt MSM - Cellular Motion aus dem MSM-Dropdown-Menü aus. Die Ergebnisse werden in einem neu erstellten 'velocity'-Verzeichnis innerhalb jedes Positionsordners 'analysis/p0', 'analysis/p1' usw. gespeichert.
-
Teil 1: Quantifizierung der Hydrogelverformung
- Quantifizierung von Zell-ECM und Zell-Zell-Kräften
HINWEIS: Die manuelle Eingabe in diesem Schritt umfasst die Identifizierung des Datenverzeichnisses, die Angabe der Hydrogelsteifigkeit und die Reaktion auf Aufforderungen zur Zellclustersegmentierung, um Zellen aus dem zelllosen Bereich zu erkennen. Zu den wichtigsten zuvor erstellten Dateien, die in diesem Schritt verwendet werden, gehören 'p*/displacement/*_disp.dat'. Zu den wichtigsten neuen Dateien, die in diesem Schritt erstellt wurden, gehören: "p*/traction/*_trac.dat", das die von den Zellen auf das Hydrogel ausgeübten Kräfte auflistet; "p*/traction/*_domain.dat", in dem die Lage der Gitterpunkte aufgeführt ist, die Zellen enthalten; und Eingabedateien 'p*/traction.in, p*/clusterInput.txt' und 'p*/prestress.in', die Die Benutzerauswahl für diesen Schritt aufzeichnen. Nach der ersten quantitativen Bewertung von Zell-ECM und Zell-Zell-Kräften wurden mehrere Variationen der Technik entwickelt1,15. Die Variationen konzentrieren sich auf bestimmte Fälle von Substraten, Zellen, experimentellen Bedingungen oder numerischen Werkzeugen7,8,21,22. Siehe Abbildung 8 für die visuelle Beschreibung, die über AnViM, die Fourier-Transformations-Traktionsmikroskopie und die Monolayer-Stressmikroskopie-Analyse auf die Hydrogelverformungsdaten eingreift1,2,15.- Starten Sie die Fiji-Software und wählen Sie im Dropdown-Menü MSM - Cell-ECM und Cell-Cell Forces (dritte Option im Dropdown-Menü) aus.
- Wählen Sie im Dialogfeld des Dateibrowsers das übergeordnete Verzeichnis des Ordners "analysis" aus, der die Ordner "tnimgs" und "analysis" mit den analysierten Daten enthält. Klicken Sie auf Auswählen.
- Geben Sie im Fenster Parameter zur Berechnung der Zellkräfte den Schermodul, die Dicke des Hydrogels und den erwarteten Geräuschpegel ein. Wählen Sie OK aus. Auf diese Weise kann die Traktion über die MATLAB-Funktion ausgeführt werden.
HINWEIS: Nach diesen Eingaben berechnet AnViM die Zell-ECM-Kräfte. - Geben Sie danach an, ob die Monoschicht konfluent ist. Wenn das gesamte Zellenbild mit Zellen bedeckt ist, lautet die Antwort Ja. In diesem Fall werden die Zell-Zell-Kräfte über den gesamten Frame berechnet. Auf der anderen Seite, wenn ein Teil des Bildes keine Zellen hat, dann ist die Antwort Nein. Befolgen Sie in diesem Fall die AnViM-Anweisungen, um die Segmentierung des Bildbereichs, der Zellen enthält, zu erleichtern.
- Wenn die Antwort nein lautet, zeichnen Sie manuell ein Polygon um das Nichtzellenobjekt, wenn die Software dazu aufgefordert wird, und wählen Sie dann eine oder mehrere geeignete Methoden für die Segmentierung aus. Die Software fragt nach der Farbe (schwarz oder weiß) der Zellen.
- Aktivieren Sie die Option Spots automatisch füllen in der Software und klicken Sie auf OK.
ANMERKUNG: Die Schritte zum Segmentieren einer nicht fließenden Monoschicht werden im ersten Teil von Abschnitt 2.4 behandelt. Zell-ECM-Kräfte werden in '*_trac.dat' Dateien in einem neu angelegten Verzeichnis 'Traktion' innerhalb jedes Positionsordners gespeichert, und die Eingabe in die Cell-ECM Kraftberechnungssoftware wird in der Datei 'traction.in' gespeichert (Ergänzende Abbildung S8). Zell-Zell-Kräfte werden in '*_prestress.dat'-Dateien in einem neu erstellten Verzeichnis 'prestress' innerhalb jedes Positionsordners gespeichert, und die Eingabe in die Zellzellen-Kraftberechnungssoftware wird in der Datei 'prestress.in' gespeichert (Ergänzende Abbildung S9).
- Zuordnen von Rasterpunktwerten auf einzelne Zellen
HINWEIS: Einer der aktuellen Schwerpunkte von iTACS ist die Einführung eines einfachen Ansatzes zur Interpretation der gemessenen mechanischen Signale im Feld. Dieser Ansatz ist vorteilhaft für die Untersuchung mechanischer Wechselwirkungen zwischen benachbarten Zellen eines Clusters18. Insgesamt befinden sich die bisher quantifizierten Eigenschaften auf regelmäßig verteilten Gitterpunkten über den Zellhaufen. Diese Daten identifizieren Median, Mittelwert und Standardabweichung ausgewählter Eigenschaften innerhalb der morphologischen Grenze einzelner Zellen und ordnen diese als zelluläre physikalische Eigenschaften/Signale zu. Aus diesen wird die Standardabweichung verwendet, um die Variabilität anzuzeigen, die Differenz zwischen Mittelwert und Median wird verwendet, um die Art der Verteilung anzugeben, und der Medianwert wird verwendet, um den Gesamtzustand der Zellen für die ausgewählten Eigenschaften anzugeben.- Teil 1: Segmentierung des Bildbereichs, der Zellen enthält
Hinweis: Die manuelle Eingabe in diesen Schritt umfasst das Identifizieren des Datenverzeichnisses und das Reagieren auf die Segmentierungsaufforderungen, um Zellen aus dem Bereich ohne Zellen zu erkennen. Zu den wichtigsten zuvor erstellten Dateien, die in diesem Schritt verwendet werden, gehören 'p*/phs/*.tif'. Zu den wichtigsten neuen Dateien, die in diesem Schritt erstellt werden, gehören "p*/phs/bwImages/*.tif", bei denen es sich um binäre Bilder handelt, die Zellbereiche von Nicht-Zell-Bereichen trennen, "p*/phs/textResults/frameNum_percentHealed.txt", das den prozentualen Bildbereich auflistet, der jeweils von Zellen bedeckt ist, und "p*/clusterInput.txt", der die in der AcTrM-Schnittstelle eingegebenen Benutzerauswahl aufzeichnet. In Abbildung 9 finden Sie eine visuelle Beschreibung der folgenden Schritte zum Segmentieren des Bildbereichs, der Zellen enthält. Dieser Teil muss vor der Berechnung der Zell-Zell-Kräfte abgeschlossen werden. In diesem Fall müssen diese Schritte nicht wiederholt werden. Wenn diese Schritte vor den Zell-Zell-Kraftberechnungen durchgeführt werden, werden sie zu Beginn der Kraftberechnungen nicht angefordert.- Starten Sie die Fidschi-Software, und wählen Sie im Dropdown-Menü MSM die Option Segmentierung - Mobilfunkcluster aus.
- Wählen Sie im Dialogfeld des Dateibrowsers das Positionsverzeichnis 'analysis/p0', 'analysis/p1' usw. aus, das Eigenschaften enthält, die auf regelmäßig verteilten Rasterpunkten quantifiziert werden.
- Geben Sie an, ob die Monoschicht konfluent ist.
HINWEIS: Die folgenden Schritte werden nur aufgerufen, wenn die Monoschicht nicht konfluent ist. Folgen Sie den AnViM-Anweisungen, um den neuartigen mehrgleisigen Ansatz anzuwenden, um die Bildbereiche zu identifizieren, die Zellen enthalten. Der Prozess umfasst vier Methoden - jede nähert sich der Segmentierung auf unterschiedliche Weise. Eine oder mehrere dieser In Kombination verwendeten Methoden decken eine Vielzahl von Bildern der Zellen ab. Probieren Sie daher verschiedene Ansätze aus, um herauszufinden, welche Kombination für ihre Daten am besten geeignet ist. - Passen Sie den "Domänenverbindungsfaktor" und den "Abstrichfaktor" an, um eine optimale Segmentierung zu erhalten.
HINWEIS: Der "Smear-Faktor" vergrößert die Regionen, die Zellen enthalten. - Geben Sie an, ob die Zellen im Binärbild schwarz oder weiß angezeigt werden.
- Wählen Sie im Fenster Wegbeschreibung zum Säubern des Berandungsbildes aus, ob das Bild automatisch oder manuell bereinigt werden soll.
HINWEIS: Unerwünschte Merkmale der Bilder sind schwarze Flecken auf den Von Zellen besetzten Pixeln und weiße Flecken auf den Pixeln, die vom zu analysierenden Zellcluster getrennt sind. Die Analyse kann nur für die Regionen durchgeführt werden, die verbunden sind. Daher müssen mehrere getrennte Bereiche separat analysiert werden.
- Teil 2: Segmentierung der einzelnen Zellen in den Bildern
HINWEIS: Zu den manuellen Eingaben für diesen Schritt gehören das Identifizieren des Datenverzeichnisses und das Reagieren auf die Segmentierungsaufforderungen, um einzelne Zellen im Bild zu erkennen. Zu den wichtigsten zuvor erstellten Dateien, die in diesem Schritt verwendet werden, gehören 'p*/phs/*.tif' und 'p*/phs/bwImages/*.tif'. Zu den wichtigsten neuen Dateien, die erstellt wurden, gehören 'p*/phs/textResults/*.csv', die zelluläre morphologische Eigenschaften enthalten. In Abbildung 10 finden Sie eine visuelle Beschreibung der folgenden Schritte zum Segmentieren einzelner Zellen der Monoschicht.- Starten Sie die Fidschi-Software, und wählen Sie im Dropdown-Menü MSM die Option Segmentierung - Einzelne Zellen aus.
- Wählen Sie im Dialogfeld des Dateibrowsers das Positionsverzeichnis 'analysis/p0', 'analysis/p1' usw. aus, das Eigenschaften enthält, die auf regelmäßig verteilten Rasterpunkten quantifiziert werden.
- Geben Sie im Fenster Eingabeparameter auswählen das maximale Seitenverhältnis, die Hervorhebung dessen, wie stark sich das Zellzentrum im Vergleich zur Zellgrenze abhebt, und zwei Unschärfeparameter an, um die Erkennung der Zellen zu steuern (ergänzende Abbildung S10).
- Zeichnen Sie auf dem Bildstapel für jede Position ein Polygon auf der kleinsten normalen Zelle. Dies wird verwendet, um die Fläche zu berechnen. Alles, was kleiner ist, wird von der Software nicht als Zelle betrachtet.
- Zeichnen Sie als Nächstes ein Polygon um die größte normale Zelle und geben Sie an, ob das Zellzentrum oder die Zell-Zell-Schnittstelle heller ist.
HINWEIS: AnViM erstellt dann dateien 'phs/textResults/0001.csv', 'phs/textResults/0002.csv' usw. für jeden Frame, die Informationen zu Zellen innerhalb dieses Frames enthalten. In diesem Stadium enthält diese Datei morphologische Informationen über die Zellen, einschließlich Fläche, Schwerpunkt, Umfang, Orientierung, Zirkularität, Seitenverhältnis, Rundheit, Festigkeit, Abstand vom zellfreien Bereich. Die Längeneinheit in diesen Eigenschaften sind Pixel, und der Winkel ist in Grad. Schließlich wird diese Datei aktualisiert, um die Intensität, Bewegung und Kräfte des zellulären Pixels in den folgenden Schritten zu enthalten.
- Teil 3: Abbildung der Pixelintensitäten auf den Zellen
HINWEIS: Zu den manuellen Eingaben in diesem Schritt gehören das Identifizieren des Datenverzeichnisses und das Auswählen von Zuordnungseigenschaften und -parametern. Zu den wichtigsten zuvor erstellten Dateien, die in diesem Schritt verwendet wurden, gehören 'p*/phs/*.tif' (oder 'p*/fluo/*.tif ') und 'p*/phs/bwImages/*.tif'. Zu den wichtigsten neuen Dateien, die in diesem Schritt erstellt wurden, gehört 'p*/phs/textResults/*.csv', das zelluläre morphologische Eigenschaften auflistet. In Abbildung 11 finden Sie eine visuelle Beschreibung der folgenden Schritte, um die Pixelintensitäten in dem von einzelnen Zellen und ihrem benachbarten Bereich umgebenen Bereich zu bewerten und als Eigenschaften der Zellen zu definieren.- Starten Sie die Fidschi-Software, und wählen Sie im Dropdown-Menü MSM die Option Auf Zellen zuordnen - Intensitäten aus.
- Wählen Sie im Dialogfeld des Dateibrowsers das Positionsverzeichnis 'analysis/p0', 'analysis/p1' usw. aus, das Eigenschaften enthält, die auf regelmäßig verteilten Rasterpunkten quantifiziert werden.
- Wählen Sie Zellteilung erkennen oder Zelluläre Fluoreszenz quantifizieren aus, wenn Sie von der Software dazu aufgefordert werden. Definieren Sie die Größe der benachbarten Region. Aktivieren Sie sowohl die Sammeleigenschaften des benachbarten Bereichs als auch die Sammeleigenschaften der Zellen. Klicken Sie auf OK.
- Geben Sie im Fenster Zweck der Aufzeichnung der Zellintensität an, welche Art von Bild für die Intensitätszuordnung verwendet werden soll, die Größe der benachbarten Region und ob Daten für einzelne Zellen oder beide Zellen und ihre benachbarten Regionen gesammelt werden (ergänzende Abbildung S11).
HINWEIS: Die Abbildung der Pixelintensitäten eines Phasenkontrastbildes ermöglicht die Erkennung von Zellteilungsereignissen. Die Kartierung der Intensitäten der Fluoreszenzbilder ermöglicht den Nachweis von Fluktuationen in den fluoreszierend markierten zytoplasmatischen Molekülen. Die Ausgabe der obigen Schritte ist der Mittelwert, der Median, die Standardabweichung, die minimale und maximale Pixelintensität für einzelne Zellen. Diese Nummern werden als neue Spalten in den Dateien 'phs/textResults/0001.csv', 'phs/textResults/0002.csv' usw. eingetragen.
- Teil 4: Kartierung der Kräfte und Bewegungen auf den Zellen
HINWEIS: Zu den manuellen Eingaben für diesen Schritt gehören das Identifizieren des Datenverzeichnisses und das Auswählen von Zuordnungseigenschaften und -parametern. Zu den wichtigsten zuvor erstellten Dateien, die in diesem Schritt verwendet wurden, gehören 'p*/phs/textResults/*.csv', 'p*/velocity/*_vel.dat', 'p*/displacement/*_disp.dat', 'p*/traction/*_trac.dat', 'p*/prestress/*_prestress.dat' und 'p*/phs/bwImages/*.tif '. In diesem Schritt werden keine neuen Dateien erstellt. Stattdessen werden die neuen Informationen (Zellkraft und Bewegungseigenschaften) zu den Dateien 'p*/phs/textResults/*.csv' hinzugefügt. Siehe Abbildung 12 für die visuelle Beschreibung der Schritte, um die Kräfte und Bewegungen in dem von einzelnen Zellen und ihrem benachbarten Bereich umfassten Bereich zu bewerten und sie als Eigenschaften der Zellen zu definieren.- Wählen Sie aus dem MSM-Dropdown-Menü Map on Cells - Motion/Force Data (Ergänzende Abbildung S12).
- Führen Sie dann die in Schritt 2.4.3 aufgeführten Schritte aus.
HINWEIS: Neue Spalten werden den Dateien 'phs/textResults/0001.csv', 'phs/textResults/0002.csv' usw. hinzugefügt und enthalten den Mittelwert, den Median und die Standardabweichung der Geschwindigkeit, die Orientierung der Geschwindigkeit, die durchschnittliche Zytoskelettspannung, die Spannungsanisotropie, die Dehnungsenergie im Hydrogel, die Ausrichtung der höchsten Spannung und die Größe der Zell-ECM-Traktion (ergänzende Abbildung S13).
- Teil 1: Segmentierung des Bildbereichs, der Zellen enthält
- Visualisierung der Ergebnisse
- Teil 1: Verfolgung der zellulären Identitäten im gesamten Experiment
HINWEIS: Zu den manuellen Eingaben für diesen Schritt gehören das Identifizieren des Datenverzeichnisses und das Auswählen zugeordneter Eigenschaften zur Visualisierung. Zu den wichtigsten zuvor erstellten Dateien, die in diesem Schritt verwendet werden, gehören 'p*/phs/textResults/*.csv'. Zu den wichtigsten neuen Dateien, die während dieses Schritts erstellt werden, gehören 'p*/phs/textResults/cellTimeTrace_*.csv', die eine Zeitspur der Mobilfunkdaten enthält. In Abbildung 13 finden Sie eine visuelle Beschreibung der folgenden Schritte, um die Eigenschaften einzelner Zellen über die gesamte Dauer des Experiments zu verfolgen.- Starten Sie die Fidschi-Software, und wählen Sie im Dropdown-Menü MSM die Option Ergebnisse - Daten verfolgen aus.
- Wählen Sie im Dialogfeld des Dateibrowsers das Positionsverzeichnis 'analysis/p0', 'analysis/p1' usw. aus, das die zu verfolgenden Zellulären Eigenschaften enthält. Wenn Sie fertig sind, klicken Sie auf Auswählen.
- Wählen Sie im Fenster Startpunkt für Tracking auswählen den Startrahmen für die Überwachung und die Anzahl der gleichzeitig verfolgten Frames aus (ergänzende Abbildung S14). Beginnen Sie immer mit Frame Nummer 2, da die Geschwindigkeit für Frame Nummer 1 nicht bestimmt werden kann. Wenn Sie fertig sind, klicken Sie auf OK.
- Wählen Sie im Fenster Zu verfolgende Variablen überprüfen die Variablen aus, indem Sie entweder die Variablennamen eingeben oder die Begriffe aus den Kontrollkästchen auswählen (Ergänzende Abbildung S15). Klicken Sie dann auf OK.
HINWEIS: Das Tracking generiert 'phs/textResults/cellTimeTrace_*.csv'-Dateien, wobei jede Spalte eine eindeutige Zellennummer enthält und jede nachfolgende Zeile die fortlaufende Zeitinstanz darstellt.
- Teil 2: Generierung von Zeitspuren der bewerteten Zelleigenschaften
HINWEIS: Zu den manuellen Eingaben für diesen Schritt gehören das Identifizieren des Datenverzeichnisses und das Auswählen zugeordneter Eigenschaften zur Visualisierung. Zu den wichtigsten zuvor erstellten Dateien, die in diesem Schritt verwendet werden, gehören 'p*/phs/textResults/cellTimeTrace_*.csv'. Zu den wichtigsten neuen Dateien, die während dieses Schritts erstellt werden, gehören 'p*/phs/trackedDataPlots/*.tif', die Zeittrack-Plots enthalten, und 'p*/phs/trackedDataPlots/*.csv', die Plotdaten enthalten. In Abbildung 14 finden Sie eine visuelle Beschreibung der folgenden Schritte zum Generieren von Zeitspuren der bewerteten Zelleigenschaften.- Starten Sie die Fidschi-Software, und wählen Sie im Dropdown-Menü MSM die Option Ergebnisse - Plot aus.
- Wählen Sie im Dialogfeld des Dateibrowsers das Positionsverzeichnis 'analysis/p0', 'analysis/p1' usw. aus, das die zu zeichnenden zellularen Eigenschaften enthält.
- Wählen Sie aus, ob das Plotten auf Zellen mit ununterbrochenen Spuren beschränkt werden soll, indem Sie auf die Schaltfläche Ja oder Nein klicken.
HINWEIS: Diese Option ignoriert die Zellen, die in einer oder mehreren Zeitinstanzen nicht erkannt werden konnten. - Wählen Sie die Anzahl der Variablen aus, die gleichzeitig geplottet werden sollen, und klicken Sie auf OK.
HINWEIS: Derzeit erlaubt AnViM die Anzeige von maximal drei Variablen im selben Diagramm. - Wählen Sie einzelne Variablen für das Plotten über ein Dropdown-Menü aus.
HINWEIS: Mit diesen Schritten werden eine TIFF-Datei (Tagged Image File Format) und eine durch Kommas getrennte Datendatei im Ordner 'phs/trackedDataPlots/' erstellt. Der Dateiname besteht aus Variablennamen, die durch einen Unterstrich getrennt sind, und endet mit einer Zellennummer.
- Teil 3: Erstellung von Heatmaps der bewerteten Zelleigenschaften
HINWEIS: Zu den manuellen Eingaben in diesem Schritt gehören das Identifizieren des Datenverzeichnisses und das Auswählen zugeordneter Eigenschaften zur Visualisierung. Zu den wichtigsten zuvor erstellten Dateien, die in diesem Schritt verwendet werden, gehören 'p*/phs/textResults/cellTimeTrace_*.csv'. Zu den wichtigsten neuen Dateien, die in diesem Schritt generiert werden, gehören 'p*/phs/segmentedImages/cellPropMaps/*.tif', die die Heatmaps der Mobilfunkeigenschaften darstellen. In Abbildung 15 finden Sie eine visuelle Beschreibung der Schritte zur Erstellung von Heatmaps der bewerteten Zelleigenschaften.- Wählen Sie aus dem MSM-Dropdown-Menü Ergebnisse - Bild.
- Führen Sie die in Schritt 2.5.2 beschriebenen Schritte aus.
HINWEIS: Die Ausgabe wird als TIFF-Dateidateien im Ordner 'phs/segmentedImages/cellPropMaps/' gespeichert. Die Dateinamen sind die Zeitinstanznummer und der Variablenname, die durch einen Unterstrich getrennt sind.
- Teil 1: Verfolgung der zellulären Identitäten im gesamten Experiment
Ergebnisse
Wir stellen hier zwei der wichtigsten Ergebnisse für das demonstrierte Beispiel vor. Das erste Ergebnis ist die Zeitspur der Zellgeschwindigkeit und der Zytoskelettspannung für Zelle Nummer 1 (Abbildung 16). Die Eigenschaften werden auf einer gemeinsamen vertikalen Achse angezeigt, um die visuelle Zuordnung zwischen den Eigenschaften zu erleichtern, und die horizontale Achse gibt die Zeitinstanznummer an. In diesem Experiment wurden aufeinanderfolgende Frames in einem Intervall von 15 Minuten erfasst. Der zweite Ausgang ist ein Array von Heatmaps 1 h in das Experiment hinein (Abbildung 17). Zu den hier gezeigten Eigenschaften gehören Ausbreitungsfläche, Orientierung, Zirkularität, Geschwindigkeit, Bewegungsrichtung, maximale Spannungsorientierung, Zytoskelettspannung, Substrattraktionen und Spannungsanisotropie einzelner Zellen.

Abbildung 1: Struktur des Integrative Toolkits zur Analyse zellulärer Signale (iTACS). Zwei Schlüsselkomponenten von iTACS sind das Akquisitions- und Schulungsmodul (AcTrM) und das Analyse- und Visualisierungsmodul (AnViM). AcTrM kann verschiedene Hydrogel-Präparationstechniken verwenden, die derzeit für die Herstellung von Hydrogelen existieren, die fest auf einem Mikroskoptisch gehalten werden können, jede Zellaussaat und ein Wachstumsprotokoll, das Zellen in einer Fokusebene hält. AnViM kann verschiedene Techniken verwenden, um die Hydrogel- und Monoschichtverformung, die Zell-ECM-Kräfte und die Zell-Zell-Kräfte zu quantifizieren. Alle diese vom Benutzer bevorzugten Komponenten des Kraftmessprotokolls können in iTACS untergebracht werden und wurden mit gestrichelten Boxen identifiziert. Die mit festen Boxen identifizierten Komponenten sind neuartige Beiträge zur zellulären Kraftmesstechnik. Die Visualisierung im AnViM konzentriert sich auf den Medianwert und die Variabilität der Eigenschaften über einzelne Zellen hinweg. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
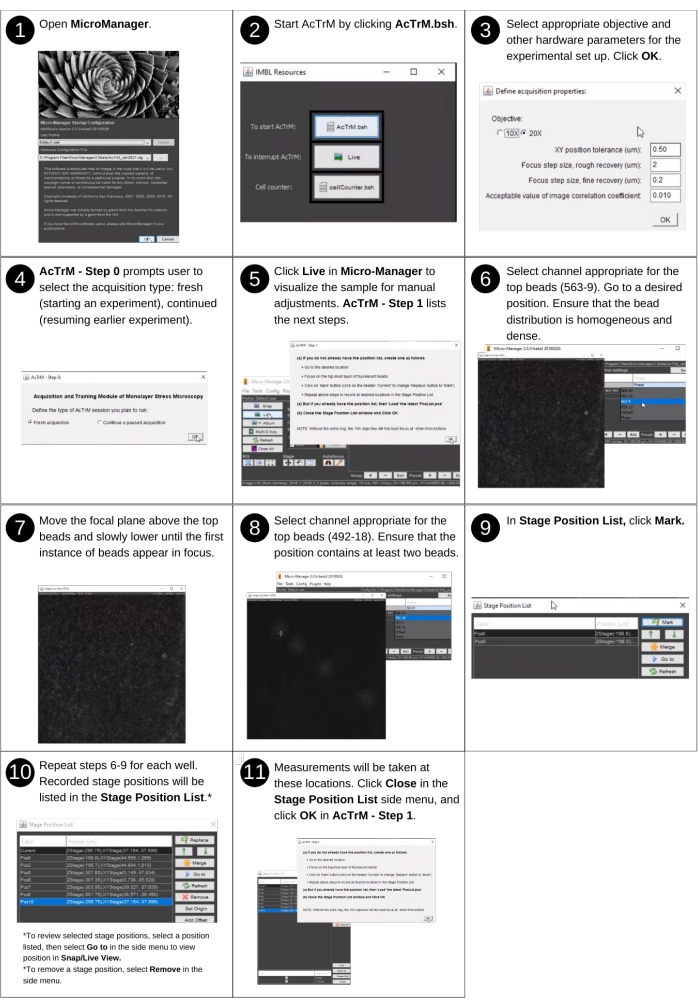
Abbildung 2: Referenzbilderfassung - Teil 1. Schritte zum Erstellen einer Positionsliste mit AcTrM. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
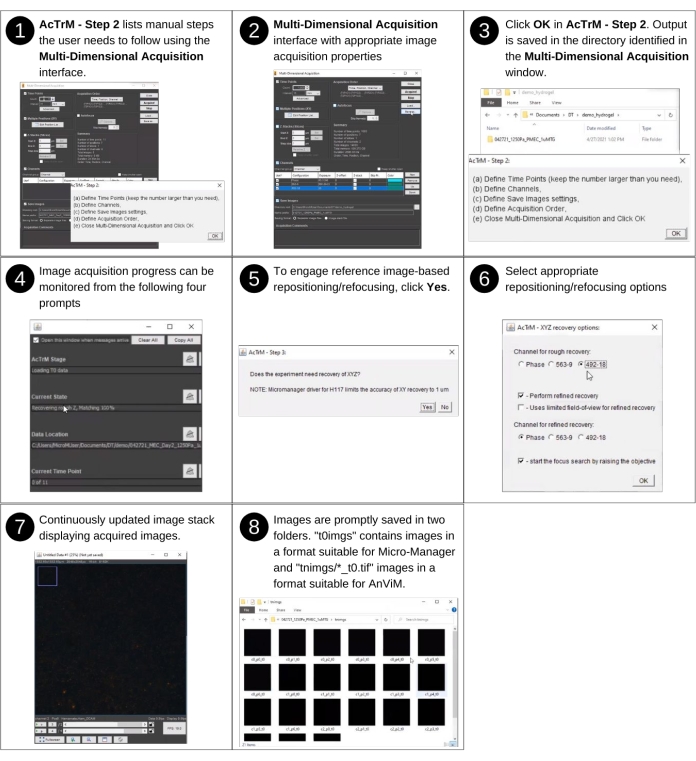
Abbildung 3: Referenzbilderfassung - Teil 2. Schritte zum Erfassen von Referenzbildern mit AcTrM. Detaillierte Ansichten der Schritte 2, 4 und 6 sind in den ergänzenden Abbildungen S2, S3 bzw. S4 dargestellt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
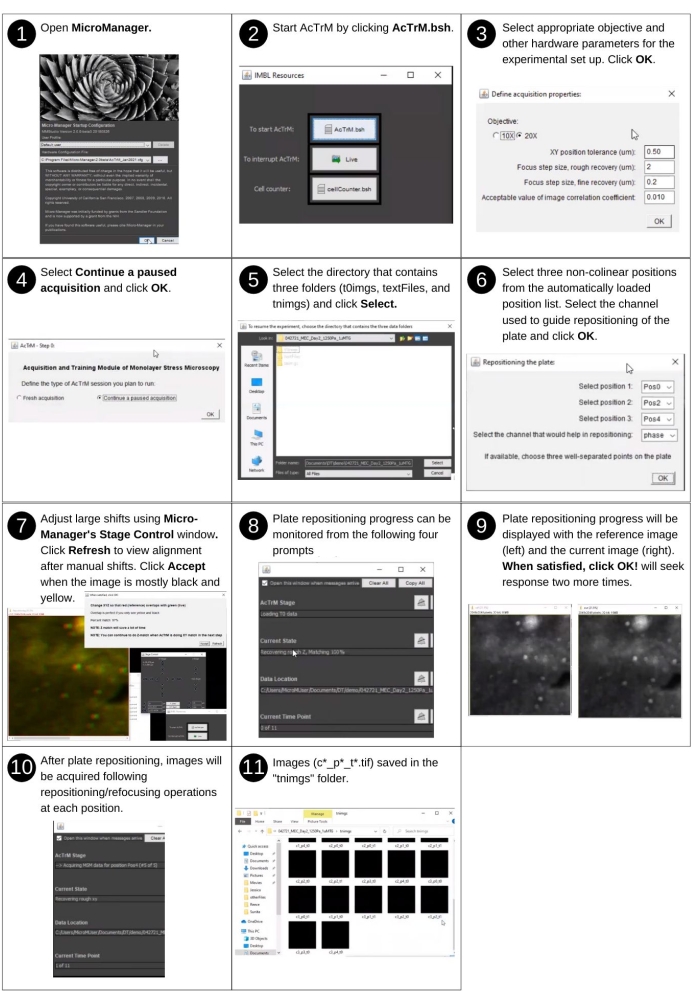
Abbildung 4: Automatisierte Bilderfassung für das verbleibende Experiment. Schritte zum Fortsetzen der Bilderfassung zur Bewertung des Mobilfunkverhaltens mit AcTrM. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 5: Einrichten der automatisierten Datenanalyse. Schritte zum Starten der automatisierten Bildanalyse mit AnViM. Die Software erkennt das von AcTrM verwendete Bildformat. Eine detaillierte Ansicht der Panels in den Schritten 3 und 5 ist in der ergänzenden Abbildung S5 bzw. der ergänzenden Abbildung S6 dargestellt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
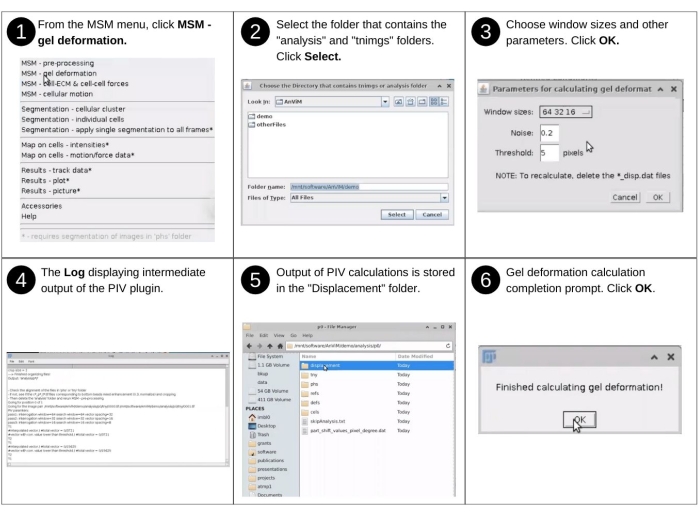
Abbildung 6: Quantifizierung der Verformung von Hydrogel und Monoschicht - Teil 1. Steps to engage, via AnViM, the Particle Image Velocimetry implementation of Tseng, Q. et al., PNAS (2012)20 to quantify deformation of the top surface of the hydrogel. Anwender können innerhalb von AnViM auch andere Ansätze zur Quantifizierung der Hydrogelverformung implementieren. Eine detaillierte Ansicht von Schritt 3 ist in der ergänzenden Abbildung S7 dargestellt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
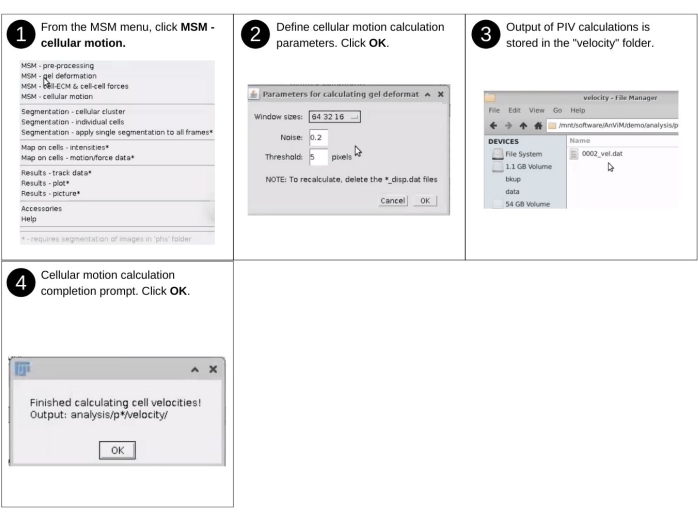
Abbildung 7: Quantifizierung der Verformung von Hydrogel und Monoschicht - Teil 2. Steps to engage, via AnViM, the Particle Image Velocimetry implementation of Tseng, Q. et al., PNAS (2012)20 to quantify the local motion of individual cells. Benutzer können innerhalb von AnViM auch andere Ansätze zur Quantifizierung der zellulären Bewegung implementieren. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
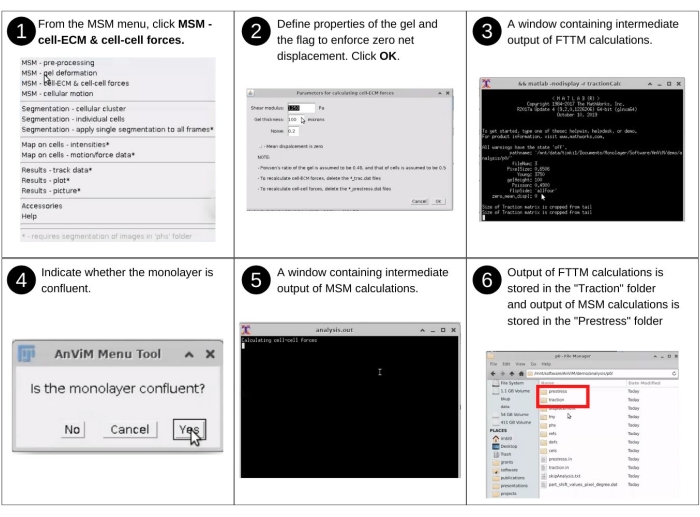
Abbildung 8: Quantifizierung von Zell-ECM und Zell-Zell-Kräften. Schritte zur Durchführung der Bildanalyse, um über AnViM die Fourier Transform Traction Microscopy-Implementierung von Trepat et al., Nature Physics (2009) 15 zur Quantifizierung der von den Zellen auf das Hydrogel ausgeübten Kräfte und die Monolayer Stress Microscopy-Implementierung von Tambe et al., Nature Materials (2011) 1 zur Quantifizierung von Kräften innerhalb einzelner Zellen und zwischen benachbarten Zellen zu verwenden. Anwender können innerhalb von AnViM auch andere Ansätze zur Quantifizierung von Zell-ECM und Zell-Zell-Kräften implementieren. Eine detaillierte Übersicht über Schritt 6 ist in ergänzender Abbildung S8 und ergänzender Abbildung S9 dargestellt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
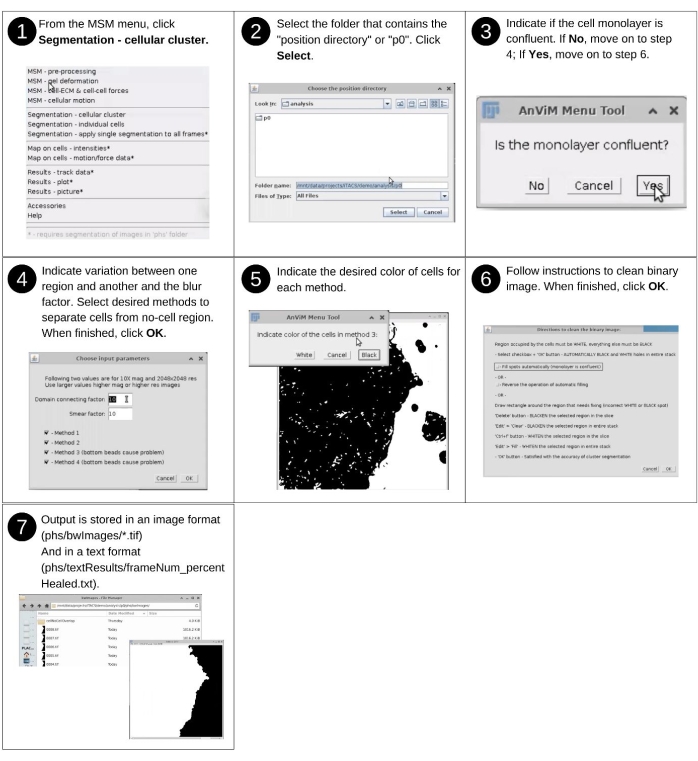
Abbildung 9: Zuordnen von Rasterpunktwerten auf einzelnen Zellen - Teil 1. Schritte zur Segmentierung der Bildbereiche, die Zellen enthalten, mit einem neuartigen mehrgleisigen Ansatz. Dieser Ansatz kann verwendet werden, um Phasenkontrast-, Hellfeld- oder Fluoreszenzbilder der Zellen zu segmentieren. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
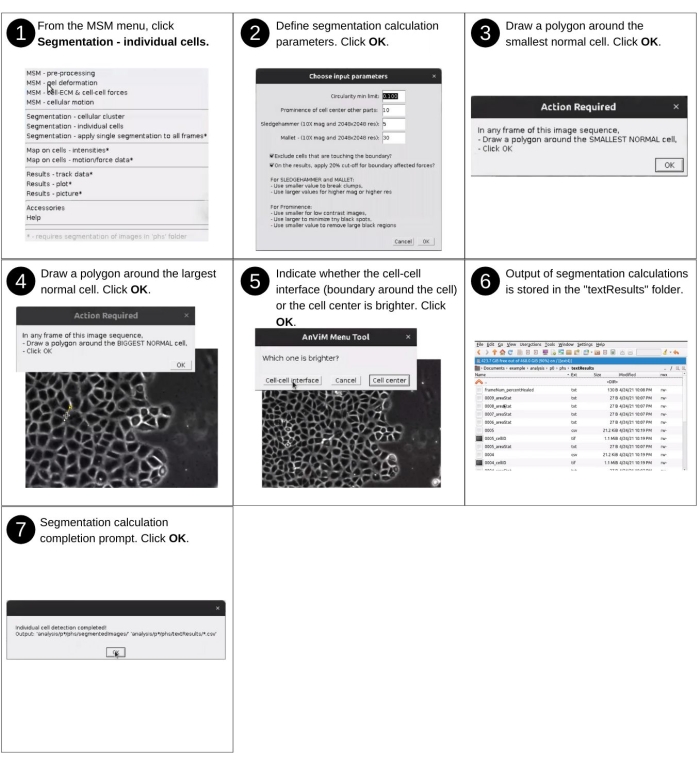
Abbildung 10: Zuordnen von Rasterpunktwerten auf einzelnen Zellen - Teil 2. Schritte zur Segmentierung einzelner Zellen einer Monoschicht unter Verwendung eines neuartigen mehrgleisigen Ansatzes, der in AnViM entwickelt wurde. Dieser Ansatz kann verwendet werden, um Phasenkontrast-, Hellfeld- oder Fluoreszenzbilder der Zellen zu segmentieren. Eine detaillierte Ansicht von Schritt 2 ist in ergänzender Abbildung S10 dargestellt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
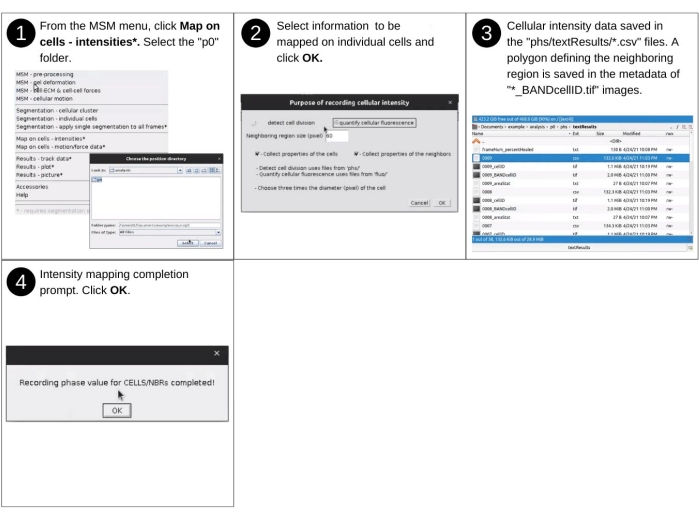
Abbildung 11: Zuordnen von Rasterpunktwerten auf einzelnen Zellen - Teil 3. Schritte zur Beurteilung der Pixelintensitäten im Bereich innerhalb einzelner Zellen und innerhalb des benachbarten Bereichs einzelner Zellen mitHilfe von AnViM. Die bewerteten Intensitäten umfassen die Durchlichtintensität und die Fluoreszenzintensität. Dieser Teil bildet den Medianwert und die Standardabweichung der Pixelintensitäten innerhalb einzelner Zellen und innerhalb eines benachbarten Bereichs einzelner Zellen ab. Eine detaillierte Ansicht von Schritt 2 ist in der ergänzenden Abbildung S11 dargestellt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
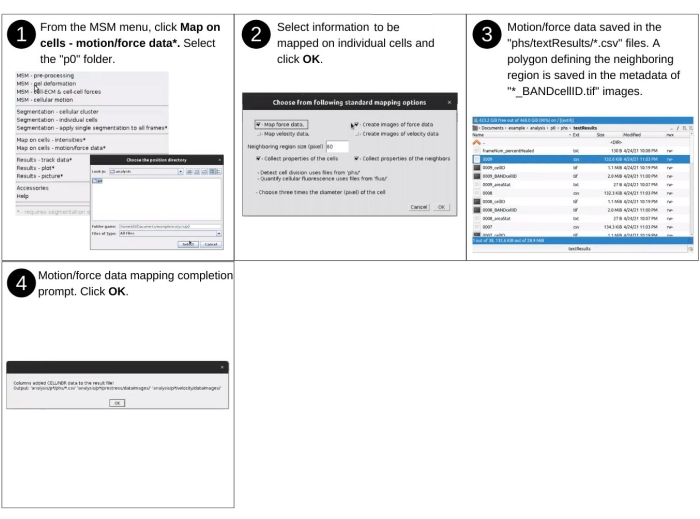
Abbildung 12: Zuordnen von Rasterpunktwerten auf einzelnen Zellen - Teil 4. Schritte zur Beurteilung von Kräften und Bewegungseigenschaften der Gitterpunkte innerhalb einzelner Zellen und innerhalb des benachbarten Bereichs einzelner Zellen mit AnViM. Dieser Teil bildet den Medianwert und die Standardabweichung der Eigenschaften innerhalb einzelner Zellen und innerhalb eines benachbarten Bereichs einzelner Zellen ab. Eine detaillierte Übersicht über die Schritte 2 und 3 finden Sie in der Ergänzenden Abbildung S12 und der Ergänzenden Abbildung S13. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
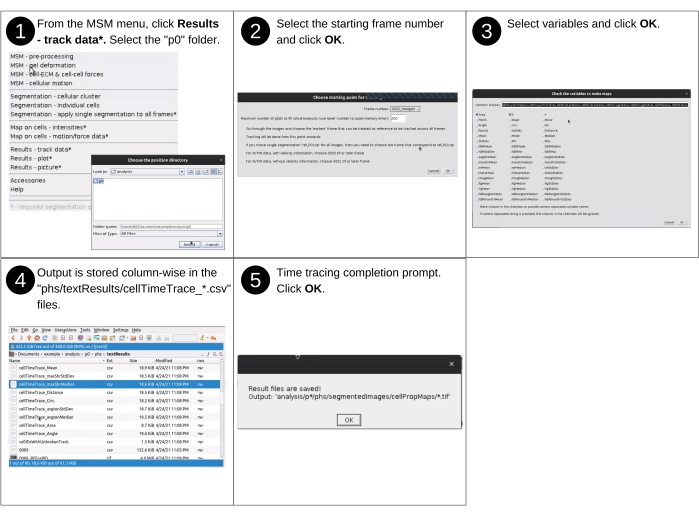
Abbildung 13: Visualisierung der Ergebnisse - Teil 1. Schritte zur Verfolgung der Eigenschaften einzelner Zellen über die gesamte Dauer des Experiments mit AnViM. Eine detaillierte Übersicht über die Schritte 2 und 3 finden Sie in der Ergänzenden Abbildung S14 und der Ergänzenden Abbildung S15. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
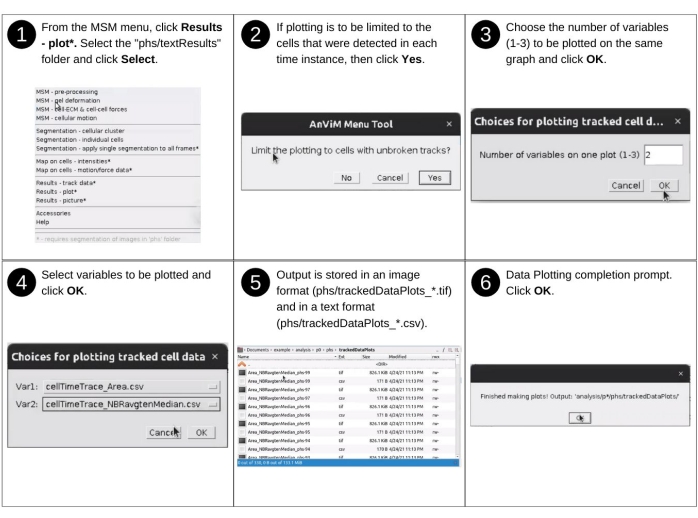
Abbildung 14: Visualisierung der Ergebnisse - Teil 2. Schritte zum Generieren von Zeitablaufverfolgungen der bewerteten Eigenschaften mit AnViM. Der Benutzer hat die Möglichkeit, bis zu drei Eigenschaften in einem Diagramm zu plotten. Zeitspuren werden entweder für alle Zellen oder nur für die Zellen generiert, für die die Verfolgung im gesamten Experiment erfolgreich war. Eine detaillierte Übersicht über Schritt 5 ist in Ergänzende Abbildung S16 und Ergänzende Abbildung S17 dargestellt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
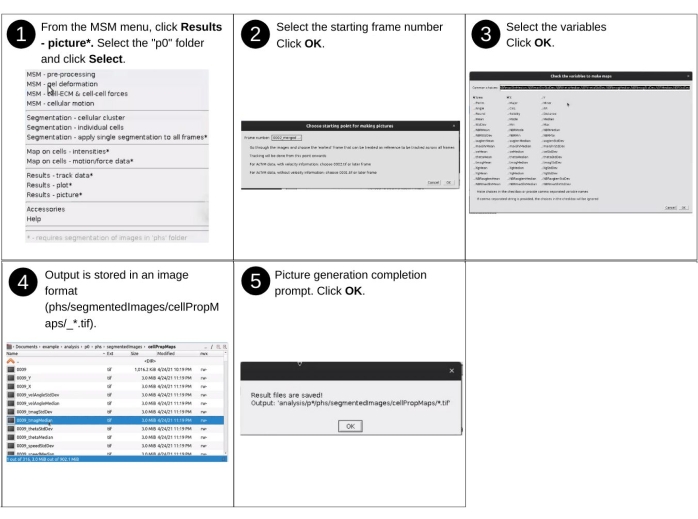
Abbildung 15: Visualisierung der Ergebnisse - Teil 3. Schritte zum Generieren von Heatmaps der bewerteten Eigenschaften mit AnViM. Heatmaps werden für alle Frames generiert, die dem Startframe und allen ausgewählten Eigenschaften folgen. Eine detaillierte Ansicht von Schritt 3 ist in der ergänzenden Abbildung S18 dargestellt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
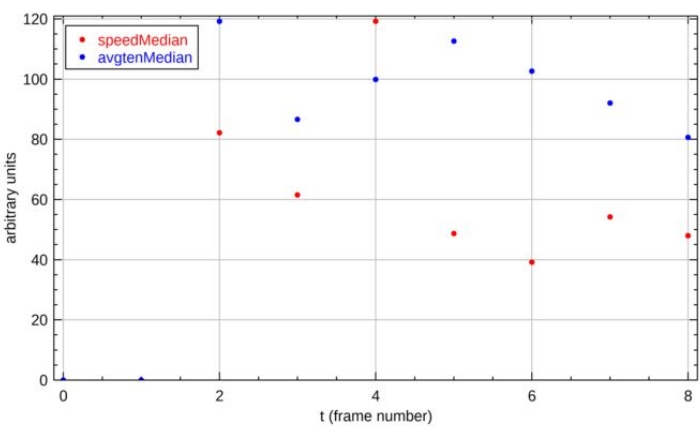
Abbildung 16: Zeitablaufverfolgungen für Zellen-ID 1. Zwei angezeigte Eigenschaften sind die zelluläre Zytoskelettspannung ("avgtenMedian") und die zelluläre Geschwindigkeit ("speedMedian"). Sowohl die zelluläre Zytoskelettspannung als auch die zelluläre Geschwindigkeit werden als Medianwert über die Gitterpunkte innerhalb der Zellen quantifiziert. Die beiden Eigenschaften werden auf derselben Achse mit beliebigen Einheiten dargestellt, um Beziehungen zwischen den bewerteten Eigenschaften zu visualisieren. Weitere Variablennamen sind in der Ergänzungstabelle S1 aufgeführt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
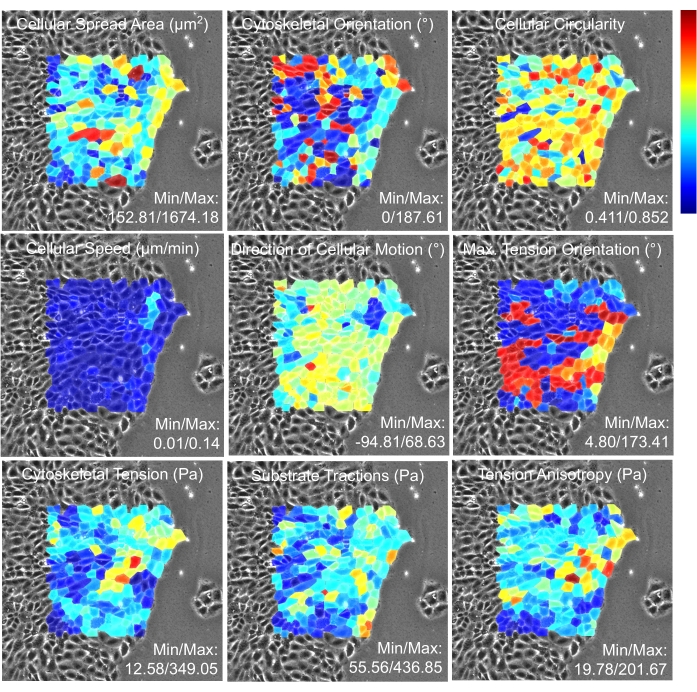
Abbildung 17: Heatmaps der Eigenschaften einzelner Zellen über die analysierte Monoschicht. Jede Zelle wird mit dem Medianwert der im Bedienfeld angegebenen Eigenschaft eingefärbt. So zeigt tiefes Rot den maximalen zellularen Wert im Farbspektrum an, und tiefes Blau zeigt den minimalen zellulären Wert über die analysierte Monoschicht an. Wie in Tambe et al., PLoS One (2013)2 beschrieben, haben die Zellen, die sich näher an der Grenze befinden, mechanische Kräfte, die von unbekannten Eigenschaften der Zellen außerhalb des Bildes beeinflusst werden. Daher wird die Heatmap für Zellen weit von der Grenze entfernt generiert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Abbildung S1: Beispielbilder der oberen und unteren fluoreszierenden Perle. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Abbildung S2: Eine detaillierte Ansicht von Schritt 2 aus Abbildung 3. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Abbildung S3: Eine detaillierte Ansicht von Schritt 4 aus Abbildung 3. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Abbildung S4 A: Detailansicht von Schritt 6 aus Abbildung 3. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Abbildung S5: Eine detaillierte Ansicht von Schritt 3 aus Abbildung 5. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Abbildung S6: Eine detaillierte Ansicht von Schritt 5 aus Abbildung 5. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Abbildung S7: Eine detaillierte Ansicht von Schritt 3 aus Abbildung 6. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Abbildung S8: Eine Detailansicht der Zell-ECM-Kraftausgabe von Schritt 6 aus Abbildung 8. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Abbildung S9: Eine detaillierte Ansicht der Zell-Zell-Kraftausgabe von Schritt 6 aus Abbildung 8. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Abbildung S10: Eine detaillierte Ansicht von Schritt 2 aus Abbildung 10. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Abbildung S11: Eine detaillierte Ansicht von Schritt 2 aus Abbildung 11. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Abbildung S12: Eine detaillierte Ansicht von Schritt 2 aus Abbildung 12. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Abbildung S13: Eine detaillierte Ansicht der Ausgabe von Schritt 3 aus Abbildung 12. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Abbildung S14: Eine detaillierte Ansicht von Schritt 2 in Abbildung 13. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Abbildung S15: Eine detaillierte Ansicht von Schritt 3 aus Abbildung 13. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Abbildung S16: Eine detaillierte Ansicht der in Schritt 5 generierten Datendateien aus Abbildung 14. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Abbildung S17: Eine detaillierte Ansicht eines Diagramms, das in Schritt 5 aus Abbildung 14 generiert wurde. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Abbildung S18: Eine detaillierte Ansicht einer Heatmap und der Datei, die den Bereich des in Schritt 4 generierten Farbspektrums aus Abbildung 15 enthält. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Tabelle S1: Eine Liste ausgewählter Eigenschaften, die von iTACS quantifiziert wurden. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Diskussion
Adhärente Zellen nutzen sowohl mechanische als auch chemische Signale, um zu überleben, zu wachsen und zu funktionieren. Eine Vielzahl von Mikroskopie-Software optimiert die Benutzererfahrung bei der Beurteilung der chemischen Signale durch fluoreszenzbasierte Bildgebung. Die Bewertung der mechanischen Signale umfasst jedoch Fähigkeiten, die in der Standard-Mikroskopiesoftware nicht verfügbar sind. Darüber hinaus ist die Bewertung mechanischer Signale am effizientesten, wenn die Datenerfassung in die Datenanalyse integriert ist. Das Fehlen einer einheitlichen Plattform, die den einzigartigen Anforderungen der mechanischen Signalbewertung gerecht wird, war eine große technologische Lücke in der experimentellen Zellbiologie. Das Integrative Toolkit to Analyze Cellular Signals (iTACS) wurde entwickelt, um diese Lücke zu schließen. Die beiden Komponenten von iTACS, AnViM und AcTrM, statten Benutzer mit den notwendigen Fähigkeiten aus, um zelluläre Eigenschaften von vier großen Kategorien zu quantifizieren: Kräfte, Bewegung, Morphologie und Fluoreszenz / Helligkeit. Über diese Kategorien hinweg ist iTACS derzeit in der Lage, mehr als 50 einzigartige Aspekte einzelner adhärenter Zellen aufzudecken. Diese Aspekte umfassen spezifische Eigenschaften jeder breiten Kategorie, einschließlich ihres repräsentativen Wertes und ihrer Variabilität in der Zelle (Ergänzende Tabelle S1). Zum Beispiel gibt es innerhalb der Kräfte Zugkräfte über das Zytoskelett, Anisotropie dieser Spannung, die Ausrichtung der maximalen Spannung und Scherspannung über die Zell-ECM-Grenzfläche, die einen tiefgreifenden Einfluss auf das Verhalten der adhärenten Zellen hat1,3,6.
Ein neuartiger Ansatz zur Untersuchung des mechanischen Verhaltens einzelner Zellen einer Monoschicht
Einzelne Zellen einer Monoschicht befinden sich in einem Austausch von Signalen chemischer und mechanischer Natur3. Diese beiden Arten von Signalen werden auf unterschiedliche Weise über die zelluläre Monoschicht übertragen23. Das Wissen über die mechanische Signalübertragung hinkt jedoch dem der chemischen Signalübertragung hinterher. Diese Wissenslücke fällt mit einem anhaltenden Mangel an einfachen und intuitiven Ansätzen zur Bewertung zellulärer mechanischer Signale zusammen. Der hier beschriebene neuartige Data-Mapping-Ansatz ist in der Lage, diese Lücke zu schließen. Eine solche Kartierung zeigt, dass die Fluktuation der intrinsischen Zytoskelettspannung in der benachbarten Region einer Zelle als Entspannungs-, Fluidisierungs- und Verankerungssignale dient, die Veränderungen in der Zellform, -größe und -geschwindigkeit regulieren18. Karten der Eigenschaften benachbarter Regionen zeigen "mehrzellige Unterteilungsmuster", bei denen Zellen innerhalb der Unterteilung einer relativ einheitlichen Mikroumgebung ausgesetzt sind und die Zellen an der Grenze der Unterteilung einer bemerkenswert ungleichmäßigen Mikroumgebung ausgesetzt sind18.
Zugänglichkeit der Kraftmesstechnik
Es gibt eine Vielzahl von Protokollen zur Herstellung von PAA-Hydrogelen, zur Analyse der Hydrogelverformung und Zellbewegung sowie zur Quantifizierung von Zell-ECM und Zell-Zell-Kräften1,2,7,8,9,13,14,15,18,20,21,24,25,26,27 ,28,29,30,31,32. Diese Entwicklungen bleiben jedoch außerhalb der Reichweite der üblichen zellbiologischen Laboratorien und beschränken sich auf Laboratorien mit technischem Know-how. Durch die Automatisierung der technischen Aspekte dieser Ansätze und deren Integration unter einer einheitlichen und benutzerfreundlichen Plattform ist es das Ziel von iTACS, die Bewertung mechanischer Signale zu einer Routineaktivität in der experimentellen zellbiologischen Forschung und Ausbildung zu machen.
ImageJ ermöglicht es Benutzern, Anwendungen mit Ansätzen zu entwickeln, die wenig oder keine Schulung erfordern11. iTACS basiert größtenteils auf einfachen Scripting-Ansätzen, um eine Community-gesteuerte Weiterentwicklung zu ermöglichen. Ein Großteil von AcTrM wird mit BeanShell-Skripten programmiert, und der Großteil von AnViM wird mit ImageJ-Makros programmiert. Diese Skripts und Anleitungen zur Implementierung dieser Funktionen auf dem Mikroskop des Benutzers sind über GitHub (https://github.com/IntegrativeMechanobiologyLaboratory/iTACS verfügbar.
Qualitätsstandardisierung der aufgenommenen Bilder
Obwohl die auf elastischen Substraten basierenden Techniken zur Quantifizierung physikalischer Kräfte in adhärenten Zellen in verschiedenen Labors entwickelt und implementiert wurden, fehlt dem Protokoll noch die Standardisierung. Ein Bereich, der am dringendsten einer Standardisierung bedarf, ist die Qualität der aufgenommenen Bilder der oberen Perlen (ergänzende Abbildung S1). Signifikante Probleme ergeben sich aus der Drift im Fokus während des gesamten Experiments. Unser neuartiger referenzbildbasierter Refokussierungsansatz macht eine solche Fokussierung zu einem objektiven Prozess. Die im allerersten Schritt von AcTrM definierten Parameter setzen notwendige objektive Qualitätsgrenzen. Weitere Standardisierungsmaßnahmen können in zukünftigen Versionen von AcTrM programmiert werden.
Die breite Anwendbarkeit von iTACS
Neben der Quantifizierung zahlreicher Aspekte adhärenter Zellen erleichtert die iTACS-Struktur die Verwendung für verschiedene experimentelle Protokolle und Bedürfnisse. AcTrM ermöglicht eine softwaregesteuerte Selbstschulung des Benutzers. Die Hochgeschwindigkeitsbildgebung, die beispielsweise durch die gleichzeitige Bewertung zytoplasmatischer Calciumfluktuationen erforderlich ist, ist derzeit durch die Geschwindigkeit der Neupositionierung und Neufokussierung der Hardware begrenzt und erfolgt am besten an einem Ort nach dem anderen. Die derzeitige Implementierung ist jedoch gut gerüstet für die Langzeitbildgebung, unterbrochene Bildgebung, bei der die Probe nicht für die gesamte Dauer des Experiments auf dem Mikroskoptisch gehalten werden kann. Da die Referenzbilder zu Beginn des Experiments aufgenommen werden, ermöglicht iTACS die Echtzeit-Bildgebung mechanischer Signale und eröffnet neue Wege im Drogenscreening. AnViM ermöglicht es Benutzern, hochtechnische Informationen in Laienbegriffen bereitzustellen. Die Fähigkeit, ein breites Spektrum zellulärer Eigenschaften zu quantifizieren und sie während des gesamten Experiments zu verfolgen, stellt entscheidende Fähigkeiten dar, die benötigt werden, um neue interzelluläre Kommunikationsmechanismen zu entdecken.
Für die zukünftige Entwicklung von iTACS haben wir vier Schwerpunktbereiche identifiziert: (1) Verbesserung der Datenerfassungs- und Datenanalysegeschwindigkeit, (2) Implementierung von Ansätzen zur Bewertung neuer Mobilfunksignale13, (3) Entwicklung von Workshops und Schulungsmodulen zur iTACS-basierten zellulären Signalbewertung, (4) Entwicklung kostengünstiger Automatisierungslösungen.
Offenlegungen
Die Autoren erklären keine Interessenkonflikte.
Danksagungen
D.T.T. dankt den Mitarbeitern des Zentrums für Lungenbiologie an der University of South Alabama für die anregenden Diskussionen über die experimentelle zellbiologischen Forschungsbedürfnisse. Diese Diskussionen waren entscheidend für die Initiierung der Entwicklung von iTACS.
Diese Arbeit wurde zum Teil durch Zuschüsse des National Institute of Health / National Heart Lung Blood Institute, P01 HL66299 und R37 HL60024 (Stevens), R01-HL118334 (Alvarez), F32-HL144040-01 (Xu) und der University of South Alabama durch den Abraham Mitchell Cancer Research Fund (Singh, Palanki, Tambe), research and Scholarly Development Grant (Tambe), Honors College und Summer Undergraduate Research Fellow (Nguyen) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Reagents and components used in prepare glass surface for hydrogel coating | |||
| (3-Aminopropyl)trimethoxysilane, 97% | Aldrich chemistry | 13822565 | |
| 2% Bis Solution | Bio-rad | 1610142 | |
| 3-(Trimethoxysilyl)propyl methacrylate,98% | Acros organics | 2530850 | |
| 40% Acrylamide Solution | Bio-rad | 1610140 | |
| Glass bottom 35 mm dish/ 6 or 12 or 24 well plates | MatTek or CellVis | ||
| Glutaraldehyde, EM Grade, 25% | Polysciences | 1909100 | |
| Sodium Hydroxide | Sigma-aldrich | 1002074706 | |
| Reagents and components used in preparing suitable hydrogel | |||
| 2% Bis Solution | Bio-rad | 1610142 | |
| 40% Acrylamide Solution | Bio-rad | 1610140 | |
| Ammonium Persulfate | Bio-rad | 1610700 | |
| Cover Slips | Electron Microscopy Sciences | 7222301 | |
| Dulbecco's Phosphate Buffered Saline (1M) | Gibco | 14190136 | |
| FluoSpheres carboxylate 0.2 um, yellow-green(505/515) | Invitrogen | F8811 | |
| FluoSpheres carboxylate 0.5 um, red(580/605) | Invitrogen | F8812 | |
| FluoSpheres carboxylate 2.0 um, red(580/605) | Invitrogen | F8826 | |
| Rain-X | |||
| TEMED | Bio-rad | 1610801 | |
| Reagents used in coating extracellular matrix on the hydrogel | |||
| Collagen Type I Rat Tail | Corning | 354236 | |
| HEPES(1M) | Gibco | 15630080 | |
| Phosphate Buffered Saline (1M) | Gibco | 10010023 | |
| Sulfo-SANPAH | CovaChem | 102568434 | |
| Microscope hardware used in the current study | |||
| Camera | Hamamatsu Flash 4.0 LT sCMOS Camera | C11440-42U | |
| H117 ProScanTM Stages | Prior Scientific | ||
| Light source- Lambda DG4 and Lambda DG5 | Sutter instrument company | ||
| Microscope | Nikon eclipse TE2000-S | 550372 | |
| ProScan III Universal Microscope Automation Controller | Prior Scientific | ||
| Stagetop incubator | ibidi | 11922 | |
| Stepper Motor Focus Drive | Prior Scientific |
Referenzen
- Tambe, D. T., et al. Collective cell guidance by cooperative intercellular forces. Nature Materials. 10 (6), 469-475 (2011).
- Tambe, D. T., et al. Monolayer stress microscopy: limitations, artifacts, and accuracy of recovered intercellular stresses. PloS One. 8 (2), 55172 (2013).
- Das, T., et al. A molecular mechanotransduction pathway regulates collective migration of epithelial cells. Nature Cell Biology. 17 (3), 276-287 (2015).
- Vedula, S. R., et al. Mechanics of epithelial closure over non-adherent environments. Nature Communications. 6, 6111 (2015).
- Lamason, R. L., et al. Rickettsia Sca4 reduces vinculin-mediated intercellular tension to promote spread. Cell. 167 (3), 670-683 (2016).
- Sunyer, R., et al. Collective cell durotaxis emerges from long-range intercellular force transmission. Science. 353 (6304), 1157-1161 (2016).
- Dong, L., Oberai, A. A. Recovery of cellular traction in three-dimensional nonlinear hyperelastic matrices. Computer Methods in Applied Mechanics and Engineering. 314, 296-313 (2017).
- Nier, V., et al. Kalman inversion stress microscopy. Biophysical Journal. 115 (9), 1808-1816 (2018).
- Serrano, R., et al. Three-dimensional Monolayer Stress Microscopy. Biophysical Journal. 117 (1), 111-128 (2019).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ: 25 years of Image Analysis. Nature Methods. 9 (7), 671-675 (2012).
- Schindelin, J., et al. Fiji - an Open Source platform for biological image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Edelstein, A., Amodaj, N., Hoover, K., Vale, R., Stuurman, N. Computer control of microscopes using microManager. Current Protocols in Molecular Biology. , 20 (2010).
- Patel, N. G., et al. Unleashing shear: Role of intercellular traction and cellular moments in collective cell migration. Biochemical and Biophysical Research Communications. 522 (2), 279-285 (2020).
- Yeung, T., et al. Effects of substrate stiffness on cell morphology, cytoskeletal structure, and adhesion. Cell Motility and the Cytoskeleton. 60 (1), 24-34 (2005).
- Trepat, X., et al. Physical forces during collective cell migration. Nature Physics. 5, 426-430 (2009).
- Aratyn-Schaus, Y., Oakes, P. W., Stricker, J., Winter, S. P., Gardel, M. L. Preparation of complaint matrices for quantifying cellular contraction. Journal of Visualized Experiments: JoVE. (46), (2010).
- King, J., et al. Structural and functional characteristics of lung macro- and microvascular endothelial cell phenotypes. Microvascular Research. 67 (2), 139-151 (2004).
- Patel, G., et al. Mechanical signaling in a pulmonary microvascular endothelial cell monolayer. Biochemical and Biophysical Research Communications. 519 (2), 337-343 (2019).
- Kähler, C. J., et al. Main results of the 4th International PIV Challenge. Experiments in Fluids. 57 (6), 97 (2016).
- Tseng, Q., et al. Spatial organization of the extracellular matrix regulates cell-cell junction positioning. Proceedings of the National Academy of Sciences of the United States of America. 109 (5), 1506-1511 (2012).
- Alvarez-Gonzalez, B., et al. Two-layer elastographic 3-D traction force microscopy. Scientific Reports. 7, 39315 (2017).
- Makarchuk, S., Beyer, N., Gaiddon, C., Grange, W., Hebraud, P. Holographic traction force microscopy. Scientific Reports. 8 (1), 3038 (2018).
- Janmey, P. A., Miller, R. T. Mechanisms of mechanical signaling in development and disease. Journal of Cell Science. 124 (1), 9-18 (2011).
- Dembo, M., Oliver, T., Ishihara, A., Jacobson, K. Imaging the traction stresses exerted by locomoting cells with the elastic substratum method. Biophysical Journal. 70 (4), 2008-2022 (1996).
- Dembo, M., Wang, Y. L. Stresses at the cell-to-substrate interface during locomotion of fibroblasts. Biophysical Journal. 76 (4), 2307-2316 (1999).
- Butler, J. P., Tolic-Norrelykke, I. M., Fabry, B., Fredberg, J. J. Traction fields, moments, and strain energy that cells exert on their surroundings. American Journal of Physiology: Cell Physiology. 282 (3), 595-605 (2002).
- Saez, A., et al. Traction forces exerted by epithelial cell sheets. Journal of Physics. Condensed Matter. 22 (19), 194119 (2010).
- Deforet, M., et al. Automated velocity mapping of migrating cell populations (AVeMap). Nature Methods. 9 (11), 1081-1083 (2012).
- Polio, S. R., Smith, M. L. Patterned hydrogels for simplified measurement of cell traction forces. Methods in Cell Biology. 121, 17-31 (2014).
- Toyjanova, J., et al. 3D Viscoelastic traction force microscopy. Soft Matter. 10 (40), 8095-8106 (2014).
- Serra-Picamal, X., Conte, V., Sunyer, R., Munoz, J. J., Trepat, X. Mapping forces and kinematics during collective cell migration. Methods in Cell Biology. 125, 309-330 (2015).
- Charrier, E. E., et al. A novel method to make viscoelastic polyacrylamide gels for cell culture and traction force microscopy. APL Bioengineering. 4 (3), 036104 (2020).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten