Method Article
Boîte à outils intégrative pour analyser les signaux cellulaires: forces, mouvement, morphologie et fluorescence
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
La plate-forme iTACS (Integrative Toolkit to Analyze Cellular Signals) automatise le processus de mesure simultanée d’une grande variété de signaux chimiques et mécaniques dans les cellules adhérentes. iTACS est conçu pour faciliter le développement communautaire et permettre aux chercheurs d’utiliser toutes les fonctionnalités de la plate-forme, quel que soit leur niveau de formation.
Résumé
L’évaluation quantitative des forces cellulaires et du mouvement a considérablement progressé au cours des quatre dernières décennies. Ces progrès ont fourni le cadre pour examiner les processus de signalisation mécanique perspicaces dans les systèmes de culture cellulaire. Cependant, le domaine est actuellement confronté à trois problèmes: le manque de normalisation de la qualité des données acquises, les erreurs techniques dans l’analyse et la visualisation des données, et peut-être plus important encore, la technologie reste largement hors de portée des laboratoires de biologie cellulaire courants. Pour surmonter ces limites, nous avons développé une nouvelle plateforme expérimentale - Integrative Toolkit to Analyze Cellular Signals (iTACS). iTACS se compose de deux composantes : le module d’acquisition et de formation (AcTrM) et le module d’analyse et de visualisation (AnViM). AcTrM est basé sur μManager - un logiciel de contrôle de microscope basé sur NIH-ImageJ - et facilite l’auto-formation des utilisateurs et l’automatisation des protocoles d’acquisition d’images courants. AnViM est basé sur NIH-ImageJ et facilite l’automatisation conviviale de l’analyse des données et la visualisation perspicace des résultats. Ces expériences consistent à cultiver des cellules adhérentes sur des hydrogels, à imager des marqueurs fiducial intégrés dans l’hydrogel et enfin à extraire de ces images une caractérisation mécanique complète des cellules. Actuellement, iTACS permet à l’utilisateur d’analyser et de suivre un large éventail de propriétés, y compris la morphologie, le mouvement, les forces cytosquelettiques et la fluorescence des cellules individuelles et de leur région voisine. La question de la normalisation de la qualité a été abordée dans AcTrM avec une technique de recentrage guidée par l’image de référence. Les problèmes techniques liés à l’analyse des données ont été abordés dans AnViM avec une procédure de segmentation d’image à plusieurs volets, une approche conviviale pour identifier les conditions aux limites et une nouvelle visualisation des données basée sur les propriétés cellulaires. AcTrM est conçu pour faciliter la transformation simple des microscopes à fluorescence de base en plates-formes expérimentales de mécanique cellulaire, et AnViM est équipé pour permettre aux utilisateurs de mesurer les signaux mécaniques cellulaires sans nécessiter de formation en ingénierie. iTACS sera disponible pour la communauté de recherche en tant que suite open source avec des capacités de développement pilotées par la communauté.
Introduction
Les outils d’imagerie optique et d’analyse de données couramment utilisés utilisent des technologies matérielles et logicielles presque désuètes. Le retard dans la traduction et la mise en œuvre des progrès dans les dispositifs électroniques, les approches informatiques et l’analyse mathématique dans les outils expérimentaux communs de biologie cellulaire est une contrainte majeure sur le rythme de croissance de nos connaissances en physiologie cellulaire. Actuellement, les chercheurs en biologie cellulaire trouvent des outils de biologie moléculaire à portée de main, mais des outils basés sur des principes d’ingénierie sont hors de portée. L’un de ces outils basés sur des principes d’ingénierie est la microscopie à contrainte monocouche (MSM)1,2. Bien que le MSM ait été adapté et étudié dans divers laboratoires à travers le monde, son utilisation se limite principalement aux laboratoires ayant une expertise en ingénierie3,4,5,6,7,8,9.
NIH-ImageJ est l’un des outils open source les plus populaires parmi les chercheurs en biologie cellulaire10. Les progrès de la communauté d’utilisateurs axés sur la contribution ont été au cœur de sa popularité11,12. ImageJ possède des fonctionnalités qui permettent aux utilisateurs de développer des applications avec un mélange d’un langage de programmation avancé et d’approches de script simplifiées. Ces fonctionnalités permettent aux utilisateurs d’acquérir des connaissances de base en programmation pour implémenter, adapter et faire progresser toute nouvelle contribution au logiciel. En nous appuyant sur ces qualités de NIH-ImageJ, nous avons développé l’Integrative Toolkit to Analyze Cellular Signals (iTACS), qui permet une intégration à faible coût des outils matériels et logiciels souhaités pour automatiser la mesure d’une grande variété de signaux chimiques et mécaniques à travers les cellules adhérentes11,12.
iTACS comprend deux composantes : le module d’acquisition et de formation (AcTrM) et le module d’analyse et de visualisation (AnViM). AcTrM est basé sur μManager - une application d’acquisition d’images basée sur NIH-ImageJ - pour permettre aux utilisateurs de configurer des mesures en accéléré des propriétés optiques traditionnelles et d’une variété de propriétés physiques des cellules adhérentes dans plusieurs échantillons12. AcTrM facilite la formation des utilisateurs grâce à des instructions concises incluses dans l’interface graphique. En outre, il dispose d’une nouvelle fonctionnalité de mise au point automatique basée sur des images de référence conçue pour faciliter les mesures en temps réel des forces physiques et permettre une normalisation de la qualité des données acquises.
AnViM est construit sur des plugins ImageJ, des logiciels accélérés et des scripts de gestion de fichiers qui permettent aux utilisateurs d’évaluer quantitativement plus de 50 propriétés, y compris la forme cellulaire, la taille, l’orientation, la vitesse et la direction du mouvement, les tractions exercées sur la matrice extracellulaire (ECM), et sur les cellules voisines, les moments contractiles et de cisaillement des cellules adhérentes individuelles et de leur région voisine. AnViM permet aux utilisateurs de quantifier les propriétés physiques cellulaires sans maîtriser le contexte technique sous-jacent11. En outre, il permet l’analyse des données en mode interactif ou par lots. Il génère des cartes thermiques révélant la variation spatiale et des graphiques montrant la variation temporelle des propriétés des cellules individuelles.
Dans une expérience typique, l’utilisateur cultive des cellules sur un hydrogel élastique avec des protéines de matrice extracellulaire appropriées sur la surface supérieure et deux types de marqueurs fluorescents intégrés. Essentiellement, les images de ces marqueurs fluorescents avant et après la culture des cellules sont suffisantes pour quantifier les forces à l’intérieur et autour des cellules individuelles2,13. AnViM mappe ces résultats sur des cellules individuelles du cluster adhérent et génère des images et des graphiques perspicaces.
Protocole
REMARQUE: Les échantillons examinés à l’aide de la plate-forme iTACS sont des cellules collées à un substrat mou. Le protocole d’évaluation des signaux mécaniques et chimiques est divisé en deux parties séquentielles : module d’acquisition et de formation (AcTrM) et module d’analyse et de visualisation (AnViM).
1. Module d’acquisition et de formation (AcTrM)
REMARQUE: AcTrM automatise le processus d’acquisition de données et d’auto-formation des utilisateurs. Avant toute acquisition de données, préparez un substrat souple capable de fournir les informations nécessaires pour quantifier les forces que les cellules exercent sur lui.
- Préparation d’hydrogel
REMARQUE: L’objectif ici est de préparer un module de cisaillement de 1 250 Pa, d’environ 100 μm d’épaisseur et d’hydrogel de polyacrylamide (PAA) de 22 mm de diamètre.- Préparer la solution de polyacrylamide en suivant la méthode de Yeung et al. et couler les hydrogels en suivant les étapes décrites par Trepat et al.14,15. Une exception à la procédure est que les perles incorporées immédiatement sous les cellules ont un diamètre de 0,5 μm et émettent une fluorescence jaune.
- Sautez l’étape consistant à fixer des billes de 2 μm à la vitre de couverture si la zone de visualisation doit avoir une grande région sans cellule.
NOTE: Le protocole de préparation de l’hydrogel est maintenant assez établi sur le terrain16. Tout au long de la description suivante, les perles de 0,5 μm sont appelées « perles supérieures » et les perles de 2 μm sont appelées « perles inférieures ». Toutefois, les perles inférieures sont facultatives lorsque la région imagée contient une grande zone sans cellule. Le motif de perle supérieure d’une telle région sans cellule servira le motif de perle inférieure. - Montez l’hydrogel avec des perles fluorescentes intégrées sur la scène du microscope.
- Prévoyez 15-20 min pour que la température de la plaque atteigne un état d’équilibre.
REMARQUE: La surface supérieure de la plaque est fonctionnalisée avec des protéines de matrice extracellulaire, mais les cellules ne sont pas encore ensemencées sur l’hydrogel.
- Acquisition d’images de référence
- Partie 1 : Création d’une liste de postes
REMARQUE: La saisie manuelle dans cette étape comprend le suivi des invites AcTrM pour choisir les emplacements avec la meilleure distribution de perles. Aucun fichier créé précédemment n’est utilisé dans cette étape. Les nouveaux dossiers clés produits dans cette étape incluent les dossiers 't0imgs', 'tnimgs' et 'textfiles', et le fichier produit inclut le fichier de liste de positions. Reportez-vous à la Figure 2 pour la description visuelle des étapes suivantes qui guident la création d’une liste de positions à l’aide d’AcTrM. L’exemple démontré acquiert des données à un grossissement de 20x.- Démarrez μManager 2.0 - Bêta.
- Exécutez AcTrM en cliquant sur le bouton AcTrM.bsh dans la fenêtre Ressources IMBL .
- Dans la fenêtre intitulée Définir les propriétés d’acquisition, sélectionnez l’objectif approprié, la tolérance pour la précision de la récupération de position latérale (c.-à-d. XY), la taille de l’étape de la mise au point approximative et raffinée (c.-à-d. z) et la valeur du coefficient de corrélation d’image acceptable pour la récupération de la mise au point.
REMARQUE: Une tolérance plus fine pour la récupération de position latérale ralentira la récupération de position, mais une tolérance substantielle nécessitera une correction de position pendant l’analyse des données et peut entraîner des difficultés de récupération de la mise au point. Une taille de pas plus petite ralentira l’opération de recentrage, mais une taille de pas très élevée peut provoquer un mouvement rapide avec des occasions de manquer la mise au point. - Dans la fenêtre AcTrM - Étape 0 , sélectionnez Nouvelle acquisition pour créer une liste de positions.
REMARQUE: La fenêtre intitulée AcTrM - Étape 1 répertorie toutes les étapes suivantes clés. Ces étapes impliquent de déplacer la scène, d’ajuster la mise au point et de cliquer sur les boutons du μManager. Ces étapes guident la construction de la liste des postes adaptés à l’acquisition de données. - Cliquez sur Live in Micromanager pour visualiser les échantillons pour les réglages manuels répertoriés dans la fenêtre AcTrM - Étape 1 .
- Suivez les étapes indiquées dans cette fenêtre.
- Maintenant, acquérez l’image des perles. Sélectionnez le canal approprié pour les perles supérieures.
- Assurez-vous également de regarder les perles inférieures. Pour ce faire, sélectionnez les canaux pour les perles inférieures. Une image floue de perles du bas est vue.
REMARQUE: Les canaux peuvent être commutés à partir du menu déroulant prédéfini de la fenêtre principale de μManager. Si les perles inférieures sont utilisées dans l’expérience, assurez-vous qu’elles sont présentes dans la position sélectionnée. Ces perles apparaîtront floues. - Après avoir sélectionné la position appropriée, cliquez sur Marquer dans la fenêtre Liste des positions de la scène pour enregistrer la position.
REMARQUE : La meilleure position est définie par une distribution dense et uniforme des perles supérieures encastrées immédiatement sous la surface supérieure (c.-à-d. les perles supérieures) et d’au moins deux perles attachées au verre de couverture (c.-à-d. les perles inférieures) (figure supplémentaire S1). La récupération de la mise au point est plus rapide si les perles inférieures apparaissent comme de grandes bagues floues. Cependant, si les expériences sont effectuées dans une position sans besoin de mise au point ou de récupération de position latérale, ignorez les instructions relatives à l’image des perles inférieures. - Suivez les étapes 6 à 9 décrites à la figure 2 pour inclure des positions supplémentaires dans la liste.
REMARQUE: Choisissez quelques positions supplémentaires afin qu’un tour d’élimination puisse permettre les meilleures positions choisies sur la plaque.
- Partie 2 : Acquisition d’images de référence
REMARQUE: Cette étape n’implique aucune saisie manuelle. Les fichiers clés précédemment créés et utilisés dans cette étape incluent 'textfiles/*.pos'. Les nouveaux fichiers et dossiers clés produits au cours de cette étape incluent ceux des dossiers 't0imgs' et 'tnimgs'. Reportez-vous à la Figure 3 et à la Figure S2 supplémentaire pour la description visuelle des étapes suivantes qui guident l’acquisition d’images de référence à l’aide d’AcTrM. La fenêtre AcTrM - Étape 2 répertorie également toutes les étapes clés.- En suivant les étapes répertoriées dans la fenêtre AcTrM - Étape 2 , faites les choix dans la fenêtre Acquisition multidimensionnelle . Par exemple, pour effectuer un laps de temps long, indiquez un certain nombre d’images à prendre, sélectionnez le premier canal comme canal de phase, puis sélectionnez le suivant pour les perles supérieures et celui qui suit est pour les perles inférieures. Cliquez sur Fermer dans la fenêtre Acquisition multidimensionnelle , puis cliquez sur OK dans la fenêtre ActrM - Étape 2 .
- Dans la fenêtre AcTrM - Étape 3 , choisissez d’activer la récupération de position XYZ basée sur l’image de référence. Le choix est généralement oui. Cependant, si les images sont acquises à une position sans place pour la mise au point ou la dérive de la scène, le choix dans la fenêtre AcTrM - Étape 3 sera Non.
- Dans la fenêtre AcTrM - Options de récupération XYZ , définissez le canal pour la récupération XYZ approximative, si vous souhaitez effectuer une récupération XYZ affinée, une région et un canal pour une récupération XYZ affinée et la direction à suivre pour commencer le recentrage (Figure supplémentaire S4). Une fois terminé, AcTrM acrégera des images de référence.
REMARQUE: Les choix typiques pour le canal seront le canal des perles inférieures. La récupération XYZ fonctionne mieux lorsqu’elle est effectuée avec l’image complète dans l’implémentation actuelle. Les images de référence comprennent généralement une image de lumière transmise de l’hydrogel, une image fluorescente des perles supérieures et une image fluorescente des perles inférieures. Le contenu de chaque ensemble d’images peut varier en fonction des choix effectués dans la fenêtre Acquisition multidimensionnelle . Néanmoins, le logiciel acquerra l’image de référence définie à chaque position déterminée à la figure 2. À la fin de cette étape, trois dossiers sont créés dans le répertoire choisi : 't0imgs', 'tnimgs' et 'textfiles'. Le dossier 't0imgs' contient des images de référence dans le format reconnu par μManager et utilisé dans les étapes suivantes; Le dossier 'tnimgs' contient des images TIFF séparées avec des noms de fichiers 'c0_p0_t0.tif', 'c1_p0_t0.tif', etc. Ici, 'c' représente le canal, 'p' représente la position et 't' représente l’heure. Ces lettres suivent respectivement le numéro de canal, le numéro de position et le numéro d’image. Le dossier 'textfiles' contient la liste des positions au format XML et les choix de l’utilisateur pour l’acquisition d’images et la récupération de position.
- Partie 1 : Création d’une liste de postes
- Ensemencement et croissance des cellules
REMARQUE: La plate-forme iTACS a la flexibilité nécessaire pour s’adapter aux protocoles de préparation d’échantillons utilisés dans l’évaluation in vitro courante du comportement mécanique des cellules adhérentes, y compris les cellules clairsemées, les monocouches entièrement confluentes, le test de formation de réseau et les monocouches avec des trous ou des lacunes importantes.- Cultiver des cellules endothéliales microvasculaires pulmonaires de rat pour confluencer dans une fiole17,18.
- Détachez les cellules à l’aide de la trypsine. Remettre en suspension les cellules dans un milieu de culture contenant 10 % de sérum fœtal bovin à une concentration de 1 x 106 cellules/mL.
- Placez une gouttelette de 5 μL des cellules remises en suspension sur une surface d’hydrogel partiellement sèche et placez-la dans l’incubateur de culture cellulaire.
REMARQUE: Après 2 jours dans le milieu de culture contenant 10% de sérum bovin fœtal, les cellules de cette gouttelette forment un îlot de cellules encombré1.
- Acquisition automatisée d’images pour l’expérience restante
REMARQUE: La saisie manuelle pour cette étape comprend le respect des invites AcTrM pour reprendre l’acquisition d’image. Les principaux fichiers précédemment créés utilisés par cette étape incluent 'textfiles/*.pos' et les fichiers image de perles inférieures dans le dossier 't0imgs'. Les nouveaux fichiers clés produits à cette étape incluent des fichiers de liste de positions mis à jour et des images 'tnimgs/*_t*.tif'.- Assurez-vous que le système de contrôle de l’environnement du microscope atteint des conditions de culture tissulaire stables.
- Montez doucement la plaque contenant les cellules cultivées sur la scène du microscope.
- Prévoir 15-20 min pour que la température et l’humidité se stabilisent.
REMARQUE : Reportez-vous à la figure 4 pour la description visuelle des étapes suivantes qui guident l’acquisition d’images pour l’expérience restante à l’aide d’AcTrM. - Démarrez μManager 2.0 - Bêta.
- Exécutez AcTrM en cliquant sur le bouton AcTrM.bsh dans la fenêtre Ressources IMBL .
Remarque : Ignorez les choix effectués dans la fenêtre Définir les propriétés d’acquisition , mais utilisez les choix effectués à l’étape précédente. - Dans la fenêtre AcTrM - Étape 0 , sélectionnez Continuer l’acquisition suspendue.
- Sélectionnez le grossissement et le répertoire identifiés dans la fenêtre Acquisition multidimensionnelle de l’étape précédente où les dossiers de données ('t0imags', 'tnimgs' et 'textfiles') ont été enregistrés.
REMARQUE : Le titre de la fenêtre guide l’utilisateur pour choisir le répertoire approprié. - Dans la fenêtre Repositionnement de la plaque , sélectionnez les choix de repositionnement de la plaque (trois positions non collinéaires) ainsi que le canal utilisé pour le repositionnement.
REMARQUE: Les images enregistrées seront visibles dans l’appareil photo. Si les images qui se chevauchent s’affichent sous forme d’image en couleur rouge, verte et noire, effectuez un réglage manuel. - Cliquez sur Accepter pour procéder à l’acquisition.
REMARQUE: Cette étape surmonte les légers changements par rapport à l’irreproductibilité de l’alignement de la plaque lorsque la plaque est remontée sur la scène du microscope. Suivez les invites AcTrM pour accélérer l’alignement à chacune des trois positions en affichant une image composite de l’image de référence en rouge (généralement des perles inférieures) et de l’image actuellement observée à travers l’objectif affiché en vert. Obtenir le vert suffisamment proche du rouge laisse moins de travail pour le logiciel. Lorsque le chevauchement est parfait, l’image composite apparaît en jaune et noir. On est généralement suffisamment proche lorsque les mêmes perles inférieures sont visibles dans les images rouges et vertes, et que les bagues rouges et vertes correspondantes se touchent. Une fois le repositionnement de la plaque terminé, AcTrM amène la scène à chaque position sélectionnée et récupère la position XYZ en faisant correspondre l’image de référence avec ce qui est actuellement vu à travers la caméra. La première position latérale (XY) est mise en correspondance, puis la mise au point (Z) est mise en correspondance. La récupération brutale est suivie d’une récupération affinée de la position et de la concentration. Les mises à jour de l’état actuel de l’expérience sont affichées à l’écran par le biais d’une fenêtre en quatre parties illustrée à la figure supplémentaire S3. Les images acquises sont enregistrées dans le dossier 'tnimgs' suivant '*_t1.tif', '*_t2.tif', indiquant le nombre de temps pour l’ensemble d’images. Si la récupération XYZ identifie une position mise à jour, la nouvelle liste de positions est générée et enregistrée dans le dossier 'textfiles'.
2. Module d’analyse et de visualisation (AnViM)
- Mise en place d’une analyse automatisée des données
REMARQUE: La saisie manuelle dans cette étape comprend l’identification de l’emplacement du dossier 'tnimgs'. Les principaux fichiers précédemment créés utilisés dans cette étape incluent 'tnimgs/*.tif'. Les nouveaux fichiers et dossiers clés produits à cette étape incluent le dossier 'analysis' dans le même dossier parent que 'tnimgs' et les dossiers pour chaque position 'analysis/p*'. Dans chaque 'analyse/p*', cette étape crée un nouveau fichier image '*.tif' dans les dossiers 'phs' et 'tny'. Ces images sont des images recadrées et dérivées de cellules et de perles supérieures. Les autres fichiers créés incluent 'analysis/analysisChoices.txt' qui répertorie les choix d’analyse, 'analysis/p*/skipAnalysis.txt', qui répertorie les cas où l’analyse est ignorée en raison d’une dérive préexistante importante, et 'analysis/p*/part_shift_values_pixel_degree.dat' qui répertorie les valeurs de dérive estimées. AnViM ne modifie pas les données brutes dans le dossier 'tnimgs'. Reportez-vous à la figure 5 pour la description visuelle des étapes suivantes qui guident la configuration de l’analyse automatisée des données à l’aide d’AnViM.- Démarrez le logiciel Fidji et sélectionnez la première option dans le menu déroulant MSM intitulé MSM - prétraitement.
- À l’aide de la boîte de dialogue de l’explorateur de fichiers, sélectionnez le dossier contenant le dossier 'tnimgs', qui contient les données analysées.
- Définissez le canal des images. En suivant les directives de la figure supplémentaire S5, identifiez les numéros de canal de l’image de lumière transmise des cellules (image de contraste de phase des cellules), l’image des perles inférieures et l’image des perles supérieures. Les trois cases à cocher suivantes (« Déplacer les fichiers vers le dossier de position », « Effectuer une correction supplémentaire pour un mouvement rigide » et « Recadrer les données et enregistrer dans le dossier de position ») sont généralement cochées. Enfin, définissez la tolérance pour le rejet des données fortement dérivées, la taille des pixels et les côtés de l’image où les cellules se croisent.
- Répondez à l’invite pour améliorer la luminosité et le contraste des images de perles inférieures afin que les perles apparaissent bien en évidence. Ajustez ceci à l’aide de la barre de défilement dans le menu et cliquez sur OK.
- Maintenant, effectuez une correction de position pour vous débarrasser des quarts de travail, le cas échéant. Une fois cela fait, un dossier d’analyse est créé.
REMARQUE: L’amélioration du contraste n’est généralement pas nécessaire, mais cette disposition est disponible pour les expériences où l’exposition aux lasers doit être minimisée. Après ce choix, AnViM copie les fichiers du dossier 'tnimgs' vers le dossier 'analysis'. Les fichiers correspondant à chaque position sont enregistrés dans les dossiers 'analysis/p0, analysis/p1', etc. Dans chacun de ces dossiers de position, AnViM crée des dossiers 'cels', 'defs' et 'refs' contenant respectivement l’image lumineuse transmise d’origine, des images de perles supérieures et des images de perles inférieures (figure supplémentaire S6). AnViM analyse ensuite le mouvement rigide dans les images des perles inférieures et crée un dossier 'phs' contenant une image de lumière transmise corrigée des cellules et un dossier 'tny' contenant des images de perles supérieures mises à jour. Enfin, les choix de l’utilisateur concernant les canaux, les opérations, la tolérance, la taille des pixels et le franchissement des limites sont enregistrés dans le fichier 'analysisChoices.txt' (Figure supplémentaire S6).
- Quantification de la déformation de l’hydrogel et de la monocouche
REMARQUE: Il est important de noter que la quantification du mouvement à partir d’une séquence d’images est un domaine en évolution rapide19. La technologie est constamment optimisée pour les fonctionnalités, y compris la vitesse, la précision, les caractéristiques spécifiques dans les images brutes et les modèles de déformation spécifiques. Par conséquent, il est probable que certains utilisateurs utilisent une approche de quantification de la déformation différente de celle présentée ici.-
Partie 1 : Quantification de la déformation de l’hydrogel
REMARQUE: Les entrées manuelles de cette étape incluent l’identification du répertoire de données et la sélection de la résolution de grille. Les principaux fichiers précédemment créés utilisés dans cette étape incluent 'p*/tny/*.tif'. Les nouveaux fichiers clés produits dans ce domaine incluent 'p*/displacement/*_disp.dat' qui répertorie les vecteurs de déplacement de la surface supérieure de l’hydrogel. Voir la Figure 6 pour la description visuelle des étapes suivantes pour engager, via AnViM, l’analyse de vélocimétrie d’image de particules sur les images de perles supérieures20.- Démarrez le logiciel Fidji et, dans le menu déroulant MSM , sélectionnez MSM - Gel Deformation.
- À partir de là, sélectionnez l’option qui convient à l’expérience.
- À l’aide de la boîte de dialogue de l’explorateur de fichiers, sélectionnez le répertoire parent du dossier 'analysis' contenant les données analysées.
- Dans la fenêtre Paramètres de calcul de la déformation du gel , sélectionnez la taille de la fenêtre de corrélation croisée, le niveau de bruit et le seuil appropriés (figure supplémentaire S7)20.
REMARQUE: Les résultats sont enregistrés dans un répertoire « déplacement » nouvellement créé dans chaque dossier de position « analyse / p0, analyse / p1 », etc. C’est là que tous les fichiers de sortie sont stockés.
-
Partie 2 : Quantification de la déformation monocouche
REMARQUE: Les entrées manuelles de cette étape incluent l’identification du répertoire de données et la sélection de la résolution de grille. Les principaux fichiers précédemment créés utilisés dans cette étape incluent 'p*/tny/*.tif'. Les nouveaux fichiers clés produits dans ce domaine incluent 'p*/velocity/*_vel.dat' qui répertorie les vecteurs de mouvement des cellules. Voir la figure 7 pour la description visuelle des étapes à suivre pour engager, via AnViM, l’analyse de vélocimétrie d’image de particules sur les images de perles supérieures20. La procédure est similaire à celle suivie pour quantifier la déformation de l’hydrogel et sélectionne MSM - Cellular Motion dans le menu déroulant MSM . Les résultats sont enregistrés dans un répertoire 'velocity' nouvellement créé dans chaque dossier de position 'analysis/p0', 'analysis/p1', etc.
-
Partie 1 : Quantification de la déformation de l’hydrogel
- Quantification de l’ECM et des forces cellule-cellule
REMARQUE: La saisie manuelle dans cette étape comprend l’identification du répertoire de données, l’indication de la rigidité de l’hydrogel et la réponse aux invites de segmentation des clusters cellulaires pour détecter les cellules de la région sans cellule. Les principaux fichiers précédemment créés utilisés dans cette étape incluent 'p*/displacement/*_disp.dat'. Les nouveaux fichiers clés produits à cette étape comprennent: « p * / traction / * _trac.dat », qui répertorie les forces exercées par les cellules sur l’hydrogel; « p*/traction/*_domain.dat », qui indique l’emplacement des points de la grille contenant des cellules; et les fichiers d’entrée 'p*/traction.in, p*/clusterInput.txt' et 'p*/prestress.in' qui enregistrent les choix de l’utilisateur pour cette étape. À la suite de la première évaluation quantitative de l’ECM cellulaire et des forces cellule-cellule, plusieurs variantes de la technique ont été développées1,15. Les variations se concentrent sur des cas particuliers de substrats, de cellules, de conditions expérimentales ou d’outils numériques7,8,21,22. Voir la figure 8 pour la description visuelle à engager via AnViM, la microscopie de traction à transformée de Fourier et l’analyse de microscopie de contrainte monocouche sur les données de déformation de l’hydrogel1,2,15.- Démarrez le logiciel Fidji et, dans le menu déroulant MSM , sélectionnez MSM - Cell-ECM et Cell-Cell Forces (troisième option dans le menu déroulant).
- À l’aide de la boîte de dialogue du navigateur de fichiers, sélectionnez le répertoire parent du dossier 'analysis' contenant les dossiers 'tnimgs' et 'analysis' contenant les données analysées. Cliquez sur Sélectionner.
- Dans la fenêtre Paramètres de calcul des forces cellulaires , entrez le module de cisaillement, l’épaisseur de l’hydrogel et le niveau de bruit prévu. Sélectionnez OK. Cela lui permet d’exécuter la traction via la fonction MATLAB.
REMARQUE: Après ces entrées, AnViM calcule les forces cell-ECM. - Après cela, indiquez si la monocouche est confluente. Si l’image entière de la cellule est recouverte de cellules, la réponse est Oui. Dans ce cas, les forces cellule-cellule seront calculées sur l’ensemble du cadre. D’autre part, si une partie de l’image n’a pas de cellules, alors la réponse est Non. Dans ce cas, suivez les invites AnViM pour faciliter la segmentation de la région d’image qui contient des cellules.
- Si la réponse est non, dessinez manuellement un polygone autour de l’objet non-cellule lorsque le logiciel vous invite à le faire, puis sélectionnez une ou plusieurs méthodes appropriées pour la segmentation. Le logiciel demandera la couleur (noir ou blanc) des cellules.
- Cochez l’option Remplir automatiquement les taches dans le logiciel et cliquez sur OK.
NOTE: Les étapes de segmentation d’une monocouche non confluente seront abordées dans la première partie de la section 2.4. Les forces Cell-ECM sont stockées dans des fichiers '*_trac.dat' dans un répertoire 'traction' nouvellement créé dans chaque dossier de position, et l’entrée dans le logiciel de calcul de force cell-ECM est enregistrée dans le fichier 'traction.in' (Figure supplémentaire S8). Les forces cellule-cellule sont stockées dans des fichiers '*_prestress.dat' dans un répertoire nouvellement créé 'prestress' dans chaque dossier de position, et l’entrée dans le logiciel de calcul de force cellule-cellule est enregistrée dans le fichier 'prestress.in' (Figure supplémentaire S9).
- Mappage des valeurs des points de grille sur des cellules individuelles
REMARQUE: L’un des objectifs actuels d’iTACS est d’introduire une approche simple pour interpréter les signaux mécaniques mesurés sur le terrain. Cette approche est bénéfique pour examiner les interactions mécaniques entre les cellules voisines d’un cluster18. Dans l’ensemble, les propriétés quantifiées jusqu’à présent se trouvent sur des points de grille régulièrement espacés à travers le cluster cellulaire. Ces données identifient la médiane, la moyenne et l’écart-type des propriétés sélectionnées à l’intérieur des limites morphologiques des cellules individuelles et les attribuent en tant que propriétés/signaux physiques cellulaires. À partir de ceux-ci, l’écart-type est utilisé pour indiquer la variabilité, la différence entre la moyenne et la médiane est utilisée pour indiquer la nature de la distribution, et la valeur médiane est utilisée pour indiquer l’état global des cellules pour les propriétés sélectionnées.- Partie 1 : Segmentation de la région d’image qui contient des cellules
REMARQUE : la saisie manuelle de cette étape comprend l’identification du répertoire de données et la réponse aux invites de segmentation pour détecter les cellules de la région sans cellule. Les principaux fichiers précédemment créés utilisés dans cette étape incluent 'p*/phs/*.tif'. Les nouveaux fichiers clés produits à cette étape incluent 'p*/phs/bwImages/*.tif' qui sont des images binaires séparant les régions cellulaires des régions sans cellule, 'p*/phs/textResults/frameNum_percentHealed.txt', qui répertorie le pourcentage de zone d’image couverte par les cellules dans chaque instance, et 'p*/clusterInput.txt', qui enregistre les choix utilisateur saisis dans l’interface AcTrM. Reportez-vous à la Figure 9 pour obtenir la description visuelle des étapes suivantes pour segmenter la région de l’image qui contient des cellules. Cette partie doit être complétée avant de calculer les forces cellule-cellule. Dans ce cas, ces étapes n’ont pas besoin d’être répétées. De plus, si ces étapes sont effectuées avant les calculs de force cellule-cellule, elles ne sont pas demandées au début des calculs de force.- Démarrez le logiciel Fidji et, dans le menu déroulant MSM , sélectionnez Segmentation - Cluster cellulaire.
- À l’aide de la boîte de dialogue de l’explorateur de fichiers, sélectionnez le répertoire de position 'analysis/p0', 'analysis/p1', etc., qui contient des propriétés quantifiées sur des points de grille régulièrement espacés.
- Indiquez si la monocouche est confluente.
REMARQUE: Les étapes ci-dessous ne sont appelées que lorsque la monocouche n’est pas confluente. Suivez les invites AnViM pour appliquer la nouvelle approche à plusieurs volets afin d’identifier les régions d’image qui contiennent des cellules. Le processus implique quatre méthodes - chacune abordant la segmentation d’une manière différente. Une ou plusieurs de ces méthodes utilisées en combinaison couvrent une grande variété d’images des cellules. Par conséquent, essayez différentes approches pour trouver la combinaison qui fonctionne le mieux pour leurs données. - Ajustez le 'Facteur de connexion au domaine' et le 'Facteur de frottis' pour obtenir une segmentation optimale.
REMARQUE: Le « facteur de frottis » rend les régions qui contiennent des cellules plus grandes. - Indiquez si les cellules apparaissent en noir ou blanc dans l’image binaire.
- Dans la fenêtre Instructions pour nettoyer l’image de frontière , choisissez de nettoyer l’image automatiquement ou manuellement.
REMARQUE: Les caractéristiques indésirables des images sont des taches noires sur les pixels occupés par les cellules et des taches blanches sur les pixels déconnectés du groupe de cellules à analyser. L’analyse ne peut être effectuée que sur les régions connectées. Ainsi, plusieurs régions déconnectées doivent être analysées séparément.
- Partie 2: Segmentation des cellules individuelles dans les images
REMARQUE: Les entrées manuelles de cette étape incluent l’identification du répertoire de données et la réponse aux invites de segmentation pour détecter les cellules individuelles dans l’image. Les principaux fichiers précédemment créés utilisés dans cette étape incluent 'p*/phs/*.tif' et 'p*/phs/bwImages/*.tif'. Les nouveaux fichiers clés produits incluent 'p*/phs/textResults/*.csv', qui contiennent des propriétés morphologiques cellulaires. Reportez-vous à la Figure 10 pour la description visuelle des étapes suivantes pour segmenter les cellules individuelles de la monocouche.- Démarrez le logiciel Fidji et, dans le menu déroulant MSM , sélectionnez Segmentation - Cellules individuelles.
- À l’aide de la boîte de dialogue de l’explorateur de fichiers, sélectionnez le répertoire de position 'analysis/p0', 'analysis/p1', etc., qui contient des propriétés quantifiées sur des points de grille régulièrement espacés.
- Dans la fenêtre Choisir les paramètres d’entrée , indiquez le rapport d’aspect maximal, la proéminence de la position du centre de la cellule par rapport à la limite de la cellule et deux paramètres de flou pour guider la détection des cellules (figure supplémentaire S10).
- Sur la pile d’images pour chaque position, dessinez un polygone sur la plus petite cellule normale. Ceci est utilisé pour calculer la zone. Tout ce qui est plus petit que cela ne sera pas considéré comme une cellule par le logiciel.
- Ensuite, dessinez un polygone autour de la plus grande cellule normale et indiquez si le centre cellulaire ou l’interface cellule-cellule est plus lumineux.
REMARQUE: AnViM crée ensuite des fichiers 'phs/textResults/0001.csv', 'phs/textResults/0002.csv', etc., pour chaque cadre contenant des informations sur les cellules de ce cadre. À ce stade, ce fichier comprend des informations morphologiques sur les cellules, y compris la surface, le centroïde, le périmètre, l’orientation, la circularité, le rapport d’aspect, l’arrondi, la solidité, la distance par rapport à la région sans cellule. L’unité de longueur dans ces propriétés est en pixels et l’angle est en degrés. Enfin, ce fichier est mis à jour pour contenir l’intensité, le mouvement et les forces des pixels cellulaires dans les étapes suivantes.
- Partie 3 : Cartographie des intensités de pixels sur les cellules
REMARQUE : Les entrées manuelles de cette étape incluent l’identification du répertoire de données et la sélection des propriétés et paramètres de mappage. Les principaux fichiers précédemment créés utilisés à cette étape incluent 'p*/phs/*.tif' (ou 'p*/fluo/*.tif ') et 'p*/phs/bwImages/*.tif'. Les nouveaux fichiers clés produits dans cette étape incluent 'p*/phs/textResults/*.csv', qui répertorie les propriétés morphologiques cellulaires. Reportez-vous à la figure 11 pour la description visuelle des étapes suivantes afin d’évaluer les intensités de pixels dans la région englobée par les cellules individuelles et leur région voisine et de les définir comme propriétés des cellules.- Démarrez le logiciel Fidji et, dans le menu déroulant MSM , sélectionnez Carte sur les cellules - Intensités.
- À l’aide de la boîte de dialogue de l’explorateur de fichiers, sélectionnez le répertoire de position 'analysis/p0', 'analysis/p1', etc., qui contient des propriétés quantifiées sur des points de grille régulièrement espacés.
- Sélectionnez Détecter la division cellulaire ou Quantifier la fluorescence cellulaire lorsque le logiciel vous y invite. Définissez la taille de la région voisine. Cochez les cases Collecter les propriétés de la région voisine et Collecter les propriétés des cellules. Cliquez sur OK.
- Dans la fenêtre But de l’enregistrement de l’intensité cellulaire, indiquez le type d’image à utiliser pour la cartographie de l’intensité, la taille de la région voisine et si des données sont collectées pour des cellules individuelles ou les deux cellules et leurs régions voisines (figure supplémentaire S11).
REMARQUE: Le mappage des intensités de pixels d’une image à contraste de phase permet de détecter les événements de division cellulaire. La cartographie des intensités d’images fluorescentes permettra de détecter les fluctuations des molécules cytoplasmiques marquées par fluorescence. La sortie des étapes ci-dessus est la moyenne, la médiane, l’écart-type, le minimum et le maximum d’intensité en pixels pour les cellules individuelles. Ces numéros sont saisis sous forme de nouvelles colonnes dans les fichiers 'phs/textResults/0001.csv', 'phs/textResults/0002.csv', etc.
- Partie 4: Cartographie des forces et du mouvement sur les cellules
REMARQUE : Les entrées manuelles de cette étape incluent l’identification du répertoire de données et la sélection des propriétés et paramètres de mappage. Les principaux fichiers précédemment créés utilisés dans cette étape incluent 'p*/phs/textResults/*.csv', 'p*/velocity/*_vel.dat', 'p*/displacement/*_disp.dat', 'p*/traction/*_trac.dat', 'p*/prestress/*_prestress.dat' et 'p*/phs/bwImages/*.tif '. Aucun nouveau fichier n’est créé au cours de cette étape. Au lieu de cela, les nouvelles informations (propriétés de force cellulaire et de mouvement) sont ajoutées aux fichiers 'p*/phs/textResults/*.csv '. Voir la figure 12 pour la description visuelle des étapes afin d’évaluer les forces et les mouvements dans la région englobée par les cellules individuelles et leur région voisine et de les définir comme propriétés des cellules.- Choisissez dans le menu déroulant MSM Map on Cells - Motion/Force Data (Figure supplémentaire S12).
- Ensuite, suivez les étapes répertoriées à l’étape 2.4.3.
REMARQUE: De nouvelles colonnes sont ajoutées aux fichiers 'phs/textResults/0001.csv', 'phs/textResults/0002.csv', etc. et incluent la moyenne, la médiane et l’écart-type de vitesse, l’orientation de la vitesse, la tension cytosquelettique moyenne, l’anisotropie de tension, l’énergie de déformation dans l’hydrogel, l’orientation de la tension la plus élevée et l’amplitude de la traction cellule-ECM (figure supplémentaire S13).
- Partie 1 : Segmentation de la région d’image qui contient des cellules
- Visualisation des résultats
- Partie 1: Suivi des identités cellulaires à travers l’expérience
REMARQUE : Les entrées manuelles de cette étape incluent l’identification du répertoire de données et la sélection des propriétés mappées à visualiser. Les principaux fichiers précédemment créés utilisés dans cette étape incluent 'p*/phs/textResults/*.csv'. Les nouveaux fichiers clés produits au cours de cette étape incluent 'p*/phs/textResults/cellTimeTrace_*.csv', qui contient une trace temporelle des données cellulaires. Reportez-vous à la figure 13 pour la description visuelle des étapes suivantes afin de suivre les propriétés des cellules individuelles pendant toute la durée de l’expérience.- Démarrez le logiciel Fidji et, dans le menu déroulant MSM , sélectionnez Résultats - Données de suivi.
- À l’aide de la boîte de dialogue du navigateur de fichiers, sélectionnez le répertoire de position 'analysis/p0', 'analysis/p1', etc., contenant les propriétés cellulaires à suivre. Lorsque vous avez terminé, cliquez sur Sélectionner.
- Dans la fenêtre Choisir le point de départ pour le suivi , choisissez l’image de départ pour la surveillance et le nombre d’images suivies simultanément (figure supplémentaire S14). Commencez toujours à partir de l’image numéro 2 car la vitesse ne peut pas être déterminée pour l’image numéro 1. Lorsque vous avez terminé, cliquez sur OK.
- Dans la fenêtre Vérifier les variables pour effectuer des pistes , choisissez les variables en tapant les noms des variables ou en cochant les termes dans les cases à cocher (Figure supplémentaire S15). Ensuite, cliquez sur OK.
REMARQUE : Le suivi génère des fichiers 'phs/textResults/cellTimeTrace_*.csv' avec chaque colonne contenant un numéro de cellule unique et chaque ligne successive représentant l’instance temporelle consécutive.
- Partie 2 : Génération de traces temporelles des propriétés cellulaires évaluées
REMARQUE : Les entrées manuelles de cette étape incluent l’identification du répertoire de données et la sélection des propriétés mappées à visualiser. Les principaux fichiers précédemment créés utilisés dans cette étape incluent 'p*/phs/textResults/cellTimeTrace_*.csv'. Les nouveaux fichiers clés produits au cours de cette étape incluent 'p*/phs/trackedDataPlots/*.tif', qui contiennent des tracés de suivi temporel et 'p*/phs/trackedDataPlots/*.csv', qui contiennent des données de tracé. Voir la figure 14 pour la description visuelle des étapes suivantes afin de générer des traces temporelles des propriétés cellulaires évaluées.- Démarrez le logiciel Fidji et, dans le menu déroulant MSM , sélectionnez Résultats - tracé.
- À l’aide de la boîte de dialogue du navigateur de fichiers, sélectionnez le répertoire de position 'analysis/p0', 'analysis/p1', etc., contenant les propriétés cellulaires suivies à tracer.
- Choisissez de limiter le tracé aux cellules avec des pistes ininterrompues en cliquant sur le bouton Oui ou Non .
Remarque : Cette option ignore les cellules, qui n’ont pas pu être détectées dans une ou plusieurs instances de temps. - Choisissez le nombre de variables à tracer simultanément et cliquez sur OK.
REMARQUE: Actuellement, AnViM permet d’afficher un maximum de trois variables dans le même tracé. - Choisissez des variables individuelles à tracer à l’aide d’un menu déroulant.
REMARQUE : ces étapes créent un fichier TIFF (tagged image file format) et un fichier de données séparé par des virgules dans le dossier 'phs/trackedDataPlots/'. Le nom de fichier est composé de noms de variables séparés par un trait de soulignement et se termine par un numéro de cellule.
- Partie 3 : Génération de cartes thermiques des propriétés cellulaires évaluées
REMARQUE : Les entrées manuelles de cette étape incluent l’identification du répertoire de données et la sélection des propriétés mappées à visualiser. Les principaux fichiers précédemment créés utilisés dans cette étape incluent 'p*/phs/textResults/cellTimeTrace_*.csv'. Les nouveaux fichiers clés générés à cette étape incluent 'p*/phs/segmentedImages/cellPropMaps/*.tif', qui sont les cartes thermiques des propriétés cellulaires. Voir la figure 15 pour la description visuelle des étapes permettant de générer des cartes thermiques des propriétés cellulaires évaluées.- Choisissez dans le menu déroulant MSM Résultats - Image.
- Suivez les étapes décrites à l’étape 2.5.2.
REMARQUE: La sortie est stockée sous forme de fichiers TIFF dans le dossier 'phs/segmentedImages/cellPropMaps/'. Les noms de fichiers sont le numéro d’instance de temps et le nom de la variable séparés par un trait de soulignement.
- Partie 1: Suivi des identités cellulaires à travers l’expérience
Résultats
Nous présentons ici deux des principaux résultats de l’exemple démontré. La première sortie est la trace temporelle de la vitesse cellulaire et de la tension cytosquelettique pour la cellule numéro 1 (Figure 16). Les propriétés sont affichées sur un axe vertical partagé pour faciliter l’association visuelle entre les propriétés, et l’axe horizontal indique le numéro d’instance temporelle. Dans cette expérience, des images successives ont été acquises à un intervalle de 15 minutes. La deuxième sortie est un tableau de cartes thermiques 1 h après le début de l’expérience (Figure 17). Les propriétés présentées ici comprennent la zone d’étalement, l’orientation, la circularité, la vitesse, la direction du mouvement, l’orientation de la tension maximale, la tension cytosquelettique, les tractions du substrat et l’anisotropie de tension des cellules individuelles.

Figure 1 : Structure de la boîte à outils intégrative pour analyser les signaux cellulaires (iTACS). Deux composants clés d’iTACS sont le module d’acquisition et de formation (AcTrM) et le module d’analyse et de visualisation (AnViM). AcTrM peut utiliser diverses techniques de préparation d’hydrogel qui existent actuellement pour préparer des hydrogels qui peuvent être maintenus fermement sur une scène de microscope, n’importe quel ensemencement cellulaire et un protocole de croissance qui retient les cellules dans un plan focal. AnViM peut utiliser diverses techniques pour quantifier la déformation de l’hydrogel et de la monocouche, les forces cellule-ECM et les forces cellule-cellule. Tous ces composants préférés par l’utilisateur du protocole de mesure de force peuvent être intégrés dans iTACS, et ils ont été identifiés avec des boîtes en pointillés. Les composants identifiés avec les boîtes solides sont de nouvelles contributions à la technologie de mesure de la force cellulaire. La visualisation dans l’AnViM se concentre sur la valeur médiane et la variabilité des propriétés entre les cellules individuelles. Veuillez cliquer ici pour voir une version agrandie de cette figure.
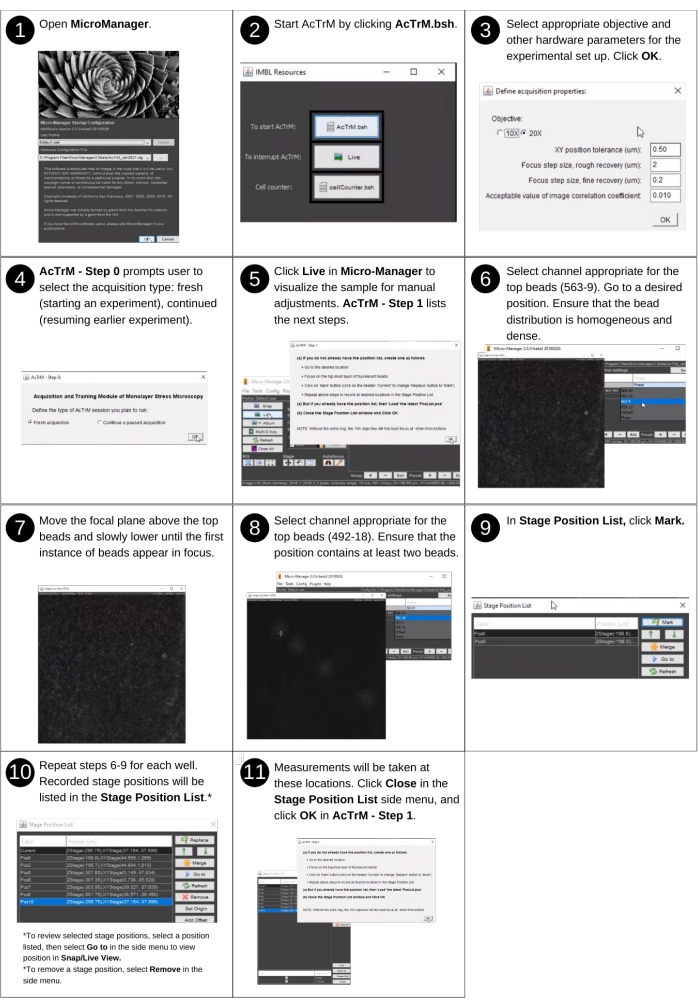
Figure 2 : Acquisition d’images de référence - partie 1. Étapes de création d’une liste de positions à l’aide d’AcTrM. Veuillez cliquer ici pour voir une version agrandie de cette figure.
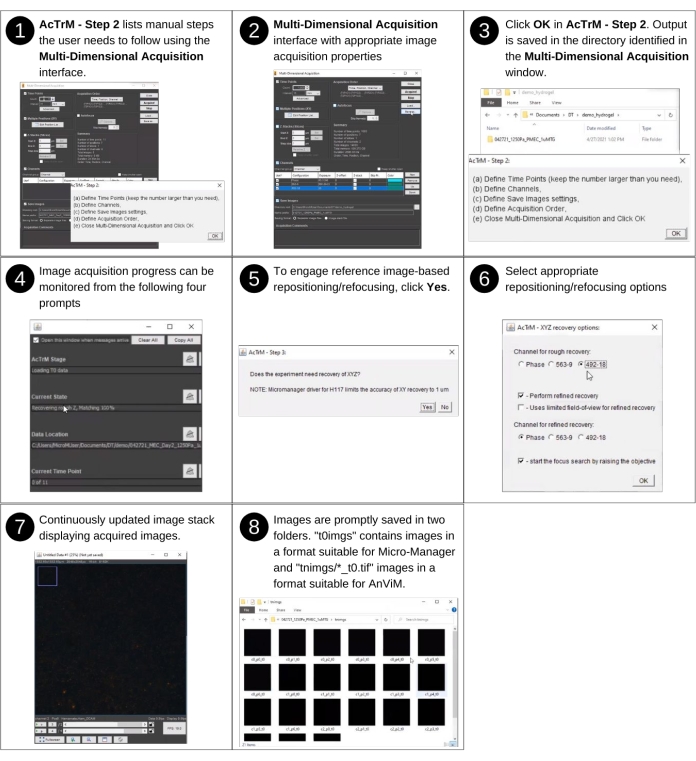
Figure 3 : Acquisition d’images de référence - partie 2. Étapes d’acquisition d’images de référence à l’aide d’AcTrM. Des vues détaillées des étapes 2, 4 et 6 sont présentées dans les figures supplémentaires S2, S3 et S4, respectivement. Veuillez cliquer ici pour voir une version agrandie de cette figure.
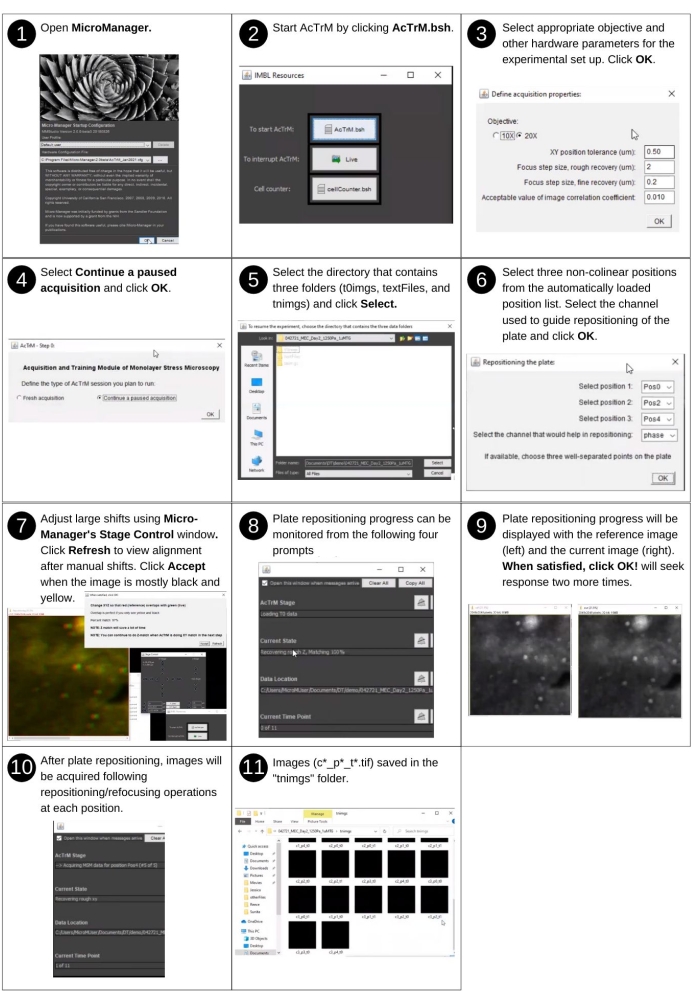
Figure 4 : Acquisition automatisée d’images pour l’expérience restante. Étapes de reprise de l’acquisition d’images pour évaluer le comportement cellulaire à l’aide d’AcTrM. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 5 : Configuration de l’analyse automatisée des données. Étapes pour commencer l’analyse automatisée d’images à l’aide d’AnViM. Le logiciel reconnaît le format d’image utilisé par AcTrM. Une vue détaillée des panneaux des étapes 3 et 5 est présentée dans la figure supplémentaire S5 et la figure supplémentaire S6, respectivement. Veuillez cliquer ici pour voir une version agrandie de cette figure.
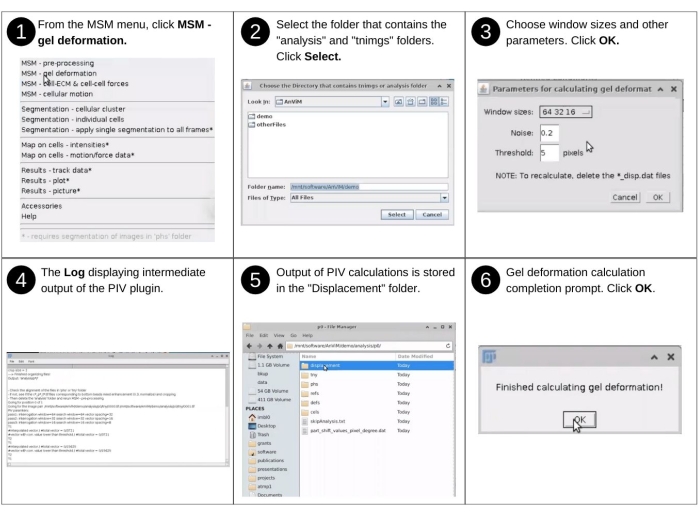
Figure 6: Quantification de la déformation de l’hydrogel et de la monocouche - partie 1. Étapes pour engager, via AnViM, l’implémentation de la vélocimétrie d’image de particules de Tseng, Q. et al., PNAS (2012)20 pour quantifier la déformation de la surface supérieure de l’hydrogel. Les utilisateurs peuvent également mettre en œuvre dans AnViM d’autres approches pour quantifier la déformation de l’hydrogel. Une vue détaillée de l’étape 3 est présentée à la figure supplémentaire S7. Veuillez cliquer ici pour voir une version agrandie de cette figure.
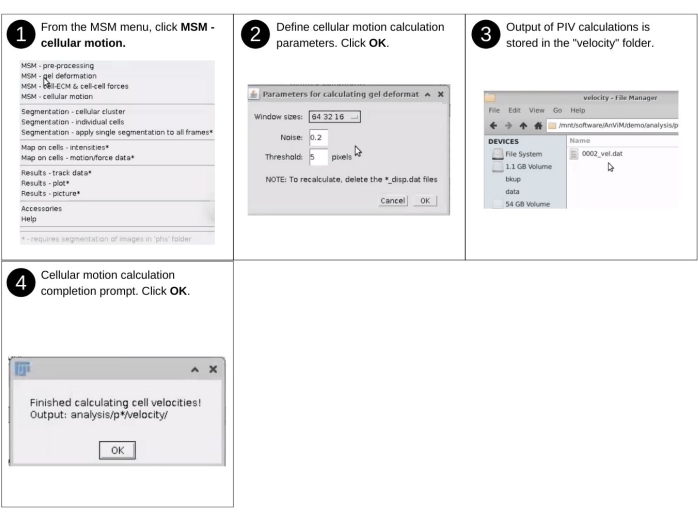
Figure 7: Quantification de la déformation de l’hydrogel et de la monocouche - partie 2. Étapes pour engager, via AnViM, l’implémentation particle Image Velocimetry de Tseng, Q. et al., PNAS (2012)20 pour quantifier le mouvement local des cellules individuelles. Les utilisateurs peuvent également implémenter dans AnViM d’autres approches pour quantifier le mouvement cellulaire. Veuillez cliquer ici pour voir une version agrandie de cette figure.
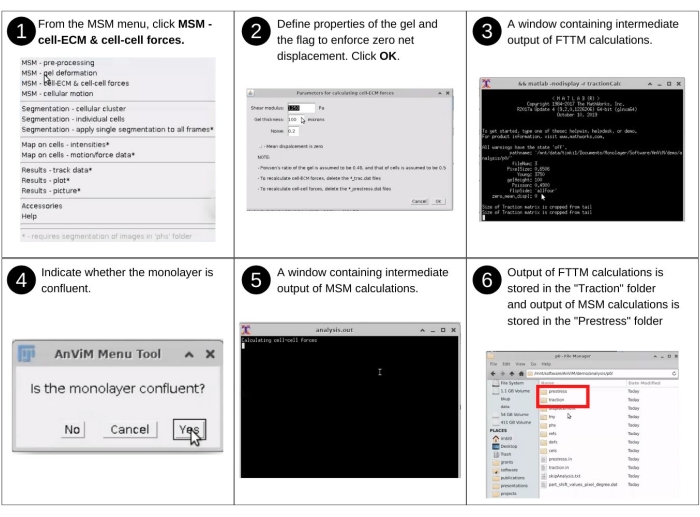
Figure 8 : Quantification des forces cellule-ECM et cellule-cellule. Étapes pour effectuer une analyse d’image pour engager, via AnViM, l’implémentation de microscopie de traction à transformée de Fourier de Trepat et al., Nature Physics (2009)15 pour quantifier les forces exercées par les cellules sur l’hydrogel, et l’implémentation monocouche de microscopie de stress de Tambe et al., Nature Materials (2011)1 pour quantifier les forces à l’intérieur des cellules individuelles et entre les cellules voisines. Les utilisateurs peuvent également mettre en œuvre dans AnViM d’autres approches pour quantifier les forces cellule-ECM et cellule-cellule. Une vue détaillée de l’étape 6 est présentée dans la figure supplémentaire S8 et la figure supplémentaire S9. Veuillez cliquer ici pour voir une version agrandie de cette figure.
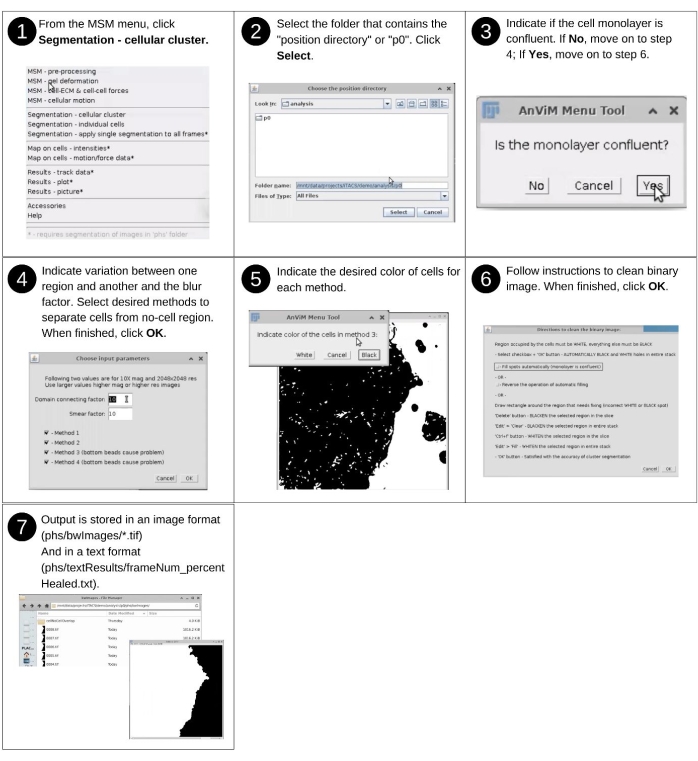
Figure 9 : Mappage des valeurs des points de la grille sur des cellules individuelles - partie 1. Étapes pour segmenter les régions d’image contenant des cellules à l’aide d’une nouvelle approche à plusieurs volets. Cette approche peut être utilisée pour segmenter les images de contraste de phase, de champ lumineux ou de fluorescence des cellules. Veuillez cliquer ici pour voir une version agrandie de cette figure.
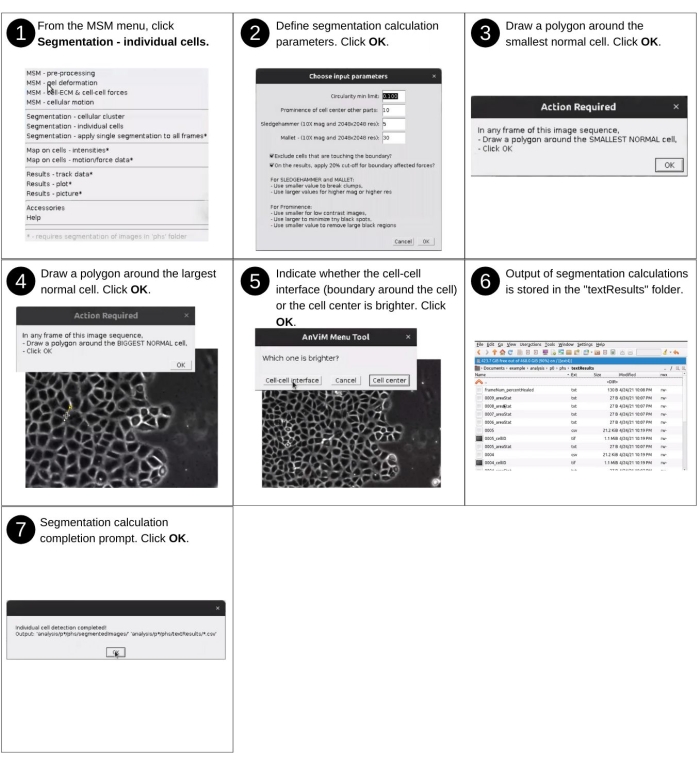
Figure 10 : Mappage des valeurs des points de la grille sur des cellules individuelles - partie 2. Étapes pour segmenter les cellules individuelles d’une monocouche à l’aide d’une nouvelle approche à plusieurs volets développée dans AnViM. Cette approche peut être utilisée pour segmenter les images de contraste de phase, de champ lumineux ou de fluorescence des cellules. Une vue détaillée de l’étape 2 est présentée à la figure supplémentaire S10. Veuillez cliquer ici pour voir une version agrandie de cette figure.
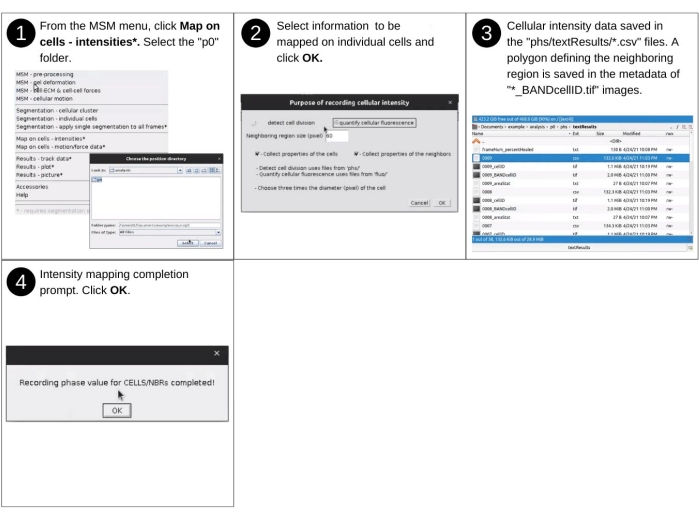
Figure 11 : Mappage des valeurs des points de la grille sur des cellules individuelles - partie 3. Étapes pour évaluer les intensités de pixels dans la région dans les cellules individuelles et dans la région voisine des cellules individuelles à l’aide d’AnViM. Les intensités évaluées comprennent l’intensité lumineuse transmise et l’intensité de fluorescence. Cette partie cartographie la valeur médiane et l’écart-type des intensités de pixels dans les cellules individuelles et dans une région voisine des cellules individuelles. Une vue détaillée de l’étape 2 est présentée à la figure supplémentaire S11. Veuillez cliquer ici pour voir une version agrandie de cette figure.
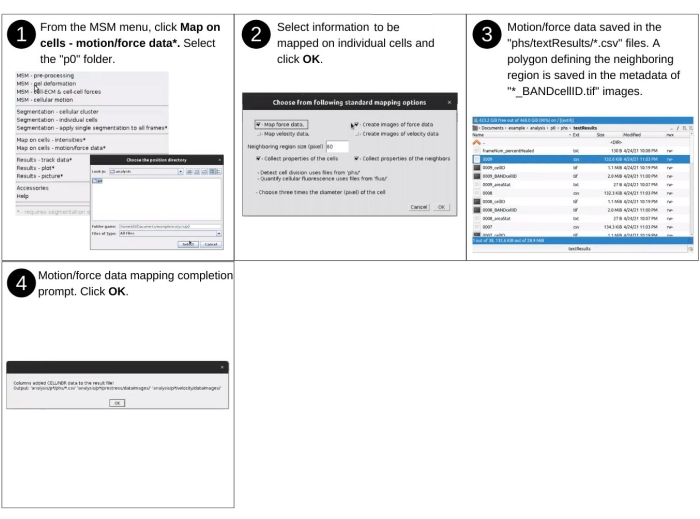
Figure 12 : Mappage des valeurs des points de la grille sur des cellules individuelles - partie 4. Étapes pour évaluer les forces et les propriétés de mouvement des points de grille dans les cellules individuelles et dans la région voisine des cellules individuelles à l’aide d’AnViM. Cette partie cartographie la valeur médiane et l’écart-type des propriétés dans les cellules individuelles et dans une région voisine des cellules individuelles. Une vue détaillée des étapes 2 et 3 est présentée à la figure supplémentaire S12 et à la figure supplémentaire S13. Veuillez cliquer ici pour voir une version agrandie de cette figure.
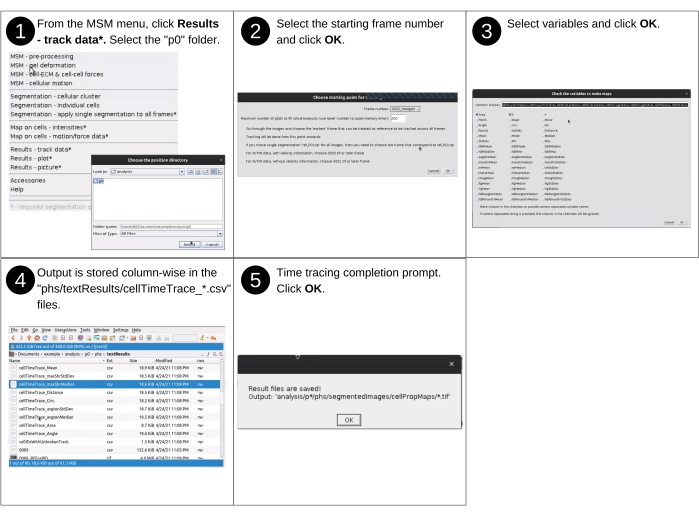
Figure 13 : Visualisation des résultats - partie 1. Étapes pour suivre les propriétés des cellules individuelles pendant toute la durée de l’expérience à l’aide d’AnViM. Une vue détaillée des étapes 2 et 3 est présentée à la figure supplémentaire S14 et à la figure supplémentaire S15. Veuillez cliquer ici pour voir une version agrandie de cette figure.
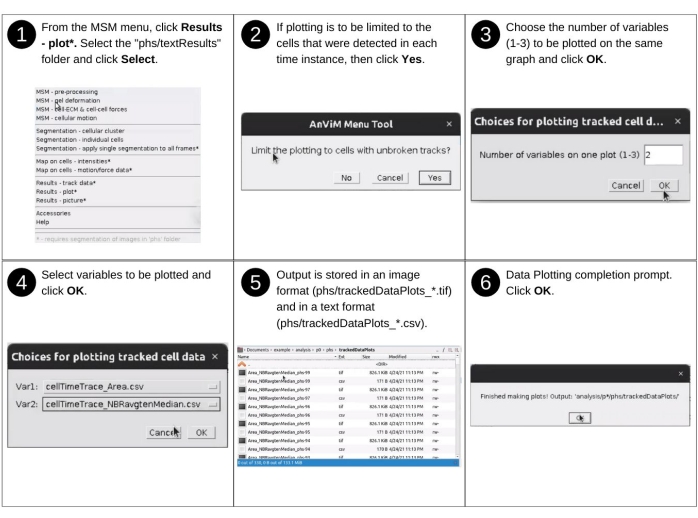
Figure 14 : Visualisation des résultats - partie 2. Étapes pour générer des traces temporelles des propriétés évaluées à l’aide d’AnViM. L’utilisateur a la possibilité de tracer jusqu’à trois propriétés dans un graphique. Des traces temporelles sont générées pour toutes les cellules ou uniquement pour les cellules pour lesquelles le suivi a été couronné de succès tout au long de l’expérience. Une vue détaillée de l’étape 5 est présentée dans la figure supplémentaire S16 et la figure supplémentaire S17. Veuillez cliquer ici pour voir une version agrandie de cette figure.
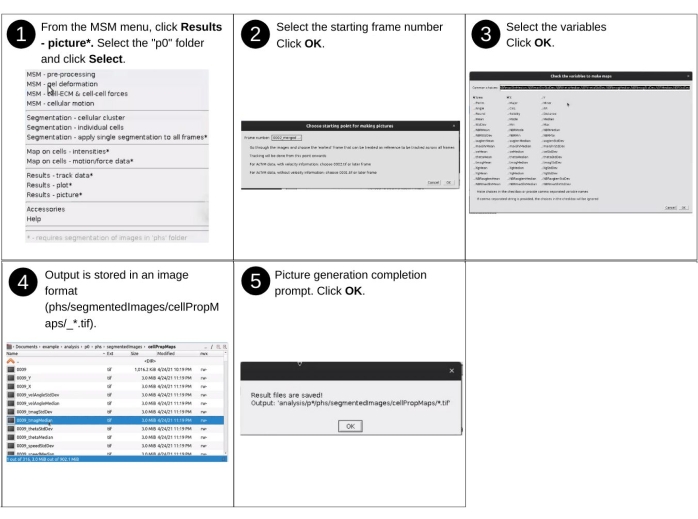
Figure 15 : Visualisation des résultats - partie 3. Étapes pour générer des cartes thermiques des propriétés évaluées à l’aide d’AnViM. Des cartes thermiques sont générées pour toutes les images suivant l’image de départ et toutes les propriétés sélectionnées. Une vue détaillée de l’étape 3 est présentée à la figure supplémentaire S18. Veuillez cliquer ici pour voir une version agrandie de cette figure.
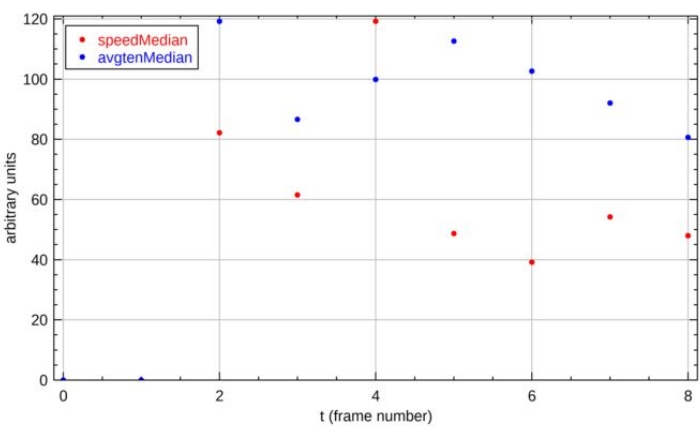
Figure 16 : Traces temporelles pour l’ID de cellule 1. Deux propriétés affichées sont la tension cytosquelettique cellulaire (« avgtenMedian ») et la vitesse cellulaire (« speedMedian »). La tension cytosquelettique cellulaire et la vitesse cellulaire sont quantifiées comme la valeur médiane à travers les points de grille dans les cellules. Les deux propriétés sont tracées sur le même axe avec des unités arbitraires pour visualiser les relations entre les propriétés évaluées. Les noms de variables supplémentaires sont répertoriés dans le tableau supplémentaire S1. Veuillez cliquer ici pour voir une version agrandie de cette figure.
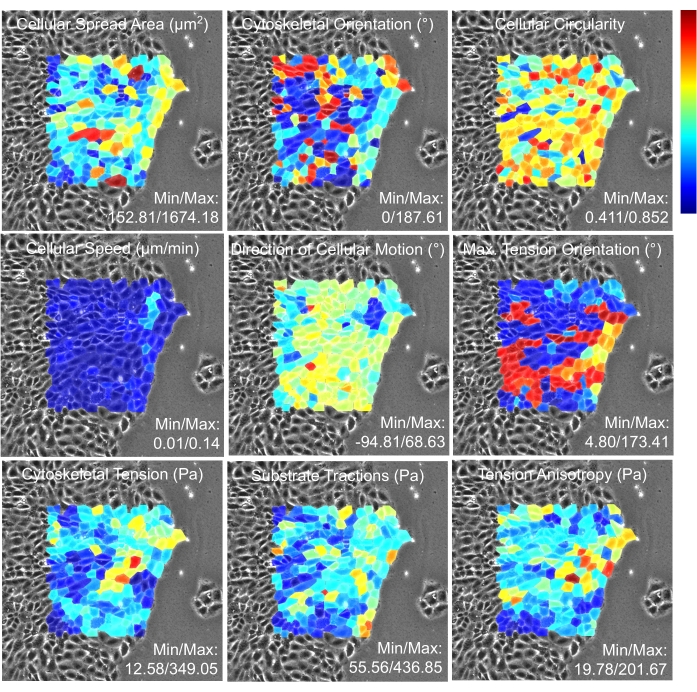
Figure 17 : Cartes thermiques des propriétés des cellules individuelles à travers la monocouche analysée. Chaque cellule est colorée avec la valeur médiane de la propriété indiquée dans le panneau. Ainsi, le rouge profond indique la valeur cellulaire maximale dans le spectre de couleurs, et le bleu profond indique la valeur cellulaire minimale sur la monocouche analysée. Comme décrit dans Tambe et al., PLoS One (2013)2, les cellules situées plus près de la frontière ont des forces mécaniques affectées par des propriétés inconnues des cellules en dehors de l’image. Par conséquent, la carte thermique est générée pour les cellules éloignées de la frontière. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Figure S1 : Exemples d’images de la perle fluorescente supérieure et inférieure. Veuillez cliquer ici pour télécharger ce fichier.
Figure S2 : Vue détaillée de l’étape 2 de la figure 3. Veuillez cliquer ici pour télécharger ce fichier.
Figure S3 : Vue détaillée de l’étape 4 de la figure 3. Veuillez cliquer ici pour télécharger ce fichier.
Figure S4 A : vue détaillée de l’étape 6 de la figure 3. Veuillez cliquer ici pour télécharger ce fichier.
Figure S5 : Vue détaillée de l’étape 3 de la figure 5. Veuillez cliquer ici pour télécharger ce fichier.
Figure S6 : Vue détaillée de l’étape 5 de la figure 5. Veuillez cliquer ici pour télécharger ce fichier.
Figure S7 : Vue détaillée de l’étape 3 de la figure 6. Veuillez cliquer ici pour télécharger ce fichier.
Figure S8 : Vue détaillée de la sortie de force cellule-ECM de l’étape 6 de la Figure 8. Veuillez cliquer ici pour télécharger ce fichier.
Figure S9 : Vue détaillée de la sortie de force cellule-cellule de l’étape 6 de la Figure 8. Veuillez cliquer ici pour télécharger ce fichier.
Figure S10 : Vue détaillée de l’étape 2 de la figure 10. Veuillez cliquer ici pour télécharger ce fichier.
Figure S11 : Vue détaillée de l’étape 2 de la figure 11. Veuillez cliquer ici pour télécharger ce fichier.
Figure S12 : Vue détaillée de l’étape 2 de la figure 12. Veuillez cliquer ici pour télécharger ce fichier.
Figure S13 : Vue détaillée de la sortie de l’étape 3 de la figure 12. Veuillez cliquer ici pour télécharger ce fichier.
Figure S14 : Vue détaillée de l’étape 2 de la figure 13. Veuillez cliquer ici pour télécharger ce fichier.
Figure S15 : Vue détaillée de l’étape 3 de la figure 13. Veuillez cliquer ici pour télécharger ce fichier.
Figure S16 : Vue détaillée des fichiers de données générés à l’étape 5 de la figure 14. Veuillez cliquer ici pour télécharger ce fichier.
Figure S17 : Vue détaillée d’un tracé généré à l’étape 5 de la figure 14. Veuillez cliquer ici pour télécharger ce fichier.
Figure S18 : Vue détaillée d’une carte thermique et du fichier contenant la gamme du spectre de couleurs généré à l’étape 4 de la figure 15. Veuillez cliquer ici pour télécharger ce fichier.
Tableau S1 : Liste des propriétés sélectionnées quantifiées par iTACS. Veuillez cliquer ici pour télécharger ce tableau.
Discussion
Les cellules adhérentes utilisent à la fois des signaux mécaniques et chimiques pour survivre, se développer et fonctionner. Une grande variété de logiciels de microscopie optimise l’expérience utilisateur dans l’évaluation des signaux chimiques grâce à l’imagerie basée sur la fluorescence. Cependant, l’évaluation des signaux mécaniques implique des capacités qui ne sont pas disponibles dans le logiciel de microscopie standard. De plus, l’évaluation des signaux mécaniques est plus efficace lorsque l’acquisition de données est intégrée à l’analyse de données. L’absence d’une plate-forme unifiée qui réponde aux besoins uniques de l’évaluation des signaux mécaniques a constitué une lacune technologique majeure dans la biologie cellulaire expérimentale. La boîte à outils intégrative pour analyser les signaux cellulaires (iTACS) est conçue pour combler cette lacune. Les deux composants d’iTACS, AnViM et AcTrM, dotent les utilisateurs des capacités nécessaires pour quantifier les propriétés cellulaires de quatre grandes catégories : forces, mouvement, morphologie et fluorescence/luminosité. Dans ces catégories, iTACS est actuellement capable de révéler plus de 50 aspects uniques des cellules adhérentes individuelles. Ces aspects comprennent des propriétés spécifiques de chaque grande catégorie, y compris leur valeur représentative et leur variabilité à travers la cellule (tableau supplémentaire S1). Par exemple, dans les forces, il y a des forces de traction à travers le cytosquelette, l’anisotropie de cette tension, l’orientation de la tension maximale et la contrainte de cisaillement à travers l’interface cellule-ECM qui a une influence profonde sur le comportement des cellules adhérentes1,3,6.
Une nouvelle approche pour examiner le comportement mécanique des cellules individuelles d’une monocouche
Les cellules individuelles d’une monocouche sont engagées dans un échange de signaux de nature chimique et mécanique3. Ces deux types de signaux sont transmis à travers la monocouche cellulaire d’une manière différente23. Cependant, la connaissance de la transmission mécanique du signal est à la traîne par rapport à celle de la transmission du signal chimique. Ce manque de connaissances coïncide avec un manque persistant d’approches simples et intuitives pour évaluer les signaux mécaniques cellulaires. La nouvelle approche de cartographie des données décrite ici est équipée pour combler cette lacune. Une telle cartographie révèle que la fluctuation de la tension cytosquelettique intrinsèque dans la région voisine d’une cellule sert de signaux de relaxation, de fluidisation et d’ancrage qui régulent les changements de forme, de taille et de vitesse cellulaires18. Les cartes des propriétés des régions voisines présentent des modèles de « subdivision multicellulaire » où les cellules de la subdivision sont exposées à un microenvironnement relativement uniforme et les cellules à la limite de la subdivision sont exposées à un microenvironnement remarquablement non uniforme18.
Accessibilité de la technologie de mesure de la force
Il existe divers protocoles pour fabriquer des hydrogels d’AAP, analyser la déformation de l’hydrogel et le mouvement cellulaire, et quantifier l’ECM cellulaire et les forces cellule-cellule1,2,7,8,9,13,14,15,18,20,21,24,25,26,27 ,28,29,30,31,32. Cependant, ces développements restent hors de portée des laboratoires communs de biologie cellulaire et confinés aux laboratoires ayant une expertise en ingénierie. En automatisant les aspects techniques de ces approches et en les intégrant sous une plateforme unifiée et conviviale, l’objectif d’iTACS est de faire de l’évaluation des signaux mécaniques une activité de routine dans la recherche et l’enseignement de la biologie cellulaire expérimentale.
ImageJ permet aux utilisateurs de développer des applications en utilisant des approches qui nécessiteraient peu ou pas de formation11. iTACS est en grande partie construit en utilisant des approches de script simples pour faciliter le développement continu piloté par la communauté. Une grande partie d’AcTrM est programmée à l’aide de scripts BeanShell, et la majeure partie d’AnViM est programmée à l’aide de macros ImageJ. Ces scripts et conseils pour implémenter ces fonctionnalités sur le microscope de l’utilisateur sont disponibles via GitHub (https://github.com/IntegrativeMechanobiologyLaboratory/iTACS).
Standardisation de la qualité des images acquises
Bien que les techniques basées sur un substrat élastique pour quantifier les forces physiques dans les cellules adhérentes aient été développées et mises en œuvre dans divers laboratoires, le protocole manque encore de standardisation. L’un des domaines qui nécessite le plus de normalisation est la qualité des images de perles supérieures acquises (figure supplémentaire S1). Des problèmes importants découlent de la dérive de la mise au point tout au long de l’expérience. Notre nouvelle approche de recentrage basée sur l’image de référence fait d’une telle focalisation un processus objectif. Les paramètres définis dans la toute première étape de l’AcTrM imposent les limites de qualité objectives nécessaires. D’autres mesures de normalisation peuvent être programmées dans les futures versions d’AcTrM.
La large applicabilité d’iTACS
En plus de quantifier de nombreux aspects des cellules adhérentes, la structure iTACS facilite son utilisation pour divers protocoles et besoins expérimentaux. AcTrM permet l’auto-formation de l’utilisateur guidée par logiciel. L’imagerie à grande vitesse requise, par exemple, par l’évaluation simultanée des fluctuations cytoplasmiques du calcium est actuellement limitée par la vitesse de repositionnement et de recentrage du matériel et il est préférable de l’effectuer à un endroit à la fois. Cependant, la mise en œuvre actuelle est bien équipée pour l’imagerie à long terme, l’imagerie interrompue, où l’échantillon ne peut pas être conservé sur la scène du microscope pendant toute la durée de l’expérience. Étant donné que les images de référence sont acquises au début de l’expérience, iTACS permet l’imagerie en temps réel des signaux mécaniques, ouvrant de nouvelles voies dans les applications de dépistage des drogues. AnViM permet aux utilisateurs de fournir des informations hautement techniques en termes simples. La capacité de quantifier un large spectre de propriétés cellulaires et de les suivre tout au long de l’expérience constitue des capacités essentielles nécessaires pour découvrir de nouveaux mécanismes de communication intercellulaire.
Pour le développement futur d’iTACS, nous avons identifié quatre domaines d’intervention : (1) l’amélioration de la vitesse d’acquisition et d’analyse des données, (2) la mise en œuvre d’approches pour évaluer les nouveaux signaux cellulaires13, (3) le développement d’ateliers et de modules éducatifs sur l’évaluation des signaux cellulaires basée sur iTACS, (4) le développement de solutions d’automatisation à faible coût.
Déclarations de divulgation
Les auteurs ne déclarent aucun conflit d’intérêts.
Remerciements
D.T.T. remercie le personnel affilié au Center for Lung Biology de l’Université de South Alabama pour avoir stimulé les discussions sur les besoins de recherche en biologie cellulaire expérimentale. Ces discussions ont été cruciales pour lancer le développement d’iTACS.
Ce travail a été soutenu en partie par des subventions du National Institute of Health / National Heart Lung Blood Institute, P01 HL66299 et R37 HL60024 (Stevens), R01-HL118334 (Alvarez), F32-HL144040-01 (Xu), et de l’Université de South Alabama par l’intermédiaire du Abraham Mitchell Cancer Research Fund (Singh, Palanki, Tambe), research and scholarly development Grant (Tambe), Honors College et Summer Undergraduate Research Fellow (Nguyen).
matériels
| Name | Company | Catalog Number | Comments |
| Reagents and components used in prepare glass surface for hydrogel coating | |||
| (3-Aminopropyl)trimethoxysilane, 97% | Aldrich chemistry | 13822565 | |
| 2% Bis Solution | Bio-rad | 1610142 | |
| 3-(Trimethoxysilyl)propyl methacrylate,98% | Acros organics | 2530850 | |
| 40% Acrylamide Solution | Bio-rad | 1610140 | |
| Glass bottom 35 mm dish/ 6 or 12 or 24 well plates | MatTek or CellVis | ||
| Glutaraldehyde, EM Grade, 25% | Polysciences | 1909100 | |
| Sodium Hydroxide | Sigma-aldrich | 1002074706 | |
| Reagents and components used in preparing suitable hydrogel | |||
| 2% Bis Solution | Bio-rad | 1610142 | |
| 40% Acrylamide Solution | Bio-rad | 1610140 | |
| Ammonium Persulfate | Bio-rad | 1610700 | |
| Cover Slips | Electron Microscopy Sciences | 7222301 | |
| Dulbecco's Phosphate Buffered Saline (1M) | Gibco | 14190136 | |
| FluoSpheres carboxylate 0.2 um, yellow-green(505/515) | Invitrogen | F8811 | |
| FluoSpheres carboxylate 0.5 um, red(580/605) | Invitrogen | F8812 | |
| FluoSpheres carboxylate 2.0 um, red(580/605) | Invitrogen | F8826 | |
| Rain-X | |||
| TEMED | Bio-rad | 1610801 | |
| Reagents used in coating extracellular matrix on the hydrogel | |||
| Collagen Type I Rat Tail | Corning | 354236 | |
| HEPES(1M) | Gibco | 15630080 | |
| Phosphate Buffered Saline (1M) | Gibco | 10010023 | |
| Sulfo-SANPAH | CovaChem | 102568434 | |
| Microscope hardware used in the current study | |||
| Camera | Hamamatsu Flash 4.0 LT sCMOS Camera | C11440-42U | |
| H117 ProScanTM Stages | Prior Scientific | ||
| Light source- Lambda DG4 and Lambda DG5 | Sutter instrument company | ||
| Microscope | Nikon eclipse TE2000-S | 550372 | |
| ProScan III Universal Microscope Automation Controller | Prior Scientific | ||
| Stagetop incubator | ibidi | 11922 | |
| Stepper Motor Focus Drive | Prior Scientific |
Références
- Tambe, D. T., et al. Collective cell guidance by cooperative intercellular forces. Nature Materials. 10 (6), 469-475 (2011).
- Tambe, D. T., et al. Monolayer stress microscopy: limitations, artifacts, and accuracy of recovered intercellular stresses. PloS One. 8 (2), 55172 (2013).
- Das, T., et al. A molecular mechanotransduction pathway regulates collective migration of epithelial cells. Nature Cell Biology. 17 (3), 276-287 (2015).
- Vedula, S. R., et al. Mechanics of epithelial closure over non-adherent environments. Nature Communications. 6, 6111 (2015).
- Lamason, R. L., et al. Rickettsia Sca4 reduces vinculin-mediated intercellular tension to promote spread. Cell. 167 (3), 670-683 (2016).
- Sunyer, R., et al. Collective cell durotaxis emerges from long-range intercellular force transmission. Science. 353 (6304), 1157-1161 (2016).
- Dong, L., Oberai, A. A. Recovery of cellular traction in three-dimensional nonlinear hyperelastic matrices. Computer Methods in Applied Mechanics and Engineering. 314, 296-313 (2017).
- Nier, V., et al. Kalman inversion stress microscopy. Biophysical Journal. 115 (9), 1808-1816 (2018).
- Serrano, R., et al. Three-dimensional Monolayer Stress Microscopy. Biophysical Journal. 117 (1), 111-128 (2019).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ: 25 years of Image Analysis. Nature Methods. 9 (7), 671-675 (2012).
- Schindelin, J., et al. Fiji - an Open Source platform for biological image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Edelstein, A., Amodaj, N., Hoover, K., Vale, R., Stuurman, N. Computer control of microscopes using microManager. Current Protocols in Molecular Biology. , 20 (2010).
- Patel, N. G., et al. Unleashing shear: Role of intercellular traction and cellular moments in collective cell migration. Biochemical and Biophysical Research Communications. 522 (2), 279-285 (2020).
- Yeung, T., et al. Effects of substrate stiffness on cell morphology, cytoskeletal structure, and adhesion. Cell Motility and the Cytoskeleton. 60 (1), 24-34 (2005).
- Trepat, X., et al. Physical forces during collective cell migration. Nature Physics. 5, 426-430 (2009).
- Aratyn-Schaus, Y., Oakes, P. W., Stricker, J., Winter, S. P., Gardel, M. L. Preparation of complaint matrices for quantifying cellular contraction. Journal of Visualized Experiments: JoVE. (46), (2010).
- King, J., et al. Structural and functional characteristics of lung macro- and microvascular endothelial cell phenotypes. Microvascular Research. 67 (2), 139-151 (2004).
- Patel, G., et al. Mechanical signaling in a pulmonary microvascular endothelial cell monolayer. Biochemical and Biophysical Research Communications. 519 (2), 337-343 (2019).
- Kähler, C. J., et al. Main results of the 4th International PIV Challenge. Experiments in Fluids. 57 (6), 97 (2016).
- Tseng, Q., et al. Spatial organization of the extracellular matrix regulates cell-cell junction positioning. Proceedings of the National Academy of Sciences of the United States of America. 109 (5), 1506-1511 (2012).
- Alvarez-Gonzalez, B., et al. Two-layer elastographic 3-D traction force microscopy. Scientific Reports. 7, 39315 (2017).
- Makarchuk, S., Beyer, N., Gaiddon, C., Grange, W., Hebraud, P. Holographic traction force microscopy. Scientific Reports. 8 (1), 3038 (2018).
- Janmey, P. A., Miller, R. T. Mechanisms of mechanical signaling in development and disease. Journal of Cell Science. 124 (1), 9-18 (2011).
- Dembo, M., Oliver, T., Ishihara, A., Jacobson, K. Imaging the traction stresses exerted by locomoting cells with the elastic substratum method. Biophysical Journal. 70 (4), 2008-2022 (1996).
- Dembo, M., Wang, Y. L. Stresses at the cell-to-substrate interface during locomotion of fibroblasts. Biophysical Journal. 76 (4), 2307-2316 (1999).
- Butler, J. P., Tolic-Norrelykke, I. M., Fabry, B., Fredberg, J. J. Traction fields, moments, and strain energy that cells exert on their surroundings. American Journal of Physiology: Cell Physiology. 282 (3), 595-605 (2002).
- Saez, A., et al. Traction forces exerted by epithelial cell sheets. Journal of Physics. Condensed Matter. 22 (19), 194119 (2010).
- Deforet, M., et al. Automated velocity mapping of migrating cell populations (AVeMap). Nature Methods. 9 (11), 1081-1083 (2012).
- Polio, S. R., Smith, M. L. Patterned hydrogels for simplified measurement of cell traction forces. Methods in Cell Biology. 121, 17-31 (2014).
- Toyjanova, J., et al. 3D Viscoelastic traction force microscopy. Soft Matter. 10 (40), 8095-8106 (2014).
- Serra-Picamal, X., Conte, V., Sunyer, R., Munoz, J. J., Trepat, X. Mapping forces and kinematics during collective cell migration. Methods in Cell Biology. 125, 309-330 (2015).
- Charrier, E. E., et al. A novel method to make viscoelastic polyacrylamide gels for cell culture and traction force microscopy. APL Bioengineering. 4 (3), 036104 (2020).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon