Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Uso del analizador de flujo metabólico celular en tiempo real para monitorear la bioenergética de osteoblastos
En este artículo
Resumen
El ensayo de flujo metabólico celular en tiempo real mide la tasa de consumo de oxígeno y la tasa de acidificación extracelular, que corresponde a la producción mitocondrial y glicolítica de trifosfato de adenosina, utilizando sensores de pH y oxígeno. El manuscrito explica un método para comprender el estado energético de los osteoblastos y la caracterización e interpretación del estado bioenergético celular.
Resumen
La formación ósea por osteoblastos es un proceso esencial para la adquisición ósea adecuada y el recambio óseo para mantener la homeostasis esquelética y, en última instancia, prevenir la fractura. En el interés tanto de optimizar el pico de masa ósea como de combatir diversas enfermedades musculoesqueléticas (es decir, osteoporosis posmenopáusica, anorexia nerviosa, diabetes mellitus tipo 1 y 2), se han realizado esfuerzos increíbles en el campo de la biología ósea para caracterizar completamente los osteoblastos a lo largo de su proceso de diferenciación. Dado el papel principal de los osteoblastos maduros para secretar proteínas de la matriz y vesículas de mineralización, se ha observado que estos procesos requieren una cantidad increíble de energía celular, o trifosfato de adenosina (ATP). El estado general de la energía celular a menudo se conoce como bioenergética celular, e incluye una serie de reacciones metabólicas que detectan la disponibilidad de sustrato para derivar ATP para satisfacer las necesidades celulares. Por lo tanto, el método actual detalla el proceso de aislamiento de las células estromales primarias de la médula ósea murina (BMSC) y el monitoreo de su estado bioenergético utilizando el analizador de flujo metabólico celular en tiempo real en varias etapas de la diferenciación de osteoblastos. Es importante destacar que estos datos han demostrado que el perfil metabólico cambia drásticamente a lo largo de la diferenciación de osteoblastos. Por lo tanto, se requiere el uso de este tipo de célula fisiológicamente relevante para apreciar completamente cómo el estado bioenergético de una célula puede regular la función general.
Introducción
La formación de hueso por el osteoblasto se acompaña de destrucción coordinada o reabsorción de huesos por osteoclastos. El equilibrio entre la formación de hueso osteoblástico y la reabsorción de osteoclastos es un proceso acoplado que describe el recambio óseo o la remodelación, que es esencial para la homeostasis esquelética. La disfunción osteoblástica conduce a una formación ósea deteriorada y da lugar a diversas enfermedades, incluida la osteoporosis 1,2,3. La diferenciación ex vivo/in vitro de células madre del estroma de la médula ósea (BMSC) a precursores de osteoblastos y osteoblastos maduros da como resultado la formación y deposición de la matriz ósea mineralizada en el vaso de cultivo a lo largo del tiempo 4,5,6. Esta formación ósea por el osteoblasto requiere una cantidad significativa de energía celular. Específicamente, se ha demostrado que la síntesis y secreción de colágeno dependen en gran medida de las proporciones celulares de ATP: ADP, y presumiblemente, el tráfico y la secreción de vesículas mineralizadas requieren ATPadicional 7,8,9,10,11. Muchos investigadores han demostrado que el proceso de osteoblastogénesis y función osteoblástica requiere un suministro adecuado de energía para satisfacer la demanda metabólica de formación ósea 12,13,14,15,16. Por lo tanto, el objetivo de este método es caracterizar el estado bioenergético de las células del estroma murino primario a lo largo de la diferenciación de osteoblastos utilizando el analizador de flujo metabólico celular en tiempo real. Estas técnicas ayudan a desarrollar una mejor comprensión de la homeostasis esquelética, lo que en última instancia puede conducir al desarrollo de nuevas opciones terapéuticas capaces de mejorar los trastornos esqueléticos.
El analizador de flujo metabólico celular en tiempo real se puede utilizar para medir la tasa de consumo de oxígeno (OCR) y la tasa de acidificación extracelular (ECAR) de los osteoblastos vivos, que corresponde a la producción de ATP mitocondrial y glicolítico, respectivamente. Fundamental para esta metodología es el hecho de que un ion H+ por lactato se libera durante la glucólisis en la conversión de glucosa en lactato, lo que altera el pH de los medios reflejado en los valores de ECAR. Por el contrario, durante el ciclo del TCA (ácido tricarboxílico), la fosforilación oxidativa a través de las mitocondrias produce CO2 utilizando o consumiendo oxígeno, y por lo tanto el monitoreo de OCR refleja este proceso metabólico. El analizador mide tanto OCR como ECAR en el microambiente extracelular simultáneamente y en tiempo real, lo que permite un tremendo potencial al estudiar la bioenergética celular 6,17. Además, realizar estos ensayos es relativamente sencillo y fácilmente personalizable dependiendo del objetivo experimental. Se han empleado técnicas similares para comprender mejor la regulación metabólica de las células T del sistema inmunitario18,19, el inicio del cáncer y la progresión20, junto con otros múltiples tipos de células que contribuyen a los síndromes metabólicos21,22.
Las ventajas del analizador de flujo metabólico en tiempo real sobre las técnicas alternativas incluyen (1) la capacidad de medir la bioenergética celular de las células vivas en tiempo real, (2) la capacidad de realizar ensayos con un número relativamente pequeño de células (requiere tan solo 5.000 células), (3) los puertos de inyección para manipular paralelamente múltiples tratamientos en un sistema de 96 pocillos de alto rendimiento, (4) el uso de imágenes celulares automatizadas sin etiquetas radiactivas para la normalización18, 23,24. Los siguientes métodos tienen como objetivo proporcionar una descripción generalizada pero detallada del monitoreo de la bioenergética celular en BMSC murinas a lo largo de la diferenciación de osteoblastos utilizando el analizador. Incluirá ensayos realizados rutinariamente; sin embargo, al igual que con muchas técnicas y métodos, se recomienda encarecidamente que los laboratorios individuales determinen detalles específicos para sus experimentos.
Selección de ensayos y diferentes tipos de ensayos disponibles: Una amplia variedad de kits de ensayo y reactivos están disponibles para estudiar la bioenergética de las células, al tiempo que se garantiza la fiabilidad y consistencia de los resultados experimentales. Además, el software de escritorio también ofrece plantillas de ensayo que se pueden personalizar fácilmente. El ensayo se puede definir en función de las necesidades del usuario para medir diferentes parámetros metabólicos. Estos ensayos pueden ser modificados de varias maneras en función del objetivo experimental y/o la cuestión científica. Por ejemplo, con cuatro puertos de inyección, se pueden inyectar múltiples compuestos en los medios de ensayo para analizar la respuesta celular específica de cada vía metabólica.
Prueba de fenotipo de energía celular: Este ensayo mide el fenotipo metabólico y el potencial metabólico de las células vivas. Este ensayo también se recomienda como el primer paso para tener una idea generalizada del metabolismo específico de la vía. Se inyecta una mezcla de oligomicina A-un inhibidor de la ATP sintasa y cianuro de carbonilo 4-(trifluorometoxi) fenilhidrazona (FCCP), un agente de desacoplamiento mitocondrial para comprender el potencial de energía celular. La inyección de oligomicina A inhibe la síntesis de ATP, lo que resulta en un aumento en la tasa de glucólisis (ECAR) para permitir que las células satisfagan sus demandas de energía; por otro lado, la inyección de FCCP resulta en un mayor OCR debido a la despolarización de la membrana mitocondrial. Esencialmente, este ensayo representa la respiración metabólica basal, y después de las inyecciones duales, empujones o tensiones, la respuesta metabólica. Sobre la base de estos parámetros, el software traza OCR y ECAR de las células clasificando las células como estado aeróbico, quiescente, glucolítico o energético a lo largo del tiempo25,26.
Ensayo de tasa de producción en tiempo real de ATP: Esto mide la producción celular de ATP simultáneamente a partir de la glucólisis y la respiración mitocondrial. Este ensayo mide cuantitativamente los cambios metabólicos de las dos vías de energía y proporciona datos sobre las tasas de producción de ATP mitocondrial y glicolítico a lo largo del tiempo. El ensayo obtiene datos basales de OCR y ECAR seguidos del cálculo de la tasa de producción de ATP mitocondrial a través de la inyección de oligomicina A y la tasa de producción de ATP glicolítico a través de la inyección de rotenona + mezcla de antimicina A (inhibición total de la función mitocondrial), lo que resulta en acidificación mitocondrial17,27.
Prueba de esfuerzo de mitocondrias celulares (o prueba de esfuerzo de mito celular): Esto mide la función mitocondrial a través de la respiración ligada al ATP, cuantifica la bioenergética celular, identifica la disfunción mitocondrial y mide la respuesta de las células al estrés. Varios parámetros, incluida la capacidad respiratoria basal y sobrante, la respiración ligada a ATP, la respiración máxima y el consumo de oxígeno no mitocondrial, se pueden obtener en un solo ensayo. Este ensayo implica inyecciones secuenciales de oligomicina A, FCCP (agente desacoplamiento mitocondrial), una mezcla de inhibidores de rotenona /antimicina A para analizar eficientemente el efecto de estos en la función mitocondrial28.
Prueba de flexibilidad de flexión de combustible mito: Esto mide la tasa de respiración mitocondrial por la oxidación de los tres combustibles mitocondriales primarios por la presencia y ausencia de sus inhibidores. La inhibición secuencial de glucosa, glutamina y ácidos grasos ayuda a medir la dependencia, capacidad y flexibilidad de las células y la dependencia de las células en varias vías celulares para satisfacer la demanda de energía. Cuando las mitocondrias no pueden satisfacer las demandas de la vía bloqueada de interés oxidando otros combustibles, las células entran en un estado de dependencia. La capacidad de las células se calcula mediante la inhibición de las otras dos vías alternativas seguida de la inhibición de la vía de interés. La flexibilidad de las células ayuda a comprender la capacidad de las mitocondrias para compensar y satisfacer las necesidades de combustible de la vía inhibida. Se calcula restando la dependencia de las células de la capacidad de las células. Tres inhibidores diferentes se utilizan de forma independiente o como una mezcla de dos para calcular eficazmente los parámetros del ensayo. El ácido 2-ciano-3-(1-fenil-1H-indol-3-il)-2-propenoico (UK5099) inhibe la oxidación de la glucosa al bloquear el portador de piruvato en la glucólisis. El sulfuro de etilo bis-2-(5-fenilacetamidado-1,3,4-tiadiazol-2-il) (BPTES) inhibe la vía de oxidación de la glutamina, y el etomoxir inhibe la oxidación de los ácidos grasos de cadena larga29.
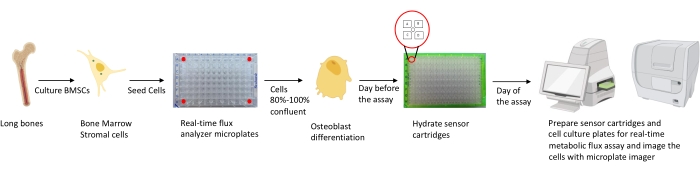
Figura 1: Representación esquemática de la metodología de cultivo y preparación de osteoblastos para su análisis. Las BMSC murinas se aíslan de huesos largos, se cultivan y se siembran en placas de 96 pocillos a una densidad de 25,000 células / pozo. El cultivo de estas células en medios específicos de osteoblastos se inicia cuando alcanzan el 80%-100% de confluencia para iniciar su diferenciación. Los ensayos se realizan en diferentes etapas de diferenciación. Las placas de los cartuchos se hidratan un día antes del ensayo. El día del ensayo, se inyectan diferentes inhibidores en los puertos de los cartuchos del sensor según los requisitos del ensayo, y se agrega un búfer de calibración a la placa de calibración de 96 pocillos. Después de la calibración, se realiza el ensayo de flujo metabólico celular en tiempo real, seguido de la obtención de imágenes de la microplaca de cultivo celular utilizando el generador de imágenes de microplacas para normalizar los datos del analizador de flujo metabólico celular en tiempo real con el recuento de células. Haga clic aquí para ver una versión más grande de esta figura.
Access restricted. Please log in or start a trial to view this content.
Protocolo
Todos los procedimientos se basaron en las pautas y la aprobación del Comité Institucional de Cuidado y Uso de Animales en el Centro Médico de la Universidad de Vanderbilt.
1. Preparación de reactivos y configuración del ensayo
- Aislamiento y cultivo de células estromales de la médula ósea (véase también el artículoanterior 30).
- Preparar medios celulares esenciales alfa mínimos (αMEM) completos complementando medios esenciales mínimos con modificación alfa con 10% de FBS (suero fetal bovino), 100 U/mL de penicilina y 100 μg/mL de estreptomicina.
- Prepare el tubo de recolección de médula ósea recortando el extremo de un tubo de microcentrífuga de 0,6 ml para que las células puedan pasar e insertándolo en un tubo de microcentrífuga de 1,5 ml que contenga 100 μL de αMEM completo.
- Sacrificar a los ratones usando el tratamiento con CO2 de la siguiente manera. Coloque al animal en la cámara de CO2 durante 2-3 minutos o hasta que cese la respiración. Espere al menos 1 minuto después de que el animal quede inconsciente para sacar a los ratones de la cámara y dislocarse cervicalmente.
- Usando fórceps estériles y un par de tijeras, corte la parte inferior del abdomen de los ratones sacrificados para hacer una pequeña incisión. Aislar los huesos largos (fémur, tibia y cresta ilíaca) de los ratones.
- Recorte los huesos largos para eliminar todo el tejido blando. Una vez que se limpie el hueso, corte ~ 1-2 mm de los extremos distal y proximal para crear una abertura para que la médula se enjuague.
NOTA: Esta abertura debe ser conservadora para evitar perder la médula y permitir que se enjuague. - Coloque los huesos en un tubo de recolección que contenga 100 μL de PBS estéril (solución salina tamponada con fosfato) para aislar la médula ósea total.
- Enjuague la médula por centrifugación a 10.000 x g durante 15-20 s a temperatura ambiente. Las células de la médula se grican en la parte inferior del tubo.
- Repita la centrifugación hasta que la cavidad ósea aparezca blanca y desprovista de la mayoría de los elementos de la médula. Resuspend la población mixta de médula ósea mediante la canalización suave hacia arriba y hacia abajo.
- Cultivar las células de un animal (tanto fémur como tibia) en un matraz de cultivo celular de 75 cm2 en 10 ml de medio de cultivo celular e incubar a 37 °C en una incubadora de cultivo celular con 5% de CO2. Si agrupa células de 2-3 animales, use un matraz de cultivo celular de 150 cm2 (recomendado).
- Después de 24-48 h de incubación de la población mixta, aspirar la población de células hematopoyéticas no adherentes contenidas dentro del medio de cultivo y lavar las células adherentes con 1x PBS.
- Siembra celular a partir de BMSC y diferenciación de osteoblastos
- Tripsinizar las células adherentes agregando suficiente tripsina-EDTA al 0,25% (aproximadamente 3-4 ml) para cubrir ligeramente la superficie del matraz, seguido de una incubación de 3 minutos a 37 °C.
- Agregue 6-7 ml de αMEM completo al matraz / tripsina para resuspendir los BMSC adherentes mediante el pipeteo cuidadoso hacia arriba y hacia abajo. Transfiera la suspensión BMSC a un tubo de centrífuga cónico.
- Retire una alícuota de 50 μL de la suspensión de BMSC y agréguele 50 μL de azul de tripano (dilución 1:1). Cuente el número total de células viables que excluyen el colorante pipeteando 10 μL de esta mezcla en un hemocitómetro y observándolo bajo el microscopio. No cuente ninguna célula muerta o no saludable que parezca de color azul (<10% de células).
- Sobre la base del recuento de células, calcule el volumen de suspensión celular en αMEM completa necesario para una concentración final de 2,4 x 106 células/ml para un volumen total de al menos 10 ml por placa.

Figura 2: La microplaca de cultivo celular, diseñada específicamente para el analizador. (A) Se destacan los cuatro pozos de corrección de fondo, A1, A12, H1, H12. Estos pocillos solo contienen medios de ensayo sin células. (B) El código de barras en el costado de la placa para escanear la placa usando el lector y analizador de imágenes. Haga clic aquí para ver una versión más grande de esta figura.
- Centrifugar las células en el tubo cónico a 1.000 x g durante 5 min y resuspendir las células a la concentración final deseada de 2,4 x 106 células/ml.
- Transfiera la suspensión celular a un depósito y, utilizando una pipeta multicanal, resuspenda cuidadosamente las células para garantizar una mezcla homogénea de células.
- Sembrar 2,5 x 104 células por pozo en la microplaca de cultivo celular de 96 pocillos con 80 μL de αMEM completa. No sembrar células en los pozos de corrección de fondo (A1, A12, H1, H12); en su lugar, simplemente agregue el medio en estos cuatro pozos.
NOTA: Los BMSC para los ensayos están chapados en microplacas de cultivo celular de 96 pocillos diseñadas para el analizador junto con los cartuchos del sensor. El área de superficie de estas placas es diferente de una placa regular de 96 pocillos. El área de superficie de cada pozo en la placa es de 0.106 cm2, que es aproximadamente el 40% del área típica de la placa de 96 pocillos. La densidad óptima de siembra celular se elige en función del tipo de célula. Por lo general, el analizador puede detectar entre 0,5-4 x 104 células por pozo. Los osteoblastos necesitan estar en contacto para diferenciarse de manera efectiva; para este propósito, se ha seleccionado el recubrimiento entre 2.0 x 10 4-3.0 x 104 BMSCs/pozo en 80 μL de αMEM completo. - Agitar suavemente la placa para asegurar la distribución uniforme de las células en los pocillos e incubar a 37 °C, 5% CO2. Compruebe el crecimiento de las células y la confluencia celular bajo el microscopio después de 48 h. Cambie el medio de cultivo celular si es necesario.
- Dependiendo del objetivo del ensayo, cuando las BMSC son 60%-80% confluentes (típicamente 48-72 h), iniciar la diferenciación de osteoblastos cambiando el medio de cultivo celular a medios de diferenciación de osteoblastos (αMEM completo suplementado con 5 mM de fosfato de β-glicerol y 50 μg/mL de ácido L-ascórbico).
- Si se van a analizar células estromales indiferenciadas (Día 0), mantenga las células bajo αMEM completa.
- Cambie los medios de diferenciación de osteoblastos cada dos días y visualice las células bajo el microscopio para asegurarse de que estén sanas hasta el día del ensayo. Preferiblemente, 24 h antes del ensayo programado, cambie los medios y mantenga un horario de cambio medio consistente (recomendado).
NOTA: Cambie cuidadosamente el medio inclinando ligeramente las placas en ángulo; esto evita el contacto accidental de las puntas de la pipeta con las placas de cultivo celular y la interrupción de la monocapa de células.
2. Preparación del cartucho del sensor para la calibración del flujo extracelular
- Hidrate los cartuchos del sensor del kit de ensayo extracelular antes del día del ensayo. Retire los cartuchos del sensor (placa verde) y coloque los sensores boca abajo.
- Usando una pipeta multicanal, agregue 200 μL de H2O a cada pocillo de la placa de utilidad. Coloque cuidadosamente los cartuchos del sensor de nuevo en la placa de utilidad e incube la placa durante la noche a temperatura ambiente.
NOTA: El fabricante recomienda incubar los cartuchos del sensor en una incubadora sin CO2 a 37 °C durante la noche. Sin embargo, puede producirse una evaporación significativa de los cartuchos del sensor. Si esto sucede, los cartuchos de sensor se pueden incubar a temperatura ambiente. Estas placas deben incubarse durante un mínimo de 4 h y un máximo de 72 h. - El día del ensayo, deseche el H2O de la placa de utilidad y agregue 200 μL de calibrante. Incubar la placa de utilidad durante al menos 1 h antes del ensayo.
3. Preparación de medios del analizador de flujo metabólico celular en tiempo real
- Use el medio DMEM libre de fenol rojo con un pH preajustado de 7.4 (recomendado) para ejecutar el ensayo con BMSC.
- Preparar 80 ml de medios de ensayo complementando los medios DMEM con 1 mM de piruvato de sodio, 2 mM de glutamina, 10 mM de glucosa, 200 nM de insulina, 50-200 μM de ácido oleico BSA.
- Incubar el medio de ensayo completo a 37 °C en un baño de agua.
4. Preparación de compuestos para los cartuchos del sensor
- Descongele la oligomicina A, la rotenona y la antimicina A en hielo. Pipetear hacia arriba y hacia abajo para solubilizar los compuestos antes de su uso.
- Añadir 3 ml de medio de ensayo preparado a cada tubo, seguido de la adición del tubo compuesto A respectivo: 26,4 μL de oligomicina A de 2,5 mM; tubo B: 3,1 μL de 12,67 mM de rotenona + 4,1 μL de 9,4 mM de antimicina A + 30 μL tinción de Hoechst.
- Cargue una concentración de 10x de estos inhibidores en el puerto correspondiente. La concentración final de soluciones de inyección necesarias es de 2 μM de oligomicina A, 1 μM de rotenona y 4,1 μM de antimicina A.
NOTA: Hoechst se agrega al puerto de inyección final para teñir fluorescentemente los núcleos con fines de imagen y normalización. Estas concentraciones se pueden optimizar en función del tipo de célula. - Cargue 20 μL de estos compuestos en las células en 180 μL de medios de ensayo.
5. Preparar la microplaca de cultivo celular para el ensayo
- Retire la microplaca de cultivo celular de la incubadora de 37 °C y observe las células bajo el microscopio.
- Retire el medio de ensayo del baño de agua.
- Lave suavemente las células con 200 μL de medio de ensayo dos veces y agregue 200 μL de medios de ensayo por pozo.
NOTA: Una vez que se agrega el medio de ensayo final a las células, el tiempo hasta que las placas entran en el analizador es crucial. Por lo tanto, no comience a reemplazar el medio hasta que se realicen los siguientes pasos dentro de 1 h. - Revise las células bajo el microscopio para asegurarse de que las células permanezcan adheridas a los pozos.
- Asegúrese de que las celdas en D5 y E8 se adhieran con una monocapa consistente y no se laven durante la etapa de lavado. El software de imágenes celulares utiliza estos dos pozos para configurar el enfoque automático y la exposición automática.
NOTA: El fabricante recomienda incubar la placa en una incubadora sin CO2 a 37 °C durante 1 h; este paso se puede omitir si se prefiere la obtención de imágenes automatizadas. Por ejemplo, el generador de imágenes de microplacas mantiene las mismas condiciones en una cámara cerrada, y las células se pueden obtener imágenes bajo un campo brillante.
6. Configuración del ensayo y la obtención de imágenes
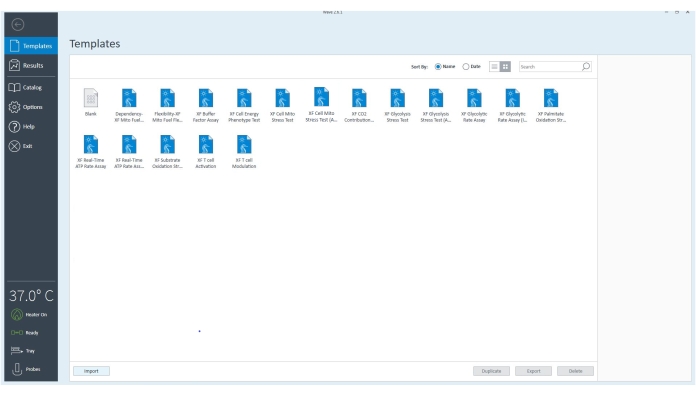
Figura 3: El software del controlador. El software verifica que el equipo está conectado y se ajusta a 37 °C. Los archivos de plantilla para diferentes ensayos que se pueden realizar con el analizador de flujo extracelular se pueden seleccionar para personalizar el ensayo aún más en función de los objetivos experimentales. Haga clic aquí para ver una versión más grande de esta figura.
- Abra el software de escritorio en la computadora junto al equipo.
- Compruebe el estado de la conexión en la esquina inferior izquierda del software del controlador.
- Vaya a Plantillas y seleccione el archivo de plantilla XF ATP Rate Assay o la plantilla de ensayo adecuada.
- Seleccione Definiciones de grupo en la parte superior de la pantalla y defina los grupos.
- Seleccione el Diseño del mapa de placas y asigne los pozos en función de los grupos definidos.
- Verifique el protocolo del instrumento, asegúrese de que los compuestos agregados se enumeran correctamente e incluya la información del proyecto para futuras referencias.
- Haga clic en Ejecutar ensayo; esto le pedirá la selección de la ubicación de almacenamiento de archivos de resultado.
- Seleccione la ubicación para guardar el archivo de resultados.
- Guarde el archivo con la fecha de ensayo y haga clic en Iniciar ejecución.
- Coloque el cartucho del sensor y la placa de utilidad en la bandeja y haga clic en Estoy listo para iniciar la calibración.
- Antes de comenzar la calibración, asegúrese de que se retira la tapa del cartucho y que el cartucho del sensor se coloca en la orientación correcta en la placa de utilidad. Este paso tomará de 10 a 20 minutos y, una vez completado, el software mostrará el cuadro de diálogo Placa de celda de carga .
7. Obtener imágenes de campo brillante
NOTA: Este paso es opcional. Si no hay ningún equipo de imágenes disponible, vaya al paso 8.

Figura 4: El software de imágenes celulares se comunica con el lector de imágenes a través de la computadora. Las células en la microplaca se pueden visualizar antes y después del ensayo, y el recuento de células / pozo se obtiene después del ensayo para normalizar los datos. Haga clic aquí para ver una versión más grande de esta figura.
- Abra el software de imágenes celulares en la computadora.
- Asegúrese de que el generador de imágenes de microplacas esté encendido y que los puertos estén conectados al equipo.
- Compruebe la barra de estado en la parte inferior izquierda de la pantalla para asegurarse de que la temperatura está establecida en 37 °C y que el estado de la conexión debe estar resaltado en verde como listo.
- Escanee el código de barras de la placa para iniciar el proceso de obtención de imágenes.
- Proporcione un nombre a la placa celular y presione Guardar (este es el nombre donde se guardarán tanto las imágenes de campo brillante como las fluorescentes). Haga clic en Realizar escaneo de campo brillante.
- La siguiente pantalla, placa y menú de escaneo, muestran las opciones para la obtención de imágenes. Antes del ensayo, seleccione Iniciar análisis de Campo Brillante.
- Coloque la microplaca de cultivo celular junto con la cubierta/tapa de la placa en el soporte de la bandeja y alinee bien A1 con la marca A1. Haga clic en Cerrar bandeja.
- Aparece la siguiente pantalla, adquisición de imágenes de campo brillante, con un mapa de placas. Haga clic en Escanear todos los pozos, que inicia el proceso de inicialización del sistema seguido de 30 a 35 minutos de un escaneo.
- Después de la exploración de campo brillante, retire la microplaca de cultivo celular y colóquela en el analizador para realizar el ensayo.
8. Ejecución del ensayo
- Una vez completada la calibración, el software muestra el cuadro de diálogo Placa de celda de carga .
- Haga clic en Abrir bandeja para reemplazar la bandeja de utilidad con una microplaca de cultivo celular. Asegúrese de que la tapa esté retirada y que el A1 de la placa encaje en la orientación correcta.
- Luego, haga clic en Placa de celda de carga para iniciar el ensayo. El cartucho del sensor permanecerá dentro del analizador para las inyecciones de ensayo.
- Espere hasta que comience el ensayo y muestre el tiempo estimado de finalización.
- Al finalizar el ensayo, el software muestra el cuadro de diálogo Descargar cartucho del sensor . Haga clic en Expulsar y retire la microplaca de cultivo celular del analizador.
- Retire con cuidado el cartucho del sensor y reemplace la tapa de la placa celular. Las células están listas para imágenes fluorescentes y conteo celular.
- Después de extraer la placa de celda y el cartucho del sensor, aparece el cuadro de diálogo Ensayo completo .
- Haga clic en Ver resultados para abrir el archivo de resultados del ensayo y normalizar los datos inmediatamente o haga clic en Inicio.
9. Obtener imágenes de fluorescencia y normalizar
NOTA: Este paso es un método opcional pero preferido para la normalización de BMSC y osteoblastos. Si no hay equipo de imágenes disponible, se debe realizar otro método de normalización, como el aislamiento y cuantificación de proteínas o ADN.

Figura 5: Imágenes representativas del software de imágenes utilizado para la normalización de los datos del ensayo. (A) Imagen de campo brillante cosida que muestra la confluencia celular en todo el pozo. (B) Imagen de fluorescencia cosida que muestra núcleos de osteoblastos teñidos de Hoechst utilizados para contar el número de células para normalizar los resultados del ensayo. Estos son osteoblastos después de 7 días de diferenciación. Haga clic aquí para ver una versión más grande de esta figura.
- Después de completar el ensayo, escanee el código de barras de la placa con el lector de códigos de barras de mano. Si la placa ya ha sido fotografiada, no requerirá un nuevo nombre.
- Seleccione Fluorescencia y recuento de celdas, coloque la placa de la celda en el soporte de la bandeja y haga clic en Cerrar bandeja.
- En la ventana de adquisición de imágenes, seleccione Escanear todos los pozos para comenzar a crear imágenes. Las imágenes de fluorescencia tardan entre 15 y 20 minutos en escanear toda la placa. Observe la marca de verificación verde que indica que se ha completado el escaneo.
- Revise las imágenes fluorescentes y los recuentos de células en la aplicación de imágenes e imágenes celulares haciendo clic al azar en un par de pozos.
NOTA: Hay una opción para ver las celdas contadas en la parte inferior derecha de la pantalla. Esta opción muestra una imagen enmascarada, resaltando los objetos que se contaron. - Una vez que se completen las imágenes de fluorescencia, exporte las imágenes para obtener referencias adicionales.
- Una vez que se completen las imágenes y el recuento de células, abra el archivo Resultados y haga clic en Normalizar. La pantalla de normalización dará el diseño de la placa y una opción para importar el recuento de celdas.
- Haga clic en Importar y seleccione Solicitar el software de escritorio para normalizar el ensayo con recuento de celdas automáticamente.
Access restricted. Please log in or start a trial to view this content.
Resultados
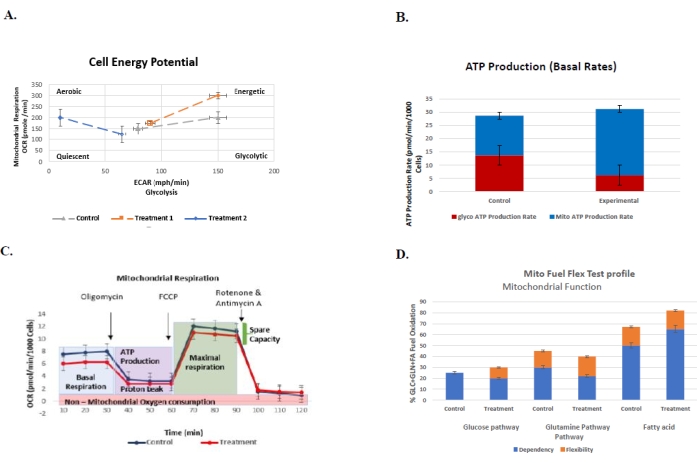
Figura 6: Gráficos representativos para ensayos realizados rutinariamente para comprender el perfil bioenergético celular del grupo de control vs. tratamiento con sus respectivos errores estándar. (A) La prueba de fenotipo de energía celular. La gráfica representa la glucólisis (ECAR) vs. respiración mitocondrial (OCR)...
Access restricted. Please log in or start a trial to view this content.
Discusión
El analizador de flujo metabólico celular en tiempo real se puede utilizar para explorar la energía celular en diferentes condiciones. El protocolo ilustra el aislamiento eficiente de las BMSC, el cultivo de células en placas de cultivo celular apropiadas y su diferenciación a osteoblastos maduros, que se pueden usar para diversos ensayos utilizando el analizador de flujo extracelular. Además, también se explican en detalle los pasos críticos del ensayo de flujo metabólico celular en tiempo real, incluida la hidr...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este trabajo fue apoyado por el Instituto Nacional de Salud (NIH) Instituto Nacional de Artritis y Enfermedades Musculoesqueléticas y de la Piel (NIAMS) Subvención AR072123 y el Instituto Nacional sobre el Envejecimiento (NIA) Subvención AG069795 (a ERR).
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| 0.25% Trypsin EDTA | Sigma-Aldrich | T4049 | |
| 2-cyano-3-(1-phenyl-1H-indol-3-yl)-2-propenoic acid | Sigma - Aldrich | PZ0160 | UK5099 |
| Antimycin A | Sigma - Aldrich | A8674 | |
| Ascorbic acid | Sigma-Aldrich | A4544-100G | |
| Bis-2-(5-phenylacetamido-1,3,4-thiadiazol-2-yl)ethyl sulfide | Sigma - Aldrich | SML0601 | BPTES |
| Carbonyl cyanide 4-(trifluoromethoxy)phenylhydrazone | Sigma - Aldrich | C2920 | FCCP |
| Cytation 5 imaging reader | BioTek | N/A | Microplate imager |
| Etomoxir sodium salt hydrate | Sigma - Aldrich | E1905 | |
| Hoechst 33342 Solution (20 mM) | Thermo Scientific | 62249 | |
| Insulin | Sigma - Aldrich | I6634 | |
| Oleic Acid-Albumin from bovine serum | Sigma - Aldrich | O3008 | |
| Oligomycin A - 5 mg | Sigma - Aldrich | 75351 | |
| Rotenone | Sigma - Aldrich | R8875-1G | |
| Seahorse XF 1.0 M Glucose Solution | Agilent Technologies | 103577-100 | |
| Seahorse XF 100mM Pyruvate Solution | Agilent Technologies | 103578-100 | |
| Seahorse XF 200mM Glutamine solution | Agilent Technologies | 103579-100 | |
| Seahorse XF DMEM media | Agilent Technologies | 103575-100 | DMEM assay media eith 5mM HEPES, pH 7.4, without phenol red, sodium bicarbonate, glucose, pyruvate, and L-glutamine |
| Seahorse XFe96 Analyzer | Agilent Technologies | S7800B | Real- Time Metabolic flux analyzer |
| Seahorse XFe96 FluxPak | Agilent Technologies | 102416-100 | Includes XFe96 Sensor cartridges, Cell culture microplates, and Seahorse XF Calibrant solution |
| The Cell imaging 1.1.0.11 software | Agilent Technologies - BioTek | ||
| Wave software 2.6.1 | Agilent Technologies | ||
| β-glycerol phosphate | Sigma-Aldrich | G9422-50G |
Referencias
- Rodan, G. A. Bone homeostasis. Proceedings of the National Academy of Sciences of the United States of America. 95 (23), 13361-13362 (1998).
- Nakahama, K. I. Cellular communications in bone homeostasis and repair. Cellular and Molecular Life Sciences. 67 (23), 4001-4009 (2010).
- Kim, J. M., Lin, C., Stavre, Z., Greenblatt, M. B., Shim, J. H. Osteoblast-osteoclast communication and bone homeostasis. Cells. 9 (9), 2073(2020).
- Gao, J., et al. SIRT3/SOD2 maintains osteoblast differentiation and bone formation by regulating mitochondrial stress. Cell Death and Differentiation. 25 (2), 229-240 (2018).
- Baron, R. Molecular mechanisms of bone resorption by the osteoclast. The Anatomical Record. 224 (2), 317-324 (1989).
- Tian, L., Rosen, C. J., Guntur, A. R. Mitochondrial Function and Metabolism of Cultured Skeletal Cells. Methods in Molecular Biology. 2230, Clifton, N.J. 437-447 (2021).
- Zanotelli, M. R., et al. Regulation of ATP utilization during metastatic cell migration by collagen architecture. Molecular Biology of the Cell. 29 (1), 1-9 (2018).
- Gonzales, S., Wang, C., Levene, H., Cheung, H. S., Huang, C. Y. C. ATP promotes extracellular matrix biosynthesis of intervertebral disc cells. Cell and Tissue Research. 359 (2), 635-642 (2015).
- Kruse, N. J., Bornstein, P. The metabolic requirements for transcellular movement and secretion of collagen. Journal of Biological Chemistry. 250 (13), 4841-4847 (1975).
- Rendina-Ruedy, E., Guntur, A. R., Rosen, C. J. Intracellular lipid droplets support osteoblast function. Adipocyte. 6 (3), 250-258 (2017).
- Sinnott-Armstrong, N., et al. A regulatory variant at 3q21.1 confers an increased pleiotropic risk for hyperglycemia and altered bone mineral density. Cell Metabolism. 33 (3), 615-628 (2021).
- Esen, E., Lee, S. Y., Wice, B. M., Long, F. PTH promotes bone anabolism by stimulating aerobic glycolysis via IGF signaling. Journal of Bone and Mineral Research. 30 (11), 1959-1968 (2015).
- Borle, A. B., Nichols, N., Nichols, G. Metabolic studies of bone in vitro: I. Normal bone. Journal of Biological Chemistry. 235, 1206-1210 (1960).
- Borle, A. B., Nichols, N., Nichols, G. Metabolic studies of bone in vitro: II. The metabolic patterns of accretion and resorption. Journal of Biological Chemistry. 235, 1211-1214 (1960).
- Adamek, G., Felix, R., Guenther, H. L., Fleisch, H. Fatty acid oxidation in bone tissue and bone cells in culture. Characterization and hormonal influences. The Biochemical Journal. 248 (1), 129-137 (1987).
- Frey, J. L., et al. Wnt-Lrp5 signaling regulates fatty acid metabolism in the osteoblast. Molecular and Cellular Biology. 35 (11), 1979-1991 (2015).
- Romero, N., Rogers, G., Neilson, A., Dranka, B. P. Quantifying cellular ATP production rate using agilent seahorse XF technology. , (2018).
- vander Windt, G., Chang, C., Pearce, E. Measuring bioenergetics in T cells using a Seahorse Extracellular Flux Analyzer. Current Protocols in Immunology. 113, 1(2016).
- Traba, J., Miozzo, P., Akkaya, B., Pierce, S. K., Akkaya, M. An optimized protocol to analyze glycolysis and mitochondrial respiration in lymphocytes. Journal of Visualized Experiments:JoVE. (117), e54918(2016).
- Noel, P., et al. Preparation and metabolic assay of 3-dimensional spheroid co-cultures of pancreatic cancer cells and fibroblasts. Journal of Visualized Experiments:JoVE. (126), e56081(2017).
- Nicholls, D., et al. Bioenergetic profile experiment using C2C12 myoblast cells. Journal of Visualized Experiments: JoVE. (46), e2511(2010).
- Sakamuri, S. S. V. P., et al. Measurement of respiratory function in isolated cardiac mitochondria using Seahorse XFe24 Analyzer: applications for aging research. GeroScience. 40 (3), 347-356 (2018).
- What are the advantages of using Seahorse XF technology. , Available from: https://www.agilent.com/en/support/cell-analysis/advantages-of-using-xf-tech (2018).
- Horan, M. P., Pichaud, N., Ballard, J. W. O. Review: Quantifying mitochondrial dysfunction in complex diseases of aging. Journals of Gerontology - Series A Biological Sciences and Medical Sciences. 67 (10), 1022-1035 (2012).
- XF cell energy phenotype test. , Available from: https://www.agilent.com/en/product/cell-analysis/real-time-cell-metabolic-analysis/xf-assay-kits-reagents-cell-assay-media/seahorse-xf-cell-energy-phenotype-test-kit-740884 (2021).
- Leung, D. T. H., Chu, S. Measurement of oxidative stress: Mitochondrial function using the seahorse system. Methods in Molecular Biology. 1710, Clifton, N.J. 285-293 (2018).
- XF ATP rate assay. , Available from: https://www.agilent.com/en/product/cell-analysis/real-time-cell-metabolic-analysis/xf-assay-kits-reagents-cell-assay-media/seahorse-xf-cell-energy-phenotype-test-kit-740889 (2021).
- XF cell mito stress test. , Available from: https://www.agilent.com/en/product/cell-analysis/real-time-cell-metabolic-analysis/xf-assay-kits-reagents-cell-assay-media/seahorse-xf-cell-energy-phenotype-test-kit-740885 (2021).
- XF cell mito fuel flex test. , Available from: https://www.agilent.com/en/product/cell-analysis/real-time-cell-metabolic-analysis/xf-assay-kits-reagents-cell-assay-media/seahorse-xf-cell-energy-phenotype-test-kit-740888 (2021).
- Maridas, D. E., Rendina-Ruedy, E., Le, P. T., Rosen, C. J. Isolation, culture, and differentiation of bone marrow stromal cells and osteoclast progenitors from mice. Journal of Visualized Experiments: JoVE. (131), e56750(2018).
- Wei, J., et al. Glucose uptake and Runx2 synergize to orchestrate osteoblast differentiation and bone formation. Cell. 161 (7), 1576-1591 (2015).
- Zoch, M. L., Abou, D. S., Clemens, T. L., Thorek, D. L. J., Riddle, R. C. In vivo radiometric analysis of glucose uptake and distribution in mouse bone. Bone Research. 4, 16004(2016).
- Divakaruni, A. S., Paradyse, A., Ferrick, D. A., Murphy, A. N., Jastroch, M. Analysis and interpretation of microplate-based oxygen consumption and pH data. Methods in Enzymology. 547, 309-354 (2014).
- Kam, Y., Jastromb, N., Clayton, J., Held, P., Dranka, B. Normalization of agilent seahorse XF data by in-situ cell counting using a BioTek cytation 5 application note. , (2017).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados