Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Utilisation d’un analyseur de flux métabolique cellulaire en temps réel pour surveiller la bioénergétique des ostéoblastes
Dans cet article
Résumé
Le test de flux métabolique cellulaire en temps réel mesure le taux de consommation d’oxygène et le taux d’acidification extracellulaire, qui correspond à la production mitochondriale et glycolytique d’adénosine triphosphate, à l’aide de capteurs de pH et d’oxygène. Le manuscrit explique une méthode pour comprendre l’état énergétique des ostéoblastes et la caractérisation et l’interprétation de l’état bioénergétique cellulaire.
Résumé
La formation osseuse par les ostéoblastes est un processus essentiel pour une acquisition osseuse et un renouvellement osseux appropriés afin de maintenir l’homéostasie squelettique et, en fin de compte, de prévenir la fracture. Dans le but d’optimiser la masse osseuse maximale et de lutter contre diverses maladies musculo-squelettiques (c.-à-d. l’ostéoporose post-ménopausique, l’anorexie mentale, le diabète sucré de type 1 et 2), des efforts incroyables ont été déployés dans le domaine de la biologie osseuse pour caractériser pleinement les ostéoblastes tout au long de leur processus de différenciation. Compte tenu du rôle principal des ostéoblastes matures dans la sécrétion de protéines matricielles et de vésicules de minéralisation, il a été noté que ces processus prennent une quantité incroyable d’énergie cellulaire, ou adénosine triphosphate (ATP). L’état énergétique cellulaire global est souvent appelé bioénergétique cellulaire, et il comprend une série de réactions métaboliques qui détectent la disponibilité du substrat pour dériver l’ATP pour répondre aux besoins cellulaires. Par conséquent, la méthode actuelle détaille le processus d’isolement des cellules stromales primaires de la moelle osseuse murine (BMSC) et de surveillance de leur état bioénergétique à l’aide de l’analyseur de flux métabolique cellulaire en temps réel à différents stades de la différenciation des ostéoblastes. Il est important de noter que ces données ont démontré que le profil métabolique change radicalement tout au long de la différenciation des ostéoblastes. Ainsi, l’utilisation de ce type de cellule physiologiquement pertinent est nécessaire pour apprécier pleinement comment l’état bioénergétique d’une cellule peut réguler la fonction globale.
Introduction
La formation d’os par l’ostéoblaste s’accompagne d’une destruction ou d’une résorption coordonnée des os par les ostéoclastes. L’équilibre entre la formation osseuse ostéoblastique et la résorption des ostéoclastes est un processus couplé décrivant le renouvellement osseux ou le remodelage, ce qui est essentiel pour l’homéostasie squelettique. Le dysfonctionnement ostéoblastique entraîne une altération de la formation osseuse et entraîne diverses maladies, y compris l’ostéoporose 1,2,3. La différenciation ex vivo/in vitro des cellules souches stromales de la moelle osseuse (BMSC) en précurseurs d’ostéoblastes et en ostéoblastes matures entraîne la formation et le dépôt de la matrice osseuse minéralisée dans le récipient de culture au fil du temps 4,5,6. Cette formation osseuse par l’ostéoblaste nécessite une quantité importante d’énergie cellulaire. Plus précisément, il a été démontré que la synthèse et la sécrétion de collagène dépendent fortement des rapports cellulaires ATP: ADP, et vraisemblablement, le trafic et la sécrétion de vésicules minéralisées nécessitent de l’ATP supplémentaire 7,8,9,10,11. De nombreux chercheurs ont démontré que le processus d’ostéoblastogenèse et de fonction ostéoblastique nécessite un apport adéquat d’énergie pour répondre à la demande métabolique de la formation osseuse 12,13,14,15,16. Par conséquent, l’objectif de cette méthode est de caractériser l’état bioénergétique des cellules stromales murines primaires tout au long de la différenciation ostéoblastique à l’aide de l’analyseur de flux métabolique cellulaire en temps réel. Ces techniques aident à développer une meilleure compréhension de l’homéostasie squelettique, ce qui pourrait finalement conduire au développement de nouvelles options thérapeutiques capables d’améliorer les troubles squelettiques.
L’analyseur de flux métabolique cellulaire en temps réel peut être utilisé pour mesurer le taux de consommation d’oxygène (OCR) et le taux d’acidification extracellulaire (ECAR) des ostéoblastes vivants, ce qui correspond à la production mitochondriale et glycolytique d’ATP, respectivement. Le fait qu’un ion H+ par lactate soit libéré lors de la glycolyse lors de la conversion du glucose en lactate, ce qui modifie le pH du milieu reflété dans les valeurs ECAR est fondamental pour cette méthodologie. Inversement, au cours du cycle TCA (acide tricarboxylique), la phosphorylation oxydative via les mitochondries produit du CO2 en utilisant ou en consommant de l’oxygène, et donc la surveillance de l’OCR reflète ce processus métabolique. L’analyseur mesure à la fois l’OCR et l’ECAR dans le microenvironnement extracellulaire simultanément et en temps réel, ce qui permet un potentiel énorme lors de l’étude de la bioénergétique cellulaire 6,17. De plus, l’exécution de ces tests est relativement simple et facilement personnalisable en fonction de l’objectif expérimental. Des techniques similaires ont été utilisées pour mieux comprendre la régulation métabolique des lymphocytes T du système immunitaire18,19, l’initiation et la progression du cancer20, ainsi que de multiples autres types de cellules contribuant aux syndromes métaboliques21,22.
Les avantages de l’analyseur de flux métabolique en temps réel par rapport aux techniques alternatives comprennent (1) la capacité de mesurer la bioénergétique cellulaire de cellules vivantes en temps réel, (2) la capacité d’effectuer des essais avec un nombre relativement faible de cellules (nécessite aussi peu que 5 000 cellules), (3) des ports d’injection pour manipuler en parallèle plusieurs traitements dans un système à haut débit de 96 puits, (4) l’utilisation d’un imageur de cellules automatisé sans étiquette radioactive pour la normalisation18, 23,24. Les méthodes suivantes visent à fournir une description généralisée mais détaillée de la surveillance de la bioénergétique cellulaire dans les BMSC murins tout au long de la différenciation des ostéoblastes à l’aide de l’analyseur. Il comprendra des tests effectués régulièrement; cependant, comme pour de nombreuses techniques et méthodes, il est fortement encouragé que les laboratoires individuels déterminent des détails spécifiques pour leurs expériences.
Sélection de tests et différents types de tests disponibles: Une grande variété de kits de dosage et de réactifs sont disponibles pour étudier la bioénergétique des cellules tout en assurant la fiabilité et la cohérence des résultats expérimentaux. En outre, le logiciel de bureau propose également des modèles de test qui peuvent être facilement personnalisés. Le test peut être défini en fonction des besoins de l’utilisateur pour mesurer différents paramètres métaboliques. Ces essais peuvent être modifiés de différentes manières en fonction de l’objectif expérimental et/ou de la question scientifique. Par exemple, avec quatre ports d’injection, plusieurs composés peuvent être injectés dans le milieu d’essai pour analyser la réponse cellulaire spécifique à chaque voie métabolique.
Test de phénotype d’énergie cellulaire: Ce test mesure le phénotype métabolique et le potentiel métabolique des cellules vivantes. Ce test est également recommandé comme première étape pour avoir une idée généralisée du métabolisme spécifique à la voie. Un mélange d’oligomycine A-un inhibiteur de l’ATP synthase et de cyanure de carbonyle 4-(trifluorométhoxy)phénylhydrazone (FCCP)-un agent de découplage mitochondrial est injecté pour comprendre le potentiel énergétique cellulaire. L’injection d’oligomycine A inhibe la synthèse de l’ATP, entraînant une augmentation du taux de glycolyse (ECAR) pour permettre aux cellules de répondre à leurs besoins énergétiques; d’autre part, l’injection de FCCP entraîne une OCR plus élevée en raison de la dépolarisation de la membrane mitochondriale. Essentiellement, ce test décrit la respiration métabolique basale et, après les doubles injections, poussées ou stress, la réponse métabolique. Sur la base de ces paramètres, le logiciel trace ensuite l’OCR et l’ECAR des cellules en classant les cellules en état aérobie, quiescent, glycolytique ou énergétique au fil du temps25,26.
Test du taux de production atp en temps réel : Cela mesure la production cellulaire d’ATP simultanément à partir de la glycolyse et de la respiration mitochondriale. Ce test mesure quantitativement les changements métaboliques des deux voies énergétiques et fournit des données sur les taux de production d’ATP mitochondrial et glycolytique au fil du temps. Le test obtient des données OCR et ECAR basales suivies du calcul du taux de production mitochondrial d’ATP par injection d’oligomycine A et du taux de production d’ATP glycolytique par injection de mélange roténone + antimycine A (inhibition totale de la fonction mitochondriale), entraînant une acidification mitochondriale17,27.
Test de stress des mitochondries cellulaires (ou test d’effort mito cellulaire): Cela mesure la fonction mitochondriale par la respiration liée à l’ATP, quantifie la bioénergétique cellulaire, identifie le dysfonctionnement mitochondrial et mesure la réponse des cellules au stress. Divers paramètres, y compris la capacité respiratoire basale et de rechange, la respiration liée à l’ATP, la respiration maximale et la consommation d’oxygène non mitochondriale, peuvent être obtenus en un seul test. Ce test implique des injections séquentielles d’oligomycine A, fccP (agent de découplage mitochondrial), un mélange d’inhibiteurs de roténone / antimycine A pour analyser efficacement l’effet de ceux-ci sur la fonction mitochondriale28.
Flexibilité mito fuel flex test: Cela mesure le taux de respiration mitochondriale par l’oxydation des trois combustibles mitochondriaux primaires par la présence et l’absence de leurs inhibiteurs. L’inhibition séquentielle du glucose, de la glutamine et des acides gras aide à mesurer la dépendance, la capacité et la flexibilité des cellules et la dépendance des cellules dans diverses voies cellulaires pour répondre à la demande d’énergie. Lorsque les mitochondries ne peuvent pas répondre aux exigences de la voie d’intérêt bloquée en oxydant d’autres combustibles, les cellules entrent dans un état de dépendance. La capacité des cellules est calculée par inhibition des deux autres voies alternatives suivies de l’inhibition de la voie d’intérêt. La flexibilité des cellules aide à comprendre la capacité des mitochondries à compenser et à répondre aux besoins en carburant de la voie inhibée. Il est calculé en soustrayant la dépendance des cellules de la capacité des cellules. Trois inhibiteurs différents sont utilisés indépendamment ou sous forme de mélange de deux pour calculer efficacement les paramètres du test. L’acide 2-cyano-3-(1-phényl-1H-indol-3-yl)-2-propénoïque (UK5099) inhibe l’oxydation du glucose en bloquant le transporteur de pyruvate dans la glycolyse. Le sulfure d’éthyle bis-2-(5-phénylacétamido-1,3,4-thiadiazol-2-yl) (BPTES) inhibe la voie d’oxydation de la glutamine et l’étomoxir inhibe l’oxydation des acides gras à longue chaîne29.
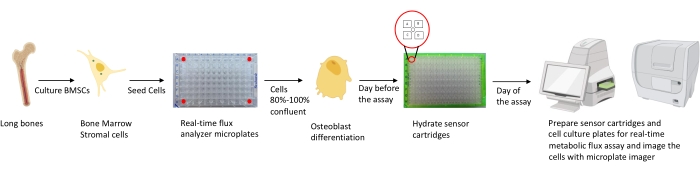
Figure 1 : Représentation schématique de la méthodologie de culture et de préparation des ostéoblastes pour analyse. Les BMSC murins sont isolés à partir d’os longs, cultivés et ensemencés dans des plaques de 96 puits à une densité de 25 000 cellules/puits. La culture de ces cellules dans des milieux spécifiques aux ostéoblastes commence lorsqu’elles atteignent une confluence de 80% à 100% pour commencer leur différenciation. Les tests sont effectués à différents stades de différenciation. Les plaques de cartouche sont hydratées un jour avant le test. Le jour du test, différents inhibiteurs sont injectés dans les orifices des cartouches de capteur en fonction des exigences du test, et un tampon d’étalonnage est ajouté à la plaque d’étalonnage de 96 puits. Après l’étalonnage, le test de flux métabolique cellulaire en temps réel est effectué, suivi de l’imagerie de la microplaque de culture cellulaire à l’aide de l’imageur de microplaques pour normaliser les données de l’analyseur de flux métabolique cellulaire en temps réel avec le nombre de cellules. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Access restricted. Please log in or start a trial to view this content.
Protocole
Toutes les procédures étaient basées sur les directives et l’approbation du comité institutionnel de soins et d’utilisation des animaux du centre médical de l’Université Vanderbilt.
1. Préparation des réactifs et configuration du test
- Isolement et culture des cellules stromales de la moelle osseuse (voir également l’article précédent30).
- Préparer un milieu de culture cellulaire alpha minimum complet (αMEM) en complétant le milieu essentiel minimum par une modification alpha avec 10 % de FBS (sérum bovin fœtal), 100 U/mL de pénicilline et 100 μg/mL de streptomycine.
- Préparer le tube de prélèvement de moelle osseuse en coupant l’extrémité d’un tube de microcentrifugation de 0,6 mL afin que les cellules puissent passer à travers et en l’insérant dans un tube de microcentrifugation de 1,5 mL contenant 100 μL d’αMEM complet.
- Euthanasier les souris en utilisant le traitement au CO2 comme suit. Placez l’animal dans la chambre à CO2 pendant 2-3 min ou jusqu’à ce que la respiration cesse. Attendez au moins 1 minute après que l’animal soit devenu inconscient pour retirer les souris de la chambre et les disloquer cervicalement.
- À l’aide d’une pince stérile et d’une paire de ciseaux, ouvrez le bas-ventre des souris euthanasiées pour faire une petite incision. Isolez les os longs (fémur, tibia et crête iliaque) des souris.
- Coupez les os longs pour enlever tous les tissus mous. Une fois l’os nettoyé, coupez environ 1 à 2 mm des extrémités distale et proximale pour créer une ouverture permettant à la moelle de rincer.
REMARQUE: Cette ouverture doit être conservatrice pour éviter de perdre la moelle tout en lui permettant de débusquer. - Placer les os dans un tube collecteur contenant 100 μL de 1x PBS stérile (solution saline tamponnée au phosphate) pour isoler la moelle osseuse totale.
- Rincer la moelle par centrifugation à 10 000 x g pendant 15-20 s à température ambiante. Les cellules de la moelle granulent au fond du tube.
- Répétez la centrifugation jusqu’à ce que la cavité osseuse apparaisse blanche et dépourvue de la plupart des éléments de la moelle. Remettre en suspension la population mixte de moelle osseuse en piquant doucement de haut en bas.
- Cultiver les cellules d’un animal (fémur et tibia) dans une fiole de culture cellulaire de 75 cm2 dans 10 mL de milieux de culture cellulaire et incuber à 37 °C dans un incubateur de culture cellulaire avec 5 % de CO2. Si vous mettez en commun des cellules de 2 à 3 animaux, utilisez une fiole de culture de 150 cmà 2 cellules (recommandé).
- Après 24 à 48 heures d’incubation de la population mixte, aspirer la population de cellules hématopoïétiques non adhérentes contenue dans le milieu de culture et laver les cellules adhérentes avec 1x PBS.
- Ensemencement cellulaire à partir de BMSC et différenciation des ostéoblastes
- Trypsiniser les cellules adhérentes en ajoutant suffisamment de trypsine-EDTA à 0,25 % (environ 3-4 mL) pour recouvrir légèrement la surface de la fiole, suivie d’une incubation de 3 min à 37 °C.
- Ajouter 6 à 7 mL d’αMEM complet à la fiole/trypsine pour remettre en suspension les BMSC adhérents en pipetant soigneusement de haut en bas. Transférer la suspension BMSC dans un tube de centrifugeuse conique.
- Retirez une aliquote de 50 μL de la suspension BMSC et ajoutez-y 50 μL de bleu de trypan (dilution 1:1). Compter le nombre total de cellules viables qui excluent le colorant en pipetant 10 μL de ce mélange sur un hémocytomètre et en l’observant au microscope. Ne comptez pas les cellules mortes ou malsaines qui semblent de couleur bleue (<10% de cellules).
- Sur la base du nombre de cellules, calculer le volume de suspension cellulaire en αMEM complet nécessaire pour une concentration finale de 2,4 x 106 cellules/mL pour un volume total d’au moins 10 mL par plaque.

Figure 2 : La microplaque de culture cellulaire, spécialement conçue pour l’analyseur. (A) Les quatre puits de correction de fond, A1, A12, H1, H12, sont mis en évidence. Ces puits ne contiennent que des milieux d’essai sans cellules. (B) Le code-barres sur le côté de la plaque pour scanner la plaque à l’aide du lecteur d’imagerie et de l’analyseur. Veuillez cliquer ici pour voir une version agrandie de cette figure.
- Centrifuger les cellules dans le tube conique à 1 000 x g pendant 5 min et remettre les cellules en suspension à la concentration finale souhaitée de 2,4 x 106 cellules/mL.
- Transférer la suspension cellulaire dans un réservoir et, à l’aide d’un pipet multicanal, remettre soigneusement en suspension les cellules pour assurer un mélange homogène de cellules.
- Ensemencez 2,5 x 104 cellules par puits dans la microplaque de culture cellulaire de 96 puits avec 80 μL d’αMEM complet. Ne pas ensemencer les cellules dans les puits de correction de fond (A1, A12, H1, H12); au lieu de cela, il suffit d’ajouter le milieu dans ces quatre puits.
REMARQUE: Les BMSC pour les tests sont plaqués dans une microplaque de culture cellulaire à 96 puits conçue pour l’analyseur en conjonction avec les cartouches de capteur. La surface de ces plaques est différente d’une plaque ordinaire de 96 puits. La surface de chaque puits dans la plaque est de 0,106 cm2, ce qui représente environ 40% de la surface typique de la plaque de 96 puits. La densité d’ensemencement cellulaire optimale est choisie en fonction du type de cellule. En règle générale, l’analyseur peut détecter entre 0,5 et 4 x 104 cellules par puits. Les ostéoblastes doivent être en contact pour se différencier efficacement; à cette fin, un placage entre 2,0 x 10 4-3,0 x 104 BMSC/puits dans 80 μL d’αMEM complet a été sélectionné. - Agiter doucement la plaque pour assurer la répartition uniforme des cellules dans les puits et incuber à 37 °C, 5% de CO2. Vérifiez la croissance des cellules et la confluence cellulaire au microscope après 48 h. Changez le milieu de culture cellulaire si nécessaire.
- Selon l’objectif du test, lorsque les BMSC sont confluents à 60% à 80% (généralement 48-72 h), initier la différenciation ostéoblastique en changeant le milieu de culture cellulaire en milieu de différenciation ostéoblastique (αMEM complet complété par 5 mM de phosphate de β-glycérol et 50 μg / mL d’acide L-ascorbique).
- Si des cellules stromales indifférenciées (jour 0) doivent être analysées, maintenir les cellules sous αMEM complet.
- Changez le milieu de différenciation des ostéoblastes tous les deux jours et visualisez les cellules au microscope pour vous assurer qu’elles sont en bonne santé jusqu’au jour du test. De préférence, 24 heures avant l’essai prévu, changer le support et maintenir un calendrier de changement moyen cohérent (recommandé).
REMARQUE: Changez soigneusement le support en inclinant légèrement les plaques à un angle; cela évite le contact accidentel des pointes de pipette avec les plaques de culture cellulaire et la perturbation de la monocouche des cellules.
2. Préparation de la cartouche de capteur pour l’étalonnage du flux extracellulaire
- Hydratez les cartouches de capteur du kit de dosage extracellulaire avant le jour du test. Retirez les cartouches du capteur (plaque verte) et placez les capteurs à l’envers.
- À l’aide d’un pipet multicanal, ajouter 200 μL deH2O à chaque puits de la plaque utilitaire. Replacez soigneusement les cartouches de capteur sur la plaque utilitaire et incubez la plaque pendant la nuit à température ambiante.
REMARQUE: Le fabricant recommande d’incuber les cartouches de capteur dans un incubateur sans CO2 37 °C pendant la nuit. Cependant, une évaporation importante des cartouches de capteur peut se produire. Si cela se produit, les cartouches de capteur peuvent être incubées à température ambiante. Ces plaques doivent être incubées pendant un minimum de 4 h et un maximum de 72 h. - Le jour de l’essai, jeter le H2O de la plaque utilitaire et ajouter 200 μL d’calibrant. Incuber la plaque d’utilité pendant au moins 1 h avant le test.
3. Préparation du milieu de l’analyseur de flux métabolique cellulaire en temps réel
- Utilisez le milieu DMEM sans rouge de phénol avec un pH préréglé de 7,4 (recommandé) pour exécuter le test avec des BMSC.
- Préparer 80 mL de milieux d’essai en complétant les milieux DMEM avec 1 mM de pyruvate de sodium, 2 mM de glutamine, 10 mM de glucose, 200 nM d’insuline, 50-200 μM d’acide oléique BSA.
- Incuber le milieu d’essai complet à 37 °C au bain-marie.
4. Préparation des composés pour les cartouches de capteur
- Décongeler l’oligomycine A, la roténone et l’antimycine A sur la glace. Pipette de haut en bas pour solubiliser les composés avant utilisation.
- Ajouter 3 mL de milieu d’essai préparé à chaque tube, suivi de l’ajout du tube composé A respectif: 26,4 μL de 2,5 mM d’oligomycine A; tube B : 3,1 μL de 12,67 mM de roténone + 4,1 μL de 9,4 mM d’antimycine A + 30 μL de coloration de Hoechst.
- Chargez une concentration de 10x de ces inhibiteurs dans le port correspondant. La concentration finale de solutions injectables nécessaires est de 2 μM d’oligomycine A, 1 μM de roténone et 4,1 μM d’antimycine A.
REMARQUE: Hoechst est ajouté à l’orifice d’injection final pour la coloration fluorescente des noyaux à des fins d’imagerie et de normalisation. Ces concentrations peuvent être optimisées en fonction du type de cellule. - Chargez 20 μL de ces composés dans les cellules dans 180 μL de milieux d’essai.
5. Préparer la microplaque de culture cellulaire pour l’essai
- Retirez la microplaque de culture cellulaire de l’incubateur à 37 °C et observez les cellules au microscope.
- Retirez le milieu d’essai du bain-marie.
- Lavez doucement les cellules avec 200 μL de milieu d’essai deux fois et ajoutez 200 μL de milieu d’essai par puits.
REMARQUE: Une fois que le support d’essai final est ajouté aux cellules, le temps jusqu’à ce que les plaques pénètrent dans l’analyseur est crucial. Par conséquent, ne commencez pas à remplacer le support avant que les étapes suivantes ne soient effectuées dans les 1 h. - Vérifiez les cellules au microscope pour vous assurer que les cellules restent adhérées aux puits.
- Assurez-vous que les cellules de D5 et E8 sont collées avec une monocouche cohérente et n’ont pas été emportées pendant l’étape de lavage. Le logiciel d’imagerie cellulaire utilise ces deux puits pour régler l’autofocus et l’exposition automatique.
REMARQUE: Le fabricant recommande d’incuber la plaque dans un incubateur sans CO2 37 °C pendant 1 h; cette étape peut être ignorée si l’imagerie automatisée est préférable. Par exemple, l’imageur à microplaques maintient les mêmes conditions dans une chambre fermée et les cellules peuvent être imagées sous un champ lumineux.
6. Configuration du test et de l’imagerie
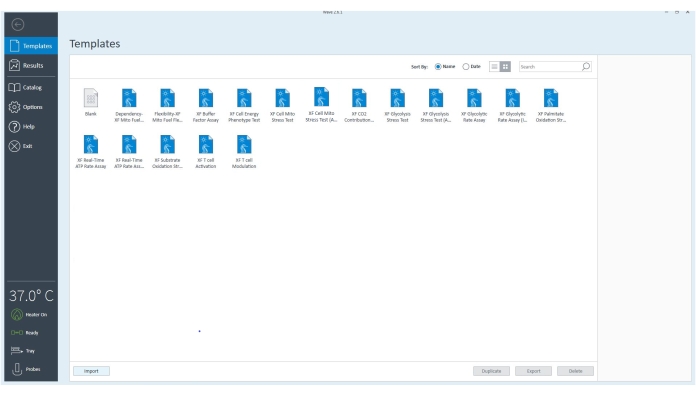
Figure 3 : logiciel du contrôleur. Le logiciel vérifie que l’équipement est connecté et réglé sur 37 °C. Les fichiers modèles pour différents tests pouvant être effectués avec l’analyseur de flux extracellulaire peuvent être sélectionnés pour personnaliser davantage le test en fonction des objectifs expérimentaux. Veuillez cliquer ici pour voir une version agrandie de cette figure.
- Ouvrez le logiciel de bureau dans l’ordinateur à côté de l’équipement.
- Vérifiez l’état de la connexion dans le coin inférieur gauche du logiciel du contrôleur.
- Accédez à Modèles et sélectionnez le fichier de modèle de test de taux ATP XF ou le modèle de test approprié.
- Sélectionnez Définitions de groupe en haut de l’écran et définissez les groupes.
- Sélectionnez la disposition de la carte des plaques et attribuez les puits en fonction des groupes définis.
- Vérifiez le protocole de l’instrument, assurez-vous que les composés ajoutés sont correctement répertoriés et incluez les informations du projet pour références futures.
- Cliquez sur Exécuter le test; cela vous invitera à sélectionner l’emplacement de stockage du fichier de résultats.
- Sélectionnez l’emplacement d’enregistrement du fichier de résultats.
- Enregistrez le fichier avec la date du test et cliquez sur Démarrer l’exécution.
- Placez la cartouche du capteur et la plaque utilitaire sur le plateau et cliquez sur Je suis prêt pour lancer l’étalonnage.
- Avant de commencer l’étalonnage, assurez-vous que le couvercle de la cartouche est retiré et que la cartouche du capteur est placée dans la bonne orientation sur la plaque utilitaire. Cette étape prendra 10 à 20 minutes et, une fois terminée, le logiciel affichera la boîte de dialogue Plaque de cellule de charge .
7. Obtenez des images en champ clair
REMARQUE : cette étape est facultative. Si aucun équipement d’imagerie n’est disponible, passez à l’étape 8.

Figure 4 : Le logiciel d’imagerie cellulaire communique avec le lecteur d’imagerie par l’intermédiaire de l’ordinateur. Les cellules de la microplaque peuvent être imagées avant et après le test, et le nombre de cellules / puits est obtenu après le test pour normaliser les données. Veuillez cliquer ici pour voir une version agrandie de cette figure.
- Ouvrez le logiciel d’imagerie cellulaire sur l’ordinateur.
- Assurez-vous que l’imageur de microplaques est allumé et que les ports sont connectés à l’ordinateur.
- Vérifiez la barre d’état en bas à gauche de l’écran pour vous assurer que la température est réglée sur 37 °C et que l’état de la connexion doit être mis en surbrillance en vert comme prêt.
- Scannez le code-barres de la plaque pour lancer le processus d’imagerie.
- Donnez un nom à la plaque de cellule et appuyez sur Enregistrer (c’est le nom où les images en champ lumineux et fluorescentes seront enregistrées). Cliquez sur Effectuer une analyse en fond clair.
- L’écran, la plaque et le menu de numérisation suivants affichent les options d’imagerie. Avant le test, sélectionnez Démarrer l’analyse en fond clair.
- Placez la microplaque de culture cellulaire avec le couvercle de la plaque sur le support du plateau et alignez bien A1 avec la marque A1. Cliquez sur Fermer le bac.
- L’écran suivant, l’acquisition d’images en champ clair, avec une carte de plaques apparaît. Cliquez sur Scan All Wells, qui lance le processus d’initialisation du système suivi de 30 à 35 minutes d’analyse.
- Après le balayage en champ lumineux, retirez la microplaque de culture cellulaire et placez-la dans l’analyseur pour effectuer le test.
8. Exécution du test
- Une fois l’étalonnage terminé, le logiciel affiche la boîte de dialogue Plaque de cellule de charge .
- Cliquez sur Ouvrir le plateau pour remplacer le plateau utilitaire par une microplaque de culture cellulaire. Assurez-vous que le couvercle est retiré et que le A1 de la plaque s’adapte à la bonne orientation.
- Ensuite, cliquez sur Load Cell Plate pour lancer le test. La cartouche du capteur restera à l’intérieur de l’analyseur pour les injections de dosage.
- Attendez que le test commence et affiche l’heure estimée d’achèvement.
- Une fois le test terminé, le logiciel affiche la boîte de dialogue Décharger la cartouche de capteur . Cliquez sur Éjecter et retirez la microplaque de culture cellulaire de l’analyseur.
- Retirez soigneusement la cartouche du capteur et remplacez le couvercle de la plaque de cellule. Les cellules sont prêtes pour l’imagerie fluorescente et le comptage cellulaire.
- Après avoir retiré la plaque de cellule et la cartouche du capteur, la boîte de dialogue Test terminé s’affiche .
- Cliquez sur Afficher les résultats pour ouvrir le fichier de résultats du test et normaliser les données immédiatement ou cliquez sur Accueil.
9. Obtenir des images de fluorescence et normaliser
REMARQUE: Cette étape est une méthode facultative mais préférée pour la normalisation des BMSC et des ostéoblastes. Si aucun équipement d’imagerie n’est disponible, une autre méthode de normalisation doit être effectuée, telle que l’isolement et la quantification des protéines ou de l’ADN.

Figure 5 : Images représentatives du logiciel d’imagerie utilisé pour la normalisation des données du test. (A) Image en champ lumineux cousu montrant la confluence cellulaire dans tout le puits. (B) Image de fluorescence cousue montrant des noyaux d’ostéoblastes colorés par Hoechst utilisés pour compter le nombre de cellules afin de normaliser les résultats du test. Ce sont des ostéoblastes après 7 jours de différenciation. Veuillez cliquer ici pour voir une version agrandie de cette figure.
- Une fois le test terminé, scannez le code-barres de la plaque avec le lecteur de code-barres portable. Si la plaque a déjà été imagée, elle ne nécessitera pas de nouveau nom.
- Sélectionnez Fluorescence & Cell Count, placez la plaque de cellule sur le support du plateau et cliquez sur Fermer le plateau.
- Dans la fenêtre d’acquisition d’image, sélectionnez Numériser tous les puits pour commencer l’imagerie. L’imagerie par fluorescence prend environ 15 à 20 minutes pour scanner toute la plaque. Observez la coche verte indiquant que l’analyse est terminée.
- Examinez les images fluorescentes et le nombre de cellules dans l’application d’imagerie et d’imagerie cellulaire en cliquant au hasard sur quelques puits.
REMARQUE: Il existe une option pour afficher les cellules comptées en bas à droite de l’écran. Cette option affiche une image masquée, mettant en surbrillance les objets qui ont été comptés. - Une fois l’imagerie par fluorescence terminée, exportez les images pour obtenir des références supplémentaires.
- Une fois l’imagerie et le nombre de cellules terminés, ouvrez le fichier Résultats et cliquez sur Normaliser. L’écran de normalisation donnera la disposition de la plaque et une option pour importer le nombre de cellules.
- Cliquez sur Importer et sélectionnez Appliquer pour le logiciel de bureau pour normaliser automatiquement le test avec le nombre de cellules.
Access restricted. Please log in or start a trial to view this content.
Résultats
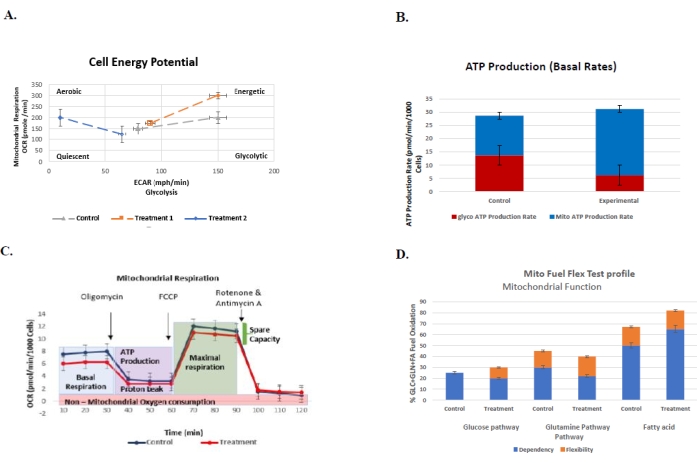
Figure 6 : Graphiques représentatifs des essais effectués régulièrement pour comprendre le profil bioénergétique cellulaire du groupe témoin par rapport au groupe de traitement avec leurs erreurs-types respectives. (A) Le test de phénotype de l’énergie cellulaire. Le graphique représente la glycolyse (ECAR) par ra...
Access restricted. Please log in or start a trial to view this content.
Discussion
L’analyseur de flux métabolique cellulaire en temps réel peut être utilisé pour explorer l’énergie cellulaire dans différentes conditions. Le protocole illustre l’isolement efficace des BMSC, la culture des cellules dans des plaques de culture cellulaire appropriées et leur différenciation en ostéoblastes matures, qui peuvent être utilisés pour divers tests à l’aide de l’analyseur de flux extracellulaire. En outre, les étapes critiques du test du flux métabolique cellulaire en temps réel, y compr...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Ce travail a été soutenu par le National Institute of Health (NIH) National Institute of Arthritis and Musculoskeletal and Skin Diseases (NIAMS) Grant AR072123 et national Institute on Aging (NIA) Grant AG069795 (à ERR).
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| 0.25% Trypsin EDTA | Sigma-Aldrich | T4049 | |
| 2-cyano-3-(1-phenyl-1H-indol-3-yl)-2-propenoic acid | Sigma - Aldrich | PZ0160 | UK5099 |
| Antimycin A | Sigma - Aldrich | A8674 | |
| Ascorbic acid | Sigma-Aldrich | A4544-100G | |
| Bis-2-(5-phenylacetamido-1,3,4-thiadiazol-2-yl)ethyl sulfide | Sigma - Aldrich | SML0601 | BPTES |
| Carbonyl cyanide 4-(trifluoromethoxy)phenylhydrazone | Sigma - Aldrich | C2920 | FCCP |
| Cytation 5 imaging reader | BioTek | N/A | Microplate imager |
| Etomoxir sodium salt hydrate | Sigma - Aldrich | E1905 | |
| Hoechst 33342 Solution (20 mM) | Thermo Scientific | 62249 | |
| Insulin | Sigma - Aldrich | I6634 | |
| Oleic Acid-Albumin from bovine serum | Sigma - Aldrich | O3008 | |
| Oligomycin A - 5 mg | Sigma - Aldrich | 75351 | |
| Rotenone | Sigma - Aldrich | R8875-1G | |
| Seahorse XF 1.0 M Glucose Solution | Agilent Technologies | 103577-100 | |
| Seahorse XF 100mM Pyruvate Solution | Agilent Technologies | 103578-100 | |
| Seahorse XF 200mM Glutamine solution | Agilent Technologies | 103579-100 | |
| Seahorse XF DMEM media | Agilent Technologies | 103575-100 | DMEM assay media eith 5mM HEPES, pH 7.4, without phenol red, sodium bicarbonate, glucose, pyruvate, and L-glutamine |
| Seahorse XFe96 Analyzer | Agilent Technologies | S7800B | Real- Time Metabolic flux analyzer |
| Seahorse XFe96 FluxPak | Agilent Technologies | 102416-100 | Includes XFe96 Sensor cartridges, Cell culture microplates, and Seahorse XF Calibrant solution |
| The Cell imaging 1.1.0.11 software | Agilent Technologies - BioTek | ||
| Wave software 2.6.1 | Agilent Technologies | ||
| β-glycerol phosphate | Sigma-Aldrich | G9422-50G |
Références
- Rodan, G. A. Bone homeostasis. Proceedings of the National Academy of Sciences of the United States of America. 95 (23), 13361-13362 (1998).
- Nakahama, K. I. Cellular communications in bone homeostasis and repair. Cellular and Molecular Life Sciences. 67 (23), 4001-4009 (2010).
- Kim, J. M., Lin, C., Stavre, Z., Greenblatt, M. B., Shim, J. H. Osteoblast-osteoclast communication and bone homeostasis. Cells. 9 (9), 2073(2020).
- Gao, J., et al. SIRT3/SOD2 maintains osteoblast differentiation and bone formation by regulating mitochondrial stress. Cell Death and Differentiation. 25 (2), 229-240 (2018).
- Baron, R. Molecular mechanisms of bone resorption by the osteoclast. The Anatomical Record. 224 (2), 317-324 (1989).
- Tian, L., Rosen, C. J., Guntur, A. R. Mitochondrial Function and Metabolism of Cultured Skeletal Cells. Methods in Molecular Biology. 2230, Clifton, N.J. 437-447 (2021).
- Zanotelli, M. R., et al. Regulation of ATP utilization during metastatic cell migration by collagen architecture. Molecular Biology of the Cell. 29 (1), 1-9 (2018).
- Gonzales, S., Wang, C., Levene, H., Cheung, H. S., Huang, C. Y. C. ATP promotes extracellular matrix biosynthesis of intervertebral disc cells. Cell and Tissue Research. 359 (2), 635-642 (2015).
- Kruse, N. J., Bornstein, P. The metabolic requirements for transcellular movement and secretion of collagen. Journal of Biological Chemistry. 250 (13), 4841-4847 (1975).
- Rendina-Ruedy, E., Guntur, A. R., Rosen, C. J. Intracellular lipid droplets support osteoblast function. Adipocyte. 6 (3), 250-258 (2017).
- Sinnott-Armstrong, N., et al. A regulatory variant at 3q21.1 confers an increased pleiotropic risk for hyperglycemia and altered bone mineral density. Cell Metabolism. 33 (3), 615-628 (2021).
- Esen, E., Lee, S. Y., Wice, B. M., Long, F. PTH promotes bone anabolism by stimulating aerobic glycolysis via IGF signaling. Journal of Bone and Mineral Research. 30 (11), 1959-1968 (2015).
- Borle, A. B., Nichols, N., Nichols, G. Metabolic studies of bone in vitro: I. Normal bone. Journal of Biological Chemistry. 235, 1206-1210 (1960).
- Borle, A. B., Nichols, N., Nichols, G. Metabolic studies of bone in vitro: II. The metabolic patterns of accretion and resorption. Journal of Biological Chemistry. 235, 1211-1214 (1960).
- Adamek, G., Felix, R., Guenther, H. L., Fleisch, H. Fatty acid oxidation in bone tissue and bone cells in culture. Characterization and hormonal influences. The Biochemical Journal. 248 (1), 129-137 (1987).
- Frey, J. L., et al. Wnt-Lrp5 signaling regulates fatty acid metabolism in the osteoblast. Molecular and Cellular Biology. 35 (11), 1979-1991 (2015).
- Romero, N., Rogers, G., Neilson, A., Dranka, B. P. Quantifying cellular ATP production rate using agilent seahorse XF technology. , (2018).
- vander Windt, G., Chang, C., Pearce, E. Measuring bioenergetics in T cells using a Seahorse Extracellular Flux Analyzer. Current Protocols in Immunology. 113, 1(2016).
- Traba, J., Miozzo, P., Akkaya, B., Pierce, S. K., Akkaya, M. An optimized protocol to analyze glycolysis and mitochondrial respiration in lymphocytes. Journal of Visualized Experiments:JoVE. (117), e54918(2016).
- Noel, P., et al. Preparation and metabolic assay of 3-dimensional spheroid co-cultures of pancreatic cancer cells and fibroblasts. Journal of Visualized Experiments:JoVE. (126), e56081(2017).
- Nicholls, D., et al. Bioenergetic profile experiment using C2C12 myoblast cells. Journal of Visualized Experiments: JoVE. (46), e2511(2010).
- Sakamuri, S. S. V. P., et al. Measurement of respiratory function in isolated cardiac mitochondria using Seahorse XFe24 Analyzer: applications for aging research. GeroScience. 40 (3), 347-356 (2018).
- What are the advantages of using Seahorse XF technology. , Available from: https://www.agilent.com/en/support/cell-analysis/advantages-of-using-xf-tech (2018).
- Horan, M. P., Pichaud, N., Ballard, J. W. O. Review: Quantifying mitochondrial dysfunction in complex diseases of aging. Journals of Gerontology - Series A Biological Sciences and Medical Sciences. 67 (10), 1022-1035 (2012).
- XF cell energy phenotype test. , Available from: https://www.agilent.com/en/product/cell-analysis/real-time-cell-metabolic-analysis/xf-assay-kits-reagents-cell-assay-media/seahorse-xf-cell-energy-phenotype-test-kit-740884 (2021).
- Leung, D. T. H., Chu, S. Measurement of oxidative stress: Mitochondrial function using the seahorse system. Methods in Molecular Biology. 1710, Clifton, N.J. 285-293 (2018).
- XF ATP rate assay. , Available from: https://www.agilent.com/en/product/cell-analysis/real-time-cell-metabolic-analysis/xf-assay-kits-reagents-cell-assay-media/seahorse-xf-cell-energy-phenotype-test-kit-740889 (2021).
- XF cell mito stress test. , Available from: https://www.agilent.com/en/product/cell-analysis/real-time-cell-metabolic-analysis/xf-assay-kits-reagents-cell-assay-media/seahorse-xf-cell-energy-phenotype-test-kit-740885 (2021).
- XF cell mito fuel flex test. , Available from: https://www.agilent.com/en/product/cell-analysis/real-time-cell-metabolic-analysis/xf-assay-kits-reagents-cell-assay-media/seahorse-xf-cell-energy-phenotype-test-kit-740888 (2021).
- Maridas, D. E., Rendina-Ruedy, E., Le, P. T., Rosen, C. J. Isolation, culture, and differentiation of bone marrow stromal cells and osteoclast progenitors from mice. Journal of Visualized Experiments: JoVE. (131), e56750(2018).
- Wei, J., et al. Glucose uptake and Runx2 synergize to orchestrate osteoblast differentiation and bone formation. Cell. 161 (7), 1576-1591 (2015).
- Zoch, M. L., Abou, D. S., Clemens, T. L., Thorek, D. L. J., Riddle, R. C. In vivo radiometric analysis of glucose uptake and distribution in mouse bone. Bone Research. 4, 16004(2016).
- Divakaruni, A. S., Paradyse, A., Ferrick, D. A., Murphy, A. N., Jastroch, M. Analysis and interpretation of microplate-based oxygen consumption and pH data. Methods in Enzymology. 547, 309-354 (2014).
- Kam, Y., Jastromb, N., Clayton, J., Held, P., Dranka, B. Normalization of agilent seahorse XF data by in-situ cell counting using a BioTek cytation 5 application note. , (2017).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon