Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Medición de la compresibilidad de célula y núcleo basada en microdispositivo acustofluídico
En este artículo
Resumen
Aquí se presenta un protocolo para construir un sistema rápido y no destructivo para medir la compresibilidad de células o núcleos basado en microdispositivos acustofluídicos. Se investigaron los cambios en las propiedades mecánicas de las células tumorales después de la transición epitelial-mesenquimal o la radiación ionizante, lo que demuestra la perspectiva de aplicación de este método en la investigación científica y la práctica clínica.
Resumen
La mecánica celular juega un papel importante en la metástasis tumoral, la transformación maligna de las células y la radiosensibilidad. Durante estos procesos, estudiar las propiedades mecánicas de las células es a menudo un desafío. Los métodos de medición convencionales basados en el contacto, como la compresión o el estiramiento, son propensos a causar daño celular, lo que afecta la precisión de la medición y el cultivo celular posterior. Las mediciones en estado adherente también pueden afectar la precisión, especialmente después de la irradiación, ya que la radiación ionizante aplanará las células y mejorará la adhesión. Aquí, se ha desarrollado un sistema de medición de mecánica celular basado en el método acustofluídico. La compresibilidad celular se puede obtener registrando la trayectoria de movimiento de la celda bajo la acción de la fuerza acústica, que puede realizar mediciones rápidas y no destructivas en estado suspendido. Este documento informa en detalle los protocolos para el diseño de chips, preparación de muestras, registro de trayectoria, extracción de parámetros y análisis. La compresibilidad de diferentes tipos de células tumorales se midió con base en este método. La medición de la compresibilidad del núcleo también se logró ajustando la frecuencia de resonancia de la cerámica piezoeléctrica y el ancho del microcanal. Combinado con la verificación a nivel molecular de los experimentos de inmunofluorescencia, se comparó la compresibilidad celular antes y después de la transición epitelial a mesenquimal inducida por fármacos (EMT). Además, se reveló el cambio de compresibilidad celular después de la irradiación de rayos X con diferentes dosis. El método de medición de la mecánica celular propuesto en este documento es universal y flexible y tiene amplias perspectivas de aplicación en la investigación científica y la práctica clínica.
Introducción
Las propiedades mecánicas celulares juegan un papel importante en la metástasis tumoral, la transformación maligna de las células y la radiosensibilidad 1,2. Para obtener una comprensión profunda del papel de las propiedades mecánicas celulares en el proceso anterior, la medición precisa de la mecánica celular es crítica, y la medición no debe causar daño a las células para su posterior cultivo y análisis. El proceso de medición debe ser lo más rápido posible, de lo contrario, la viabilidad celular puede verse afectada si las células se eliminan del entorno de cultivo durante mucho tiempo.
Los métodos de medición de mecánica celular existentes enfrentan algunas limitaciones. Algunos métodos, como la citometría de torsión magnética, las pinzas magnéticas y la microrreología de seguimiento de partículas, causan daño celular debido a la introducción de partículas en las células 3,4,5. Los métodos que miden por contacto con células, como el microscopio de fuerza atómica (AFM), la aspiración de micropipetas, la microconstricción y la técnica de placa paralela, también son propensos al daño celular y el rendimiento es difícil de aumentar 6,7,8. Además, la radiación ionizante aplanará las células y aumentará su adhesión9; Por lo tanto, es necesario medir la mecánica celular completa en suspensión.
En respuesta a los desafíos anteriores, se ha desarrollado un sistema de medición de mecánica celular basado en el método acustofluídico 10,11,12,13,14. El ancho del canal se adapta a la media longitud de onda acústica, creando así un nodo de onda estacionaria en la línea media del microcanal. Bajo la acción de la fuerza de radiación acústica, las células o perlas estándar pueden moverse al nodo de presión acústica. Dado que se conocen las propiedades físicas de las perlas estándar (tamaño, densidad y compresibilidad), se puede determinar la densidad de energía acústica. Luego, la compresibilidad celular se puede obtener registrando las trayectorias de movimiento de las células en el campo acústico. Se puede lograr una medición no destructiva de alto rendimiento de células en estado de suspensión. Este documento presentará el diseño del chip microfluídico, el establecimiento del sistema y los pasos de medición. Se ha llevado a cabo la medición de varios tipos de células tumorales para verificar la precisión del método. El ámbito de aplicación de este método se había extendido a estructuras subcelulares (como el núcleo) ajustando la frecuencia de resonancia de la cerámica piezoeléctrica y el ancho del microcanal. Además, se investigaron los cambios en la compresibilidad celular después de la EMT inducida por fármacos o la irradiación de rayos X con diferentes dosis. Los resultados demuestran la amplia aplicabilidad de este método como una poderosa herramienta para estudiar la correlación entre los cambios bioquímicos y las propiedades mecánicas celulares.
Protocolo
1. Fabricación y montaje del microdispositivo acustofluídico
- Fabricación del chip microfluídico.
- Diseñe un chip de un solo canal con una sola entrada y salida como se muestra en la Figura 1. Para las células de medición, mantener la sección transversal rectangular del microcanal a 740 μm de ancho y 100 μm de profundidad. Para medir el núcleo celular, cambie el ancho y la profundidad del microcanal a 250 μm y 100 μm, respectivamente.
- Prepare el microcanal en la oblea de silicio mediante grabado iónico reactivo. Selle la parte superior del microcanal con una pieza de vidrio transparente resistente al calor mediante unión anódica15. Lave las patatas fritas con un limpiador ultrasónico durante 10 minutos. Séquelos en un horno de secado a 50 °C para su uso posterior.
- Fabricar bloques de polidimetilsiloxano (PDMS).
- Agregue 30 ml de prepolímero a un plato de vidrio de 100 mm (de diámetro). Agregue 3 ml de agente de curado al prepolímero con una jeringa.
NOTA: La relación de volumen del agente de curado y el prepolímero es de 1:10. - Mezcle vigorosamente el prepolímero PDMS y el agente de curado con una varilla de vidrio durante unos 10 minutos. Busque burbujas de aire pequeñas y uniformemente separadas en la solución, que indican que el prepolímero PDMS y el agente de curado están bien mezclados.
- Coloque el plato de vidrio en un desecador al vacío y evacúe durante 15-25 s. Repita este proceso hasta que no haya burbujas de aire en la mezcla.
- Colocar el plato de vidrio en un horno de secado a 50 °C durante 1 h para permitir que la mezcla se cure. Después de la incubación, use un bisturí para cortar el PDMS en bloques de tamaño adecuado de aproximadamente 1,2 cm de largo y 1 cm de ancho.
NOTA: La longitud del bloque PDMS es consistente con el ancho del chip, y el ancho se selecciona para garantizar que haya suficiente espacio en el medio para la cerámica piezoeléctrica cuando se adhieren dos bloques PDMS en el chip.
- Agregue 30 ml de prepolímero a un plato de vidrio de 100 mm (de diámetro). Agregue 3 ml de agente de curado al prepolímero con una jeringa.
- Enlace el bloque PDMS al chip.
- Perfore agujeros en el bloque PDMS para puertos de entrada y salida con una aguja hueca de 1 mm de diámetro. Coloque los bloques PDMS y el chip (parte posterior hacia arriba) en un limpiador de plasma durante 1 minuto.
- Alinee los orificios de los bloques PDMS con la entrada y salida del chip. Presione suavemente los bloques PDMS al chip durante 15 s. Esto debería causar que se produzca una unión entre los bloques PDMS y la superficie del chip.
- Conecte el catéter de politetrafluoroetileno (PTFE) al chip (Figura 2B).
- Corte dos piezas de catéter de PTFE con un diámetro interior de 0,8 mm y una longitud de 10 cm. Doblar una aguja de acero inoxidable con un diámetro interior de 0,7 mm y una longitud de 1,5 cm por 90° en forma de L. Conéctelo a un extremo del catéter. Prepare dos de estos catéteres con agujas.
- Inserte las agujas de acero inoxidable en los orificios de los bloques PDMS. Para la entrada, conecte una aguja dispensadora de 19 G al otro extremo del catéter como conector para una jeringa.
- Después de completar los pasos anteriores, inyecte agua desionizada para probar la estanqueidad del canal general. Impermeable al agua significa un buen sello.
- Conjunto cerámico piezoeléctrico (Figura 2C)
- Utilice un cortador de alambre de diamante para cortar láminas de cerámica piezoeléctrica con un diámetro de 2 cm en cuatro tiras con un ancho de 5 mm.
- Asegúrese de que la frecuencia de resonancia de la cerámica piezoeléctrica coincida con el ancho del microcanal del chip. Para el microcanal de 740 μm y 250 μm de ancho, utilice cerámicas piezoeléctricas con frecuencias de resonancia de 1 MHz y 3 MHz, respectivamente.
- Suelde los cables en ambos lados de la cerámica piezoeléctrica en un extremo.
- Pegue la cerámica piezoeléctrica al centro de la parte posterior del chip con pegamento de cianoacrilato.
- Para extender el pegamento uniformemente, coloque una gota de pegamento sobre la cerámica piezoeléctrica, alise el pegamento con un palillo de dientes y retire el exceso de pegamento. Luego, presiónelo rápidamente en el chip y continúe presionando durante aproximadamente 1 minuto. Asegúrese de que la cerámica piezoeléctrica y el chip estén firmemente unidos y en contacto uniforme.
- Monte el microdispositivo (Figura 2D).
- Corte una pieza de PDMS (aproximadamente 1,5 cm de largo y 1 cm de ancho) como base del microdispositivo. Con cinta adhesiva de doble cara, pegue un lado de la base a los bloques PDMS de entrada y salida, y el otro lado a una corredera de vidrio transparente. Fije todo el microdispositivo a la etapa del microscopio para mantener el chip en un plano focal.
2. Preparación de la muestra
- Preparación de soluciones patrón de partículas de poliestireno.
- Añadir 0,05 ml de solución de partículas de poliestireno (6 μm de diámetro) (2,1 x 108 partículas/ml) a 10 ml de solución salina tamponada con fosfato (PBS) y mezclar bien.
NOTA: Para reducir el error de medición causado por el cambio de la densidad de energía acústica, la solución de partículas de poliestireno se mezcló con la solución de muestra en cada experimento como calibración.
- Añadir 0,05 ml de solución de partículas de poliestireno (6 μm de diámetro) (2,1 x 108 partículas/ml) a 10 ml de solución salina tamponada con fosfato (PBS) y mezclar bien.
- Preparación de suspensiones celulares.
- Lave las células adherentes (por ejemplo, MCF7, MDA-MB-231, HCT116) al 90% de confluencia (~5 x 105 células) con PBS. Añadir 500 μL de tripsina al 0,25% (1x) durante 1-2 min a temperatura ambiente (25 °C). Retire la tripsina, agregue 1 ml de medio completo y forme una suspensión celular por pipeteo.
- Centrifugar la suspensión celular a 100 x g durante 5 min. Retire el sobrenadante y vuelva a suspender en 0.5-1 mL de PBS para obtener una suspensión celular. Las células se contaron con un hemocitómetro y la concentración fue de aproximadamente 3-5 x 105 células/ml.
- Preparación de la suspensión del núcleo celular
- Realice el paso 2.2. Luego, retire el sobrenadante y agregue 200 μL de reactivo de extracción de proteínas citoplasmáticas A (suplementado con PMSF al 1%) por pellet de células de 20 μL (aproximadamente 5 millones de células) y mezcle bien.
- Vortex la mezcla anterior a 220 x g durante 5 s, y luego colóquela en un baño de hielo durante 10 min. Después de la incubación, añadir 10 μL de reactivo B de extracción de proteínas citoplasmáticas a la solución.
- Vórtice a 220 x g durante 5 s. Colocar en baño de hielo durante 1 min y volver a vorámice a 220 x g durante 5 s. Luego, finalmente centrifugar a 1.000 x g durante 5 min a 4 °C.
NOTA: La relación de volumen de los reactivos de extracción de proteínas citoplasmáticas A y B es de 20:1. - Retire el sobrenadante y vuelva a suspender el pellet en 1 ml de PBS. A continuación, centrifugar a 1.000 x g a 4 °C durante 4 min. Retirar el sobrenadante y resuspender en 100 μL de PBS como suspensión del núcleo celular.
- Añadir azul de tripano a la suspensión del núcleo celular anterior y teñir a temperatura ambiente (25 °C) durante 4 min. La relación de volumen de la solución de azul de tripano a la suspensión del núcleo es de 1:1. Cuente el número de núcleos bajo el microscopio invertido con un objetivo 10x.
NOTA: Para identificar claramente los núcleos celulares bajo el microscopio, se requiere tinción de azul de tripano. La solución de azul de tripano debe estar en un baño de agua a 37 °C durante 10 minutos antes de usarla para una tinción efectiva. - Diluir la suspensión del núcleo celular anterior con tampón PBS a una concentración de 2-3 x 105 núcleos/ml. Filtrar la suspensión del núcleo celular a través de un tamiz de 70 μm.
3. Medición de la compresibilidad de la célula y el núcleo
- Configurar el sistema de medición (Figura 3)
- Encienda la fuente de luz del microscopio y abra el software de la cámara. Utilice el objetivo 4x para encontrar la posición media del microcanal, es decir, la posición de la cerámica piezoeléctrica.
- Conecte los cables y suéldelos a los terminales positivo y negativo de la salida del generador de señal en la cerámica piezoeléctrica, respectivamente.
- Coloque la jeringa en la bomba de microinyección y conéctela al catéter de entrada. Coloque un recipiente pequeño en el extremo del catéter de salida para contener el líquido que fluye fuera del microcanal.
- Determinar los parámetros de medición
- Aspire la solución de partículas de poliestireno con la jeringa e inyéctela en el microcanal del chip. Evite las burbujas de aire en el microcanal del chip para garantizar una medición precisa. Asegúrese de que las partículas se distribuyan uniformemente en el microcanal del chip.
NOTA: La medición se puede realizar sin flujo o bomba de jeringa. Si es necesario, el caudal de la bomba de microinyección debe ajustarse a un valor adecuado. Aquí, el rango de caudal es de 0-20 μL / h. - Ajuste la salida del generador de señal a una señal sinusoidal con una frecuencia de 1 MHz (3 MHz para la medición del núcleo celular) y una tensión pico a pico (Vpp) de 10 V.
- Ajuste la frecuencia de la señal hasta que se observe que las partículas se mueven hacia la línea media del microcanal y permanecen en movimiento hacia adelante a lo largo de la línea media después de alcanzar la línea media (Figura 4).
NOTA: La velocidad de las partículas que se mueven hacia la línea media está determinada por la amplitud del voltaje, que se puede ajustar entre 5 Vpp y 20 Vpp.
- Aspire la solución de partículas de poliestireno con la jeringa e inyéctela en el microcanal del chip. Evite las burbujas de aire en el microcanal del chip para garantizar una medición precisa. Asegúrese de que las partículas se distribuyan uniformemente en el microcanal del chip.
- Medir células y núcleos
- Mezcle 1 ml de suspensión de células o núcleos con la solución de partículas estándar en una proporción de 1:1 e inyecte en el microcanal con una jeringa.
- Comience a grabar con la cámara CCD cuando las células o núcleos entren en el campo de visión. Luego, encienda el generador de señales. Deje de grabar cuando las células o núcleos lleguen a la línea media.
- Enjuague el microcanal con agua desionizada, alcohol al 75% y agua desionizada en secuencia para su uso posterior.
4. Tratamiento de datos
- Mapear trayectorias de partículas o células.
- Importe el video capturado al software ImageJ: Archivo > Abrir> Seleccione Carpeta. Haga clic en la forma de elipse en la barra de herramientas del software ImageJ para seleccionar una celda de interés y su partícula adyacente (Figura 5).
- Como se muestra en la Figura 5, parámetros de medición preestablecidos en el software ImageJ como Analizar→ Establecer medición →Área, Centroide, Etiqueta de visualización.
- Tomando el marco donde la célula o partícula objetivo sufre desplazamiento longitudinal como marco inicial; Registre la posición del píxel y el tamaño de la celda o partícula en cada fotograma hasta que llegue a la línea media del microcanal. Exporte los datos como un archivo de hoja de cálculo y repita el paso hasta que se obtengan trayectorias para todas las celdas de interés.
- Coordinar la transformación y corrección.
- Registre las coordenadas de píxeles de las cuatro esquinas del microcanal en este campo de visión como (0, y1), (0, y2), (x3, y3), (x4, y4). Aquí x3 = x4.
- Para cada punto de medición (xi, yi), calcule la nueva coordenada (xi', yi') después de la corrección de rotación utilizando las siguientes fórmulas:
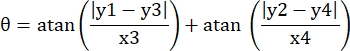
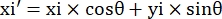

- Convierte coordenadas de píxeles en coordenadas de tamaño real. Las coordenadas reales se pueden obtener multiplicando las coordenadas de píxeles por la proporción. La relación era el ancho real del microcanal dividido por el ancho de píxeles (H) del microcanal.
- Transformar y corregir las coordenadas en píxeles de las células y partículas obtenidas en el paso 4.1 a los datos de trayectoria de movimiento final. Todas las coordenadas menos las coordenadas de la esquina inferior izquierda, es decir, (0, y2). La velocidad de fotogramas del vídeo es de 40 fotogramas por segundo, por lo que multiplica el número de fotogramas correspondientes a cada coordenada por 0,025 s para obtener el tiempo de movimiento de las partículas, obteniendo así el cambio de la posición en la dirección y con el tiempo.
- Calcule la densidad de energía acústica (Figura 6A, B).
- El movimiento de la célula o partícula en la dirección Y es impulsado por la fuerza acústica F ac y la fuerza hidrodinámica FD. Calcula la trayectoria del movimiento utilizando las siguientes fórmulas:
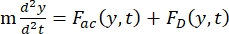 (1)
(1)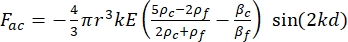 (2)
(2)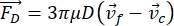 (3)
(3)
donde r y D son el radio y el diámetro de la célula o partícula, ρ y β son la densidad y la compresibilidad, ν es el vector velocidad. Los subíndices c y f denotan la célula y el fluido, respectivamente. d es la distancia desde el nodo de presión acústica más cercano, μ es la viscosidad dinámica del fluido, k es el número de onda y E es la densidad de energía acústica.
NOTA: La densidad de MCF7, HCT116, A549 y núcleos celulares fue de 1068 kg/m 3, 1077 kg/m 3, 1073 kg/m 3 y 1155 kg/m 3, respectivamente 12,16,17. - De acuerdo con las fórmulas descritas en el paso 4.3.1, utilice el software MATLAB para obtener la solución numérica para la trayectoria de partículas estándar bajo el método de campo acústico con diferencia finita.
- Dentro del rango de campo acústico preestablecido, cambie la densidad de energía acústica y ajuste la solución numérica (obtenida en el paso 4.3.2) y la trayectoria de movimiento medida (obtenida en el paso 4.2) para la partícula estándar. Seleccione el mejor resultado de ajuste de acuerdo con el error cuadrático medio de ajuste. La densidad de energía acústica obtenida aquí se utiliza como parámetro para el cálculo posterior de la compresibilidad celular.
- El movimiento de la célula o partícula en la dirección Y es impulsado por la fuerza acústica F ac y la fuerza hidrodinámica FD. Calcula la trayectoria del movimiento utilizando las siguientes fórmulas:
- Calcule la compresibilidad celular (Figura 6C, D).
- Ajuste la densidad de energía acústica al valor obtenido en el paso 4.3.3.
- De acuerdo con las fórmulas descritas en el paso 4.3.1, utilice el software MATLAB para obtener la solución numérica para la trayectoria celular bajo el campo acústico con el método de diferencia finita.
- Similar al paso 4.3.3, dentro del rango de compresibilidad preestablecido, cambie la compresibilidad y ajuste la solución numérica (obtenida en el paso 4.4.2) y la trayectoria de movimiento medida para la celda (obtenida en el paso 4.2). Utilice el coeficiente de compresibilidad correspondiente al resultado de mejor ajuste como la compresibilidad celular medida.
Resultados
Aquí, el trabajo presentó un protocolo para la construcción de un sistema de medición de compresibilidad celular rápido y no destructivo basado en un microdispositivo acustofluídico y demostró sus ventajas para medir células y núcleos en diferentes situaciones. La figura 1 muestra el esquema del canal microfluídico. Los componentes y el ensamblaje del microdispositivo acustofluídico se muestran en la Figura 2. La figura 3...
Discusión
Los métodos de medición de mecánica celular comúnmente utilizados son AFM, aspiración de micropipetas, métodos microfluídicos, técnica de placa paralela, pinzas ópticas, camilla óptica y métodos acústicos20. Los métodos de microfluídica pueden funcionar con tres enfoques: microconstricción, flujo extensional y flujo de corte. Entre ellos, la camilla óptica, las pinzas ópticas, los métodos acústicos, el flujo extensional y los enfoques de flujo cortante son mediciones sin contact...
Divulgaciones
Los autores no tienen intereses financieros contrapuestos u otros conflictos de intereses.
Agradecimientos
Este estudio fue apoyado por la Fundación Nacional de Ciencias Naturales de China (números de subvención 12075330 y U1932165) y la Fundación de Ciencias Naturales de la provincia de Guangdong, China (número de subvención 2020A1515010270).
Materiales
| Name | Company | Catalog Number | Comments |
| 0.25% trypsin(1x) | GIBCO | 15050-065 | |
| 502 glue | Evo-bond | cyanoacrylate glue | |
| A549 | ATCC | CCL-185 | lung adenocarcinoma |
| Cytonucleoprotein and cytoplasmic protein extraction kit | Beyotime | P0027 | Contains cytoplasmic protein extraction reagents A and B |
| Dulbecco’s modified Eagle medium (DMEM) | corning | 10-013-CVRC | |
| Fetal Bovine Srum(FBS) | AUSGENEX | FBS500-S | |
| HCT116 | ATCC | CCL247 | colorectal carcinoma |
| Heat-resistant glass | Pyrex | ||
| Leibovitz’s L-15 medium | GIBCO | 11415-064 | |
| MCF-7 | ATCC | HTB-22 | breast Adenocarcinoma |
| MDA-MB-231 | ATCC | HTB-26 | breast Adenocarcinoma |
| Minimum Essential Medium (MEM) | corning | 10-010-CV | |
| Penicillin-Streptomycin | GIBCO | 15140-122 | |
| Phosphate buffer | corning | 21-040-cvc | |
| PMSF | Beyotime | ST506 | 100mM |
| Polybead Polystyrene Red Dyed Microsphere | polysciences | 15714 | The diameter of microshpere is 6.00µm |
| propidium iodide(PI) | Sigma-Aldrich | P4170 | |
| SYLGARD 184Silicone ELASTOMER | Dow-Corning | 1673921 | Contains prepolymers and curing agents |
| Trypan Blue | Beyotime | C0011 |
Referencias
- Wirtz, D., Konstantopoulos, K., Searson, P. C. The physics of cancer: the role of physical interactions and mechanical forces in metastasis. Nature Reviews. Cancer. 11 (7), 512-522 (2011).
- Frame, F. M., et al. HDAC inhibitor confers radiosensitivity to prostate stem-like cells. British Journal of Cancer. 109 (12), 3023-3033 (2013).
- Tseng, Y., Kole, T. P., Wirtz, D. Micromechanical mapping of live cells by multiple-particle-tracking microrheology. Biophysical Journal. 83 (6), 3162-3176 (2002).
- Möller, W., Brown, D. M., Kreyling, W. G., Stone, V. Ultrafine particles cause cytoskeletal dysfunctions in macrophages: role of intracellular calcium. Particle and Fibre Toxicology. 2, 7 (2005).
- Wang, X., et al. A three-dimensional magnetic tweezer system for intraembryonic navigation and measurement. IEEE Transactions on Robotics. 34 (1), 240-247 (2018).
- Machida, S., et al. Direct manipulation of intracellular stress fibres using a hook-shaped AFM probe. Nanotechnology. 21 (38), 385102 (2010).
- Bufi, N., et al. Human primary immune cells exhibit distinct mechanical properties that are modified by inflammation. Biophysical Journal. 108 (9), 2181-2190 (2015).
- Hogan, B., Babataheri, A., Hwang, Y., Barakat, A. I., Husson, J. Characterizing cell adhesion by using micropipette aspiration. Biophysical Journal. 109 (2), 209-219 (2015).
- Jung, J. -. W., et al. Ionising radiation induces changes associated with epithelial-mesenchymal transdifferentiation and increased cell motility of A549 lung epithelial cells. European Journal of Cancer. 43 (7), 1214-1224 (2007).
- Hartono, D., et al. On-chip measurements of cell compressibility via acoustic radiation. Lab-on-a-Chip. 11 (23), 4072-4080 (2011).
- Sitters, G., et al. Acoustic force spectroscopy. Nature Methods. 12 (1), 47-50 (2015).
- Augustsson, P., Karlsen, J. T., Su, H. -. W., Bruus, H., Voldman, J. Iso-acoustic focusing of cells for size-insensitive acousto-mechanical phenotyping. Nature Communications. 7 (1), 11556 (2016).
- Cushing, K. W., et al. Ultrasound characterization of microbead and cell suspensions by speed of sound measurements of neutrally buoyant samples. Analytical Chemistry. 89 (17), 8917-8923 (2017).
- Riaud, A., Wang, W., Thai, A. L. P., Taly, V. Mechanical characterization of cells and microspheres sorted by acoustophoresis with in-line resistive pulse sensing. Physical Review Applied. 13 (3), 034058 (2020).
- Petersson, F., Aberg, L., Swärd-Nilsson, A. -. M., Free Laurell, T. flow acoustophoresis: microfluidic-based mode of particle and cell separation. Analytical Chemistry. 79 (14), 5117-5123 (2007).
- Griwatz, C., Brandt, B., Assmann, G., Zänker, K. S. An immunological enrichment method for epithelial cells from peripheral blood. Journal of Immunological Methods. 183 (2), 251-265 (1995).
- Katholnig, K., Poglitsch, M., Hengstschläger, M., Weichhart, T. Lysis gradient centrifugation: a flexible method for the isolation of nuclei from primary cells. Methods in Molecular Biology. 1228, 15-23 (2015).
- Fu, Q., Zhang, Y., Huang, T., Liang, Y., Liu, Y. Measurement of cell compressibility changes during epithelial-mesenchymal transition based on acoustofluidic microdevice. Biomicrofluidics. 15 (6), 064101 (2021).
- Zhang, Y., et al. Ionizing radiation-induced DNA damage responses affect cell compressibility. Biochemical and Biophysical Research Communications. 603, 116-122 (2022).
- Hao, Y., et al. Mechanical properties of single cells: Measurement methods and applications. Biotechnology Advances. 45, 107648 (2020).
- Yousafzai, M., et al. Effect of neighboring cells on cell stiffness measured by optical tweezers indentation. Journal of Biomedical Optics. 21 (5), 057004 (2016).
- Wei, M. -. T., et al. A comparative study of living cell micromechanical properties by oscillatory optical tweezers. Optics Express. 16 (12), 8594-8603 (2008).
- Khan, Z. S., Vanapalli, S. A. Probing the mechanical properties of brain cancer cells using a microfluidic cell squeezer device. Biomicrofluidics. 7 (1), 011806 (2013).
- Hirawa, S., Masudo, T., Okada, T. Acoustic recognition of counterions in ion-exchange resins. Analytical Chemistry. 79 (7), 3003-3007 (2007).
- Joosse, S. A., Gorges, T. M., Biology Pantel, K. detection, and clinical implications of circulating tumor cells. EMBO Molecular Medicine. 7 (1), 1-11 (2015).
- Martin, O. A., Anderson, R. L., Narayan, K., MacManus, M. P. Does the mobilization of circulating tumour cells during cancer therapy cause metastasis. Nature Reviews Clinical Oncology. 14 (1), 32-44 (2017).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados