Method Article
Análisis fenotípico semiautomatizado de cultivos celulares esferoides funcionales en 3D
En este artículo
Resumen
Presentamos un protocolo para el cultivo de esferoides de alta reproducibilidad y su caracterización fenotípica mediante captura de imágenes y proteómica.
Resumen
Presentamos un protocolo que describe las propiedades y ventajas del uso de una incubadora de clinostato independiente para el cultivo, tratamiento y monitorización de cultivos celulares en 3D. El clinostato imita un entorno en el que las células pueden ensamblarse como esferoides altamente reproducibles con bajas fuerzas de cizallamiento y difusión activa de nutrientes. Demostramos que tanto los hepatocitos cancerosos como los no cancerosos (líneas celulares HepG2/C3A y THLE-3) requieren 3 semanas de crecimiento antes de alcanzar funcionalidades comparables a las de las células hepáticas. Este protocolo destaca la conveniencia de utilizar incubadoras para células 3D con cámaras que monitorean el crecimiento celular, ya que se pueden tomar instantáneas para contar y medir los esferoides durante el tratamiento. Describimos la comparación de las líneas celulares THLE-3 y HepG2/C3A, mostrando cómo se pueden cultivar líneas celulares no cancerosas, así como células cancerosas inmortalizadas. Demostramos e ilustramos cómo se pueden llevar a cabo experimentos de proteómica a partir de unos pocos esferoides, que se pueden recolectar sin perturbar la señalización celular, es decir, sin necesidad de tripsinización. Demostramos que el análisis proteómico se puede utilizar para monitorear el fenotipo hepático típico del metabolismo de la cadena respiratoria y la producción de proteínas involucradas en la desintoxicación de metales y describimos un sistema semiautomatizado para contar y medir el área del esferoide. En conjunto, el protocolo presenta una caja de herramientas que comprende una caracterización fenotípica a través de la captura de imágenes y una línea de proteómica para experimentar en modelos de cultivo celular en 3D.
Introducción
Se ha demostrado que los cultivos celulares in vitro son necesarios e invaluables para establecer conocimientos fundamentales en biología. Gran parte del conocimiento científico en biología y específicamente en cáncer proviene del sistema de cultivo 2D, es decir, células que crecen en una monocapa. Aunque el cultivo 2D ha sido el sistema de cultivo celular dominante, tiene muchas desventajas que pueden sofocar un mayor progreso biológico. Por ejemplo, los cultivos 2D carecen de interacciones célula-célula importantes para la señalizacióny proliferación celular. Hasta la fecha, se ha demostrado que los sistemas de cultivo 3D modelan mejor la diferenciación, la respuesta a los fármacos, la invasión tumoral y la biología 2,3,4,5. El modelado 3D de cánceres malignos es especialmente vital debido al aumento del envejecimiento de la población y la mortalidad por cáncer. El carcinoma hepatocelular (CHC) es una de las principales causas de mortalidad relacionada con el cáncer en todo el mundo y con frecuencia tiene un pronóstico pésimo6. Se sabe que el CHC tiene una baja tasa de curación, una respuesta deficiente a los medicamentos y una alta tasa de recurrencia 6,7,8. Se han desarrollado varios modelos 3D para hígado normal y CHC que imitan la fisiología del tejido hepático normal y maligno in vivo 9,10.
Algunos de los sistemas 3D actuales incluyen recubrimientos de líquidos, biorreactores, hidrogel, andamios y estructuras impresas en 3D. Los esferoides generados en los biorreactores proporcionan específicamente ventajas únicas porque imitan la distribución tumoral de la exposición a nutrientes, el intercambio de gases y la proliferación/quiescencia celular11. Los biorreactores son especialmente adecuados para modelos de cáncer debido a su facilidad de uso, gran escalabilidad, difusión de nutrientes y accesibilidad11. Además, los biorreactores pueden permitir experimentos de alto rendimiento, mayor reproducibilidad y disminución del error humano. El biorreactor utilizado en este estudio es único porque simula un sistema de gravedad reducida, lo que minimiza las fuerzas de cizallamiento disruptivas aplicadas en los biorreactores típicos, lo que permite una mejor reproducibilidad12. La gravedad omnidireccional y la reducción de las fuerzas de cizallamiento permiten que las células se desarrollen de una manera más fisiológica. Como evidencia, las células HepG2/C3A cultivadas bajo esta metodología desarrollan orgánulos esféricos que producen in vivo niveles de ATP, adenilato quinasa, urea y colesterol13,14. Además, los tratamientos farmacológicos en este sistema 3D son más avanzados y automatizados en comparación con los cultivos 2D. En los cultivos 2D, los tratamientos farmacológicos a menudo deben tener un curso de tiempo corto debido a la necesidad de tripsinizar y mantener la salud celular. Sin embargo, en nuestro caso, podemos realizar tratamientos farmacológicos a largo plazo de esferoides sin necesidad de alterar la estructura y la fisiología de las células. Por lo tanto, es necesario pasar de los cultivos 2D a los 3D para modelar mejor los fenómenos biológicos in vivo y seguir desarrollando la ciencia.
En este artículo se presenta una metodología para el cultivo de esferoides de alta reproducibilidad (Figura 1 y Figura 2) y se muestra un sistema semiautomatizado para caracterizar fenotípicamente estructuras 3D (Figura 3). A nivel de imagen, proporcionamos información sobre el conteo y la medición del área de los esferoides (Figura 3). Mediante el uso de métodos de espectrometría de masas, mostramos cómo se puede utilizar la proteómica para evaluar funciones biológicas específicas (Figura 4). Al recopilar y analizar estos datos, esperamos mejorar la comprensión de la biología detrás de los sistemas de cultivo celular en 3D.
Protocolo
1. Tampones y reactivos
- Medios de crecimiento celular para células HepG2/C3A: Prepare el medio de Eagle modificado de Dulbecco (DMEM, 4,5 g/L de glucosa) que contiene un 10 % de suero fetal bovino (FBS), aminoácidos no esenciales (1 % v/v), L-glutamina (1 % v/v) y penicilina/estreptomicina (0,5 % v/v). Almacene el sustrato a 4 °C.
- Medios de crecimiento celular para células THLE-3: Prepare un medio de crecimiento de células epiteliales bronquiales [BEGM] que contenga sus suplementos (extracto de hipófisis bovina [BPE], hidrocortisona, factor de crecimiento epidérmico humano [hEGF], epinefrina, transferrina, insulina, ácido retinoico, triyodotironina, gentamicina sulfato-anfotericina [GA]), así como 10% de FBS, 5 ng/mL de hEGF y 70 ng/mL de fosfoetanolamina.
- 500 mM de DL-Ditiotreitol (DTT): Para preparar 10 mL, resuspender 0,771 g de DTT en 10 mL de agua de grado HPLC. Almacenar 100 μl de alícuotas a -20 °C.
- 200 mM de yodoacetamida: Para preparar 1 mL, resuspender 36 mg de yodoacetamida en 1 mL de bicarbonato amónico 50 mM. No almacene la yodoacetamida resuspendida.
- Dodecil sulfato de sodio (SDS) al 5%: Para preparar 50 mL, resuspender 2,5 g de SDS en 50 mL de bicarbonato amónico 50 mM. Conservar a 4 °C.
- Ácido fosfórico al 12%: Para preparar 100 ml, diluya 14,1 ml de ácido fosfórico al 85% en 85,9 ml de agua de grado HPLC. Conservar en una botella de vidrio a temperatura ambiente (RT).
- Tampón aglutinante: Para preparar 1 L, agregue 900 mL de metanol concentrado a 100 mL de bicarbonato amónico 10 mM o TEAB.
- Solución de ácido trifluoroacético (TFA) al 0,1 %: Agregue 1 ml de TFA concentrado a 999 ml de agua de grado HPLC. Conservar a 4 °C.
- Solución de acetonitrilo al 60 %/TFA al 0,1 %: Agregue 600 ml de acetonitrilo de grado HPLC (60 % v/v) a 399 ml de agua de grado HPLC. A continuación, agregue 1 ml de TFA concentrado a esta solución, luego guárdelo a 4 °C.
- Fase A móvil (MPA) - 0,1 % de ácido fórmico: Agregue 1 ml de ácido fórmico concentrado a 999 ml de agua de grado HPLC y mezcle bien.
- Fase B móvil (MPB) - 80 % de acetonitrilo de grado HPLC + 0,1 % de ácido fórmico: Agregue 800 ml de acetonitrilo de grado HPLC a 199 ml de agua de grado HPLC. A continuación, agregue 1 ml de ácido fórmico concentrado a esta solución y mezcle.
2. Preparación de esferoides
NOTA: La Figura 1A representa los pasos iniciales para preparar y cultivar esferoides 3D a partir de líneas celulares.
- Descongele las células HepG2/C3A y THLE-3 congeladas y cultive como una monocapa utilizando medios de crecimiento estándar en un matraz o placa de cultivo de tejidos hasta que alcancen aproximadamente el 80% de confluencia.
NOTA: Cuando las células alcancen la confluencia, verifique la morfología celular general y los patrones de crecimiento con un microscopio. No se recomienda utilizar celdas con un número de paso alto. - Lave las celdas dos veces con la solución salina equilibrada de Hank (HBSS, use 5 ml para un matraz T75 cm2 o un plato de 10 cm).
- Añadir 5 mL de tripsina-EDTA al 0,05% diluido en HBSS (dilución 1:2) e incubar durante 5 min a 37 °C con CO2 al 5%. Use un microscopio para evaluar el desprendimiento celular y agregue 3 ml de FBS o medios de crecimiento (que contengan 10% de FBS) para neutralizar la reacción de tripsina.
- Transfiera la suspensión celular a un tubo de 15 ml. Girar a 270 x g a RT durante 5 min.
- Aspirar el sobrenadante y resuspender las células en 5 ml de medio de crecimiento completo.
NOTA: Si hay demasiadas células, diluya la suspensión celular antes de contar. - Contar el número de células y diluir la suspensión celular en medios de crecimiento completos para obtener 1 x 106 en un volumen máximo de 1,5 mL.
- Equilibre una placa inferior redonda de 24 pocillos de fijación ultra baja que contiene micropocillos.
- Lave los pocillos con 0,5 ml de sustrato.
- Centrifugar la placa a 3.000 x g durante 5 min (esto elimina las burbujas de aire de la superficie de los pocillos).
- Transfiera la suspensión celular (preparada en el paso 1.4) a la placa y centrifugue durante 3 minutos a 120 x g.
NOTA: El volumen de suspensión celular puede variar según el recuento de células, pero es importante limitarlo a 1,5 ml. - Incubar la placa durante 24 h a 37 °C con 5% de CO2 para iniciar la formación de esferoides.
3. Cultivo de esferoides en biorreactores (Figura 2)
NOTA: Para preservar la estructura de los esferoides, utilice puntas de diámetro ancho cuando manipule esferas 3D.
- Equilibre el biorreactor llenando la cámara de humedad con 25 mL de agua estéril y la cámara celular con 9 mL de medio de crecimiento 24 h antes de transferir los esferoides a ella (Figura 2A). Asegúrese de usar una jeringa de 10 ml acoplada a una aguja larga cuando llene la humedad y las cámaras celulares.
- Incubar el biorreactor, girando (15 rpm) en la incubadora 3D (Figura 2D,F), durante 24 h a 37 °C con 5% de CO2.
- Con puntas de 1 ml de diámetro ancho, pipetee suavemente hacia arriba y hacia abajo para separar los esferoides de la placa de fijación ultrabaja y transfiera los esferoides a una placa de cultivo de tejidos.
- Lave la placa de fijación ultra baja con 0,5 ml de medio de crecimiento precalentado para capturar los esferoides sobrantes y transferirlos al mismo plato del paso 3.3.
- Evalúe el tamaño, la compacidad y la redondez de los esferoides utilizando un microscopio óptico (aumento de 4x) y seleccione los que estén suficientemente formados.
NOTA: Las células están suficientemente formadas cuando son compactas y no se deshacen durante la manipulación. También es importante que los esferoides sean una sola unidad y no estén agrupados. Se prefiere un tamaño de esferoide entre 100 y 200 μm cuando se transfiere al biorreactor. - Transfiera los esferoides al biorreactor equilibrado lleno de 5 ml de medios de crecimiento frescos. Después de transferir los esferoides, llene completamente el biorreactor con medios de crecimiento nuevos y asegúrese de evitar agregar burbujas al biorreactor cuando lo llene con medios.
- Coloque el biorreactor en la incubadora 3D y ajuste la velocidad de rotación con la unidad de control (Figura 2C). Para esferoides HepG2/C3A, ajuste la velocidad de rotación a 10-11 rpm; para esferoides THLE-3, ajústelo a 11-12 rpm.
NOTA: La velocidad de rotación se establece correctamente cuando los esferoides se distribuyen uniformemente en el centro del biorreactor y no tocan sus paredes. La Figura 2E muestra un biorreactor rotativo. - Cambie los medios de crecimiento cada 2-3 días retirando 10 ml de medios viejos y reemplazándolos con 10 ml de medios nuevos; asegúrese de no eliminar los esferoides al intercambiar medios (Figura 2B).
NOTA: Se recomienda tener una rutina de intercambio de medios (por ejemplo, 48 h/48 h/72 h) y mantener un registro detallado. - Ajuste la velocidad de rotación cada vez que se cambie el soporte. Aumente la velocidad a medida que los esferoides crecen en tamaño y número.
- Después de 15 días en cultivo, dividió los esferoides en dos nuevos biorreactores.
NOTA: Dependiendo de la línea celular y la tasa de crecimiento, el tiempo puede variar y debe optimizarse individualmente. - Después de 20 días, los esferoides están listos para su recolección.
4. Captura de imágenes y conteo de esferoides
NOTA: La canalización simplificada para el recuento de esferoides se muestra en la Figura 3A. Para el recuento de esferoides y la determinación del área (sección 5), es fundamental evaluar la compacidad de las estructuras 3D. Esto contribuirá a un contraste de color más mejorado, lo cual es necesario para que el método sea preciso.
- Abra la aplicación 3D instalada en la tableta.
- Seleccione el biorreactor y tome una instantánea.
NOTA: Alternativamente, se puede tomar una fotografía. Se deben tener en cuenta los siguientes parámetros.- Coloque un fondo negro detrás del biorreactor (se proporciona un soporte negro en la caja del biorreactor).
- Asegúrese de que no haya reflejo de luz en la cámara de la celda.
- Tome la foto lo más cerca posible del biorreactor.
- Abra una imagen en FIJI (ImageJ).
- Prepare la imagen para el análisis:
- Seleccione la herramienta Óvalo . Dibuja un círculo alrededor del enchufe.
- Haga clic en Eliminar en el teclado para que todo lo que esté dentro del círculo sea negro. Dibuja un círculo alrededor de la cámara de la celda. Asegúrese de que todos los esferoides estén dentro del círculo.
- Ejecute la macro para contar esferoides:
- Haga clic en Plugins > Macro > Record. Copie el texto de la macro del archivo complementario 1 en la grabadora.
- Presione Crear y se abrirá una nueva ventana. Pulse Ejecutar para analizar la imagen abierta.
- Se abrirá una nueva ventana con los resultados (recuento, área total, tamaño promedio y área porcentual).
- Cierre la imagen y repita los pasos 4.4 y 4.5 para cada imagen para la que se necesite el recuento de esferoides. Para un procesamiento más fácil y rápido, guarde la macro y ejecútela haciendo clic en Complementos > Macro > Ejecutar y seleccione la macro guardada.
5. Determinación planimétrica del área del esferoide
NOTA: La tubería simplificada para determinar el área del esferoide se muestra en la Figura 3B.
- Tome imágenes como se describe en el paso 3.5.
- Antes de comenzar, establezca una escala global para medir el área de los esferoides.
- Abra en FIJI una imagen con una barra de escala con el aumento deseado. Seleccione la herramienta de línea. Dibuja sobre la barra de escala (la línea debe ser tan larga como la barra de escala).
- Abra Analizar > Establecer escala. Escriba la distancia conocida (la longitud de la línea se ingresa automáticamente en la Distancia en píxeles. Asegúrese de marcar Global.
- Presione OK. Ahora se establece la escala para la imagen que se analizará.
- Para iniciar la determinación planimétrica, ejecute la macro (Archivo complementario 2).
- Haga clic en Procesar > Lote > Macro. Elija la carpeta que contiene las imágenes que se van a analizar. Esta carpeta será la entrada.
- Elija la carpeta creada en el paso 5.3.1 como Salida. Presione Proceso.
- Como control de calidad, evalúe si el área del esferoide medida corresponde a la imagen original.
NOTA: Macro excluye los esferoides en el borde donde solo se puede medir una parte del esferoide.
6. Colección de esferoides
NOTA: Se recomienda encarecidamente que los esferoides se recojan utilizando puntas de diámetro ancho para preservar su estructura 3D. La recolección se puede realizar utilizando el tapón en la parte frontal del biorreactor (Figura 2A).
El tamaño de los esferoides en el momento de la recolección puede variar según la línea celular, el número inicial de células y el proceso de división (días en cultivo, número de esferoides por biorreactor y proporción de división).
- Para recolectar esferoides, extraiga 5 ml de medio del biorreactor a través del puerto superior con una jeringa acoplada a una aguja larga. Asegúrese de dejar que los esferoides se hundan en la parte inferior central del biorreactor (cerca del puerto inferior).
- Abra el puerto frontal y recoja los esferoides con una punta de 1 ml de diámetro ancho. Coloque los esferoides en tubos de microcentrífuga. Centrifugar los esferoides a 500 x g durante 5 min y desechar los medios.
- Lave los esferoides con 200 μL de HBSS para eliminar el FBS. Centrifugar a 500 x g durante 5 min y desechar el sobrenadante.
- Congele rápidamente el gránulo esferoide con nitrógeno líquido y guárdelo a -80 °C hasta su procesamiento.
7. Viabilidad de los esferoides
NOTA: La viabilidad de los esferoides se determinó midiendo la actividad de la adenilato quinasa (AK) liberada por las células dañadas (Figura 4A). Debido al gradiente de difusión, la medición de AK es eficiente cuando los esferoides tienen un diámetro inferior a 900 μm12. Si los esferoides se hacen más grandes, o si hay alguna duda con respecto a la medición de viabilidad, se puede realizar un ensayo de ATP15.
- Recoja el sobrenadante esferoide y transfiera 20 μL a una placa de paredes blancas de 96 pocillos (fondo plano transparente). Agregue 100 μL de reactivo de detección de adenilato quinasa a cada pocillo y homogeneice pipeteando suavemente hacia arriba y hacia abajo.
- Eliminar las burbujas por centrifugación en el vacío de velocidad durante 2 min en RT. Incubar las placas durante 20 min en RT.
- Coloque la placa en el luminómetro (lector de placas, modo de luminiscencia) e inicie el programa. Configure los parámetros de acuerdo con las instrucciones del fabricante del kit.
NOTA: En los ensayos de viabilidad bioluminiscente, la salida de luz del luminómetro directo (comúnmente RLU) se puede usar para calcular la respuesta celular. Como la adenilato quinasa solo se filtrará de las células cuya integridad celular se ha visto comprometida, es posible lograr un control total de la adenilato quinasa mediante el uso de un reactivo de lisis.
8. Extracción de proteínas
NOTA: La Figura 1B representa el flujo de trabajo para el procesamiento de esferoides y la extracción de proteínas.
- Recoja esferoides (sección 6) en el día 36. Resuspender los esferoides en 25 μL de SDS al 5% para lisar las células.
- Después de agregar un 5% de SDS, homogeneice el gránulo pipeteando hacia arriba y hacia abajo. En algunos casos, cuando sea difícil lisar los esferoides, use un mortero pequeño.
- Incubar las muestras con 20 mM de TDT durante 1 h para reducir las proteínas. Mezclar pipeteando hacia arriba y hacia abajo.
- Incubar muestras con 40 mM de yodoacetamida durante 30 min protegidas de la luz a proteínas alquiladas. Mezclar pipeteando hacia arriba y hacia abajo.
- Añadir una solución de ácido fosfórico al 12% (10x) a las muestras a una concentración final del 1,2%.
- Diluir las muestras en seis volúmenes de tampón aglutinante y mezclar suavemente.
- Cargue la muestra en una placa de filtro S-Trap y centrifuga a 500 x g durante 30 s.
NOTA: Si el volumen total de la muestra en el paso 8.4 excede la capacidad del volumen de la columna, cargue las muestras en lotes y repita el paso 8.6 hasta que todo pase a través de la columna. - Lavar las muestras dos veces con 150 μL de tampón aglutinante. Después de cada lavado, centrifugar a 500 x g durante 30 s y desechar el flujo.
- Incubar las muestras con 1 μg de tripsina de grado secuencial diluido en bicarbonato amónico 50 mM durante la noche a 37 °C.
NOTA: Se recomienda al menos 20 μg de proteína como material de partida para el análisis completo del proteoma. Para ello, 1 μg de tripsina es la cantidad ideal para una digestión eficiente. Elimine las burbujas entre el búfer y el lecho de la columna. - Eluir péptidos con 40 μL de bicarbonato amónico de 50 mM y agruparlos en el mismo tubo de recogida.
- Girar a 500 x g durante 30 s. Eluir péptidos con 40 μL de AGT al 0,1% y agruparlos en el mismo tubo de recolección.
- Girar a 500 x g durante 30 s. Eluir los péptidos con 40 μL de acetonitrilo al 60 % y TFA al 0,1 % y agruparlos en el mismo tubo de recogida.
- Girar a 500 x g durante 30 s. Vaporizar los eluidos agrupados en el vacío rápido y almacenar las muestras a -80 °C hasta su procesamiento.
9. Limpieza de muestras
NOTA: Antes de proceder al análisis proteómico, es necesario eliminar la sal presente en las muestras. Las sales pueden interferir con el análisis de cromatografía líquida de alta resolución y espectrometría de masas (HPLC-MS) a medida que se ionizan durante la electropulverización, suprimiendo la señal de los péptidos. La configuración para la desalinización utilizada en este estudio fue demostrada previamente por Joseph-Chowdhury y sus colegas16.
- Antes de comenzar, asegúrese de que haya una placa de recolección limpia de 96 pocillos para recolectar el flujo durante los siguientes pasos. Mezcle la resina C18 (50 mg/ml en acetonitrilo al 100%) en una placa de agitación magnética.
- Agregue 70 μL de la suspensión de resina C18 por pocillo de la placa filtrante de 96 pocillos, hágalo rápidamente para asegurarse de que la resina C18 no se acumule en el fondo de la pipeta.
- Encienda la aspiradora suavemente para evitar salpicaduras. Deseche el flujo recogido en la placa de recolección de 96 pocillos.
- Lavar la resina con 100 μL de AGT al 0,1%. Encienda la aspiradora suavemente para evitar salpicaduras y mezcla de muestras y deseche el flujo.
- Vuelva a suspender cada muestra en 100 μL de AGT al 0,1%. Verifique y asegúrese de que el pH esté entre 2-3.
- Cargue cada muestra en cada pocillo de la placa de filtro. Encienda la aspiradora suavemente para evitar salpicaduras y mezcla de muestras y deseche el flujo.
- Lavar con 100 μL de AGT al 0,1%. Encienda la aspiradora suavemente para evitar salpicaduras y mezclas de muestras. Deseche el flujo a través.
- Reemplace la placa de recolección de 96 pocillos por una nueva placa de 96 pocillos para recolectar las muestras desalinizadas.
- Añadir 60 μL de acetonitrilo/0,1% de TFA por pocillo. Para eluir las muestras de la resina C18, encienda la aspiradora suavemente para evitar salpicaduras.
- Recoja el flujo y séquelo en un vacío rápido a RT. Proceda a LC-MS/MS o almacene la placa a -80 °C hasta que esté lista para procesar las muestras.
10. Análisis proteómico mediante cromatografía líquida acoplada a espectrometría de masas
NOTA: Para generar los datos de este manuscrito, se utilizó un sistema nLC-MS/MS con una configuración de sistema de dos columnas con una columna de trampa C18 de 300 μm de diámetro interior x 0,5 cm y una nanocolumna analítica de 75 μm de diámetro interno x 25 cm de C18-AQ (3 μm) empaquetada internamente.
- Prepare las fases móviles para que se ejecuten en la HPLC:
Fase A móvil (MPA): 0,1% de ácido fórmico en agua de grado HPLC
Fase B móvil (MPB): 0,1% de ácido fórmico en acetonitrilo de grado HPLC - Programe el método HPLC de la siguiente manera: 4%-34% MPB durante 120 min, 34%-90% MPB durante 5 min, isocrático 90% MPB durante 5 min; caudal: 300 nL/min.
- Configure el método de adquisición de MS para realizar la adquisición independiente de datos (DIA) para generar espectros de MS/MS de las señales peptídicas detectadas.
- Vuelva a suspender 1 μg de muestras en 10 μl de AGT al 0,1 % antes de colocar las muestras en el muestreador automático nLC.
- Ejecute el método nLC-MS según lo programado para la adquisición de proteómica canónica:
- Ajuste el escaneo MS completo a 300-1100 m/z en el orbitrap, con una resolución de 120.000 (a 200 m/z) y un objetivo de control automático de ganancia (AGC) de 125.
- Establezca MS/MS en el orbitrap con una ventana de aislamiento secuencial de 50 m/z con un objetivo AGC de 400 y una energía de disociación colisional de mayor energía (HCD) de 30.
11. Análisis de datos
- Importe los archivos de datos sin procesar nLC-MS/MS en un software de detección de picos compatible con la plataforma MS utilizada.
- Seleccione la base de datos adecuada (humano, mouse, etc.).
- Establezca la acetilación N-terminal como modificación variable y la carbamidometilcisteína como modificación fija.
- Especifique tripsina como la enzima digestiva con dos escisiones perdidas permitidas.
- Establezca la tolerancia de masa de acuerdo con el analizador de masas utilizado para la adquisición de espectros.
- Exporte el análisis como una hoja de cálculo y procese los datos según sea necesario.
Resultados
En este protocolo, describimos las propiedades de una innovadora incubadora de células 3D libre de estrés, un sistema diseñado específicamente para el cultivo de esferoides 3D (Figura 2). Optimizamos el protocolo para el cultivo 3D de las líneas celulares THLE-3 y HepG2/C3A. El protocolo descrito aquí es fácil de usar y permite la reproducibilidad y el cultivo rentable de > 100 esferoides por biorreactor. Una vez en el biorreactor, los esferoides se tratan de manera similar a las células mantenidas en cultivo 2D. Las condiciones óptimas de crecimiento se logran intercambiando los medios dos o tres veces por semana (Figura 2B) y ajustando la velocidad de rotación de acuerdo con el crecimiento y el tamaño de los esferoides (Figura 2C). Este sistema, en el que los esferoides 3D se cultivan en biorreactores rotativos (Figura 2E, F), proporciona un entorno de crecimiento óptimo para las estructuras 3D al exponer el esferoide a una cantidad igual y muy baja de fuerza de cizallamiento.
Anteriormente hemos mostrado cómo se pueden utilizar los esferoides para el análisis de la modificación de la cromatina16. Aquí, demostramos en detalle cómo obtener esferoides hepáticos y cómo se pueden realizar experimentos de proteómica para el análisis del proteoma completo (Figura 1). En resumen, el protocolo se inició mediante el uso de células planas THLE-3 o HepG2/C3A hasta que el cultivo alcanzó el 80% de confluencia. Para cultivar células como esferoides, aproximadamente 2.000 células se colocaron en una placa de fijación ultra baja que contenía micropocillos para permitirles autoagregarse, y luego, los esferoides formados se transfirieron a un biorreactor (Figura 1A). A pesar de que son funcionalmente activos después de 3 semanas en cultivo, como se demostró previamente17, mostramos resultados de esferoides recolectados después de 36 días en cultivo para este protocolo. Después de la recolección, los esferoides se centrifugaron y tanto los gránulos como el sobrenadante se almacenaron para su análisis. La viabilidad celular fue evaluada a partir del sobrenadante para la cuantificación de la adenilato quinasa liberada por las células dañadas, como se describió previamente17. Las proteínas celulares se extrajeron del gránulo celular y el proteoma completo se analizó mediante espectrometría de masas de alta resolución (Figura 1B).
Este protocolo también demuestra un método semiautomatizado para el conteo de esferoides (Figura 3A) utilizando el programa de procesamiento de imágenes públicas FIJI (Fiji Is Just ImageJ)18. Para el análisis se debe tomar una imagen de buena calidad del esferoide y se deben considerar algunos parámetros como se menciona en la sección 5. A continuación, después de preparar la imagen para el análisis, se utiliza un script de macros (Archivo suplementario 1) para contar los esferoides. La macro funciona creando primero una carpeta llamada FIJI Spheroids counting, dentro de la carpeta donde se encuentran las imágenes de esferoides. En esta carpeta, se guarda toda la información del análisis; esto incluye una imagen de los esferoides que se contaron, con un número de identificación en cada esferoide. También incluye un archivo de Excel llamado conteo de esferoides. Este archivo contiene el área de píxeles y el número de identificación de cada esferoide que se ha contado. Los datos correspondientes a una imagen analizada se presentan en cada pestaña del archivo. La pestaña está etiquetada de acuerdo con el nombre de la imagen analizada. Dado que el tamaño de los esferoides puede verse afectado por muchos factores, incluido el número de estructuras dentro de un vaso y el tratamiento farmacológico, también es importante controlar su superficie (planimetría). El script de macros que se presenta aquí (Archivo Suplementario 2) funciona midiendo las áreas negras, que corresponden a los esferoides de la imagen (Figura 3B). La salida se recopila en un archivo llamado planimetría.xlsx, que contiene el área medida, el perímetro y el diámetro de cada esferoide. También existe una medida llamada Feret, que se utiliza para calcular el diámetro. Feret es el diámetro más largo posible, mientras que minFeret es el más corto. El diámetro es el promedio de estos dos. Dentro de la carpeta de salida, además del archivo planimetría .xlsx, también hay una imagen de los esferoides que se midieron.
Antes de proceder al análisis del proteoma, se evaluó la viabilidad de los esferoides a lo largo del tiempo de cultivo. Los niveles de AK aumentan hasta el día 17, alcanzando aproximadamente el 7% de la muerte celular, y luego la muerte disminuye a niveles inferiores al 5% (Figura 4A), lo que concuerda con el trabajo publicado previamente17. Este protocolo también muestra el análisis completo del proteoma para monitorizar el fenotipo celular. En primer lugar, se compararon los proteomas de las células planas THLE-3 y HepG2/C3A y los esferoides. Al analizar el primer componente principal (PC1), es evidente que existe una estricta separación de las muestras de esferoides de los cultivos celulares planos, y parece que la correlación del tipo de célula (THLE-3 y HepG2/C3A) no es relevante (Figura 4B). Aunque los esferoides THLE-3 y HepG2/C3A no se agrupan, comparten perfiles similares consistentes con la función hepática. Demostramos en este protocolo el ejemplo de las metalotioneínas, que tienen un papel en la desintoxicación de metales realizada por el hígado. En el análisis proteómico identificamos 2 isoformas sobreexpresadas en esferoides en comparación con células planas (MT1E y MT1X) (Figura 4C). También mostramos el enriquecimiento de Gene Ontology (GO) de ambas líneas celulares cultivadas como esferoides. El proceso metabólico de los hidratos de carbono, que comprende el ciclo del ácido tricarboxílico (ciclo TCA), la cadena de transporte de electrones (respiración celular) y el metabolismo del piruvato, es un término frecuente y está enriquecido en los esferoides HepG2/C3A y THLE-3 (Figura 4D,E). La desintoxicación celular, los ácidos grasos y el metabolismo del colesterol son otras funciones enriquecidas en ambos esferoides. Juntas, se sabe que estas funciones son cruciales para la función hepática.
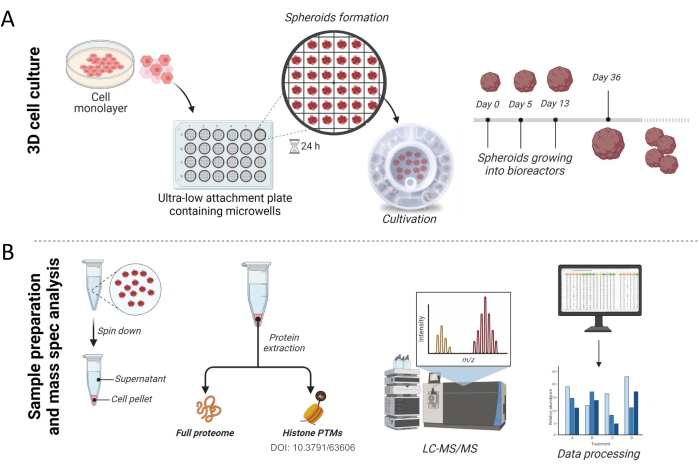
Figura 1: Flujo de trabajo para el cultivo de esferoides y la preparación de muestras . (A) Enfoque experimental de cultivo celular en 3D. Los cultivos celulares planos en la confluencia deseada se tripsinizaron y se sembraron en una placa de 24 pocillos de fijación ultra baja que contenía micropocillos, donde las células se autoensamblan en esferoides. Después de 24 h, los esferoides se transfirieron a un biorreactor y se cultivaron hasta que estuvieron listos para el análisis. (B) Después de la recolección, los esferoides se peletizaron y tanto el gránulo como el sobrenadante de cultivo se almacenaron hasta su procesamiento. Se extrajeron las histonas16y las proteínas no histonas, se digierieron en péptidos y se analizaron mediante espectrometría de masas de alta resolución. Los archivos sin procesar obtenidos de la espectrometría de masas se buscaron en la base de datos humana y los datos se procesaron posteriormente. Haga clic aquí para ver una versión más grande de esta figura.
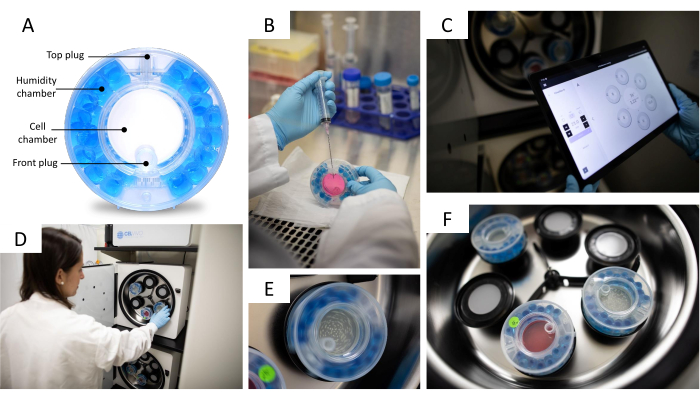
Figura 2: Sistema de cultivo celular 3D . (A) Partes del biorreactor. El biorreactor está compuesto por una cámara de intercambio de gases y humidificación que contiene perlas de agua y una cámara celular que se puede abrir con dos tapones para el intercambio de medios y la recolección de esferoides. (B) Intercambio de medios de biorreactores. El biorreactor se llena con 10 ml de medio de crecimiento utilizando una jeringa con una aguja. (C) La aplicación de control del sistema. La velocidad de rotación, el nivel de CO2, la temperatura, el registro de alarmas y otras funcionalidades se pueden controlar mediante la unidad de control. (D) Colocación del biorreactor en la incubadora 3D. Cada biorreactor tiene un motor asociado que puede hacer girar el biorreactor lentamente. (E) Biorreactor en movimiento con la velocidad (rpm) controlada por una tableta (C). La velocidad (rpm) se ajusta de acuerdo con el tamaño de los esferoides. (F) Biorreactores dentro de la incubadora 3D. La incubadora 3D puede albergar hasta 6 biorreactores controlados individualmente. Foto cortesía de Jason Torres Photography. Haga clic aquí para ver una versión más grande de esta figura.
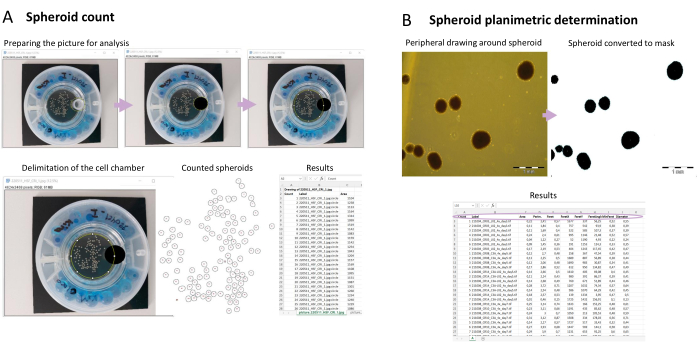
Figura 3: Caracterización fenotípica de esferoides mediante captura de imágenes . (A) Recuento semiautomatizado de esferoides. Después de tomar instantáneas de los esferoides en el biorreactor, la imagen se prepara para su análisis en FIJI. Se cuenta cada esferoide y se proporciona un número de identificación para cada uno de ellos. Se utiliza una macro y los resultados se muestran mostrando el identificador del esferoide contado, la etiqueta (nombre de la imagen que se analizó) y el área (el número de píxeles contados en el esferoide). (B) Determinación planimétrica del área esferoide. Con una macro, se determinan el área, el perímetro y el diámetro de un esferoide específico. Haga clic aquí para ver una versión más grande de esta figura.
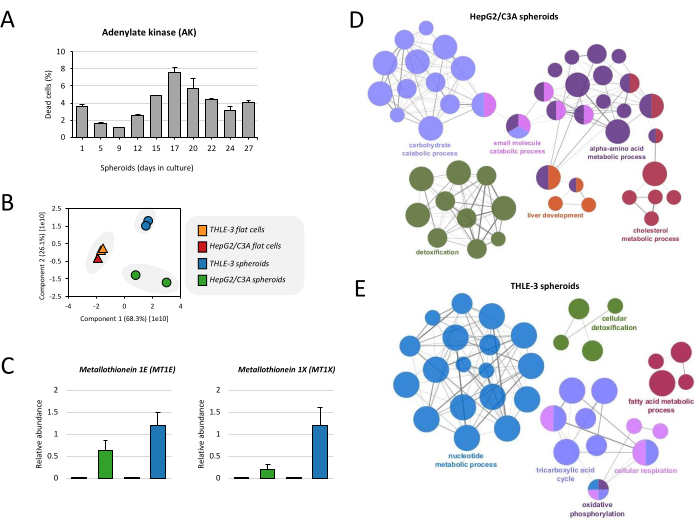
Figura 4: Análisis del proteoma de los esferoides hepáticos. (A) La viabilidad de los esferoides se calculó a partir de la liberación de adenilato quinasa (AK) en el sobrenadante del cultivo. Los resultados son las medias de los puntos de datos duplicados ± DE. (B) Se realizó un análisis de componentes principales (PCA) para comparar el proteoma de las células planas y esferoides THLE-3 y HepG2/C3A. (C) Abundancia relativa de metalotioneínas, que son proteínas expresadas por el hígado humano. Los datos se representan como medios ± SEM. (D) Las redes agrupadas funcionalmente muestran el enriquecimiento de GO para los esferoides HepG2/C3A y (E) los esferoides THLE-3, donde solo se muestra la etiqueta del término más significativo por grupo. La red se construyó utilizando ClueGo19. El tamaño del nodo representa la importancia del enriquecimiento del término. Haga clic aquí para ver una versión más grande de esta figura.
Archivo complementario 1: Script de macros para el conteo de esferoides. Haga clic aquí para descargar este archivo.
Archivo complementario 2: Macro para la determinación planimétrica de esferoides. Haga clic aquí para descargar este archivo.
Discusión
Comprender la biología detrás de las estructuras celulares tridimensionales (3D) es extremadamente importante para un conocimiento más completo de sus funcionalidades. Existe un creciente interés en el uso de modelos 3D para estudiar biología compleja y realizar cribados de toxicidad. Al cultivar células en 3D hay que tener en cuenta muchos factores, incluida la evaluación fenotípica del sistema modelo. Un fenotipo se define como un conjunto de características observables de un organismo específico, como la morfología, el comportamiento, las propiedades fisiológicas y bioquímicas20.
En este protocolo, demostramos cómo se pueden realizar experimentos de proteómica a partir de unos pocos esferoides y se pueden usar para monitorear el fenotipo hepático típico. La espectrometría de masas se ha convertido en un método ampliamente aplicado para la caracterización celular en 3D, permitiendo la investigación de una variedad de cuestiones biológicas 12,16,21,22. Para un análisis completo del proteoma, se recomienda utilizar al menos 20 μg de material de partida proteico, de los cuales se inyecta 1 μg en el espectrómetro de masas. Es importante mencionar que agregar menos muestra podría conducir a la pérdida de sensibilidad, y agregar más empeoraría gradualmente la calidad de la cromatografía y, finalmente, conduciría al bloqueo de la columna. En este estudio, demostramos que los esferoides HepG2/C3A y THLE-3 están enriquecidos con proteínas importantes de la glucólisis y del ciclo del TCA, que son vías hepáticas específicas y son críticas para mantener los niveles de glucosa en sangre y para la producción de energía23,24. En realidad, el análisis por espectrometría de masas no solo proporciona información a nivel de proteína, sino que también permite investigar las modificaciones postraduccionales de proteínas, como lo demostró previamente nuestro grupo16.
Otro aspecto a tener en cuenta en los estudios fenotípicos 3D es el número y tamaño de los esferoides. Además de hacer que los experimentos sean más reproducibles, contar el número de esferoides y determinar su tamaño es esencial para determinar cuándo dividir el cultivo en múltiples biorreactores, ya que el número de estructuras 3D dentro de un recipiente puede afectar el tamaño de los esferoides y los niveles de actividad metabólica. Sin embargo, es importante destacar que el número y el tamaño de los esferoides dependen de la línea celular, el número inicial de células, el proceso de división y el momento de la recolección. Los detalles del cultivo de esferoides HepG2/C3A, como el número de células por esferoides, el contenido de proteínas y el tamaño en función de la edad, fueron proporcionados por Fey, Korzeniowska y Wrzesinski25. Para un análisis preciso y exitoso utilizando el método semiautomatizado descrito aquí, el paso más crítico es una buena imagen de los esferoides. Para simplificar, la foto se puede tomar con un teléfono o tableta, pero su resolución debe mantenerse lo más alta posible. Como las imágenes se adquieren rápidamente, permiten realizar experimentos de cribado a gran escala para visualizar características fenotípicas específicas o investigar las respuestas al tratamiento farmacológico. Por lo tanto, debido al creciente número de ensayos basados en células, en los últimos 10 años se han desarrollado varios programas informáticos de código abierto para el análisis de imágenes26. En este protocolo, describimos un sistema semiautomatizado que utiliza el software FIJI18 para contar y medir el tamaño de los esferoides. Presentamos scripts (comandos de programación simples) para definir una secuencia de operaciones algorítmicas que se pueden aplicar a una colección de imágenes, haciendo que el análisis sea un proceso fácil y rápido. Sin embargo, dependiendo de la característica del esferoide, se debe emplear una medición manual. Por ejemplo, si los esferoides son demasiado translúcidos, el script FIJI será impreciso. Por cierto, uno de los criterios más importantes para que este método funcione es la compacidad de los esferoides. Esta característica contribuirá a un contraste de color más mejorado entre los esferoides y el fondo, lo cual es necesario para que el método sea preciso.
En resumen, además de presentar una metodología para el cultivo de esferoides de alta reproducción, también se describió un sistema semiautomatizado acoplado a la caracterización fenotípica mediante captura de imágenes y proteómica. Esperamos que esta caja de herramientas para analizar células 3D sea más robusta con un software de análisis de imágenes totalmente automatizado y espectrómetros de masas de próxima generación.
Divulgaciones
Helle Sedighi Frandsen trabaja como científica investigadora en CelVivo ApS, el productor del sistema ClinoStar. Karoline Mikkelsen es una estudiante de doctorado industrial que trabaja en CelVivo ApS y realiza sus estudios de doctorado en SDU, Odense, Dinamarca. Todos los demás autores no tienen intereses financieros contrapuestos.
Agradecimientos
El laboratorio de Sidoli agradece a la Fundación para la Investigación de la Leucemia (Beca de Investigación para Nuevos Investigadores Hollis Brownstein), AFAR (premio Sagol Network GerOmics), Deerfield (premio Xseed), Relay Therapeutics, Merck y la Oficina del Director de los NIH (1S10OD030286-01).
Materiales
| Name | Company | Catalog Number | Comments |
| 1.5 mL microcentrifuge tubes | Bio-Rad | 2239480 | |
| 10 mL syringe | Fisher Scientific | 1481754 | Luer lock tip, graduated to 12 mL |
| 1000 µL wide bore pipet tips | Fisher Scientific | 14222703 | |
| 200 µL wide bore pipet tips | Fisher Scientific | 14222730 | |
| 96-well Orochem filter plate | Orochem | OF1100 | |
| 96-well skirted plate | Axygen | PCR-96-FS-C | |
| 96-well vacuum manifold | Millipore | MAVM0960R | |
| Ammonium bicarbonate | Sigma | A6141-25G | |
| Bronchial Epithelial Cell Growth Medium (BEGM) | Lonza | CC-3170 | |
| Cell culture grade water | Corning | 25-055-CV | |
| ClinoReactor | CelVivo | N/A | Bioreactor for 3D cell culture |
| ClinoStar incubator | CelVivo | N/A | CO2 incubator for 3D cell culture |
| DTT | Sigma | D0632-5G | |
| Dulbecco's Modified Eagle's Medium (DMEM) | Fisher Scientific | MT17205CV | |
| Elplasia 24-well round bottom ultra-low attachment plate containing microwells | Corning | 4441 | |
| Fetal Bovine Serum | Fisher Scientific | MT35010CV | |
| Formic acid | Thermo | 28905 | |
| Hank's Balanced Salt Solution (HBSS) | Fisher Scientific | MT21022CV | |
| hEGF | Corning | 354052 | |
| HERAcell vios 160i | Thermo | 51033557 | CO2 incubator for 2D cell culture |
| HPLC grade acetonitrile | Fisher Scientific | A955-4 | |
| HPLC grade methanol | Fisher Scientific | A452-1 | |
| HPLC grade water | Fisher Scientific | W5-4 | |
| Iodoacetamide | Sigma | I1149-5G | |
| L-glutamine | Fisher Scientific | MT25015CI | |
| Non-essential amino acids | Fisher Scientific | MT25025CI | |
| Oasis HLB Resin 30 µm | Waters | 186007549 | |
| Orbitrap Fusion Lumos Tribrid mass spectrometer | Thermo | IQLAAEGAAPFADBMBHQ | High resolution mass spectrometer |
| PAULA microscope | Leica | ||
| Penicillin-Streptomycin | Fisher Scientific | MT3002CI | |
| PerkinElmer Victor X2 multilabel microplate reader | PerkinElmer | ||
| pH paper | Hydrion | 93 | |
| Phosphoetanolamine | Sigma | P0503 | |
| Phosphoric acid | Fisher Scientific | A260-500 | |
| Pipette gun | Eppendorf | Z666467 (Milipore Sigma) | |
| Refrigerated centrifuge | Thermo | 75-217-420 | |
| Reprosil-Pur resin | MSWIL | R13.AQ.003 | 120 Å pore size, C18-AQ phase, 3 μM bead size |
| SDS | Bio-Rad | 1610301 | |
| Sequencing grade modified trypsin | Promega | V511A | |
| SpeedVac vacuum concentrator (96-well plates) | Thermo | 15308325 | Savant SPD1010 |
| Sterile hood | Thermo | 1375 | |
| Sterile serological pipettes | Fisher Scientific | 1367549 | |
| S-trap | Protifi | C02-micro-80 | |
| Syringe needle (18 G) | Fisher Scientific | 14817100 | 3" length, 0.05" diameter |
| Trifluoroacetic acid (TFA) | Thermo | 28904 | |
| Trypsin-EDTA | Gibco | 25300-054 | |
| Vortex | Sigma | Z258415 | |
| Water bath | Fisher Scientific | FSGPD10 |
Referencias
- Ravi, M., Paramesh, V., Kaviya, S. R., Anuradha, E., Solomon, F. D. 3D cell culture systems: advantages and applications. Journal of Cellular Physiology. 230 (1), 16-26 (2015).
- Nirmalanandhan, V. S., Duren, A., Hendricks, P., Vielhauer, G., Sittampalam, G. S. Activity of anticancer agents in a three-dimensional cell culture model. Assay and Drug Development Technologies. 8 (5), 581-590 (2010).
- Erickson, I. E., Huang, A. H., Chung, C., Li, R. T., Burdick, J. A., Mauck, R. L. Differential maturation and structure-function relationships in mesenchymal stem cell- and chondrocyte-seeded hydrogels. Tissue Engineering Part A. 15 (5), 1041-1052 (2009).
- Vinci, M., et al. Advances in establishment and analysis of three-dimensional tumor spheroid-based functional assays for target validation and drug evaluation. BMC Biology. 10 (29), (2012).
- Liu, J., Abate, W., Xu, J., Corry, D., Kaul, B., Jackson, S. K. Three-dimensional spheroid cultures of A549 and HepG2 cells exhibit different lipopolysaccharide (LPS) receptor expression and LPS-induced cytokine response compared with monolayer cultures. Innate Immunity. 17 (3), 245-255 (2011).
- Khafaga, A. F., Mousa, S. A., Aleya, L., Abdel-Daim, M. M. Three-dimensional (3D) cell culture: a valuable step in advancing treatments for human hepatocellular carcinoma. Cancer Cell International. 22 (1), 243 (2022).
- Llovet, J. M., Burroughs, A., Bruix, J. Hepatocellular carcinoma. The Lancet. 362 (9399), 1907-1917 (2003).
- Sia, D., Llovet, J. M. Liver cancer: Translating '-omics' results into precision medicine for hepatocellular carcinoma. Nature Reviews Gastroenterology & Hepatology. 14 (10), 571-572 (2017).
- Tang, J., et al. A three-dimensional cell biology model of human hepatocellular carcinoma in vitro. Tumour Biology. 32 (3), 469-479 (2011).
- van Zijl, F., Mikulits, W. Hepatospheres: Three dimensional cell cultures resemble physiological conditions of the liver. World Journal of Hepatology. 2 (1), 1-7 (2010).
- Chaicharoenaudomrung, N., Kunhorm, P., Noisa, P. Three-dimensional cell culture systems as an in vitro platform for cancer and stem cell modeling. World Journal of Stem Cells. 11 (12), 1065-1083 (2019).
- Wrzesinski, K., Fey, S. J. Metabolic reprogramming and the recovery of physiological functionality in 3D cultures in micro-bioreactors. Bioengineering(Basel, Switzerland). 5 (1), 22 (2018).
- Breslin, S., O'Driscoll, L. Three-dimensional cell culture: The missing link in drug discovery. Drug Discovery Today. 18 (5-6), 240-249 (2013).
- Kapalczynska, M., et al. 2D and 3D cell cultures - a comparison of different types of cancer cell cultures. Archives of Medical Science: AMS. 14 (4), 910-919 (2018).
- Wrzesinski, K., Frandsen, H. S., Calitz, C., Gouws, C., Korzeniowska, B., Fey, S. J. Clinostat 3D cell culture: Protocols for the preparation and functional analysis of highly reproducible, large, uniform spheroids and organoids. Methods in Molecular Biology. 2273, 17-62 (2021).
- Joseph-Chowdhury, J. N., et al. Global level quantification of histone post-translational modifications in a 3D cell culture model of hepatic tissue. Journal of Visualized Experiments: JoVE. 183, 63606 (2022).
- Wrzesinski, K., Fey, S. J. After trypsinisation, 3D spheroids of C3A hepatocytes need 18 days to re-establish similar levels of key physiological functions to those seen in the liver. Toxicology Research. 2, 123-135 (2013).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Bindea, G., et al. ClueGO: a Cytoscape plug-in to decipher functionally grouped gene ontology and pathway annotation networks. Bioinformatics. 25 (8), 1091-1093 (2009).
- Houle, D., Govindaraju, D. R., Omholt, S. Phenomics: The next challenge. Nature Reviews. Genetics. 11 (12), 855-866 (2010).
- Gonneaud, A., Asselin, C., Boudreau, F., Boisvert, F. M. Phenotypic analysis of organoids by proteomics. Proteomics. 17 (20), (2017).
- Avelino, T. M., et al. Mass spectrometry-based proteomics of 3D cell culture: A useful tool to validate culture of spheroids and organoids. SLAS Discovery. 27 (3), 167-174 (2022).
- Chiang, J., McManus, L. M., MitchellIn, R. N. . Liver physiology: Metabolism and Detoxification. Pathobiology of Human Disease. , 1770-1782 (2014).
- Begriche, K., Massart, J., Robin, M. A., Borgne-Sanchez, A., Fromenty, B. Drug-induced toxicity on mitochondria and lipid metabolism: Mechanistic diversity and deleterious consequences for the liver. Journal of Hepatology. 54 (4), 773-794 (2011).
- Fey, S. J., Korzeniowska, B., Wrzesinski, K. Response to and recovery from treatment in human liver-mimetic clinostat spheroids: a model for assessing repeated-dose drug toxicity. Toxicology Research. 9 (4), 379-389 (2020).
- Smith, K., et al. Phenotypic image analysis software tools for exploring and understanding big image data from cell-based assays. Cell Systems. 6 (6), 636-653 (2018).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados