Method Article
Analisi fenotipica semi-automatizzata di colture cellulari sferoidi 3D funzionali
In questo articolo
Riepilogo
Presentiamo un protocollo per la coltivazione di sferoidi ad alta riproducibilità e la loro caratterizzazione fenotipica utilizzando l'acquisizione di immagini e la proteomica.
Abstract
Presentiamo un protocollo che descrive le proprietà e i vantaggi dell'utilizzo di un incubatore clinostatico autonomo per la coltivazione, il trattamento e il monitoraggio di colture cellulari 3D. Il clinostato imita un ambiente in cui le cellule possono assemblarsi come sferoidi altamente riproducibili con basse forze di taglio e diffusione attiva dei nutrienti. Dimostriamo che sia gli epatociti tumorali che quelli non tumorali (linee cellulari HepG2/C3A e THLE-3) richiedono 3 settimane di crescita prima di raggiungere funzionalità paragonabili alle cellule epatiche. Questo protocollo evidenzia la convenienza di utilizzare incubatori per cellule 3D con telecamere che monitorano la crescita cellulare, in quanto è possibile scattare istantanee per contare e misurare gli sferoidi durante il trattamento. Descriviamo il confronto tra le linee cellulari THLE-3 e HepG2/C3A, mostrando come le linee cellulari non cancerose possono essere coltivate e immortalizzate nelle cellule tumorali. Dimostriamo e illustriamo come gli esperimenti di proteomica possano essere condotti da pochi sferoidi, che possono essere raccolti senza perturbare la segnalazione cellulare, cioè senza bisogno di tripsinizzazione. Mostriamo che l'analisi proteomica può essere utilizzata per monitorare il fenotipo tipico del metabolismo della catena respiratoria e la produzione di proteine coinvolte nella detossificazione dei metalli e descriviamo un sistema semi-automatizzato per contare e misurare l'area dello sferoide. Nel complesso, il protocollo presenta una cassetta degli attrezzi che comprende una caratterizzazione fenotipica tramite acquisizione di immagini e una pipeline proteomica per sperimentare su modelli di colture cellulari 3D.
Introduzione
È stato dimostrato che le colture cellulari in vitro sono necessarie e preziose per stabilire le conoscenze fondamentali della biologia. Gran parte della comprensione scientifica in biologia e cancro in particolare è venuta dal sistema di coltura 2D, cioè cellule che crescono in un monostrato. Sebbene la coltura 2D sia stata il sistema di coltura cellulare dominante, presenta molti svantaggi che possono potenzialmente soffocare ulteriori progressi biologici. Ad esempio, le colture 2D mancano di interazioni cellula-cellula importanti per la segnalazione e la proliferazione cellulare1. Ad oggi, i sistemi di coltura 3D hanno dimostrato di modellare meglio la differenziazione, la risposta ai farmaci, l'invasione tumorale e la biologia 2,3,4,5. La modellazione 3D dei tumori maligni è particolarmente importante a causa dell'aumento dell'invecchiamento della popolazione e della mortalità per cancro. Il carcinoma epatocellulare (HCC) è una delle principali cause di mortalità correlata al cancro in tutto il mondo e spesso ha una prognosi abissale6. L'HCC è noto per avere un basso tasso di guarigione, una scarsa risposta ai farmaci e un alto tasso di recidiva 6,7,8. Sono stati sviluppati diversi modelli 3D per il fegato normale e l'HCC che imitano la fisiologia del tessuto epatico normale e maligno in vivo 9,10.
Alcuni degli attuali sistemi 3D includono sovrapposizioni liquide, bioreattori, idrogel, scaffold e strutture stampate in 3D. Gli sferoidi generati nei bioreattori offrono vantaggi unici in particolare perché imitano la distribuzione tumorale dell'esposizione ai nutrienti, dello scambio gassoso e della proliferazione/quiescenza cellulare11. I bioreattori sono particolarmente adatti per i modelli di cancro grazie alla loro facilità d'uso, all'ampia scalabilità, alla diffusione dei nutrienti e all'accessibilità11. Inoltre, i bioreattori possono consentire esperimenti ad alto rendimento, una maggiore riproducibilità e una riduzione dell'errore umano. Il bioreattore utilizzato in questo studio è unico perché simula un sistema di gravità ridotta, che riduce al minimo le forze di taglio dirompenti applicate nei tipici bioreattori, consentendo una migliore riproducibilità12. La gravità omnidirezionale e la riduzione delle forze di taglio consentono alle cellule di svilupparsi in modo più fisiologico. Come prova, le cellule HepG2/C3A cresciute con questa metodologia sviluppano organelli sferici che producono livelli in vivo di ATP, adenilato chinasi, urea e colesterolo13,14. Inoltre, i trattamenti farmacologici in questo sistema 3D sono più avanzati e automatizzati rispetto alle colture 2D. Nelle colture 2D, i trattamenti farmacologici devono spesso avere un breve decorso temporale a causa della necessità di tripsinizzare e mantenere la salute delle cellule. Tuttavia, nel nostro caso, possiamo eseguire trattamenti farmacologici a lungo termine degli sferoidi senza la necessità di interrompere la struttura e la fisiologia delle cellule. Pertanto, è necessario un passaggio dalle colture 2D a quelle 3D per modellare meglio i fenomeni biologici in vivo e l'ulteriore sviluppo scientifico.
Questo articolo presenta una metodologia per la coltivazione di sferoidi ad alta riproducibilità (Figura 1 e Figura 2) e mostra un sistema semi-automatizzato per caratterizzare fenotipicamente strutture 3D (Figura 3). A livello di immagine, forniamo informazioni sul conteggio e la misurazione dell'area degli sferoidi (Figura 3). Utilizzando metodi di spettrometria di massa, mostriamo come la proteomica può essere utilizzata per valutare specifiche funzioni biologiche (Figura 4). Raccogliendo e analizzando questi dati, speriamo di migliorare la comprensione della biologia alla base dei sistemi di coltura cellulare 3D.
Protocollo
1. Tamponi e reagenti
- Terreni di crescita cellulare per cellule HepG2/C3A: Preparare il terreno di coltura modificato di Dulbecco (DMEM, 4,5 g/L di glucosio) contenente il 10% di siero fetale bovino (FBS), aminoacidi non essenziali (1% v/v), L-glutammina (1% v/v) e penicillina/streptomicina (0,5% v/v). Conservare il substrato di coltura a 4 °C.
- Terreni di crescita cellulare per cellule THLE-3: Preparare il terreno di crescita delle cellule epiteliali bronchiali [BEGM] contenente i suoi integratori (estratto ipofisario bovino [BPE], idrocortisone, fattore di crescita epidermico umano [hEGF], epinefrina, transferrina, insulina, acido retinoico, triiodotironina, gentamicina solfato-amfotericina [GA]) e 10% FBS, 5 ng/mL hEGF e 70 ng/mL di fosfoetanolammina.
- 500 mM di DL-ditiotreitolo (DTT): per preparare 10 mL, risospendere 0,771 g di DTT in 10 mL di acqua HPLC. Conservare aliquote da 100 μL a -20 °C.
- 200 mM di iodoacetammide: per preparare 1 mL, risospendere 36 mg di iodoacetammide in 1 mL di bicarbonato di ammonio 50 mM. Non conservare iodoacetammide in sospensione.
- 5% sodio dodecil solfato (SDS): per preparare 50 mL, risospendere 2,5 g di SDS in 50 mL di bicarbonato di ammonio 50 mM. Conservare a 4 °C.
- Acido fosforico al 12%: per preparare 100 mL, diluire 14,1 mL di acido fosforico all'85% in 85,9 mL di acqua per HPLC. Conservare in bottiglia di vetro a temperatura ambiente (RT).
- Tampone legante: Per preparare 1 L, aggiungere 900 mL di metanolo concentrato a 100 mL di bicarbonato di ammonio 10 mM o TEAB.
- Soluzione di acido trifluoroacetico (TFA) allo 0,1%: aggiungere 1 mL di TFA concentrato a 999 mL di acqua HPLC. Conservare a 4 °C.
- Soluzione di acetonitrile al 60% e TFA allo 0,1%: aggiungere 600 mL di acetonitrile (60% v/v) a 399 mL di acqua per HPLC. Successivamente, aggiungere 1 mL di TFA concentrato a questa soluzione, quindi conservare a 4 °C.
- Fase mobile A (MPA) - 0,1% di acido formico: aggiungere 1 mL di acido formico concentrato a 999 mL di acqua per HPLC e mescolare bene.
- Mobile Phase B (MPB) - 80% acetonitrile HPLC + 0,1% acido formico: aggiungere 800 mL di acetonitrile HPLC a 199 mL di acqua HPLC. Successivamente, aggiungere 1 mL di acido formico concentrato a questa soluzione e mescolare.
2. Preparazione degli sferoidi
NOTA: La Figura 1A rappresenta i passaggi iniziali per la preparazione e la coltura di sferoidi 3D da linee cellulari.
- Scongelare le cellule HepG2/C3A e THLE-3 congelate e farle crescere come monostrato utilizzando terreni di crescita standard in un pallone o in un piatto di coltura tissutale fino a raggiungere circa l'80% di confluenza.
NOTA: Quando le cellule raggiungono la confluenza, controllare la morfologia cellulare generale e i modelli di crescita utilizzando un microscopio. Non è consigliabile utilizzare celle con numeri di passaggio elevati. - Lavare le cellule due volte con la soluzione salina bilanciata di Hank (HBSS, utilizzare 5 ml per un pallone T75 cm2 o un piatto da 10 cm).
- Aggiungere 5 mL di tripsina-EDTA allo 0,05% diluita in HBSS (diluizione 1:2) e incubare per 5 minuti a 37 °C con il 5% di CO2 . Utilizzare un microscopio per valutare il distacco cellulare e aggiungere 3 mL di FBS o terreni di crescita (contenenti il 10% di FBS) per neutralizzare la reazione della tripsina.
- Trasferire la sospensione cellulare in una provetta da 15 ml. Girare a 270 x g a RT per 5 min.
- Aspirare il surnatante e risospendere le cellule in 5 mL di terreno di coltura completo.
NOTA: Se ci sono troppe cellule, diluire la sospensione cellulare prima di contare. - Contare il numero di cellule e diluire la sospensione cellulare in un terreno di coltura completo per ottenere 1 x 106 in un volume massimo di 1,5 mL.
- Equilibrare una piastra inferiore rotonda a 24 pozzetti con attacco ultrabasso contenente micropozzetti.
- Lavare i pozzetti con 0,5 mL di terreno di coltura.
- Centrifugare la piastra a 3.000 x g per 5 minuti (questo rimuove le bolle d'aria dalla superficie dei pozzetti).
- Trasferire la sospensione cellulare (preparata al punto 1.4) nella piastra e centrifugare per 3 minuti a 120 x g.
NOTA: Il volume della sospensione cellulare può variare a seconda del numero di cellule, ma è importante limitarlo a 1,5 mL. - Incubare la piastra per 24 ore a 37 °C con il 5% di CO2 per iniziare la formazione degli sferoidi.
3. Coltura di sferoidi in bioreattori (Figura 2)
NOTA: Per preservare la struttura degli sferoidi, utilizzare punte a foro largo quando si maneggiano sfere 3D.
- Equilibrare il bioreattore riempiendo la camera di umidità con 25 mL di acqua sterile e la camera cellulare con 9 mL di terreno di coltura 24 ore prima di trasferire gli sferoidi (Figura 2A). Assicurarsi di utilizzare una siringa da 10 ml accoppiata a un ago lungo quando si riempiono le camere di umidità e cellule.
- Incubare il bioreattore, ruotando (15 giri/min) nell'incubatore 3D (Figura 2D,F), per 24 ore a 37 °C con il 5% di CO2 .
- Utilizzando puntali con foro largo 1 mL, pipettare delicatamente verso l'alto e verso il basso per staccare gli sferoidi dalla piastra di attacco ultra-basso e trasferire gli sferoidi in una piastra di coltura tissutale.
- Lavare la piastra di attacco ultra-bassa con 0,5 mL di terreno di coltura preriscaldato per catturare gli sferoidi rimanenti e trasferirli nella stessa piastra del passaggio 3.3.
- Valutare le dimensioni, la compattezza e la rotondità degli sferoidi utilizzando un microscopio ottico (ingrandimento 4x) e selezionare quelli sufficientemente formati.
NOTA: Le celle sono sufficientemente formate quando sono compatte e non si sfaldano durante la manipolazione. È anche importante che gli sferoidi siano un'unica unità e non raggruppati insieme. Una dimensione dello sferoide compresa tra 100 e 200 μm è preferibile quando viene trasferita al bioreattore. - Trasferire gli sferoidi nel bioreattore bilanciato riempito con 5 mL di terreno di coltura fresco. Dopo aver trasferito gli sferoidi, riempire completamente il bioreattore con nuovi terreni di crescita e assicurarsi di evitare di aggiungere bolle al bioreattore quando lo si riempie di terreno.
- Posizionare il bioreattore nell'incubatore 3D e regolare la velocità di rotazione utilizzando l'unità di controllo (Figura 2C). Per gli sferoidi HepG2/C3A, impostare la velocità di rotazione a 10-11 giri/min; per gli sferoidi THLE-3, impostarlo a 11-12 giri/min.
NOTA: La velocità di rotazione è impostata correttamente quando gli sferoidi sono distribuiti uniformemente al centro del bioreattore e non toccano le sue pareti. La Figura 2E mostra un bioreattore rotante. - Sostituire i terreni di coltura ogni 2-3 giorni rimuovendo 10 mL di terreno vecchio e sostituendolo con 10 mL di terreno fresco; assicurarsi di non rimuovere gli sferoidi durante lo scambio di fluidi (Figura 2B).
NOTA: Si consiglia di avere una routine di scambio di supporti (ad esempio, 48 ore/48 ore/72 ore) e di tenere un registro dettagliato. - Regolare la velocità di rotazione ogni volta che si cambia supporto. Aumenta la velocità man mano che gli sferoidi crescono di dimensioni e numero.
- Dopo 15 giorni di coltura, dividere gli sferoidi in due nuovi bioreattori.
NOTA: A seconda della linea cellulare e del tasso di crescita, il tempo può variare e deve essere ottimizzato individualmente. - Dopo 20 giorni, gli sferoidi sono pronti per la raccolta.
4. Acquisizione e conteggio delle immagini degli sferoidi
NOTA: La pipeline semplificata per il conteggio degli sferoidi è mostrata nella Figura 3A. Per la conta degli sferoidi e la determinazione dell'area (sezione 5), è fondamentale valutare la compattezza delle strutture 3D. Ciò contribuirà a un contrasto di colore più avanzato, necessario affinché il metodo sia accurato.
- Apri l'app 3D installata sul tablet.
- Seleziona il bioreattore e scatta un'istantanea.
NOTA: In alternativa, è possibile scattare una foto. Devono essere considerati i seguenti parametri.- Posizionare uno sfondo nero dietro il bioreattore (il supporto nero è fornito nella scatola del bioreattore).
- Assicurarsi che non vi siano riflessi di luce sulla camera cellulare.
- Scatta la foto il più vicino possibile al bioreattore.
- Aprire un'immagine in FIJI (ImageJ).
- Preparare l'immagine per l'analisi:
- Selezionate lo strumento Ovale . Disegna un cerchio attorno alla spina.
- Fai clic su Elimina sulla tastiera per rendere nero tutto ciò che si trova all'interno del cerchio. Disegna un cerchio attorno alla camera cellulare. Assicurati che tutti gli sferoidi siano all'interno del cerchio.
- Eseguire la macro per contare gli sferoidi:
- Fare clic su Plug-in > Macro > Record. Copiare il testo macro dal file supplementare 1 nel registratore.
- Premi Crea e si aprirà una nuova finestra. Premere Esegui per analizzare l'immagine aperta.
- Si aprirà una nuova finestra con i risultati (conteggio, area totale, dimensione media e area percentuale).
- Chiudere l'immagine e ripetere i passaggi 4.4 e 4.5 per ogni immagine per la quale è necessario il conteggio degli sferoidi. Per un'elaborazione più semplice e veloce, salvare la macro ed eseguirla facendo clic su Plug-in > Macro > Esegui e selezionare la macro salvata.
5. Determinazione planimetrica dell'area sferoidale
NOTA: La pipeline semplificata per la determinazione dell'area sferoidale è mostrata nella Figura 3B.
- Scattare le immagini come descritto al punto 3.5.
- Prima di iniziare, imposta una scala globale per misurare l'area degli sferoidi.
- Apri in FIJI un'immagine con una barra di scala con l'ingrandimento desiderato. Selezionate lo strumento linea. Disegna sopra la barra della scala (la linea deve essere lunga quanto la barra della scala).
- Aprite Analizza (Analyze) > Imposta scala (Set Scale). Scrivi la distanza nota (la lunghezza della linea viene inserita automaticamente nella sezione Distanza in pixel. Assicurati di selezionare Globale.
- Premere OK. Ora la scala è impostata per l'immagine che verrà analizzata.
- Per avviare la determinazione planimetrica, eseguire la macro (File supplementare 2).
- Fare clic su Elabora > Batch > macro. Scegliere la cartella che contiene le immagini da analizzare. Questa cartella sarà l'input.
- Scegliere la cartella creata nel passaggio 5.3.1 come Output. Premere Processo.
- Come controllo di qualità, valutare se l'area sferoidale misurata corrisponde all'immagine originale.
NOTA: Macro esclude gli sferoidi sul bordo in cui è possibile misurare solo una parte dello sferoide.
6. Raccolta di sferoidi
NOTA: Si consiglia vivamente di raccogliere gli sferoidi utilizzando punte a foro largo per preservarne la struttura 3D. La raccolta può essere effettuata utilizzando il tappo nella parte anteriore del bioreattore (Figura 2A).
La dimensione degli sferoidi al momento della raccolta può variare a seconda della linea cellulare, del numero iniziale di cellule e del processo di scissione (giorni in coltura, numero di sferoidi per bioreattore e rapporto di divisione).
- Per raccogliere gli sferoidi, rimuovere 5 mL di terreno dal bioreattore attraverso la porta superiore utilizzando una siringa accoppiata a un ago lungo. Assicurati di lasciare che gli sferoidi affondino in basso al centro del bioreattore (vicino alla porta inferiore).
- Aprire la porta anteriore e raccogliere gli sferoidi utilizzando una punta larga 1 mL. Mettere gli sferoidi in provette per microcentrifuga. Centrifugare gli sferoidi a 500 x g per 5 minuti ed eliminare i terreni.
- Lavare gli sferoidi con 200 μL di HBSS per rimuovere l'FBS. Centrifugare a 500 x g per 5 minuti ed eliminare il surnatante.
- Congelare istantaneamente il pellet sferoidale con azoto liquido e conservare a -80 °C fino alla lavorazione.
7. Vitalità degli sferoidi
NOTA: La vitalità degli sferoidi è stata determinata misurando l'attività dell'adenilato chinasi (AK) rilasciata dalle cellule danneggiate (Figura 4A). A causa del gradiente di diffusione, la misurazione dell'AK è efficiente quando gli sferoidi sono più piccoli di 900 μm di diametro12. Se gli sferoidi diventano più grandi, o se c'è qualche dubbio sulla misurazione della vitalità, può essere eseguito un test dell'ATP15.
- Raccogliere il surnatante sferoide e trasferire 20 μL in una piastra a 96 pozzetti con pareti bianche (fondo piatto trasparente). Aggiungere 100 μL di reagente per il rilevamento dell'adenilato chinasi a ciascun pozzetto e omogeneizzare pipettando delicatamente verso l'alto e verso il basso.
- Rimuovere le bolle mediante centrifugazione nel vuoto a velocità per 2 minuti a RT. Incubare le piastre per 20 minuti a RT.
- Posizionare la lastra nel luminometro (lettore di targhe, modalità luminescenza) e avviare il programma. Impostare i parametri secondo le istruzioni del produttore del kit.
NOTA: Nei saggi di vitalità bioluminescente, l'emissione luminosa diretta del luminometro (comunemente RLU) può essere utilizzata per calcolare la risposta cellulare. Poiché l'adenilato chinasi fuoriesce solo dalle cellule la cui integrità cellulare è stata compromessa, è possibile ottenere un controllo totale dell'adenilato chinasi utilizzando un reagente di lisi.
8. Estrazione delle proteine
NOTA: La Figura 1B rappresenta il flusso di lavoro per l'elaborazione degli sferoidi e l'estrazione delle proteine.
- Raccogliere gli sferoidi (sezione 6) il giorno 36. Risospendere gli sferoidi in 25 μL di SDS al 5% per lisare le cellule.
- Dopo aver aggiunto il 5% di SDS, omogeneizzare il pellet pipettando su e giù. In alcuni casi, quando è difficile lisare gli sferoidi, utilizzare un piccolo pestello.
- Incubare i campioni con 20 mM DTT per 1 ora per ridurre le proteine. Mescolare pipettando su e giù.
- Incubare i campioni con iodoacetammide 40 mM per 30 minuti al riparo da proteine leggere o alchilate. Mescolare pipettando su e giù.
- Aggiungere una soluzione di acido fosforico al 12% (10x) ai campioni a una concentrazione finale dell'1,2%.
- Diluire i campioni in sei volumi di tampone di legame e mescolare delicatamente.
- Caricare il campione su una piastra filtrante S-Trap e centrifugare a 500 x g per 30 s.
NOTA: Se il volume totale del campione al punto 8.4 supera la capacità del volume della colonna, caricare i campioni in lotti e ripetere il passaggio 8.6 fino a quando tutto non è passato attraverso la colonna. - Lavare i campioni due volte con 150 μL di tampone legante. Dopo ogni lavaggio, centrifugare a 500 x g per 30 s ed eliminare il flusso.
- Incubare i campioni con 1 μg di tripsina sequenziabile diluita in 50 mM di bicarbonato di ammonio per una notte a 37 °C.
NOTA: Si raccomandano almeno 20 μg di proteine come materiale di partenza per un'analisi completa del proteoma. Per questo, 1 μg di tripsina è la quantità ideale per una digestione efficiente. Rimuovere eventuali bolle tra il tampone e il letto della colonna. - Eluire i peptidi con 40 μL di bicarbonato di ammonio 50 mM e raggrupparli nella stessa provetta di raccolta.
- Girare verso il basso a 500 x g per 30 s. Eluire i peptidi con 40 μL di TFA allo 0,1% e raggrupparli nella stessa provetta di raccolta.
- Girare verso il basso a 500 x g per 30 s. Eluire i peptidi con 40 μL di acetonitrile al 60% e TFA allo 0,1% e raggrupparli nella stessa provetta di raccolta.
- Girare verso il basso a 500 x g per 30 s. Essiccare gli eluati in pool nel vuoto veloce e conservare i campioni a -80 °C fino al trattamento.
9. Pulizia del campione
NOTA: Prima di procedere all'analisi proteomica è necessario rimuovere il sale presente nei campioni. I sali possono interferire con l'analisi della cromatografia liquida-spettrometria di massa (HPLC-MS) ad alte prestazioni poiché si ionizzano durante l'elettrospray, sopprimendo il segnale dei peptidi. La configurazione per la desalinizzazione utilizzata in questo studio è stata precedentemente dimostrata da Joseph-Chowdhury e colleghi16.
- Prima di iniziare, assicurarsi che ci sia una piastra di raccolta pulita a 96 pozzetti per raccogliere il flusso durante i passaggi successivi. Mescolare la resina C18 (50 mg/mL in acetonitrile al 100%) su una piastra magnetica.
- Aggiungere 70 μL di sospensione di resina C18 per pozzetto della piastra filtrante a 96 pozzetti, eseguire questa operazione rapidamente per assicurarsi che la resina C18 non si accumuli sul fondo della pipetta.
- Accendi delicatamente l'aspirapolvere per evitare schizzi. Scartare il flusso continuo raccolto sulla piastra di raccolta a 96 pozzetti.
- Lavare la resina con 100 μL di TFA allo 0,1%. Accendere delicatamente il vuoto per evitare schizzi e miscelazione dei campioni e scartare il flusso.
- Risospendere ogni campione in 100 μL di TFA allo 0,1%. Controllare e assicurarsi che il pH sia compreso tra 2-3.
- Caricare ogni campione in ciascun pozzetto della piastra filtrante. Accendere delicatamente il vuoto per evitare schizzi e miscelazione dei campioni e scartare il flusso.
- Lavare con 100 μL di TFA allo 0,1%. Accendere delicatamente l'aspirapolvere per evitare schizzi e miscelazione dei campioni. Scartare il flusso.
- Sostituire la piastra di raccolta a 96 pozzetti con una nuova piastra a 96 pozzetti per raccogliere i campioni dissalati.
- Aggiungere 60 μL di acetonitrile/0,1% TFA per pozzetto. Per eluire i campioni dalla resina C18, accendere delicatamente l'aspirapolvere per evitare schizzi.
- Raccogliere il flusso e asciugare in un vuoto a velocità a RT. Procedere a LC-MS/MS o conservare la piastra a -80 °C fino al momento di elaborare i campioni.
10. Analisi proteomica mediante cromatografia liquida accoppiata a spettrometria di massa
NOTA: Per generare i dati per questo manoscritto, è stato utilizzato un sistema nLC-MS/MS con una configurazione di sistema a due colonne con una colonna trappola C18 ID 300 μm x 0,5 cm e una nanocolonna analitica C18-AQ (3 μm) ID 75 μm x 25 cm confezionata internamente.
- Preparare le fasi mobili per l'esecuzione sull'HPLC:
Fase mobile A (MPA): 0,1% di acido formico in acqua per HPLC
Fase mobile B (MPB): 0,1% di acido formico in acetonitrile di grado HPLC - Programmare il metodo HPLC come segue: 4%-34% MPB su 120 min, 34%-90% MPB su 5 min, isocratico 90% MPB su 5 min; portata: 300 nL/min.
- Impostare il metodo di acquisizione MS per eseguire l'acquisizione indipendente dai dati (DIA) per generare spettri MS/MS dei segnali peptidici rilevati.
- Risospendere 1 μg di campioni in 10 μL di TFA allo 0,1% prima di inserire i campioni nell'autocampionatore nLC.
- Eseguire il metodo nLC-MS come programmato per l'acquisizione della proteomica canonica:
- Impostare la scansione MS completa a 300-1100 m/z nell'orbitrap, con una risoluzione di 120.000 (a 200 m/z) e un target di controllo automatico del guadagno (AGC) di 125.
- Impostare MS/MS nell'orbitrap con una finestra di isolamento sequenziale di 50 m/z con un target AGC di 400 e un'energia di dissociazione collisionale (HCD) ad alta energia di 30.
11. Analisi dei dati
- Importare i file di dati grezzi nLC-MS/MS in un software di rilevamento dei picchi compatibile con la piattaforma MS utilizzata.
- Seleziona il database appropriato (umano, mouse, ecc.).
- Impostare l'acetilazione N-terminale come modifica variabile e la carbamidometilcisteina come modifica fissa.
- Specificare la tripsina come enzima digestivo con due scissioni mancate consentite.
- Impostare la tolleranza di massa in base all'analizzatore di massa utilizzato per l'acquisizione degli spettri.
- Esporta l'analisi come foglio di calcolo ed elabora ulteriormente i dati in base alle esigenze.
Risultati
In questo protocollo, descriviamo le proprietà di un innovativo incubatore cellulare 3D senza stress, un sistema progettato specificamente per la coltura di sferoidi 3D (Figura 2). Abbiamo ottimizzato il protocollo per la coltura 3D di linee cellulari THLE-3 e HepG2/C3A. Il protocollo qui descritto è semplice da usare e consente la riproducibilità e la coltura economica di > 100 sferoidi per bioreattore. Una volta nel bioreattore, gli sferoidi vengono trattati in modo simile alle cellule mantenute in coltura 2D. Le condizioni di crescita ottimali si ottengono scambiando il terreno due o tre volte alla settimana (Figura 2B) e regolando la velocità di rotazione in base alla crescita e alle dimensioni degli sferoidi (Figura 2C). Questo sistema, in cui gli sferoidi 3D vengono coltivati in bioreattori rotanti (Figura 2E,F), fornisce un ambiente di crescita ottimale per le strutture 3D esponendo lo sferoide a una quantità uguale e molto bassa di forza di taglio.
Abbiamo precedentemente mostrato come gli sferoidi possono essere utilizzati per l'analisi della modificazione della cromatina16. Qui dimostriamo in dettaglio come ottenere sferoidi epatici e come possono essere condotti esperimenti di proteomica per l'analisi dell'intero proteoma (Figura 1). In breve, il protocollo è stato avviato utilizzando cellule piatte THLE-3 o HepG2/C3A fino a quando la coltura non ha raggiunto l'80% di confluenza. Per coltivare le cellule come sferoidi, circa 2.000 cellule sono state placcate in una piastra a bassissimo attacco contenente micropozzetti per consentire loro di auto-aggregarsi, quindi gli sferoidi formati sono stati trasferiti in un bioreattore (Figura 1A). Sebbene siano funzionalmente attivi dopo 3 settimane di coltura, come dimostrato in precedenza17, mostriamo i risultati degli sferoidi raccolti dopo 36 giorni di coltura per questo protocollo. Dopo la raccolta, gli sferoidi sono stati centrifugati e sia il pellet che il surnatante sono stati conservati per l'analisi. La vitalità cellulare è stata valutata dal surnatante per la quantificazione dell'adenilato chinasi rilasciata dalle cellule danneggiate, come descritto in precedenza17. Le proteine cellulari sono state estratte dal pellet cellulare e l'intero proteoma è stato analizzato mediante spettrometria di massa ad alta risoluzione (Figura 1B).
Questo protocollo dimostra anche un metodo semi-automatico per il conteggio degli sferoidi (Figura 3A) utilizzando il programma di elaborazione delle immagini pubbliche FIJI (Fiji Is Just ImageJ)18. Per l'analisi deve essere scattata un'immagine di buona qualità dello sferoide e devono essere considerati alcuni parametri come menzionato nel paragrafo 5. Quindi, dopo aver preparato l'immagine per l'analisi, viene utilizzato uno script macro (File supplementare 1) per contare gli sferoidi. La macro funziona creando prima una cartella chiamata FIJI Spheroids counting, all'interno della cartella in cui si trovano le immagini degli sferoidi. In questa cartella vengono salvate tutte le informazioni dell'analisi; questo include un'immagine degli sferoidi che sono stati contati, con un numero ID su ogni sferoide. Include anche un file Excel chiamato conteggio degli sferoidi. Questo file contiene l'area dei pixel e il numero ID per ogni sferoide che è stato conteggiato. I dati corrispondenti a un'immagine analizzata vengono presentati in ogni scheda del file. La scheda è etichettata in base al nome dell'immagine analizzata. Poiché le dimensioni degli sferoidi possono essere compromesse da molti fattori, tra cui il numero di strutture all'interno di un vaso e il trattamento farmacologico, è anche importante monitorare la loro superficie (planimetria). Lo script macro qui presentato (File supplementare 2) funziona misurando le aree nere, che corrispondono agli sferoidi nell'immagine (Figura 3B). L'output viene raccolto in un file chiamato planimetry.xlsx, che contiene l'area, il perimetro e il diametro misurati di ogni sferoide. C'è anche una misura chiamata Feret, utilizzata per calcolare il diametro. Feret è il diametro più lungo possibile, mentre minFeret è il più corto. Il diametro è la media di questi due. All'interno della cartella di output, oltre al file planimetry.xlsx, c'è anche un'immagine degli sferoidi che sono stati misurati.
Prima di procedere all'analisi del proteoma, è stata valutata la vitalità degli sferoidi nel corso del tempo di coltura. I livelli di AK aumentano fino al giorno 17, raggiungendo circa il 7% della morte cellulare, e poi la morte diminuisce a livelli inferiori al 5% (Figura 4A), che è in accordo con il lavoro precedentemente pubblicato17. Questo protocollo mostra anche l'analisi completa del proteoma per il monitoraggio del fenotipo cellulare. In primo luogo, sono stati confrontati i proteomi delle cellule piatte e degli sferoidi THLE-3 e HepG2/C3A. Analizzando il primo componente principale (PC1), è evidente che esiste una rigorosa separazione dei campioni di sferoidi dalle colture cellulari piatte e sembra che la correlazione del tipo di cellula (THLE-3 e HepG2/C3A) non sia rilevante (Figura 4B). Sebbene gli sferoidi THLE-3 e HepG2/C3A non si raggruppino insieme, condividono profili simili coerenti con la funzionalità epatica. Dimostriamo in questo protocollo l'esempio delle metallotioneine, che hanno un ruolo nella disintossicazione dei metalli effettuata dal fegato. Nell'analisi proteomica sono state identificate 2 isoforme sovraespresse negli sferoidi rispetto alle cellule piatte (MT1E e MT1X) (Figura 4C). Mostriamo anche l'arricchimento dell'ontologia genica (GO) di entrambe le linee cellulari cresciute come sferoidi. Il processo metabolico dei carboidrati, che comprende il ciclo dell'acido tricarbossilico (ciclo TCA), la catena di trasporto degli elettroni (respirazione cellulare) e il metabolismo del piruvato, è un termine frequente ed è arricchito sia negli sferoidi HepG2/C3A che THLE-3 (Figura 4D,E). La disintossicazione cellulare, gli acidi grassi e il metabolismo del colesterolo sono altre funzioni arricchite in entrambi gli sferoidi. Insieme, queste funzioni sono note per essere cruciali per la funzionalità epatica.
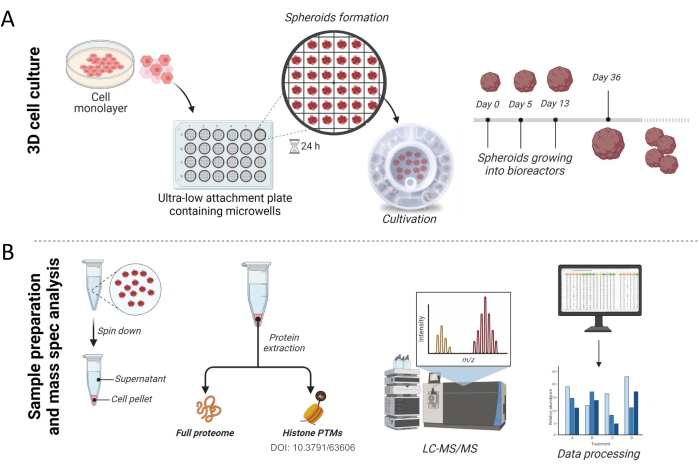
Figura 1: Flusso di lavoro per la coltura di sferoidi e la preparazione del campione . (A) Approccio sperimentale di colture cellulari 3D. Le colture cellulari piatte alla confluenza desiderata sono state tripsinizzate e seminate su una piastra a 24 pozzetti a bassissimo attacco contenente micropozzetti, dove le cellule si auto-assemblano in sferoidi. Dopo 24 ore, gli sferoidi sono stati trasferiti in un bioreattore e coltivati fino a quando non sono pronti per l'analisi. (B) Dopo la raccolta, gli sferoidi sono stati pellettati e sia il pellet che il surnatante di coltura sono stati conservati fino alla lavorazione. Gli istoni16e le proteine non istoniche sono stati estratti, digeriti in peptidi e analizzati mediante spettrometria di massa ad alta risoluzione. I file grezzi ottenuti dalla spettrometria di massa sono stati confrontati con il database umano e i dati sono stati ulteriormente elaborati. Fare clic qui per visualizzare una versione più grande di questa figura.
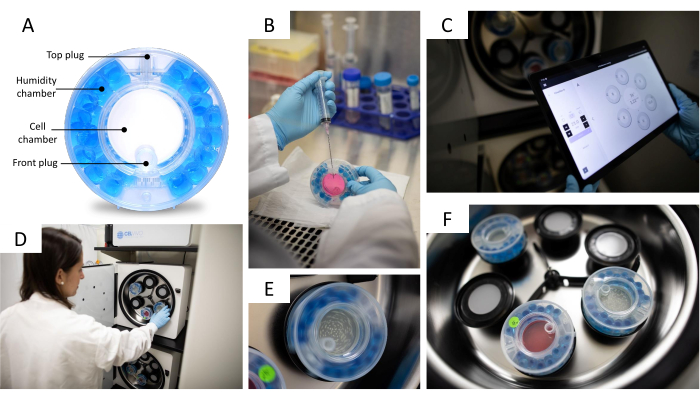
Figura 2: Sistema di coltura cellulare 3D . (A) Parti del bioreattore. Il bioreattore è composto da una camera di scambio gassoso e umidificazione contenente perle d'acqua e da una camera cellulare apribile con due tappi per lo scambio di terreni e la raccolta degli sferoidi. (B) Scambio di terreni per bioreattori. Il bioreattore viene riempito con 10 mL di terreno di coltura utilizzando una siringa con un ago. (C) L'app di controllo del sistema. La velocità di rotazione, il livello di CO2, la temperatura, il registro degli allarmi e altre funzionalità possono essere controllate utilizzando l'unità di controllo. (D) Posizionamento del bioreattore nell'incubatore 3D. Ogni bioreattore ha un motore associato che può far girare lentamente il bioreattore. (E) Bioreattore in movimento con la velocità (giri/min) controllata da una compressa (C). La velocità (giri/min) viene regolata in base alle dimensioni degli sferoidi. (F) Bioreattori all'interno dell'incubatore 3D. L'incubatore 3D può ospitare fino a 6 bioreattori controllati individualmente. Foto per gentile concessione di Jason Torres Photography. Fare clic qui per visualizzare una versione più grande di questa figura.
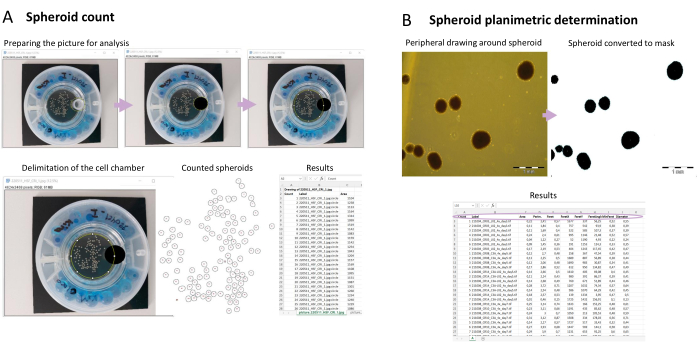
Figura 3: Caratterizzazione fenotipica degli sferoidi tramite acquisizione di immagini . (A) Conta semiautomatica degli sferoidi. Dopo aver scattato istantanee degli sferoidi nel bioreattore, l'immagine viene preparata per l'analisi nelle FIJI. Ogni sferoide viene contato e per ciascuno di essi viene fornito un numero ID. Viene utilizzata una macro e vengono visualizzati i risultati che mostrano l'ID per lo sferoide conteggiato, l'etichetta (nome dell'immagine analizzata) e l'area (il numero di pixel contati nello sferoide). (B) Determinazione planimetrica dell'area sferoidale. Utilizzando una macro, vengono determinati l'area, il perimetro e il diametro di uno sferoide specifico. Fare clic qui per visualizzare una versione più grande di questa figura.
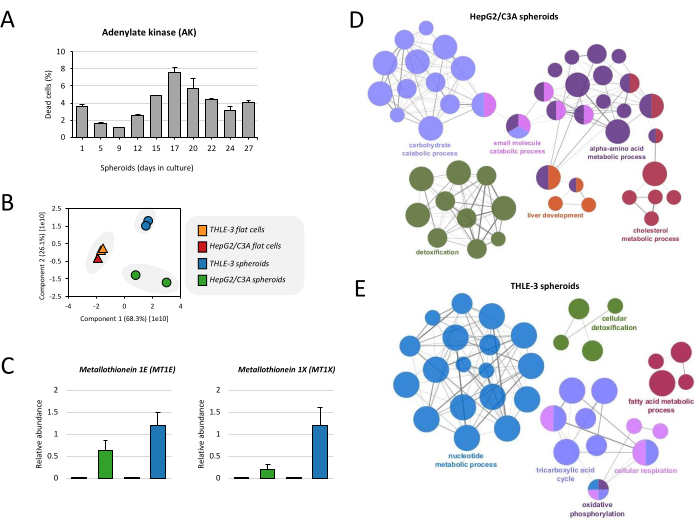
Figura 4: Analisi del proteoma degli sferoidi epatici . (A) La vitalità degli sferoidi è stata calcolata in base al rilascio di adenilato chinasi (AK) sul surnatante della coltura. I risultati sono la media di punti dati duplicati ± SD. (B) L'analisi delle componenti principali (PCA) è stata eseguita per confrontare il proteoma di cellule piatte e sferoidi THLE-3 e HepG2/C3A. (C) Abbondanza relativa di metallotioneine, che sono proteine espresse dal fegato umano. I dati sono rappresentati come medie ± SEM. (D) Le reti funzionalmente raggruppate mostrano l'arricchimento di GO per gli sferoidi HepG2/C3A e (E) gli sferoidi THLE-3, dove viene mostrata solo l'etichetta del termine più significativo per gruppo. La rete è stata costruita utilizzando ClueGo19. La dimensione del nodo rappresenta il significato dell'arricchimento del termine. Fare clic qui per visualizzare una versione più grande di questa figura.
File supplementare 1: Script macro per il conteggio degli sferoidi. Fare clic qui per scaricare il file.
File supplementare 2: Macro per la determinazione planimetrica degli sferoidi. Fare clic qui per scaricare il file.
Discussione
Comprendere la biologia alla base delle strutture cellulari tridimensionali (3D) è estremamente importante per una conoscenza più completa delle loro funzionalità. C'è un crescente interesse nell'utilizzo di modelli 3D per lo studio di biologia complessa e l'esecuzione di screening della tossicità. Quando si coltivano cellule in 3D è necessario considerare molti fattori, tra cui la valutazione fenotipica del sistema modello. Un fenotipo è definito come un gruppo di caratteristiche osservabili di uno specifico organismo, come la morfologia, il comportamento, le proprietà fisiologiche e biochimiche20.
In questo protocollo, dimostriamo come gli esperimenti di proteomica possono essere condotti da pochi sferoidi e possono essere utilizzati per monitorare il fenotipo tipico del fegato. La spettrometria di massa è diventata un metodo ampiamente applicato per la caratterizzazione cellulare 3D, consentendo l'indagine di una varietà di questioni biologiche 12,16,21,22. Per un'analisi completa del proteoma, si consiglia di utilizzare almeno 20 μg di materiale di partenza proteico, da cui 1 μg viene iniettato nello spettrometro di massa. È importante ricordare che l'aggiunta di meno campione potrebbe portare a una perdita di sensibilità e l'aggiunta di più peggiorerebbe gradualmente la qualità della cromatografia e alla fine porterebbe al blocco della colonna. In questo studio, abbiamo dimostrato che gli sferoidi HepG2/C3A e THLE-3 sono arricchiti con importanti proteine provenienti dalla glicolisi e dal ciclo TCA, che sono vie epatiche specifiche e sono fondamentali per il mantenimento dei livelli di glucosio nel sangue e per la produzione di energia23,24. In realtà, l'analisi di spettrometria di massa fornisce non solo informazioni a livello proteico, ma permette anche di studiare le modificazioni post-traduzionali delle proteine, come mostrato in precedenza dal nostro gruppo16.
Un altro aspetto da considerare negli studi fenotipici 3D è il numero e la dimensione degli sferoidi. Oltre a rendere gli esperimenti più riproducibili, contare il numero di sferoidi e determinarne le dimensioni è essenziale per determinare quando dividere la coltura in più bioreattori, poiché il numero di strutture 3D all'interno di un recipiente può influire sulle dimensioni degli sferoidi e sui livelli di attività metabolica. Tuttavia, è importante sottolineare che il numero e le dimensioni degli sferoidi dipendono dalla linea cellulare, dal numero iniziale di cellule, dal processo di scissione e dal tempo di raccolta. I dettagli della coltura di sferoidi HepG2/C3A, come il numero di cellule per sferoidi, il contenuto proteico e le dimensioni in funzione dell'età, sono stati forniti da Fey, Korzeniowska e Wrzesinski25. Per un'analisi accurata e di successo utilizzando il metodo semi-automatico qui descritto, il passo più critico è una buona immagine degli sferoidi. Per semplicità, la foto può essere scattata con un telefono o un tablet, ma la sua risoluzione dovrebbe essere mantenuta il più alta possibile. Poiché le immagini sono rapide da acquisire, consentono esperimenti di screening su larga scala per visualizzare specifiche caratteristiche fenotipiche o indagare le risposte al trattamento farmacologico. Pertanto, a causa del crescente numero di saggi basati su cellule, negli ultimi 10 anni è stato sviluppato un certo numero di software open-source per l'analisi delle immagini26. In questo protocollo, descriviamo un sistema semi-automatizzato che utilizza il software FIJI18 per contare e misurare le dimensioni degli sferoidi. Abbiamo presentato degli script (semplici comandi di programmazione) per definire una sequenza di operazioni algoritmiche che possono essere applicate ad una collezione di immagini, rendendo l'analisi un processo facile e veloce. Tuttavia, a seconda delle caratteristiche dello sferoide, è necessario utilizzare una misurazione manuale. Ad esempio, se gli sferoidi sono troppo traslucidi, l'alfabeto FIJI sarà impreciso. A proposito, uno dei criteri più importanti per il funzionamento di questo metodo è la compattezza degli sferoidi. Questa caratteristica contribuirà a un contrasto di colore più elevato tra gli sferoidi e lo sfondo, necessario affinché il metodo sia accurato.
In sintesi, oltre a presentare una metodologia per la coltivazione di sferoidi ad alta riproducibilità, è stato descritto anche un sistema semi-automatizzato accoppiato alla caratterizzazione fenotipica tramite acquisizione di immagini e proteomica. Ci aspettiamo che questo toolbox per l'analisi delle cellule 3D diventi più robusto con un software di analisi delle immagini completamente automatizzato e spettrometri di massa di nuova generazione.
Divulgazioni
Helle Sedighi Frandsen lavora come ricercatrice presso CelVivo ApS, l'azienda produttrice del sistema ClinoStar. Karoline Mikkelsen è una dottoranda industriale impiegata presso CelVivo ApS e sta svolgendo il suo dottorato di ricerca presso la SDU, Odense, Danimarca. Tutti gli altri autori non hanno interessi finanziari concorrenti.
Riconoscimenti
Il laboratorio Sidoli ringrazia la Leukemia Research Foundation (Hollis Brownstein New Investigator Research Grant), l'AFAR (premio Sagol Network GerOmics), Deerfield (premio Xseed), Relay Therapeutics, Merck e l'Ufficio del Direttore NIH (1S10OD030286-01).
Materiali
| Name | Company | Catalog Number | Comments |
| 1.5 mL microcentrifuge tubes | Bio-Rad | 2239480 | |
| 10 mL syringe | Fisher Scientific | 1481754 | Luer lock tip, graduated to 12 mL |
| 1000 µL wide bore pipet tips | Fisher Scientific | 14222703 | |
| 200 µL wide bore pipet tips | Fisher Scientific | 14222730 | |
| 96-well Orochem filter plate | Orochem | OF1100 | |
| 96-well skirted plate | Axygen | PCR-96-FS-C | |
| 96-well vacuum manifold | Millipore | MAVM0960R | |
| Ammonium bicarbonate | Sigma | A6141-25G | |
| Bronchial Epithelial Cell Growth Medium (BEGM) | Lonza | CC-3170 | |
| Cell culture grade water | Corning | 25-055-CV | |
| ClinoReactor | CelVivo | N/A | Bioreactor for 3D cell culture |
| ClinoStar incubator | CelVivo | N/A | CO2 incubator for 3D cell culture |
| DTT | Sigma | D0632-5G | |
| Dulbecco's Modified Eagle's Medium (DMEM) | Fisher Scientific | MT17205CV | |
| Elplasia 24-well round bottom ultra-low attachment plate containing microwells | Corning | 4441 | |
| Fetal Bovine Serum | Fisher Scientific | MT35010CV | |
| Formic acid | Thermo | 28905 | |
| Hank's Balanced Salt Solution (HBSS) | Fisher Scientific | MT21022CV | |
| hEGF | Corning | 354052 | |
| HERAcell vios 160i | Thermo | 51033557 | CO2 incubator for 2D cell culture |
| HPLC grade acetonitrile | Fisher Scientific | A955-4 | |
| HPLC grade methanol | Fisher Scientific | A452-1 | |
| HPLC grade water | Fisher Scientific | W5-4 | |
| Iodoacetamide | Sigma | I1149-5G | |
| L-glutamine | Fisher Scientific | MT25015CI | |
| Non-essential amino acids | Fisher Scientific | MT25025CI | |
| Oasis HLB Resin 30 µm | Waters | 186007549 | |
| Orbitrap Fusion Lumos Tribrid mass spectrometer | Thermo | IQLAAEGAAPFADBMBHQ | High resolution mass spectrometer |
| PAULA microscope | Leica | ||
| Penicillin-Streptomycin | Fisher Scientific | MT3002CI | |
| PerkinElmer Victor X2 multilabel microplate reader | PerkinElmer | ||
| pH paper | Hydrion | 93 | |
| Phosphoetanolamine | Sigma | P0503 | |
| Phosphoric acid | Fisher Scientific | A260-500 | |
| Pipette gun | Eppendorf | Z666467 (Milipore Sigma) | |
| Refrigerated centrifuge | Thermo | 75-217-420 | |
| Reprosil-Pur resin | MSWIL | R13.AQ.003 | 120 Å pore size, C18-AQ phase, 3 μM bead size |
| SDS | Bio-Rad | 1610301 | |
| Sequencing grade modified trypsin | Promega | V511A | |
| SpeedVac vacuum concentrator (96-well plates) | Thermo | 15308325 | Savant SPD1010 |
| Sterile hood | Thermo | 1375 | |
| Sterile serological pipettes | Fisher Scientific | 1367549 | |
| S-trap | Protifi | C02-micro-80 | |
| Syringe needle (18 G) | Fisher Scientific | 14817100 | 3" length, 0.05" diameter |
| Trifluoroacetic acid (TFA) | Thermo | 28904 | |
| Trypsin-EDTA | Gibco | 25300-054 | |
| Vortex | Sigma | Z258415 | |
| Water bath | Fisher Scientific | FSGPD10 |
Riferimenti
- Ravi, M., Paramesh, V., Kaviya, S. R., Anuradha, E., Solomon, F. D. 3D cell culture systems: advantages and applications. Journal of Cellular Physiology. 230 (1), 16-26 (2015).
- Nirmalanandhan, V. S., Duren, A., Hendricks, P., Vielhauer, G., Sittampalam, G. S. Activity of anticancer agents in a three-dimensional cell culture model. Assay and Drug Development Technologies. 8 (5), 581-590 (2010).
- Erickson, I. E., Huang, A. H., Chung, C., Li, R. T., Burdick, J. A., Mauck, R. L. Differential maturation and structure-function relationships in mesenchymal stem cell- and chondrocyte-seeded hydrogels. Tissue Engineering Part A. 15 (5), 1041-1052 (2009).
- Vinci, M., et al. Advances in establishment and analysis of three-dimensional tumor spheroid-based functional assays for target validation and drug evaluation. BMC Biology. 10 (29), (2012).
- Liu, J., Abate, W., Xu, J., Corry, D., Kaul, B., Jackson, S. K. Three-dimensional spheroid cultures of A549 and HepG2 cells exhibit different lipopolysaccharide (LPS) receptor expression and LPS-induced cytokine response compared with monolayer cultures. Innate Immunity. 17 (3), 245-255 (2011).
- Khafaga, A. F., Mousa, S. A., Aleya, L., Abdel-Daim, M. M. Three-dimensional (3D) cell culture: a valuable step in advancing treatments for human hepatocellular carcinoma. Cancer Cell International. 22 (1), 243 (2022).
- Llovet, J. M., Burroughs, A., Bruix, J. Hepatocellular carcinoma. The Lancet. 362 (9399), 1907-1917 (2003).
- Sia, D., Llovet, J. M. Liver cancer: Translating '-omics' results into precision medicine for hepatocellular carcinoma. Nature Reviews Gastroenterology & Hepatology. 14 (10), 571-572 (2017).
- Tang, J., et al. A three-dimensional cell biology model of human hepatocellular carcinoma in vitro. Tumour Biology. 32 (3), 469-479 (2011).
- van Zijl, F., Mikulits, W. Hepatospheres: Three dimensional cell cultures resemble physiological conditions of the liver. World Journal of Hepatology. 2 (1), 1-7 (2010).
- Chaicharoenaudomrung, N., Kunhorm, P., Noisa, P. Three-dimensional cell culture systems as an in vitro platform for cancer and stem cell modeling. World Journal of Stem Cells. 11 (12), 1065-1083 (2019).
- Wrzesinski, K., Fey, S. J. Metabolic reprogramming and the recovery of physiological functionality in 3D cultures in micro-bioreactors. Bioengineering(Basel, Switzerland). 5 (1), 22 (2018).
- Breslin, S., O'Driscoll, L. Three-dimensional cell culture: The missing link in drug discovery. Drug Discovery Today. 18 (5-6), 240-249 (2013).
- Kapalczynska, M., et al. 2D and 3D cell cultures - a comparison of different types of cancer cell cultures. Archives of Medical Science: AMS. 14 (4), 910-919 (2018).
- Wrzesinski, K., Frandsen, H. S., Calitz, C., Gouws, C., Korzeniowska, B., Fey, S. J. Clinostat 3D cell culture: Protocols for the preparation and functional analysis of highly reproducible, large, uniform spheroids and organoids. Methods in Molecular Biology. 2273, 17-62 (2021).
- Joseph-Chowdhury, J. N., et al. Global level quantification of histone post-translational modifications in a 3D cell culture model of hepatic tissue. Journal of Visualized Experiments: JoVE. 183, 63606 (2022).
- Wrzesinski, K., Fey, S. J. After trypsinisation, 3D spheroids of C3A hepatocytes need 18 days to re-establish similar levels of key physiological functions to those seen in the liver. Toxicology Research. 2, 123-135 (2013).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Bindea, G., et al. ClueGO: a Cytoscape plug-in to decipher functionally grouped gene ontology and pathway annotation networks. Bioinformatics. 25 (8), 1091-1093 (2009).
- Houle, D., Govindaraju, D. R., Omholt, S. Phenomics: The next challenge. Nature Reviews. Genetics. 11 (12), 855-866 (2010).
- Gonneaud, A., Asselin, C., Boudreau, F., Boisvert, F. M. Phenotypic analysis of organoids by proteomics. Proteomics. 17 (20), (2017).
- Avelino, T. M., et al. Mass spectrometry-based proteomics of 3D cell culture: A useful tool to validate culture of spheroids and organoids. SLAS Discovery. 27 (3), 167-174 (2022).
- Chiang, J., McManus, L. M., MitchellIn, R. N. . Liver physiology: Metabolism and Detoxification. Pathobiology of Human Disease. , 1770-1782 (2014).
- Begriche, K., Massart, J., Robin, M. A., Borgne-Sanchez, A., Fromenty, B. Drug-induced toxicity on mitochondria and lipid metabolism: Mechanistic diversity and deleterious consequences for the liver. Journal of Hepatology. 54 (4), 773-794 (2011).
- Fey, S. J., Korzeniowska, B., Wrzesinski, K. Response to and recovery from treatment in human liver-mimetic clinostat spheroids: a model for assessing repeated-dose drug toxicity. Toxicology Research. 9 (4), 379-389 (2020).
- Smith, K., et al. Phenotypic image analysis software tools for exploring and understanding big image data from cell-based assays. Cell Systems. 6 (6), 636-653 (2018).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon