Method Article
Evidencia de EpCAM y citoqueratina que expresan células epiteliales en sangre humana y murina normal y médula ósea
En este artículo
Resumen
Este artículo presenta un método reproducible con nuevos hallazgos sobre la presencia de células epiteliales en sangre y médula ósea humana y de ratón normal utilizando citometría de flujo y microscopía de inmunofluorescencia. Se utilizaron ratones transgénicos Krt1-14;mTmG como método in vivo para confirmar estos hallazgos.
Resumen
Se han identificado células epiteliales en la sangre y la médula ósea de pacientes con cáncer y otras enfermedades. Sin embargo, la presencia de células epiteliales normales en la sangre y la médula ósea de individuos sanos aún no se ha identificado de manera consistente. Aquí se presenta un método reproducible para aislar células epiteliales de sangre y médula ósea (BM) humana sana y murina utilizando citometría de flujo y microscopía de inmunofluorescencia (IF). Las células epiteliales en individuos sanos se identificaron y aislaron por primera vez mediante citometría de flujo utilizando la molécula de adhesión de células epiteliales (EpCAM). Se confirmó que estas células EpCAM + expresan queratina utilizando microscopía de inmunofluorescencia en ratones transgénicos Krt1-14;mTmG. Las muestras de sangre humana tenían 0,18% ± 0,0004 células EpCAM+ (SEM; n = 7 réplicas biológicas, 4 réplicas experimentales). En BM humana, el 3,53% ± 0,006 (SEM; n = 3 réplicas biológicas, 4 réplicas experimentales) de células mononucleares fueron EpCAM +. En sangre de ratón, las células EpCAM+ constituyeron 0,45% ± 0,0006 (SEM; n = 2 réplicas biológicas, 4 réplicas experimentales), y en BM de ratón, 5,17% ± 0,001 (SEM; n = 3 réplicas biológicas, 4 réplicas experimentales) fueron EpCAM +. En ratones, todas las células EpCAM + fueron inmunorreactivas a la pancitoqueratina, según lo determinado por microscopía IF. Los resultados se confirmaron utilizando ratones transgénicos Krt1-14;mTmG, con un número bajo (8,6 células GFP+ nativas por 106 células analizadas; 0,085% de células viables), pero números significativos (p < 0,0005) de células GFP+ presentes en la BM murina normal, que no fueron el resultado de la aleatoriedad en comparación con múltiples controles negativos. Además, las células EpCAM + en la sangre del ratón fueron más heterogéneas que las células CD45 + (0.58% en BM; 0.13% en sangre). Estas observaciones concluyen que las células que expresan proteínas citoqueratina son reproduciblemente detectables entre las células mononucleares de sangre humana y murina y BM. Demostramos un método de recolección de tejidos, citometría de flujo e inmunotinción que se puede utilizar para identificar y determinar la función de estas células epiteliales pancitoqueratina en individuos sanos.
Introducción
Las células epiteliales se encuentran en las barreras físicas entre nuestros cuerpos y el medio ambiente y son capaces de reconocer y responder a los cambios en su microambiente1. Tienen un nicho de células madre proliferantes que proporciona una forma de voltear tejido nuevo y reparar el daño2. Nuestro laboratorio estudia células madre en los folículos pilosos de la piel; La piel es un buen modelo para estudiar el tejido epitelial y la proliferación de células madre porque es fácilmente visible y hay una renovación constante de las células. Los cánceres epiteliales son la forma más común de cáncer, posiblemente debido a que los tejidos epiteliales, como la piel, son la primera línea de defensa contra los carcinógenos ambientales, lo que lleva a altas tasas de rotación y la proliferación de células epiteliales3. La mayor parte de la epidermis de la piel, la capa protectora superior, está compuesta por queratinocitos, que expresan diferentes tipos de queratinas para proporcionar soporte y estructura4. Los pacientes con cánceres epiteliales a menudo tienen células epiteliales presentes en la sangre y la médula ósea que también expresan queratinas. Las biopsias líquidas son una forma no invasiva de detectar y monitorizar estas células epiteliales en diferentes fluidos corporales5. Las células epiteliales circulantes, también llamadas células tumorales circulantes (CTC), se encuentran en la sangre periférica y pueden ser biomarcadores para el pronóstico del cáncer, así como guiar los tratamientos de terapia individualizados. Las CTC también pueden indicar la progresión de la enfermedad, la eficacia del tratamiento y la supervivencia general del paciente 5,6.
La molécula de adhesión de células epiteliales (EpCAM) es un marcador clínicamente utilizado para CTC y puede identificar tumores de origen epitelial en pacientes con cáncer. La EpCAM desempeña un papel en la adhesión, migración, señalización, proliferación y diferenciación celular6. Para que una célula epitelial circulante se clasifique como CTC, debe ser positiva para citoqueratinas 8, 18 y 19, y negativa para CD45, un marcador leucocitario común6. Las CTC generalmente se identifican mediante el agotamiento de CD45 con microperlas magnéticas, seguido de pruebas de EpCAM y citoqueratina 19 utilizando microscopía de inmunofluorescencia7. La principal limitación en la detección de CTC es su rareza; Constituyen menos del 0,01% de todas las células de la sangre, y muy pocas sobreviven en circulación para llegar a órganos distantes 8,9,10. Se debe tener en cuenta el diseño de experimentos y técnicas para aislar e identificar estas células debido a su naturaleza rara. Actualmente, solo hay un clasificador automatizado de células individuales aprobado por la Administración de Alimentos y Medicamentos (FDA) que se utiliza para identificar CTC, y utiliza EpCAM como biomarcador. Otros métodos incluyen la separación magnética de perlas y la citometría de flujo, o una combinación de estos métodos. Se necesitan nuevas técnicas que tengan mayor sensibilidad y especificidad para la detección de CTC raras11.
La citometría de flujo es un método preferido para la detección de poblaciones de células raras en la sangre, la médula ósea y otras muestras de tejido. Estas células raras pueden incluir células madre, células endoteliales circulantes, CTC y células de enfermedades residuales. La citometría de flujo permite mediciones cuantitativas de cada tipo de célula y clasifica estas células para realizar pruebas adicionales 7,9. Se realizaron recuentos incrementales para garantizar una evaluación precisa de estas células raras. La capacidad de usar la compuerta para excluir células de análisis posteriores es una forma de aumentar la especificidad al analizar células. Las limitaciones de la citometría de flujo son el tiempo requerido para el análisis de muestras grandes y la falta de confirmación visual de la identidad celular. Para superar esto, se realizó microscopía de inmunofluorescencia en las células clasificadas para confirmar sus identidades.
Anteriormente, nuestro laboratorio ha demostrado que en ratones las células de la médula ósea son reclutadas y contribuyen a los tumores de piel12. Estas células derivadas de la médula ósea son positivas para pancitoqueratina y citoqueratina epidérmica. Para dilucidar aún más el papel de las células madre epiteliales, las células derivadas de la médula ósea y las células que expresan citoqueratina en la progresión tumoral, se buscaron células positivas para citoqueratina EpCAM + en muestras normales de sangre y médula ósea murina y humana. Al igual que con la mayoría de los experimentos, este método se desarrolló a través de múltiples iteraciones. Anteriormente, se utilizaron ratones transgénicos junto con recuentos incrementales para buscar células que expresan K14GFP en la médula ósea12. A medida que se contaron más células, se pudo identificar una población representativa de células raras, como se muestra en la Figura 1 en la sección de resultados representativos. La justificación para investigar las células epiteliales en sangre y médula ósea normales se basó en la literatura temprana sobre CTC, donde los donantes sanos normales tenían niveles de fondo de células EpCAM+13. Como se mencionó anteriormente, la caracterización de EpCAM a menudo comienza con el agotamiento de CD45. Este paso se omitió porque algunas células hematopoyéticas tienen expresión de EpCAM y citoqueratina por razones desconocidas que deben investigarse más a fondo. Por lo tanto, las células se clasificaron en función de la presencia o ausencia de EpCAM, independientemente del linaje celular, y luego se inmunotiñeron para la citoqueratina. El siguiente protocolo y el flujo de trabajo que se muestra en la Figura 2 describen una técnica que utiliza citometría de flujo con compensación y controles, métodos estadísticos e imágenes de inmunofluorescencia para aislar e identificar estas poblaciones raras de células epiteliales.
Protocolo
Todos los protocolos con animales fueron aprobados por el Comité Institucional de Cuidado y Uso de Animales de la Universidad de Minnesota de acuerdo con los NIH y las pautas federales. La sangre humana fresca y la médula ósea se compraron de fuentes comerciales. La compañía recolectó muestras bajo un protocolo IRB aprobado de donantes negativos para VIH, hepatitis B, hepatitis C y se sometieron a pruebas de detección de COVID-19. Todas las muestras fueron primero desidentificadas y anonimizadas antes de ser enviadas por la compañía. Dado que estos se obtuvieron comercialmente, no se requirió la aprobación del IRB.
1. Preparación de soluciones
NOTA: Todas las soluciones deben prepararse en una campana biológica con un ambiente estéril. Obtener la certificación de nivel de bioseguridad 2 (BSL2) para el trabajo con sangre humana y médula ósea.
- Prepare la solución de recolección de médula ósea agregando 1 ml de gentamicina y 5 ml de suero fetal bovino (FBS) a 500 ml de solución salina de equilibrio de Hank (HBSS).
- Prepare la solución tampón de tinción agregando 50 ml de FBS a 500 ml de HBSS.
- Prepare 1x tampón de lisis combinando 10 ml de tampón de lisis 10x con 90 ml de agua estéril.
2. Preparación de la campana
- Encienda el capó y déjelo funcionar durante unos minutos para obtener un flujo de aire adecuado.
- Recolectar todos los materiales para realizar la recolección de médula ósea. Rocíelos dentro de la campana con etanol al 70% para mantener un ambiente estéril. Rocíe las manos cada vez antes de llevarlas dentro de la capucha.
- Coloque la bandeja esterilizada en autoclave en la capucha junto con tijeras esterilizadas en autoclave, un bisturí ensamblado, pinzas y pinzas curvas en una taza pequeña llena con etanol al 70% para mantenerlas estériles.
- Agregue 10 ml de solución de recolección de médula ósea a un tubo de centrífuga de 50 ml y etiquételo como "Extremidades". Etiquete otro tubo de centrífuga de 50 ml como "Médula ósea".
- Extraiga 10 ml de solución de extracción de médula ósea en una jeringa y luego coloque una aguja de 26 G.
3. Recolección de médula ósea de ratones
- Eutanasia del ratón mediante asfixia porCO2 . La muerte se confirma por la ausencia de latidos del corazón y pellizcar los pies para asegurarse de que no haya reacción. La luxación cervical puede realizarse como un medio secundario de eutanasia completa.
- Coloque el ratón en un recipiente de 500 ml y agregue suficiente yodo para cubrir la mitad del ratón. Agite suavemente y agite el recipiente para garantizar una cobertura completa. Enjuagar con agua desionizada. Realice un lavado más de yodo, y luego dos lavados con etanol al 70% siguiendo los mismos pasos.
- Coloque el ratón en la capucha y colóquelo boca arriba en la bandeja. Agarre las patas traseras y sepárelas hasta que se sienta un estallido; Esto es para separar las piernas de la columna vertebral.
- Haga una incisión de 1 cm en la piel del ratón cerca del área de la ingle. Inserte un par de tijeras cerradas en la incisión y luego abra las tijeras debajo de la piel para separarla del peritoneo.
- Corte la piel de la pierna alrededor del muslo y luego corte la piel por la pierna para exponer los músculos y los huesos.
- Retire las patas traseras cortando alrededor de la articulación de la cadera, asegurándose de no cortar a través del fémur. Coloque las extremidades en el tubo etiquetado como "Extremidades" Cuando se retiren ambas piernas, coloque el ratón en una bolsa de cadáver y póngalo en el congelador.
NOTA: El hueso de la cadera debe palparse primero antes de cortar para guiar las tijeras y evitar cortar el hueso del fémur, ya que la mayor parte de la médula ósea se obtiene del fémur. - Comience a cortar los músculos, el tejido y la grasa a lo largo de una de las extremidades con las tijeras. Cortar paralelo al hueso. Luego, use el bisturí y retire la grasa o músculo restante, usando un movimiento de raspado perpendicular contra el hueso.
- Una vez que el hueso se haya limpiado adecuadamente, separe el fémur y la tibia en la rodilla. Use el bisturí para hacer cortes en ambos extremos del fémur y la tibia donde no hay médula ósea visible.
- Inserte la jeringa y la aguja preparadas en el hueso. Si hay resistencia, corte un poco más del extremo del hueso hasta que no haya más resistencia.
- Mientras sostiene el hueso por encima del tubo etiquetado como "médula ósea", expulse la solución de recolección de médula ósea a través del hueso para enjuagar la médula ósea en el tubo. Repita en el otro extremo del hueso para obtener el resto de la médula ósea, hasta que el hueso aparezca todo blanco o vacío.
- Repita los pasos 7-9 para todos los huesos y extremidades restantes; Toda la médula se enjuagará en el mismo tubo cónico.
- Con una jeringa vacía de 10 ml y una aguja de 20 G, rompa los grupos de médula ósea en el tubo extrayendo la médula ósea hacia arriba y hacia abajo en la jeringa 5-10 veces.
- Agregue un filtro estéril de 40 μm a un tubo de centrífuga limpio de 50 ml y enjuáguelo con 1 ml de solución de recolección de médula ósea. Luego, filtre la médula ósea para eliminar cualquier grumo restante. Etiquete este tubo como "Médula ósea filtrada".
- Guarde la médula ósea filtrada en el refrigerador o en hielo hasta que la use. Debe usarse el mismo día de la cosecha. Si no se usa el mismo día, congele las células con un crioconservante, como dimetilsulfóxido (DMSO).
PRECAUCIÓN: Todos los residuos de estos experimentos deben eliminarse adecuadamente en un contenedor de residuos de riesgo biológico. Todos los objetos punzantes deben ir en un recipiente de objetos punzantes de riesgo biológico debidamente etiquetado.
4. Lisis de glóbulos rojos de la médula ósea
- Centrifugar las células de la médula ósea a 170 x g durante 10 min a temperatura ambiente. Aspire y deseche el sobrenadante. Resuspender las células en 10 ml de 1x tampón de lisis de glóbulos rojos e incubar durante 4 min.
- Agregue 30 ml de solución salina tamponada con fosfato de Dulbecco (DPBS) para detener la reacción del tampón de lisis. Centrifugar las células durante 8 min a 170 x g a temperatura ambiente. Aspire y deseche el sobrenadante. Resuspender en 10-20 mL de tampón de tinción.
5. Recuento de células
- Combine 500 μl de células con 9,5 ml de solución de recolección de médula ósea para obtener una dilución 1:20.
NOTA: Esto se utilizará para el recuento de células y no necesita mantenerse estéril. - Extraiga 200 μL de células de la dilución 1:20, agréguelas a 200 μL de azul de tripano y mezcle bien.
- Agregue la mezcla a un hemocitómetro y cuente las células vivas y muertas en ambos lados. Calcule la suspensión de celdas original usando esta tabla (que se muestra en el Archivo Suplementario 1) y las ecuaciones:
Células por mililitro: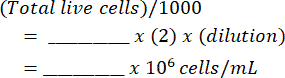
Total de celdas: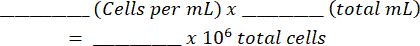
- Girar las células a 170 x g durante 10 min a 4 °C y resuspender hasta una concentración de 10 x 10 6 por 1 ml (1 x 106 células por 100 μL), según las sugerencias del fabricante para diluciones de anticuerpos.
- Proceda a la citometría de flujo (paso 8).
6. Recolección de sangre de ratones
- Eutanasia al ratón usando gasCO2 o dislocación cervical.
- Realice una extracción de sangre cardíaca en un ángulo de 45° con una aguja de 26 G conectada a una jeringa de 1 ml recubierta con ácido etilendiaminotetraacético (EDTA) 0,5 M.
- Gire la sangre y vuelva a suspenderla en medios o tampón de tinción.
NOTA: Las células se girarán y se resuspenderán a la concentración final en el tampón de tinción después de la lisis de glóbulos rojos y el recuento de células. - Realice la lisis de glóbulos rojos (paso 4).
- Realice el recuento de celdas (paso 5).
- Proceda a la citometría de flujo (paso 8).
7. Procesamiento de muestras de sangre humana y médula ósea
- Realice todos los procedimientos bajo una capucha certificada BSL2 mientras usa equipo de protección personal.
- Recoja las muestras la noche anterior y realice la lisis de glóbulos rojos. Asegúrese de que las muestras sean anónimas y anónimas antes de enviar las muestras mononucleares durante la noche en hielo.
- A su llegada, haga girar las células vivas a 170 x g a 4 °C durante 10 min.
NOTA: Estas son muestras BSL2 que deben seguir las pautas locales. - Resuspender las células en medios o tampón de tinción.
NOTA: Las células deben girarse y resuspenderse a la concentración final después del recuento celular con tampón de tinción. - Realice el recuento de celdas (paso 5).
- Proceda a la citometría de flujo (paso 8).
8. Citometría de flujo
- Tinción
- Divida las células en tubos marcados individualmente según el panel de tinción utilizado, incluidos los controles de compensación (sin teñir, controles de isotipo, teñidos únicos y fluorescencia menos los [FMO]). Congele las células restantes o utilícelas para la clasificación celular activada por fluorescencia. Ver Tabla 1 para un ejemplo de un panel de tinción con las diluciones de anticuerpos utilizadas.
NOTA: Por cada fluoróforo adicional agregado, se deben agregar FMO en combinación con fluoróforos restantes junto con una sola tinción de ese fluoróforo. Los controles de isotipo se pueden utilizar para bloquear la tinción no específica. Cada nuevo anticuerpo utilizado debe ser titulado con las muestras para una dilución óptima. - Agregue anticuerpos de acuerdo con las diluciones recomendadas por el fabricante y mezcle bien moviendo la parte inferior de los tubos. Las concentraciones de anticuerpos generalmente se administran como 1 x 106 células por 100 μL. Las diluciones con anticuerpos y controles se muestran en la Tabla 1. Los anticuerpos se conjugan con fluoróforos y deben protegerse de la luz.
NOTA: Es mejor utilizar EpCAM conjugada con ficoeritrina (PE) para permitir la visualización de poblaciones bajas de células (título de 3-5 μL por 1 millón de células, como se indica en la Tabla 1). PE-CD49f se utilizó como un control de PE de tinción única (20 μL por 1 millón de células), ya que estas células son más prevalentes que PE-EpCAM y, por lo tanto, más fáciles de usar para la compensación. - Incubar durante 30 min a 4 °C en la oscuridad.
- Después de la incubación, vuelva a colocar los tubos en la campana BSL2 y agregue 1 ml de tampón de tinción a cada tubo.
- A continuación, centrifugar los tubos a 170 x g a 4 °C durante 5-10 min.
- Después de la centrifugación, lleve los tubos tapados con las células de nuevo a la campana BSL2 y aspire el sobrenadante. Asegúrese de cambiar las puntas de la pipeta de aspiración entre cada tubo para evitar la contaminación cruzada.
- Vuelva a suspender el pellet celular en 1 ml de tampón de tinción y mueva la parte inferior de los tubos para mezclar.
- Lave las celdas dos veces más repitiendo los pasos 8.1.4-8.1.7.
NOTA: No utilice pipetas para resuspender, para evitar la pérdida de células. - Resuspender en 500 μL de tampón de tinción para la resuspensión final antes de la citometría de flujo. Agregue 4′,6-diamidino-2-fenilindol (DAPI) o discriminador de células muertas, de acuerdo con las recomendaciones del fabricante.
- Divida las células en tubos marcados individualmente según el panel de tinción utilizado, incluidos los controles de compensación (sin teñir, controles de isotipo, teñidos únicos y fluorescencia menos los [FMO]). Congele las células restantes o utilícelas para la clasificación celular activada por fluorescencia. Ver Tabla 1 para un ejemplo de un panel de tinción con las diluciones de anticuerpos utilizadas.
- Citómetro de flujo
- Después de configurar la máquina, utilice los controles y FMO para configurar la compensación y las puertas. Use la solución salina balanceada de Hank (HBSS) como líquido de la vaina para ayudar a sostener las células frágiles. Utilice PBS si HBSS no está disponible.
NOTA: El citómetro de flujo utilizado y el software se enumeran en la Tabla de materiales. - Cargue las muestras de una en una y recopile puntos de datos para 50.000, 100.000, 500.000 y un millón de eventos. Mantenga los tubos en hielo o refrigerados hasta su uso.
NOTA: Se puede establecer un límite de tiempo al recolectar para garantizar la viabilidad de la muestra. - Recoja las células en 3 ml de RPMI con 10% de FBS en tubos de centrífuga de 15 ml.
- Si realiza inmunofluorescencia en células EpCAM+, use el citómetro de flujo para clasificar de 800 a 1,000 células en cada pocillo de un portaobjetos de 8 pocillos.
- Clasifique la población de células EpCAM en portaobjetos separados como un control negativo para usar cuando se tiñe la inmunofluorescencia.
NOTA: Las células también se pueden clasificar en tubos de 15 ml que contienen FBS y girar en un portaobjetos usando la citocentrífuga o una pipeta para evitar que las células se rompan. - Fijar los portaobjetos en una mezcla de metanol/50% de acetona a -20 °C durante 10 min. Conservar los portaobjetos a -20 °C hasta que se tiñan con inmunofluorescencia.
- Después de configurar la máquina, utilice los controles y FMO para configurar la compensación y las puertas. Use la solución salina balanceada de Hank (HBSS) como líquido de la vaina para ayudar a sostener las células frágiles. Utilice PBS si HBSS no está disponible.
- Análisis de citometría de flujo
- Abra el software de análisis de citometría de flujo con licencia.
- Archivos estándar de citometría de flujo de carga (FCS) adquiridos del citómetro de flujo con controles y muestras.
- Haga clic en Crear grupo y use palabras clave para colocar muestras y controles en un grupo .
NOTA: Utilice el control "sin mancha" para compuerta en el software de análisis de citometría de flujo, como se muestra en la Figura 3. - Haga doble clic en la muestra de control sin manchas para abrir un gráfico sin cifrar.
- Establezca el eje y del gráfico en SSC-A y el eje x en FSC-A (área de dispersión lateral por área de dispersión hacia adelante), lo que indica el tamaño de la celda y la complejidad interna.
- Haga clic en la forma del polígono en el panel superior izquierdo y cree una puerta alrededor de las celdas. Asegúrese de excluir las células en la periferia, ya que lo más probable es que sean desechos. Etiquete esta puerta como "Celdas", como se muestra en la figura 3A.
- Una vez que las celdas estén cerradas, haga doble clic dentro de la forma del polígono para abrir otro gráfico en una nueva ventana.
- Establezca el eje y del nuevo gráfico en SSC-A y el eje x en SSC-W para la discriminación doble. Haga clic en la forma del polígono y cree un rectángulo alrededor de las celdas individuales. Etiquete esto como "Celdas individuales", como se muestra en la Figura 3B.
- Una vez que las celdas estén cerradas, haga doble clic dentro de la forma del polígono para abrir otro gráfico en una nueva ventana.
- Establezca el eje y en FSC-A y el eje x en DAPI-A (o el discriminador de celda muerta que se utilice).
- Cree una puerta poligonal alrededor de las celdas negativas DAPI en el lado izquierdo del gráfico. Etiquete estas "células vivas", como se muestra en la Figura 3C.
- Una vez que las celdas estén cerradas, haga doble clic dentro de la forma del polígono para abrir otro gráfico en una nueva ventana.
- Establezca el eje y en SSC-A y el eje x en EpCAM-PE (o cualquier fluoróforo que se utilice para identificar las células EpCAM). Las celdas de la izquierda son negativas para EpCAM. Si se muestra alguna celda a la derecha, son EpCAM positivas.
- Utilice el gráfico EpCAM-PE para crear una puerta poligonal, que excluye las celdas de la izquierda y solo incluye el espacio vacío de la derecha. Etiquete el área vacía a la derecha dentro del polígono "EpCAM", como se muestra en la Figura 3D.
NOTA: Este es un control sin manchas, por lo que no debe haber expresión EpCAM. Utilice el control sin tinción para determinar qué células son positivas para EpCAM. Esto completa la estrategia de cierre para la compensación, por lo que todos los gráficos pueden cerrarse en este momento. - En la página del espacio de trabajo, resalte el linaje sin manchas con "Células", "Células individuales", "Células vivas" y "EpCAM". Haga clic derecho y seleccione copiar análisis para agrupar. Esto copia la estrategia de acceso a todas las demás muestras cargadas dentro del grupo creado anteriormente. Si un grupo no se creó inicialmente, se puede crear ahora seleccionando Crear grupo en la parte superior izquierda.
- Revise las muestras dentro del grupo y verifique las puertas de polígono dibujadas previamente para asegurarse de que encajan con todas las muestras y controles. Esto proporciona el número porcentual y el número de celdas.
- Haga clic en el botón L del editor de diseño en la esquina superior izquierda sobre "Espacio de trabajo". Estos gráficos se pueden organizar y exportar utilizando el editor de diseño.
- Preparación de reactivos de inmunofluorescencia
- Prepare el diluyente de anticuerpos combinando 1 g de albúmina sérica bovina (BSA), 2 g de leche descremada, 10 ml de solución salina tamponada con tris (TBS) 10 x , 100 μL de Tween y 90 ml de agua destilada.
- Prepare TBST combinando 50 ml de 20x TBS, 950 ml de agua destilada y 200 μL de Tween.
- Preparar suero normal de caballo al 10% (NHS) combinando 1 ml de NHS con 9 ml de diluyente de anticuerpos (paso 8.4.1).
- Prepare 1% NHS combinando 1 ml de 10% NHS con 9 mL de TBST.
- Tinción por inmunofluorescencia
- Retire los portaobjetos a -20 °C y deje que se calienten a temperatura ambiente.
- Lave los toboganes tres veces en agua destilada durante 5 minutos cada una.
- Bloquear los portaobjetos durante 1 h a temperatura ambiente en NHS al 10% en diluyente de anticuerpos (paso 8.4.3).
- Diluir el anticuerpo primario (pancitoqueratina) a 1:750 en NHS al 1% en TBST (paso 8.4.4). Incubar portaobjetos en el anticuerpo primario diluido durante la noche a 4 °C.
- Al día siguiente, lave las diapositivas tres veces con un tampón de lavado 1x (PBS o TBST) durante 5 minutos cada una.
- Diluir el anticuerpo secundario a 1:1.000 en NHS al 1% en TBST (paso 8.4.4). Incubar los portaobjetos en el anticuerpo secundario diluido durante 1 h a temperatura ambiente.
- Lave las diapositivas tres veces con 1x tampón de lavado durante 5 minutos cada una. Monte y cubra las guías con soportes de montaje rígidos con DAPI. Use un hisopo de algodón para extender suavemente cualquier burbuja debajo del cubreobjetos.
Resultados
Usando estos métodos, se visualizaron poblaciones raras de células epiteliales en la sangre y la médula ósea de humanos y ratones normales. Con las compensaciones y controles adecuados como se describe, los resultados consistentes que mostraron que 4% -5% de las células en la médula ósea murina eran EpCAM +, independientemente de cuántas células se contaron, como se muestra en la Figura 4 y la Figura 5. En muestras de sangre murinas, menos del 0,5% de las células eran EpCAM+, como se muestra en la Figura 6. En muestras de médula ósea humana, el 2% -5% de las células eran EpCAM+, como se muestra en la Figura 7 y la Figura 8. Si bien 2% -5% es un rango grande, los porcentajes dentro de cada donante individual fueron consistentes a medida que se contaron gradualmente más células. En muestras de sangre humana, alrededor del 0,3% de las células eran EpCAM+, como se muestra en la Figura 9. Nuestras muestras de control (sin tinción, control de isotipo y FMO) arrojaron muy pocos resultados falsos positivos de EpCAM+, como se muestra en la Figura 4 y la Figura 7. Las células de los grupos EpCAM+ y EpCAM- que se clasificaron en portaobjetos mostraron tinción positiva para pancitoqueratina en muestras de EpCAM+, y fueron negativas para pancitoqueratina en muestras de EpCAM-, como se muestra en la Figura 10. Estos resultados indican que los experimentos fueron diseñados adecuadamente y reproducibles.
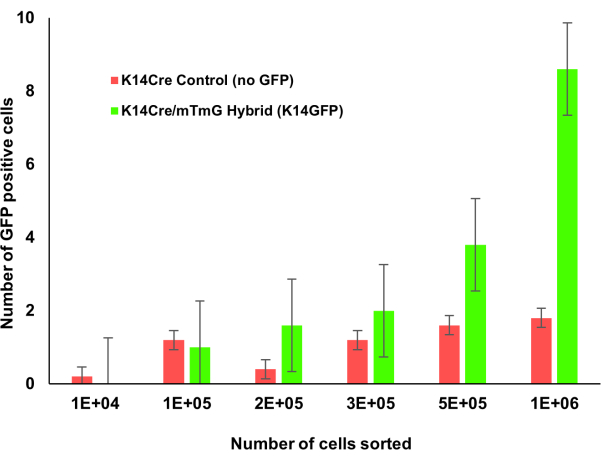
Figura 1: Ratones transgénicos Krt14Cre;mTmG con recuentos incrementales de médula ósea. La médula ósea de ratones Krt1-14;mTmG se contó incrementalmente. Las células positivas para GFP indican expresión de queratina 14 y se identificaron mediante citometría de flujo. A medida que se contaron más células de médula ósea, esta población de células positivas para queratina 14 fue más fácilmente identificable. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Flujo de trabajo para células EpCAM+ y citoqueratina+. La médula ósea y las células sanguíneas se clasificaron primero utilizando la clasificación de células activadas por fluorescencia (FACS) para separar las células EpCAM + y EpCAM-. Estas células se clasificaron en dos tubos de ensayo diferentes, así como en dos portaobjetos diferentes. Las células clasificadas en tubos de ensayo se hicieron girar en portaobjetos utilizando una citocentrífuga. Luego, los portaobjetos se tiñeron con un anticuerpo primario pancitoqueratina y luego se tiñeron con un anticuerpo secundario. Los portaobjetos se analizaron mediante microscopía de fluorescencia para observar la expresión de pancitoqueratina. Haga clic aquí para ver una versión más grande de esta figura.
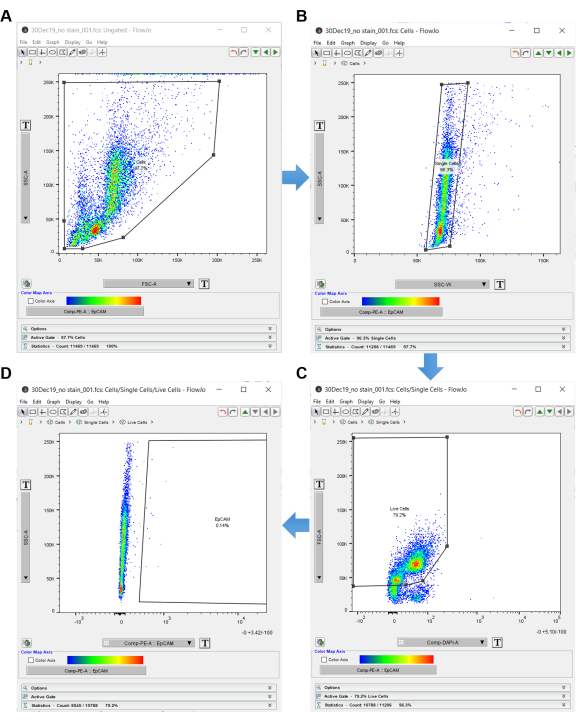
Figura 3: Software de análisis de citometría de flujo. Al analizar los datos de citometría de flujo utilizando un software de análisis, se utiliza el control sin tinción para seleccionar las células de interés. Las células se seleccionan primero con SSC-A y FSC-A, que muestran la complejidad interna y el tamaño de las células. (A) Se dibuja una puerta de polígono alrededor de las celdas. (B) Las células individuales son adquiridas por SSC-W por SSC-W. (C) Las células vivas se adquieren mediante la activación FSC-A frente a DAPI. (D) Las células EpCAM negativas se excluyen mediante gating a la derecha de las células EpCAM negativas. Haga clic aquí para ver una versión más grande de esta figura.
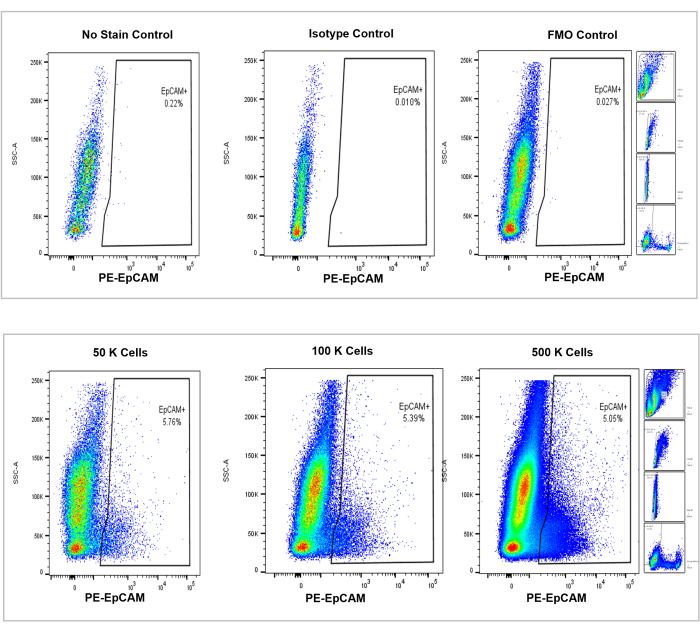
Figura 4: Análisis citométrico de flujo de células EpCAM+ en médula ósea de ratón. Los controles de no tinción, isotipo y FMO se muestran en el panel superior, con los recuentos incrementales de 50,000, 100,000 y 500,000 celdas mostrados en el panel inferior. Estos gráficos visualizan la consistencia en los porcentajes entre los recuentos, a pesar del aumento general en el total de celdas contadas. Los paneles de la derecha indican la estrategia de compuerta, como se discutió anteriormente en la sección de análisis de citometría de flujo y como se muestra en la Figura 3. Haga clic aquí para ver una versión más grande de esta figura.
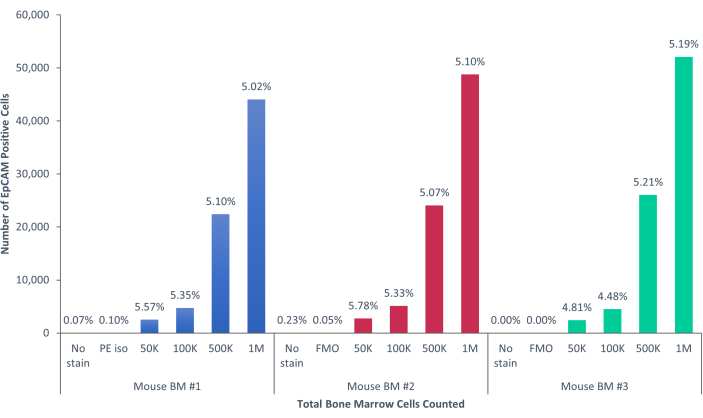
Figura 5: Las células EpCAM+ en médula ósea de ratón comprenden el 5,17% ± el 0,001% de la población. Se analizaron las células de la médula ósea de tres ratones individuales. Se incluyeron los controles apropiados para un adecuado rigor científico. El porcentaje de células positivas se mantuvo constante en todas las muestras, debido a que los ratones eran genéticamente idénticos. Haga clic aquí para ver una versión más grande de esta figura.
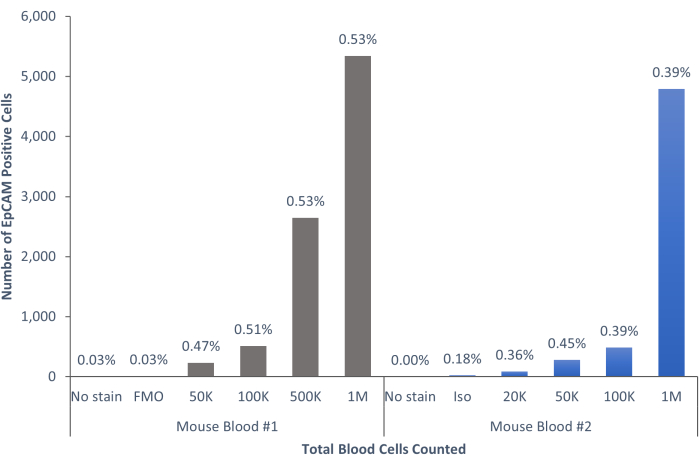
Figura 6: Las células EpCAM+ en sangre de ratón comprenden el 0,45% ± el 0,0006% de la población. Se analizaron las células sanguíneas de dos ratones individuales. Se incluyeron controles para mostrar que se siguió el procedimiento adecuado para producir resultados concluyentes. Haga clic aquí para ver una versión más grande de esta figura.
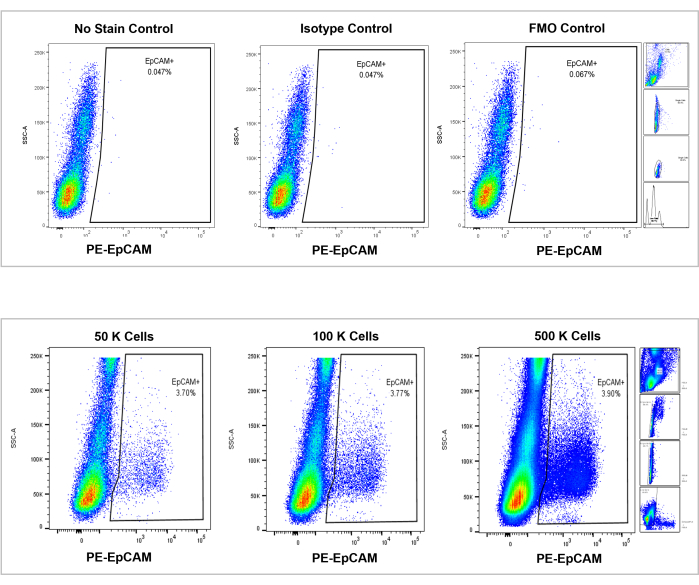
Figura 7: Análisis citométrico de flujo en médula ósea humana EpCAM+. Los controles de no tinción, isotipo y FMO se muestran en el panel superior, y los recuentos incrementales de 50,000, 100,000 y 500,000 celdas se muestran en el panel inferior. Estos gráficos visualizan la consistencia en los porcentajes entre los recuentos, a pesar del aumento general del total de celdas contadas. Los paneles de la derecha indican la estrategia de compuerta, como se discutió anteriormente en la sección de análisis de citometría de flujo y como se muestra en la Figura 3. Haga clic aquí para ver una versión más grande de esta figura.

Figura 8: Las células EpCAM+ en la médula ósea humana comprenden el 3,53% ± el 0,006% de la población. Se analizaron tres muestras diferentes de médula ósea humana. Se incluyeron controles apropiados para el rigor científico. El porcentaje de células positivas varía debido a la heterogeneidad genética entre los seres humanos. Haga clic aquí para ver una versión más grande de esta figura.
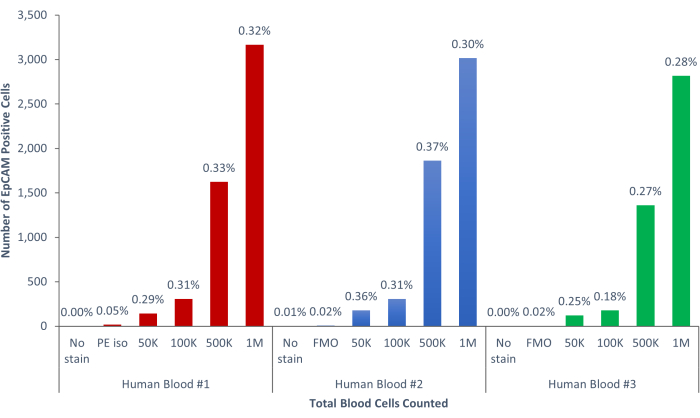
Figura 9: Las células EpCAM+ de la sangre humana comprenden el 0,18% ± el 0,0004% de la población. Se analizaron tres muestras diferentes de sangre humana. Se incluyeron controles apropiados para el rigor científico. Haga clic aquí para ver una versión más grande de esta figura.
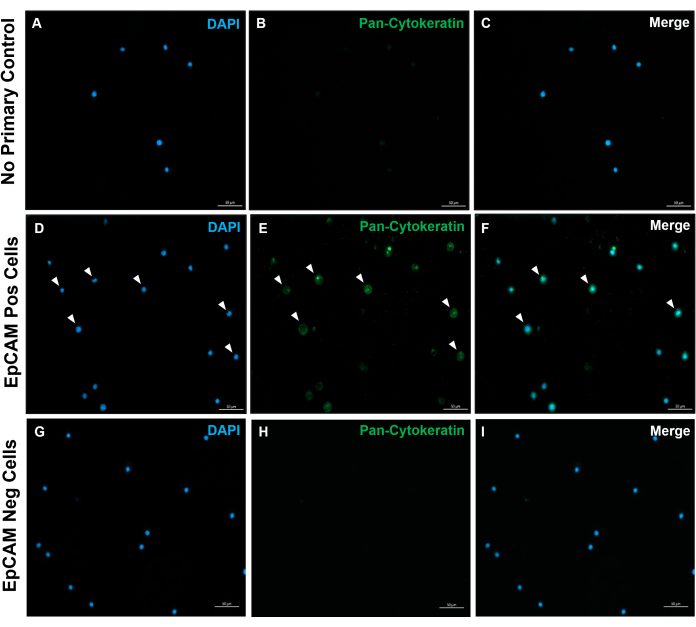
Figura 10: Inmunofluorescencia de portaobjetos EpCAM+ y EpCAM. Se utilizó FACS para separar las células EpCAM+ y EpCAM-. La pancitoqueratina se tiñó para usar el anticuerpo pancitoqueratina DAKO. No se utilizó control primario de anticuerpos en suero normal, ya que la pancitoqueratina es un anticuerpo policlonal. Estos resultados confirman la exactitud del sistema de control de los bienes sobre el terreno. Haga clic aquí para ver una versión más grande de esta figura.
| Médula ósea humana o sangre | |||
| Anticuerpo | Tubo # | # de Celdas | AB Conc |
| Sin manchas | 1 | 1x106 | X |
| Solo DAPI | 2 | 1x106 | 1uL/mL |
| Control del isotipo PE | 3 | 1x106 | 1 uL |
| CD49f-PE Control de tinción única | 4 | 1x106 | 20 uL en 100uL por 1x106 |
| EpCAM-PE baja titulación | 5 | 1x106 | 3 uL en 100uL por 1x106 |
| Titulación media EpCAM-PE | 6 | 1x106 | 4 uL en 100uL por 1x106 |
| Alta titulación EpCAM-PE | 7 | 1x106 | 5 uL en 100uL por 1x106 |
| EpCAM-PE High Sort en portaobjetos | 8 | 10x106 | 50 uL en 1 ml |
Tabla 1: Panel de tinción por citometría de flujo. Un ejemplo de un panel de tinción de citometría de flujo para células mononucleares de sangre o médula ósea. Los controles incluidos son DAPI solamente, control no teñido y control de tinción única de PE (PE-CD49f se utilizó como un mejor control positivo). La fluorescencia menos uno no está incluida en este panel, ya que es de un solo color (PE). Al agregar más colores fluoróforos, como isotiocianato de fluoresceína (FITC) o aloficocianina (APC), se deben incluir FMO excluyendo un fluoróforo para cada control FMO.
Archivo complementario 1: Conteo de células vivas con hematocitómetro. Haga clic aquí para descargar este archivo.
Discusión
Existe alguna evidencia en la literatura de la presencia de células epiteliales en la médula ósea. Anteriormente, los artículos han investigado típicamente el papel de las células epiteliales en el contexto de enfermedades y lesiones, como en el hígado, los pulmones, el tracto gastrointestinal (GI), el timo y la piel14,15,16,17. Sin embargo, no se sabe mucho sobre la presencia de estas células epiteliales en la médula ósea de individuos sanos. Este trabajo busca establecer un método reproducible, con el objetivo de identificar y aislar células epiteliales de sangre y médula ósea normales. Este método impulsará el campo hacia adelante para identificar por qué las células epiteliales están presentes y cuál es su papel en la sangre y la médula ósea en ausencia de enfermedad; Tal vez estas células son parte del mantenimiento normal del tejido o se activan en momentos de lesión. La médula ósea es un depósito de células madre; sin embargo, no está claro cuál puede ser el linaje de estas células epiteliales. Un artículo reciente discute las células epiteliales derivadas de la médula ósea en el timo, que primero expresa EpCAM y el marcador hematopoyético CD45, y luego pierde su expresión de CD45 con el tiempo después de la lesión15. La investigación de nuestro laboratorio también ha confirmado la presencia de células CD45+ EpCAM+ en sangre sana y médula ósea en ausencia de lesión12. Sin embargo, el papel de estas células aún no se ha determinado.
Se necesitaba un método reproducible para examinar las células epiteliales dentro de la médula ósea sana. El método descrito ayudará a caracterizar estas células aún más en su estado normal. Hay pasos importantes en este método para mantener la reproducibilidad. Uno de los pasos más críticos en este protocolo es mantener un ambiente estéril en la capucha mientras se cosecha la médula ósea de los ratones. Si se cosecha de varios ratones, se evita la contaminación cruzada entre muestras mediante el uso de nuevas agujas y jeringas para cada ratón. Esto también asegura que la muestra esté limpia y libre de cualquier contaminante que pueda afectar los resultados. Además, se debe aumentar el número de jeringas preparadas y tubos cónicos etiquetados para cada ratón adicional. Otro paso importante consiste en enjuagar los huesos hasta que se muestre un color blanco limpio para garantizar que se hayan eliminado la mayoría de las células de la médula ósea; Las posibilidades de detectar células raras aumentan en una muestra más pura. Se realizó una modificación para optimizar el protocolo de lisis de glóbulos rojos; Se probaron varios tampones de lisis para encontrar el correcto que diera una viabilidad consistentemente alta, ya que casi la mitad de las células se perdían durante este paso. Es posible que sea necesario optimizar diferentes reactivos y períodos de incubación para obtener mejores resultados en otros entornos.
La limitación más significativa de este protocolo es el uso de citometría de flujo para encontrar poblaciones de células raras. Como se discutió anteriormente, la adición de controles y recuentos incrementales ayuda a aumentar la especificidad y precisión del análisis. Otra limitación es que los marcadores apropiados para la población de interés deben identificarse de antemano. Por lo tanto, es necesario conocer los marcadores clave, los anticuerpos para la citometría de flujo y los anticuerpos compatibles con la especie objetivo.
Estos métodos son una mejora con respecto a los métodos existentes, ya que permiten el análisis de células individuales a una fracción del costo del aislador automático de CTC existente y la secuenciación de ARN de una sola célula. Además, la citometría de flujo está más fácilmente disponible. FACS mantuvo una mayor viabilidad para las células en comparación con los resultados informados anteriormente utilizando la separación magnética de microperlas. Por último, estas técnicas permiten la separación de células para análisis posteriores, como la secuenciación masiva de ARN, la secuenciación de ARNsc o el cultivo celular.
Divulgaciones
No se conocen conflictos de intereses.
Agradecimientos
Josh Monts, técnico del citómetro de flujo de las instalaciones centrales, Instituto Hormel
Todd Schuster, Gerente de Instalaciones Principales, The Hormel Institute
Derek Gordon, Estadístico, Universidad de Rutgers
Nos gustaría agradecer a Karen Klein de Clarus Editorial Services, Santa Fe, NM por su asistencia editorial.
Este trabajo fue apoyado en parte por el Instituto Nacional de Artritis y Enfermedades Musculoesqueléticas y de la Piel de los Institutos Nacionales de Salud bajo el Número de Premio R21 AR075281, y una Subvención en Ayuda de Investigación, Arte y Beca, Oficina del Vicepresidente de Investigación, Universidad de Minnesota (Propuesta # 324240). Agradecemos el apoyo del Instituto Hormel.
Materiales
| Name | Company | Catalog Number | Comments |
| 40 μm Cell Strainer | Falcon | 352340 | Used to filter out any large clumps of cells from bone marrow |
| 5 mL Polystyrene Round-bottom tube | Falcon | 352054 | Tubes used for flow cytometry |
| 500 mL jar with lid | Nalgene | 11-823-32 | Used to wash the mouse |
| 190 proof Ethanol | Decon laboratories Inc | 2801 | Diluted to 70% with dH2O |
| Alexa Fluor 488 Goat anti-rabbit IgG (H+L) | Invitrogen | A11008 | Dilution: 1:1000 |
| Brightline Hemacytometer | Hausser Scientific | 02-671-10 | Used to count alive cells |
| Bovine Serum Albumin | Jackson ImmunoResearch | 001-000-162 | Component of antibody diluent |
| Characterized Fetal Bovine Serum (FBS) | Hyclone | SH30071.03 | Component of staining buffer and harvesting solution |
| Curity gauze sponges | Covidien | 2187 | Used to keep a clean work area when harvesting limbs and bone marrow |
| Curved Forceps | Miltex | 18-784 | Used during harvest |
| Cytokeratin, Wide Spectrum Screening | Dako | Z0622 | Dilution 1:750 *Now discontinued |
| Dulbecco's Phosphate Buffered Saline (DPBS 1x) | Gibco | 14190-144 500mL | Used to stop the red blood cell lysis reaction |
| Ethylenediaminetetraacetic acid | Sigma Aldrich | E9884-100G | Used at 0.5 M |
| Gentamicin sulfate | Lonza | 17-518Z | Component of harvesting solution |
| Hank's Balanced Salt Solution (HBSS 1x) | Gibco | 14175-095 500mL | Component of harvesting solution and staining buffer solution |
| Luer-Lok tip 10 mL syringe | Becton, Dickinson and Co | 309604 | Used with 26G needle to flush bones, used with 20G needle to break up clumps |
| Magic touch 2 ice bucket | BelArt | M16807-2001 | Used to store the specimens on ice |
| Nonfat dry milk | Apex | 20-241 | Component of Antibody Diluent |
| Normal horse serum | Vector | ZE0122 | Component of Antibody Diluent |
| PE anti-human CD 326 (EpCAM) | Biolegend | 324206 | Dilution: 5 µl in 100 µl per 1x106 cells |
| PE anti-mouse CD 326 (EpCAM) | Biolegend | 118205 | Dilution: 3 µl in 100 µl per 1x106 cells |
| PE mouse IgG2b, κ isotype control | Biolegend | 400313 | Dilution: 1 µl in 100 µl per 1x106 cells |
| PE rat anti-human CD49f | BD Biosciences | 555763 | Dilution: 20 µl in 100 µl per 1x106 cells |
| PE rat IgG2am, κ Isotype control | Biolegend | 400508 | Dilution: 1 µl in 100 µl per 1x106 cells |
| Polypropylene Conical Centrifuge Tubes 50 mL | Basix | 14-955-240 | Used in the centrifuge |
| Povidone-Iodine Scrub | Aplicare | 82-227 | Antiseptic used to sterilize the mice |
| PrecisionGlide Needle 20G x 1 1/2 | Becton, Dickinson and Co | 305176 | 20G needle used to break up bone marrow clumps |
| PrecisionGlide Needle 26G x 1/2 | Becton, Dickinson and Co | 305111 | 26G needle used to flush bone marrow from bones |
| PTFE Printed Slides | Electron Microscopy Services | 63422-06 | 8 well slides cells sorted onto for immunofluorescence |
| RBC Lysis buffer 10X | Invitrogen | 00-4300-54 | Dilution 1:10 using sterile deionized water |
| Scissors | Roboz | RS-6762 | Used during harvest |
| Stainless steel surgical blade #4 | Bard-Parker | 371222 | Used during harvest |
| Sterile tray | Polar ware | 10F | Used during harvest |
| StretchEase Powder-free nitrile examination gloves | Denville Scientific | G4161 | Used during harvest |
| Surgical Scalpel handle #4 | Fischer | 12-000-164 | Used with surgical blade |
| TBS 20x | Thermo | Component of TBST, used at 1X | |
| Trypan Blue Stain (0.4%) | Gibco | 15250-061 | Used to count dead cells. Filtered with .45 µm |
| Tween 20 | Sigma Aldrich | P1379-500mL | Component of TBST |
| Tweezers | Miltex | 6-8 | Used during harvest |
| Vectashield Vibrance Antifade Mounting Medium with DAPI | Vector | H-1800 | Nuclear stain for immunofluorescence |
Referencias
- Larsen, S. B., Cowley, C. J., Fuchs, E. Epithelial cells: liaisons of immunity. Current Opinion in Immunology. 62, 45-53 (2020).
- Blanpain, C., Fuchs, E. Plasticity of epithelial stem cells in tissue regeneration. Science. 344 (6189), 1242281 (2014).
- Coradini, D., Casarsa, C., Oriana, S. Epithelial cell polarity and tumorigenesis: New perspectives for cancer detection and treatment. Acta Pharmacologica Sinica. 32 (5), 552-564 (2011).
- Dmello, C., et al. Multifaceted role of keratins in epithelial cell differentiation and transformation. Journal of Biosciences. 44 (2), 33 (2019).
- Poulet, G., Massias, J., Taly, V. Liquid biopsy: General concepts. Acta Cytologica. 63 (6), 449-455 (2019).
- Eslami-S, Z., Cortés-Hernández, L. E., Alix-Panabières, C. Epithelial cell adhesion molecule: an anchor to isolate clinically relevant circulating tumor cells. Cells. 9 (8), 1836 (2020).
- Morris, R. J. Circulating tumor cells: quintessential precision oncology presenting challenges for biology. NPJ Precision Oncology. 1 (1), 16 (2017).
- Lin, D., et al. Circulating tumor cells: biology and clinical significance. Signal Transduction and Targeted Therapy. 6 (1), 404 (2021).
- Spence, M., Adai, S., Landon, T., Richter, G. Few and far between: Tools and strategies for rare-event detection using flow cytometry. BioprobesJournal of Cell Biology. 71, 14-18 (2015).
- Yu, M., Stott, S., Toner, M., Maheswaran, S., Haber, D. A. Circulating tumor cells: approaches to isolation and characterization. Journal of Cell Biology. 192 (3), 373-382 (2011).
- Ye, Q., Ling, S., Zheng, S., Xu, X. Liquid biopsy in hepatocellular carcinoma: circulating tumor cells and circulating tumor DNA. Molecular Cancer. 18 (1), 114 (2019).
- Park, H., et al. marrow-derived epithelial cells and hair follicle stem cells contribute to development of chronic cutaneous neoplasms. Nature Communications. 9 (1), 5293 (2018).
- Marsman, W. A., et al. Epithelial cells in bone marrow: do they matter. Gut. 54 (12), 1821-1822 (2005).
- Borue, X., et al. marrow-derived cells contribute to epithelial engraftment during wound healing. The American Journal of Pathology. 165 (5), 1767-1772 (2004).
- Chakrabarti, S., et al. marrow-derived cells contribute to the maintenance of thymic stroma including TECs. Journal of Immunology Research. 2022, 6061746 (2022).
- Krause, D. S., et al. Multi-organ, multi-lineage engraftment by a single bone marrow-derived stem cell. Cell. 105 (3), 369-377 (2001).
- Wong, A. P., et al. Targeted cell replacement with bone marrow cells for airway epithelial regeneration. American Journal of Physiology-Lung Cellular and Molecular Physiology. 293 (3), L740-L752 (2007).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados