Method Article
Données probantes sur l’expression d’EpCAM et de cytokératine dans le sang et la moelle osseuse humains et murins normaux
Dans cet article
Résumé
Cet article présente une méthode reproductible avec de nouvelles découvertes sur la présence de cellules épithéliales dans le sang et la moelle osseuse humains et souris normaux en utilisant la cytométrie en flux et la microscopie d’immunofluorescence. Des souris transgéniques Krt1-14;mTmG ont été utilisées comme méthode in vivo pour confirmer ces résultats.
Résumé
Des cellules épithéliales ont été identifiées dans le sang et la moelle osseuse de patients atteints de cancer et d’autres maladies. Cependant, la présence de cellules épithéliales normales dans le sang et la moelle osseuse d’individus en bonne santé n’a pas encore été identifiée de manière cohérente. Présenté ici est une méthode reproductible pour isoler les cellules épithéliales du sang humain et murin sain et de la moelle osseuse (BM) en utilisant la cytométrie de flux et la microscopie d’immunofluorescence (IF). Les cellules épithéliales chez des individus sains ont d’abord été identifiées et isolées par cytométrie de flux à l’aide d’une molécule d’adhésion cellulaire épithéliale (EpCAM). Il a été confirmé que ces cellules EpCAM+ expriment de la kératine par microscopie d’immunofluorescence chez des souris transgéniques Krt1-14;mTmG. Les échantillons de sang humain contenaient 0,18 % ± 0,0004 cellules EpCAM+ (MEB ; n = 7 répétitions biologiques, 4 répétitions expérimentales). Dans la BM humaine, 3,53% ± 0,006 (MEB; n = 3 répétitions biologiques, 4 réplicats expérimentaux) de cellules mononucléaires étaient EpCAM+. Dans le sang de souris, les cellules EpCAM+ constituaient 0,45 % ± 0,0006 (MEB ; n = 2 réplicats biologiques, 4 réplicats expérimentaux), et chez la BM de souris, 5,17 % ± 0,001 (MEB ; n = 3 réplicats biologiques, 4 répétitions expérimentales) étaient des EpCAM+. Chez la souris, toutes les cellules EpCAM+ étaient immunoréactives à la pancytokératine, comme déterminé par microscopie IF. Les résultats ont été confirmés en utilisant des souris transgéniques Krt1-14;mTmG, avec un nombre faible (8,6 cellules GFP+ natives pour 106 cellules analysées; 0,085% des cellules viables), mais un nombre significatif (p < 0,0005) de cellules GFP+ présentes dans la BM murine normale, qui n’étaient pas le résultat du caractère aléatoire par rapport à plusieurs témoins négatifs. De plus, les cellules EpCAM+ dans le sang de souris étaient plus hétérogènes que les cellules CD45+ (0,58 % dans BM; 0,13 % dans le sang). Ces observations concluent que les cellules exprimant des protéines de cytokératine sont détectables de manière reproductible parmi les cellules mononucléaires du sang humain et murin et de la BM. Nous démontrons une méthode de prélèvement tissulaire, de cytométrie en flux et d’immunomarquage qui peut être utilisée pour identifier et déterminer la fonction de ces cellules épithéliales pancytokératine chez des individus en bonne santé.
Introduction
Les cellules épithéliales se trouvent dans les barrières physiques entre notre corps et l’environnement et sont capables de reconnaître et de répondre aux changements dans leur microenvironnement1. Ils ont une niche de cellules souches proliférante qui fournit un moyen de retourner de nouveaux tissus et de réparer les dommages2. Notre laboratoire étudie les cellules souches dans les follicules pileux de la peau; La peau est un bon modèle pour étudier la prolifération des tissus épithéliaux et des cellules souches, car elle est facilement visible et il y a un renouvellement constant des cellules. Les cancers épithéliaux sont la forme la plus courante de cancer, peut-être due aux tissus épithéliaux, tels que la peau, qui constituent la première ligne de défense contre les carcinogènes environnementaux, entraînant des taux de renouvellement élevés et la prolifération des cellules épithéliales3. La majeure partie de l’épiderme cutané, la couche protectrice supérieure, est composée de kératinocytes, qui expriment différents types de kératines pour fournir un soutien et une structure4. Les patients atteints de cancers épithéliaux ont souvent des cellules épithéliales présentes dans leur sang et leur moelle osseuse qui expriment également des kératines. Les biopsies liquides sont un moyen non invasif de détecter et de surveiller ces cellules épithéliales dans différents fluides corporels5. Les cellules épithéliales circulantes, également appelées cellules tumorales circulantes (CTC), se trouvent dans le sang périphérique et peuvent être des biomarqueurs du pronostic du cancer, ainsi que guider les traitements individualisés. Les CTC peuvent également indiquer la progression de la maladie, l’efficacité du traitement et la survie globaledes patients 5,6.
La molécule d’adhésion cellulaire épithéliale (EpCAM) est un marqueur cliniquement utilisé pour les CTC et peut identifier les tumeurs d’origine épithéliale chez les patients cancéreux. L’EpCAM joue un rôle dans l’adhésion, la migration, la signalisation, la prolifération et la différenciationcellulaires 6. Pour qu’une cellule épithéliale circulante soit classée comme CTC, elle doit être positive pour les cytokératines 8, 18 et 19, et négative pour CD45, un marqueur leucocytairecommun 6. Les CTC sont généralement identifiés en déplénant d’abord CD45 avec des microbilles magnétiques, puis en testant l’EpCAM et la cytokératine 19 par microscopie d’immunofluorescence7. La principale limite de la détection des CTC est leur rareté; Ils représentent moins de 0,01% de toutes les cellules dans le sang, et très peu survivent dans la circulation pour atteindre des organes distants 8,9,10. Il faut tenir compte de la conception d’expériences et de techniques permettant d’isoler et d’identifier ces cellules en raison de leur nature rare. Actuellement, il n’y a qu’un seul trieur automatisé à cellule unique approuvé par la Food and Drug Administration (FDA) utilisé pour identifier les CTC, et il utilise EpCAM comme biomarqueur. D’autres méthodes incluent la séparation des billes magnétiques et la cytométrie en flux, ou une combinaison de ces méthodes. Il est nécessaire de mettre au point de nouvelles techniques plus sensibles et plus spécifiques pour la détection des CTC rares11.
La cytométrie de flux est une méthode privilégiée pour la détection de populations de cellules rares dans le sang, la moelle osseuse et d’autres échantillons de tissus. Ces cellules rares peuvent inclure les cellules souches, les cellules endothéliales circulantes, les CTC et les cellules pathologiques résiduelles. La cytométrie en flux permet des mesures quantitatives de chaque type de cellule et trie ces cellules pour des tests supplémentaires 7,9. Des comptages progressifs ont été effectués pour assurer une évaluation précise de ces cellules rares. La possibilité d’utiliser le contrôle pour exclure les cellules d’une analyse plus approfondie est un moyen d’accroître la spécificité lors de l’analyse des cellules. Les limites de la cytométrie en flux sont le temps requis pour l’analyse de grands échantillons et l’absence de confirmation visuelle de l’identité cellulaire. Pour surmonter cela, une microscopie d’immunofluorescence a été réalisée sur les cellules triées pour confirmer leur identité.
Auparavant, notre laboratoire a montré que chez la souris, les cellules de la moelle osseuse sont recrutées et contribuent aux tumeurs cutanées12. Ces cellules dérivées de la moelle osseuse sont positives pour la pancytokératine et la cytokératine épidermique. Pour mieux élucider le rôle des cellules souches épithéliales, des cellules dérivées de la moelle osseuse et des cellules exprimant la cytokératine dans la progression tumorale, des cellules EpCAM+ cytokératine positives dans des échantillons murins normaux et humains et de moelle osseuse ont été recherchés. Comme pour la plupart des expériences, cette méthode a été développée par de multiples itérations. Auparavant, des souris transgéniques étaient utilisées avec des numérations incrémentielles pour rechercher des cellules exprimant K14GFP dans la moelle osseuse12. Au fur et à mesure que davantage de cellules ont été comptées, une population représentative de cellules rares a pu être identifiée, comme le montre la figure 1 dans la section des résultats représentatifs. La justification de l’étude des cellules épithéliales dans le sang normal et la moelle osseuse était basée sur la littérature ancienne sur les CTC, où les donneurs sains normaux avaient des niveaux de fond de cellules EpCAM+13. Comme mentionné précédemment, la caractérisation de l’EpCAM commence souvent par l’épuisement de CD45. Cette étape a été omise parce que certaines cellules hématopoïétiques ont une expression d’EpCAM et de cytokératine pour des raisons inconnues qui doivent être étudiées plus avant. Par conséquent, les cellules ont été triées en fonction de la présence ou de l’absence d’EpCAM, quelle que soit la lignée cellulaire, puis immunocolorées pour la cytokératine. Le protocole ci-dessous et le flux de travail illustré à la figure 2 décrivent une technique qui utilise la cytométrie en flux avec compensation et contrôles, des méthodes statistiques et l’imagerie par immunofluorescence pour isoler et identifier ces populations rares de cellules épithéliales.
Protocole
Tous les protocoles sur les animaux ont été approuvés par le comité institutionnel de soin et d’utilisation des animaux de l’Université du Minnesota conformément aux directives des NIH et fédérales. Du sang humain frais et de la moelle osseuse ont été achetés auprès de sources commerciales. Des échantillons ont été prélevés par l’entreprise dans le cadre d’un protocole approuvé de la CISR auprès de donneurs négatifs pour le VIH, l’hépatite B et l’hépatite C et soumis à un dépistage de la COVID-19. Tous les échantillons ont d’abord été anonymisés et anonymisés avant d’être expédiés par l’entreprise. Comme il s’agissait d’une autorisation commerciale, aucune approbation de la CISR n’était requise.
1. Préparation des solutions
NOTE: Toutes les solutions doivent être préparées dans une hotte biologique avec un environnement stérile. Obtenir la certification de niveau de biosécurité 2 (BSL2) pour le travail avec du sang humain et de la moelle osseuse.
- Préparer la solution de récolte de moelle osseuse en ajoutant 1 mL de gentamicine et 5 mL de sérum fœtal bovin (FBS) à 500 mL de solution saline équilibrée de Hank (HBSS).
- Préparez la solution tampon de coloration en ajoutant 50 ml de FBS à 500 ml de HBSS.
- Préparer 1x tampon de lyse en combinant 10 ml de tampon de lyse 10x avec 90 ml d’eau stérile.
2. Préparation de la hotte
- Allumez le capot et laissez-le fonctionner pendant quelques minutes pour obtenir une bonne circulation de l’air.
- Recueillir tout le matériel pour effectuer le prélèvement de moelle osseuse. Vaporisez-les à l’intérieur de la hotte avec de l’éthanol à 70% pour maintenir un environnement stérile. Vaporisez les mains à chaque fois avant qu’elles ne soient prises à l’intérieur de la hotte.
- Placez le plateau autoclavé dans la hotte avec des ciseaux autoclavés, un scalpel assemblé, une pince à épiler et des pinces incurvées dans une petite tasse remplie d’éthanol à 70% pour les garder stériles.
- Ajouter 10 mL de solution de prélèvement de moelle osseuse à un tube centrifuge de 50 ml et étiqueter « Membres ». Étiquetez un autre tube à centrifuger de 50 ml comme « moelle osseuse ».
- Prélever 10 ml de solution de prélèvement de moelle osseuse dans une seringue, puis fixer une aiguille de 26 g.
3. Prélèvement de moelle osseuse sur des souris
- Euthanasier la souris par asphyxie au CO2 . La mort est confirmée par l’absence de battement de cœur et le pincement des pieds pour s’assurer qu’il n’y a pas de réaction. La luxation cervicale peut être pratiquée comme moyen secondaire d’euthanasie complète.
- Placez la souris dans un contenant de 500 ml et ajoutez suffisamment d’iode pour couvrir la moitié de la souris. Secouez doucement et agitez le récipient pour assurer une couverture complète. Rincer à l’eau désionisée. Effectuez un autre lavage à l’iode, puis deux lavages avec de l’éthanol à 70% en suivant les mêmes étapes.
- Placez la souris dans le capot et posez-la sur le dos sur le plateau. Attrapez les pattes postérieures et écartez-les jusqu’à ce qu’un pop se fasse sentir; Il s’agit de séparer les jambes de la colonne vertébrale.
- Faites une incision de 1 cm sur la peau de la souris près de la région de l’aine. Insérez une paire de ciseaux fermée dans l’incision, puis ouvrez les ciseaux sous la peau pour la séparer du péritoine.
- Coupez la peau de la jambe autour de la cuisse, puis coupez la peau le long de la jambe pour exposer les muscles et les os.
- Enlevez les pattes postérieures en coupant autour de l’articulation de la hanche, en veillant à ne pas couper à travers le fémur. Placez les membres dans le tube étiqueté comme « Membres » Lorsque les deux pattes sont retirées, placez la souris dans un sac de carcasse et mettez-la au congélateur.
REMARQUE: L’os de la hanche doit être palpé avant la coupe pour guider les ciseaux et éviter de couper l’os du fémur, car la majeure partie de la moelle osseuse est obtenue à partir du fémur. - Commencez à couper les muscles, les tissus et la graisse le long de l’un des membres à l’aide des ciseaux. Couper parallèlement à l’os. Ensuite, utilisez le scalpel et retirez la graisse ou le muscle restant, en utilisant un mouvement de grattage perpendiculaire contre l’os.
- Une fois que l’os est nettoyé correctement, séparez le fémur et le tibia au niveau du genou. Utilisez le scalpel pour faire des coupures aux deux extrémités du fémur et du tibia où il n’y a pas de moelle osseuse visible.
- Insérez la seringue préparée et l’aiguille dans l’os. S’il y a de la résistance, coupez un peu plus l’extrémité de l’os jusqu’à ce qu’il n’y ait plus de résistance.
- Tout en tenant l’os au-dessus du tube étiqueté « moelle osseuse », expulsez la solution de prélèvement de la moelle osseuse à travers l’os pour rincer la moelle osseuse dans le tube. Répétez à l’autre extrémité de l’os pour obtenir le reste de la moelle osseuse, jusqu’à ce que l’os apparaisse tout blanc ou vide.
- Répétez les étapes 7 à 9 pour tous les os et membres restants; Toute la moelle sera rincée dans le même tube conique.
- À l’aide d’une seringue vide de 10 ml et d’une aiguille de 20 g, briser les amas de moelle osseuse dans le tube en aspirant la moelle osseuse de haut en bas dans la seringue 5 à 10 fois.
- Ajouter un filtre stérile de 40 μm dans un tube centrifuge propre de 50 ml et rincer avec 1 mL de solution de prélèvement de moelle osseuse. Ensuite, filtrez la moelle osseuse pour éliminer les touffes restantes. Étiquetez ce tube « Moelle osseuse filtrée ».
- Conservez la moelle osseuse filtrée au réfrigérateur ou sur de la glace jusqu’à utilisation. Il doit être utilisé le jour même de la récolte. S’il n’est pas utilisé le même jour, congelez les cellules à l’aide d’un agent de conservation cryoglycémique, tel que le diméthylsulfoxyde (DMSO).
ATTENTION : Tous les déchets issus de ces expériences doivent être éliminés correctement dans un conteneur à déchets présentant un risque biologique. Tous les objets tranchants doivent être placés dans un contenant d’objets tranchants présentant un risque biologique correctement étiqueté.
4. Lyse globule rouge de la moelle osseuse
- Centrifuger les cellules de la moelle osseuse à 170 x g pendant 10 min à température ambiante. Passer l’aspirateur et jeter le surnageant. Remettez les cellules en suspension dans 10 mL de 1x tampon de lyse des globules rouges et incuber pendant 4 min.
- Ajouter 30 mL de solution saline tamponnée au phosphate (DPBS) de Dulbecco pour arrêter la réaction du tampon de lyse. Centrifuger les cellules pendant 8 min à 170 x g à température ambiante. Passer l’aspirateur et jeter le surnageant. Resuspendre dans 10-20 mL de tampon de coloration.
5. Nombre de cellules
- Combiner 500 μl de cellules avec 9,5 mL de solution de prélèvement de moelle osseuse pour obtenir une dilution de 1:20.
REMARQUE: Il sera utilisé pour le comptage des cellules et n’a pas besoin d’être gardé stérile. - Extraire 200 μL de cellules de la dilution 1:20, les ajouter à 200 μL de bleu de trypan et bien mélanger.
- Ajouter le mélange à un hémocytomètre et compter les cellules vivantes et mortes des deux côtés. Calculez la suspension cellulaire originale à l’aide de ce tableau (présenté dans le fichier supplémentaire 1) et des équations suivantes :
Cellules par millilitre: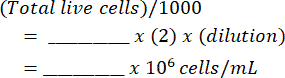
Nombre total de cellules :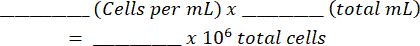
- Faire tourner les cellules à 170 x g pendant 10 minutes à 4 °C et remettre en suspension à une concentration de 10 x 10 6 par 1 mL (1 x 106 cellules par 100 μL), en fonction des suggestions du fabricant pour les dilutions d’anticorps.
- Passez à la cytométrie en flux (étape 8).
6. Prélèvement de sang sur des souris
- Euthanasier la souris à l’aide de gaz CO2 ou d’une luxation cervicale.
- Effectuer une prise de sang cardiaque à un angle de 45° avec une aiguille de 26 G fixée à une seringue de 1 mL recouverte d’acide éthylènediaminetétraacétique (EDTA) 0,5 M.
- Faites tourner le sang vers le bas et remettez-le en suspension dans un milieu ou un tampon de coloration.
REMARQUE: Les cellules seront filées vers le bas et remises en suspension à la concentration finale dans le tampon de coloration après la lyse des globules rouges et le nombre de cellules. - Effectuez la lyse des globules rouges (étape 4).
- Effectuez le comptage des cellules (étape 5).
- Passez à la cytométrie en flux (étape 8).
7. Traitement des échantillons de sang humain et de moelle osseuse
- Effectuer toutes les procédures sous une cagoule certifiée BSL2 tout en portant un équipement de protection individuelle.
- Prélever les échantillons la nuit précédente et effectuer la lyse des globules rouges. S’assurer que les échantillons sont anonymisés et anonymisés avant d’expédier les échantillons mononucléaires pendant la nuit sur la glace.
- À l’arrivée, faire tourner les cellules sous tension à 170 x g à 4 °C pendant 10 min.
REMARQUE: Ce sont des échantillons BSL2 qui doivent suivre les directives locales. - Remettez les cellules en suspension dans un support ou un tampon de coloration.
REMARQUE: Les cellules doivent être tournées vers le bas et remises en suspension à la concentration finale après comptage des cellules avec tampon de coloration. - Effectuez le comptage des cellules (étape 5).
- Passez à la cytométrie en flux (étape 8).
8. Cytométrie de flux
- Coloration
- Divisez les cellules en tubes étiquetés individuellement en fonction du panneau de coloration utilisé, y compris les contrôles de compensation (non colorés, témoins d’isotype, coloration simple et fluorescence moins un [FMO]). Congelez toutes les cellules restantes ou utilisez-les pour le tri cellulaire activé par fluorescence. Voir le tableau 1 pour un exemple de panneau de coloration avec les dilutions d’anticorps utilisées.
REMARQUE : Pour chaque fluorophore supplémentaire ajouté, les FMO en combinaison avec les fluorophores restants doivent être ajoutés avec une seule coloration de ce fluorophore. Les contrôles d’isotypes peuvent être utilisés pour bloquer les colorations non spécifiques. Chaque nouvel anticorps utilisé doit être titré avec les échantillons pour une dilution optimale. - Ajouter les anticorps selon les dilutions recommandées par le fabricant et bien mélanger en agitant le fond des tubes. Les concentrations d’anticorps sont généralement indiquées sous la forme de 1 x 106 cellules par 100 μL. Les dilutions avec les anticorps et les témoins sont indiquées dans le tableau 1. Les anticorps sont conjugués avec des fluorophores et doivent être protégés de la lumière.
REMARQUE : Il est préférable d’utiliser l’EpCAM conjugué à la phycoérythrine (PE) pour permettre la visualisation de faibles populations de cellules (titrer à 3-5 μL pour 1 million de cellules, comme indiqué dans le tableau 1). Le PE-CD49f a été utilisé comme contrôle de PE à coloration unique (20 μL pour 1 million de cellules), car ces cellules sont plus répandues que le PE-EpCAM et donc plus faciles à utiliser pour la compensation. - Incuber pendant 30 min à 4 °C dans l’obscurité.
- Après l’incubation, ramenez les tubes dans la hotte BSL2 et ajoutez 1 ml de tampon de coloration dans chaque tube.
- Ensuite, centrifuger les tubes à 170 x g à 4 °C pendant 5-10 min.
- Après la centrifugation, ramenez les tubes bouchés avec les cellules dans la hotte BSL2 et aspirez le surnageant. Assurez-vous de changer les extrémités de la pipette d’aspiration entre chaque tube pour éviter la contamination croisée.
- Remettez en suspension la pastille de cellule dans 1 mL de tampon de coloration et agitez le fond des tubes pour mélanger.
- Lavez les cellules deux fois de plus en répétant les étapes 8.1.4-8.1.7.
REMARQUE: N’utilisez pas de pipettes pour remettre en suspension, afin d’éviter de perdre des cellules. - Resuspendre dans 500 μL de tampon de coloration pour la remise en suspension finale avant cytométrie en flux. Ajouter du 4′,6-diamidino-2-phénylindole (DAPI) ou un discriminateur à cellules mortes, selon les recommandations du fabricant.
- Divisez les cellules en tubes étiquetés individuellement en fonction du panneau de coloration utilisé, y compris les contrôles de compensation (non colorés, témoins d’isotype, coloration simple et fluorescence moins un [FMO]). Congelez toutes les cellules restantes ou utilisez-les pour le tri cellulaire activé par fluorescence. Voir le tableau 1 pour un exemple de panneau de coloration avec les dilutions d’anticorps utilisées.
- Cytomètre en flux
- Après avoir configuré la machine, utilisez les commandes et les FMO pour régler la compensation et les portes. Utilisez la solution saline équilibrée de Hank (HBSS) comme liquide de gaine pour aider à soutenir les cellules fragiles. Utilisez PBS si HBSS n’est pas disponible.
REMARQUE: Le cytomètre en flux utilisé et le logiciel sont répertoriés dans le tableau des matériaux. - Chargez les échantillons un par un et collectez des points de données pour 50 000, 100 000, 500 000 et un million d’événements. Conserver les tubes sur de la glace ou réfrigérés jusqu’à utilisation.
REMARQUE : Une limite de temps peut être fixée lors du prélèvement pour assurer la viabilité de l’échantillon. - Recueillir les cellules dans 3 mL de RPMI avec 10% FBS dans des tubes à centrifuger de 15 mL.
- Si vous effectuez une immunofluorescence sur des cellules EpCAM+, utilisez le cytomètre en flux pour trier 800 à 1 000 cellules dans chaque puits d’une lame de 8 puits.
- Trier la population de cellules EpCAM sur des lames séparées comme témoin négatif à utiliser lors de la coloration pour l’immunofluorescence.
REMARQUE: Les cellules peuvent également être triées dans des tubes de 15 mL contenant FBS et filées sur une lame à l’aide du cytocentrifugeuse ou d’une pipette pour empêcher les cellules d’éclater. - Fixer les lames dans un mélange 50 % de méthanol/50 % d’acétone à -20 °C pendant 10 min. Conserver les lames à -20 °C jusqu’à coloration par immunofluorescence.
- Après avoir configuré la machine, utilisez les commandes et les FMO pour régler la compensation et les portes. Utilisez la solution saline équilibrée de Hank (HBSS) comme liquide de gaine pour aider à soutenir les cellules fragiles. Utilisez PBS si HBSS n’est pas disponible.
- Analyse de cytométrie en flux
- Ouvrez le logiciel d’analyse de cytométrie en flux sous licence.
- Fichiers standard de cytométrie en flux de charge (FCS) acquis à partir du cytomètre en flux avec des contrôles et des échantillons.
- Cliquez sur Créer un groupe et utilisez des mots-clés pour placer des échantillons et des contrôles dans un groupe.
REMARQUE : Utilisez le contrôle « sans tache » pour ouvrir une grille dans le logiciel d’analyse de cytométrie en flux, comme illustré à la figure 3. - Double-cliquez sur l’exemple de contrôle sans tache pour ouvrir un graphique non contrôlé.
- Définissez l’axe y du graphique sur SSC-A et l’axe des x sur FSC-A (zone de diffusion latérale par zone de diffusion directe), ce qui indique la taille de la cellule et la complexité interne.
- Cliquez sur la forme du polygone dans le panneau supérieur gauche et créez une porte autour des cellules. Assurez-vous d’exclure les cellules à la périphérie, car il s’agit très probablement de débris. Étiquetez cette porte « Cellules », comme illustré à la figure 3A.
- Une fois les cellules fermées, double-cliquez à l’intérieur de la forme du polygone pour ouvrir un autre graphique dans une nouvelle fenêtre.
- Définissez l’axe y du nouveau graphique sur SSC-A et l’axe des x sur SSC-W pour la discrimination doublet. Cliquez sur la forme du polygone et créez un rectangle autour des cellules individuelles. Étiquetez cette « Cellules individuelles », comme illustré à la figure 3B.
- Une fois les cellules fermées, double-cliquez à l’intérieur de la forme du polygone pour ouvrir un autre graphique dans une nouvelle fenêtre.
- Définissez l’axe y sur FSC-A et l’axe x sur DAPI-A (ou tout autre discriminateur de cellules mortes utilisé).
- Créez une porte polygonale autour des cellules négatives DAPI sur le côté gauche du graphique. Étiquetez ces « cellules vivantes », comme illustré à la figure 3C.
- Une fois les cellules fermées, double-cliquez à l’intérieur de la forme du polygone pour ouvrir un autre graphique dans une nouvelle fenêtre.
- Réglez l’axe y sur SSC-A et l’axe x sur EpCAM-PE (ou le fluorophore utilisé pour identifier les cellules EpCAM). Les cellules de gauche sont EpCAM négatif. Si des cellules sont montrées à droite, elles sont positives à l’EpCAM.
- Utilisez le graphique EpCAM-PE pour créer une porte polygonale, qui exclut les cellules de gauche et inclut uniquement l’espace vide à droite. Étiquetez la zone vide à droite à l’intérieur du polygone « EpCAM », comme illustré à la figure 3D.
Remarque : Il s’agit d’un contrôle sans tache, il ne doit donc pas y avoir d’expression EpCAM. Utilisez le contrôle sans tache pour déterminer quelles cellules sont positives à l’EpCAM. Cela complète la stratégie de points de contrôle pour la rémunération, de sorte que tous les graphiques peuvent être fermés à ce moment-là. - Sur la page de l’espace de travail, mettez en surbrillance la lignée sans tache avec « Cellules », « Cellules uniques », « Cellules dynamiques » et « EpCAM ». Cliquez avec le bouton droit de la souris et sélectionnez Copier l’analyse pour grouper. Cette opération copie la stratégie de contrôle à tous les autres échantillons chargés au sein du groupe créé précédemment. Si un groupe n’a pas été créé initialement, il peut être créé maintenant en sélectionnant Créer un groupe en haut à gauche.
- Parcourez les échantillons au sein du groupe et vérifiez les portes polygonales dessinées précédemment pour vous assurer qu’elles correspondent à tous les échantillons et contrôles. Cela fournit le pourcentage et le nombre de cellules.
- Cliquez sur le bouton L de l’éditeur de mise en page dans le coin supérieur gauche au-dessus de « Espace de travail ». Ces graphiques peuvent ensuite être organisés et exportés à l’aide de l’éditeur de mise en page.
- Préparation de réactifs d’immunofluorescence
- Préparer le diluant d’anticorps en combinant 1 g d’albumine sérique bovine (BSA), 2 g de lait écrémé, 10 mL de solution saline tamponnée 10x tris (TBS), 100 μL de Tween et 90 mL d’eau distillée.
- Préparer le TBST en combinant 50 mL de TBS 20x, 950 mL d’eau distillée et 200 μL de Tween.
- Préparer du sérum normal pour chevaux (NHS) à 10 % en combinant 1 mL de NHS avec 9 mL de diluant d’anticorps (étape 8.4.1).
- Préparer l’ENM à 1 % en combinant 1 mL de NHS à 10 % avec 9 mL de TBST.
- Coloration par immunofluorescence
- Retirez les lames de -20 °C et laissez-les se réchauffer à température ambiante.
- Lavez les lames trois fois à l’eau distillée pendant 5 minutes chacune.
- Bloquer les lames pendant 1 h à température ambiante dans du NHS à 10 % dans un diluant d’anticorps (étape 8.4.3).
- Diluer l’anticorps primaire (pancytokératine) à 1:750 dans le NHS à 1 % dans le TBST (étape 8.4.4). Incuber les lames dans l’anticorps primaire dilué pendant une nuit à 4 °C.
- Le lendemain, lavez les lames trois fois avec un tampon de lavage 1x (PBS ou TBST) pendant 5 minutes chacune.
- Diluer l’anticorps secondaire à 1:1 000 dans le NHS à 1 % dans le TBST (étape 8.4.4). Incuber les lames dans l’anticorps secondaire dilué pendant 1h à température ambiante.
- Lavez les lames trois fois avec 1x tampon de lavage pendant 5 minutes chacune. Montez et couverssez les glissières avec un support de montage rigide avec DAPI. Utilisez un coton-tige pour étaler doucement les bulles sous la lame.
Résultats
En utilisant ces méthodes, des populations rares de cellules épithéliales dans le sang et la moelle osseuse d’humains et de souris normaux ont été visualisées. Avec les compensations et les contrôles appropriés tels que décrits, les résultats ont systématiquement montré que 4% à 5% des cellules dans la moelle osseuse murine étaient EpCAM+, quel que soit le nombre de cellules comptées, comme le montrent la figure 4 et la figure 5. Dans les échantillons de sang murin, moins de 0,5 % des cellules étaient EpCAM+, comme le montre la figure 6. Dans les échantillons de moelle osseuse humaine, 2 % à 5 % des cellules étaient EpCAM+, comme le montrent les figures 7 et 8. Bien que 2 % à 5 % soit une grande fourchette, les pourcentages au sein de chaque donneur individuel étaient cohérents à mesure que davantage de cellules étaient comptées. Dans les échantillons de sang humain, environ 0,3% des cellules étaient EpCAM+, comme le montre la figure 9. Nos échantillons témoins (pas de coloration, contrôle d’isotype et FMO) ont donné très peu de résultats EpCAM+ faussement positifs, comme le montrent la figure 4 et la figure 7. Les cellules des groupes EpCAM+ et EpCAM- qui ont été triées sur des lames ont montré une coloration positive pour la pan-cytokératine dans les échantillons EpCAM+, et étaient négatives pour la pan-cytokératine dans les échantillons EpCAM-, comme le montre la figure 10. Ces résultats indiquent que les expériences ont été conçues de manière appropriée et reproductibles.
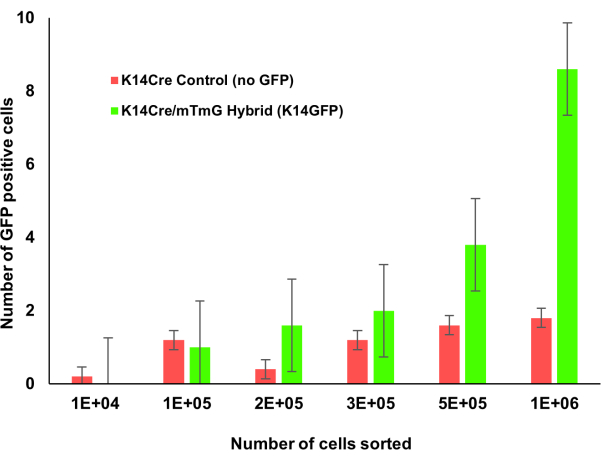
Figure 1 : Souris transgéniques Krt14Cre;mTmG avec numération incrémentielle de la moelle osseuse. La moelle osseuse des souris Krt1-14;mTmG a été comptée progressivement. Les cellules GFP positives indiquent l’expression de la kératine 14 et ont été identifiées par cytométrie en flux. Comme plus de cellules de moelle osseuse ont été comptées, cette population de cellules positives à la kératine 14 était plus facilement identifiable. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 2 : Flux de travail pour les cellules EpCAM+ et cytokératine+. La moelle osseuse et les cellules sanguines ont d’abord été triées à l’aide du tri cellulaire activé par fluorescence (FACS) pour séparer les cellules EpCAM+ et EpCAM-. Ces cellules ont été triées dans deux tubes à essai différents, ainsi que sur deux lames différentes. Les cellules triées dans des tubes à essai ont été filées sur des lames à l’aide d’une cytocentrifugeuse. Les lames ont ensuite été colorées à l’aide d’un anticorps primaire pan-cytokératine, puis colorées avec un anticorps secondaire. Les lames ont été analysées par microscopie à fluorescence pour observer l’expression de la pancytokératine. Veuillez cliquer ici pour voir une version agrandie de cette figure.
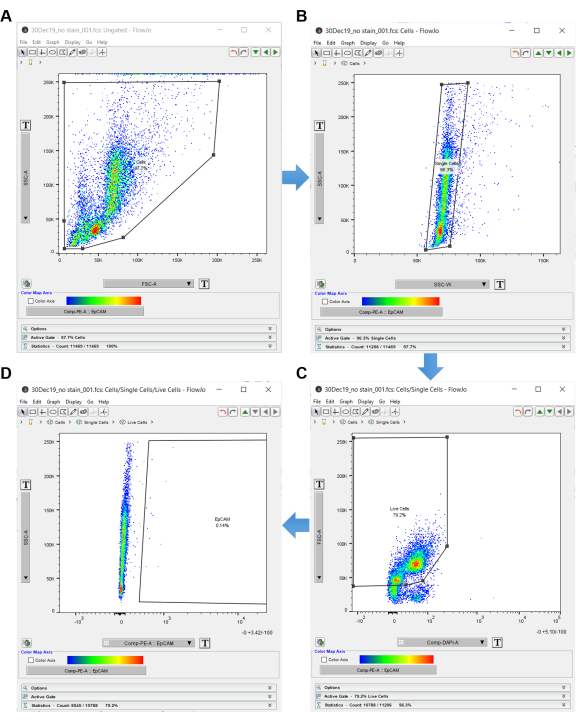
Figure 3 : Logiciel d’analyse de cytométrie en flux. Lors de l’analyse des données de cytométrie en flux à l’aide d’un logiciel d’analyse, le contrôle sans tache est utilisé pour sélectionner les cellules d’intérêt. Les cellules sont d’abord sélectionnées avec SSC-A et FSC-A, qui montrent la complexité interne et la taille des cellules. (A) Une porte polygonale est dessinée autour des cellules. (B) Les cellules individuelles sont acquises par contrôle SSC-A par SSC-W. (C) Les cellules vivantes sont acquises en comparant le FSC-A au DAPI. (D) Les cellules EpCAM négatives sont exclues en gisant à droite des cellules EpCAM négatives. Veuillez cliquer ici pour voir une version agrandie de cette figure.
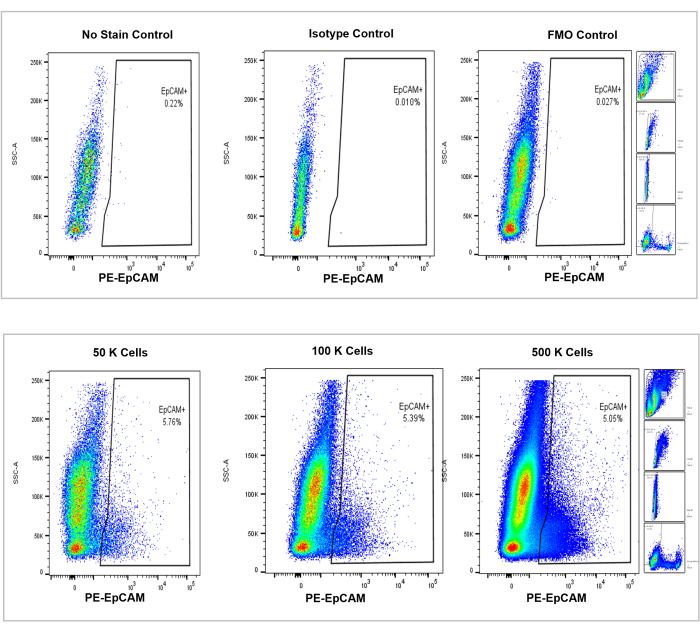
Figure 4 : Analyse cytométrique en flux de cellules EpCAM+ dans la moelle osseuse de souris. Les contrôles d’absence de tache, d’isotype et de FMO sont affichés dans le panneau supérieur, avec les comptes incrémentiels de 50 000, 100 000 et 500 000 cellules affichés dans le panneau inférieur. Ces graphiques visualisent la cohérence des pourcentages entre les nombres, malgré l’augmentation globale du nombre total de cellules comptées. Les panneaux de droite indiquent la stratégie de contrôle, telle que discutée précédemment dans la section analyse de cytométrie en flux et comme illustré à la figure 3. Veuillez cliquer ici pour voir une version agrandie de cette figure.
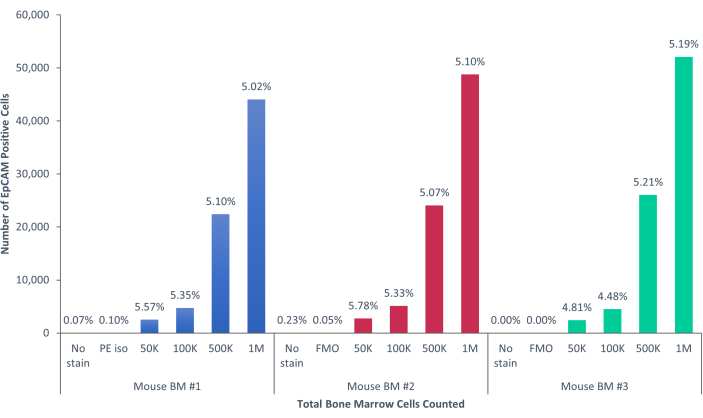
Figure 5 : Les cellules EpCAM+ dans la moelle osseuse de souris représentent 5,17 % ± 0,001 % de la population. Les cellules de la moelle osseuse de trois souris individuelles ont été analysées. Les contrôles appropriés ont été inclus pour une rigueur scientifique appropriée. Le pourcentage de cellules positives est resté constant dans tous les échantillons, car les souris étaient génétiquement identiques. Veuillez cliquer ici pour voir une version agrandie de cette figure.
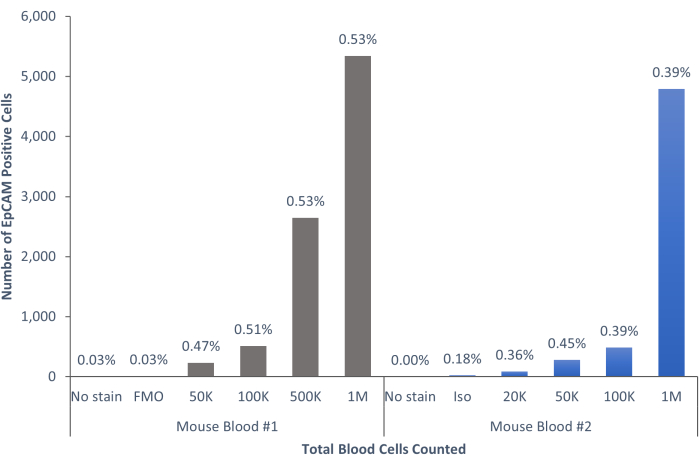
Figure 6 : Les cellules EpCAM+ dans le sang de souris représentent 0,45 % ± 0,0006 % de la population. Les cellules sanguines de deux souris individuelles ont été analysées. Des contrôles ont été inclus pour montrer que la procédure appropriée a été suivie pour produire des résultats concluants. Veuillez cliquer ici pour voir une version agrandie de cette figure.
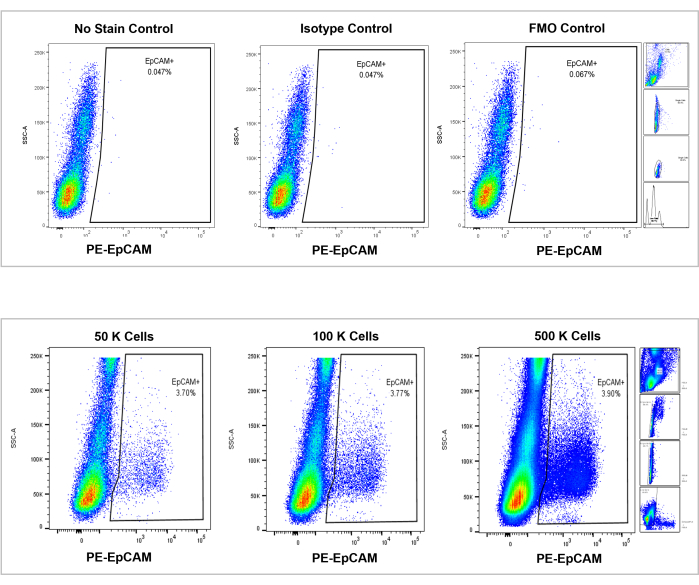
Figure 7 : Analyse cytométrique en flux sur la moelle osseuse humaine EpCAM+. Les contrôles d’absence de tache, d’isotype et de FMO sont affichés dans le panneau supérieur, et les comptes incrémentiels de 50 000, 100 000 et 500 000 cellules sont affichés dans le panneau inférieur. Ces graphiques visualisent la cohérence des pourcentages entre les nombres, malgré l’augmentation globale du nombre total de cellules comptées. Les panneaux de droite indiquent la stratégie de contrôle, telle que discutée précédemment dans la section analyse de cytométrie en flux et comme illustré à la figure 3. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 8 : Les cellules EpCAM+ dans la moelle osseuse humaine représentent 3,53 % ± 0,006 % de la population. Trois échantillons différents de moelle osseuse humaine ont été analysés. Des contrôles appropriés pour la rigueur scientifique ont été inclus. Le pourcentage de cellules positives varie en raison de l’hétérogénéité génétique chez l’homme. Veuillez cliquer ici pour voir une version agrandie de cette figure.
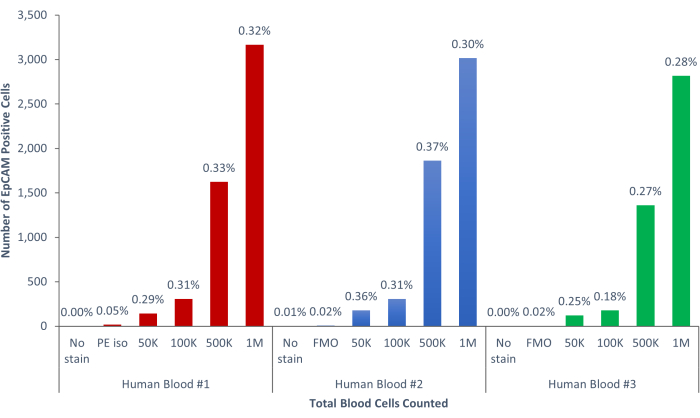
Figure 9 : Les cellules EpCAM+ du sang humain représentent 0,18 % ± 0,0004 % de la population. Trois échantillons de sang humain différents ont été analysés. Des contrôles appropriés pour la rigueur scientifique ont été inclus. Veuillez cliquer ici pour voir une version agrandie de cette figure.
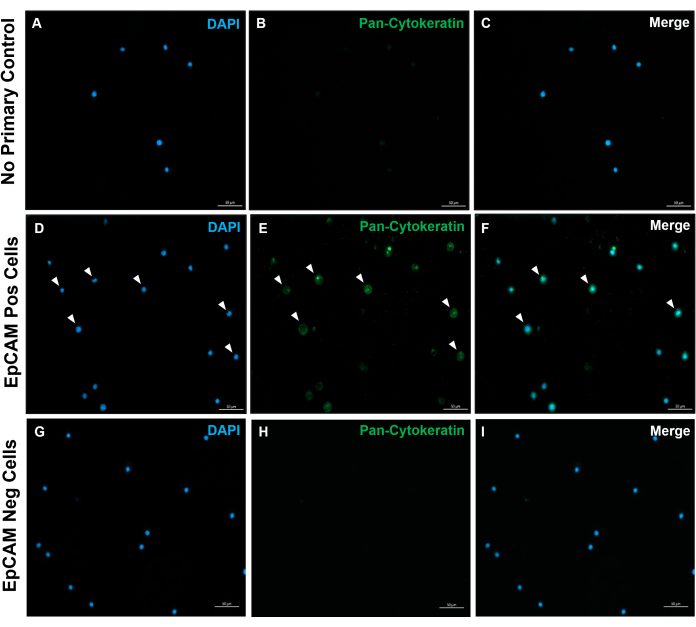
Figure 10 : Immunofluorescence des lames EpCAM+ et EpCAM-. FACS a été utilisé pour séparer les cellules EpCAM+ et EpCAM-. La pan-cytokératine a été colorée pour l’utilisation de l’anticorps pancytokératine DAKO. Aucun contrôle d’anticorps primaire dans le sérum normal n’a été utilisé, car la pancytokératine est un anticorps polyclonal. Ces résultats confirment l’exactitude du FACS. Veuillez cliquer ici pour voir une version agrandie de cette figure.
| Moelle osseuse humaine ou sang | |||
| Anticorps | Tube # | # de cellules | AB Conc |
| Non taché | 1 | 1x106 | X |
| DAPI uniquement | 2 | 1x106 | 1uL/mL |
| Contrôle de l’isotype PE | 3 | 1x106 | 1 uL |
| CD49f-PE Contrôle des taches simples | 4 | 1x106 | 20 uL dans 100uL par 1x106 |
| EpCAM-PE Low Titration | 5 | 1x106 | 3 uL dans 100uL par 1x106 |
| Titrage moyen EpCAM-PE | 6 | 1x106 | 4 uL dans 100uL par 1x106 |
| Titrage élevé EpCAM-PE | 7 | 1x106 | 5 uL dans 100uL par 1x106 |
| EpCAM-PE Tri élevé sur les diapositives | 8 | 10x106 | 50 uL dans 1 mL |
Tableau 1 : Panneau de coloration par cytométrie en flux. Un exemple de panneau de coloration par cytométrie en flux pour les cellules mononucléaires du sang ou de la moelle osseuse. Les témoins inclus sont DAPI uniquement, contrôle non coloré et contrôle des taches uniques PE (PE-CD49f a été utilisé comme meilleur contrôle positif). La fluorescence moins un n’est pas incluse dans ce panneau car il ne s’agit que d’une seule couleur (PE). Lors de l’ajout de colorants fluorophores supplémentaires, tels que l’isothiocyanate de fluorescéine (FITC) ou l’allophycocyanine (APC), les FMO doivent être inclus en excluant un fluorophore pour chaque témoin FMO.
Fichier supplémentaire 1 : Comptage des cellules vivantes à l’aide d’un hématocytomètre. Veuillez cliquer ici pour télécharger ce fichier.
Discussion
Il existe des preuves dans la littérature de la présence de cellules épithéliales dans la moelle osseuse. Auparavant, les articles ont généralement étudié le rôle des cellules épithéliales dans le contexte de maladies et de blessures, telles que dans le foie, les poumons, le tractus gastro-intestinal (GI), le thymus et la peau14,15,16,17. Cependant, on ne sait pas grand-chose sur la présence de ces cellules épithéliales dans la moelle osseuse d’individus en bonne santé. Cet article cherche à établir une méthode reproductible, dans le but d’identifier et d’isoler les cellules épithéliales du sang normal et de la moelle osseuse. Cette méthode permettra d’identifier pourquoi les cellules épithéliales sont présentes et quel est leur rôle dans le sang et la moelle osseuse en l’absence de maladie; Peut-être que ces cellules font partie de l’entretien normal des tissus ou sont activées en cas de blessure. La moelle osseuse est un dépôt de cellules souches; Cependant, on ne sait pas quelle peut être la lignée de ces cellules épithéliales. Un article récent traite des cellules épithéliales dérivées de la moelle osseuse dans le thymus, qui expriment d’abord EpCAM et le marqueur hématopoïétique CD45, puis perdent leur expression de CD45 au fil du temps après une lésion15. Les recherches de notre laboratoire ont également confirmé la présence de cellules CD45+ EpCAM+ dans le sang sain et la moelle osseuse en l’absence de blessure12. Cependant, le rôle de ces cellules reste à déterminer.
Il était nécessaire de mettre au point une méthode reproductible pour examiner les cellules épithéliales dans la moelle osseuse saine. La méthode décrite aidera à caractériser davantage ces cellules dans leur état normal. Il y a des étapes importantes dans cette méthode pour maintenir la reproductibilité. L’une des étapes les plus critiques de ce protocole consiste à maintenir un environnement stérile dans le capot tout en prélevant la moelle osseuse de souris. En cas de prélèvement sur plusieurs souris, la contamination croisée entre les échantillons est évitée en utilisant de nouvelles aiguilles et seringues pour chaque souris. Cela garantit également que l’échantillon est propre et exempt de tout contaminant susceptible d’affecter les résultats. En outre, le nombre de seringues préparées et de tubes coniques marqués pour chaque souris supplémentaire doit être augmenté. Une autre étape importante consiste à rincer les os jusqu’à ce qu’une couleur blanche propre soit évidente pour s’assurer que la plupart des cellules de la moelle osseuse ont été enlevées; Les chances de détecter des cellules rares augmentent dans un échantillon plus pur. Une modification a été apportée pour optimiser le protocole de lyse des globules rouges; Plusieurs tampons de lyse ont été testés pour trouver le bon qui donnait une viabilité élevée et constante, car près de la moitié des cellules étaient perdues au cours de cette étape. Il peut être nécessaire d’optimiser différents réactifs et périodes d’incubation pour obtenir de meilleurs résultats dans d’autres contextes.
La limitation la plus importante de ce protocole est l’utilisation de la cytométrie en flux pour trouver des populations de cellules rares. Comme nous l’avons mentionné précédemment, l’ajout de contrôles et de comptes supplémentaires contribue à accroître la spécificité et l’exactitude de l’analyse. Une autre limite est que les marqueurs appropriés pour la population d’intérêt doivent être identifiés à l’avance. Ainsi, il faut connaître les marqueurs clés, les anticorps pour la cytométrie en flux et les anticorps compatibles avec les espèces cibles.
Ces méthodes constituent une amélioration par rapport aux méthodes existantes car elles permettent une analyse unicellulaire à une fraction du coût de l’isolateur automatique CTC existant et du séquençage de l’ARN unicellulaire. De plus, la cytométrie en flux est plus facilement disponible. FACS a maintenu une viabilité plus élevée pour les cellules par rapport aux résultats rapportés antérieurement en utilisant la séparation magnétique par microbilles. Enfin, ces techniques permettent la séparation des cellules pour les analyses en aval, telles que le séquençage de l’ARN en vrac, le séquençage de l’ARNc ou la culture cellulaire.
Déclarations de divulgation
Il n’y a pas de conflits d’intérêts connus.
Remerciements
Josh Monts, technicien en cytomètre en flux de l’installation centrale, The Hormel Institute
Todd Schuster, gestionnaire des installations principales, The Hormel Institute
Derek Gordon, statisticien, Université Rutgers
Nous tenons à remercier Karen Klein de Clarus Editorial Services, Santa Fe, NM pour son aide éditoriale.
Ce travail a été soutenu en partie par le National Institute of Arthritis and Musculoskeletal and Skin Diseases des National Institutes of Health sous le numéro d’attribution R21 AR075281, et une subvention d’aide à la recherche, à l’art et à l’érudition, Bureau du vice-président à la recherche, Université du Minnesota (proposition #324240). Nous remercions le Hormel Institute pour son soutien.
matériels
| Name | Company | Catalog Number | Comments |
| 40 μm Cell Strainer | Falcon | 352340 | Used to filter out any large clumps of cells from bone marrow |
| 5 mL Polystyrene Round-bottom tube | Falcon | 352054 | Tubes used for flow cytometry |
| 500 mL jar with lid | Nalgene | 11-823-32 | Used to wash the mouse |
| 190 proof Ethanol | Decon laboratories Inc | 2801 | Diluted to 70% with dH2O |
| Alexa Fluor 488 Goat anti-rabbit IgG (H+L) | Invitrogen | A11008 | Dilution: 1:1000 |
| Brightline Hemacytometer | Hausser Scientific | 02-671-10 | Used to count alive cells |
| Bovine Serum Albumin | Jackson ImmunoResearch | 001-000-162 | Component of antibody diluent |
| Characterized Fetal Bovine Serum (FBS) | Hyclone | SH30071.03 | Component of staining buffer and harvesting solution |
| Curity gauze sponges | Covidien | 2187 | Used to keep a clean work area when harvesting limbs and bone marrow |
| Curved Forceps | Miltex | 18-784 | Used during harvest |
| Cytokeratin, Wide Spectrum Screening | Dako | Z0622 | Dilution 1:750 *Now discontinued |
| Dulbecco's Phosphate Buffered Saline (DPBS 1x) | Gibco | 14190-144 500mL | Used to stop the red blood cell lysis reaction |
| Ethylenediaminetetraacetic acid | Sigma Aldrich | E9884-100G | Used at 0.5 M |
| Gentamicin sulfate | Lonza | 17-518Z | Component of harvesting solution |
| Hank's Balanced Salt Solution (HBSS 1x) | Gibco | 14175-095 500mL | Component of harvesting solution and staining buffer solution |
| Luer-Lok tip 10 mL syringe | Becton, Dickinson and Co | 309604 | Used with 26G needle to flush bones, used with 20G needle to break up clumps |
| Magic touch 2 ice bucket | BelArt | M16807-2001 | Used to store the specimens on ice |
| Nonfat dry milk | Apex | 20-241 | Component of Antibody Diluent |
| Normal horse serum | Vector | ZE0122 | Component of Antibody Diluent |
| PE anti-human CD 326 (EpCAM) | Biolegend | 324206 | Dilution: 5 µl in 100 µl per 1x106 cells |
| PE anti-mouse CD 326 (EpCAM) | Biolegend | 118205 | Dilution: 3 µl in 100 µl per 1x106 cells |
| PE mouse IgG2b, κ isotype control | Biolegend | 400313 | Dilution: 1 µl in 100 µl per 1x106 cells |
| PE rat anti-human CD49f | BD Biosciences | 555763 | Dilution: 20 µl in 100 µl per 1x106 cells |
| PE rat IgG2am, κ Isotype control | Biolegend | 400508 | Dilution: 1 µl in 100 µl per 1x106 cells |
| Polypropylene Conical Centrifuge Tubes 50 mL | Basix | 14-955-240 | Used in the centrifuge |
| Povidone-Iodine Scrub | Aplicare | 82-227 | Antiseptic used to sterilize the mice |
| PrecisionGlide Needle 20G x 1 1/2 | Becton, Dickinson and Co | 305176 | 20G needle used to break up bone marrow clumps |
| PrecisionGlide Needle 26G x 1/2 | Becton, Dickinson and Co | 305111 | 26G needle used to flush bone marrow from bones |
| PTFE Printed Slides | Electron Microscopy Services | 63422-06 | 8 well slides cells sorted onto for immunofluorescence |
| RBC Lysis buffer 10X | Invitrogen | 00-4300-54 | Dilution 1:10 using sterile deionized water |
| Scissors | Roboz | RS-6762 | Used during harvest |
| Stainless steel surgical blade #4 | Bard-Parker | 371222 | Used during harvest |
| Sterile tray | Polar ware | 10F | Used during harvest |
| StretchEase Powder-free nitrile examination gloves | Denville Scientific | G4161 | Used during harvest |
| Surgical Scalpel handle #4 | Fischer | 12-000-164 | Used with surgical blade |
| TBS 20x | Thermo | Component of TBST, used at 1X | |
| Trypan Blue Stain (0.4%) | Gibco | 15250-061 | Used to count dead cells. Filtered with .45 µm |
| Tween 20 | Sigma Aldrich | P1379-500mL | Component of TBST |
| Tweezers | Miltex | 6-8 | Used during harvest |
| Vectashield Vibrance Antifade Mounting Medium with DAPI | Vector | H-1800 | Nuclear stain for immunofluorescence |
Références
- Larsen, S. B., Cowley, C. J., Fuchs, E. Epithelial cells: liaisons of immunity. Current Opinion in Immunology. 62, 45-53 (2020).
- Blanpain, C., Fuchs, E. Plasticity of epithelial stem cells in tissue regeneration. Science. 344 (6189), 1242281 (2014).
- Coradini, D., Casarsa, C., Oriana, S. Epithelial cell polarity and tumorigenesis: New perspectives for cancer detection and treatment. Acta Pharmacologica Sinica. 32 (5), 552-564 (2011).
- Dmello, C., et al. Multifaceted role of keratins in epithelial cell differentiation and transformation. Journal of Biosciences. 44 (2), 33 (2019).
- Poulet, G., Massias, J., Taly, V. Liquid biopsy: General concepts. Acta Cytologica. 63 (6), 449-455 (2019).
- Eslami-S, Z., Cortés-Hernández, L. E., Alix-Panabières, C. Epithelial cell adhesion molecule: an anchor to isolate clinically relevant circulating tumor cells. Cells. 9 (8), 1836 (2020).
- Morris, R. J. Circulating tumor cells: quintessential precision oncology presenting challenges for biology. NPJ Precision Oncology. 1 (1), 16 (2017).
- Lin, D., et al. Circulating tumor cells: biology and clinical significance. Signal Transduction and Targeted Therapy. 6 (1), 404 (2021).
- Spence, M., Adai, S., Landon, T., Richter, G. Few and far between: Tools and strategies for rare-event detection using flow cytometry. BioprobesJournal of Cell Biology. 71, 14-18 (2015).
- Yu, M., Stott, S., Toner, M., Maheswaran, S., Haber, D. A. Circulating tumor cells: approaches to isolation and characterization. Journal of Cell Biology. 192 (3), 373-382 (2011).
- Ye, Q., Ling, S., Zheng, S., Xu, X. Liquid biopsy in hepatocellular carcinoma: circulating tumor cells and circulating tumor DNA. Molecular Cancer. 18 (1), 114 (2019).
- Park, H., et al. marrow-derived epithelial cells and hair follicle stem cells contribute to development of chronic cutaneous neoplasms. Nature Communications. 9 (1), 5293 (2018).
- Marsman, W. A., et al. Epithelial cells in bone marrow: do they matter. Gut. 54 (12), 1821-1822 (2005).
- Borue, X., et al. marrow-derived cells contribute to epithelial engraftment during wound healing. The American Journal of Pathology. 165 (5), 1767-1772 (2004).
- Chakrabarti, S., et al. marrow-derived cells contribute to the maintenance of thymic stroma including TECs. Journal of Immunology Research. 2022, 6061746 (2022).
- Krause, D. S., et al. Multi-organ, multi-lineage engraftment by a single bone marrow-derived stem cell. Cell. 105 (3), 369-377 (2001).
- Wong, A. P., et al. Targeted cell replacement with bone marrow cells for airway epithelial regeneration. American Journal of Physiology-Lung Cellular and Molecular Physiology. 293 (3), L740-L752 (2007).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon