Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Cuantificación de la citotoxicidad celular dependiente de anticuerpos en un modelo de esferoide tumoral: aplicación para el descubrimiento de fármacos
En este artículo
Resumen
Aquí, presentamos un método para identificar compuestos que modulan el mecanismo ADCC, un importante mecanismo de destrucción de células cancerosas de anticuerpos antitumorales. El efecto citotóxico de las células NK se mide en esferoides de células de cáncer de mama en presencia de trastuzumab. El análisis de imágenes identifica células asesinas y diana vivas y muertas en esferoides.
Resumen
La inmunoterapia basada en anticuerpos monoclonales dirigida a los antígenos tumorales es ahora un pilar del tratamiento del cáncer. Uno de los mecanismos de acción clínicamente relevantes de los anticuerpos es la citotoxicidad celular dependiente de anticuerpos (ADCC, por sus siglas en inglés), en la que el anticuerpo se une a las células cancerosas y activa el componente celular del sistema inmunitario, por ejemplo, las células asesinas naturales (NK), para destruir las células tumorales. La eficacia de estas terapias podría mejorarse mediante la identificación de compuestos adyuvantes que aumenten la sensibilidad de las células cancerosas o la potencia de las células inmunitarias. Además, las interacciones farmacológicas no descubiertas en pacientes de cáncer comedicados por afecciones previas o síntomas asociados con el cáncer pueden determinar el éxito de la terapia con anticuerpos; por lo tanto, es necesario eliminar estas interacciones farmacológicas no deseadas. Con estos objetivos en mente, creamos un modelo de ADCC para el cáncer y describimos aquí un protocolo simple para encontrar fármacos moduladores de ADCC. Dado que los modelos 3D, como los esferoides de células cancerosas, son superiores a los cultivos 2D en la predicción de las respuestas in vivo de los tumores a las terapias contra el cáncer, los cocultivos esferoides de células de cáncer de mama HER2+ JIMT-1 que expresan EGFP y el NK92. Se establecieron líneas celulares CD16 y se indujeron con trastuzumab, un anticuerpo monoclonal clínicamente aprobado contra el cáncer de mama HER2 positivo. Se permitió que los esferoides JIMT-1 se formaran en placas de 96 pocillos con fondo en U repelentes a las células. El día 3 se añadieron células NK y trastuzumab. A continuación, los esferoides se tiñeron con anexina V-Alexa 647 para medir la muerte celular apoptótica, que se cuantificó en la zona periférica de los esferoides con un microscopio automatizado. La aplicabilidad de nuestro ensayo para identificar moléculas moduladoras de ADCC se demuestra al mostrar que Sunitinib, un inhibidor del receptor tirosina quinasa aprobado por la FDA contra el cáncer metastásico, elimina casi por completo el ADCC. La generación de esferoides y las canalizaciones de adquisición y análisis de imágenes son compatibles con el cribado de alto rendimiento de compuestos moduladores de ADCC en esferoides de células cancerosas.
Introducción
Los esferoides tumorales multicelulares (MCTS) son modelos tridimensionales (3D) ampliamente utilizados que se forman debido a la tendencia de las células adherentes a agregarse y representan una herramienta importante para obtener información mecanicista sobre la biología de las células cancerosas. Pueden generarse a partir de una amplia gama de tipos celulares mediante numerosas técnicas, como los cultivos 3D basados en líquidos y en andamios1. Su principal ventaja sobre los modelos 2D monocapa es que recapitulan las principales características de los tumores in vivo , a saber, la organización estructural y la hipoxia, imitando el comportamiento biológico de las células tumorales, especialmente los mecanismos que conducen al escape terapéutico y la resistencia a los fármacos2. Por lo tanto, dado que los MCTS pueden mejorar la previsibilidad de la toxicidad y la sensibilidad a los medicamentos, se utilizan ampliamente para estudiar cánceres en 3D y podrían mejorar el desarrollo de medicamentos efectivos para diferentes tipos de cáncer3.
Para estudiar cualquier enfermedad, existe una necesidad crítica de modelos relevantes y convenientes. La creación de modelos para estudios de inmunología del cáncer es un reto porque el sistema inmunitario está formado por varios tipos de células. Cada tipo de célula tiene varios subtipos y un amplio espectro de estados de activación. Estos diferentes tipos de células inmunitarias interactúan con las células cancerosas y otros componentes tumorales, lo que en última instancia influye en el resultado de la enfermedad. Los métodos de cultivo celular in vitro 2D no logran recapitular estas complejas interacciones celulares, ya que carecen de traducibilidad y son incapaces de predecir la acción de un fármaco a nivel de sistema (por ejemplo, en tejidos)4,5. Además, los modelos de ratón también tienen graves limitaciones debido a las diferencias fundamentales entre el sistema inmunitario humano y el murino. Los sistemas de cultivo 3D pueden, por lo tanto, llenar los vacíos actuales en los modelos disponibles, proporcionando un método alternativo y mejorando nuestra comprensión de la inmunología del cáncer6. En concreto, los modelos de esferoides podrían utilizarse para probar inmunoterapias, principalmente para evaluar la eficacia del cribado de fármacos y los anticuerpos terapéuticos para mejorar la infiltración de células inmunitarias y los efectos antitumorales contra las dianas esferoides7. Además, se ha demostrado ampliamente el potencial de los MCTS compuestos por células en diferentes estados metabólicos y proliferativos para estudiar las interacciones entre las células del estroma (por ejemplo, linfocitos, macrófagos, fibroblastos) y las células cancerosas y para el desarrollo de nuevas estrategias contra el cáncer8. Por lo tanto, existe una necesidad vital de corroborar plataformas predictivas y precisas para impulsar el proceso de prueba de fármacos, teniendo en cuenta la fisiopatología del microambiente tumoral.
El cáncer de mama (CM) es el cáncer más frecuente diagnosticado en mujeres a nivel mundial. La clasificación clínica de esta enfermedad heterogénea se basa en la presencia de receptores transmembrana, por ejemplo, receptores de estrógeno (RE) y progesterona (PR) (denominados colectivamente receptores hormonales, HR) junto con la sobreexpresión o amplificación de la proteína/oncogén del receptor 2 del factor de crecimiento epidérmico humano (HER2). Con base en la expresión inmunohistoquímica de estos receptores, se reconocen comúnmente cuatro subtipos: cáncer de mama luminal A (HR+/HER2-), luminal B (HR+/HER2+), HER2-positivo (HR-/HER2+) y triple negativo (HR-/HER2-). El grupo HER2+ constituye el 10-15% de los casos de CM y se caracteriza por una alta expresión de HER2 con ausencia de RE y PR, con peor pronóstico en comparación con los tumores luminales y requiriendo fármacos específicos dirigidos contra la proteína HER2/neu9.
El desarrollo del CM es un proceso de varios pasos, y un diagnóstico precoz es esencial para un tratamiento exitoso de la enfermedad10. Sin embargo, a pesar de las opciones de tratamiento personalizadas de la CM que han surgido recientemente (p. ej., terapias endocrinas y de anticuerpos anti-HER2), la CM sigue siendo un reto para los oncólogos. Al igual que la cirugía, la quimioterapia y la radioterapia, estas terapias personalizadas también pueden tener efectos adversos graves y los pacientes pueden desarrollar resistencia a estos agentes, lo que hace que sea un desafío a largo plazo determinar la mejor estrategia11,12. Por lo tanto, una mejor comprensión de la interacción entre el tumor y su microambiente es esencial y se espera que proporcione nuevas direcciones para el desarrollo de nuevos tratamientos que tengan en cuenta las especificidades de los diferentes subtipos de CM13. Se está estudiando una nueva ola de inmunoterapias, como los conjugados anticuerpo-fármaco, las terapias adoptivas de células T, las vacunas y los nuevos anticuerpos monoclonales (mAb) dirigidos a HER2 en una amplia población de pacientes con tumores que expresan HER214.
El trastuzumab, por ejemplo, representa una modalidad de tratamiento eficaz para el CM HER2+. Como parte de su modo de acción, el trastuzumab media las actividades dependientes del receptor gamma cristalizable de fragmentos (FcγR). Los FcγR se distinguen por su afinidad por el fragmento Fc y la respuesta inmunitaria que inician. La activación de FcγRIIIa (CD16A) en las células asesinas naturales (NK) es crucial para mediar la citotoxicidad celular dependiente de anticuerpos (ADCC), mientras que la activación de FcγRIIa (CD32A) y FcγRIIIa en macrófagos induce fagocitosis celular dependiente de anticuerpos (ADCP)15. Los estudios en modelos animales mostraron que los ratones que carecían de receptores FcγRI (CD64) y FcγRIII (CD16) no pudieron iniciar respuestas inmunitarias protectoras contra antígenos específicos del tumor, lo que revela que ADCC es probablemente un mecanismo de acción importante para el mAb Trastuzumab16.
Dado que las células NK recurren a los Abs unidos a las células tumorales para la destrucción de las células cancerosas por ADCC, la expresión de los receptores Fc es fundamental para un tratamiento eficaz con Trastuzumab17 (Figura 1). Además, su acción se equilibra eficientemente mediante la estimulación de los receptores activadores e inhibidores, por ejemplo, los receptores similares a las inmunoglobulinas de las células asesinas (KIR)18.
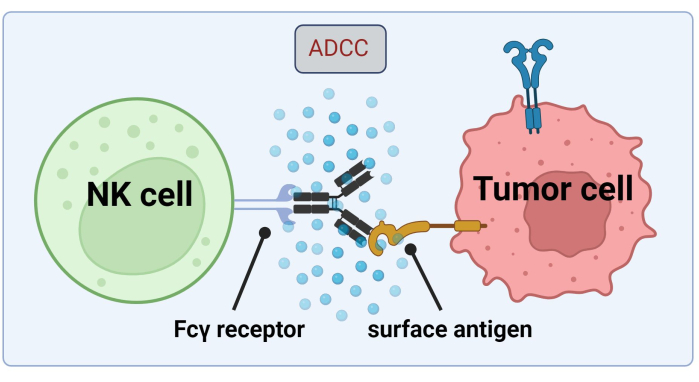
Figura 1. Mecanismo de ADCC en el contexto de una respuesta antitumoral. El receptor Fcγ de una célula asesina natural (NK) reconoce la región Fc de un anticuerpo, que previamente se había unido a un antígeno de superficie en una célula cancerosa. Esta sinapsis inmunológica conduce a la degranulación de la célula NK, que libera mediadores citotóxicos como las granzimas y la perforina. Estas moléculas contribuyen a la formación de poros en la membrana celular y activan las vías apoptóticas provocando la muerte celular programada de la célula diana (imagen creada con Biorender.com). Haga clic aquí para ver una versión más grande de esta figura.
El desarrollo de inmunoterapia para HER2+ BC representa un campo en evolución. En este caso, se deben considerar las interacciones entre varios componentes del sistema inmunológico. Además, publicaciones anteriores han probado ampliamente terapias combinadas que involucran todo tipo de terapias tradicionales, inmunes o celulares para identificar combinaciones sinérgicas19.
Varios modelos 3D de HER2+ BC se han utilizado previamente para el descubrimiento de fármacos. Por ejemplo, Balalaeva et al. utilizaron esferoides SKBR-3 que sobreexpresan HER2 para evaluar la citotoxicidad de la inmunotoxina 4D5scFv-PE40 dirigida a HER220. En otro estudio, se estableció un sistema de cultivo de HER2+ BC basado en 3D Matrigel, para medir el crecimiento celular en respuesta a Trastuzumab y agentes endocrinos21. Estos estudios ponen de manifiesto la importancia de los modelos de esferoides tumorales de células cancerosas con sobreexpresión de HER2 para representar una estrategia eficaz para mejorar clínicamente las respuestas terapéuticas22.
Nuestro grupo identificó previamente Sunitinib, un inhibidor de la tirosina quinasa multidirigido, como un inhibidor de ADCC dependiente de Trastuzumab en células JIMT-1 HER2+ BC en un ensayo de cultivo 2D. El estudio reveló que Sunitinib induce la autofagia y perjudica la función de destrucción de las células NK, regulando a la baja la expresión de HER2 y mejorando la adhesión a la superficie de las células JIMT-117.
Aquí establecimos un nuevo modelo ADCC esferoide en 3D (células cancerosas NK.92.CD16 + Trastuzumab + JIMT-1-EGFP) que se utilizará para aplicaciones de cribado de alto rendimiento y, con el fin de validar los hallazgos mencionados anteriormente, se utilizó Sunitinib como compuesto modelo. En primer lugar, generamos células EGFP que expresan JIMT-117 y cultivamos esferoides a partir de estas células. La ADCC fue inducida por células NK junto con Trastuzumab, y los esferoides se mantuvieron en cultivo en presencia o ausencia de compuestos de prueba durante 24 h (Figura 2). La cuantificación de ADCC se basa en la detección de la muerte de células cancerosas apoptóticas (tinción de anexina V) mediante un sistema de análisis de alto contenido.
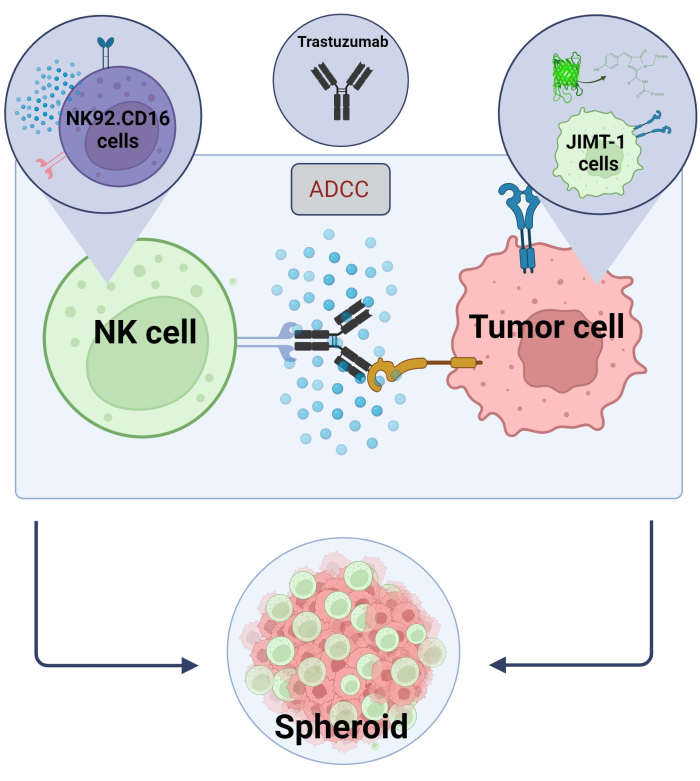
Figura 2. ADCC en un sistema de cocultivo de esferoides 3D. Nuestros entornos experimentales se basan en un sistema de esferoides 3D que puede modelar con mayor precisión el microentorno in vivo en comparación con los modelos 2D. Las células de cáncer de mama JIMT-1 EGFP se sembraron en un fondo cóncavo repelente de células para formar un grupo celular de forma redonda, llamado esferoide. A continuación, se inició ADCC añadiendo NK92. Células asesinas naturales CD16 (relación E:T = 20:1) y un anticuerpo monoclonal anti-HER2, Trastuzumab. El modelo experimental ha demostrado ser eficiente y fácilmente aplicable para la identificación de compuestos de prueba modificadores de ADCC (imagen creada con Biorender.com). Haga clic aquí para ver una versión más grande de esta figura.
Demostramos que la adquisición de datos de esta manera se puede hacer en tiempo real y es estadísticamente robusta para su uso en el cribado de alto contenido en el descubrimiento de fármacos contra el cáncer. Es importante destacar que este modelo permite una validación extendida de un conjunto más grande de compuestos y se puede aplicar a varios ensayos de interés.
Protocolo
1. Configuración del modelo de esferoide de proteína fluorescente mejorada con JIMT-1 (EGFP)
- Para formar un fondo repelente de células en forma de U, cubra la placa de 96 pocillos con una solución de agarosa-PBS al 0,5 % (30 μL/pocillo). Incubar la placa a temperatura ambiente durante aproximadamente 30-45 min.
- Lavar las células JIMT-1-EGFP dos veces con 2 mL de PBS estéril (la generación de la línea celular JIMT1-EGFP se informó en una publicación anterior17). Utilizar matraces de cultivo de tejidos T25 y medios JIMT-1 (medio DMEM/F-12 suplementado con suero fetal bovino (FBS) al 20%, insulina 0,3 U/ml (100 UI/ml, Humulin R) y penicilina-estreptomicina al 1%) para el cultivo celular.
- Añadir 2 mL de tripsina-EDTA al matraz e incubarlo durante 10 min en una incubadora de CO2 .
- Golpee el matraz para comprobar si las células JIMT-1 se desprendieron después del tiempo de incubación.
- Use 2 ml de medio JIMT-1 para detener la digestión y recoja la suspensión celular en un tubo de 15 ml.
- Cuente las células en una cámara de Bürker con azul de tripán al 0,4 % (80 μL del colorante + 20 μL de la suspensión celular) y ajuste el número de células a 20.000 células/mL.
- Pipetear 100 μL de la suspensión celular (2000 células/pocillo) en cada pocillo de la placa de 96 pocillos (pre-recubierto con solución de agarosa-PBS al 0,5% como se indica en 1.1).
- Deje que las células se agrupen durante un tiempo de incubación de 3 días a 37 °C en una incubadora de CO2 .
- Compruebe regularmente el tamaño y la forma de los esferoides con un microscopio invertido.
2. Recubrimiento de la placa HCS
NOTA: Para evitar la adhesión de esferoides JIMT-1-EGFP a la superficie de vidrio de la placa, es crucial recubrir la placa de cribado de alto contenido (HCS) (de lo contrario, no sería posible realizar un análisis de alto contenido).
- El día 3 después de la inducción de esferoides, recubra la placa de cribado de alto contenido de 96 pocillos con Pluronic-F127 (0,5% en DMSO, 50 μL/pocillo) e incube la placa durante 45 min a temperatura ambiente.
- Aspire la solución de recubrimiento y lave los pocillos dos veces con medio libre de suero DMEM/F-12 (100 μL/pocillo).
3. Transferencia de esferoides a la placa HCS
- Con una pipeta de 1 ml, transfiera los esferoides por triplicado a la placa HCS de 96 pocillos con fondo de vidrio.
4. Pretratamiento de esferoides JIMT-1 EGFP con compuestos problema
- Añadir el compuesto de prueba (p. ej., Sunitinib diluido en DMSO a una concentración de 40 μM) pipeteando 10 μL/pocillo y añadir 10 μL de medio JIMT-1 fresco a los pocillos de control (CTL).
- Incubar la placa durante 1 h en una incubadora deCO2 a 37 °C.
5. Inducción de ADCC mediante la adición de las células efectoras
NOTA: Las células CD16.176V.NK92 (en lo sucesivo denominadas células NK) se cultivaron en α-MEM suplementadas con 20% de FBS, 1% de MEM-NEAA, 1% de Na-piruvato, 1% de glutamina, 1% de penicilina-estreptomicina y 100 UI/ml de IL-2.
- Recoja las células NK del matraz en un tubo de 15 ml. Cuente las células con azul de tripano (80 μL del colorante + 20 μL de la suspensión celular) y ajuste la densidad celular a una relación efector-objetivo (E:T) de 20:1 (40.000 células NK/pocillo).
- Tiñir las células NK con 10 μM Cell Tracker Blue (CTB, 1 μL en 1 mL de medio α-MEM NK) y colocarlas en una incubadora de CO2 a 37 °C durante 1 h.
- Centrifugar las células NK a 150 x g durante 3 min a temperatura ambiente dos veces para lavar el exceso del colorante con 1 mL de medio α-MEM.
- Vuelva a suspender el gránulo de células NK en 1 ml de medio JIMT-1 fresco.
- Agregue las células NK teñidas junto con el anticuerpo anti-HER2 (Ab) (trastuzumab, disuelto en H2O destilado estéril) a los esferoides JIMT-1 diana pipeteando 55 μL/pocillo de células NK teñidas con CTB y 55 μL/pocillo de 10 μg/ml de Ab en medio JIMT-1 (el volumen total de tratamiento añadido para el ADCC es de 110 μL/pocillo y la concentración final de Sunitinib es de 20 μM).
NOTA: Para la selección de la concentración de Ab y la relación E:T, nos basamos en nuestra publicación anterior17. Se realizaron experimentos preliminares con diferentes relaciones E:T y concentraciones de Trastuzumab para evaluar cuál era el más eficaz en la inducción de ADCC en esferoides (Figura suplementaria S1). - Agregue 110 μL/pocillo de medio JIMT-1 fresco a los pocillos de control (CTL).
- Incubar la placa durante 24 h en una incubadora de CO2 a 37 °C.
NOTA: Dado que se sabe que las células NK92 ejercen funciones citotóxicas no específicas, para el control se utilizan células JIMT-1 coincubadas con células NK solas y con un control de isotipo o un Ab F(ab')2. En este caso, se utilizó F(ab')2-trastuzumab (TR-F(ab')2) como CTL negativo. El fragmento TR-F(ab')2 se preparó según lo informado por Tóth et al.19 y se agregó en el mismo volumen que Trastuzumab junto con células NK como se describe en 5.5. La concentración se ajustó a 6,6 μg/mL correspondiente a una concentración equimolar con Trastuzumab23.
6. Tinción de anexina V-647 de esferoides
- Para medir la muerte celular apoptótica, tiñir los esferoides con el conjugado de anexina V-Alexa Fluor 647 durante 1 h en medio JIMT-1 (1:100) pipeteando 50 μL/pocillo.
7. Imágenes
NOTA: La imagen de la placa se obtiene a las 24 h después de la adición de las células efectoras NK a las células diana. Para la obtención de imágenes, se utilizan un analizador de alto contenido y un software de análisis de imágenes.
- Seleccione el tipo de microplaca de la lista de placas mediante la opción Tipo de placa . Utilice una placa de cribado de alto contenido (HCS) de 96 pocillos.
- Seleccione Enfoque automático de dos picos, ya que el ensayo se lleva a cabo en placas con objetivo de 10x con apertura numérica (NA) de 0,3, utilizando las opciones Enfoque automático y Objetivo , respectivamente. Elija el modo confocal con la opción Modo de opción y aplique la agrupación 2 con la opción Agrupación .
- Para obtener imágenes de esferoides, seleccione los canales apropiados mediante la opción Selección de canal . Para detectar las células JIMT-1 transducidas por EGFP, elija EGFP (tiempo de integración de 200 ms, potencia del láser del 50%, altura de la pila de 2,0 μm; por ejemplo: 488 nm em: 500-550 nm), y para detectar células apoptóticas utilice Alexa 647 (tiempo de integración de 100 ms, potencia del láser del 50%, altura de la pila de 10,0 μm; por ejemplo: 640 nm em: 650-760 nm). Para visualizar las células NK dentro de los esferoides, elija el siguiente canal: DAPI (tiempo de integración 100 ms, potencia láser 50%, altura de la pila 2,0 μm; ej: 405 nm em: 435-480 nm).
- En la opción Selección de diseño , seleccione pilas Z, ya que las células de los esferoides tienden a estar en diferentes planos focales del microscopio. 10 planos (con una distancia de 10 μm) son suficientes para cubrir toda la región esferoide. Establezca los valores del primer plano y del último plano en 0 μm y 90 μm, respectivamente.
NOTA: Antes de iniciar la medición, se pueden tomar imágenes de muestra con la función de instantánea para verificar la configuración correcta. - Establezca el número de pozos y campos para la creación de imágenes mediante la opción Definir diseño .
8. Análisis de alto contenido (HCA)
NOTA: Analice las imágenes con el software Harmony o exporte las imágenes para analizarlas con un software de terceros preferido. Para el análisis de la eficiencia de ADCC, se mide la intensidad de fluorescencia de la anexina V 647. Las células diana eliminadas por ADCC aparecen en el área periférica de los esferoides. Por lo tanto, las células positivas para la anexina V se miden en este "anillo" esferoide. Con el fin de validar este método, se realizaron análisis con diferentes parámetros para evaluar cuál era el más fiable y el que producía mejores resultados (Figura complementaria S2). En el vídeo se muestra un pocillo ADCC para demostrar paso a paso el proceso de análisis de imágenes.
- Identifique los esferoides por la fluorescencia EGFP de las células JIMT-1 utilizando la opción Buscar regiones de textura y fíltrelos por tamaño (> 25.000 μm2).
- Elimine los objetos de borde mediante la opción Seleccionar población .
- Dado que las células positivas a la anexina V (apoptóticas) aparecen en la periferia de los esferoides, mida la intensidad de la anexina en este "anillo" apoptótico, seleccionado por la opción Seleccionar región (borde exterior -90%) para determinar la muerte celular apoptótica.
- Exprese los valores de intensidad de la anexina V como intensidad media.
Resultados
Se generaron células JIMT-1 que expresan EGFP y se cultivaron esferoides a partir de estas células. El sunitinib se utilizó como compuesto de prueba, ya que anteriormente se había demostrado que afectaba al curso de ADCC17. Los esferoides se dejaron agrupar durante 72 h. En el día 3, se añadieron 10 μg/ml de trastuzumab (o equimolar 6,6 μg/mL de TR-F(ab')219) y células NK (20:1) a los esferoides en presencia o ausencia de 20 μM de sunitinib (1 h de pret...
Discusión
A pesar de las mejoras significativas en el tratamiento de la CM en las últimas décadas, los pacientes todavía desarrollan regularmente resistencia a la medicación o experimentan efectos secundarios negativos24. La alta morbilidad y mortalidad ligada a la CM exige una investigación continua de los mecanismos moleculares subyacentes, así como plataformas de cribado robustas para identificar nuevas moléculas procesables para el desarrollo terapéutico25. Estas estrateg...
Divulgaciones
Los autores declararon no tener conflictos de intereses.
Agradecimientos
LV recibió financiación de las subvenciones de la Oficina Nacional de Investigación, Desarrollo e Innovación GINOP-2.3.2-15-2016-00010 TUMORDNS", GINOP-2.3.2-15-2016-00048-STAYALIVE, OTKA K132193 y K147482. Este proyecto ha recibido financiación de la Red Húngara de Investigación HUN-REN. Las células CD16.176V.NK-92 se obtuvieron del Dr. Kerry S. Campbell (Fox Chase Center, Filadelfia, PA, en nombre de Brink Biologics, lnc. San Diego, CA), están protegidos por patentes en todo el mundo y fueron licenciados por Nantkwest, lnc. (www.nantkwest.com). Los autores agradecen a György Vereb y Árpád Szöőr su ayuda con el uso de la línea celular NK-92 y el TR-F(ab')2, y por el asesoramiento técnico.
Materiales
| Name | Company | Catalog Number | Comments |
| 96-well glass bottom Cell Carrier Ultra microplates | PerkinElmer, Waltham, MA, USA | LLC 6055302 | for spheroids measurements |
| 96-well tissue culture plates | TPP | 92096 | for cell seeding |
| α-MEM medium | Sigma | M8042 | in NK medium |
| Agarose | Sigma | A9539 | for spheroids seeding |
| Annexin V-Alexa Fluo 647 conjugate | Invitrogen-ThermoFisher Scientific | A23204 | for apoptosis measurement with HCS |
| CD16.176 V.NK-92 cells | Dr. Kerry S. Campbell (the Fox Chase Cancer Center, Philadelphia, PA on behalf of Brink Biologics, Inc. San Diego, CA) | ATCC CRL-2407 | for cell culture |
| Cell Tracker Blue | Invitrogen-Thermo Scientific, Waltham, MA, USA) | C2110 | for staining of NK cells |
| DMEM/F-12 medium | Sigma | D8437 | in JIMT1-EGFP medium |
| Dimethyl sulfoxide (DMSO) | Sigma | D8418 | for coating HCS plate before transfering the spheroids |
| Fetal bovine serum (FBS) | Biosera | FB-1090/500 | JIMT-1-EGFP and NK medium |
| Glutamine | Gibco | 35,050–061 | in NK medium |
| GraphPad Prism 8.0.1 | GraphPad Software Inc., San Diego, CA, USA | for statistical analysis | |
| Harmony software | PerkinElmer, Waltham, MA, USA | for HCA | |
| IL-2 | Proleukin, Novartis Hungária Kft., Budapest, Hungary | PHC0026 | in NK medium |
| Insulin (Humulin R) | Eli Lilly | HI0219 | JIMT-1-EGFP medium |
| JIMT-1 breast cancer cells | for cell culture | ||
| MEM Non-essential Amino Acids (MEM-NEAA) | Gibco | 11,140–050 | in NK medium |
| Na-pyruvate | Lonza | BE13-115E | in NK medium |
| Opera Phenix High-Content Analysis equipment | PerkinElmer, Waltham, MA, USA | HH14001000 | for HCA |
| PBS (Posphate buffered saline) | Lonza | BE17-517Q | for washing the cells |
| Penicillin-Streptomycin | Biosera | LM-A4118 | JIMT-1-EGFP and NK medium |
| pLP-1, pLP-2, pLP-VSV-G, pWOXEGFP | Invitrogen, (Prof. József T zsér, University of Debrecen) zsér, University of Debrecen) | for JIMT-1-EGFP cell line | |
| Pluronic-F127 | Sigma | P2443 | for coating HCS plate before transfering the spheroids |
| Sunitinib malate | SigmaAldrich | PZ0012 | for treatments |
| Trastuzumab Ab (humanized anti-HER2 monoclonal antibody) | Herzuma®, EGIS Pharmaceuticals, Budapest, Hungary | NDC-63459-303-43 | for treatments |
| Trastuzumab-F(ab')2 | Gift from Prof. György Vereb and Árpád Szö r r | Department of Biophysics and Cell Biology, University of Debrecen | for treatments |
| Trypan blue 0.4% solution | Sigma | T8154 | for cell counting |
| Trypsin-EDTA 1X in PBS | Biosera | LM-T1706 | for cells detachment |
Referencias
- Jubelin, C., et al. Three-dimensional in vitro culture models in oncology research. Cell Biosci. 12 (1), 155 (2022).
- Mittler, F., et al. High-content monitoring of drug effects in a 3D spheroid model. Front Oncol. 7, 293 (2017).
- Badr-Eldin, S. M., Aldawsari, H. M., Kotta, S., Deb, P. K., Venugopala, K. N. Three-dimensional in vitro cell culture models for efficient drug discovery: Progress so far and future prospects. Pharmaceuticals (Basel). 15 (8), 926 (2022).
- Brancato, V., Oliveira, J. M., Correlo, V. M., Reis, R. L., Kundu, S. C. Could 3D models of cancer enhance drug screening. Biomaterials. 232, 119744 (2020).
- Pinto, B., Henriques, A. C., Silva, P. M. A., Bousbaa, H. Three-dimensional spheroids as in vitro preclinical models for cancer research. Pharmaceutics. 12 (12), 1186 (2020).
- Fitzgerald, A. A., Li, E., Weiner, L. M. 3D culture systems for exploring cancer immunology. Cancers (Basel). 13 (1), 56 (2020).
- Zanoni, M., et al. 3D tumor spheroid models for in vitro therapeutic screening: A systematic approach to enhance the biological relevance of data obtained. Sci Rep. 6, 19103 (2016).
- Boucherit, N., Gorvel, L., Olive, D. 3D tumor models and their use for the testing of immunotherapies. Front Immunol. 11, 603640 (2020).
- Li, Y., et al. Recent progress on immunotherapy for breast cancer: Tumor microenvironment, nanotechnology and more. Front Bioeng Biotechnol. 9, 680315 (2021).
- Lo Nigro, C., et al. NK-mediated antibody-dependent cell-mediated cytotoxicity in solid tumors: Biological evidence and clinical perspectives. Ann Transl Med. 7 (5), 105 (2019).
- Sun, Y. S., et al. Risk factors and preventions of breast cancer. Int J Biol Sci. 13 (11), 1387-1397 (2017).
- Liu, P. H., Wei, J. C., Wang, Y. H., Yeh, M. H. Female breast cancer incidence predisposing risk factors identification using nationwide big data: A matched nested case-control study in taiwan. BMC Cancer. 22 (1), 849 (2022).
- Burguin, A., Diorio, C., Durocher, F. Breast cancer treatments: Updates and new challenges. J Pers Med. 11 (8), 808 (2021).
- Costa, R. L. B., Czerniecki, B. J. Clinical development of immunotherapies for HER2(+) breast cancer: A review of HER2-directed monoclonal antibodies and beyond. NPJ Breast Cancer. 6, 10 (2020).
- Musolino, A., et al. Role of fcgamma receptors in HER2-targeted breast cancer therapy. J Immunother Cancer. 10 (1), e003171 (2022).
- Petricevic, B., et al. Trastuzumab mediates antibody-dependent cell-mediated cytotoxicity and phagocytosis to the same extent in both adjuvant and metastatic HER2/NEU breast cancer patients. J Transl Med. 11, 307 (2013).
- Guti, E., et al. The multitargeted receptor tyrosine kinase inhibitor sunitinib induces resistance of HER2 positive breast cancer cells to Trastuzumab-mediated ADCC. Cancer Immunol Immunother. 71 (9), 2151-2168 (2022).
- Barok, M., et al. Trastuzumab decreases the number of circulating and disseminated tumor cells despite Trastuzumab resistance of the primary tumor. Cancer Lett. 260 (1-2), 198-208 (2008).
- Toth, G., et al. The combination of Trastuzumab and Pertuzumab administered at approved doses may delay development of Trastuzumab resistance by additively enhancing antibody-dependent cell-mediated cytotoxicity. MAbs. 8 (7), 1361-1370 (2016).
- Ehlers, F. a. I., et al. ADCC-inducing antibody Trastuzumab and selection of KIR-HLA ligand mismatched donors enhance the NK cell anti-breast cancer response. Cancers (Basel). 13 (13), 3232 (2021).
- Gangadhara, S., Smith, C., Barrett-Lee, P., Hiscox, S. 3d culture of Her2+ breast cancer cells promotes AKT to MAPK switching and a loss of therapeutic response. BMC Cancer. 16, 345 (2016).
- Balalaeva, I. V., Sokolova, E. A., Puzhikhina, A. D., Brilkina, A. A., Deyev, S. M. Spheroids of Her2-positive breast adenocarcinoma for studying anticancer immunotoxins in vitro. Acta Naturae. 9 (1), 38-43 (2017).
- Selis, F., et al. Pegylated Trastuzumab fragments acquire an increased in vivo stability but show a largely reduced affinity for the target antigen. Int J Mol Sci. 17 (4), 491 (2016).
- Johnston, R. L., et al. High content screening application for cell-type specific behaviour in heterogeneous primary breast epithelial subpopulations. Breast Cancer Res. 18 (1), 18 (2016).
- Kandaswamy, C., Silva, L. M., Alexandre, L. A., Santos, J. M. High-content analysis of breast cancer using single-cell deep transfer learning. J Biomol Screen. 21 (3), 252-259 (2016).
- Esquer, H., Zhou, Q., Abraham, A. D., Labarbera, D. V. Advanced high-content-screening applications of clonogenicity in cancer. SLAS Discov. 25 (7), 734-743 (2020).
- Pickl, M., Ries, C. H. Comparison of 3D and 2D tumor models reveals enhanced Her2 activation in 3D associated with an increased response to trastuzumab. Oncogene. 28 (3), 461-468 (2009).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados