Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Nuevo protocolo de cuantificación para la progresión de la calcificación cardiovascular mediante imágenes longitudinales de MicroPET/MicroCT
En este artículo
Resumen
Este novedoso protocolo implica la cuantificación de la progresión de la calcificación cardiovascular a partir de imágenes seriadas de microtomografía por emisión de positrones (PET)/microtomografía computarizada (TC) en pequeños animales de investigación.
Resumen
La tomografía por emisión de micropositrones (PET) y la microtomografía computarizada (TC) son herramientas de investigación poderosas e ideales para seguir la progresión de la calcificación cardiovascular. Debido a su naturaleza no invasiva, se pueden obtener imágenes de pequeños animales de investigación en múltiples puntos de tiempo. El reto radica en la cuantificación precisa de la calcificación cardiovascular. Aquí, proporcionamos un protocolo, utilizando imágenes de las etapas posteriores de la enfermedad como plantilla, para cuantificar con precisión la progresión de la calcificación cardiovascular en estudios longitudinales. El protocolo implica 1) la alineación del área del tórax en múltiples imágenes del mismo animal durante un estudio longitudinal como primer paso, 2) la definición de una región de interés (ROI) situada dentro del corazón y la aorta en el sitio de los depósitos de calcio más grandes que se hacen evidentes en imágenes posteriores, y 3) segmentación y cuantificación simultáneas de los depósitos de calcio en todas las imágenes adquiridas durante el estudio longitudinal. Este método simplificado mejora la precisión del análisis de imágenes en el seguimiento de la progresión de la calcificación cardiovascular al mejorar la precisión de la definición del ROI y reducir la variabilidad asociada con las técnicas anteriores que analizan las exploraciones individuales de forma independiente.
Introducción
Las enfermedades cardiovasculares son una de las principales causas mundiales de morbilidad y mortalidad, lo que exige una exploración rigurosa para descubrir sus mecanismos y diseñar estrategias preventivas y terapéuticas eficaces. La calcificación de las arterias coronarias (CAC) es ampliamente reconocida por los expertos en la materia como un factor predictivo de enfermedad cardiovascular, elevando significativamente el riesgo de mortalidad cardiovascular 1,2,3,4,5. Las calcificaciones microscópicas se consideran las etapas más tempranas de la aterosclerosis calcificada, y el término "microcalcificaciones" se ha utilizado para referirse a depósitos de calcio de entre 0,5 y 50 μm de 6,7,8,9 de diámetro. Se cree que estas pequeñas calcificaciones se fusionan para formar depósitos de calcio más grandes, lo que alimenta la progresión de la placa calcificante 6,7.
La tomografía por emisión de positrones (PET) y la tomografía computarizada (TC) son valiosas herramientas de investigación, frecuentemente empleadas para la evaluación no invasiva de la calcificación cardiovascular in vivo 5,10,11,12,13,14,15,16,17,18,19 . Estas modalidades de imagen resultan particularmente ventajosas para el seguimiento de la progresión de la calcificación vascular en estudios longitudinales con pequeños animales de investigación 11,12,13,19. La microTC ha demostrado su eficacia en la obtención de imágenes anatómicas de depósitos de calcio relativamente grandes 11,12,13,19,20.Sin embargo, su utilidad para obtener imágenes de pequeños depósitos de calcio en animales vivos está limitada por su resolución espacial de ~100 μm 8,14, lo que dificulta la investigación de la calcificación durante sus fases iniciales.
Un avance notable es la adopción de imágenes combinadas de microPET/microCT con el trazador PET, fluoruro de sodio marcado con fluoruro-18 (18F-NaF), como método estándar para la detección de calcificación basada en su unión al área superficial mineral. Este enfoque utiliza 18F-NaF radiomarcado, que se ha demostrado que identifica la superficie mineral de calcio10,13 a medida que los iones de fluoruro se unen covalentemente a la hidroxiapatita de calcio, reemplazando a los grupos hidroxilo para formar fluoroapatita21. De acuerdo con la tasa de cambio más lenta en relación con la desintegración radiactiva del 18F (vida media ~ 110 min) y con el aclaramiento del trazador a través de los riñones22, Irkle y sus colegas13 encontraron que la unión del 18F-NaF a los especímenes de carótida calcificada se limitaba a la superficie en el momento de la detección. Por lo tanto, la absorción de trazadores debe relacionarse directamente con el área de superficie mineral, que es mayor cuando una cantidad dada de mineral se encuentra en múltiples focos pequeños o en forma porosa que cuando está presente en pocos depósitos grandes y sólidos. Al acentuar las etapas más tempranas de la mineralización con alta sensibilidad, las imágenes PET con 18F-NaF pueden proporcionar información valiosa sobre las etapas tempranas de la enfermedad, lo que las hace particularmente útiles en el estudio de estrategias preventivas y terapéuticas 13,14,15.
Incluso con los avances recientes en la combinación de imágenes microPET/microCT de la calcificación vascular, existen oportunidades para mejorar la precisión del análisis de imágenes en estudios longitudinales de calcificación cardiovascular. Los enfoques convencionales utilizan la delineación manual de regiones de interés (ROI) laboriosas alrededor de cada región calcificada visible en cada ratón en cada punto de tiempo individual a lo largo del estudio longitudinal. Este método reduce la precisión, particularmente en las primeras etapas de la enfermedad, cuando los tamaños de los depósitos de calcio se acercan a los límites de detección de los escáneres, lo que puede invisibilizar algunas áreas con depósitos minúsculos dispuestos en baja densidad.
En el ámbito de la imagen, la alineación generalmente se refiere a la alineación espacial de una serie de imágenes. Al introducir la alineación como una solución novedosa a los desafíos existentes, nuestro método propuesto permite a los investigadores utilizar una ubicación consistente para seguir la progresión de la calcificación en imágenes en serie de sujetos individuales a lo largo de un estudio longitudinal. Dado que se sabe que la calcificación de los tejidos surge de vesículas de matriz de tamaño nanométrico (50-150 nm), que se unen para formar un mineral de hidroxiapatita microscópico y luego macroscópico23, se puede identificar retrospectivamente dónde se habrían ubicado las microcalcificaciones en las primeras imágenes antes de que sean discernibles.
Seguir esa misma ubicación a lo largo del tiempo, lo que es posible gracias a la alineación de la imagen, es la base de este método. Evita la necesidad de identificar directamente las primeras etapas de calcificación, ya que el ROI se asigna en función de las últimas etapas en las que se identifica fácilmente la mineralización. En este protocolo, presentamos un método de análisis de datos mejorado y simplificado que incorpora la alineación de una serie temporal de imágenes como un paso vital, mejorando la cuantificación precisa de la deposición de calcio en estudios de imágenes longitudinales, combinados, microPET/microCT (Figura 1). Si bien utilizamos el análisis de datos PET/CT como ejemplo, este método se puede aplicar a los análisis de otros datos de imágenes longitudinales, incluida la tomografía computarizada por emisión de fotón único (SPECT), la resonancia magnética (RM) y la imagen óptica24.
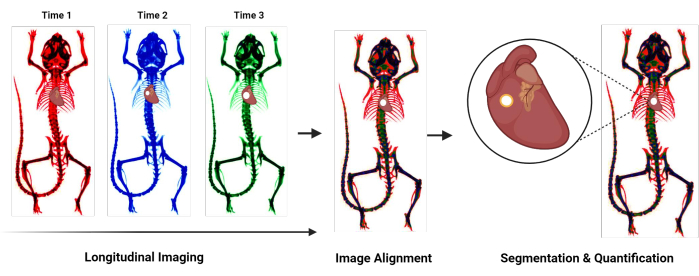
Figura 1: Diagrama de flujo de descripción general del protocolo. Diagrama de flujo que resume el novedoso protocolo para cuantificar la calcificación cardiovascular. Los pasos generales incluyen la obtención de imágenes longitudinales, la alineación de imágenes adquiridas en diferentes puntos de tiempo y la segmentación y cuantificación de la región calcificada. Haga clic aquí para ver una versión más grande de esta figura.
Protocolo
Las imágenes representativas presentan un ratón hembra con deficiencia de apolipoproteína E (Apoe-/-). Los protocolos experimentales fueron revisados y aprobados por el Comité Institucional de Cuidado y Uso de Animales de la Universidad de California, Los Ángeles.
1. Escaneo de animales
- Entre las dos adquisiciones de imágenes, alimente al ratón con una dieta estándar sin ninguna intervención. Sin embargo, antes de la primera imagen a los 15 meses de edad, cambie a una dieta occidental (21% de grasa, 0.2% de colesterol) de los 12 a los 14 meses de edad para inducir la calcificación aórtica basal.
- Asegúrese de que el escáner de microPET/CT se calibre de forma rutinaria para una adquisición óptima de datos de PET y co-registro de CT. Realice las calibraciones de acuerdo con las instrucciones del fabricante.
- Anestesiar al ratón con gas isoflurano al 2% en una cámara durante 10 minutos antes de la toma de imágenes. Confirme la anestesia pellizcando el dedo del pie del animal sin respuesta.
- Adquiera imágenes de microPET (350-650 keV, 10 min de tiempo de escaneo) y 100 μm de microCT (80 kVp, 150 μA, 720 de proyección, 1 min de tiempo de escaneo) en un escáner combinado de microPET/CT 60 min después de la inyección intravenosa de 3.7 MBq de 18F-NaF. Adquiera las exploraciones de microCT inmediatamente después de las exploraciones de microPET.
NOTA: La cama del escáner movió al animal del modal de PET al modal de TC mientras el animal permanecía bajo anestesia y en la misma posición. - Reconstruya imágenes microPET mediante un algoritmo de maximización de expectativas de subconjuntos ordenados en 3D (24 subconjuntos y 3 iteraciones), con corrección aleatoria, de atenuación y de decaimiento. Reconstruya las imágenes de TC utilizando un algoritmo de Feldkamp modificado.
NOTA: Después de la toma de imágenes, el ratón fue monitoreado hasta que recuperó la conciencia suficiente para mantener la decúbito esternal. Al final del estudio longitudinal, todos los ratones fueron sacrificados.
2. Importar archivos DICOM en el software de visualización DICOM
NOTA: Si bien este protocolo representativo utiliza el software ORS Dragonfly bajo una licencia no comercial, su flexibilidad se extiende a otras opciones de software de visor DICOM.
- Inicie el software del visor DICOM haciendo doble clic en la aplicación.
- Vaya a Archivo en la esquina superior izquierda. En el menú desplegable, seleccione Importar imágenes DICOM... para que aparezca la ventana Administrar imágenes DICOM (Archivo complementario 1- Figura complementaria S1A).
- Dentro de la ventana Administrar imágenes DICOM , haga clic en la pestaña Contenido de la carpeta . En el lado derecho, seleccione Abrir carpeta... para elegir las carpetas que contienen los archivos DICOM de interés en un estudio longitudinal (Archivo Suplementario 1- Figura Suplementaria S1B).
- Identifique las carpetas que contienen los archivos DICOM. Haga clic en Seleccionar carpeta para importar la primera carpeta a la ventana Administrar imágenes DICOM (Archivo complementario 1- Figura complementaria S1C).
- Seleccione los archivos DICOM PET ("PT") y CT ("CT") en la ventana Administrar imágenes DICOM . A continuación, haga clic en Ver estudio en el lado derecho para abrir todas las imágenes DICOM importadas (Archivo complementario 1- Figura complementaria S1D).
- Repita los pasos 2.4 y 2.5 para cada carpeta que contenga imágenes DICOM relacionadas con el mismo sujeto en el estudio longitudinal.
3. Ajuste la configuración del visor DICOM para optimizar la visualización de imágenes
- Cambie el diseño para mostrar cuatro vistas (3D, transversal, coronal, sagital), como se ilustra en el Archivo Suplementario 1- Figura Suplementaria S2. Dentro del menú desplegable Diseño en el lado izquierdo, busque Vistas (en la escena seleccionada) y seleccione un diseño que muestre cuatro vistas.
- Cambie el nombre de las imágenes para indicar los puntos de tiempo y si son imágenes PET o CT. Seleccione el nombre de la imagen y haga doble clic para cambiar el nombre de cada imagen.
- Para activar una imagen, acceda al menú desplegable Propiedades y configuración de datos en el lado derecho. Haga clic en el icono del ojo a la izquierda de cada nombre de imagen para alternar la visibilidad (Archivo complementario 1- Figura complementaria S2)
- Ajuste el brillo y el contraste de cada imagen de TC, individualmente.
NOTA: El brillo y el rango de contraste óptimos de la TC pueden variar según el sujeto, el protocolo de imagen, el escáner y los parámetros de reconstrucción25,26; Sin embargo, todos los ajustes deben ser consistentes para un solo sujeto en un estudio longitudinal.- Seleccione y active una imagen de TC haciendo clic en el nombre de la imagen en Propiedades y configuración de datos (Archivo complementario 1- Figura complementaria S2).
- Busque el histograma dentro del menú desplegable Nivelación de ventana ubicado en la pestaña Principal en el lado izquierdo de la pantalla de visualización. Active LogY encima del histograma; luego, proceda a hacer clic y deslizar los indicadores de rango amarillo para abarcar completamente el segundo pico en el histograma (Archivo Suplementario 1- Figura Suplementaria S3A). Realice ligeros ajustes hasta alcanzar el brillo/contraste óptimos; consulte la Figura 2B para ver un ejemplo de una imagen de TC ajustada por brillo/contraste.
- Busque el cuadro Rango seleccionado debajo del histograma de nivelación de ventana (Archivo complementario 1- Figura suplementaria S3B). Tome nota de los valores de Rango seleccionado para utilizarlos en los pasos siguientes.
- Seleccione y encienda una segunda imagen de TC (Archivo Suplementario 1- Figura Suplementaria S3C). Ingrese los valores de rango seleccionados de la primera imagen de TC en el cuadro Rango seleccionado para la segunda imagen de TC (Archivo complementario 1- Figura complementaria S3B-S3D). Repita el procedimiento para todas las imágenes consecutivas.
- Refina cada imagen PET ajustando el filtro Tabla de búsqueda, individualmente.
NOTA: El rango óptimo de la escala de colores de la PET puede variar según el sujeto, el protocolo de imagen, el escáner y los parámetros de reconstrucción27; Sin embargo, todos los ajustes deben ser consistentes para un solo sujeto en un estudio longitudinal.- Seleccione y active una imagen PET haciendo clic en el nombre de la imagen en Propiedades y configuración de datos (Archivo complementario 1- Figura complementaria S2).
- En el menú desplegable Nivelación de ventana en la pestaña Principal , busque Tabla de búsqueda (LUT) y desplácese para encontrar PET en el menú desplegable LUT y seleccione el filtro PET (Archivo complementario 1- Figura complementaria S4A).
- Ajuste el rango seleccionado utilizando el histograma en el menú desplegable Nivelación de ventana para lograr una visualización óptima. Active LogY encima del histograma; luego, proceda a hacer clic y deslice los indicadores de rango amarillo hacia la posición que se muestra en la Figura Suplementaria S4B (Archivo Suplementario 1). Realice ligeros ajustes hasta que se logre el rango seleccionado óptimo para la visualización de imágenes PET; consulte la Figura 2D para ver un ejemplo de una imagen de PET/CT ajustada para el rango seleccionado óptimo.
- Busque el menú desplegable Rango seleccionado en el histograma de nivelación de la ventana (Archivo complementario 1- Figura complementaria S4B). Tome nota de los valores de rango seleccionados para utilizarlos en los pasos siguientes.
- Seleccione y encienda una segunda imagen PET (Archivo complementario 1- Figura S4C). Repita el paso 3.5.2 para la segunda imagen.
- Introduzca los valores de rango seleccionados de la primera imagen PET en el cuadro Rango seleccionado para la segunda imagen PET (Archivo complementario 1- Figura S4B-S4D). Repita el procedimiento para todas las imágenes consecutivas.
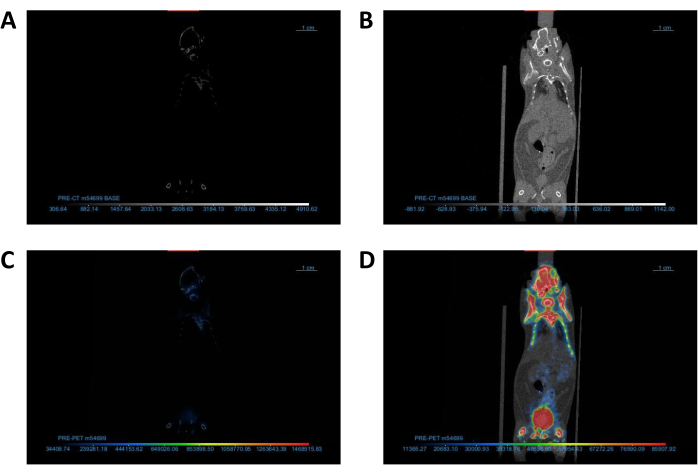
Figura 2: Ajuste de la configuración del visor DICOM para optimizar la visualización de imágenes. (A,B) Vista coronal de imágenes de TC (A) antes y (B) después del ajuste de contraste/brillo. (C,D) Vista coronal de las imágenes PET/CT (C) antes y (D) después del ajuste de la tabla de búsqueda. Abreviaturas: TC = tomografía computarizada; PET = tomografía por emisión de positrones. Haga clic aquí para ver una versión más grande de esta figura.
4. Alinee el área del tórax en las imágenes de TC
NOTA: Para simplificar, una imagen de TC servirá como imagen "base" y no se traducirá ni se rotará. La segunda imagen de TC (y eventualmente las imágenes seriadas subsiguientes) se traducirá y/o rotará para alinearse con la imagen base; A esto se le llamará imagen "superpuesta" para el propósito de la demostración. A lo largo de la alineación, es importante distinguir entre la imagen base y todas las demás imágenes superpuestas.
- Apague todas las imágenes PET y active la imagen CT base haciendo clic en el icono del ojo a la izquierda de cada nombre de imagen en el menú desplegable Propiedades y configuración de datos .
- Cambie la opacidad de la imagen CT a ~50%. Haga clic en una de las vistas 2D. En el cuadro Nivelación de ventana , busque el menú desplegable Mapeo de opacidad y deslice la escala de opacidad hacia el centro (Archivo complementario 1- Figura S5).
- Encienda la imagen de TC superpuesta y repita el paso 4.2.
- Cambie el filtro de la imagen de TC superpuesta para distinguir entre la imagen de TC base y la imagen de TC superpuesta. En el menú desplegable Nivelación de ventana en la pestaña Principal , busque Tabla de búsqueda (LUT). En el menú desplegable LUT, desplázate y haz clic en el filtro rojo .
NOTA: Ahora debería haber dos imágenes de TC en cada vista, como se ilustra en la Figura Suplementaria S5 (Archivo Suplementario 1). - Utilice las herramientas Trasladar/Rotar en las vistas 2D para alinear el área del pecho de la imagen superpuesta lo más cerca posible de la imagen base. Refiérase a la caja torácica, la columna vertebral superior y el esternón como indicadores de alineación del tórax en la imagen de TC.
NOTA: Las siguientes acciones solo se pueden ejecutar en vistas 2D (Transversal, Coronal o Sagital) (Archivo Suplementario 1- Figura Suplementaria S1). Para obtener el mejor resultado, cambie entre las tres vistas 2D y utilice las herramientas de traslado/rotación para alinear el área del pecho en cada vista.- Seleccione la imagen de superposición en el menú desplegable Propiedades y configuración de datos y haga clic una vez para seleccionar una de las vistas 2D.
- Busque el menú desplegable Traducir/Rotar en la pestaña Principal y haga clic en el icono Desplazar (Archivo complementario 1- Figura S6A).
- Haga clic y arrastre el cuadro de traducción en la esquina inferior izquierda y gire el punto de pivote en el centro de la vista 2D seleccionada para mover la imagen superpuesta para alinearla aproximadamente con la imagen base (Archivo complementario 1 - Figura complementaria S6B). Consulte Estructuras óseas para referencia de la alineación.
- Busque la herramienta Pista en el menú desplegable Manipular y mueva el eje al área del pecho haciendo clic y arrastrando el centro del eje. Acérquese al área del pecho haciendo clic en el botón de desplazamiento central del mouse de la computadora y arrastrando el mouse fuera de la pantalla (Archivo Suplementario 1 - Figura Suplementaria S6C).
- Ajuste la alineación para un posicionamiento preciso con las estructuras torácicas como la caja torácica, la columna superior y el esternón mediante ajustes adicionales de traslación y rotación. Cambie entre las vistas 2D y repita los pasos 4.5.3-4.5.4 hasta que las dos imágenes estén alineadas para parecerse a la Figura 3.
- Una vez alineadas las imágenes de TC base y la primera superposición, vuelva a cambiar el filtro de la imagen de TC superpuesta a escala de grises en el menú desplegable LUT. Repita la sección 4 para todas las imágenes de TC superpuestas del mismo animal, utilizando una imagen base constante.
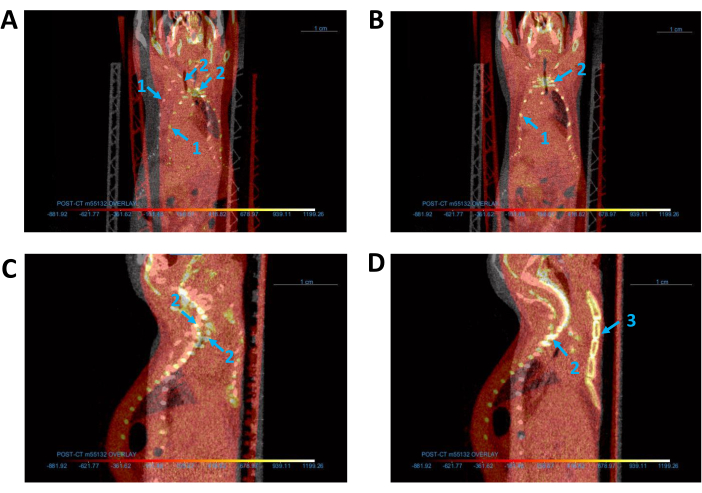
Figura 3: Alineación de todas las imágenes de TC. (A,B) Vista coronal de las imágenes de TC base (gris) y superpuestas (rojo) (A) antes y (B) después de la alineación. (C,D) Vista sagital de las imágenes de TC (C) antes y (D) después de la alineación. Las flechas azules indican (1) la jaula torácica del ratón, (2) la columna vertebral y (3) el esternón. Abreviatura: TC = tomografía computarizada. Haga clic aquí para ver una versión más grande de esta figura.
5. Registrar conjuntamente las imágenes PET con las imágenes CT correspondientes
NOTA: La alineación de la imagen de TC superpuesta y la imagen de TC base inicialmente resultará en una desalineación de la imagen de TC superpuesta con su respectiva imagen PET que se ve en la Figura 4. La imagen PET debe registrarse conjuntamente con su imagen CT correspondiente una vez más.
- Active la imagen CT superpuesta y su respectiva imagen PET haciendo clic en el icono del ojo a la izquierda del nombre de cada imagen.
- Cambie la opacidad de las imágenes PET y CT a ~50%. En el menú desplegable Nivelación de ventana , busque el menú desplegable Mapeo de opacidad y deslice la escala de opacidad hacia el centro (Archivo complementario 1 - Figura complementaria S5).
- Utilice las herramientas Trasladar/Rotar en vistas 2D para alinear la imagen PET con su imagen CT correspondiente, en función de la estructura ósea de la imagen CT y la superficie ósea de 18F-NaF en la imagen PET.
- Seleccione la imagen PET correspondiente a la imagen CT superpuesta en el menú desplegable Propiedades y configuración de datos y haga clic una vez para seleccionar una de las vistas 2D.
- Busque el menú desplegable Traducir/Rotar en la pestaña Principal y haga clic en el icono Desplazar (Archivo complementario 1 - Figura complementaria S6A).
- Haga clic y arrastre el cuadro de traducción en la esquina inferior izquierda y gire el punto de pivote en el centro de la vista 2D seleccionada (Archivo complementario 1 - Figura complementaria S6B) para mover la imagen PET para que coincida con la imagen CT.
- Repita la sección 5 para todas las imágenes de PET y CT superpuestas que no estén registradas conjuntamente con sus imágenes de TC.
NOTA: Si se completa el registro conjunto, las imágenes de PET/CT correspondientes deben alinearse a lo largo de toda la longitud del cuerpo, como se muestra en la Figura 4.
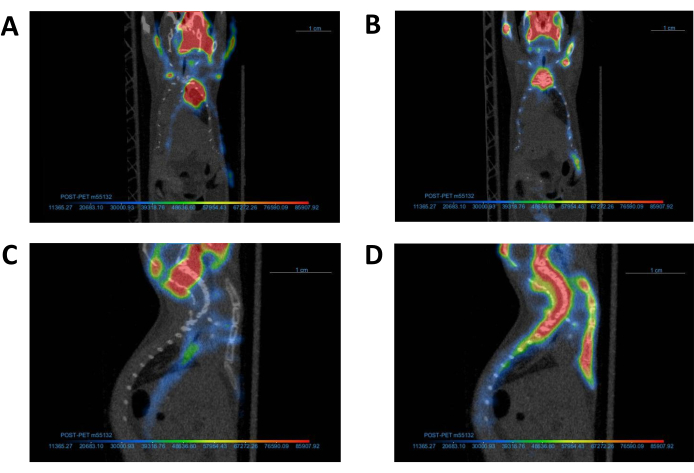
Figura 4: Alineación de las imágenes PET con la imagen CT correspondiente. Una imagen PET representativa y su correspondiente imagen CT (A, C) antes y (B, D) después del co-registro. Una vez finalizado el corregistro, las imágenes de PET y TC deben alinearse como se muestra en el Panel B. Abreviaturas: TC = tomografía computarizada; PET = tomografía por emisión de positrones. Haga clic aquí para ver una versión más grande de esta figura.
6. Identificar la calcificación cardiovascular
- Identifique el punto de tiempo con las regiones calcificadas predichas más grandes en el estudio longitudinal (es decir, punto de tiempo de calcificación tardía o sujetos pretratados). Seleccione la imagen correspondiente a este punto de tiempo para que sirva como imagen de "referencia", enfatizando la calcificación más claramente visible. Esta imagen elegida funcionará como plantilla para la comparación con todas las demás imágenes que contengan áreas de calcificación más pequeñas en diferentes puntos de tiempo.
- Encienda la imagen de TC de referencia haciendo clic en el icono del ojo a la izquierda de la imagen. Seleccione la herramienta Seguimiento en el menú desplegable Manipular y mueva el centro del eje alrededor de la región cardíaca.
- Acérquese para encontrar las regiones calcificadas (sitios pequeños y densos brillantes) superpuestas sobre la silueta cardíaca entre la caja torácica, el esternón y la columna vertebral (Figura 5). Si las regiones calcificadas no son visibles inmediatamente, desplácese por las capas de cada imagen seleccionando una vista 2D y desplazándose con el ratón.
- Mueva el eje Seguimiento para desplazarse sobre la región calcificada, lo que garantiza su visibilidad en todas las vistas 2D. Para obtener una referencia visual de las regiones de calcificación en una imagen de TC de ratón, consulte la Figura 5.
- Encienda la imagen PET correspondiente para validar la presencia de calcificación.
NOTA: Si la configuración de la imagen PET y el registro conjunto se ajustaron adecuadamente, la imagen PET debería mostrar actividad alrededor de la región calcificada, como se ejemplifica en la Figura 5D.
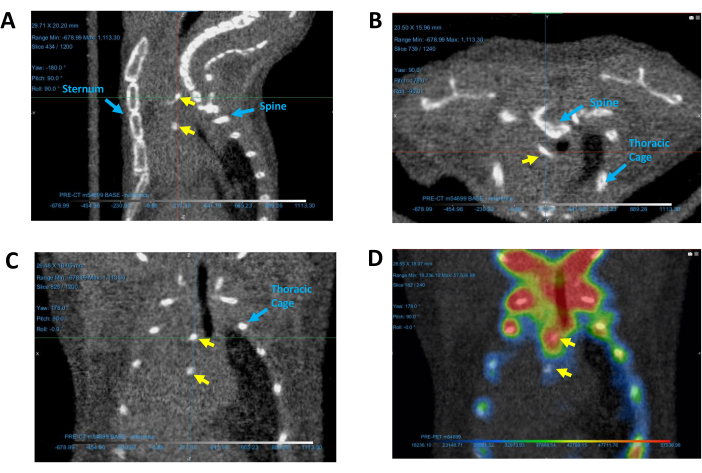
Figura 5: Identificación de regiones calcificadas en el corazón. Regiones de calcificación representadas en imágenes representativas de TC (A) sagital, (B) transversal y (C) coronal, así como (D) una imagen PET/CT coronal. Las flechas amarillas indican depósitos de calcio. Las flechas azules identifican las estructuras de referencia utilizadas para determinar la ubicación de la calcificación. Abreviaturas: TC = tomografía computarizada; PET = tomografía por emisión de positrones. Haga clic aquí para ver una versión más grande de esta figura.
7. Dibuje ROI alrededor de las regiones calcificadas
- Desactive la imagen de referencia PET. Encienda solo la imagen de referencia de TC haciendo clic en el icono del ojo a la izquierda de la imagen.
- Busque el menú desplegable Formas y elija la forma Esfera (Archivo complementario 1 - Figura complementaria S7). Ajusta las dimensiones de la esfera haciendo clic y arrastrando sus bordes para modificar el tamaño. Vuelva a colocar la esfera haciendo clic y arrastrando el cuadrado central.
- Desplácese por las vistas 2D para colocar cuidadosamente la esfera. La esfera debe encapsular toda la región de calcificación identificada y alguna área circundante, pero evitar los huesos circundantes. Para obtener una guía visual sobre el dibujo óptimo de la esfera, consulte la Figura Suplementaria S7 (Archivo Suplementario 1) para la ubicación adecuada de la esfera.
- En el menú desplegable Propiedades y configuración de datos , haga clic con el botón derecho en el nombre de la esfera y elija Agregar al ROI... (Archivo complementario 1 - Figura complementaria S8A), seguido de la selección de Nuevo ROI (Archivo complementario 1 - Figura complementaria S8B).
- Asigne un nombre adecuado al ROI recién creado. En Geometría, seleccione la imagen CT para la que se va a generar un ROI (Archivo complementario 1 - Figura complementaria S8C).
- Active el ROI haciendo clic en el icono del ojo a la izquierda del nombre del ROI y observe un ROI coloreado correspondiente al tamaño de la esfera dibujada anteriormente, como se ilustra en la Figura Suplementaria S8D (Archivo Suplementario 1).
- Busque la pestaña Segmento a la izquierda de la pantalla, junto a la pestaña principal (Figura 6A). La pestaña Segmento permite la creación y edición de ROI.
NOTA: Los siguientes tres pasos están orientados a la creación de un ROI con una precisión que resalta exclusivamente las áreas calcificadas dentro de la esfera designada.- Seleccione el ROI de referencia en las Propiedades y configuración de datos y busque el menú desplegable Rango en la pestaña Segmento . Haga clic para marcar Definir rango (Figura 6A).
- Cambie el rango definido utilizando el cuadro Rango seleccionado (Figura 6A). Asegúrese de que el rango definido encapsule todos los píxeles no calcificados con valores de unidad de Hounsfield (HU) por debajo del umbral de calcio. Esta demostración utiliza un umbral de 300 HU para el calcio, y el píxel de menor densidad en esta imagen es -2.626; por lo tanto, el rango seleccionado es -2,626-300 HU.
- Una vez configurado el rango seleccionado, haga clic en Eliminar (Figura 6A) para eliminar todos los píxeles por debajo del umbral especificado del ROI de la esfera.
NOTA: Esta acción da como resultado un nuevo ROI que resalta exclusivamente las regiones calcificadas dentro de la esfera, lo que simplifica el enfoque para un análisis posterior. El ROI completado debe parecerse al ROI representado en la Figura 6B.
- Repita los pasos 7.3-7.4 para todas las imágenes de TC consecutivas para crear nuevos ROI únicos utilizando una esfera de referencia común. Nombra cada ROI para que se corresponda con la identificación de su imagen de TC específica, asegurando un análisis conciso y organizado.
NOTA: NO debería ser necesario crear una nueva esfera para el análisis de ROI de cada imagen porque la esfera de referencia debe encapsular la región calcificada más grande de todas las imágenes, suponiendo que la alineación sea precisa.
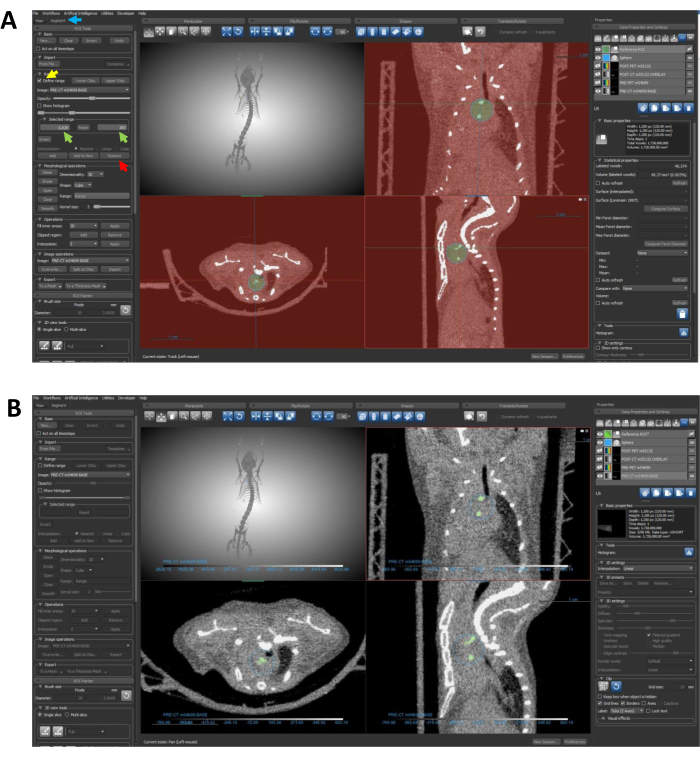
Figura 6: Uso de la segmentación para restringir el ROI a regiones calcificadas. (A) Las flechas guían a través de los pasos necesarios para eliminar los datos de densidad no deseados del ROI. La flecha azul apunta a la pestaña Segmento ; la flecha amarilla resalta la función Definir rango ; las flechas verdes indican las entradas de rango seleccionadas; y la flecha roja apunta al botón Eliminar . (B) Después de completar los pasos del protocolo 7.4-7.4.3, el ROI debe delinear específicamente las regiones calcificadas, como se muestra en este panel. Abreviatura: ROI = región de interés. Haga clic aquí para ver una versión más grande de esta figura.
8. Cuantifica el ROI de cada imagen
- Seleccione el ROI que se cuantificará en el menú desplegable Propiedades y configuración de datos .
- Busque el menú desplegable Propiedades estadísticas en el lado derecho, debajo del menú desplegable Propiedades básicas (Figura 7).
- Busque la tabla Conjunto de datos en Propiedades estadísticas. Usando el menú desplegable de la derecha, elija el conjunto de datos de imágenes apropiado que se correlacione con el ROI que se va a cuantificar (Figura 7).
- Al cuantificar los datos de TC dentro del ROI, elija el nombre de la imagen de TC que se correlacione con el ROI.
- Al cuantificar los datos de PET dentro del ROI, elija el nombre de la imagen de PET que se corresponda con el ROI.
- Haga clic en Actualizar para obtener valores cuantificados de la región calcificada seleccionada. Anote los valores mínimo, máximo y medio según la tabla del conjunto de datos. Utilice los valores medios para la cuantificación (Figura 7).
- Localice el cálculo de volumen en la parte superior del cuadro Propiedades estadísticas . Utilice este volumen para la cuantificación (Figura 7).
- En el caso de los conjuntos de datos de TC, los valores se indican en HU. Calcular el contenido volumétrico de calcio (vHU) utilizando el producto del valor medio del ROI (HU) y el volumen del ROI (mm3)12.
- En el caso de los conjuntos de datos PET, los valores se indican en Becquerels (Bq), que se corresponden con las decaimientos/segundo. Convertir a Bq/mm3 dividiendo el valor medio del ROI (Bq) por el volumen del ROI (mm3), para la concentración de absorción del trazador.

Figura 7: Cuantificación del retorno de la inversión. La pestaña Propiedades estadísticas de un ROI seleccionado. Las flechas rojas ilustran los pasos necesarios para obtener información estadística de un conjunto de datos basada en el ROI. Los recuadros rojos identifican el volumen y el valor unitario medio de Hounsfield, esencial para los cálculos posteriores del análisis. Abreviatura: ROI = región de interés. Haga clic aquí para ver una versión más grande de esta figura.
Resultados
Métodos de análisis
Esta sección ilustra la utilización exitosa a través de resultados representativos. Aquí, mostramos la imagen combinada de microPET / microCT de un solo ratón escaneado a los 15 y 18 meses de edad, después de haber sido sometido a una dieta occidental (21% de grasa, 0,2% de colesterol) de 12 a 14 meses de edad. Siguiendo las secciones 2-8 del protocolo para la cuantificación de la calcificación, cuatro investigadores independientes midier...
Discusión
Este novedoso protocolo es un enfoque mejorado para la cuantificación de la calcificación cardiovascular. Debido a la naturaleza no invasiva de las imágenes, se pueden adquirir imágenes de microTC longitudinales para seguir la progresión de la calcificación cardiovascular en animales pequeños. Aunque las imágenes de microTC por sí solas pueden mostrar la progresión del contenido de calcio, las imágenes de microPET, cuando están disponibles, pueden proporcionar niveles adicion...
Divulgaciones
Los autores no tienen conflictos de intereses que declarar.
Agradecimientos
Agradecemos a todos los miembros del Centro de Tecnología de Imágenes Preclínicas Crump de UCLA por su ayuda con la adquisición y procesamiento de datos, así como con el mantenimiento de equipos e infraestructura. Agradecemos a Jeffrey Collins por su ayuda con la operación del ciclotrón y la síntesis de 18F-NaF. Agradecemos al Grupo de Consultoría Estadística de UCLA por su ayuda con el análisis estadístico. Este trabajo cuenta con el apoyo de la Subvención de Apoyo para Centros Oncológicos de los NIH (2 P30 CA016042-44 a MT) y los Institutos Nacionales de Salud, el Instituto del Corazón, los Pulmones y la Sangre (HL137647 y HL151391 a YT y LLD). El escáner GNEXT PET/CT fue financiado por la subvención NIH S10 Shared Instrumentation for Animal Research (1S10OD026917-01A1 a Arion Chatziioannou).
Materiales
| Name | Company | Catalog Number | Comments |
| 0.5 cc Sterile Insulin Syringes | Exel International | #26028 | Used for IV injection of 18F-NaF PET Tracer |
| 18F-NaF PET Tracer | CNSI Cyclotron | ||
| Biorender | Biorender | Used for figure 1 | |
| Female Apoe-/- mouse | Jackson Laboratories | #002052 | B6.129P2-Apoetm1Unc/J |
| GNEXT PET/CT | Sofie Biosciences, Dulles, Virginia | ||
| Isoflurane | Piramal Critical Care | Used as anesthesia for mouse imaging | |
| ORS Dragonfly | Comet Technologies Canada Inc. | ||
| SPSS Statistics | IBM | ||
| Western diet for mice | Envigo | #TD88139 | 21% fat, 0.2% cholesterol |
Referencias
- Rennenberg, R. J. M. W., et al. Vascular calcifications as a marker of increased cardiovascular risk: A meta-analysis. Vasc Health Risk Man. 5, 185-197 (2009).
- Budoff, M. J., et al. Long-term prognosis associated with coronary calcification - observations from a registry of 25,253 patients. J Am Coll Cardiol. 49 (18), 1860-1870 (2007).
- Polonsky, T. S., et al. Coronary artery calcium score and risk classification for coronary heart disease prediction. JAMA. 303 (16), 1610-1616 (2010).
- Gepner, A. D., et al. Comparison of coronary artery calcium presence, carotid plaque presence, and carotid intima-media thickness for cardiovascular disease prediction in the multi-ethnic study of atherosclerosis. Circ Cardiovasc Imaging. 8 (1), e002262 (2015).
- Pillai, I. C. L., et al. Cardiac fibroblasts adopt osteogenic fates and can be targeted to attenuate pathological heart calcification. Cell Stem Cell. 20 (2), 218-232.e5 (2017).
- Mori, H., et al. Coronary artery calcification and its progression: What does it really mean. JACC Cardiovasc Imaging. 11 (1), 127-142 (2018).
- Mohan, J., Bhatti, K., Tawney, A., Zeltser, R. . StatPearls [Internet]. , (2023).
- Wang, Y., Osborne, M. T., Tung, B., Li, M., Li, Y. Imaging cardiovascular calcification. J Am Heart Assoc. 7 (13), e008564 (2018).
- Fletcher, A. J., et al. Quantifying microcalcification activity in the thoracic aorta. J Nucl Cardiol. 29 (3), 1372-1385 (2022).
- Derlin, T., et al. Feasibility of 18F-sodium fluoride PET/CT for imaging of atherosclerotic plaque. J Nucl Med. 51 (6), 862-865 (2010).
- Hsu, J. J., et al. Changes in microarchitecture of atherosclerotic calcification assessed by 18F-NaF PET and CT after a progressive exercise regimen in hyperlipidemic mice. J Nucl Cardiol. 28 (5), 2207-2214 (2021).
- Hsu, J. J., et al. Effects of teriparatide on morphology of aortic calcification in aged hyperlipidemic mice. Am J Physiol Heart Circ Physiol. 314 (6), H1203-H1213 (2018).
- Irkle, A., et al. Identifying active vascular microcalcification by 18F-sodium fluoride positron emission tomography. Nat Commun. 6, 7495 (2015).
- Syed, M. B. J., Doris, M., Dweck, M., Forsythe, R., Newby, D. E. Chapter 9 - Imaging vascular calcification: Where are we headed. Coronary Calcium: A Comprehensive Understanding of Its Biology, Use in Screening, and Interventional Management. , (2019).
- Joshi, N. V., et al. 18F-fluoride positron emission tomography for identification of ruptured and high-risk coronary atherosclerotic plaques: A prospective clinical trial. Lancet. 383 (9918), 705-713 (2014).
- Fiz, F., et al. 18F-NaF uptake by atherosclerotic plaque on PET/CT imaging: Inverse correlation between calcification density and mineral metabolic activity. J Nucl Med. 56 (7), 1019-1023 (2015).
- Kruithof, B. P., et al. An in vivo map of bone morphogenetic protein 2 post-transcriptional repression in the heart. Genesis. 49 (11), 841-850 (2011).
- Sheen, C. R., et al. Pathophysiological role of vascular smooth muscle alkaline phosphatase in medial artery calcification. J Bone Miner Res. 30 (5), 824-836 (2015).
- Tsai, M. T., Chen, Y. Y., Chang, W. J., Li, S. Y. Warfarin accelerated vascular calcification and worsened cardiac dysfunction in remnant kidney mice. J Chin Med Assoc. 81 (4), 324-330 (2018).
- Wait, J. M., et al. Detection of aortic arch calcification in apolipoprotein e-null mice using carbon nanotube-based micro-ct system. J Am Heart Assoc. 2 (1), e003358 (2013).
- White, D. J., et al. 19F MAS-NMR and solution chemical characterization of the reactions of fluoride with hydroxyapatite and powdered enamel. Acta Odontol Scand. 46 (6), 375-389 (1988).
- Czernin, J., Satyamurthy, N., Schiepers, C. Molecular mechanisms of bone 18F-NaF deposition. J Nucl Med. 51 (12), 1826-1829 (2010).
- Chen, N. X., O'neill, K. D., Dominguez, J. M., Moe, S. M. Regulation of reactive oxygen species in the pathogenesis of matrix vesicles induced calcification of recipient vascular smooth muscle cells. Vasc Med. 26 (6), 585-594 (2021).
- Klose, A. D., Paragas, N. Automated quantification of bioluminescence images. Nat Commun. 9 (1), 4262 (2018).
- Chen-Mayer, H. H., et al. Standardizing ct lung density measure across scanner manufacturers. Med Phys. 44 (3), 974-985 (2017).
- Coxson, H. O. Sources of variation in quantitative computed tomography of the lung. J Thorac Imaging. 28 (5), 272-279 (2013).
- Rogasch, J. M. M., et al. Influences on pet quantification and interpretation. Diagnostics (Basel). 12 (2), 451 (2022).
- Liljequist, D., Elfving, B., Skavberg Roaldsen, K. Intraclass correlation - a discussion and demonstration of basic features. PLoS One. 14 (7), e0219854 (2019).
- Koo, T. K., Li, M. Y. A guideline of selecting and reporting intraclass correlation coefficients for reliability research. J Chiropr Med. 15 (2), 155-163 (2016).
- Moss, A. J., et al. Molecular coronary plaque imaging using (18)f-fluoride. Circ Cardiovasc Imaging. 12 (18), e008574 (2019).
- Moss, A., et al. Coronary atherosclerotic plaque activity and future coronary events. JAMA Cardiol. 8 (8), 755-764 (2023).
- Badea, C., Hedlund, L. W., Johnson, G. A. Micro-ct with respiratory and cardiac gating. Med Phys. 31 (12), 3324-3329 (2004).
- Yang, Y., Rendig, S., Siegel, S., Newport, D. F., Cherry, S. R. Cardiac pet imaging in mice with simultaneous cardiac and respiratory gating. Phys Med Biol. 50 (13), 2979-2989 (2005).
- Liao, W., Deserno, T. M., Spitzer, K. Evaluation of free non-diagnostic dicom software tools. Proc Spie. 6919, (2008).
- Haak, D., Page, C. E., Deserno, T. M. A survey of dicom viewer software to integrate clinical research and medical imaging. J Digit Imaging. 29 (2), 206-215 (2016).
- Aiello, M., et al. How does dicom support big data management? Investigating its use in medical imaging community. Insights Imaging. 12 (1), 164 (2021).
- Kristanto, W., Van Ooijen, P. M., Groen, J. M., Vliegenthart, R., Oudkerk, M. Small calcified coronary atherosclerotic plaque simulation model: Minimal size and attenuation detectable by 64-MDCT and microCT. Int J Cardiovasc Imaging. 28 (4), 843-853 (2012).
- Dehmeshki, J., et al. Volumetric quantification of atherosclerotic plaque in ct considering partial volume effect. IEEE Trans Med Imaging. 26 (3), 273-282 (2007).
- Demer, L. L., Tintut, Y., Nguyen, K. L., Hsiai, T., Lee, J. T. Rigor and reproducibility in analysis of vascular calcification. Circ Res. 120 (8), 1240-1242 (2017).
- Alluri, K., et al. Scoring of coronary artery calcium scans: History, assumptions, current limitations, and future directions. Atherosclerosis. 239 (1), 109-117 (2015).
- Zhang, L., et al. Advances in CT techniques in vascular calcification. Front Cardiovasc Med. 8, 716822 (2021).
- Achenbach, S., Raggi, P. Imaging of coronary atherosclerosis by computed tomography. Eur Heart J. 31 (12), 1442-1448 (2010).
- Agatston, A. S., et al. Quantification of coronary artery calcium using ultrafast computed tomography. J Am Coll Cardiol. 15 (4), 827-832 (1990).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados