É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Novo protocolo de quantificação para progressão da calcificação cardiovascular usando imagens longitudinais de MicroPET/MicroCT
Neste Artigo
Resumo
Este novo protocolo envolve a quantificação da progressão da calcificação cardiovascular a partir de imagens seriadas de microtomografia por emissão de pósitrons (PET)/microtomografia computadorizada (TC) em pequenos animais de pesquisa.
Resumo
A microtomografia por emissão de pósitrons (PET) e a microtomografia computadorizada (TC) são ferramentas de pesquisa poderosas e ideais para acompanhar a progressão da calcificação cardiovascular. Devido à sua natureza não invasiva, pequenos animais de pesquisa podem ser fotografados em vários momentos. O desafio está na quantificação precisa da calcificação cardiovascular. Aqui, fornecemos um protocolo, usando imagens dos estágios posteriores da doença como modelo, para quantificar com precisão a progressão da calcificação cardiovascular em estudos longitudinais. O protocolo envolve 1) o alinhamento da área do tórax em várias imagens do mesmo animal durante um estudo longitudinal como a primeira etapa, 2) a definição de uma região de interesse (ROI) situada dentro do coração e da aorta no local de depósitos maiores de cálcio que se tornam aparentes em imagens posteriores, e 3) segmentação e quantificação simultâneas de depósitos de cálcio em todas as imagens adquiridas durante o estudo longitudinal. Esse método simplificado aumenta a precisão da análise de imagens no acompanhamento da progressão da calcificação cardiovascular, melhorando a precisão da definição do ROI e reduzindo a variabilidade associada a técnicas anteriores que analisam exames individuais de forma independente.
Introdução
A doença cardiovascular é uma das principais causas globais de morbidade e mortalidade, exigindo uma exploração rigorosa para descobrir seus mecanismos e elaborar estratégias preventivas e terapêuticas eficazes. A calcificação da artéria coronária (CAC) é amplamente reconhecida pelos especialistas da área como um fator preditivo para doença cardiovascular, elevando significativamente o risco de mortalidade cardiovascular 1,2,3,4,5. As calcificações microscópicas são consideradas os estágios iniciais da aterosclerose calcificada, e o termo "microcalcificações" tem sido usado para se referir a depósitos de cálcio entre 0,5 a 50 μm 6,7,8,9 de diâmetro. Acredita-se que essas pequenas calcificações coalescem para formar depósitos maiores de cálcio, alimentando a progressão da placa calcificada 6,7.
A tomografia por emissão de pósitrons (PET) e a tomografia computadorizada (TC) são ferramentas valiosas de pesquisa, frequentemente empregadas para a avaliação não invasiva da calcificação cardiovascular in vivo 5,10,11,12,13,14,15,16,17,18,19 . Essas modalidades de imagem mostram-se particularmente vantajosas para rastrear a progressão da calcificação vascular em estudos longitudinais envolvendo pequenos animais de pesquisa 11,12,13,19. A microTC demonstrou eficácia no fornecimento de imagens anatômicas de depósitos de cálcio relativamente grandes 11,12,13,19,20.No entanto, sua utilidade para imagens de pequenos depósitos de cálcio em animais vivos é limitada por sua resolução espacial de ~ 100 μm 8,14, tornando difícil investigar a calcificação durante suas fases iniciais.
Um avanço notável é a adoção de imagens combinadas de microPET/microCT com o traçador PET, fluoreto de sódio marcado com flúor 18 (18F-NaF), como o método padrão para a detecção de calcificação com base em sua ligação à área de superfície mineral. Essa abordagem usa F-NaF radiomarcado 18, mostrado para identificar a superfície mineral de cálcio10,13 à medida que os íons fluoreto se ligam covalentemente à hidroxiapatita de cálcio, substituindo grupos hidroxila para formar fluoroapatita21. Consistente com a taxa de troca mais lenta em relação ao decaimento radioativo do 18F (meia-vida ~ 110 min) e à depuração do traçador através dos rins22, Irkle e colegas13 descobriram que a ligação de 18F-NaF a amostras de carótidas calcificadas estava limitada à superfície no momento da detecção. Assim, a absorção do traçador deve estar diretamente relacionada com a área de superfície mineral, que é maior quando uma determinada quantidade de mineral ocorre em vários pequenos focos ou na forma porosa do que quando presente em poucos depósitos sólidos grandes. Ao acentuar os estágios iniciais da mineralização com alta sensibilidade, 18a imagem F-NaF PET pode fornecer informações valiosas sobre os estágios iniciais da doença, tornando-a particularmente útil no estudo de estratégias preventivas e terapêuticas 13,14,15.
Mesmo com os avanços recentes na combinação de imagens microPET/microCT de calcificação vascular, há oportunidades para melhorar a precisão da análise de imagens em estudos longitudinais de calcificação cardiovascular. As abordagens convencionais usam delineamento manual e trabalhoso de regiões de interesse (ROI) em torno de cada região calcificada visível em cada camundongo em cada ponto de tempo individual ao longo do estudo longitudinal. Este método reduz a precisão, particularmente nos estágios iniciais da doença, quando os tamanhos dos depósitos de cálcio se aproximam dos limites de detecção dos scanners, potencialmente tornando invisíveis algumas áreas com depósitos minúsculos dispostos em baixa densidade.
No campo da imagem, o alinhamento geralmente se refere ao alinhamento espacial de uma série de imagens. Introduzindo o alinhamento como uma nova solução para os desafios existentes, nosso método proposto permite que os investigadores usem um local consistente para acompanhar a progressão da calcificação em imagens seriadas de indivíduos individuais ao longo de um estudo longitudinal. Dado que a calcificação tecidual é conhecida por surgir de vesículas de matriz nanométricas (50-150 nm), que coalescem para formar o mineral hidroxiapatita microscópico e macroscópico23, pode-se identificar retrospectivamente onde as microcalcificações teriam sido localizadas nas primeiras imagens antes de serem discerníveis.
Seguir esse mesmo local ao longo do tempo, possibilitado pelo alinhamento da imagem, é a base para esse método. Isso evita a necessidade de identificar diretamente os primeiros estágios de calcificação, pois o ROI é atribuído com base nos estágios mais recentes, quando a mineralização é prontamente identificada. Neste protocolo, apresentamos um método de análise de dados aprimorado e simplificado que incorpora o alinhamento de uma série temporal de imagens como uma etapa vital, aprimorando a quantificação precisa da deposição de cálcio em estudos de imagem longitudinal, combinados, microPET/microCT (Figura 1). Embora usemos a análise de dados PET/CT como exemplo, esse método pode ser aplicado às análises de outros dados de imagem longitudinal, incluindo tomografia computadorizada por emissão de fóton único (SPECT), ressonância magnética (MRI) e imagem óptica24.
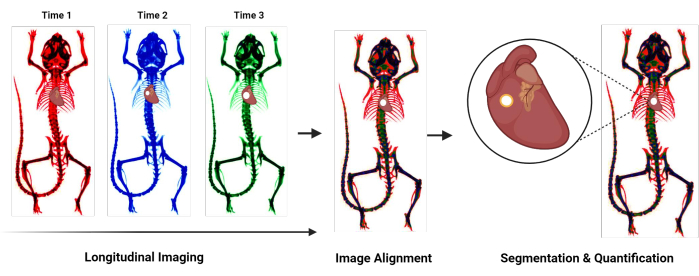
Figura 1: Fluxograma de visão geral do protocolo. Fluxograma resumindo o novo protocolo para quantificar a calcificação cardiovascular. As etapas gerais incluem imagens longitudinais, alinhamento de imagens adquiridas em diferentes momentos e segmentação e quantificação da região calcificada. Clique aqui para ver uma versão maior desta figura.
Protocolo
As imagens representativas apresentam uma fêmea de camundongo com deficiência de apolipoproteína-E (Apoe-/-). Os protocolos experimentais foram revisados e aprovados pelo Comitê Institucional de Cuidados e Uso de Animais da Universidade da Califórnia, Los Angeles.
1. Escaneamento de animais
- Entre as duas aquisições de imagem, alimente o camundongo com uma dieta padrão sem qualquer intervenção. No entanto, antes da primeira imagem aos 15 meses de idade, mude para uma dieta ocidental (21% de gordura, 0.2% de colesterol) dos 12 aos 14 meses de idade para induzir a calcificação aórtica basal.
- Certifique-se de que o scanner microPET/CT seja calibrado rotineiramente para aquisição ideal de dados PET e co-registro de TC. Realize as calibrações de acordo com as instruções do fabricante.
- Anestesiar o camundongo usando gás isoflurano a 2% em uma câmara por 10 min antes da imagem. Confirme a anestesia beliscando o dedo do pé do animal sem resposta.
- Adquira imagens microPET (350-650 keV, tempo de varredura de 10 min) e microCT de 100 μm (80 kVp, 150 μA, projeção de 720, tempo de varredura de 1 min) em um scanner combinado de microPET/CT 60 min após a injeção intravenosa de 3,7 MBq de 18F-NaF. Adquira as varreduras microCT imediatamente após as varreduras microPET.
NOTA: O leito do scanner moveu o animal do modal PET para o modal CT enquanto o animal permanecia sob anestesia e na mesma posição. - Reconstrua imagens microPET usando um algoritmo de maximização de expectativa de subconjunto ordenado em 3D (24 subconjuntos e 3 iterações), com correção aleatória, atenuação e decaimento. Reconstrua as imagens de TC usando um algoritmo de Feldkamp modificado.
NOTA: Após a imagem, o camundongo foi monitorado até recuperar a consciência suficiente para manter a decúbito esternal. Ao final do estudo longitudinal, todos os camundongos foram sacrificados.
2. Importe arquivos DICOM no software visualizador DICOM
NOTA: Embora este protocolo representativo use o software ORS Dragonfly sob uma licença não comercial, sua flexibilidade se estende a outras opções de software visualizador DICOM.
- Inicie o software DICOM viewer clicando duas vezes no aplicativo.
- Navegue até Arquivo no canto superior esquerdo. No menu suspenso, selecione Importar imagens DICOM... para solicitar o aparecimento da janela Gerenciar imagens DICOM (Arquivo Suplementar 1 - Figura Suplementar S1A).
- Na janela Gerenciar imagens DICOM , clique na guia Conteúdo da pasta . No lado direito, selecione Abrir pasta... para escolher as pastas que contêm os arquivos DICOM de interesse em um estudo longitudinal (Arquivo suplementar 1 - Figura suplementar S1B).
- Identifique as pastas que contêm os arquivos DICOM. Clique em Selecionar pasta para importar a primeira pasta para a janela Gerenciar imagens DICOM (Arquivo suplementar 1 - Figura suplementar S1C).
- Selecione os arquivos PET ("PT") e CT ("CT") DICOM na janela Gerenciar imagens DICOM . Em seguida, clique em Exibir estudo no lado direito para abrir todas as imagens DICOM importadas (Arquivo Suplementar 1- Figura Suplementar S1D).
- Repita as etapas 2.4 e 2.5 para cada pasta que contenha imagens DICOM relacionadas ao mesmo assunto no estudo longitudinal.
3. Ajuste as configurações do visualizador DICOM para otimizar a visualização da imagem
- Altere o layout para exibir quatro vistas (3D, transversal, coronal, sagital), conforme ilustrado no Arquivo Suplementar 1 - Figura Suplementar S2. No menu suspenso Layout no lado esquerdo, localize Exibições (na cena selecionada) e selecione um layout que exiba quatro exibições.
- Renomeie as imagens para indicar pontos de tempo e se são imagens PET ou CT. Selecione o nome da imagem e clique duas vezes para renomear cada imagem.
- Ative uma imagem acessando o menu suspenso Propriedades e configurações de dados no lado direito. Clique no ícone de olho à esquerda de cada nome de imagem para alternar a visibilidade (Arquivo Suplementar 1 - Figura Suplementar S2)
- Ajuste o brilho e o contraste em cada imagem de TC, individualmente.
NOTA: O brilho ideal da TC e a faixa de contraste podem variar dependendo do sujeito, protocolo de imagem, scanner e parâmetros de reconstrução25,26; no entanto, todos os ajustes devem ser consistentes para um único sujeito em um estudo longitudinal.- Selecione e ative uma imagem de TC clicando no nome da imagem em Propriedades e configurações de dados (Arquivo suplementar 1 - Figura suplementar S2).
- Encontre o histograma no menu suspenso Nivelamento de janela localizado na guia Principal no lado esquerdo da tela de exibição. Ative LogY acima do histograma; em seguida, clique e deslize os indicadores de faixa amarela para abranger completamente o segundo pico no histograma (Arquivo Suplementar 1 - Figura Suplementar S3A). Faça pequenos ajustes até atingir o brilho/contraste ideal; consulte a Figura 2B para obter um exemplo de uma imagem de TC ajustada para brilho/contraste.
- Localize a caixa Intervalo selecionado no histograma de nivelamento de janela (Arquivo suplementar 1 - Figura suplementar S3B). Anote os valores de Intervalo selecionado a serem usados nas etapas seguintes.
- Selecione e ative uma segunda imagem de TC (Arquivo Suplementar 1 - Figura Suplementar S3C). Insira os valores de intervalo selecionados da primeira imagem de TC na caixa Intervalo selecionado para a segunda imagem de TC (Arquivo Suplementar 1 - Figura Suplementar S3B-S3D). Repita para todas as imagens consecutivas.
- Refine cada imagem PET ajustando o filtro Tabela de Pesquisa, individualmente.
NOTA: A faixa ideal da escala de cores PET pode variar dependendo do assunto, protocolo de imagem, scanner e parâmetros de reconstrução27; no entanto, todos os ajustes devem ser consistentes para um único sujeito em um estudo longitudinal.- Selecione e ative uma imagem PET clicando no nome da imagem em Propriedades e configurações de dados (Arquivo suplementar 1 - Figura suplementar S2).
- Na lista suspensa Nivelamento de janela na guia Principal , localize Tabela de pesquisa (LUT) e role para encontrar PET na lista suspensa LUT e selecione o filtro PET (Arquivo Suplementar 1 - Figura Suplementar S4A).
- Ajuste o intervalo selecionado usando o histograma no menu suspenso Nivelamento de janela para obter uma visualização ideal. Ative LogY acima do histograma; em seguida, clique e deslize os indicadores de faixa amarela em direção à posição mostrada na Figura Suplementar S4B (Arquivo Suplementar 1). Faça pequenos ajustes até que a faixa selecionada ideal seja alcançada para visualização de imagem PET; consulte a Figura 2D para obter um exemplo de uma imagem PET/CT ajustada para a faixa selecionada ideal.
- Localize o menu suspenso Intervalo selecionado no histograma de nivelamento de janela (Arquivo suplementar 1 - Figura suplementar S4B). Anote os valores de intervalo selecionados a serem usados nas etapas seguintes.
- Selecione e ative uma segunda imagem PET (Supplemental File 1 - Figura S4C). Repita a etapa 3.5.2 para a segunda imagem.
- Insira os valores de intervalo selecionados da primeira imagem PET na caixa Intervalo selecionado para a segunda imagem PET (Arquivo suplementar 1 - Figura S4B-S4D). Repita para todas as imagens consecutivas.
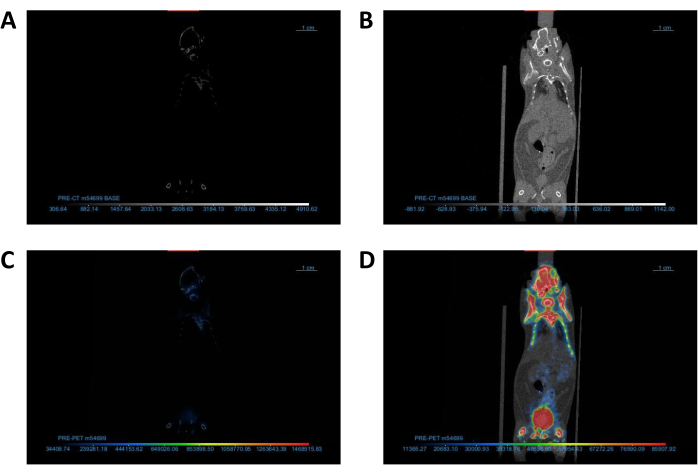
Figura 2: Ajuste das configurações do visualizador DICOM para otimizar a visualização da imagem. (A, B) Visão coronal das imagens de TC (A) antes e (B) após o ajuste de contraste/brilho. (C, D) Vista coronal das imagens PET/CT (C) antes e (D) depois do ajuste da tabela de pesquisa. Abreviaturas: TC = tomografia computadorizada; PET = tomografia por emissão de pósitrons. Clique aqui para ver uma versão maior desta figura.
4. Alinhe a área do tórax nas imagens de TC
NOTA: Para simplificar, uma imagem de TC servirá como imagem "base" e não será transladada ou girada. A segunda imagem de TC (e, eventualmente, imagens seriais subsequentes) será transladada e/ou girada para se alinhar com a imagem base; Isso será chamado de imagem de "sobreposição" para fins de demonstração. Ao longo do alinhamento, é importante distinguir entre a imagem base e todas as outras imagens de sobreposição.
- Desative todas as imagens PET e ative a imagem de TC base clicando no ícone de olho à esquerda de cada nome de imagem no menu suspenso Propriedades e configurações de dados .
- Altere a opacidade da imagem da TC para ~ 50%. Clique em uma das visualizações 2D. Na caixa Nivelamento de janela , localize o menu suspenso Mapeamento de opacidade e deslize a escala de opacidade para o centro (Arquivo suplementar 1 - Figura S5).
- Ligue a imagem de TC de sobreposição e repita a etapa 4.2.
- Altere o filtro da imagem de TC de sobreposição para distinguir entre a imagem de TC de base e a imagem de TC de sobreposição. Na lista suspensa Nivelamento de janela na guia Principal , localize Tabela de pesquisa (LUT). No menu suspenso LUT, role e clique no filtro vermelho .
NOTA: Agora deve haver duas imagens de TC em cada visualização, conforme ilustrado na Figura Suplementar S5 (Arquivo Suplementar 1). - Use as ferramentas Traduzir/Girar em visualizações 2D para alinhar a área do peito na imagem de sobreposição o mais próximo possível da imagem base. Consulte a caixa torácica, a parte superior da coluna e o esterno como indicadores de alinhamento torácico na imagem de TC.
NOTA: As ações a seguir só podem ser executadas em visualizações 2D (Transversal, Coronal ou Sagital) (Supplemental File 1- Supplemental Figure S1). Para obter o melhor resultado, alterne entre as três visualizações 2D e use as ferramentas de translação/rotação para alinhar a área do peito em cada visualização.- Selecione a imagem de sobreposição no menu suspenso Propriedades e configurações de dados e clique uma vez para selecionar uma das exibições 2D.
- Localize o menu suspenso Traduzir/Girar na guia Principal e clique no ícone Deslocar (Arquivo Suplementar 1 - Figura S6A).
- Clique e arraste na caixa de translação no canto inferior esquerdo e gire o ponto de pivô no centro da vista 2D selecionada para mover a imagem de sobreposição para alinhá-la aproximadamente com a imagem base (Arquivo Suplementar 1 - Figura Suplementar S6B). Consulte as estruturas ósseas para referência de alinhamento.
- Encontre a ferramenta Rastrear no menu suspenso Manipular e mova o eixo para a área do peito clicando e arrastando o centro do eixo. Aumente o zoom na área do peito clicando no botão de rolagem do meio no mouse do computador e arrastando o mouse para longe da tela (Arquivo Suplementar 1 - Figura Suplementar S6C).
- Ajuste o alinhamento para um posicionamento preciso com estruturas torácicas, como a caixa torácica, a parte superior da coluna e o esterno, por meio de ajustes adicionais de translação e rotação. Alterne entre as visualizações 2D e repita as etapas 4.5.3 a 4.5.4 até que as duas imagens estejam alinhadas para se parecerem com a Figura 3.
- Depois que as imagens de TC de base e a primeira sobreposição estiverem alinhadas, altere o filtro da imagem de TC de sobreposição de volta para tons de cinza no menu suspenso LUT. Repita a seção 4 para todas as imagens de TC sobrepostas do mesmo animal, usando uma imagem de base constante.
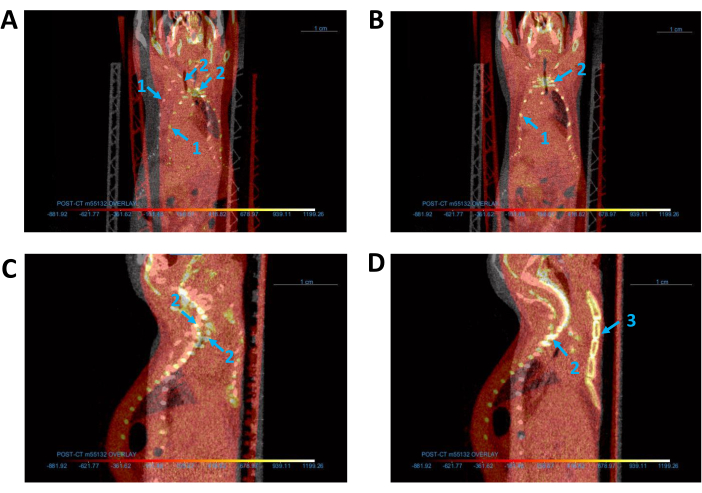
Figura 3: Alinhamento de todas as imagens de TC. (A, B) Visão coronal das imagens de TC de base (cinza) e sobreposição (vermelho) (A) antes e (B) após o alinhamento. (C, D) Visão sagital das imagens de TC (C) antes e (D) após o alinhamento. As setas azuis indicam o camundongo (1) caixa torácica, (2) coluna vertebral e (3) esterno. Abreviatura: TC = tomografia computadorizada. Clique aqui para ver uma versão maior desta figura.
5. Co-registre imagens PET com imagens de TC correspondentes
NOTA: O alinhamento da imagem de TC de sobreposição e da imagem de TC de base resultará inicialmente em desalinhamento da imagem de TC de sobreposição com sua respectiva imagem PET vista na Figura 4. A imagem PET precisa ser co-registrada com sua imagem de TC correspondente mais uma vez.
- Ative a imagem de TC de sobreposição e sua respectiva imagem PET clicando no ícone de olho à esquerda de cada nome de imagem.
- Altere a opacidade das imagens PET e CT para ~50%. No menu suspenso Nivelamento de janela , localize o menu suspenso Mapeamento de opacidade e deslize a escala de opacidade para o centro (Arquivo suplementar 1 - Figura suplementar S5).
- Use as ferramentas Translate/Rotate em visualizações 2D para alinhar a imagem PET com sua imagem de TC correspondente, com base na estrutura óssea na imagem de TC e na superfície óssea de 18F-NaF na imagem PET.
- Selecione a imagem PET correspondente à imagem de TC de sobreposição no menu suspenso Propriedades e configurações de dados e clique uma vez para selecionar uma das visualizações 2D.
- Localize o menu suspenso Traduzir/Girar na guia Principal e clique no ícone Deslocar (Arquivo Suplementar 1 - Figura Suplementar S6A).
- Clique e arraste na caixa de translação no canto inferior esquerdo e gire o ponto de pivô no centro da visualização 2D selecionada (Arquivo Suplementar 1 - Figura Suplementar S6B) para mover a imagem PET para o corregistro com a imagem de TC.
- Repita a seção 5 para todas as imagens PET e CT sobrepostas que não estão co-registradas com suas imagens CT.
NOTA: Se o co-registro for concluído, as imagens PET/CT correspondentes devem ser alinhadas ao longo de todo o comprimento do corpo, conforme mostrado na Figura 4.
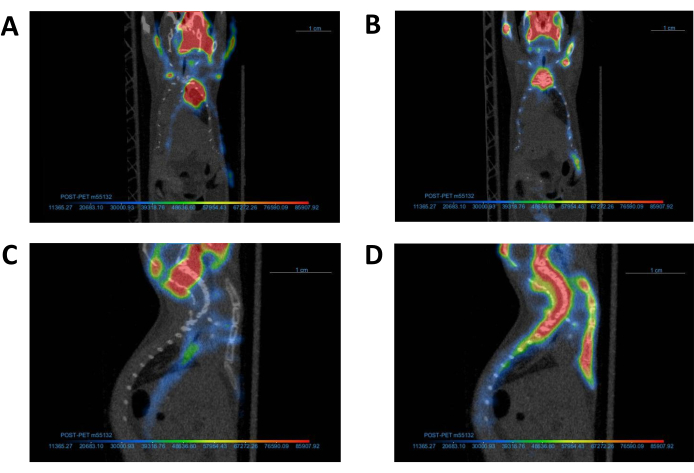
Figura 4: Alinhamento das imagens PET com a imagem de TC correspondente. Uma imagem PET representativa e sua imagem de TC correspondente (A, C) antes e (B, D) após o co-registro. Após a conclusão do co-registro, as imagens PET e CT devem ser alinhadas conforme demonstrado no Painel B. Abreviaturas: TC = tomografia computadorizada; PET = tomografia por emissão de pósitrons. Clique aqui para ver uma versão maior desta figura.
6. Identifique a calcificação cardiovascular
- Identifique o ponto de tempo com as maiores regiões calcificadas previstas no estudo longitudinal (ou seja, ponto de tempo de calcificação tardia ou indivíduos pré-tratados). Selecione a imagem correspondente a este ponto de tempo para servir como imagem de "referência", enfatizando a calcificação mais distintamente visível. Esta imagem escolhida funcionará como um modelo para comparação com todas as outras imagens contendo áreas de calcificação menores em diferentes pontos de tempo.
- Ative a imagem de TC de referência clicando no ícone de olho à esquerda da imagem. Selecione a ferramenta Rastrear no menu suspenso Manipular e mova o centro do eixo ao redor da região cardíaca.
- Aumente o zoom para encontrar as regiões calcificadas (pequenos locais densos e brilhantes) sobrepostas à silhueta cardíaca entre a caixa torácica, o esterno e a coluna vertebral (Figura 5). Se as regiões calcificadas não estiverem imediatamente visíveis, percorra as camadas de cada imagem selecionando uma visualização 2D e rolando com o mouse.
- Mova o eixo Trilha para passar o mouse sobre a região calcificada, garantindo sua visibilidade em todas as vistas 2D. Para uma referência visual das regiões de calcificação em uma imagem de tomografia computadorizada de camundongo, consulte a Figura 5.
- Ligue a imagem PET correspondente para validar a presença de calcificação.
NOTA: Se as configurações da imagem PET e o co-registro foram ajustados adequadamente, a imagem PET deve exibir atividade ao redor da região calcificada, conforme exemplificado na Figura 5D.
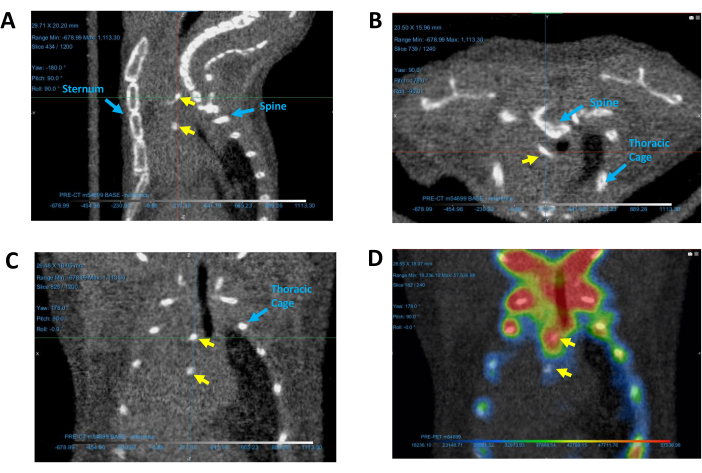
Figura 5: Identificação de regiões calcificadas no coração. Regiões de calcificação representadas em imagens representativas (A) sagitais, (B) transversais e (C) coronais, bem como (D) uma imagem coronal de PET/CT. Setas amarelas indicam depósitos de cálcio. As setas azuis identificam as estruturas de referência usadas para determinar a localização da calcificação. Abreviaturas: TC = tomografia computadorizada; PET = tomografia por emissão de pósitrons. Clique aqui para ver uma versão maior desta figura.
7. Desenhe ROIs em torno das regiões calcificadas
- Desative a imagem de referência PET. Ative apenas a imagem de referência da TC clicando no ícone de olho à esquerda da imagem.
- Localize a lista suspensa Formas e escolha a forma Esfera (Arquivo Suplementar 1 - Figura Suplementar S7). Ajuste as dimensões da esfera clicando e arrastando suas bordas para modificar o tamanho. Reposicione a esfera clicando e arrastando o quadrado central.
- Navegue pelas vistas 2D para posicionar cuidadosamente a esfera. A esfera deve encapsular toda a região de calcificação identificada e alguma área circundante, mas evitar quaisquer ossos circundantes. Para obter um guia visual sobre o desenho ideal da esfera, consulte a Figura Suplementar S7 (Arquivo Suplementar 1) para o posicionamento adequado da esfera.
- No menu suspenso Propriedades e configurações de dados , clique com o botão direito do mouse no nome da esfera e escolha Adicionar ao ROI... (Arquivo Suplementar 1 - Figura Suplementar S8A), seguido pela seleção de Novo ROI (Arquivo Suplementar 1 - Figura Suplementar S8B).
- Atribua um nome apropriado ao ROI recém-criado. Em Geometria, selecione a imagem de TC para a qual um ROI deve ser gerado (Arquivo Suplementar 1 - Figura Suplementar S8C).
- Ative o ROI clicando no ícone de olho à esquerda do nome do ROI e observe um ROI colorido correspondente ao tamanho da esfera desenhada anteriormente, conforme ilustrado na Figura Suplementar S8D (Arquivo Suplementar 1).
- Encontre a guia Segmento à esquerda da tela de exibição, ao lado da guia principal (Figura 6A). A guia Segmento permite a criação e edição de ROIs.
NOTA: As três etapas a seguir são voltadas para a criação de um ROI com uma precisão que destaca exclusivamente as áreas calcificadas dentro da esfera designada.- Selecione o ROI de referência nas Propriedades e configurações de dados e localize o menu suspenso Intervalo na guia Segmento . Clique para verificar Definir intervalo (Figura 6A).
- Altere o intervalo definido usando a caixa Intervalo selecionado (Figura 6A). Certifique-se de que o intervalo definido encapsule todos os pixels não calcificados com valores de unidade Hounsfield (HU) abaixo do limite de cálcio. Esta demonstração usa um limite de 300 HU para cálcio, e o pixel de densidade mais baixa nesta imagem é -2.626; portanto, o intervalo selecionado é -2.626-300 HU.
- Depois que o intervalo selecionado estiver configurado, clique em Remover (Figura 6A) para eliminar todos os pixels abaixo do limite especificado do ROI da esfera.
NOTA: Esta ação resulta em um novo ROI que destaca exclusivamente as regiões calcificadas dentro da esfera, simplificando o foco para análises posteriores. Os ROIs concluídos devem se assemelhar ao ROI representado na Figura 6B.
- Repita as etapas 7.3 a 7.4 para todas as imagens de TC consecutivas para criar novos ROIs exclusivos usando uma esfera de referência comum. Nomeie cada ROI para corresponder à identificação de sua imagem de TC específica, garantindo uma análise concisa e organizada.
NOTA: NÃO deve ser necessário criar uma nova esfera para a análise de ROI de cada imagem, pois a esfera de referência deve encapsular a maior região calcificada de todas as imagens, supondo que o alinhamento seja preciso.
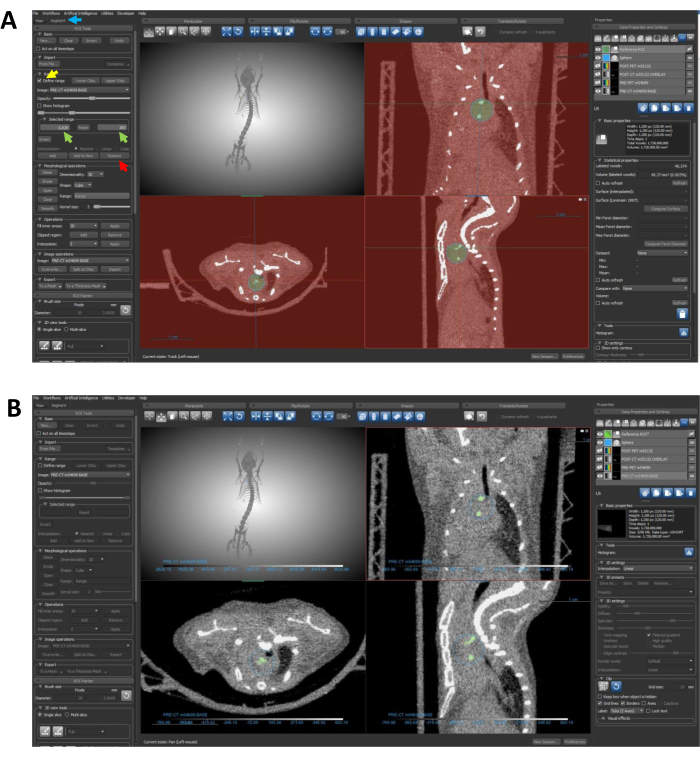
Figura 6: Usando segmentação para restringir o ROI a regiões calcificadas. (A) As setas guiam pelas etapas necessárias para eliminar dados de densidade indesejados do ROI. A seta azul aponta para a guia Segmento ; a seta amarela destaca o recurso Definir intervalo ; setas verdes indicam as entradas de intervalo selecionadas; e a seta vermelha aponta para o botão Remover . (B) Após a conclusão das etapas do protocolo 7.4-7.4.3, o ROI deve delinear especificamente as regiões calcificadas, conforme descrito neste painel. Abreviatura: ROI = região de interesse. Clique aqui para ver uma versão maior desta figura.
8. Quantifique o ROI de cada imagem
- Selecione o ROI a ser quantificado no menu suspenso Propriedades e configurações de dados .
- Localize o menu suspenso Propriedades estatísticas no lado direito abaixo do menu suspenso Propriedades básicas (Figura 7).
- Localize a tabela Conjunto de dados em Propriedades estatísticas. Usando o menu suspenso à direita, escolha o conjunto de dados de imagem apropriado que se correlaciona com o ROI a ser quantificado (Figura 7).
- Ao quantificar os dados de TC dentro do ROI, escolha o nome da imagem de TC que se correlaciona com o ROI.
- Ao quantificar os dados PET dentro do ROI, escolha o nome da imagem PET que corresponde ao ROI.
- Clique em Atualizar para adquirir valores quantificados da região calcificada selecionada. Observe os valores Min, Max e Mean de acordo com a tabela do conjunto de dados. Use os valores médios para quantificação (Figura 7).
- Localize o cálculo de volume na parte superior da caixa Propriedades estatísticas . Use este volume para quantificação (Figura 7).
- Para conjuntos de dados de TC, os valores são relatados em HU. Calcule o teor volumétrico de cálcio (vHU) usando o produto do valor médio da ROI (HU) e do volume da ROI (mm3)12.
- Para conjuntos de dados PET, os valores são relatados em Becquerels (Bq), que correspondem a decaimentos/segundo. Converta em Bq/mm3 dividindo o valor médio da ROI (Bq) pelo volume da ROI (mm3), para a concentração de absorção do marcador.

Figura 7: Quantificação do ROI. A guia Propriedades estatísticas de um ROI selecionado. As setas vermelhas ilustram as etapas necessárias para obter informações estatísticas para um conjunto de dados com base no ROI. As caixas vermelhas identificam o volume e o valor unitário médio de Hounsfield, essencial para cálculos de análise posteriores. Abreviatura: ROI = região de interesse. Clique aqui para ver uma versão maior desta figura.
Resultados
Métodos de análise
Esta seção ilustra a utilização bem-sucedida por meio de resultados representativos. Aqui, mostramos a imagem combinada de microPET / microCT de um único camundongo escaneado aos 15 e 18 meses de idade, após ser submetido a uma dieta ocidental (21% de gordura, 0,2% de colesterol) dos 12 aos 14 meses de idade. Seguindo as seções 2-8 do protocolo para quantificação de calcificação, quatro pesquisadores independentes mediram separadamente...
Discussão
Este novo protocolo é uma abordagem aprimorada para a quantificação da calcificação cardiovascular. Devido à natureza não invasiva da imagem, imagens longitudinais de microTC podem ser adquiridas para acompanhar a progressão da calcificação cardiovascular em pequenos animais. Embora as imagens de microCT sozinhas possam mostrar a progressão do conteúdo de cálcio, as imagens de microPET, quando disponíveis, podem fornecer níveis adicionais de informação, especialmente a d...
Divulgações
Os autores não têm conflitos de interesse a declarar.
Agradecimentos
Agradecemos a todos os membros do Centro de Tecnologia de Imagens Pré-clínicas Crump da UCLA por sua ajuda na aquisição e procissão de dados, bem como na manutenção de equipamentos e infraestrutura. Agradecemos a Jeffrey Collins por sua ajuda na operação do ciclotron e na síntese de 18F-NaF. Agradecemos ao Grupo de Consultoria Estatística da UCLA por sua ajuda com a análise estatística. Este trabalho é apoiado pelo NIH Cancer Center Support Grant (2 P30 CA016042-44 para MT) e National Institutes of Health, Heart, Lung and Blood Institute (HL137647 e HL151391 para YT e LLD). O scanner GNEXT PET/CT foi financiado pela Bolsa de Instrumentação Compartilhada para Pesquisa Animal NIH S10 (1S10OD026917-01A1 para Arion Chatziioannou).
Materiais
| Name | Company | Catalog Number | Comments |
| 0.5 cc Sterile Insulin Syringes | Exel International | #26028 | Used for IV injection of 18F-NaF PET Tracer |
| 18F-NaF PET Tracer | CNSI Cyclotron | ||
| Biorender | Biorender | Used for figure 1 | |
| Female Apoe-/- mouse | Jackson Laboratories | #002052 | B6.129P2-Apoetm1Unc/J |
| GNEXT PET/CT | Sofie Biosciences, Dulles, Virginia | ||
| Isoflurane | Piramal Critical Care | Used as anesthesia for mouse imaging | |
| ORS Dragonfly | Comet Technologies Canada Inc. | ||
| SPSS Statistics | IBM | ||
| Western diet for mice | Envigo | #TD88139 | 21% fat, 0.2% cholesterol |
Referências
- Rennenberg, R. J. M. W., et al. Vascular calcifications as a marker of increased cardiovascular risk: A meta-analysis. Vasc Health Risk Man. 5, 185-197 (2009).
- Budoff, M. J., et al. Long-term prognosis associated with coronary calcification - observations from a registry of 25,253 patients. J Am Coll Cardiol. 49 (18), 1860-1870 (2007).
- Polonsky, T. S., et al. Coronary artery calcium score and risk classification for coronary heart disease prediction. JAMA. 303 (16), 1610-1616 (2010).
- Gepner, A. D., et al. Comparison of coronary artery calcium presence, carotid plaque presence, and carotid intima-media thickness for cardiovascular disease prediction in the multi-ethnic study of atherosclerosis. Circ Cardiovasc Imaging. 8 (1), e002262 (2015).
- Pillai, I. C. L., et al. Cardiac fibroblasts adopt osteogenic fates and can be targeted to attenuate pathological heart calcification. Cell Stem Cell. 20 (2), 218-232.e5 (2017).
- Mori, H., et al. Coronary artery calcification and its progression: What does it really mean. JACC Cardiovasc Imaging. 11 (1), 127-142 (2018).
- Mohan, J., Bhatti, K., Tawney, A., Zeltser, R. . StatPearls [Internet]. , (2023).
- Wang, Y., Osborne, M. T., Tung, B., Li, M., Li, Y. Imaging cardiovascular calcification. J Am Heart Assoc. 7 (13), e008564 (2018).
- Fletcher, A. J., et al. Quantifying microcalcification activity in the thoracic aorta. J Nucl Cardiol. 29 (3), 1372-1385 (2022).
- Derlin, T., et al. Feasibility of 18F-sodium fluoride PET/CT for imaging of atherosclerotic plaque. J Nucl Med. 51 (6), 862-865 (2010).
- Hsu, J. J., et al. Changes in microarchitecture of atherosclerotic calcification assessed by 18F-NaF PET and CT after a progressive exercise regimen in hyperlipidemic mice. J Nucl Cardiol. 28 (5), 2207-2214 (2021).
- Hsu, J. J., et al. Effects of teriparatide on morphology of aortic calcification in aged hyperlipidemic mice. Am J Physiol Heart Circ Physiol. 314 (6), H1203-H1213 (2018).
- Irkle, A., et al. Identifying active vascular microcalcification by 18F-sodium fluoride positron emission tomography. Nat Commun. 6, 7495 (2015).
- Syed, M. B. J., Doris, M., Dweck, M., Forsythe, R., Newby, D. E. Chapter 9 - Imaging vascular calcification: Where are we headed. Coronary Calcium: A Comprehensive Understanding of Its Biology, Use in Screening, and Interventional Management. , (2019).
- Joshi, N. V., et al. 18F-fluoride positron emission tomography for identification of ruptured and high-risk coronary atherosclerotic plaques: A prospective clinical trial. Lancet. 383 (9918), 705-713 (2014).
- Fiz, F., et al. 18F-NaF uptake by atherosclerotic plaque on PET/CT imaging: Inverse correlation between calcification density and mineral metabolic activity. J Nucl Med. 56 (7), 1019-1023 (2015).
- Kruithof, B. P., et al. An in vivo map of bone morphogenetic protein 2 post-transcriptional repression in the heart. Genesis. 49 (11), 841-850 (2011).
- Sheen, C. R., et al. Pathophysiological role of vascular smooth muscle alkaline phosphatase in medial artery calcification. J Bone Miner Res. 30 (5), 824-836 (2015).
- Tsai, M. T., Chen, Y. Y., Chang, W. J., Li, S. Y. Warfarin accelerated vascular calcification and worsened cardiac dysfunction in remnant kidney mice. J Chin Med Assoc. 81 (4), 324-330 (2018).
- Wait, J. M., et al. Detection of aortic arch calcification in apolipoprotein e-null mice using carbon nanotube-based micro-ct system. J Am Heart Assoc. 2 (1), e003358 (2013).
- White, D. J., et al. 19F MAS-NMR and solution chemical characterization of the reactions of fluoride with hydroxyapatite and powdered enamel. Acta Odontol Scand. 46 (6), 375-389 (1988).
- Czernin, J., Satyamurthy, N., Schiepers, C. Molecular mechanisms of bone 18F-NaF deposition. J Nucl Med. 51 (12), 1826-1829 (2010).
- Chen, N. X., O'neill, K. D., Dominguez, J. M., Moe, S. M. Regulation of reactive oxygen species in the pathogenesis of matrix vesicles induced calcification of recipient vascular smooth muscle cells. Vasc Med. 26 (6), 585-594 (2021).
- Klose, A. D., Paragas, N. Automated quantification of bioluminescence images. Nat Commun. 9 (1), 4262 (2018).
- Chen-Mayer, H. H., et al. Standardizing ct lung density measure across scanner manufacturers. Med Phys. 44 (3), 974-985 (2017).
- Coxson, H. O. Sources of variation in quantitative computed tomography of the lung. J Thorac Imaging. 28 (5), 272-279 (2013).
- Rogasch, J. M. M., et al. Influences on pet quantification and interpretation. Diagnostics (Basel). 12 (2), 451 (2022).
- Liljequist, D., Elfving, B., Skavberg Roaldsen, K. Intraclass correlation - a discussion and demonstration of basic features. PLoS One. 14 (7), e0219854 (2019).
- Koo, T. K., Li, M. Y. A guideline of selecting and reporting intraclass correlation coefficients for reliability research. J Chiropr Med. 15 (2), 155-163 (2016).
- Moss, A. J., et al. Molecular coronary plaque imaging using (18)f-fluoride. Circ Cardiovasc Imaging. 12 (18), e008574 (2019).
- Moss, A., et al. Coronary atherosclerotic plaque activity and future coronary events. JAMA Cardiol. 8 (8), 755-764 (2023).
- Badea, C., Hedlund, L. W., Johnson, G. A. Micro-ct with respiratory and cardiac gating. Med Phys. 31 (12), 3324-3329 (2004).
- Yang, Y., Rendig, S., Siegel, S., Newport, D. F., Cherry, S. R. Cardiac pet imaging in mice with simultaneous cardiac and respiratory gating. Phys Med Biol. 50 (13), 2979-2989 (2005).
- Liao, W., Deserno, T. M., Spitzer, K. Evaluation of free non-diagnostic dicom software tools. Proc Spie. 6919, (2008).
- Haak, D., Page, C. E., Deserno, T. M. A survey of dicom viewer software to integrate clinical research and medical imaging. J Digit Imaging. 29 (2), 206-215 (2016).
- Aiello, M., et al. How does dicom support big data management? Investigating its use in medical imaging community. Insights Imaging. 12 (1), 164 (2021).
- Kristanto, W., Van Ooijen, P. M., Groen, J. M., Vliegenthart, R., Oudkerk, M. Small calcified coronary atherosclerotic plaque simulation model: Minimal size and attenuation detectable by 64-MDCT and microCT. Int J Cardiovasc Imaging. 28 (4), 843-853 (2012).
- Dehmeshki, J., et al. Volumetric quantification of atherosclerotic plaque in ct considering partial volume effect. IEEE Trans Med Imaging. 26 (3), 273-282 (2007).
- Demer, L. L., Tintut, Y., Nguyen, K. L., Hsiai, T., Lee, J. T. Rigor and reproducibility in analysis of vascular calcification. Circ Res. 120 (8), 1240-1242 (2017).
- Alluri, K., et al. Scoring of coronary artery calcium scans: History, assumptions, current limitations, and future directions. Atherosclerosis. 239 (1), 109-117 (2015).
- Zhang, L., et al. Advances in CT techniques in vascular calcification. Front Cardiovasc Med. 8, 716822 (2021).
- Achenbach, S., Raggi, P. Imaging of coronary atherosclerosis by computed tomography. Eur Heart J. 31 (12), 1442-1448 (2010).
- Agatston, A. S., et al. Quantification of coronary artery calcium using ultrafast computed tomography. J Am Coll Cardiol. 15 (4), 827-832 (1990).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados