Methods Article
Cuantificación de genomas virales adenoasociados en muestras de vectores purificadas mediante reacción en cadena de la polimerasa por gotas digitales
En este artículo
Resumen
La cuantificación precisa de las copias del genoma del vector de virus adenoasociados (AAV) es fundamental, pero aún no se ha establecido un protocolo estandarizado. Este protocolo describe un método validado para preparar muestras purificadas de AAV y realizar una reacción en cadena de la polimerasa (dd_PCR) digital para cuantificar de forma fiable el título del genoma viral.
Resumen
El virus adenoasociado (AAV) es un virus no patógeno que se utiliza como vehículo de administración para transferir genes terapéuticos a los pacientes. La cuantificación precisa del número de copias del genoma de AAV en preparaciones de vectores es esencial para la optimización de bioprocesos y el cálculo de dosis en estudios preclínicos y clínicos de productos de terapia génica basados en AAV. Actualmente, no existe un protocolo consensuado para la titulación del genoma viral de AAV. En este trabajo se presenta un protocolo de PCR digital en gotas (dd_PCR) para la cuantificación de genomas virales en muestras de vectores purificadas. Las muestras se tratan con DNasa I para eliminar el ADN contaminante no empaquetado. A continuación, las muestras tratadas con DNasa se mezclan con un conjunto adecuado de cebador-sonda (diseñado según el genoma objetivo de AAV) y reactivos de PCR, y luego se cargan en un generador de gotas. Las gotas preparadas se transfieren a una placa de PCR, donde se realiza y analiza la amplificación de PCR. El título del genoma viral se calcula en función de la concentración (copias/μL), teniendo en cuenta las diluciones de la muestra. Una medición exitosa muestra una clara separación de las nubes de gotas positivas y negativas, tiene al menos 10.000 gotas aceptadas, muestra un valor entre 10 copias/μL y 10.000 copias/μL, y tiene un coeficiente de variación (CV) entre repeticiones inferior al 20%. La titulación fiable del genoma viral ayudará en el desarrollo de productos de terapia génica seguros y eficaces basados en AAV.
Introducción
La terapia génica es una modalidad terapéutica comúnmente utilizada para tratar trastornos genéticos. El diseño de cualquier terapia génica es específico de la patología asociada a la indicación diana, pero todas las terapias génicas implican la entrega intracelular de material genético a las células diana con el fin de provocar un efecto terapéutico1. La terapia génica se puede clasificar a su vez en varias categorías, incluyendo el reemplazo de genes para mutaciones de pérdida de función, el silenciamiento de genes para anomalías de ganancia de función y técnicas de edición de genes. Independientemente de la estrategia específica empleada, el material de ácido nucleico terapéutico (denominado transgén) debe empaquetarse dentro de un vector para lograr una administración intracelular dirigida2.
A pesar de que se dispone de una variedad de sistemas de vectores virales y no virales para el desarrollo de la terapia génica, los virus adenoasociados (AAVs) son frecuentemente elegidos debido al amplio tropismo viral y a la baja inmunogenicidad asociada a este grupo de virus 1,2. Hasta la fecha, siete terapias génicas que utilizan AAV para la administración terapéutica de genes han logrado la aprobación de la Agencia Europea de Medicamentos (EMA) o de la Administración de Alimentos y Medicamentos (FDA) dirigidas a enfermedades que van desde la hemofilia (por ejemplo, Roctavian) hasta la atrofia muscular espinal (por ejemplo, Zolgensma)3.
La producción de terapias génicas basadas en AAV se deriva de la comprensión del propio AAV de tipo salvaje. El AAV es un pequeño virus de ADN de la familia Parvoviridae que comprende 13 serotipos principales (AAV1-13)3. El genoma AAV comprende una molécula de ADN monocatenario de ~4,7 kb que contiene dos marcos de lectura abiertos (ORF) principales que codifican los genes virales esenciales necesarios para la replicación del genoma, el ensamblaje de la cápside y el empaquetamiento (rep, cap). El genoma viral está flanqueado en los extremos 5' y 3' por secuencias de nucleótidos palindrómicos, denominadas repeticiones terminales invertidas (ITR). Estos ITR forman estructuras en forma de horquilla que desempeñan un papel crucial en la replicación del genoma y el empaquetamiento de los genomas virales de novo en cápsidas virales recién sintetizadas. El AAV es un virus dependiente de un auxiliar y, por lo tanto, requiere la expresión de genes auxiliares de otros virus, como el virus del herpes simple (VHS) o el adenovirus (AdV) para ser competente para la replicación1.
Con el fin de producir AAV, se emplea un sistema de expresión basado en células adecuado para facilitar la expresión de las proteínas de la cápside viral y su posterior ensamblaje en partículas virales de novo, seguidas de la encapsulación de un transgén seleccionado flanqueado por ITR (también conocido como genoma vectorial). Este proceso comúnmente utiliza un sistema de plásmido triple, que comprende (1) un plásmido que alberga genes auxiliares derivados de un virus auxiliar, (2) un plásmido que codifica los elementos virales esenciales (rep / cap) y (3) un plásmido que lleva el casete de expresión terapéutica (comúnmente conocido como plásmido de transferencia)4. La presencia única de señales de empaquetamiento de repetición terminal invertida (ITR) que flanquean el casete de expresión terapéutica en el plásmido de transferencia garantiza un empaquetamiento específico del transgén, al tiempo que excluye en su mayoría los genes virales presentes en los otros plásmidos. La co-transfección de este sistema de tres plásmidos en una plataforma de expresión basada en células (típicamente HEK293T células) da como resultado la producción de partículas virales competentes para la transducción y deficientes en la replicación, adecuadas para su uso en aplicaciones de terapia génica 3,4.
Hay una serie de atributos críticos de calidad (CQA) asociados con la producción de terapias génicas basadas en AAV que deben evaluarse para garantizar la potencia, pureza y seguridad del medicamento previsto4. Estos CQA incluyen el título de virus, el contenido de cápside y la agregación. El título del virus en sí es una combinación del número de partículas virales (título de la cápside) y el número de genomas de vectores (título del genoma del vector) presentes en cualquier preparación dada. Idealmente, la relación entre estos dos títulos debería ser de 1:1, ya que cada partícula viral debería contener un genoma vectorial, pero las ineficiencias en el empaquetamiento del genoma vectorial durante la biosíntesis dan lugar a la coproducción de cápsidas vacías o parcialmente llenas (aquellas que contienen secuencias parciales del genoma vectorial y/o secuencias del genoma no vectorial)5. La presencia de tales impurezas puede evocar potencialmente respuestas inmunitarias injustificadas y competir por los sitios de unión a los vectores, aumentando así el riesgo de inmunotoxicidad y reduciendo la tasa de transducción de cápsidas completas6. Por lo tanto, la cuantificación precisa de los genomas de AAV es esencial para establecer el título del virus y el contenido de cápside. Esto afecta tanto a la investigación básica como a la industria de la terapia génica, que requiere una dosificación precisa para mantener tanto la seguridad como la eficacia de los medicamentos.
La reacción en cadena de la polimerasa (dd_PCR) en gotas digitales se ha asociado estrechamente con la cuantificación del título del virus, ya que se puede utilizar para determinar el número de genomas de vectores presentes encualquier preparación 7. La PCR digital se introdujo por primera vez en la década de 1990 8,9 y dd_PCR es una mejora de esta tecnología que permite el procesamiento de muestras de alto rendimiento 10,11. En dd_PCR, una reacción de PCR en tiempo real de 20 μL se divide en aproximadamente 20.000 gotas recubiertas de aceite, lo que proporciona hasta 96 reacciones de este tipo cuando se aloja en una placa estándar de 96 pocillos. En comparación con la PCR cuantitativa convencional (qPCR), dd_PCR ofrece varias ventajas, incluida una mayor sensibilidad, una mayor precisión y una cuantificación más directa y absoluta de las secuencias objetivo sin necesidad de curvas estándar. Además, el alto nivel de partición en dd_PCR reduce el impacto de los inhibidores de PCR y minimiza el potencial de sesgo de la amplificación preferencial de ciertos moldes, lo que lo convierte en una opción atractiva para la medición analítica de la valoración del genoma del vector.
Protocolo
El protocolo descrito aquí está diseñado para cuantificar el título del genoma viral de un vector AAV9 purificado y producido internamente con proteína verde fluorescente (GFP) como transgén12 con un alto nivel de precisión (Tabla 1). Sin embargo, este protocolo es aplicable a cualquier serotipo de AAV y a cualquier diseño de genoma vectorial, siempre que los conjuntos de cebadores/sondas estén diseñados para dirigirse al genoma específico del vector de interés. Los detalles de los reactivos, cebadores, sondas y equipos utilizados en este estudio se enumeran en la Tabla de Materiales.
1. Preparación de soluciones madre
NOTA: Prepare todas las soluciones madre necesarias para la dilución de muestras en una estación de trabajo de PCR para evitar la contaminación por ADN extraño.
- Prepare 10 tampones de ensayo de PCR, que consta de 500 mM de KCl, 100 mM de Tris-HCl, 15 mM de MgCl2 y 0,01 % (p/v) de BSA. Mida el pH de la solución y ajústelo a 8.3.
NOTA: Esta solución se puede mantener a temperatura ambiente hasta por 1 mes. - Prepare el tampón de dilución AAV, que consta de 1x tampón de ensayo de PCR, 0,2 ng/μL de ADN esquilado con esperma de salmón (sss) y 0,1 % de F-68 plurónico. Prepare esta solución fresca con cada ensayo y manténgala a 4 °C.
NOTA: Si no se dispone de F-68 plurónico, se podría utilizar una alternativa como el tensioactivo no iónico Poloxamer 188.
2. Preparación de muestras: tratamiento con DNasa I y dilución en serie
NOTA: Con el fin de eliminar los contaminantes del ADN que podrían reducir la precisión de la valoración del genoma del vector, el ADN libre presente en la muestra de AAV (incluido el ADN plasmídico restante o los genomas virales no encapsulados) se puede eliminar antes de la amplificación por PCR utilizando DNasa. Esto es posible porque los genomas de los vectores están encapsulados dentro de las cápsidas de AAV y, por lo tanto, no son accesibles hasta la etapa de desnaturalización de la propia reacción de PCR (ver paso 5). Además, dado que se desconoce el número del genoma del vector en una muestra determinada, es necesario realizar una dilución en serie de las muestras para garantizar que las mediciones permanezcan dentro de los límites superior e inferior detectables. Realice todas las manipulaciones de muestras en una estación de trabajo de PCR. Mantenga todas las muestras en hielo a menos que se especifique lo contrario.
- Mezcle la muestra de AAV seleccionada para el análisis mediante un breve vórtice y luego centrifugado para asegurarse de que todo el líquido permanezca en el fondo del tubo que contiene la muestra.
- Para el tratamiento con DNasa, agregue 45 μL de una solución que contenga 1x tampón de DNasa I, 0,1% de F-68 plurónico y 0,04 U/μL de DNasa I en agua libre de DNasa en un tubo de tira de PCR de 8 tubos de 0,2 mL.
- Transfiera 5 μL de muestra al tubo que contiene la solución de ADNasa. Agite las muestras en vórtice y centrifuga brevemente para asegurarse de que todo el líquido permanezca en el fondo.
- En un termociclador, incube las muestras durante 1 h a 37 °C, luego enfríe a 4 °C. Colocar en hielo tan pronto como sea posible después de alcanzar los 4 °C.
- Con una nueva tira de PCR de 0,2 mL de 8 tubos, realice las diluciones adecuadas de la muestra tratada con DNasa en tampón de dilución AAV. Las diluciones de muestra recomendadas dependen del título de vg esperado (Tabla 2), pero apunte a al menos 2 diluciones diferentes por muestra y ejecute cada dilución por duplicado. Asegúrese de vortexar rigurosamente (2.000-3.000 rpm) las muestras durante 5 s antes de sacar cualquier volumen.
- Después de la dilución en serie, agite las muestras y centrifugue para asegurarse de que todo el líquido permanezca en el fondo de los tubos.
- Incluya un control positivo del que se conozca el título vg y un control negativo (solo tampón de dilución AAV) que también se denomina control sin plantilla (NTC).
NOTA: El control positivo puede ser un AAV de producción propia o un material de referencia comercial de AAV, que incluye la secuencia objetivo de los cebadores/sonda.
3. Preparación de dd_PCR mezcla maestra
NOTA: Seleccione una secuencia diana apropiada dentro del genoma del vector y diseñe un cebador directo, un cebador inverso y sondas de hidrólisis marcadas con fluoróforos reporteros FAM o HEX siguiendo las directrices publicadas13. Los cebadores que se dirigen al transgén son preferibles a los cebadores que se dirigen a los ITR, ya que el transgén es específico del diseño del genoma del vector, y la estructura secundaria asociada con la formación de horquillas de los ITR puede dificultar la unión eficiente de los cebadores. Además, los cebadores ITR podrían sobrestimar los títulos de AAV si las cápsidas parcialmente llenas que contienen fragmentos truncados del genoma del vector están presentes y aún albergan alguna de las secuencias ITR14. Prepare la mezcla maestra dd_PCR en una estación de trabajo separada y dedicada (sala de pre-PCR). Esta sala debe estar separada de aquella en la que se preparan las muestras para evitar la contaminación cruzada.
- Deje que el cebador, la sonda y dd_PCR reactivos de supermezcla alcancen la temperatura ambiente (20-25 °C). Después, haga un vórtice de todos los reactivos y centrifugue brevemente los tubos para recoger todo el líquido en el fondo del tubo.
- Prepare un volumen de dd_PCR mezcla maestra en un tubo de microcentrífuga separado de acuerdo con el número requerido de reacciones combinando los cebadores directos e inversos, la sonda, la supermezcla dd_PCR y el agua libre de DNasa de acuerdo con los volúmenes y concentraciones descritos en la Tabla 3.
- Agite la mezcla maestra y luego centrifugue brevemente el tubo para asegurarse de que todo el líquido permanezca en el fondo del tubo.
NOTA: Prepare siempre dd_PCR volumen de mezcla maestra adicional al requerido (n + 1) para tener en cuenta la pérdida de volumen de pipeteo.
- Agite la mezcla maestra y luego centrifugue brevemente el tubo para asegurarse de que todo el líquido permanezca en el fondo del tubo.
- Transfiera 19,8 μL de la mezcla maestra a cada tubo en una tira nueva de PCR de 8 tubos de 0,2 mL.
4. Generación de gotas
NOTA: Las muestras y dd_PCR mezcla maestra se mezclan por separado antes de cargarlas en un cartucho generador de gotas. Realizar todas las manipulaciones en una estación de trabajo de PCR, preferiblemente una diferente de donde se prepararon las muestras, para evitar la contaminación por amplicones. Alternativamente, limpie a fondo la estación de trabajo de PCR después de preparar las muestras.
- Transfiera 2,2 μL de la muestra diluida al volumen de 19,8 μL de dd_PCR mezcla maestra en cada tubo de la tira de 8 tubos de PCR y mezcle bien. Vórtice y centrifugue brevemente para asegurarse de que todo el líquido permanezca en el fondo.
- Transfiera 20 μL de esta solución a los pocillos contenidos en la fila central de "muestras" de un cartucho generador de gotas. Evite las burbujas en el cartucho generador de gotas; Esto puede hacer que el instrumento dé errores y no se generen gotas.
NOTA: Dado que los cartuchos están en formato de 8 pocillos, al igual que las tiras de tubo de PCR, se recomienda utilizar una pipeta de 8 canales. - Transfiera 60 μL del aceite generador de gotas a los pocillos contenidos en la fila inferior de "aceite" del cartucho generador de gotas.
NOTA: Nunca deje vacío un pocillo del cartucho generador de gotas; Llene el pocillo de muestra vacío con dd_PCR control de tampón para sondas (con el volumen correspondiente descrito anteriormente). - Coloque una junta de goma sobre el cartucho generador de gotas y colóquelo en el generador de gotas.
- Una vez generadas las gotas, utilice una pipeta de 8 canales para transferir lentamente una solución de 42,5 μL desde el chip de dd_PCR a una placa de PCR de pocillos múltiples.
NOTA: Pipetee lentamente y en un ángulo de 45 grados para evitar el cizallamiento de gotas en el líquido durante el pipeteo. Inspeccione visualmente la placa para que todos los pocillos se llenen con la misma cantidad de líquido y las gotas sean visibles como una capa opaca en el pozo. - Selle la placa con una cubierta de papel de aluminio en una máquina de termosellado durante 5 s a 180 °C.
NOTA: Inspeccione visualmente que todos los pozos estén sellados. De lo contrario, repita otros 5 s a 180 °C. La placa sellada puede mantenerse hasta 4 h a 4 °C antes de dd_PCR amplificación en un termociclador.
5. Amplificación dd_PCR
NOTA: El termociclador debe colocarse en una sala separada de la sala de pre-PCR para separar espacialmente las actividades de pre-PCR de la amplificación de PCR y evitar resultados falsos positivos debido a la contaminación del amplicón.
- Coloque la placa en un termociclador (con un módulo de reacción de 96 pocillos de profundidad) para la amplificación por PCR y ciérrela de forma segura.
- Configure el termociclador para que funcione en las condiciones descritas en la Tabla 4 y a un volumen de reacción de 40 μL. Incluya un paso de precalentamiento en la configuración del programa y asegúrese de que la tapa se caliente a 99 °C.
- Una vez finalizado el programa de PCR, mantenga la placa a 4 °C durante al menos 15 minutos para asegurarse de que la placa se enfríe por completo.
NOTA: La placa puede conservarse durante 48-72 h a 4 °C antes de que se tengan que leer las gotas.
6. Lectura de gotas
- Cargue la placa en un lector de gotas, ingrese la siguiente información en el software del sistema y continúe la lectura.
NOTA: Tipo de experimento = Cuantificación directa; Supermix = dd_PCR supermix para sondas (sin dUTP); Tipo de ensayo = objetivo único por canal; Información de destino, canal de señal 1 = canal FAM o canal HEX según la sonda utilizada. - Anote las muestras de modo que la posición de cada muestra se pueda determinar e identificar de manera efectiva a partir del archivo de datos.
7. Análisis de datos
- Realice el análisis de datos utilizando el software compatible proporcionado por el fabricante del lector de gotas.
- Llevar a cabo una prueba de idoneidad del sistema para evaluar el rendimiento general (y, por lo tanto, la fiabilidad) del ensayo.
- Verifique el conteo de eventos (número de gotas) medido para cada pocillo en la placa de PCR. Lo ideal es que el número de eventos esté entre 15.000 y 20.000. Si el recuento de eventos es <10.000, este valor debe excluirse del análisis.
- Evalúe el gráfico de amplitud 1D o 2D y compruebe la presencia de gotas de lluvia.
- Vaya a la pestaña de la tabla de datos y exporte los datos a un archivo de Excel.
- Calcule el título vg siguiendo los pasos a continuación:
- El rango de trabajo de dd_PCR es de 10 a 10 000 copias/μL. Excluya cualquier valor que sea de <10 copias/μL del análisis. El rango recomendado para el equipo de dd_PCR utilizado en este documento es de 25 a 5000 copias/μL.
NOTA: El software de análisis de datos proporcionará un número de genoma vectorial de acuerdo con el volumen de reacción de PCR (40 μL), pero esto no tiene en cuenta los factores de dilución aplicados durante la preparación de la muestra. - Calcule el título de vg de la muestra por volumen (normalmente expresado como vg/mL) teniendo en cuenta los diversos factores de dilución a lo largo de la preparación de la muestra utilizando la siguiente relación:
NOTA: Concentración (valor obtenido del análisis de datos) x 10 (dilución previa al tratamiento de DNasa I) x 10 (dilución de mezcla maestra por PCR) x 1000 (μL a mL) dilución de muestra seleccionada (en tampón de dilución AAV, por ejemplo, Tabla 2). - Calcular el coeficiente de variación (%CV) entre las réplicas y entre las diferentes diluciones de una misma muestra.
NOTA: El %CV se calcula dividiendo la desviación estándar por la media. Si el %CV entre las diferentes diluciones es >20 %, el valor no debe considerarse exacto. - Compruebe los valores del control negativo y positivo. El control negativo debe ser inferior a 5 copias/μL.
- El rango de trabajo de dd_PCR es de 10 a 10 000 copias/μL. Excluya cualquier valor que sea de <10 copias/μL del análisis. El rango recomendado para el equipo de dd_PCR utilizado en este documento es de 25 a 5000 copias/μL.
Resultados
Los resultados se pueden visualizar utilizando el software adecuado. Para cada pocillo, el gráfico de amplitud 1D muestra todas las gotas en relación con su amplitud. Se espera una clara separación entre las gotas positivas y negativas. Si más del 10% del total de gotas se encuentra entre las nubes de gotas positivas y negativas (un fenómeno conocido como lluvia de gotas), es necesario volver a medir la muestra (ver Figura 1). Información adicional sobre la lluvia de gotas está disponible en la discusión.
Se puede crear una tabla de datos para resumir toda la información registrada, incluido el nombre de la muestra, el número de gotas aceptadas (recuentos de eventos) y la concentración (copias/μL). Lo ideal es que el recuento de eventos se sitúe entre 15.000 y 20.000 gotas aceptadas. Si el recuento de eventos de un pozo es inferior a 10.000, el punto de datos debe excluirse del análisis. En el Cuadro 5 se proporciona un ejemplo de los datos de salida.
El título vg se puede calcular en función de la concentración (copias/μL), teniendo en cuenta las diluciones de la muestra. El rango de trabajo de dd_PCR es de 10-10.000 copias/μL. Los valores inferiores a 10 copias/μL deben excluirse del análisis. Se debe calcular el título vg de las muestras, así como los controles positivos y negativos. El valor medido del testigo positivo debe tener un coeficiente de variación (%CV) inferior al 20% en relación con el valor teórico. El testigo negativo debe ser inferior a 5 copias/μL. Además, se debe calcular el %CV entre repeticiones y diferentes diluciones para cada muestra. Si el %CV entre las diferentes diluciones supera el 20%, el valor puede considerarse inexacto y puede ser necesario volver a medir la muestra.
Una medición exitosa se caracteriza por una clara separación de las nubes de gotas positivas y negativas, al menos 10.000 gotas aceptadas, un valor entre 10 copias/μL y 10.000 copias/μL, y un %CV entre repeticiones inferior al 20%.
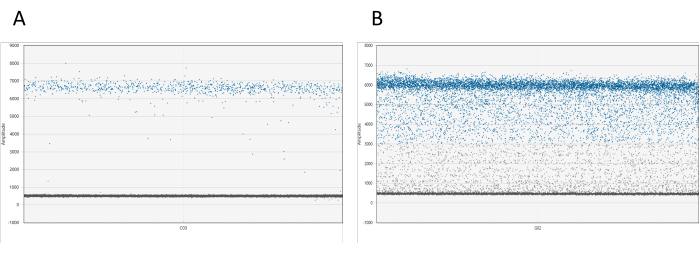
Figura 1: Visualización de dd_PCR gotas. (A) El gráfico de amplitud 1D muestra una clara separación de gotas positivas y negativas, lo que indica una partición exitosa de las gotas. (B) El gráfico muestra una mala separación de gotas positivas y negativas, conocida como lluvia de gotas, lo que sugiere una partición subóptima o problemas potenciales con el ensayo. Haga clic aquí para ver una versión más grande de esta figura.
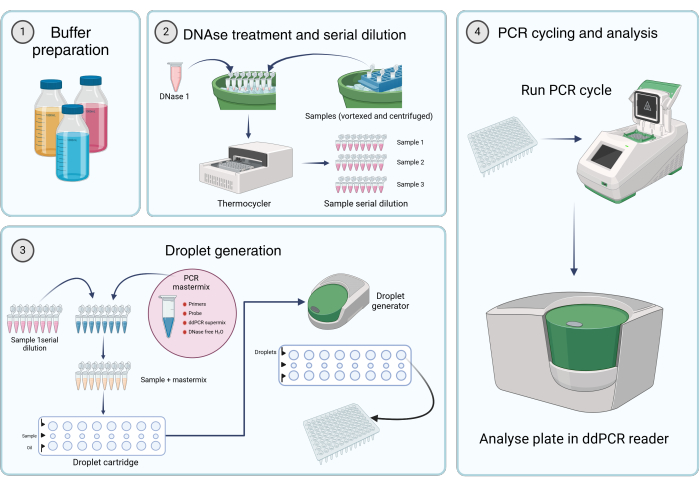
Figura 2: Flujo de trabajo para cuantificar genomas virales mediante PCR digital en gotas. (1) Los tampones, reactivos y soluciones se preparan de acuerdo con las instrucciones del fabricante o el paso 1 del protocolo (Preparación de soluciones madre). (2) Se alícuota 45 μL de la solución de DNasa I en cada tubo de una tira de PCR de 8 tubos. Después de vórtice y centrifugar brevemente las muestras, se añaden 5 μL de la muestra a uno de los tubos. La DNasa I y la tira de PCR de ocho tubos que contiene la muestra se incuban en un termociclador durante 1 h a 37 °C, seguidas de diluciones en serie de las muestras. (3) La mezcla maestra de PCR se prepara como se describe y se alícuota 19,8 μL en cada tubo de una tira de PCR de 8 tubos. Las diluciones en serie de muestra del paso 2 se añaden a la mezcla maestra. 20 μL de mastermix plus
la solución de muestra se carga en la fila central del cartucho y se transfieren 60 μL de aceite generador de gotas a los pocillos inferiores de un cartucho generador de gotas. A continuación, el cartucho se coloca en el generador de gotas y funciona de acuerdo con las condiciones especificadas. Después de la generación de gotas, 42,5 μL de la fila superior del cartucho se transfieren a una placa de PCR de pocillos múltiples. (4) La placa PCR se carga en un termociclador PCR y se ejecuta de acuerdo con las condiciones proporcionadas. La placa se analiza utilizando un lector de dd_PCR. Haga clic aquí para ver una versión más grande de esta figura.
Tabla 1: Datos de precisión. Esta tabla presenta datos de precisión de cuatro conjuntos de muestras de control de calidad (QC), cada uno con cinco concentraciones (QC1-QC5). Cada control de calidad se midió cinco veces. Se muestra el coeficiente de variación (%CV) entre diferentes corridas, que representa la repetibilidad (A), y entre diferentes conjuntos de muestras, que representa la precisión intermedia (B). Haga clic aquí para descargar esta tabla.
Tabla 2: Diluciones de la muestra. La tabla muestra las diluciones de muestra recomendadas en función del título esperado del genoma viral (vg/mL). Se recomienda un total de tres diluciones para garantizar que al menos dos valores se encuentren dentro del rango de trabajo del ensayo. Haga clic aquí para descargar esta tabla.
Tabla 3: dd_PCR composición de la mezcla maestra. Esta tabla describe la composición de la mezcla maestra de dd_PCR, que incluye un cebador directo e inverso (909 nM), una sonda (227 nM) y una supermezcla dd_PCR para sondas (sin dUTP, 1x). Haga clic aquí para descargar esta tabla.
Tabla 4: Condiciones de termociclado. La tabla detalla el programa de PCR recomendado, que incluye: (1) una incubación de 10 minutos a 95 °C para la disrupción de la cápside y la activación de enzimas, (2) 40 ciclos de 30 s a 94 °C para la desnaturalización del ADN, y 1 minuto a 60 °C para el recocido y la extensión, (3) una incubación de 10 minutos a 98 °C para la desactivación de enzimas, y (4) manteniéndose a 4 °C. La temperatura de recocido puede requerir una optimización basada en el conjunto de cebador/sonda utilizado. Haga clic aquí para descargar esta tabla.
Tabla 5: Ejemplo de datos de salida de una ejecución real de AAV. La tabla proporciona un ejemplo de los datos de salida de una ejecución real de AAV, donde se midió una muestra en dos diluciones diferentes, cada una duplicada. El título del genoma viral (vg) (en vg/mL) se calcula utilizando la siguiente fórmula: 10 (pretratamiento con DNasa I) x 10 (dilución en la mezcla maestra) x 1.000 (μL a mL) x dilución en tampón de dilución AAV. STDEV representa la desviación estándar y CV indica el coeficiente de variación. Haga clic aquí para descargar esta tabla.
Discusión
La cuantificación precisa de las copias del genoma del vector AAV en preparaciones de vectores es esencial para el desarrollo de productos de terapia génica basados en AAV. Existen varios métodos para determinar el título de vg, siendo la PCR cuantitativa (qPCR) y la dd_PCR las dos técnicas más utilizadas y aceptadas. A menudo se prefiere dd_PCR sobre la qPCR debido a su independencia de la eficiencia de la amplificación, su mayor precisión y su mayor robustez15. En la literatura se dispone de numerosos protocolos para la titulación del genoma del vector por dd_PCR, cada uno con sus propios métodos de preparación de muestras 15,16,17. Sin embargo, existe una falta de un protocolo de consenso calificado. Este artículo presenta un protocolo de dd_PCR validado para la cuantificación de genomas de vectores AAV en muestras de vectores purificadas (Figura 2).
Es fundamental prestar mucha atención a la manipulación de muestras al realizar este protocolo. La contaminación cruzada puede plantear un desafío significativo en la evaluación precisa de los títulos de vg; por lo tanto, las muestras se procesan mejor en una estación de trabajo de PCR para evitar la contaminación con ADN extraño. Además, la ejecución adecuada de la incubación de la DNasa es crucial para eliminar el ADN contaminante no empaquetado sin alterar la cápside y potencialmente digerir el genoma del vector empaquetado. Muchos protocolos incluyen la inactivación por calor y el tratamiento con proteinasa K 16,17,18. Sin embargo, durante el desarrollo interno del método, se encontró que el calentamiento excesivo era perjudicial para la titulación de vg, y el tratamiento con proteinasa K era innecesario (datos no mostrados).
A medida que dd_PCR se vuelve cada vez más popular para cuantificar genomas virales, los fabricantes han publicado guías de aplicación detalladas con pautas sobre el diseño y la optimización de ensayos de dd_PCR19. También está disponible información para la solución de problemas. En la práctica, cuando un ensayo está correctamente diseñado, los problemas más frecuentes incluyen la lluvia de gotas y el bajo recuento de gotas. La lluvia de gotas a menudo es causada por una mala accesibilidad del cebador/sondas al amplicón, pero varias estrategias pueden abordar este problema. Por ejemplo, un experimento de gradiente de temperatura puede ayudar a establecer la temperatura óptima de recocido. Además, diluir las muestras para reducir la cantidad de ADN (se recomienda que sea inferior a 66 ng) o realizar una digestión de restricción con enzimas específicas que cortan fuera de la región del amplicón puede mejorar la accesibilidad de la plantilla. Una recomendación de 10 U de enzima de restricción por μg de ADN suele ser efectiva. El problema del bajo recuento de gotas (menos de 10.000) se debe con frecuencia a un pipeteo deficiente de la muestra y al aceite de los cartuchos. Se recomienda un pipeteo lento con las puntas adecuadas para evitar el cizallamiento de las gotas. Si bien el ensayo dd_PCR es sólido, una limitación es el tiempo prolongado para obtener resultados. El proceso desde el tratamiento de la muestra hasta la lectura de las gotas tarda varias horas, lo que puede ser un inconveniente cuando se necesitan resultados rápidamente.
Es necesaria una cuantificación precisa de las copias del genoma del AAV en preparaciones de vectores en todas las etapas del ciclo de vida de la terapia génica con AAV. En concreto, es importante para optimizar los procesos de producción y purificación, realizar estudios preclínicos y determinar las dosis clínicas de los productos de terapia génica basados en AAV. El protocolo dd_PCR presentado aquí es ampliamente aplicable y se puede utilizar en productos AAV purificados con diferentes serotipos y transgenes.
Divulgaciones
IVH, BM y EH son cofundadores de Tavira Therapeutics. Los demás autores declaran no tener conflicto de intereses.
Agradecimientos
Esta investigación ha sido posible gracias a la subvención «Flandes Resilience» del Gobierno flamenco, procedente del «Mecanismo Europeo de Recuperación y Resiliencia» (MRR) (VV021/13). La figura 2 se crea con Biorender.com.
Materiales
| Name | Company | Catalog Number | Comments |
| 8-channel pipette 10 µL | Eppendorf | 3,12,50,00,010 | |
| 8-channel pipette 200 µL | Eppendorf | 3,12,50,00,036 | |
| 8-channel pipette 300 µL | Eppendorf | 3,12,50,00,052 | |
| 8-well PCR strip | Sarstedt | 72.991.002 | |
| Bovine Serum Albumin (BSA) | Sigma-Aldrich | A9418 | |
| C1000 Touch Thermal Cycler with 96–Deep Well Reaction Module | Bio-Rad | 1851197 | |
| ddPCR Buffer Control for Probes 9 mL (2 x 4.5 mL) | Bio-Rad | 1863052 | |
| ddPCR Supermix for Probes (No dUTP) (2 x 1 mL) | Bio-Rad | 1863023 | |
| ddPCR 96-Well Plates (pkg of 25) | Bio-Rad | 12001925 | |
| ddPCR Droplet Reader Oil (2 x 1L) | Bio-Rad | 1863004 | |
| DG8 Cartridge Holder | Bio-Rad | 1863051 | |
| DG8 Cartridges for QX200/QX100 (pkg of 24) | Bio-Rad | 1864008 | |
| DG8 Gaskets for QX200/QX100 (pkg of 24) | Bio-Rad | 1863009 | |
| DNase I (10U/µL) + buffer | Roche | 4716728001 | |
| Droplet Generation Oil for Probes (10 x 7 mL) | Bio-Rad | 1863005 | |
| Eppendorf ep Dualfilter T.I.P.S. Filter Tip, 0.1-10 μL, 34 mm, Rack, PCR Clean, STERILE | Eppendorf | 30078500 | |
| Eppendorf ep Dualfilter T.I.P.S. Filter Tip, 20-300 μL, 55 mm, Rack, PCR Clean, STERILE | Eppendorf | 30078560 | |
| Eppendorf ep Dualfilter T.I.P.S. Filter Tip, 2-100 μL, 53 mm, Rack, PCR Clean, STERILE | Eppendorf | 30078543 | |
| Forward lyophilized primers and respective master stocks at 100 mM | IDT | GFP as target sequence. Forward primer: 5'-GAACGGCATCAAGGTGAACT-3' | |
| Lyopohilized probe and master stock at 100 µM | IDT | GFP as target sequence. PrimeTime Eco Probe: /56-FAM/CAAGATCCG/ZEN/CCACAACATCGAGGA/3IABkFQ/ | |
| Magnesium Chloride (MgCl2) | Chemlab Analytical | CL00.1381 | |
| Nuclease free water | IDT | 11-04-02-01 | |
| PCR Plate Heat Seal, foil, pierceable (pkg of 100) | Bio-Rad | 1814040 | |
| Pluronic F-68 non-ionic surfactant (100x) | Thermo Fisher Scientific | 24040032 | |
| Potassium Chloride (KCl) | Honeywell research chemicals | 31248 | |
| QX manager software | Bio-Rad | Software to analyse ddPCR data | |
| QX200 Droplet Generator | Bio-Rad | 1864002 | |
| QX200 Droplet Reader | Bio-Rad | 1864003 | |
| Reagent reservoir | VWR | 613-1181 | |
| Reverse lyophilized primers and respective master stocks at 100 mM | IDT | GFP as target sequence. Reverse primer: 5'-TGCTCAGGTAGTGGTTGTCG-3' | |
| SafeSeal reaction tube, 1.5 mL | Sarstedt | 72.706.200 | |
| Salmon Sperm DNA, sheared (10 mg/mL) | Thermo Fisher Scientific | AM9680 | |
| TE buffer | IDT | Accompanied by primers when ordering | |
| Tris hydrochloride (Tris-HCl) | Roche | 10812846001 |
Referencias
- Balakrishnan, B., Jayandharan, G. R. Basic biology of adeno-associated virus (AAV) vectors used in gene therapy. Curr Gene Ther. 14 (2), 86-100 (2014).
- Srivastava, A. In vivo tissue-tropism of adeno-associated viral vectors. Curr Opin Virol. 21, 75-80 (2016).
- Pupo, A., et al. AAV vectors: The Rubik's cube of human gene therapy. Mol Ther. 30 (12), 3515-3541 (2022).
- Gimpel, A. L., et al. Analytical methods for process and product characterization of recombinant adeno-associated virus-based gene therapies. Mol Ther Methods Clin Dev. 20, 740-754 (2021).
- Brimble, M. A., et al. Preventing packaging of translatable P5-associated DNA contaminants in recombinant AAV vector preps. Mol Ther Methods Clin Dev. 24, 280-291 (2022).
- Wright, J. F. Product-related impurities in clinical-grade recombinant AAV vectors: Characterization and risk assessment. Biomedicines. 2 (1), 80-97 (2014).
- Dobnik, D., et al. Accurate quantification and characterization of adeno-associated viral vectors. Front Microbiol. 10, (2019).
- Sidransky, D., et al. Identification of ras oncogene mutations in the stool of patients with curable colorectal tumors. Science. 256 (5053), 102-105 (1992).
- Vogelstein, B., Kinzler, K. W. Digital PCR. Proc Natl Acad Sci USA. 96 (16), 9236-9241 (1999).
- Hindson, B. J., et al. High-throughput droplet digital PCR system for absolute quantitation of DNA copy number. Anal Chem. 83 (22), 8604-8610 (2011).
- Pinheiro, L. B., et al. Evaluation of a droplet digital polymerase chain reaction format for DNA copy number quantification. Anal Chem. 84 (2), 1003-1011 (2012).
- Sanmiguel, J., Gao, G., Vandenberghe, L. H. Quantitative and digital droplet-based AAV genome titration. Methods Mol Biol. 1950, 51-83 (2019).
- . Planning droplet digital PCR experiments Available from: https://www.bio-rad.com/en-in/life-science/learning-center/introduction-to-digital-pcr/planning-ddpcr-experiments (2024)
- Dorange, F., Le Bec, C. Analytical approaches to characterize AAV vector production & purification: Advances and challenges. Cell Gene Ther Insights. 4 (2), 119-129 (2018).
- Lock, M., et al. Absolute determination of single-stranded and self-complementary adeno-associated viral vector genome titers by droplet digital PCR. Hum Gene Ther Methods. 25 (2), 115-125 (2014).
- Prantner, A., Maar, D. Genome concentration, characterization, and integrity analysis of recombinant adeno-associated viral vectors using droplet digital PCR. PLoS One. 18 (1), e0280242 (2023).
- Dobnik, D., et al. Accurate quantification and characterization of adeno-associated viral vectors. Front Microbiol. 10, 1570 (2019).
- Suoranta, T., Laham-Karam, N., Yla-Herttuala, S. Optimized protocol for accurate titration of adeno-associated virus vectors. Hum Gene Ther. 32 (19-20), 1270-1279 (2021).
- Droplet digital PCR applications guide. Bulletin 6407 Available from: https://www.bio-rad.com/webroot/web/pdf/lsr/literature/Bulletin_6407.pdf (2024)
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados