Method Article
Digital Droplet Polymerase Chain Reaction에 의한 정제된 벡터 샘플의 Adeno-Associated Viral Genomes 정량화
요약
아데노 관련 바이러스(AAV) 벡터 게놈 복제의 정확한 정량화는 매우 중요하지만 표준화된 프로토콜은 아직 확립되지 않았습니다. 이 프로토콜은 바이러스 게놈 역가를 신뢰성 있게 정량화하기 위해 정제된 AAV 샘플을 준비하고 디지털 액적 중합효소 연쇄 반응(dd_PCR)을 수행하기 위한 검증된 방법을 설명합니다.
초록
아데노부속바이러스(AAV)는 치료용 유전자를 환자에게 전달하기 위한 전달 수단으로 사용되는 비병원성 바이러스입니다. 벡터 제제에서 AAV 게놈 복제 수의 정확한 정량화는 AAV 기반 유전자 치료제의 전임상 및 임상 연구 모두에서 생물공정 최적화 및 투여량 계산에 필수적입니다. 현재 AAV 바이러스 게놈 적정에 대한 합의 프로토콜이 부족합니다. 여기에서는 정제된 벡터 샘플에서 바이러스 게놈의 정량화를 위한 디지털 액적 PCR(dd_PCR) 프로토콜을 제시합니다. 샘플은 DNase I로 처리되어 포장되지 않은 오염 물질 DNA를 제거합니다. 그런 다음 DNase 처리된 샘플을 적절한 프라이머-프로브 세트(표적 AAV 게놈에 따라 설계됨) 및 PCR 시약과 혼합한 다음 액적 발생기에 로드합니다. 준비된 액적은 PCR 플레이트로 옮겨져 PCR 증폭이 수행되고 분석됩니다. 바이러스 게놈 역가는 시료 희석을 고려한 농도(copies/μL)를 기준으로 계산됩니다. 성공적인 측정은 양극 및 음극 액적 구름의 명확한 분리를 보여주고, 최소 10,000개의 허용 액적이 있으며, 10 copies/μL 및 10,000 copies/μL 사이의 값을 보여주고, 반복 간 변동 계수(CV)가 20% 미만임을 보여줍니다. 신뢰할 수 있는 바이러스 게놈 적정은 안전하고 효과적인 AAV 기반 유전자 치료 제품의 개발에 도움이 될 것입니다.
서문
유전자 치료는 유전 질환을 치료하는 데 일반적으로 사용되는 치료 방식입니다. 주어진 유전자 치료제의 설계는 표적 적응증의 관련 병리학에 따라 다르지만, 모든 유전자 치료는 치료 효과를 이끌어내기 위해 유전 물질을 표적 세포에 세포 내 전달하는 것을 포함합니다1. 유전자 치료는 기능 상실 돌연변이에 대한 유전자 대체, 기능 향상 이상에 대한 유전자 침묵, 유전자 편집 기술 등 여러 범주로 분류할 수 있습니다. 채택된 특정 전략에 관계없이, 치료용 핵산 물질(전이유전자라고 함)은 표적 세포내 전달을 달성하기 위해 벡터 내에 패키징되어야 합니다2.
유전자 치료제 개발을 위해 다양한 바이러스 및 비바이러스 벡터 시스템을 사용할 수 있지만, 아데노 관련 바이러스(AAV)는 이 바이러스 그룹과 관련된 광범위한 바이러스 영양성과 낮은 면역원성으로 인해 자주 선택됩니다 1,2. 현재까지 치료용 유전자 전달을 위해 AAV를 사용하는 7개의 유전자 치료제가 유럽의약청(EMA) 또는 식품의약국(FDA)으로부터 혈우병(예: Roctavian)에서 척수성 근위축증(예: Zolgensma)에 이르는 질병을 대상으로 하는 승인을 받았습니다3.
AAV 기반 유전자 치료제의 생산은 야생형 AAV 자체에 대한 이해에서 비롯됩니다. AAV는 13개의 주요 혈청형(AAV1-13)으로 구성된 Parvoviridae 계열의 작은 DNA 바이러스입니다3. AAV 게놈은 게놈 복제, 캡시드 조립 및 패키징(rep, cap)에 필요한 필수 바이러스 유전자를 암호화하는 두 개의 주요 개방형 판독 프레임(ORF)을 포함하는 ~4.7kb의 단일 가닥 DNA 분자로 구성됩니다. 바이러스 게놈은 5'와 3' 말단 양쪽 측면에 ITR(inverted terminal repeats)이라고 하는 회문 뉴클레오티드 서열이 있습니다. 이러한 ITR은 게놈 복제 및 de novo 바이러스 게놈을 새로 합성된 바이러스 캡시드로 패키징하는 데 중요한 역할을 하는 헤어핀과 같은 구조를 형성합니다. AAV는 도우미 의존성 바이러스(helper-dependent virus)이기 때문에 복제 능력이 있으려면 단순 포진 바이러스(herpes simplex virus, HSV) 또는 아데노바이러스(adenovirus, AdV)와 같은 다른 바이러스의 보조 유전자 발현이 필요하다1.
AAV를 생산하기 위해, 적절한 세포 기반 발현 시스템을 사용하여 바이러스 캡시드 단백질의 발현을 촉진하고 후속 de novo 바이러스 입자로의 조립을 촉진한 후, ITR-측면 선택된 전이 유전자(벡터 게놈이라고도 함)의 캡슐화를 수행합니다. 이 과정은 일반적으로 (1) 도우미 바이러스에서 유래한 도우미 유전자를 보유하는 플라스미드, (2) 필수 바이러스 요소(rep/cap)를 암호화하는 플라스미드, (3) 치료 발현 카세트(일반적으로 전달 플라스미드라고 함)를 운반하는 플라스미드로 구성된 트리플 플라스미드 시스템을 사용합니다4. transfer plasmid에서 therapeutic expression cassette 측면에 있는 inverted terminal repeat (ITR) packaging 신호의 고유한 존재는 transgene의 특정 packaging을 보장하는 동시에 다른 plasmid에 존재하는 바이러스 유전자를 대부분 배제합니다. 이 3-플라스미드 시스템을 세포 기반 발현 플랫폼(일반적으로 HEK293T 세포)으로 동시 transfection하면 유전자 치료 응용 분야에 사용하기에 적합한 transduction-competent, replication-deficient viral particles 가 생성됩니다 3,4.
AAV 기반 유전자 치료제의 생산과 관련된 여러 가지 중요한 품질 속성(CQA)이 있으며, 이는 의도된 의약품의 효능, 순도 및 안전성을 보장하기 위해 평가되어야 합니다4. 이러한 CQA에는 바이러스 역가, 캡시드 함량 및 응집이 포함됩니다. 바이러스 역가 자체는 주어진 제제에 존재하는 바이러스 입자의 수(캡시드 역가)와 벡터 게놈의 수(벡터 게놈 역가)의 조합입니다. 이상적으로는 각 바이러스 입자가 하나의 벡터 게놈을 포함해야 하기 때문에 이 두 역가 사이의 비율은 1:1이어야 하지만, 생합성 중 벡터 게놈 패키징의 비효율성으로 인해 비어 있거나 부분적으로 채워진 캡시드(부분 벡터 게놈 서열 및/또는 비벡터 게놈 서열을 포함하는 캡시드)가 함께 생성됩니다5. 이러한 불순물의 존재는 잠재적으로 부당한 면역 반응을 유발하고 벡터 결합 부위를 놓고 경쟁할 수 있으며, 이로 인해 면역독성의 위험이 증가하고 전체 캡시드의 transduction 속도가 감소할 수 있습니다6. 따라서 AAV 게놈의 정확한 정량화는 바이러스 역가 및 캡시드 함량을 확립하는 데 필수적입니다. 이는 기초 연구뿐만 아니라 유전자 치료 산업 모두에 영향을 미치며, 의약품의 안전성과 효능을 모두 유지하기 위해 정확한 투여량이 필요합니다.
디지털 액적 중합효소 연쇄 반응(dd_PCR)은 주어진 제제에 존재하는 벡터 게놈의 수를 측정하는 데 사용할 수 있기 때문에 바이러스 역가의 정량화와 밀접한 관련이 있습니다7. 디지털 PCR 자체는 1990년대에 처음 도입되었으며8,9 dd_PCR 이 기술이 향상되어 고처리량 시료 처리가 가능합니다10,11. dd_PCR에서 20 μL Real-Time PCR 반응은 약 20,000개의 오일로 둘러싸인 액적으로 분할되어 표준 96웰 플레이트에 수용될 때 최대 96개의 이러한 반응을 제공합니다. 기존의 정량적 PCR(qPCR)과 비교했을 때, dd_PCR은 향상된 감도, 향상된 정밀도, 표준 곡선 없이도 표적 염기서열의 보다 직접적이고 절대적인 정량화를 포함한 여러 가지 이점을 제공합니다. 또한, dd_PCR의 높은 수준의 partitioning은 PCR inhibitor의 영향을 줄이고 특정 template의 우선적 증폭으로 인한 바이어스 가능성을 최소화하여 벡터 게놈 적정의 분석 측정에 매력적인 옵션입니다.
프로토콜
여기에 설명된 프로토콜은 녹색 형광 단백질(GFP)을 전이유전자12 로 사용하여 정제된 자체 생산 AAV9 벡터의 바이러스 게놈 역가를 높은 정밀도로 정량화하도록 설계되었습니다(표 1). 그러나 이 프로토콜은 프라이머/프로브 세트가 관심 있는 특정 벡터 게놈을 표적으로 하도록 설계된 경우 모든 AAV 혈청형 및 모든 벡터 게놈 설계에 적용할 수 있습니다. 이 연구에 사용된 시약, 프라이머, 프로브 및 장비에 대한 자세한 내용은 재료 표에 나열되어 있습니다.
1. 원액 준비
참고: 외부 DNA로 인한 오염을 방지하기 위해 PCR 워크스테이션에서 시료 희석에 필요한 모든 원액을 준비하십시오.
- 500mM의 KCl, 100mM의 Tris-HCl, 15mM의 MgCl2 및 0.01%(w/v) BSA로 구성된 10x PCR 분석 완충액을 준비합니다. 용액의 pH를 측정하고 8.3으로 조정합니다.
알림: 이 용액은 실온에서 최대 1개월 동안 보관할 수 있습니다. - 1x PCR 분석 완충액, 0.2ng/μL 연어 정자 전단(sss) DNA 및 0.1% 플루로닉 F-68로 구성된 AAV 희석 완충액을 준비합니다. 각 분석과 함께 이 용액을 신선하게 만들고 4°C에서 보관하십시오.
알림: pluronic F-68을 사용할 수 없는 경우 Poloxamer 188 비이온성 계면활성제와 같은 대안을 사용할 수 있습니다.
2. 시료 준비 - DNase I 처리 및 연속 희석
참고: 벡터 게놈 적정의 정확도를 낮출 수 있는 DNA 오염 물질을 제거하기 위해 AAV 샘플 내에 존재하는 유리 DNA(남아 있는 플라스미드 DNA 또는 캡슐화되지 않은 바이러스 게놈 포함)를 DNase를 사용한 PCR 증폭 전에 제거할 수 있습니다. 이는 벡터 게놈이 AAV 캡시드 내에 캡슐화되어 있기 때문에 PCR 반응 자체의 변성 단계까지 접근할 수 없기 때문에 가능합니다(5단계 참조). 또한 주어진 샘플의 벡터 게놈 수를 알 수 없기 때문에 측정값이 검출 가능한 상한 및 하한 범위 내에서 유지되도록 하기 위해 샘플의 연속 희석을 수행해야 합니다. PCR 워크스테이션에서 모든 시료 조작을 수행합니다. 달리 명시되지 않는 한 모든 샘플을 얼음에 보관하십시오.
- 분석을 위해 선택한 AAV 시료를 잠시 소용돌이친 다음 원심분리하여 모든 액체가 시료가 들어 있는 튜브 바닥에 남아 있도록 혼합합니다.
- DNase 처리의 경우 DNase가 없는 물에 1x DNase I 완충액, 0.1% pluronic F-68 및 0.04U/μL DNase I이 포함된 용액 45μL를 0.2mL PCR 8-튜브 스트립 튜브에 추가합니다.
- 5μL의 시료를 DNAse 용액이 들어 있는 튜브로 옮깁니다. 샘플을 소용돌이치게 하고 모든 액체가 바닥에 남아 있는지 확인하기 위해 잠시 원심분리합니다.
- Thermocycler에서 37°C에서 1시간 동안 샘플을 배양한 다음 4°C로 냉각합니다. 4 °C에 도달한 후 가능한 한 빨리 얼음 위에 놓으십시오.
- 새로운 0.2mL PCR 8-튜브 스트립을 사용하여 AAV 희석 완충액에서 DNase 처리된 샘플을 적절하게 희석합니다. 권장되는 시료 희석량은 예상 vg 역가(표 2)에 따라 다르지만, 시료당 최소 2개의 다른 희석을 목표로 하고 각 희석을 중복하여 실행합니다. 엄격하게(2,000-3,000rpm) s를 소용돌이치십시오.amp부피를 꺼내기 전에 5초 동안.
- 연속 희석 후 샘플과 원심분리기를 소용돌이치게 하여 모든 액체가 튜브 바닥에 남아 있도록 합니다.
- vg titer가 알려진 positive control과 no template control(NTC)이라고도 하는 negative control(AAV 희석 버퍼만 해당)을 포함합니다.
참고: 포지티브 컨트롤은 자체 생산된 AAV 또는 프라이머/프로브의 타겟 염기서열을 포함하는 상용 AAV 표준 물질일 수 있습니다.
3. 마스터 믹스dd_PCR 준비
참고: 벡터 게놈 내에서 적절한 표적 염기서열을 선택하고 발표된 지침13에 따라 FAM 또는 HEX 리포터 형광단으로 표지된 전방 프라이머, 역방향 프라이머 및 가수분해 프로브를 설계합니다. 전이유전자를 표적으로 하는 프라이머는 ITR을 표적으로 하는 프라이머보다 선호되는데, 이는 전이유전자가 벡터 게놈 디자인에 특이적이고, ITR의 헤어핀 형성과 관련된 2차 구조가 효율적인 프라이머 결합을 방해할 수 있기 때문이다. 더욱이, ITR 프라이머는 잘린 벡터 게놈 단편을 포함하는 부분적으로 채워진 캡시드가 존재하고 여전히 ITR 서열(14) 중 하나를 수용하는 경우 AAV 역가를 과대평가할 수 있습니다. dd_PCR 마스터 믹스를 별도의 전용 워크스테이션(pre-PCR 룸)에서 준비합니다. 이 방은 교차 오염을 피하기 위해 검체가 준비되는 방과 분리되어야 합니다.
- 프라이머, 프로브 및 dd_PCR 슈퍼믹스 시약이 실온(20-25°C)에 도달하도록 합니다. 그 후, 모든 시약을 소용돌이치고 튜브를 잠시 원심분리하여 튜브 바닥에 모든 액체를 수집합니다.
- 표 3에 기재된 부피 및 농도에 따라 정방향 및 역방향 프라이머, 프로브, dd_PCR 슈퍼믹스 및 DNase-free 물을 결합하여 필요한 반응 횟수에 따라 별도의 마이크로 원심분리기 튜브에 dd_PCR 마스터 믹스를 준비합니다.
- 마스터 혼합물을 소용돌이친 다음 튜브를 잠시 원심분리하여 모든 액체가 튜브 바닥에 남아 있는지 확인합니다.
참고: 피펫팅 부피 손실을 설명하기 위해 항상 필요한 것보다 더 많은 dd_PCR 마스터 믹스 부피(n + 1)를 준비하십시오.
- 마스터 혼합물을 소용돌이친 다음 튜브를 잠시 원심분리하여 모든 액체가 튜브 바닥에 남아 있는지 확인합니다.
- 19.8 μL의 마스터 믹스를 새로운 0.2 mL PCR 8-튜브 스트립의 각 튜브에 전달합니다.
4. 액적 생성
참고: 샘플과 dd_PCR 마스터 믹스는 액적 생성 카트리지에 로드하기 전에 별도로 혼합됩니다. 앰플리콘 오염을 방지하기 위해 PCR 워크스테이션에서 모든 조작을 수행하며, 가급적이면 샘플이 준비된 곳과 다른 워크스테이션에서 수행합니다. 또는 샘플을 준비한 후 PCR 워크스테이션을 철저히 청소합니다.
- 희석된 샘플 2.2μL를 PCR 8-튜브 스트립의 각 튜브에 있는 마스터 믹스의 19.8μL 부피dd_PCR 옮기고 잘 혼합합니다. 소용돌이와 잠시 원심 분리기를 사용하여 모든 액체가 바닥에 남아 있는지 확인합니다.
- 이 용액의 20μL를 액적 생성 카트리지의 중간 "샘플" 행에 포함된 웰로 옮깁니다. 물방울 생성 카트리지에 기포가 생기지 않도록 하십시오. 이로 인해 기기에서 오류가 발생하고 물방울이 생성되지 않을 수 있습니다.
참고: 카트리지는 PCR 튜브 스트립과 마찬가지로 8-웰 형식이므로 8-채널 피펫을 사용하는 것이 좋습니다. - 액적 생성 오일의 60μL를 액적 생성 카트리지의 하단 "오일" 행에 포함된 웰로 이송합니다.
알림: 물방울 생성 카트리지의 웰을 비워 두지 마십시오. 빈 샘플을 프로브용 dd_PCR Buffer Control로 잘 채웁니다(위에서 설명한 해당 볼륨 포함). - 액적 생성 카트리지 위에 고무 개스킷을 놓고 액적 발생기에 넣습니다.
- 액적이 생성된 후 8채널 피펫을 사용하여 dd_PCR 칩의 42.5μL 용액을 멀티웰 PCR 플레이트로 천천히 옮깁니다.
알림: 피펫팅하는 동안 액체에서 물방울이 깎이지 않도록 천천히 45도 각도로 피펫팅하십시오. 모든 웰이 동일한 양의 액체로 채워지고 물방울이 웰에서 불투명 층으로 보이도록 플레이트를 육안으로 검사합니다. - 열 밀봉 기계에서 알루미늄 호일 덮개로 플레이트를 5°C에서 180초 동안 밀봉합니다.
알림: 모든 웰이 밀봉되어 있는지 육안으로 검사하십시오. 그렇지 않은 경우 180°C에서 5초를 더 반복합니다. 밀봉된 플레이트는 열 순환기에서 증폭을 dd_PCR하기 전에 4°C에서 최대 4시간 동안 보관할 수 있습니다.
5. dd_PCR 증폭
참고: 열 순환기는 PCR 증폭과 pre-PCR 활동을 공간적으로 분리하고 amplicon 오염으로 인한 위양성 결과를 방지하기 위해 pre-PCR 룸과 별도의 방에 배치해야 합니다.
- PCR 증폭을 위해 플레이트를 열 순환기(96-deep well reaction module 포함)에 넣고 단단히 닫습니다.
- 표 4에 설명된 조건과 40μL의 반응 부피에서 작동하도록 Thermocycler를 구성합니다. 프로그램 설정에 예열 단계를 포함하고 뚜껑이 99°C로 가열되었는지 확인합니다.
- PCR 프로그램이 끝나면 플레이트가 완전히 냉각되었는지 확인하기 위해 플레이트를 4°C에서 최소 15분 동안 유지하십시오.
알림: 플레이트는 물방울을 판독하기 전에 48°C에서 72-4시간 동안 보관할 수 있습니다.
6. 액적 판독
- 플레이트를 물방울 판독기에 넣고 시스템 소프트웨어에 다음 정보를 입력한 다음 판독을 계속합니다.
참고: 실험 유형 = 직접 정량화; 슈퍼믹스 = 프로브용 슈퍼믹스 dd_PCR개(dUTP 없음); 분석 유형 = 채널당 단일 타겟; 대상 정보, 신호 채널 1 = 사용된 프로브에 따른 FAM 채널 또는 HEX 채널. - 데이터 파일에서 각 샘플의 위치를 효과적으로 결정하고 식별할 수 있도록 샘플에 주석을 추가합니다.
7. 데이터 분석
- 액적 리더 제조업체에서 제공하는 호환 가능한 소프트웨어를 사용하여 데이터 분석을 수행합니다.
- 시스템 적합성 테스트를 수행하여 분석의 전반적인 성능(및 신뢰성)을 평가합니다.
- PCR 플레이트의 각 웰에 대해 측정된 이벤트 수(액적 수)를 확인합니다. 이상적으로 이벤트 수는 15,000에서 20,000 사이입니다. 이벤트 수가 <10,000인 경우 이 값을 분석에서 제외해야 합니다.
- 1D 또는 2D 진폭 플롯을 평가하고 물방울 비의 존재 여부를 확인합니다.
- 데이터 테이블 탭으로 이동하여 데이터를 Excel 파일로 내보냅니다.
- 아래 단계에 따라 vg 역가를 계산합니다.
- dd_PCR의 작동 범위는 10-10 000 copies/μL입니다. <10 copies/μL인 값은 분석에서 제외합니다. 여기에 사용된 dd_PCR 장비의 권장 범위는 25-5000 copies/μL입니다.
참고: 데이터 분석 소프트웨어는 PCR 반응 부피(40 μL)에 따라 벡터 게놈 번호를 제공하지만, 이는 시료 전처리 중에 적용되는 희석 인자를 고려하지 않습니다. - 다음 관계를 사용하여 시료 전처리 전반에 걸쳐 다양한 희석 인자를 고려하여 부피당 시료 vg 역가(일반적으로 vg/mL로 표시)를 계산합니다.
참고: 농도(데이터 분석에서 얻은 값) x 10(DNase I 전처리 희석) x 10(PCR 마스터 믹스 희석) x 1000(μL - mL) 선택된 시료 희석(AAV 희석 완충액 - 예: 표 2). - 반복실험 사이와 동일한 샘플의 서로 다른 희석액 간의 변동 계수(%CV)를 계산합니다.
참고: %CV는 표준 편차를 평균으로 나누어 계산합니다. 서로 다른 희석 사이의 %CV가 >20%인 경우 값이 정확한 것으로 간주되지 않아야 합니다. - 네거티브 및 포지티브 컨트롤의 값을 확인합니다. 네거티브 컨트롤은 5 copies/μL 미만이어야 합니다.
- dd_PCR의 작동 범위는 10-10 000 copies/μL입니다. <10 copies/μL인 값은 분석에서 제외합니다. 여기에 사용된 dd_PCR 장비의 권장 범위는 25-5000 copies/μL입니다.
결과
적절한 소프트웨어를 사용하여 결과를 시각화할 수 있습니다. 각 웰에 대해 1D 진폭 플롯은 해당 진폭을 기준으로 모든 물방울을 표시합니다. 양성 및 음성 비말 간의 명확한 구분이 예상됩니다. 전체 물방울의 10% 이상이 양수와 음의 물방울 구름 사이에 있는 경우(물방울 비라고 하는 현상) 샘플을 다시 측정해야 합니다( 그림 1 참조). 물방울 비에 대한 추가 정보는 토론에서 확인할 수 있습니다.
데이터 테이블을 생성하여 시료 이름, 허용된 액적 수(이벤트 수) 및 농도(copies/μL)를 포함하여 기록된 모든 정보를 요약할 수 있습니다. 이상적으로 이벤트 수는 15,000개에서 20,000개 사이의 허용되는 방울 사이여야 합니다. 우물에 대한 이벤트 수가 10,000 미만인 경우 데이터 점을 분석에서 제외해야 합니다. 출력 데이터의 예는 표 5에 나와 있습니다.
vg titer는 시료 희석을 고려하여 농도(copies/μL)를 기준으로 계산할 수 있습니다. dd_PCR의 작동 범위는 10-10,000 copies/μL이며, 10 copies/μL 미만의 값은 분석에서 제외해야 합니다. 샘플에 대한 vg 역가와 양성 및 음성 대조군을 계산해야 합니다. 포지티브 대조군의 측정값은 이론적 값에 비해 변동 계수(%CV)가 20% 미만이어야 합니다. 음성 대조군은 5 copies/μL 미만이어야 합니다. 또한 각 샘플에 대한 반복과 다른 희석 사이의 %CV를 계산해야 합니다. 서로 다른 희석 사이의 %CV가 20%를 초과하면 값이 부정확한 것으로 간주될 수 있으며 샘플을 다시 측정해야 할 수 있습니다.
성공적인 측정은 양성 및 음극 액적 구름의 명확한 분리, 최소 10,000개의 허용 액적, 10 copies/μL 및 10,000 copies/μL 사이의 값, 20% 미만의 반복 간 %CV를 특징으로 합니다.
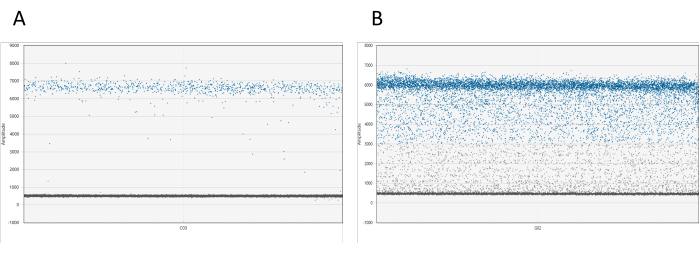
그림 1: dd_PCR 방울의 시각화. (A) 1D 진폭 플롯은 양극과 음극 방울의 명확한 분리를 보여주며, 이는 성공적인 입자 분할을 나타냅니다. (B) 플롯은 물방울 비로 알려진 양수와 음의 물방울이 제대로 분리되지 않은 것을 보여주며, 이는 최적화되지 않은 분할 또는 분석의 잠재적인 문제를 시사합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
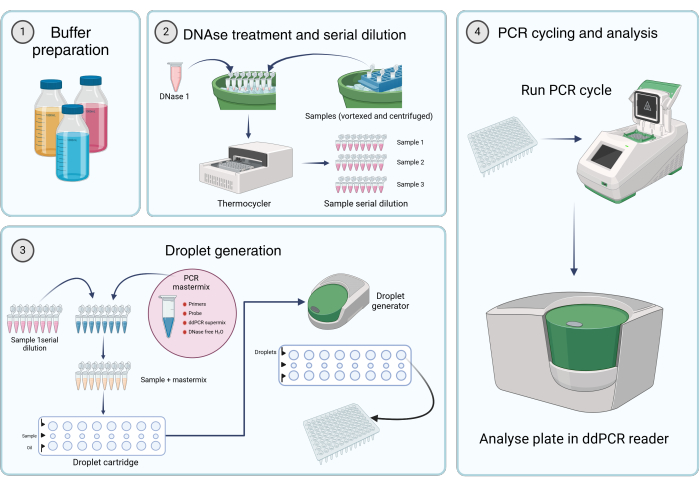
그림 2: 디지털 액적 PCR을 사용하여 바이러스 게놈을 정량화하기 위한 워크플로우. (1) 완충액, 시약 및 용액은 제조업체의 지침 또는 프로토콜의 1단계(스톡 용액 준비)에 따라 준비됩니다. (2) 45 μL의 DNase I 용액을 8-튜브 PCR 스트립의 각 튜브에 분주합니다. 샘플을 볼텍싱하고 간략하게 원심분리한 후 샘플의 5μL를 튜브 중 하나에 추가합니다. DNase I 및 시료 함유 8-튜브 PCR 스트립을 37°C에서 1시간 동안 열순환기에서 배양한 후 시료를 연속 희석합니다. (3) PCR 마스터믹스는 설명된 대로 제조하고, 19.8μL를 8-튜브 PCR 스트립의 각 튜브에 분주합니다. 2단계의 샘플 연속 희석액을 마스터믹스에 첨가합니다. 20 μL의 마스터믹스 플러스
샘플 용액을 카트리지의 중간 줄에 로드하고 60μL의 액적 생성 오일을 액적 생성 카트리지의 하부 웰로 옮깁니다. 그런 다음 카트리지를 액적 발생기에 넣고 지정된 조건에 따라 실행합니다. 액적 생성 후 카트리지의 맨 윗줄에서 42.5μL를 멀티웰 PCR 플레이트로 옮깁니다. (4) PCR 플레이트를 PCR thermocycler에 로드하고 제공된 조건에 따라 실행합니다. 플레이트는 dd_PCR 판독기를 사용하여 분석됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
표 1: 정밀도 데이터. 이 표는 각각 5가지 농도(QC1-QC5)를 가진 4개의 품질 관리(QC) 샘플 세트의 정밀 데이터를 제공합니다. 각 QC는 5 번 측정되었습니다. 반복성(A)을 나타내는 서로 다른 런 간의 변동 계수(%CV)와 중간 정밀도(B)를 나타내는 서로 다른 샘플 세트 간의 변동 계수(%CV)가 표시됩니다. 이 표를 다운로드하려면 여기를 클릭하십시오.
표 2: 샘플 희석. 표는 예상되는 바이러스 게놈 역가(vg/mL)를 기반으로 권장되는 샘플 희석을 보여줍니다. 최소 두 개의 값이 분석의 작업 범위 내에 포함되도록 하기 위해 총 3개의 희석이 권장됩니다. 이 표를 다운로드하려면 여기를 클릭하십시오.
표 3: dd_PCR 마스터 믹스 구성. 이 표는 정방향 및 역방향 프라이머(909nM), 프로브(227nM) 및 프로브용 슈퍼믹스(dUTP 제외, 1x)를 포함하는 dd_PCR 마스터 믹스dd_PCR 구성을 간략하게 보여줍니다. 이 표를 다운로드하려면 여기를 클릭하십시오.
표 4: 열순환 조건. 표는 권장되는 PCR 프로그램에 대해 자세히 설명하고 있습니다: (1) 캡시드 파괴 및 효소 활성화를 위한 95°C에서 10분 배양, (2) DNA 변성을 위한 94°C에서 30초의 40 사이클, 어닐링 및 확장을 위한 60°C에서 1분, (3) 효소 비활성화를 위한 98°C에서 10분 배양, (4) 4 °C에서 유지. 어닐링 온도는 사용된 프라이머/프로브 세트에 따라 최적화가 필요할 수 있습니다. 이 표를 다운로드하려면 여기를 클릭하십시오.
표 5: 실제 AAV 실행의 출력 데이터 예. 이 표는 실제 AAV 실행의 출력 데이터 예를 제공하며, 여기서 하나의 샘플은 각각 중복된 두 개의 서로 다른 희석으로 측정되었습니다. 바이러스 게놈(vg) 역가(vg/mL)는 10(DNase I 전처리) x 10(마스터 믹스에서 희석) x 1,000(μL to mL) x AAV 희석 완충액에서 희석 공식을 사용하여 계산됩니다. STDEV는 표준 편차를 나타내고 CV는 변동 계수를 나타냅니다. 이 표를 다운로드하려면 여기를 클릭하십시오.
토론
벡터 제제에서 AAV 벡터 게놈 사본의 정확한 정량화는 AAV 기반 유전자 치료제 개발에 필수적입니다. vg 역가를 측정하기 위한 여러 가지 방법이 존재하며, 정량적 PCR(qPCR)과 dd_PCR는 가장 널리 사용되고 허용되는 두 가지 기술입니다. dd_PCR는 증폭 효율로부터의 독립성, 더 높은 정밀도 및 더 큰 견고성으로 인해 qPCR보다 선호되는 경우가 많습니다15. dd_PCR에 의한 벡터 게놈 적정을 위한 수많은 프로토콜이 문헌에서 사용 가능하며, 각 프로토콜에는 고유한 샘플 준비 방법 15,16,17이 있습니다. 그러나 자격을 갖춘 합의 프로토콜이 부족합니다. 이 기사에서는 정제된 벡터 샘플에서 AAV 벡터 게놈을 정량화하기 위한 검증된 목적에 맞는 dd_PCR 프로토콜을 제시합니다(그림 2).
이 프로토콜을 수행할 때 시료 처리에 세심한 주의를 기울이는 것이 중요합니다. 교차 오염은 vg 역가를 정확하게 평가하는 데 상당한 어려움을 초래할 수 있습니다. 따라서 샘플은 외부 DNA에 의한 오염을 피하기 위해 PCR 워크스테이션에서 처리하는 것이 가장 좋습니다. 또한 DNase 배양의 적절한 실행은 캡시드를 방해하지 않고 잠재적으로 패키징된 벡터 게놈을 분해하지 않고 패키징되지 않은 오염 물질 DNA를 제거하는 데 중요합니다. 많은 프로토콜에는 열 불활화 및 proteinase K 처리 16,17,18이 포함됩니다. 그러나 자체 분석법 개발 과정에서 과도한 가열은 vg 적정에 해롭고 proteinase K 처리가 불필요하다는 사실이 밝혀졌습니다(데이터 표시 안 됨).
바이러스 게놈을 정량화하는 데 dd_PCR 인기가 높아짐에 따라 제조업체는 dd_PCR 분석 설계 및 최적화에 대한 지침이 포함된 자세한 응용 분야 가이드를 발행했습니다19. 문제 해결 정보도 사용할 수 있습니다. 실제로, 분석이 올바르게 설계되었을 때 가장 빈번한 문제에는 비방울 비와 낮은 액적 수가 포함됩니다. 물방울 비는 앰플리콘에 대한 프라이머/프로브의 접근성이 좋지 않아 발생하는 경우가 많지만 몇 가지 전략으로 이 문제를 해결할 수 있습니다. 예를 들어, 온도 구배 실험은 최적의 어닐링 온도를 설정하는 데 도움이 될 수 있습니다. 또한 DNA 양을 낮추기 위해 샘플을 희석하거나(66ng 미만 권장) 앰플리콘 영역 외부를 절단하는 특정 효소로 제한 분해를 수행하면 템플릿 접근성을 개선할 수 있습니다. DNA μg당 10U의 제한 효소를 권장하는 것이 일반적으로 효과적입니다. 낮은 액적 수(10,000개 미만) 문제는 카트리지의 샘플과 오일의 피펫팅 불량으로 인해 발생하는 경우가 많습니다. 액적의 전단을 방지하기 위해 적절한 팁을 사용하여 천천히 피펫팅하는 것이 좋습니다. dd_PCR 분석은 강력하지만 한 가지 제한 사항은 결과 도출 시간이 길다는 것입니다. 시료 처리에서 액적 판독까지의 과정은 몇 시간이 소요되며, 이는 결과가 빠르게 필요한 경우 단점이 될 수 있습니다.
벡터 제제에서 AAV 게놈 사본의 정확한 정량화는 AAV 유전자 치료 수명 주기의 모든 단계에서 필요합니다. 특히, 생산 및 정제 공정을 최적화하고, 전임상 연구를 수행하고, AAV 기반 유전자 치료 제품에 대한 임상 투여량을 결정하는 데 중요합니다. 여기에 제시된 dd_PCR 프로토콜은 광범위하게 적용할 수 있으며 다양한 혈청형 및 전이유전자를 가진 정제된 AAV 제품에 사용할 수 있습니다.
공개
IVH, BM 및 EH는 Tavira Therapeutics의 공동 설립자입니다. 다른 저자는 이해 상충이 없다고 선언합니다.
감사의 말
이 연구는 '유럽 회복 및 회복력 시설'(RRF)(VV021/13)에서 시작된 플랑드르 정부의 '플랑드르 회복력(Flanders Resilience)' 보조금으로 가능했습니다. 그림 2 는 Biorender.com 로 작성되었습니다.
자료
| Name | Company | Catalog Number | Comments |
| 8-channel pipette 10 µL | Eppendorf | 3,12,50,00,010 | |
| 8-channel pipette 200 µL | Eppendorf | 3,12,50,00,036 | |
| 8-channel pipette 300 µL | Eppendorf | 3,12,50,00,052 | |
| 8-well PCR strip | Sarstedt | 72.991.002 | |
| Bovine Serum Albumin (BSA) | Sigma-Aldrich | A9418 | |
| C1000 Touch Thermal Cycler with 96–Deep Well Reaction Module | Bio-Rad | 1851197 | |
| ddPCR Buffer Control for Probes 9 mL (2 x 4.5 mL) | Bio-Rad | 1863052 | |
| ddPCR Supermix for Probes (No dUTP) (2 x 1 mL) | Bio-Rad | 1863023 | |
| ddPCR 96-Well Plates (pkg of 25) | Bio-Rad | 12001925 | |
| ddPCR Droplet Reader Oil (2 x 1L) | Bio-Rad | 1863004 | |
| DG8 Cartridge Holder | Bio-Rad | 1863051 | |
| DG8 Cartridges for QX200/QX100 (pkg of 24) | Bio-Rad | 1864008 | |
| DG8 Gaskets for QX200/QX100 (pkg of 24) | Bio-Rad | 1863009 | |
| DNase I (10U/µL) + buffer | Roche | 4716728001 | |
| Droplet Generation Oil for Probes (10 x 7 mL) | Bio-Rad | 1863005 | |
| Eppendorf ep Dualfilter T.I.P.S. Filter Tip, 0.1-10 μL, 34 mm, Rack, PCR Clean, STERILE | Eppendorf | 30078500 | |
| Eppendorf ep Dualfilter T.I.P.S. Filter Tip, 20-300 μL, 55 mm, Rack, PCR Clean, STERILE | Eppendorf | 30078560 | |
| Eppendorf ep Dualfilter T.I.P.S. Filter Tip, 2-100 μL, 53 mm, Rack, PCR Clean, STERILE | Eppendorf | 30078543 | |
| Forward lyophilized primers and respective master stocks at 100 mM | IDT | GFP as target sequence. Forward primer: 5'-GAACGGCATCAAGGTGAACT-3' | |
| Lyopohilized probe and master stock at 100 µM | IDT | GFP as target sequence. PrimeTime Eco Probe: /56-FAM/CAAGATCCG/ZEN/CCACAACATCGAGGA/3IABkFQ/ | |
| Magnesium Chloride (MgCl2) | Chemlab Analytical | CL00.1381 | |
| Nuclease free water | IDT | 11-04-02-01 | |
| PCR Plate Heat Seal, foil, pierceable (pkg of 100) | Bio-Rad | 1814040 | |
| Pluronic F-68 non-ionic surfactant (100x) | Thermo Fisher Scientific | 24040032 | |
| Potassium Chloride (KCl) | Honeywell research chemicals | 31248 | |
| QX manager software | Bio-Rad | Software to analyse ddPCR data | |
| QX200 Droplet Generator | Bio-Rad | 1864002 | |
| QX200 Droplet Reader | Bio-Rad | 1864003 | |
| Reagent reservoir | VWR | 613-1181 | |
| Reverse lyophilized primers and respective master stocks at 100 mM | IDT | GFP as target sequence. Reverse primer: 5'-TGCTCAGGTAGTGGTTGTCG-3' | |
| SafeSeal reaction tube, 1.5 mL | Sarstedt | 72.706.200 | |
| Salmon Sperm DNA, sheared (10 mg/mL) | Thermo Fisher Scientific | AM9680 | |
| TE buffer | IDT | Accompanied by primers when ordering | |
| Tris hydrochloride (Tris-HCl) | Roche | 10812846001 |
참고문헌
- Balakrishnan, B., Jayandharan, G. R. Basic biology of adeno-associated virus (AAV) vectors used in gene therapy. Curr Gene Ther. 14 (2), 86-100 (2014).
- Srivastava, A. In vivo tissue-tropism of adeno-associated viral vectors. Curr Opin Virol. 21, 75-80 (2016).
- Pupo, A., et al. AAV vectors: The Rubik's cube of human gene therapy. Mol Ther. 30 (12), 3515-3541 (2022).
- Gimpel, A. L., et al. Analytical methods for process and product characterization of recombinant adeno-associated virus-based gene therapies. Mol Ther Methods Clin Dev. 20, 740-754 (2021).
- Brimble, M. A., et al. Preventing packaging of translatable P5-associated DNA contaminants in recombinant AAV vector preps. Mol Ther Methods Clin Dev. 24, 280-291 (2022).
- Wright, J. F. Product-related impurities in clinical-grade recombinant AAV vectors: Characterization and risk assessment. Biomedicines. 2 (1), 80-97 (2014).
- Dobnik, D., et al. Accurate quantification and characterization of adeno-associated viral vectors. Front Microbiol. 10, (2019).
- Sidransky, D., et al. Identification of ras oncogene mutations in the stool of patients with curable colorectal tumors. Science. 256 (5053), 102-105 (1992).
- Vogelstein, B., Kinzler, K. W. Digital PCR. Proc Natl Acad Sci USA. 96 (16), 9236-9241 (1999).
- Hindson, B. J., et al. High-throughput droplet digital PCR system for absolute quantitation of DNA copy number. Anal Chem. 83 (22), 8604-8610 (2011).
- Pinheiro, L. B., et al. Evaluation of a droplet digital polymerase chain reaction format for DNA copy number quantification. Anal Chem. 84 (2), 1003-1011 (2012).
- Sanmiguel, J., Gao, G., Vandenberghe, L. H. Quantitative and digital droplet-based AAV genome titration. Methods Mol Biol. 1950, 51-83 (2019).
- Bio-Rad Laboratories Inc. Planning droplet digital PCR experiments. , https://www.bio-rad.com/en-in/life-science/learning-center/introduction-to-digital-pcr/planning-ddpcr-experiments (2024).
- Dorange, F., Le Bec, C. Analytical approaches to characterize AAV vector production & purification: Advances and challenges. Cell Gene Ther Insights. 4 (2), 119-129 (2018).
- Lock, M., et al. Absolute determination of single-stranded and self-complementary adeno-associated viral vector genome titers by droplet digital PCR. Hum Gene Ther Methods. 25 (2), 115-125 (2014).
- Prantner, A., Maar, D. Genome concentration, characterization, and integrity analysis of recombinant adeno-associated viral vectors using droplet digital PCR. PLoS One. 18 (1), e0280242(2023).
- Dobnik, D., et al. Accurate quantification and characterization of adeno-associated viral vectors. Front Microbiol. 10, 1570(2019).
- Suoranta, T., Laham-Karam, N., Yla-Herttuala, S. Optimized protocol for accurate titration of adeno-associated virus vectors. Hum Gene Ther. 32 (19-20), 1270-1279 (2021).
- Bio-Rad. Droplet digital PCR applications guide. Bulletin 6407. , https://www.bio-rad.com/webroot/web/pdf/lsr/literature/Bulletin_6407.pdf (2024).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유