Method Article
Ingénierie cellulaire perméable protéines
Dans cet article
Résumé
Transduction des protéines permet la livraison directe de protéines biologiquement actives dans les cellules. Contrairement aux méthodes conventionnelles telles que la transfection d'ADN ou de transduction virale ce paradigme non invasive permet une manipulation très efficace cellulaires de façon titrable contourner la toxicité cellulaire et le risque de transformation oncogénique par modification génétique permanente.
Résumé
La technique de transduction de protéines permet la livraison directe du matériel biologiquement active dans les cellules de mammifères [pour revue, voir 1,2]. Pour cela, on peut faire usage de la capacité translocation des peptides cellule dite pénétrante (CPP), également désigné comme domaines de transduction protéique (DAP). Le TAT-RPC dérivés du type de virus de l'immunodéficience humaine 1 (VIH-1) Tat (trans-activateur de la transcription) des protéines a été largement utilisé. Les chargés positivement TAT favorise la perméabilité des cellules permettant ainsi de surmonter les barrières de la membrane cellulaire par endocytose et / ou la pénétration de membrane directe 2. En combinaison avec un signal de localisation nucléaire (NLS) des protéines de fusion sont capables d'entrer dans le noyau de fonctionnalité exposantes. Notre vidéo de présentation démontre, comme une ampliation de l'ingénierie des protéines cellulaires perméables, la construction, la production et l'application d'une version de cellules-perméable de la CRE ADN modification enzymatique.
Cre est une recombinase site-spécifique qui est capable de reconnaître et de se recombiner 34 sites loxP dans paires de base des cellules de mammifères in vitro et in vivo. Par conséquent, le système Cre / loxP est largement utilisé pour induire des mutations conditionnellement dans le génome de 3,4 cellules vivantes. La livraison des actifs de la recombinase Cre dans les cellules, cependant, représente une limitation.
Nous décrivons le système vectoriel pSESAME, ce qui permet une insertion directe du gène d'intérêt et fournit une plateforme pour cloner rapidement les différents domaines et les tags utilisés dans le vecteur d'une manière commode et standardisés. Réorganisation des balises différentes a été montré pour modifier les propriétés biochimiques des protéines de fusion offrant une possibilité d'atteindre un rendement supérieur et une meilleure solubilité. Nous démontrons comment exprimer et de purifier les cellules recombinantes perméants protéines à l'intérieur et de E. coli. La fonctionnalité de la protéine recombinante Cre est finalement validé en culture cellulaire en évaluant son activité recombinase intracellulaire.
Protocole
Construction du vecteur d'expression et d'expression:
Le vecteur d'expression pSESAME-Cre a été construit en insérant un fragment de Cre-encodage dans pSESAME via des sites de restriction Avril et Nhel utilisant des méthodes de clonage standard. pSESAME code pour une protéine de fusion constituée d'une histidine-tag, TAT-domaine, séquence NLS et la CRE, en abrégé HTNCre. Pour l'expression de HTNCre pSESAME-Cre a été transformé en TUNER (DE3) Placi et utilisés pour préparer un stock de glycérol.
- Une culture durant la nuit a été inoculé avec une pointe de pipette enduits de bactéries transformées par le stock de glycérol. La culture du jour au lendemain se composait de milieu LB supplémenté en glucose 0,5% [v / v] et carbénicilline à une concentration finale de 50 pg / ml et a été autorisé à croître à 37 ° C pendant 16 heures.
- Le lendemain, le plus densément cultivée nuit de la culture a été utilisée pour inoculer la culture d'expression à un ratio de 1 à 40 et a été mis dans un incubateur à 37 ° C. Expression de la culture se composait des médias tuberculose complété par du glucose à 0,5% [v / v] et l'ampicilline à une concentration finale de 100 pg / mL.
- A une DO 595 de 1,5 à la culture d'expression a été induite avec 0,5 mM d'IPTG pendant 1 h.
- Par la suite les bactéries ont été recueillies par centrifugation à 5000 rpm pendant 10 minutes dans un rotor SLA3000.
- Les bactéries ont été granulés stockés à -20 ° C jusqu'à la purification.
Purification des cellules perméables protéines:
- Frozen granulés bactéries ont été remises en suspension dans 10 ml de tampon de lyse par culture flacon litre pendant 15 minutes à température ambiante.
- La suspension a été ensuite incubés avec 1 mg / ml de lysozyme pour d'autres 15 minutes tout en mélangeant à température ambiante.
- 25 U / mL a été ajouté par la suite benzonase et incubé tout en mélangeant pendant 15 minutes à température ambiante.
- Après sonication sur glace pendant 1,5 minutes avec des impulsions s 0,5 à 45% de la puissance, 1 ml de tampon froid sel tartrique (BST) par ml de suspension a été soigneusement ajouté tout en mélangeant et incubé pendant 5 min sur la glace. SDS-PAGE de la fraction de l'échantillon lysat (L) a été prise.
- Lysat clarifié a été obtenu par centrifugation à 4 ° C pendant 30 min à 30000 g. SDS-PAGE des échantillons de fractions solubles (S) et insoluble (I) ont été prises.
- Le surnageant a été transféré dans de nouveaux tubes de 50 ml Falcon et a ensuite été délicatement mélangés pendant 1 h à 4 ° C avec 2 ml de 50% de Ni-NTA boue par litre de culture d'expression initiale.
- La suspension a été emballé dans une colonne à écoulement par gravité EconoPac (SDS-PAGE de l'échantillon de l'écoulement à travers la fraction (FT) a été prise) et lavé deux fois avec 5 volumes de lit de tampon de lavage. SDS-PAGE des échantillons des deux fractions de lavage (W1 et W2) ont été collectés.
- HTNCre fractions contenant ont été élues avec 3 lits volumes de tampon d'élution et l'échantillon de la fraction d'éluat (E) pour analyse SDS-PAGE a été prise.
- Imidazole a été éliminée par dialyse contre le tampon d'élution fraction élevée de sel à deux reprises.
- La solution de protéine a été encore concentré par dialyse contre du tampon glycérol deux fois. Dans toutes les étapes de dialyse le ratio de tampon d'échantillon a été d'au moins 50. Cette procédure a abouti à une solution stock de glycérol contenant HTNCre à une concentration d'habitude entre 200 et 450 uM, soit 1 litre de culture d'expression se traduira par environ 12 mg de protéine. Exemple de stock glycérol (GS) pour analyse SDS-PAGE ont été recueillis. Solution stock HTNCre peuvent être stockés à moins 20 ° C.
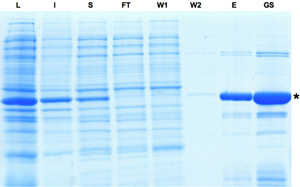
Figure 1: analyse SDS-PAGE des échantillons prélevés pendant le processus de purification de recombinase Cre. L'induction de l'expression de Cre est indiquée par la bande dominante de la fraction lysat. Bien qu'une partie de la protéine est insoluble la protéine Cre peut encore être enrichi comme on le voit dans l'éluat et les fractions d'actions de glycérol. L: lysat, I: Insoluble, S: surnageant, FT: Flow-through, W: linge, E: éluat, GS: Stock Gylcerol. S'il vous plaît cliquez ici pour voir une version agrandie de la figure 1.
Transduction des protéines dans murin souches embryonnaires (ES) cellules:
- Les cellules ES portant une conditionnelle β-galactosidase journaliste de 5 construire ont été ensemencées en utilisant des cellules individuelles TrypLE ™ Express pour la dissociation des cellules adhérentes. Après 4 à 6 heures, les cellules ont re-joint et le milieu a été enlevé.
- Par la suite les cellules ES ont été incubées avec un milieu contenant du HTNCre pendant 16 heures.
- Une quantité appropriée de protéines HTNCre (correspondant à 10 pM) sur le stock de glycérol a été dilué dans le milieu de ES et filtré par la suite stériles (0,22 m).
- Après moyennes de transduction de protéines a été modifié pour revenir à un milieu de croissance normal.
- Après deux jours cellules ont été lavées avec du PBS et fixées avec du paraformaldéhyde 4% (PFA) pendant 10 minutes.
- Deux suppléinternationale étapes de lavage avec du PBS ont été exécutés avant X-Gal coloration a été réalisée.
- Les cellules fixées ont été couverts d'une couche de solution de coloration X-Gal 6 et incubée pendant la nuit à 37 ° C.
Les résultats représentatifs:
Suivant une solution de coloration X-Gal jour a été aspiré et les cellules ont été recouvertes d'une couche de PBS pour l'analyse microscopique. 80 à 100% de cellules recombinées pu être observé dans les cellules ES murines jugés par β-galactosidase.
Discussion
Pendant le processus de purification de la protéine fusion Cre il est important de ne pas omettre l'ajout de la glace froide tampon TBS avant la centrifugation. Sinon recombinase Cre tend à précipiter dans la mémoire tampon glycérol.
Si la fraction de l'éluat semble devenir trouble dû à la forte concentration de protéine de fusion élution tampon supplémentaire doit être ajouté jusqu'à ce que la solution a autorisé à nouveau.
L'application de 10 uM de la protéine fusion Cre entraîne habituellement une efficacité de recombinaison de 80 à 100%. Sérum de veau foetal (FCS) étant une composante importante du milieu de cellules ES inhibe fortement la transduction des protéines. Par conséquent une forte concentration de la recombinase Cre a dû être utilisé. Lorsque vous travaillez dans un milieu sans sérum moins de protéines (0,5 - 2 M) peut être utilisé pour réaliser des économies recombinaison similaires.
Avec le système vectoriel pSESAME à portée de main, on peut appliquer la technique de transduction protéique à d'autres protéines, y compris les facteurs de transcription tels que Oct4 et Sox2 7 et 8 Scl/Tal1.
Remerciements
Nous remercions Olivier Brüstle et tous les membres du groupe d'ingénierie de cellules souches de l'Université de Bonn, de soutien et de discussions fructueuses. Nous remercions Sabine Schenk pour la préparation du SDS-PAGE et le soutien continu tout au long du projet. Nicole Russ et Magerhans Anna fourni un excellent soutien technique. Par ailleurs, nous tenons à remercier Andreas Bär et Mertens Sheila pour la production du film. Ce travail a été soutenu par des subventions de la Fondation Volkswagen (Az I/77864) et le ministère allemand de l'Education et la Recherche (BMBF, 01 GN 0813).
matériels
| Name | Company | Catalog Number | Comments |
| TUNER (DE3) pLacI | Novagen, EMD Millipore | 70625 | |
| Glycerol | Carl Roth GmbH | 3783.2 | |
| Na2HPO4 | Carl Roth Gmbh | T876.1 | |
| Trizma Base | Sigma-Aldrich | T1503 | |
| HCl | Carl Roth Gmbh | 4625.1 | |
| Imidazol | Carl Roth Gmbh | X998.4 | |
| NaCl | Carl Roth Gmbh | 9265.2 | |
| Yeast Extract | Carl Roth Gmbh | 2363.4 | |
| Trypton/Pepton | Carl Roth Gmbh | 8952.4 | |
| K2HPO4 | Carl Roth Gmbh | P749.2 | |
| KH2PO4 | Carl Roth Gmbh | 3904.1 | |
| Ampicillin | Sigma-Aldrich | A9518 | |
| Carbenicillin | Sigma-Aldrich | 6344.2 | |
| HEPES | Sigma-Aldrich | H3375 | |
| Lysozyme | Sigma-Aldrich | 62971 | |
| Benzonase | Novagen, EMD Millipore | ||
| L-Tartaric acid, disodium salt | Sigma-Aldrich | ||
| 50% Ni-NTA slurry | Invitrogen | R901-15 | |
| EconoPac columns | Bio-Rad | 732-1010 | |
| Sterile filter 0,22μm | Whatman, GE Healthcare | ||
| Paraformaldehyde (PFA) | Sigma-Aldrich | ||
| LB medium | Yeast extract, Trypton/Pepton, NaCl | ||
| TB medium | Yeast extract, Trypton/Pepton, Glycerol, K2HPO4, KH2PO4 | ||
| Lysis Buffer | 50 mM Na2HPO4, 5 mM Tris, pH 7.8 | ||
| Tartaric Salt Buffer (TSB) | PTB containing 2 M L-Tartaric acid, disodium salt, and 20 mM Imidazol | ||
| Washing Buffer | PTB, 500 mM NaCl, 15 mM Imidazol | ||
| Elution Buffer | PTB, 500 mM NaCl, 250 mM Imidazol | ||
| High Salt Buffer | 600 mM NaCl, 20 mM HEPES, pH 7.4 | ||
| Gylcerol Buffer | 50% glycerol, 500 mM NaCl, 20 mM HEPES, pH 7.4 | ||
| TrypLE™ Express | Invitrogen | ||
| ESGRO (LIF) | EMD Millipore | ||
| NEAA | GIBCO, by Life Technologies | 11140035 | |
| L-Glutamin | GIBCO, by Life Technologies | 25030024 | |
| β-Mercapt–thanol | GIBCO, by Life Technologies | 31350010 | |
| DMEM | GIBCO, by Life Technologies | 11960044 | |
| PBS | GIBCO, by Life Technologies | ||
| Fetal Calf Serum (FCS) | PAA Laboratories | ||
| X-Gal staining solution: | 4 mM K3(FeIII(CN)6), 4 mM K4(FeII(CN)6), 2mM MgCl2 0.4 mg/mL X-Gal solved in PBS | ||
| K3(FeIII(CN)6) | Sigma-Aldrich | P-3367 | |
| K4 (FeII(CN)6) | Sigma-Aldrich | P-9387 | |
| MgCl2 | Sigma-Aldrich | M8266 | |
| X-Gal | Sigma-Aldrich | B4252 |
Références
- Gump, J. M., Dowdy, S. F. TAT transduction: the molecular mechanism and therapeutic prospects. Trends Mol Med. 13 (10), 443-448 (2007).
- Edenhofer, F. Protein transduction revisited: novel insights into the mechanism underlying intracellular delivery of proteins. Curr Pharm Des. 14 (34), 3628-3636 (2008).
- Branda, C. S., Dymecki, S. M. Talking about a revolution: The impact of site-specific recombinases on genetic analyses in mice. Dev Cell. 6 (1), 7-28 (2004).
- Nolden, L. Site-specific recombination in human embryonic stem cells induced by cell-permeant Cre recombinase. Nat Methods. 3 (6), 461-467 (2006).
- Zhang, Y. Inducible site-directed recombination in mouse embryonic stem cells. Nucleic Acids Res. 24 (4), 543-548 (1996).
- Peitz, M. Enhanced purification of cell-permeant Cre and germline transmission after transduction into mouse embryonic stem cells. Genesis. 45 (8), 508-517 (2007).
- Bosnali, M., Edenhofer, F. Generation of transducible versions of transcription factors Oct4 and Sox2. Biol Chem. 389 (7), 851-861 (2008).
- Landry, J. R. Runx genes are direct targets of Scl/Tal1 in the yolk sac and fetal liver. Blood. 111 (6), 3005-3014 (2008).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon