Method Article
Engineering zellpermeablen Protein
In diesem Artikel
Zusammenfassung
Protein Transduktion ermöglicht die direkte Abgabe von biologisch aktiven Proteinen in Zellen. Im Gegensatz zu herkömmlichen Methoden wie DNA-Transfektion oder virale Transduktion dieser nicht-invasiven Paradigma ermöglicht hoch effiziente zelluläre Manipulation in eine titrierbare Weise umgehen zelluläre Toxizität und das Risiko der onkogenen Transformation durch permanente genetische Veränderung.
Zusammenfassung
Das Protein Transduktion Technik ermöglicht die direkte Abgabe von biologisch aktiven Materials in Säugerzellen [zur Übersicht siehe 1,2]. Für diese Verwendung des translozierenden Fähigkeit der sogenannten Zell-penetrierende Peptide (CPP) machen kann, auch als Proteintransduktionsdomänen (PTDs) bezeichnet. Das TAT-CPP aus dem humanen Immundefizienz-Virus Typ 1 (HIV-1) Tat (trans-Aktivator der Transkription)-Protein wurde in großem Umfang eingesetzt abgeleitet. Die positiv geladenen TAT fördert Zellpermeabilität damit die Überwindung der Barrieren der Zellmembran durch Endozytose und / oder direkte Membranpenetration 2. In Kombination mit einem Kernlokalisierungssignal (NLS) Fusionsproteine sind in der Lage, den Kern aufweisen Funktionalität geben. Unsere Video-Präsentation demonstriert, wie eine Veranschaulichung für das Engineering von zellpermeablen Proteine, die Konstruktion, Produktion und Anwendung eines zellpermeablen Version des DNA-modifizierende Enzyme Cre.
Cre ist eine ortsspezifische Rekombinase, die in der Lage zu erkennen und zu rekombinieren 34 Basenpaar loxP-Stellen in Säugetierzellen in vitro und in vivo ist. Deshalb ist die Cre / loxP-System ist weit verbreitet bedingt induzieren Mutationen im Genom lebender Zellen 3,4. Die Lieferung von aktiven Cre-Rekombinase zu Zellen, stellt jedoch eine Einschränkung.
Wir beschreiben die pSESAME Vektor-System, das eine direkte Insertion des Gens-of-Interest können und bietet eine Plattform, um schnell Klon unterschiedlichen Domänen und-Tags innerhalb des Vektors in eine komfortable und standardisierte Art und Weise verwendet. Umstellen der verschiedenen tags wurde gezeigt, dass die biochemischen Eigenschaften der Fusionsproteine bietet die Möglichkeit, höhere Ausbeute und bessere Löslichkeit zu erreichen ändern. Wir zeigen, wie zu äußern und zu reinigen rekombinanten Zell-Permeationsmittel Proteine in und aus E. coli. Die Funktionalität des rekombinanten Proteins ist Cre schließlich in der Zellkultur durch die Bewertung ihrer intrazellulären Rekombinase-Aktivität geprüft.
Protokoll
Bau von Expressionsvektor und Ausdruck:
Die pSESAME-Cre Expressionsvektor wurde durch Einfügen eines Cre-encoding-Fragment in pSESAME über AvrII und NheI-Restriktionsstellen mit Standard-Klonierungsverfahren konstruiert. pSESAME kodiert für ein Fusionsprotein, bestehend aus einem Histidin-Tag, TAT-Domäne, NLS-Sequenz und Cre, abgekürzt HTNCre. Für die Expression von HTNCre die pSESAME-Cre wurde in TUNER (DE3) Placi umgewandelt und verwendet, um eine Glycerin Lager vorzubereiten.
- Eine Über-Nacht-Kultur beimpft mit einer Pipettenspitze mit transformierten Bakterien aus dem Glycerin Lager beschichtet. Die Über-Nacht-Kultur bestand aus LB-Medium mit 0,5% Glucose [v / v] und Carbenicillin in einer Endkonzentration von 50 ug / mL und durfte bei 37 ° C wachsen für 16 Stunden.
- Am nächsten Tag dicht gewachsen über Nacht Kultur wurde verwendet, um den Ausdruck Kultur bei einem Verhältnis von 1 bis 40 zu impfen und wurde in einem Brutschrank bei 37 ° C stellen Expression Kultur bestand aus TB-Medien mit 0,5% Glucose [v / v] und Ampicillin in einer Endkonzentration von 100 ug / ml ergänzt.
- Bei einer OD 595 von 1.5 die Expression Kultur wurde mit 0,5 mM IPTG für 1 h induzierten
- Anschließend wurden die Bakterien durch Zentrifugation bei 5000 rpm für 10 Minuten in einem SLA3000 Rotor gesammelt.
- Bakterien-Pellets wurden bei minus 20 ° C bis zur Reinigung gespeichert.
Reinigung von zellpermeablen Protein:
- Gefrorene Bakterien-Pellets wurden in 10 mL Lysispuffer pro Liter Flasche Kultur für 15 Minuten bei Raumtemperatur resuspendiert.
- Suspension wurde dann mit 1 mg / ml Lysozym für weitere 15 Minuten inkubiert beim Mischen bei Raumtemperatur.
- 25 U / ml Benzonase wurde später und fügte hinzu, inkubiert beim Mischen für 15 Minuten bei Raumtemperatur.
- Nach Ultraschallbehandlung auf Eis für 1,5 min mit 0,5 s Impulse bei 45% der Leistung, 1 ml kaltem Wein-Salz-Puffer (TSB) pro ml Suspension wurde sorgfältig während zugegeben, die Vermischung und inkubiert für 5 min auf Eis. SDS-PAGE-Probenpuffer Lysat Fraktion (L) aufgenommen wurde.
- Cleared Lysat wurde durch Zentrifugation bei 4 ° C für 30 min bei 30.000 g erhalten SDS-PAGE-Proben von löslichen (S) und unlöslichen Fraktionen (I) aufgenommen wurden.
- Der Überstand wurde in frische 50 ml-Falcon-Röhrchen überführt und wurde dann vorsichtig für 1 h bei 4 ° C gemischt mit 2 ml 50% Ni-NTA Gülle pro Liter ersten Ausdruck Kultur.
- Die Suspension wurde in ein Fließ EconoPac Spalte (SDS-PAGE Stichprobe von Flow-Through-Fraktion (FT) aufgenommen wurde) verpackt und zweimal mit 5-Bett-Waschpuffer. SDS-PAGE-Proben der beiden Waschfraktionen (W1 & W2) gesammelt wurden.
- HTNCre-haltigen Fraktionen wurden mit 3-Bett-Elutionspuffer und Probe Eluatfraktion (E) für die SDS-PAGE-Analyse eluiert wurde.
- Imidazol wurde durch Dialyse Elutionsfraktion gegen hohe Salz-Puffer zweimal entfernt.
- Die Protein-Lösung wurde durch Dialyse gegen Glycerin-Puffer zweimal konzentriert. In aller Dialysepatienten Schritte das Verhältnis von Puffer zu Probe wurde mindestens 50. Dieses Verfahren führte zu einem Glycerin-Stammlösung enthält HTNCre in einer üblichen Konzentration zwischen 200 und 450 uM, dh 1 Liter Ausdruck Kultur in ~ 12 mg des Proteins führen. Beispiel Glycerin Lager (GS) für die SDS-PAGE-Analyse gesammelt. HTNCre Stammlösung kann bei minus 20 ° C gelagert werden
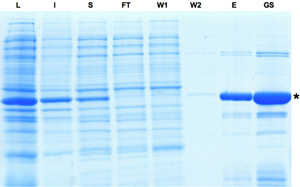
Abbildung 1: SDS-PAGE-Analyse der Proben während des Reinigungsprozesses der Rekombinase Cre gesammelt. Induktion der Cre Expression wird durch dominante Bande in Lysat Bruchteil angezeigt. Obwohl ein Teil des Proteins ist unlöslich das Cre-Protein kann weiter angereichert werden, wie in Eluat und Glycerin Lager Fraktionen gesehen. L: Lysate, I: Unlöslich, S: Überstand, FT: Flow-through, W: Waschen, E: Eluat, GS: Gylcerol Lager. Bitte klicken Sie hier, um eine größere Version der Abbildung 1 zu sehen.
Protein Transduktion in murine embryonale Stammzellen (ES-Zellen):
- ES-Zellen, die eine bedingte β-Galactosidase-Reporter 5. Konstruieren Sie wurden als einzelne Zellen mit Hilfe TrypLE ™ Express für die Dissoziation von anhaftenden Zellen ausgesät. Nach 4 bis 6 Stunden wurden die Zellen hatten wieder angebracht und das Medium wurde entfernt.
- Anschließend ES-Zellen wurden mit HTNCre-haltigem Medium für 16 Stunden inkubiert.
- Eine entsprechende Menge HTNCre Protein (entspricht 10 nM) aus dem Glycerin Lager wurde in ES-Medium und anschließend sterilfiltriert (0,22 um) verdünnt.
- Nach Proteintransduktionsdomäne Medium wurde wieder auf das normale Wachstum Medium gewechselt.
- Nach zwei Tagen wurden die Zellen mit PBS gewaschen und mit 4% Paraformaldehyd (PFA) für 10 Minuten.
- Zwei zusätzlichezusätzliche Waschschritte mit PBS wurden hingerichtet, bevor X-Gal-Färbung durchgeführt wurde.
- Fixierten Zellen wurden mit einer Schicht von X-Gal Färbelösung 6 abgedeckt und inkubiert über Nacht bei 37 ° C.
Repräsentative Ergebnisse:
Am nächsten Tag X-Gal Färbelösung wurde angestrebt und die Zellen wurden mit einer Schicht von PBS für die Mikroskopie Analyse abgedeckt. 80 bis 100% der rekombinierten Zellen konnten in den murinen ES-Zellen durch β-Galactosidase-Aktivität gemessen beobachtet werden.
Diskussion
Während des Reinigungsprozesses des Cre-Fusionsprotein ist es wichtig, nicht auf die Zugabe von eiskaltem TBS-Puffer vor dem Zentrifugieren weglassen. Ansonsten Rekombinase Cre tendenziell Niederschlag in der Glycerin-Puffer.
Wenn die Eluatfraktion scheint trüb aufgrund der hohen Konzentration des Fusionsproteins zusätzliche Elutionspuffer sollte hinzugefügt werden, bis die Lösung wieder gelöscht hat.
Die Anwendung von 10 pM Cre-Fusionsprotein führt in der Regel eine Rekombination Effizienz von 80 bis 100%. Fötalem Kälberserum (FCS), ein wichtiger Bestandteil der ES-Zell-Medium stark hemmt Proteintransduktionsdomäne. Deshalb hohe Konzentration von Cre-Rekombinase eingesetzt werden mussten. Bei der Arbeit in serum-freien Bedingungen weniger Protein (0,5 bis 2 M) kann verwendet werden, um ähnliche Rekombination Wirkungsgrade zu erreichen.
Mit dem pSESAME Vektor-System zur Hand kann man die Technik der Proteintransduktionsdomäne an andere Proteine einschließlich Transkriptionsfaktoren wie Oct4 und Sox2 7 und 8 gelten Scl/Tal1.
Danksagungen
Wir danken Oliver Brüstle und alle Mitglieder des Stem Cell Engineering Group, Universität Bonn, für die Unterstützung und wertvolle Diskussionen. Wir bedanken uns bei Sabine Schenk für die Vorbereitung des SDS-PAGE und dauerhafte Unterstützung während des gesamten Projekts. Nicole Russ und Anna Magerhans vorgesehen ausgezeichneten technischen Support. Darüber hinaus möchten wir Andreas Bär und Sheila Mertens für die Produktion des Films zu danken. Diese Arbeit wurde durch Zuschüsse aus dem Volkswagen-Stiftung (Az I/77864) und dem deutschen Bundesministerium für Bildung und Forschung (BMBF, 01 GN 0813) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| TUNER (DE3) pLacI | Novagen, EMD Millipore | 70625 | |
| Glycerol | Carl Roth GmbH | 3783.2 | |
| Na2HPO4 | Carl Roth Gmbh | T876.1 | |
| Trizma Base | Sigma-Aldrich | T1503 | |
| HCl | Carl Roth Gmbh | 4625.1 | |
| Imidazol | Carl Roth Gmbh | X998.4 | |
| NaCl | Carl Roth Gmbh | 9265.2 | |
| Yeast Extract | Carl Roth Gmbh | 2363.4 | |
| Trypton/Pepton | Carl Roth Gmbh | 8952.4 | |
| K2HPO4 | Carl Roth Gmbh | P749.2 | |
| KH2PO4 | Carl Roth Gmbh | 3904.1 | |
| Ampicillin | Sigma-Aldrich | A9518 | |
| Carbenicillin | Sigma-Aldrich | 6344.2 | |
| HEPES | Sigma-Aldrich | H3375 | |
| Lysozyme | Sigma-Aldrich | 62971 | |
| Benzonase | Novagen, EMD Millipore | ||
| L-Tartaric acid, disodium salt | Sigma-Aldrich | ||
| 50% Ni-NTA slurry | Invitrogen | R901-15 | |
| EconoPac columns | Bio-Rad | 732-1010 | |
| Sterile filter 0,22μm | Whatman, GE Healthcare | ||
| Paraformaldehyde (PFA) | Sigma-Aldrich | ||
| LB medium | Yeast extract, Trypton/Pepton, NaCl | ||
| TB medium | Yeast extract, Trypton/Pepton, Glycerol, K2HPO4, KH2PO4 | ||
| Lysis Buffer | 50 mM Na2HPO4, 5 mM Tris, pH 7.8 | ||
| Tartaric Salt Buffer (TSB) | PTB containing 2 M L-Tartaric acid, disodium salt, and 20 mM Imidazol | ||
| Washing Buffer | PTB, 500 mM NaCl, 15 mM Imidazol | ||
| Elution Buffer | PTB, 500 mM NaCl, 250 mM Imidazol | ||
| High Salt Buffer | 600 mM NaCl, 20 mM HEPES, pH 7.4 | ||
| Gylcerol Buffer | 50% glycerol, 500 mM NaCl, 20 mM HEPES, pH 7.4 | ||
| TrypLE™ Express | Invitrogen | ||
| ESGRO (LIF) | EMD Millipore | ||
| NEAA | GIBCO, by Life Technologies | 11140035 | |
| L-Glutamin | GIBCO, by Life Technologies | 25030024 | |
| β-Mercapt–thanol | GIBCO, by Life Technologies | 31350010 | |
| DMEM | GIBCO, by Life Technologies | 11960044 | |
| PBS | GIBCO, by Life Technologies | ||
| Fetal Calf Serum (FCS) | PAA Laboratories | ||
| X-Gal staining solution: | 4 mM K3(FeIII(CN)6), 4 mM K4(FeII(CN)6), 2mM MgCl2 0.4 mg/mL X-Gal solved in PBS | ||
| K3(FeIII(CN)6) | Sigma-Aldrich | P-3367 | |
| K4 (FeII(CN)6) | Sigma-Aldrich | P-9387 | |
| MgCl2 | Sigma-Aldrich | M8266 | |
| X-Gal | Sigma-Aldrich | B4252 |
Referenzen
- Gump, J. M., Dowdy, S. F. TAT transduction: the molecular mechanism and therapeutic prospects. Trends Mol Med. 13 (10), 443-448 (2007).
- Edenhofer, F. Protein transduction revisited: novel insights into the mechanism underlying intracellular delivery of proteins. Curr Pharm Des. 14 (34), 3628-3636 (2008).
- Branda, C. S., Dymecki, S. M. Talking about a revolution: The impact of site-specific recombinases on genetic analyses in mice. Dev Cell. 6 (1), 7-28 (2004).
- Nolden, L. Site-specific recombination in human embryonic stem cells induced by cell-permeant Cre recombinase. Nat Methods. 3 (6), 461-467 (2006).
- Zhang, Y. Inducible site-directed recombination in mouse embryonic stem cells. Nucleic Acids Res. 24 (4), 543-548 (1996).
- Peitz, M. Enhanced purification of cell-permeant Cre and germline transmission after transduction into mouse embryonic stem cells. Genesis. 45 (8), 508-517 (2007).
- Bosnali, M., Edenhofer, F. Generation of transducible versions of transcription factors Oct4 and Sox2. Biol Chem. 389 (7), 851-861 (2008).
- Landry, J. R. Runx genes are direct targets of Scl/Tal1 in the yolk sac and fetal liver. Blood. 111 (6), 3005-3014 (2008).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten