Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Les changements d'équilibre dans la structure de l'ARN de surveillance par «peroxydation» et «oxydatif» Hydroxyl Footprinting Radical
Dans cet article
Résumé
Ce protocole décrit comment quantifier les Mg (II)-dépendante de la formation de l'ARN structure tertiaire par deux méthodes d'empreinte radical hydroxyle.
Résumé
Molécules d'ARN jouent un rôle essentiel dans la biologie. En plus de transmettre l'information génétique, l'ARN peut incorporer uniques structures tertiaires remplir un rôle de régulateur biologiques spécifiques, liant ou de catalyseur. Information sur la formation de contacts tertiaire est essentielle pour comprendre la fonction des molécules d'ARN. Les radicaux hydroxyles (OH •) sont des sondes unique de la structure des acides nucléiques en raison de leur grande réactivité et une petite taille. 1 Lorsqu'il est utilisé comme une sonde d'empreinte, les radicaux hydroxyles cartographier la surface accessible au solvant de l'squelette phosphodiester de l'ADN et l'ARN 1 2 avec aussi fine que la résolution seul nucléotide. Hydroxyle empreinte radical peut être utilisé pour identifier les nucléotides au sein d'une surface de contact intermoléculaires, par exemple en ADN-protéines et d'ARN une teneur en protéines complexes. Equilibre et cinétique 3 4 transitions peut être déterminé en effectuant des empreintes hydroxyles radicaux en fonction d'une solutisur la variable ou l'heure, respectivement. Une caractéristique clé de l'empreinte est que l'exposition limitée à la sonde (par exemple, "la cible unique cinétiques») des résultats de l'échantillonnage uniforme de chaque nucléotide du polymère. 5
Dans cet article, vidéo, nous utilisons le domaine P4-P6 du ribozyme de Tetrahymena pour illustrer la préparation des échantillons d'ARN et la détermination d'un Mg (II)-médiée isothermes pliantes. Nous décrivons l'utilisation du protocole bien connu l'empreinte de radicaux hydroxyles qui nécessite H 2 O 2 (nous appelons cela la "peroxydante« protocole) et une valeur, mais pas très connu, alternative qui utilise naturellement dissous O 2 (nous appelons cela «l' oxydatif "protocole). Un aperçu de la réduction des données, la transformation et les procédures de l'analyse est présentée.
Protocole
1. Préparation des réactifs Footprinting
- Préparer un tampon de réaction 10x contenant 100 mM cacodylate de sodium, EDTA 1 mM, et 1 M KCl. Ajuster le pH à 7,4. Filtrer le tampon en utilisant un dispositif de filtre acétate de 0,2 pM (Nalgene). Remarque: ne pas ARN pipette directement dans la mémoire tampon 10x.
- Préparer le mélange réactif de titrage pour chaque réaction, comme indiqué dans le tableau 1. Le volume du mélange de titration (1x tampon et Mg (II) à la concentration souhaitée) doit être de 90 ul, avant d'ajouter 10 ul d'ARN dans un tampon de 1x.
- Préparer un tampon de digestion RNAse T1 contenant de l'urée 6.63M, citrate de sodium 20 mM, 1 mM EDTA, 0,25 pg / ul ARNt, cyanol xylène 0,025%, et le bleu de bromophénol 0,025%. Ce tampon peut être conservé à 4 ° C pendant jusqu'à 6 mois.
- La réaction de Fenton nécessite fraîchement préparés solutions aqueuses de 250 mM de Fe (NH 4) 2 (SO 4) 2, 250 mM EDTA et 500 mM de sodium L-ascorbate. Les concentrations indiquées dans le chapitre 10.5 sont optimales pour des expériences de l'équilibre dans un tampon de cacodylate de sodium, comme cela ne veut pas piéger les radicaux et divers concentrations en ions divalents.
- Réactifs pour la réaction empreinte (1.5.2) peroxydante (1.5.1) et oxydante.
- La réaction de Fenton peroxydante nécessite fraîchement préparés solutions aqueuses de 100 mM de Fe-EDTA et 50 mM de sodium L-ascorbate et de 1,5% H 2 O 2. La solution Fe-EDTA est préparée en mélangeant Fe (NH 4) 2 (SO 4) 2 avec de l'EDTA à avoir 1,1 fois plus de l'EDTA sur le fer.
- Juste avant d'initier la réaction de clivage oxydatif (3.4.2), de créer le mélange réaction de Fenton en combinant 2 pi de Fe (NH 4) 2 (SO 4) 2 (250 mm), 2,2 l de EDTA (250 mM), 62,5 ul L-ascorbate de sodium (500 mM), 22,5 pi de tampon de réaction 10x et 135,5 ul de H 2 O pour atteindre les concentrations finales dans le mélange réactionnel empreinte (3.4.2) de 0.10 mM, 0,11 mM et 6,61 mM, respectivement.
2. Préparation de l'ARN pour l'expérience d'empreintes
- L'ARN peut être produite par la norme de transcription in vitro de 6 modèles de l'ADN ou, si courte que 50 nucléotides, être achetés (par exemple chez Integrated DNA Technologies, www.idtdna.com). Suivez les procédures recommandées pour la propreté de laboratoire afin de maintenir l'intégrité des échantillons d'ARN au cours des expériences empreintes 7.
- La déphosphorylation 8 du transcrit in vitro d'ARN est nécessaire avant d'étiquetage P 32 de l'extrémité 5 'de l'ARN. 10 pmol d'ARN sont marqués aux extrémités par des kinasing 9. La solution devrait être nuageux après inactivation de la phosphatase succès.
- Purifier l'ARN radiomarqué en utilisant l'électrophorèse sur gel dénaturant. L'ARN est récupéré par deux extractions ultérieures gel en utilisant l'acétate de sodium 0,3 M (pH 5,5). L'ARN est précipité en mélangeant 0,5 ml de solution aqueuse à 1 ml d'éthanol et ultérieuresd'incubation de 1 minute sur la glace sèche. Spin échantillon à 4 ° C, 12 500 rpm pendant 0,5 heure. Jeter le surnageant et laver avec de l'éthanol culot d'ARN de 70%. Retirer le surnageant après centrifugation supplémentaire et culot sec dans le vide.
- Dissoudre le 32 P-étiqueté les échantillons d'ARN dans 330 ul de 1x Buffer.Pipet 30 pl de la solution de l'ARN dans un tube de réaction, un précipité avec l'éthanol et de l'ARN à sec dans le vide; garder ce tube pour générer l'échelle de référence (étape 3.5). Dénaturer la solution tamponnée ARN par chauffage à 95 ° C pendant 2 min. Refroidir les échantillons à température ambiante pendant 15 minutes et donner un tour rapide pour faire baisser les condensats sur les couvercles des tubes dans la solution.
3. L'expérience d'empreintes
- Le peigne électrophorèse détermine le nombre maximal de points de données par gel analytique. Nous utilisons un peigne à 30 puits. L'échelle de référence occupe 2 puits et le contrôle non clivée. Préparer 27 tubes de réaction. La finale Mg (II) concentrations doivent être espacés uniformément sur une échelle logarithmique couvrant plusieurs ordres de grandeur autour du point médian de titrage.
- Par ailleurs, incuber l'ARN et le Mg (II) des solutions à 50 ° C pendant 5 min. Mélanger 10 ul de la solution d'ARN avec 90 ul de la quantité correspondante de Mg (II) en 1x tampon pour atteindre un volume final de 100 pl. Incuber les solutions pendant 30 min à 50 ° C.
- Laissez les solutions s'équilibrer à 25 ° C pendant 1 heure tout en pliage se produit.
- Peroxydante (3.4.1) ou oxydatif (3.4.2) hydroxyle réaction empreinte radicale.
- Démarrer la réaction peroxydante en plaçant des gouttelettes de 2 pl Fe-EDTA (100 mm), 2 ul de sodium L-ascorbate (50 mm) et 2 ul H 2 O 2 (1,5%), séparés les uns des autres à l'intérieur haut de la réaction tube contenant la solution d'ARN et de démarrer la réaction empreinte en mélangeant vigoureusement. Arrêter la réaction après 15 secondes en ajoutant 300 ul d'éthanol froid absolu. Tournez les tubes 3-5 fois. Précipité, lavez etsèche le culot comme à l'étape 2.4.
- Démarrez le oxydatif footprintingreaction radical hydroxyle en ajoutant 5 ul du mélange de réaction de Fenton fraîchement préparés (1,6). Incuber pendant 30 min à 25 ° C. Ajouter 300 ul d'éthanol absolu froid pour stopper la réaction et mélanger en tournant les tubes 3-5 fois. Précipité, laver et sécher le culot, comme indiqué dans l'étape 2.4.
- Générer un échantillon de référence RNase T1 digérer, conformément aux procédures standard de 10 en utilisant la solution préparée à l'étape 1.3.
- Dissoudre culots d'ARN dans les 8 ul du gel de chargement II colorant (Ambion) et confirmer l'ARN est remis en suspension par l'aide du compteur Geiger.
- Préparer une dénaturation, un gel de polyacrylamide 8% séquençage selon des protocoles standard. 11 Charger les échantillons dont deux références et un contrôle. Séparer des fragments d'ARN à 60 W - 75 W pendant 2,5 heures. Exposer gel séché sur un écran de phosphore de stockage pendant la nuit. Balayage écran phosphorique avec un système d'imagerie pour autoradiographie sans film, par exemple,. Tempête 865 (GE Healthcare) ou Typhoon (GE Healthcare). Transfert de fichier image du gel sur votre ordinateur.
4. Analyse des données
- La réactivité du radical hydroxyle de chaque nucléotide est déterminé par la quantification de l'intensité de chaque bande sur le gel analytique. Téléchargez, installez et ouvert SAFA, un outil facile à utiliser un logiciel open source pour le montage et la quantification seule bande. 12, 13 Suivez les instructions du manuel utilisateur. Brièvement, la charge de la séquence d'ARN sous. Txt suivie par l'image gel comme. Gel de fichier. Ajustez intensités des bandes, de définir les voies, choisissez une voie d'ancrage, et d'effectuer un alignement de gel. Attribution des bandes de nucléotides se produit en référence à l'échelle de digestion RNase T1. Quantifier les intégrales bande et utiliser la fonction de normalisation / colorplot à normaliser et à attribuer des résidus potentiellement invariant. Enregistrez le résultat dans un fichier. Txt.
- SAFA sorties d'un tableur contenant des colonnes représentant voies sur le gel et des lignes représentant l'enINTÉGRÉ densité de la bande des bandes individuelles correspondant au fragment d'ARN. Tout d'abord, identifier les sites de protection qui affichent un changement notable dans l'accessibilité de solvant en comparant le profil de protection dérivée de la non Mg (II) de l'échantillon avec le profil du point de fin de Mg (II) de concentration (50 mM dans notre exemple). La baisse de la valeur, le plus protégé le nucléotide est contre l'attaque des radicaux hydroxyles et vice versa.
- Créer isothermes pliage de la bande normalisée de intégrales ou des groupes de nucléotides par rapport Mg (II) de concentration. Les isothermes sont individuellement mis à l'échelle de la saturation fractionnelle par fi = L + (U - L) •
 où f représente la densité intégrée de la bande (s) en cours d'analyse, L et U représentent les limites inférieures et supérieures à la transition et
où f représente la densité intégrée de la bande (s) en cours d'analyse, L et U représentent les limites inférieures et supérieures à la transition et 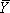 est la saturation fractionnelle. 14 Les données sont adaptées en utilisant un L non-linéaireprogramme d'analyse de l'Est carrés (Nous utilisons soit l'origine (OriginLab) ou GraphPad (GraphPad Software, Inc.)) à l'équation de Hill (1),
est la saturation fractionnelle. 14 Les données sont adaptées en utilisant un L non-linéaireprogramme d'analyse de l'Est carrés (Nous utilisons soit l'origine (OriginLab) ou GraphPad (GraphPad Software, Inc.)) à l'équation de Hill (1), 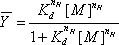
où K d est la constante de dissociation d'équilibre, [M] correspond à la concentration de la variable qui intervient dans la réaction de repliement, Mg (II) dans cet exemple, et n H est le coefficient de Hill. Cette procédure d'échelles de la transition vers la saturation fractionnelle, détermine son milieu et fournit un test phénoménologique de savoir si une transition est sigmoïde. Isothermes dérivées de gels de séquençage (figure 3A) qui ont été analysés par la SAFA (figure 3B) et ajustement tel que décrit ci-dessus sont présentés dans la figure 3C. Les isothermes de saturation fractionnelle montre la figure 3 ont été générés par l'échelonnement dans un tableur les valeurs générées par la SAFA contre le meilleur ajustement des limites supérieure et inférieure (U et L) en utilisant la fraction équation de valeur = / (U - L) - L / (U - L).
5. Les résultats représentatifs:
La figure 3 montre des résultats représentatifs de P4 à P6 ARN • expériences empreinte OH. L'image du gel (à gauche) indique que le clivage de fond) est minime (voie de droite), b) l'affectation simple nucléotide est possible grâce à des bandes bien définies dans la voie de T1 (T1 RNase clive à chaque G), et c) du radical hydroxyle la fragmentation induite par l'ARN est bien au-dessus de fond. La transition de faible à haute Mg (II) est affiliée à diminuer la densité intégrée de la bande individuels et les groupes de bandes indiquant la formation d'ARN structure tertiaire. Simple ou des groupes de nucléotides contigus dont le clivage des changements de façon concomitante sont considérés comme une «protection». Les protections • OH caractéristique du Mg (II) la médiation de pliage de l'ARN P4-P6 correspondent étroitement aux régions inaccessibles solvant de la molécule observée dans sa structure cristalline. 15
Plié molécules d'ARN c une ont des degrés très différents (à savoir, comment proche de fond le déclin bande de densité) de protection contre les contacts tertiaires qui n'ont pas été corrélés avec phénomène structurel ou dynamique claire. Ainsi, certaines molécules d'ARN se montrent bien défini des transitions structurelles, comme le montre la figure 3, et d'autres non. Intensités des bandes sont quantifiés et normalisés par SAFA 12 analyse (figure 3B). La sortie est un "thermique" complot visualiser le degré relatif de protection contre • OH. La transition de couleur décrit les changements d'accessibilité lors de Mg (II) plus. Blanc à rouge ou bleu montre nucléotides plus accessible ou plus protégés, respectivement. Chaque degré d'ombrage est affilié à une valeur numérique qui peut être tracée comme une courbe de protection (figure 3C) et analysées par un modèle de liaison tels que l'équation de Hill (1). La constante d'équilibre de dissociation affilié à la protection de 153 à 155 est à peu près le double de la valeur correspondante de protection 163-164.
ONTENU ">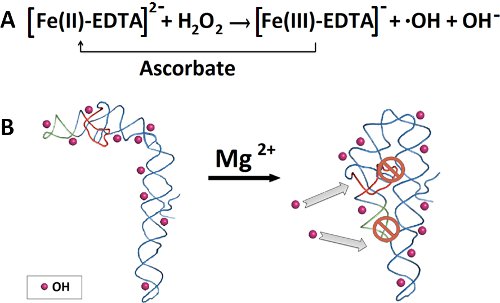
Figure 1. Production de radicaux hydroxyles et P4-P6 pliage ARN. A) le Fe (II) catalyse la production de radicaux hydroxyles par le peroxyde d'hydrogène. L'ascorbate réduit le Fe (III) Retour au fer ferreux. B) En l'absence de Mg (II), aucune structure P4-P6 tertiaire est formé, ce qui permet de pénétrer les radicaux hydroxyles et cliver toutes les positions armature accessible. L'addition de Mg (II) initie pliage de P4 à P6, permettant seulement l'épine dorsale accessible au solvant à être clivée par les radicaux hydroxyles.
Figure 2. Schéma pour une expérience empreinte radical hydroxyle. Un Dephophorylation) et 32 P-5'-end d'étiquetage de l'ARN. B) Purification de 32 P-ARN marqué sur un gel de polyacrylamide dénaturant. C) L'excision de la bande de l'ARN, après l'extraction d'ARN, et précipitation à l'éthanol. D) et Prefoldingrepliement de l'ARN. E) Ajout d'fraîchement préparés mélange réaction de Fenton pour générer les radicaux hydroxyles. F) la séparation fragment d'ARN par électrophorèse sur gel dénaturant de polyacrylamide. G) La quantification de fragments d'ARN par un logiciel de SAFA.
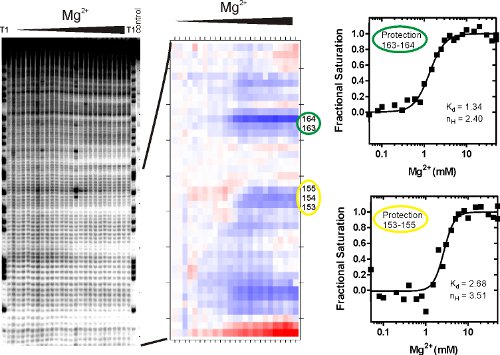
Figure 3. Analyse de fragments d'ARN après réaction empreinte peroxydante Fenton. (A) L'ARN a été exposé à des radicaux hydroxyles et les produits de clivage ont été résolues en utilisant l'électrophorèse sur gel dénaturant de polyacrylamide (PAGE). La photo montre des produits de clivage peroxydante avec une concentration croissante de Mg (II). Voies de référence et de contrôle sont étiquetés. Ce fichier image est l'entrée pour le programme SAFA, qui quantifie le volume de chaque bande. (B) Le «thermique» intrigue générée par la SAFA. (C) Un tableur des valeurs relatives densité de la bande intégrés à la sortie du numéro de nucléotides SAFA est analysée par un modèle de liaison tels que la Colline equatisur (1). Régions de protection ont été choisis en fonction de l'augmentation de la densité de la bande intégrale comme une fonction de Mg (II) de concentration. Le Kd est le Mg (II) la concentration à laquelle la moitié de l'ARN est plié sur le site à surveiller. Le coefficient de Hill, n H, est une mesure de la pente de la courbe qui donne des informations sur la coopérativité de liaison et fournit une estimation plus faible pour le nombre d'ions magnésium impliquée dans le pliage de ce site particulier.
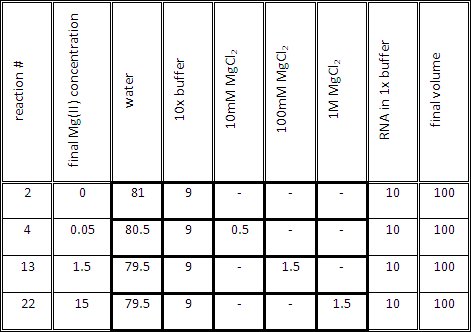
Tableau 1. Volumes Représentant pour générer des échantillons d'ARN contenant différents Mg (II) sur les concentrations.
Discussion
Hydroxyle empreinte radicale est un outil précieux pour évaluer la surface accessible au solvant des acides nucléiques. La formation quantitative et qualitative de la structure tertiaire 14 peut être suivie en fonction de paramètres tels que le type et la concentration d'ions, pH, température, protéines de liaison ou de pliage des co-facteurs. La combinaison convaincante d'un protocole simple et peu coûteux et l'accessibilité résultant de solvant et de l'information pliage sur un seu...
Déclarations de divulgation
Pas de conflits d'intérêt déclarés.
Remerciements
Ce travail a été soutenu par des subventions du National Institute of Health RO1-GM085130 et la National Science Foundation MCB0929394. Nous remercions le Dr Marion Schmidt pour son hospitalité et pour nous permettre de filmer dans son laboratoire.
matériels
| Name | Company | Catalog Number | Comments |
| Name | Company | Cat# | |
| Sodium Cacodylate(Caution! Toxic) | Sigma-Aldrich | C4945-25g | |
| EDTA (0.5 M) | Ambion | AM9260G | |
| DEPC treated water | Ambion | AM9915G | |
| Sodium Acetate (3 M) | Ambion | AM9740 | |
| MgCl2 (1 M) | Ambion | AM9530G | |
| Urea | Ambion | AM9902 | |
| Sodium Citrate | Sigma-Aldrich | W302600 | |
| tRNA | Sigma-Aldrich | R-7876 | |
| Sodium-L-ascorbate | Sigma-Aldrich | A7631-25g | |
| Fe(NH4)2(SO4)2 . 6 H2O | Sigma-Aldrich | F1543-500g | |
| RNase T1 | Fermentas | EN0541 | |
| Hydrogen Peroxide (30%) | Sigma-Aldrich | 349887 |
Références
- Tullius, T. D., Dombroski, B. A. Hydroxyl radical "footprinting": high-resolution information about DNA-protein contacts and application to lambda repressor and Cro protein. Proc. Natl. Acad. Sci. U.S.A. 83, 5469-5473 (1986).
- Celander, D. W., Cech, T. R. Iron(II)-ethylenediaminetetraacetic acid catalyzed cleavage of RNA and DNA oligonucleotides: similar reactivity toward single- and double-stranded forms. Biochemistry. 29, 1355-1361 (1990).
- Celander, D. W., Cech, T. R. Visualizing the higher order folding of a catalytic RNA molecule. Science. 251, 401-407 (1991).
- Sclavi, B., Sullivan, M., Chance, M. R., Brenowitz, M., Woodson, S. A. RNA folding at millisecond intervals by synchrotron hydroxyl radical footprinting. Science. 279, 1940-1943 (1998).
- Tullius, T. D., Dombroski, B. A., Churchill, M., Kam, L. Hydroxyl radical footprinting: a high-resolution method for mapping protein-DNA contacts. Methods. Enzym. 155, 537-558 (1987).
- Milligan, J. F., Groebe, D. R., Witherell, G. W., Uhlenbeck, O. C. Oligoribonucleotide synthesis using T7 RNA polymerase and synthetic DNA templates. Nucleic. Acids. Res. 15, 8783-8798 (1987).
- Schlatterer, J. C., Brenowitz, M. Complementing global measures of RNA folding with local reports of backbone solvent accessibility by time resolved hydroxyl radical footprinting. Methods. 49, 142-147 (2009).
- Sambrook, J., Russell, D. W. . Molecular Cloning: A Laboratory Manual. , (2001).
- Zaug, A. J., Grosshans, C. A., Cech, T. R. Sequence-specific endoribonuclease activity of the Tetrahymena ribozyme: enhanced cleavage of certain oligonucleotide substrates that form mismatched ribozyme-substrate complexes. Biochemistry. 27, 8924-8931 (1988).
- Knapp, G. Enzymatic approaches to probing of RNA secondary and tertiary structure. Methods. Enzym. 180, 192-212 (1989).
- Slatko, B. E., Albright, L. M. Denaturing gel electrophoresis for sequencing. Curr. Protoc. Mol. Biol. Chapter 7, Unit 7.6-Unit 7.6 (2001).
- Das, R., Laederach, A., Pearlman, S. M., Herschlag, D., Altman, R. B. SAFA: semi-automated footprinting analysis software for high-throughput quantification of nucleic acid footprinting experiments. RNA. 11, 344-354 (2005).
- Simmons, K., Martin, J. S., Shcherbakova, I., Laederach, A. Rapid quantification and analysis of kinetic *OH radical footprinting data using SAFA. Methods. Enzym. 468, 47-66 (2009).
- Uchida, T., He, Q., Ralston, C. Y., Brenowitz, M., Chance, M. R. Linkage of monovalent and divalent ion binding in the folding of the P4-P6 domain of the Tetrahymena ribozyme. Biochemistry. 41, 5799-5806 (2002).
- Cate, J. H., Gooding, A. R., Podell, E., Zhou, K., Golden, B. L., Kundrot, C. E., Cech, T. R., Doudna, J. A. Crystal structure of a group I ribozyme domain: principles of RNA packing. Science. 273, 1678-1685 (1996).
- Petri, V., Brenowitz, M. Quantitative nucleic acids footprinting: thermodynamic and kinetic approaches. Curr. Opin. Biotechnol. 8, 36-44 (1997).
- Takamoto, K., Das, R., He, Q., Doniach, S., Brenowitz, M., Herschlag, D., Chance, M. R. Principles of RNA compaction: insights from the equilibrium folding pathway of the P4-P6 RNA domain in monovalent cations. J. Mol. Biol. 343, 1195-1206 (2004).
- Brenowitz, M., Senear, D. F., Shea, M. A., Ackers, G. K. Quantitative DNase footprint titration: a method for studying protein-DNA interactions. Methods. Enzym. 130, 132-181 (1986).
- Shcherbakova, I., Mitra, S. Hydroxyl-radical footprinting to probe equilibrium changes in RNA tertiary structure. Methods. Enzym. 468, 31-46 (2009).
- Howlett, G. J., Minton, A. P., Rivas, G. Analytical ultracentrifugation for the study of protein association and assembly. Curr. Opinion. Chem. Biol. 10, 430-436 (2006).
- McGinnis, J. L., Duncan, C. D., Weeks, K. M. High-throughput SHAPE and hydroxyl radical analysis of RNA structure and ribonucleoprotein assembly. Methods. Enzym. 468, 67-89 (2009).
- Jonikas, M. A., Radmer, R. J., Laederach, A., Das, R., Pearlman, S., Herschlag, D., Altman, R. B. Coarse-grained modeling of large RNA molecules with knowledge-based potentials and structural filters. RNA. 15, 189-199 (2009).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon