Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
À haut débit Générateur expression des protéines utilisant une plate-forme microfluidique
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Nous présentons une approche microfluidique pour l'expression de puces à protéines. Le dispositif se compose de milliers de chambres de réaction contrôlées par des micro-mécaniques vannes. Le dispositif microfluidique est couplé à une bibliothèque de gènes microréseau imprimé. Ces gènes sont ensuite transcrits et traduits sur puce, résultant en un réseau de protéines prêt à l'emploi expérimental.
Résumé
Domaines en croissance rapide, tels que la biologie des systèmes, exigent l'élaboration et la mise en œuvre de nouvelles technologies, permettant des mesures à haut débit et haute-fidélité de grands systèmes. Microfluidique promet d'accomplir beaucoup de ces exigences, comme la réalisation d'expériences de criblage à haut débit sur puce, englobant des dosages biochimiques, biophysiques et basée sur des cellules 1. Depuis les premiers jours de dispositifs microfluidiques, ce domaine a considérablement évolué, conduisant à l'élaboration de microfluidique intégration à grande échelle 2,3. Cette technologie permet l'intégration de milliers de vannes micromécaniques sur un seul appareil avec une empreinte d'affranchissement entreprises (figure 1). Nous avons développé une plate-forme microfluidique à haut débit pour générer l'expression in vitro de puces à protéines (figure 2) nommés PING (Générateur de réseau d'interactions protéiques). Ces tableaux peuvent servir de modèle pour de nombreuses expériencescomme protéine-protéine 4, protéine-ARN 5 ou protéine-ADN 6 interactions.
Le dispositif se composent de milliers de chambres de réaction, qui sont programmés individuellement à l'aide de puces à ADN a. Alignement de ces puces microfluidiques imprimés à dispositifs programmes chaque chambre avec un seul point d'éliminer la contamination potentielle ou réactivité croisée ailleurs, générant des puces utilisant des techniques de puces à ADN standards de repérage est également très modulaire, permettant le rangement de protéines de l'ADN 7, 8, petites molécules, et même des suspensions colloïdales. L'impact potentiel de la microfluidique sur les sciences biologiques est significative. Un certain nombre d'essais basés sur la microfluidique ont déjà fourni des informations nouvelles sur la structure et la fonction des systèmes biologiques, et le domaine de la microfluidique continueront d'influer sur la biologie.
Protocole
1. Fabrication de périphériques
- Acheté DTPA-D SU-8 moisissure et la moisissure contrôle SPR220-7 découlent de la Fonderie Stanford Microfluidique ( www.stanford.edu / group / fonderie ).
- Exposer les moules en silicone à chlorotriméthylsilane (TMCS) la vapeur pendant 10 min pour favoriser la libération élastomère après les étapes de cuisson 9.
- Préparer un mélange à base d'élastomère de silicone et l'agent de durcissement (bien mélanger) dans deux rapports différents 5:1 et 20:1 pour le contrôle de flux et de moules, respectivement. Les différents rapports sont nécessaires pour une bonne adhérence des couches multiples.
- Verser le PDMS 5:1 sur la couche de contrôle (environ 5 mm de hauteur). Degas la couche de contrôle et faire cuire pendant 30 min à 80 ° C. Dépôt à la tournette (Laurell, France) le mélange 20:1 PDMS sur le débit de la couche à 2.600 tours par minute pendant 60 secondes, puis le faire cuire à 80 ° C pendant 30 min. La vitesse tournette est optimisé pour la commande de la soupape à une pressionde 15 psi. Filage rapide se traduira par une mince couche de plus faible pression d'activation, et inversement. Les appareils que nous utilisons ont une limite de 25 psi dans le contrôle et 10 psi dans le flux, au-dessus duquel le détachement du substrat peut se produire.
- Séparer la couche de contrôle du moule. Faites-le lentement et veillez à ne pas peler le modèle SU8. Ensuite, couper l'appareil autour de son périmètre et de percer des trous pour accéder aux canaux de contrôle.
- Aligner le flux et niveaux de contrôle manuellement sous un stéréoscope. Commencez par aligner le coin supérieur gauche, la valve bouton (couche de contrôle) doit être au milieu de la chambre de réaction (couche d'écoulement). Ensuite, aligner la première rangée et relâchez doucement la rangée couche de contrôle par ligne. Assurez-vous que toutes les vannes des boutons sont dans le milieu des chambres de réaction et que les valves d'entrée d'adresse et de traverser les canaux d'écoulement à la position correcte. Le processus peut être répété jusqu'à ce que toutes les lignes locales sont alignés. A la fin, relâcher toute tension dans le b PDMSy soulevant avec soin sur les côtés et dans les coins. Ne soulevez pas trop ou mauvais alignement peut se produire.
- Faire cuire pendant 2 heures à 80 ° C.
- Couper autour du périmètre de l'appareil et le zeste de l'appareil à deux couches (puce) du moule débit. Percer des trous pour accéder aux canaux d'écoulement (Figure 1).
2. Rangement de l'ADN et l'alignement de périphériques
- Produire des gènes synthétiques par PCR d'assemblage. Les gènes synthétiques sont composer du promoteur T7, site de liaison ribosomique (RBS), ORF avec deux marqueurs épitopiques (un à chaque extrémité), et terminateur T7 4. Les gènes peuvent varier en longueur de 100 pb jusqu'à, au moins, 5.000 pb.
- Préparer les gènes synthétiques pour ranger: préparer un mélange d'éthylène glycol de poly (1,25%) et D-tréhalose dihydrate (125 mg / ml) et de distribuer 2 pi par réaction en plaque 384 puits. Cette solution réduit la liaison irréversible de l'ADN de la vitre, ainsi que pour la visualisation lors de l'alignement 10.
- Transférer les gènes de synthèse de la plaque 384 puits. Habituellement concentrations d'ADN peut varier de 10 ng / ul à 100 ng / ul concentration finale. Ajouter dH 2 O pour un volume final de 20 pi (dépend de puces à ADN et le type de broche).
- Détecter une série de gènes synthétiques sur des substrats en verre revêtus époxy en utilisant un puces à ADN. Nous utilisons MicroGrid 610 (Bio Robotics) avec des épingles silicone SMT-S75 (Synthèse parallèle, Etats-Unis). Communiquer avec l'impression de l'ADN, avec ces repères spécifiques, des résultats de taches d'un diamètre d'environ 100 um sur la surface du verre. Chaque chargement broches est suffisant pour environ 100 places. Assurez-vous que les emplacements colonnes et de lignes correspondent à l'appareil utilisé. Le dispositif que nous utilisons contient 16 colonnes et 40 lignes avec un terrain de 680 um de 320 um, respectivement.
- Aligner manuellement le dispositif microfluidique pour l'ensemble de gènes sous un stéréoscope. Le spot ADN doit être dans le milieu des chambres d'ADN. Début de l'alignement en plaçant la première rangée de taches below la première rangée de chambres d'ADN. Ensuite, mettre en ligne le reste des lignes de finition avec un réglage fin de l'ensemble du dispositif. Une fois que le tableau est aligné des précautions doivent être prises pour atténuer le stress tout en PDMS, en le soulevant au niveau local. Si la tension est laissé dans le PDMS il ne peut pas bien adhérer à la puce.
- Enfin, le dispositif de liaison de la lame de verre par incubation d'une nuit sur une plaque chauffante à 80 ° C.
3. D'amorçage et activer le dispositif
- Les vannes de la couche de contrôle sont actionnés à partir de l'ordinateur en utilisant LabVIEW. Le script LabVIEW contrôle une série de microélectrovannes via un boîtier de commande électronique (acheté chez Stanford Microfluidique Foundry). Le collecteur d'électrovanne est reliée à l'air comprimé, qui commandent le flux d'air et la pression dans les soupapes de commande de l'appareil.
- Connecter le dispositif à la tubulure d'électrovannes à l'aide d'un tube flexible en matière plastique d'un diamètre intérieur de 0,02 "(Tygon) Et goupille en acier inoxydable (New England Petit Tubes Corporation).
- Remplir les tubes avec DDW et insérer la broche dans les trous d'accès de la couche de commande. Assurez-vous que chaque tube est relié à son canal de commande correspondant.
- Exécutez l'application LabVIEW sur l'ordinateur pour activer les canaux de contrôle. En activant la vanne à partir du script LabVIEW, nous appliquons une pression d'air, qui à son tour va pousser l'eau dans les canaux de contrôle PDMS. Régler la pression d'air à 5 psi. Il est recommandé de remplir le «sandwich» et les vannes première adresse, afin d'identifier croisées pourparlers entre les canaux de commande des appareils défectueux. En cas de contre-discussions, l'actionnement de la ligne «sandwich» de contrôle conduira à l'actionnement de l'une des lignes du cou ou du contrôle Button. Ceci est similaire à un court-circuit dans un circuit électrique. Dispositif de diaphonie est défectueux et ne peut pas être utilisé.
- Une fois que tous les canaux de contrôle et les vannes sont pleins de DDW, les vannes sont amorcées et raisonsdy pour bloquer les canaux d'écoulement en dessous. Augmenter la pression de l'air à 15 psi. Activation de la vanne est commandée à partir d'un programme LabVIEW grâce à un ensemble de commutateurs ON / OFF connectés à des électrovannes individuelles. Chaque interrupteur commande une ligne de commande spécifique par son électrovanne correspondante. Allumer l'interrupteur (dans le script) appliquer une pression d'air dans le tube correspondant. La pression d'air va pousser le DDW dans la ligne de commande et se traduira par l'expansion des vannes microfluidiques, en bloquant le canal en dessous. La désactivation d'une vanne de régulation, libérera la pression de l'air et ensuite libérer le blocage du canal d'écoulement respectif.
- Afin de s'assurer que toutes les vannes sont ouvertes, raccorder un tuyau à l'une des entrées de débit et de débit d'air dans le dispositif à psi 4-5. Cela libérera des soupapes grippées de la lame de verre.
- Enfin, le dispositif est amorcée et prête. Fermez toutes les entrées et "cou" vannes, pour commencer la chimie de surface.
- Afin de faciliter l'auto-assemblage d'un réseau de protéines à la surface et empêcher l'adsorption non spécifique dans le dispositif microfluidique, la surface est modifiée chimiquement:
- Un composant de s'écouler à travers le dispositif, connecter un nouveau tube avec la solution requise à l'un des canaux d'écoulement dans le dispositif. Relier le côté libre du tube collecteur à l'emploi et ouvrir le flux d'air sous pression (5 psi).
- Chargez 40 pl de la biotine-BSA (1 pg / pl) dans un nouveau tube et le débit environ la moitié de celui-ci pendant 20 min à travers le dispositif, la BSA va se lier à la surface époxy.
- Utiliser Hepes (50 mM) pour le lavage de substrat n'ayant pas réagi entre chacune des différentes étapes de chimie de surface.
- Débit 25 streptavidine ul (0,5 pg / pl) pendant 20 minutes au-dessus de la biotine-BSA.
- Laver avec de l'HEPES pendant 5 min.
- Fermer le bouton «soupape et s'écouler le reste de la biotine-BSA (comme décrit ciLit ci-dessus), la passivation de la surface entourant le bouton.
- Laver avec de l'HEPES pendant 5 min.
- Relâchez le bouton «soupape et le débit 30 ul de penta-His-biotine (0,16 pg / pl) pendant 20 min. L'anticorps se lie à la streptavidine exposés, plus précisément à l'aire sous la «touche» la création d'un anti His-tag tableau.
5. Expression de la protéine
- Exprimer les protéines de l'appareil à l'aide de réticulocytes de lapin couplé transcription rapide et réaction de traduction. L'expression des protéines à partir des gènes synthétiques tachetés sur l'appareil crée un tableau de protéines prêtes à l'emploi. Par exemple, une telle utilisation est pour un écran protéine de liaison.
- Ouvrez les "cou" vannes et de réticulocytes de lapin flux de transcription rapide et couplé réaction de traduction à travers le dispositif dans la chambre de l'ADN. Ensuite, fermez les vannes 'sandwich' et séparer chaque gène de son environnement. Incuber l'appareil sur une plaque chauffante pendant 2,5 heures à 30 ° C. Exprimé proteins se diffuse à partir de la chambre de l'ADN à la chambre de réaction spontanée (vanne col est ouvert) et se lient à l'anticorps anti-His sous le bouton «soupape de blocage de la protéine par l'intermédiaire de son extrémité C-terminale variable.
- Étiqueter les protéines avec C-myc Cy3. L'anticorps se lie à son épitope correspondant situé à l'extrémité N-terminale des protéines.
- Déterminer les niveaux d'expression de protéines avec un scanner de microréseau (LS Reloaded, Tecan) en utilisant un laser à 532 nm et 575 nm d'émission filtre.
6. Les résultats représentatifs
1. Fabrication de périphériques
Une conception graphique pour le périphérique a été créé par AutoCAD en fonction de nos besoins expérimentaux. La conception a été imprimé sur un film transparent par une flasheuse haute résolution. Cette transparence constitue un photomasque en photolithographie contact. Une technique de micro-usinage de surface a été utilisée pour créer des modèles 3-D sur une tranche de silicium déterminée par les motifs inscrits sur til masque utilisé.
Le dispositif microfluidique a été fabriqué sur moule en silicone de moulage en élastomère de silicone (PDMS, SYLGARD 184, Dow Corning, USA) (Figure 1). Chaque dispositif est constitué de deux couches de PDMS alignés, le débit et la couche de contrôle. Le moule a été exposé à chlorotriméthylsilane (TMCS, Aldrich) la vapeur pendant 10 min pour favoriser la libération élastomère après les étapes de cuisson. Un mélange à base d'élastomère de silicone et l'agent de durcissement est préparé en deux taux différents 5:1 et 20:1 pour le contrôle de flux et de moules, respectivement. La couche de contrôle a été dégazé et cuit pendant 30 min à 80 ° C. La couche d'écoulement a d'abord été appliquée par centrifugation (Laurell, USA) à 2.600 tours par minute pendant 60 sec et cuit à 80 ° C pendant 30 min. La couche de contrôle a été séparé de son moule et de contrôle des trous ont été percés de canaux d'accès. Ensuite, les couches de flux et de contrôle ont été alignées manuellement sous un stéréoscope et cuire pendant 2 heures à 80 ° C. Le dispositif à deux couches a été pelées from le moule débit et trous d'écoulement d'accès canaux ont été percés.
2. Description de l'appareil
La capacité de l'appareil peut varier de 500 à 10.000 cellules unitaires (figure 2). Le dispositif est constitué de deux couches, une couche d'écoulement (gris) et de la couche de commande (couleur). La couche de contrôle contiennent une variété de vannes avec fonction différente. Chaque cellule élémentaire, dans la couche d'écoulement, est composé d'une ADN et une chambre de réaction et est commandé par trois types de vannes micromécaniques; «col», «bouton», et «sandwich» (figure 2A). «Gène synthétique» Un tacheté déposé dans la chambre de l'ADN est bloqué à partir de la chambre de réaction par le «cou» de vanne (vert). Lavage de piégeage mécanique et de protéines de surface reliés à la chambre de réaction est réalisée par le «bouton» de soupape (bleu). Le «sandwich» permet à chaque vanne réaction se produise dans sa cellule propre unité (rouge). Les vannes d'adresses peut diviser l'appareil jusqu'à 8 sections indépendantes, des différeanalyse la condition nt. En outre, la couche de commande de soupapes d'entrée contient permettant l'écoulement de fluide sélectionné dans la couche d'écoulement. Les canaux de contrôle PDMS sont pleines de GFI. Quand une valve est actionnée de l'augmentation de pression (15 psi) entraîne l'expansion du PDMS. Dans les endroits où la membrane est assez mince (c.-à-croisement des lignes de commande et de débit), cela suffit pour bloquer complètement la ligne d'écoulement. Hauteur moyenne de cellule unitaire est de 10 um, et le volume moyen de cellule unitaire est inférieure à 1 nl.
3. Expression des protéines et la détection
Les protéines ont été exprimées sur le périphérique à l'aide de réticulocytes de lapin couplé transcription rapide et réaction de traduction (Promega). L'expression des protéines créé un réseau de protéines prêts à l'emploi dans un écran de liaison (figure 3). Un mélange d'expression (12,5 pi) a été chargé dans l'appareil, puis inondées dans les chambres de l'ADN en ouvrant les «cou» vannes. Ensuite, les 'sandwich' vannes étaientfermé en laissant chaque cellule unitaire séparé de ses cellules voisines et le dispositif a été incubé sur une plaque chauffante pendant 2,5 heures à 30 ° C. Protéines exprimées diffusé à travers la chambre de gène dans la chambre de réaction, où l'extrémité C-terminale His pouvant lier le anticorps anti-His (Qiagen) localisé sous le bouton «soupape de blocage de la protéine à la surface. Les protéines ont été marquées avec un Cy3 C-myc (Sigma), qui limite à son épitope correspondant situé à l'extrémité N-terminale des protéines et étiquetés. Les niveaux d'expression des protéines ont été déterminées avec un scanner de microréseau (LS Reloaded, Tecan) à l'aide d'un laser 532 nm et 575 nm filtre. Le réseau de protéines résultant consiste à faire varier les niveaux d'expression des protéines. Généralement, environ 20% d'une banque de gènes ne pas exprimer à des niveaux détectables. Aucune corrélation avec la taille de la protéine a été observée 3. Niveaux de référence ont été déterminés en utilisant des chambres qui n'ont pas été repérés avec l'ADN. Ainsi, les signaux issus des chambres de protéines correspondantes sont dues soit à noise ou adsorption non spécifique des anticorps d'étiquetage.
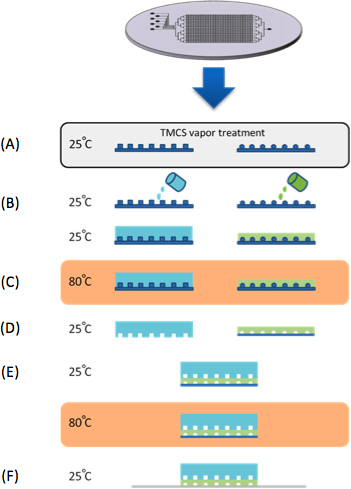
Figure 1. Fabrication puce. (A) Les moules ont été traités avec des vapeurs TMCS pendant 10 min. (B) PDMS est coulé sur le contrôle et moules en silicone de débit. (C) deux moules de commande d'écoulement et sont cuits en 80 ° C pendant 30 min. (D) la couche de contrôle du moule pelées, découpées et les entrées de commande sont perforées. (E) la couche de commande est alignée sur la couche d'écoulement sous un stéréoscope et ensuite cuite dans 80 ° C pendant 2 heures. En parallèle à la fabrication de dispositifs en PDMS, une série de gènes synthétiques sont microarrayed époxy sur des substrats de verre (Associés CEL). (F) Le dispositif est alors aligné sur la puce à ADN piéger les «gènes synthétiques» dans les chambres d'ADN.

Figure 2. Une photo de l'appareil PING. (A) Le dispositif consist de deux couches de PDMS alignés (contrôle et les flux). Le débit de la couche contenant l'ADN et parallèles chambres de réaction (gris) commandés par des soupapes micromécaniques ("bouton", "sandwich" et "col") dans la couche de commande. (B) Les couches sont alignées sur un microréseau imprimé, dont les programmes de chaque chambre de l'ADN avec un seul point. Cela élimine tout risque de contamination.

Figure 3. Images fluorescentes d'un réseau de protéines Créé avec une puce microfluidique. Les gènes imprimés sur place ont été exprimés à des protéines au sein de l'appareil (dans la chambre de l'ADN) et ont tiré vers le bas à la surface (en dessous du "bouton" valve dans la chambre de réaction) à travers leur Son extrémité C-terminale tag. L'expression des protéines a été détectée à l'aide de leur C-myc tag extrémité N-terminale et un anticorps fluorescent spécifique. Différentes intensités de signal indiquent différents niveaux d'expression des protéines. Un-spotted chambres ont servi de témoins pour le fond levels.
Access restricted. Please log in or start a trial to view this content.
Discussion
Dans cet article, nous présentons une méthode de génération de puces à protéines à haut débit en utilisant une plate-forme microfluidique. La génération tableau sont basées sur le microréseau d'impression de matrices d'ADN et l'expression de protéines in vitro de l'ADN dans le dispositif microfluidique.
Notre nouvelle plate-forme microfluidique présente plusieurs avantages importants par rapport aux méthodes actuellement utilisées, qui en fon...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Aucun conflit d'intérêt déclaré.
Remerciements
Ce travail a été soutenu par Marie Curie international prime de réintégration.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| Réactif / équipement | Entreprise | Numéro de catalogue | |
| PDMS-Sylgard 184 | Dow Corning USA | ESSEX-DC | |
| Chlorotriméthylsilane (TMCS | Sigma-Aldrich | C72854 | |
| Substrats de verre enduits époxy | Associés CEL USA | VEPO-25C | |
| Poly éthylène glycol (PEG) | Sigma-Aldrich | 81260 | |
| D-tréhalose dihydraté | Sigma-Aldrich | T9531 | |
| Biotinylé-BSA | Percer | PIR-29130 | |
| Neutravidine | Percer | 31050 | |
| penta-His-biotine | Qiagen | 34440 | |
| Hepes | Industries biologiques | 03-025-1B | |
| TNT-T7 | Promega | L5540 | |
| C-myc Cy3 | Sigma-Aldrich | ||
| Boîtier de commande | Stanford Microfluidique Foundry | ||
| Moule | Stanford Microfluidique Foundry | ||
| Pin | New England Petit Tubes Corporation | ||
| Tygon tube microbore | Tygon | S-54-HL | |
| Puces à ADN | Bio Robotique | MicroGrid 610 | |
| Broches en silicone | Parallèlement Synthèse | SMT-S75 |
Références
- Maerkl, S. J. Integration column: Microfluidic high-throughput screening. Integrative biology quantitative biosciences from nano to macro. 1, 19-29 (2009).
- Hong, J. W., Quake, S. R. Integrated nanoliter systems. Nature. 21, 1179-1183 (2003).
- Unger, M. A Monolithic Microfabricated Valves and Pumps by Multilayer Soft Lithography. Science. 288, 113-116 (2000).
- Gerber, D., Maerkl, S. J., Quake, S. R. An in vitro microfluidic approach to generating protein-interaction networks. Nature. 6, 71-74 (2009).
- Einav, S. Discovery of a hepatitis C target and its pharmacological inhibitors by microfluidic affinity analysis. Nature. 26, 1019-1027 (2008).
- Fordyce, P. M. De novo identification and biophysical characterization of transcription-factor binding sites with microfluidic affinity analysis. Nature Biotechnology. 28, 962-967 (2010).
- Zhu, H. Global analysis of protein activities using proteome chips. Science (New York, N.Y.). 293, 2101-2105 (2001).
- Ramachandran, N. Self-assembling protein microarrays. Science (New York, N.Y.). 305, 86-90 (2004).
- Zhong, J. F. A microfluidic processor for gene expression profiling of single human embryonic stem cells. Lab on a chip. 8, 68-74 (2008).
- Kusnezow, W., Hoheisel, J. D. Solid supports for microarray immunoassays. Journal of molecular recognition JMR. 16, 165-176 (2003).
- Lundin, M., Monne, M., Widell, A., Von Heijne, G., Persson, M. A. A. Topology of the membrane-associated hepatitis C virus protein NS4B. Journal of virology. 77, 5428(2003).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon