Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Analyser l'internalisation cellulaire de nanoparticules et de bactéries multi-spectrale par cytométrie de flux d'imagerie
Dans cet article
Résumé
Dans cet article, nous décrivons une méthode utilisant multi-spectrale cytométrie de flux d'imagerie pour quantifier l'internalisation des nanoparticules ou des bactéries polyanhydride par des cellules RAW 264.7.
Résumé
Systèmes Nanoparticulate ont émergé comme des outils précieux dans la livraison des vaccins par leur capacité à fournir efficacement des marchandises, y compris les protéines, les cellules présentatrices d'antigènes 1-5. L'internalisation des nanoparticules (NP) par les cellules présentatrices de l'antigène est une étape cruciale pour générer une réponse immunitaire efficace à l'antigène encapsulé. Pour déterminer comment les changements dans la fonction incidence des nanoparticules formulation, nous avons cherché à développer un débit élevé, quantitative protocole expérimental qui était compatible avec la détection des nanoparticules internalisées ainsi que les bactéries. À ce jour, deux techniques indépendantes, la microscopie et cytométrie de flux, ont été les méthodes utilisées pour étudier la phagocytose des nanoparticules. La nature à haut débit de la cytométrie de flux génère données statistiques solides. Toutefois, en raison de faible résolution, il ne parvient pas à quantifier avec précision par rapport à des cellules internalisées nanoparticules liées. Microscopie génère des images à haute résolution spatiale; houtefois, cela prend du temps et implique de petits échantillons 6-8. Multi-spectrale cytométrie de flux d'imagerie (MIFC) est une nouvelle technologie qui intègre les aspects de la microscopie et cytométrie en flux qui effectue multicolore imagerie spectrale de fluorescence champ et lumineux à la fois à travers un noyau laminaire. Cette capacité fournit une analyse précise des intensités de signaux fluorescents et les relations spatiales entre les différentes structures et fonctions cellulaires à haute vitesse.
Nous décrivons ici une méthode utilisant MIFC pour caractériser les populations de cellules qui ont intériorisé des nanoparticules polyanhydrides ou Salmonella enterica sérotype Typhimurium. Nous décrivons également la préparation de suspensions de nanoparticules, l'étiquetage, l'acquisition de la cellule sur un système ImageStream X et l'analyse des données en utilisant l'application IDÉES. Nous démontrons également l'application d'une technique qui peut être utilisé pour différencier l'internalisation pathways pour nanoparticules et les bactéries à l'aide cytochalasine-D comme inhibiteur de l'actine-phagocytose.
Protocole
1. RAW 264.7 de la culture cellulaire
- Récolte des cellules RAW 264.7 de leurs flacons quand ils atteignent la confluence en les grattant délicatement avec un grattoir à cellules. Le comte et la plaque eux dans un plat de 24 puits de culture cellulaire à une densité de 5 x 10 5 cellules / puits dans 0,5 ml de milieu complet Eagle modifié par Dulbecco (cDMEM; 10% inactivé par la chaleur du sérum fœtal bovin (FBS), 2 mM Glutamax, et 10 mM d'HEPES) et incuber une nuit à 37 ° C dans un 5% de CO 2 incubateur.
2. Salmonella enterica sérotype Typhimurium pathogène 14028 Transformation et de la Culture
- Présentez plasmides recombinants exprimant la protéine fluorescente verte (GFP) à S. Typhimurium par électroporation. Tout d'abord, préparer la culture de Salmonella enterica sérotype Typhimurium (ATCC 14028) dans un bouillon LB et incuber une nuit à 37 ° C avec aération.
- Le lendemain, pastille 1 ml de bactéries à la vitesse maximale dans une microcentrifugeuse. Laver les cellules four fois avec 1 ml de MOPS ou une solution de glycérol froid (1 mM 3 - (N-morpholino) propanesulfonique (MOPS) de mémoire tampon dans 20% de glycérol) en exerçant une légère remise en suspension du culot avec une pipette entre chaque lavage 9. Après le lavage final, remettre en suspension le culot dans 50 ul de MOPS / glycérol dans lequel ~~~V~~HEAD=NNS 0,5 pg de pAKgfp1 ou pAKgfplux1 ADN plasmidique a été ajouté 10.
- Transférer la suspension cellulaire à un pré-réfrigéré 1 cuvette d'électroporation mm écart et électroporer en utilisant les paramètres: 1800 V, 200 W, 25 mF. Immédiatement après l'accouchement d'impulsion, ajouter 1 ml de bouillon LB et de transférer la suspension cellulaire à une nouvelle tube de 15 ml. Laisser les cellules à se redresser à 37 ° C avec aération. Après une heure, concentrer les cellules par centrifugation et remettre en suspension dans ~ 100 ul de surnageant restant.
- Enfin, la plaque des cellules sur des plaques LB contenant 100 ug / ml d'ampicilline et incuber une nuit à 37 ° C. Des colonies isolées sont purifiées par restreaking avec le choix des antibiotiques et d'expression de la GFP ia confirmé par illumination avec un isothiocyanate de fluorescéine standard (FITC) jeu de filtres.
- Inoculer 10 ml de bouillon LB contenant 25 ug / ml d'ampicilline (ou antibiotique approprié) avec une anse de Salmonella qui a été transformé en expriment la GFP de façon stable.
- Cultiver les bactéries nuit à 37 ° C.
- Diluer à 1:10 de Salmonella dans un tube de culture frais contenant 10 ml de LB additionné d'ampicilline et incuber pendant 5 h à 37 ° C.
- Mesurer l'absorbance de la culture à 600 nm et calculer la concentration de la Salmonella par ml en utilisant un préalable établir la courbe de croissance.
- Diluer la Salmonella dans les cDMEM (0,5 ml / puits) pour obtenir une multiplicité d'infection (MOI) de 100 par RAW 264.7 cellule.
3. Préparation de la suspension de nanoparticules
- Fabriquer 1% FITC-nanoparticules chargées telles que décrites précédemment 11. En bref, les particules sont fabriqués par polyanhydrideanti-solvant nanoencapsulation, dans lequel le polymère est dissous dans du chlorure de méthylène (4 ° C à une concentration de 25 mg / ml) et précipité dans le pentane (-30 ° C à une ratio de 1:200 chlorure de méthylène: pentane). Récupérer des nanoparticules par filtration sous vide. Après évaporation de tout solvant résiduel, peser le polyanhydride séchée nanoparticules à l'aide stérilisés peser le papier.
- Ajouter 5 mg de nanoparticules à 0,5 ml de phosphate froid (PBS, de calcium et de magnésium libre, pH 7,4) dans un tube de 1,5 ml et le garder sur la glace jusqu'à ce que les nanoparticules sont ajoutées aux cellules RAW 264.7.
- Soniquer la suspension de nanoparticules (tout en gardant sur la glace) à l'aide d'un processeur à ultrasons liquide équipé d'une micropointe pour environ 25 s 4 à 6 joules.
4. Essai phagocytose
- Prétraiter un sous-ensemble de cellules RAW 264.7 à 5 pg / ml cytochalasine D-1 h avant l'introduction de NP ou Salmonella en aspirant moyen et replace avec cDMEM frais complété avec l'inhibiteur. Retour des cultures à 37 ° C incubateur.
- Après l'incubation de 1 h avec l'inhibiteur, enlever les plaques de l'incubateur, vortex de la suspension de nanoparticules, et ajouter 10 ul dans les puits appropriés.
- Vortex de Salmonella et d'infecter les cellules RAW 264.7 avec une MOI de 100 en ajoutant des bactéries dans les puits appropriés.
- Appuyez sur les temps de la plaque à quelques mélanger et incuber à 37 ° C ou 4 ° C (contrôle) pendant 45 min.
- Retirez les plaques de l'incubateur ou d'un réfrigérateur et le lieu sur la glace. Laver les cellules deux fois avec de la glace du PBS froid (sans Ca 2 + et Mg 2 +) par aspiration et le rejet l'ancien milieu pour éliminer les particules non consolidées, de Salmonella, et morts ou individuelle des cellules RAW 264.7.
- Pour récolter les cellules RAW 264.7, après le second lavage, ajouter 250 ul de PBS glacé et grattez délicatement les puits.
- Pipeter les cellules récoltées en clair-vue SNAP CAP microcentrifuger les tubes et les garder sur la glace.
- Laver les cellules en ajoutant 1 ml de tampon de lavage à froid (FBS 2% de la chaleur inactivé, l'azoture de sodium à 0,1% dans du PBS) et centrifuger à 250 x g pendant 10 min à 4 ° C.
- Jeter le surnageant et éliminer le tampon résiduel en tapant le microtube inversée sur une serviette en papier. Remettre en suspension le culot cellulaire en faisant délicatement ratisser le microtube dans un rack tube à essai.
- Fixer les cellules RAW 264.7 par l'ajout de 100 uL de paraformaldéhyde à 4% (PFA) dans du PBS et permettre aux cellules de reposer pendant 15 min à température ambiante (RT).
- Laver les cellules RAW 264.7 en ajoutant 1 ml de Perm / Wash tampon (BD Biosciences) et centrifuger à 250 x g pendant 10 min à 4 ° C.
- Répétez l'étape 4.9.
- Colorer les cellules RAW 264.7 pour l'actine par l'ajout de 100 ul de Perm / Wash buffer contenant Alexa Fluor phalloïdine 660 (AF 660; 1:150 de dilution) pendant 15 min à température ambiante. Répétez les étapes 4.11 et 4.12.
- Remettre en suspension des cellules RAW 264.7 dans 50 pl dePBS contenant 1% et PFA de les stocker dans l'obscurité à 4 ° C jusqu'à l'acquisition.
Trucs & astuces:
- Simple échantillons fluorescents de couleur doivent être préparés à cette étape pour être utilisé comme contrôle de la rémunération lors de la configuration de l'instrument ImageStream X. La rémunération contrôle inclus dans cette expérience étaient les suivants: des cellules RAW 264.7 (pas marqué pour l'actine) incubées avec des nanoparticules; des cellules RAW 264.7 (pas marqué pour l'actine) incubé par la bactérie Salmonella; actine marqués ou étiquetés cellules RAW 264.7.
- Pour les études d'inhibiteurs, il est important d'inclure le contrôle du véhicule dans lequel l'inhibiteur a été dissoute en tant que contrôle. Dans cette expérience, la cytochalasine-D a été dissous dans du DMSO à 100%. Par conséquent, nous cellules incubées dans cDMEM contenant du DMSO puis évalué l'internalisation des nanoparticules ou la salmonelle. Nous n'avons pas détecté de différence significative entre le groupe moyen et le contrôle DMSO (données non présentées).
- La SAguide de préparation mple disponible sur le site Amnis ( https://www.amnis.com/images/1__Sample_Prep_Guide_3L_-_0418081.pdf ) est une bonne ressource pour vérifier la compatibilité de fluorophores avec le X ImageStream. Pour accéder à ce document, les utilisateurs doivent d'abord s'inscrire (gratuit-de-charge) avec l'AMIS.
5. L'acquisition des échantillons sur le X ImageStream
- Augmentez la puissance de ImageStream X et le lancement INSPIRE.
- Initialiser Fluidics. A la fin de cette SpeedBeads de script doit être exécuté.
- Dans le menu Fichier, choisissez Charger modèle par défaut.
- Dans le menu Affichage Image Gallery, sélectionnez TOUT et appuyez sur Exécuter pour démarrer l'installation d'imagerie des perles.
- Régler de base de suivi pour les images du centre latéralement (si nécessaire).
- Sélectionnez le fond clair (BF) de canal et cliquez sur Set intensité.
- Attendez jusqu'à ce que le CV vitesse d'écoulement est toujours less de 0,2%.
- Calibrer l'appareil tous les jours. Dans l'onglet ASSIST, cliquez sur Démarrer pour exécuter les étalonnages Tous et aux tests et vérifier que tous ont réussi.
- Appuyez sur Verrouiller Flush et de la charge (FLL) pour charger le premier échantillon. Chargez la plus brillante échantillon dans l'expérience qui entre en fluorescence avec chaque fluorochrome utilisé. Il est essentiel que vous exécutez ce premier échantillon d'établir les paramètres de l'instrument et puis ne pas les changer pour toute l'expérience.
- Tournez sur chaque laser utilisé dans l'expérience et mis la puissance du laser de sorte que chaque fluorochrome a des valeurs de pixels max entre 100 et 4000 points, tels que mesurés dans les diagrammes de dispersion.
- Définir des critères de classification de cellules, d'éliminer collection d'objets indésirables. Pour la collecte de données uniquement à partir des cellules, sélectionnez Limite de la zone Basse dans le canal BF à 50 um. Objets avec superficie inférieure à 50 microns sera considéré comme débris et ne sera pas acquise. Sélectionnez les canaux doivent être collectées.
- Entrez le nom du fichier, le dossier de destination, Séquence mis# À 1 et le nombre d'événements à acquérir.
- Cliquez sur Exécuter Acquérir pour collecter et enregistrer la première expérience fichier de données.
- Cliquez FLL et exécuter l'exemple suivant expérimentale. Répétez jusqu'à ce que tous les échantillons expérimentaux ont été recueillis.
- Cliquez sur Paramètres Comp (s'éteint clair et laser de dispersion et permet la collecte de tous les canaux) et de recueillir 500 cellules positives à partir de chaque échantillon unique teinté pour chaque fluorophore dans l'expérience pour développer une matrice de compensation.
Pour résumer, les échantillons peuvent être exécutés dans l'ordre suivant:
- Brightest premier échantillon.
- Échantillons d'essai restants.
- Rémunération contrôle contenant la fluorescence de couleur unique.
Trucs & astuces:
- Un modèle INSPIRE peuvent être sauvegardés et rechargés pour définir les réglages de l'instrument. Une fois les modèles sont enregistrés, le passeur d'échantillons peut être utilisé pour un fonctionnement sans surveillance pour exécuter les échantillons expérimentaux avec un modèle et le compensation contrôle avec un modèle pour chaque contrôle de la couleur seule.
- La configuration ImageStream X pour ces expériences: 488 et 658 nm lasers d'excitation; 785 nm laser dispersent; 40X (0,75 NA) objectif, 6 système de canal avec une pile ISX filtre standard. L'option passeur d'échantillons a été utilisée pour permettre l'acquisition des échantillons sans surveillance.
- Les paramètres suivants ont été utilisés pour l'expérience. Les nanoparticules ont été imagées avec le laser de 488 nm mis à 10mW, le laser 658 nm fixé à 50 mW, le laser 785 nm fixé à 2 mW et BF dans le canal 1. Cellules infectées par les salmonelles ont été imagées avec le laser de 488 nm réglé à 20 mW, 658 nm laser fixé à 50 mW, le laser 785 nm fixé à 2 mW, et BF dans le canal 1. Contrôles de compensation ont été collectées en l'absence de BF et 785. Au moins 5000 événements (c.-à-cellules) ont été imagées pour chacun des échantillons d'essai.
6. Analyse d'Image
- Lancer des idées et double-cliquez sur l'assistant internalisation et de la charge sur lese de l'échantillon d'essai. fichiers rif.
- Créer une matrice de compensation en cliquant sur 'Matrix Nouveau' à l'étape 2. L'assistant de compensation est lancé. Ajouter des fichiers pour les commandes d'une seule couleur dans l'expérience. Cliquez sur Suivant dans l'assistant en suivant les instructions jusqu'à ce que le fichier de la matrice de rémunération est sauvegardé et chargé dans la boîte à l'étape 2 de l'Assistant internalisation.
- Cliquez sur Suivant et suivez les instructions jusqu'à ce qu'un fichier. Daf est généré.
- Définir les propriétés d'affichage d'image en sélectionnant les chaînes d'images utilisés lors de l'acquisition. Cliquez sur Ch. 2 (FITC) et Ch. 5 (AF 660). (BF et SSC sont sélectionné par défaut).
- Sélectionner le canal sur l'image pour la fabrication de la limite de cellule (CH01) et le canal dans lequel des nanoparticules ou des bactéries ont été collectées (CH02).
- Un diagramme de dispersion de la zone par rapport Brightfield Aspect Ratio fond clair de l'ensemble des cellules est générée. Définir la population seule cellule en cliquant sur les points individuels et de déclenchement autour de maillots. Les cellules individuelles ont un rapport d'aspect d'un1 er tour et doublets d'environ 0,5 (figure 1A).
- Un histogramme de la racine de dégradé sur fond clair Mean Square (RMS) de l'image en fond clair est généré et le point de vue de la population dans la galerie d'image est réglée bin sélectionné (figure 1B). Cliquez sur les bacs afin de déterminer où les cellules de meilleure mise au point de début et de dessiner une zone de la ligne de cellules de grille ciblées. Plus le gradient RMS, le mieux ciblée. Passer à l'étape suivante que si il ya des taches autres que vous voulez à la porte de suite.
- Un diagramme de dispersion de l'intensité nouvelle du canal 2 sur le Pixel axe des x par rapport à Max de Ch2 sur l'axe des y est générée (figure 1C). Cliquez sur les points et voir les images pour vous aider à tirer la région autour des cellules qui sont positives pour les nanoparticules ou des bactéries.
- Un histogramme de la fonctionnalité de l'internalisation est généré avec une région qui commence à 0, qui doit être réglée par l'observation des images (figure 1D). La fonction de l'internalisation est un rapport de lal'intensité à l'intérieur de la cellule à l'intensité de la cellule entière. Il est mis à l'échelle de telle sorte que lors d'une valeur de 0 environ la moitié de l'intensité est à l'intérieur. L'assistant a créé une région pour désigner l'intérieur en faisant un masque qui a utilisé l'entrée image de cellule de l'étape 5 de trouver la surface cellulaire et érodé ce par 4 pixels. Notez que ce masque peut être réglée manuellement pour différents types de cellules lorsque cela est nécessaire. Dans cette expérience, nous avons réglé manuellement la fonctionnalité en créant un masque d'objet sur l'image en fond clair d'abord et érodé ce par 4 pixels. La fonction de l'internalisation a ensuite été calculé sur la base de ce masque: 4 pixel objet érodée. Cette fonctionnalité nous permet de distinguer les particules internalisées et les bactéries, qui ont la majorité de leur signal de fluorescence dans la limite de masque à partir de particules liée à la surface et les bactéries, qui ont la majorité de leur signal de fluorescence en dehors du périmètre de masque (Figure 2).
- Créer un nouvel histogramme avec l'internalisation nouvelle FEAture sur la base de l'objet masque érodée. Dessiner une région à la porte sur les cellules internalisées en visionnant les images en mode bac sélectionné. Dans nos expériences, nous avons mis cette porte à 0,3. Les cellules avec un score inférieur à 0,3 ont été considérés comme des cellules de surface de particules liés positifs.
- Enfin, pour éliminer les cellules avec l'étiquetage de fond et à identifier des nanoparticules internalisées ou des bactéries, la fonctionnalité idées comptent spot a été utilisé. Nombre de Spot est une fonction qui compte le nombre de composantes connexes ou des masques petites dans une image. Les fonctions de masque, Spot, Peak et de l'intensité ont été utilisés pour définir les taches. Le masque au comptant trouve les détails lumineux dans une image qui ont un rayon d'utilisateur spécifié et le seuil au-dessus du fond local, le masque est affinée par la séparation des intensités élevées dans les endroits individuels en utilisant la fonction de pointe puis des taches ci-dessus d'une intensité de 200 chefs d'accusation ont été inclus . Voir le guide place Amnis de masquage pour de plus amples informations (figure 1E). Une nouvelle statistiques modèle de port a été générée par diverses caractéristiques, y compris dans le menu Rapports.
- Ce fichier a été enregistré en tant que fichier de modèle à utiliser pour l'analyse des lots de tous les fichiers expérimentales.
- Dans le logiciel IDEAS, cliquez sur Outils → données par lots des fichiers et des entrées de tous les fichiers. Rif. Ajoutez le fichier matrice de compensation (. CTM) et le fichier modèle (. Ast) dans les sections correspondantes. Envoyer le lot pour le traitement. Après l'étape de traitement, tous les fichiers. Rif sont analysés et les fichiers. Daf sont générées pour chacun des fichiers individuels crus. Un fichier rapport final est généré avec des statistiques pour tous les échantillons.
Trucs & astuces:
- L'analyse d'image a été réalisée en utilisant le 4,0 IDÉES version du logiciel et l'assistant internalisation avec quelques modifications. L'assistant est auto-instructif et l'ouptput. Fichier daf peuvent être générés en suivant les instructions de l'assistant.
7. Les résultats représentatifs
"> Les images représentatives de la figure 2 montrent que MIFC peut être utilisée avec succès pour distinguer entre intériorisée (à gauche du panneau) par rapport à la surface limite (panneau de droite) NP (figure 2A) ou Salmonella (figure 2B). L'internalisation des NP et de Salmonella ont été réduits de deux actine inhiber et d'abaissement de la température de 4 ° C (Fig. 3A). le pourcentage de cellules positives pour la surface liée NP augmenté d'environ 8% à 37 ° C à plus de 35% après que l'une cytochalasine D-ou 4 ° C traitement ( figure 3B). En revanche, le pourcentage de cellules avec Salmonella surface liée a été réduit de 35% à 15% après la cytochalasine D-traitement. L'incubation des cellules RAW 264.7 par Salmonella à 4 ° C a diminué internalisation sans une augmentation apparente de la quantité de liée à la surface des bactéries par rapport à la 37 ° C de contrôle. Ensemble, ces données démontrent que Salmonella et NP sont internalisées par un processus cellulaire similaire qui nécessite d'actine et dépend de la température. En outre, les données indiquent que l'attachement durable de Salmonella aux macrophages nécessite polymérisation de l'actine.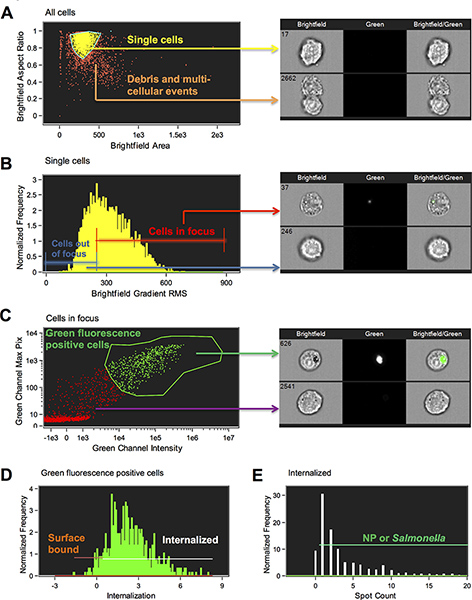
Figure 1. Schéma de la stratégie de déclenchement utilisées pour déterminer intériorisé par rapport nanoparticules liée à la surface et de Salmonella. (A) Pour limiter l'analyse à des cellules individuelles, il est important pour éliminer les débris cellulaires et multi-événements. Les cellules individuelles et pourpoints ont été séparés de agrégats multicellulaires en utilisant la zone IDÉES caractéristiques et l'aspect ratio de l'image en fond clair (M01). Secteur est la taille de l'image en microns carrés et rapport d'aspect est le petit axe divisée par l'axe principal et par conséquent une mesure de circularité (un cercle parfait aura un rapport d'aspect de 1; doublets généralement asperatios de l'ordre de 0,5 ct et des agrégats multicellulaires sont typiquement moins de 0,5). Une région a été élaboré à la porte sur les événements cellule unique (Étape 6.6). (B) Pour la porte sur les cellules de mise au point, les idées de fonctionnalités Dégradé RMS de l'image en fond clair est tracé dans un histogramme. Le dispositif de gradient RMS mesure la qualité de netteté d'une image par détection des changements de valeurs de pixels dans l'image. Une plus grande valeur RMS dégradé indique une image plus ciblée (Étape 6.7). (C) de fluorescence verte des cellules positives ont été sélectionnés par déclenchement sur les cellules avec des valeurs élevées de pixels Max et de l'intensité dans le canal de fluorescence verte (étape 6.8). (D) avec des cellules NP intériorisée ou Salmonella ont été sélectionnés en choisissant la population de cellules avec un score internalisation égale ou supérieure à 0,3. Les cellules ayant obtenu une note inférieure à 0,3 ont été considérés comme liée à la surface (étape 6.9). (E) Les cellules de la "intériorisé" porte ont été en outre caractérisé sur la base du nombre de places (NP ou STM) parce AUTRESe sont des cellules avec une coloration de fond qui ont été comptés comme intériorisée, mais avait une valeur au comptant de zéro (étape 6.11). Images représentatifs sont présentés pour chaque porte. Cliquez ici pour agrandir la figure .
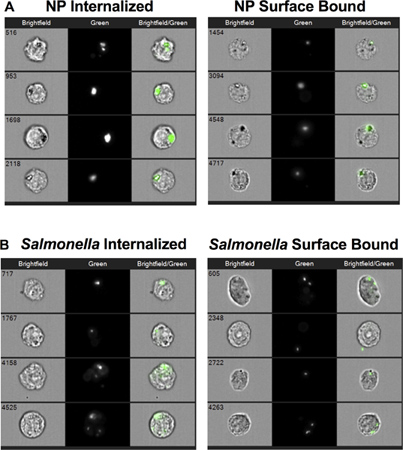
Figure 2. Images de cellules de surface de nanoparticules internalisées et consolidés (NP) ou Salmonella. (A) des images représentatives de cellules RAW 264.7 que intériorisée NP (panneau de gauche) et les cellules à laquelle ont été NP liés à leur surface, mais pas intériorisée (à droite). (B) des images représentatives de cellules RAW 264.7 que Salmonella intériorisée (à gauche) et les cellules à laquelle Salmonella ont été liés à leur surface, mais pas intériorisée (à droite).
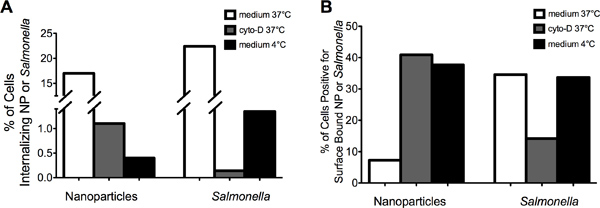
Figure 3. Cytochalale traitement le péché-D de cellules inhibées internalisation des NP et de Salmonella. (A) Le prétraitement des cellules RAW 264.7 par la cytochalasine-D ou l'incubation à 4 ° C réduit l'incidence de l'internalisation du NP ou des Salmonella par rapport à des cellules RAW 264.7 incubées à 37 ° C dans un milieu. RAW 264.7 cellules incubées dans un milieu contenant du DMSO (c.-à-véhicule de contrôle) ont montré des niveaux d'internalisation similaires pour le NP et Salmonella par rapport à un milieu seul (données non présentées). (B) Le prétraitement des cellules RAW 264.7 avec la cytochalasine-D a augmenté le pour cent des cellules avec une surface liée NP tout en diminuant le pour cent des cellules avec de Salmonella de surface lié.
Discussion
Des études ont montré que les nanoparticules biodégradables à base de poly (lactique-co-acide glycolique (PLGA) ou polyanhydrides peuvent être utilisés pour délivrer des antigènes encapsulés ou des médicaments à des cellules cibles. L'adoption de ces nanoparticules par les cellules phagocytaires est important pour leur efficacité, ce qui rend quantitative . analyse de l'internalisation crucial dans la conception de nouveaux systèmes de nanoparticules de livraison En utilisant cette méthode, l'a...
Déclarations de divulgation
Sherree L. ami est employé par Amnis Corporation, qui fabrique le système X ImageStream.
Remerciements
Les auteurs tiennent à remercier le Prix ONR-Muri (NN00014-06-1-1176) et le US Army Medical Research et du matériel de commande (Numéros de subvention W81XWH-09-1-0386 et W81XWH-10-1-0806) pour les financiers soutenir.
matériels
| Name | Company | Catalog Number | Comments |
| Nom du réactif | Entreprise | Numéro de catalogue | Commentaires |
| RAW 264.7 lignée cellulaire | American Type Culture Collection (ATCC) | TIB-71 | |
| Eagle modifié par Dulbecco (DMEM) | Cellgro | 10-013-CV | |
| Sérum de veau fœtal | Atlanta Biologicals | S 11150 | Premium Grade |
| Glutamax | Gibco | 35050-061 | |
| HEPES | Gibco | 15630-080 | |
| 24 et la plaque | PPT | 92024 | |
| Flacons de culture cellulaire | PPT | 90151 | |
| Racloir à cellules | PPT | 99002 | 24 cm |
| Salmonella enterica sérotype Typhimurium | ATCC | 14028 | |
| BTX ECM630 Manipulateur cellule électro | BTX Harvard Apparatus | ||
| MOPS | Fisher Scientific | BP308 | |
| Tampon phosphate salin (PBS) | Cellgro | 21-040-CV | |
| Ultrasons processeur liquide | Misonix | S-4000 | |
| Cytochalasine-D | Sigma-Aldrich, | C8273 | |
| Formaldéhyde | Polysciences | 04018 | |
| Le tampon de lavage | FBS 2% de la chaleur inactivé, l'azoture de sodium à 0,1% dans du PBS. | ||
| Perm / tampon de lavage | BD Biosciences | 554714 | |
| Clear-voir microtubes Snap Cap | Sigma | T4816 | |
| Alexa Fluor 660 phalloïdine | Invitrogen | A22285 | |
| X ImageStream | Amnis Corporationtion | 100200 | Options: 658nm laser, passeur d'échantillons |
| L'azoture de sodium | Fisher Scientific | S-500 227I |
Références
- Ulery, B. D., Kumar, D., Ramer-Tait, A. E., Metzger, D. W., Wannemuehler, M. J., Narasimhan, B. Design of a protective single-dose intranasal nanoparticle-based vaccine platform for respiratory infectious diseases. PLoS One. 6, e17642 (2011).
- Kasturi, S. P., Skountzou, I., Albrecht, R. A., Koutsonanos, D., Hua, T., Nakaya, H. I., Ravindran, R., Stewart, S., Alam, M., Kwissa, M., Villinger, F., Murthy, N., Steel, J., Jacob, J., Hogan, R. J., García-Sastre, A., Compans, R., Pulendran, B. Programming the magnitude and persistence of antibody responses with innate immunity. Nature. 470, 543-547 (2011).
- Rice-Ficht, A. C., Arenas-Gamboa, A. M., Kahl-McDonagh, M. M., Ficht, T. A. Polymeric particles in vaccine delivery. Curr. Opin. Microbiol. 13, 106-112 (2010).
- Jain, J. P., Chitkara, D., Kumar, N. Polyanhydrides as localized drug delivery carrier: an update. Expert. Opin. Drug. Deliv. 5, 889-907 (2008).
- Pfeifer, B. A., Burdick, J. A., Little, S. R., Langer, R. Poly(ester-anhydride):poly(beta-amino ester) micro- and nanospheres: DNA encapsulation and cellular transfection. Int. J. Pharm. 304, 210-219 (2005).
- Ahmed, F., Friend, S., George, T. C., Barteneva, N., Lieberman, J. Numbers matter: quantitative and dynamic analysis of the formation of an immunological synapse using imaging flow cytometry. J. Immunol. Methods. 347, 79-86 (2009).
- Hampton, M. B., Winterbourn, C. C. Methods for quantifying phagocytosis and bacterial killing by human neutrophils. J. Immunol. Methods. 232, 15-22 (1999).
- Rieger, A. M., Hall, B. E., Barreda, D. R. Macrophage activation differentially modulates particle binding, phagocytosis and downstream antimicrobial mechanisms. Dev. Comp. Immunol. 34, 1144-1159 (2010).
- Murphy, K. C., Campellone, K. G. Lambda Red-mediated recombinogenic engineering of enterohemorrhagic and enteropathogenic E. coli. BMC. Mol. Biol. 4, 11 (2003).
- Karsi, A., Lawrence, M. L. Broad host range fluorescence and bioluminescence expression vectors for Gram-negative bacteria. Plasmid. 57, 286-295 (2007).
- Ulery, B. D., Phanse, Y., Sinha, A., Wannemuehler, M. J., Narasimhan, B., Bellaire, B. H. Polymer chemistry influences monocytic uptake of polyanhydride nanospheres. Pharm. Res. 26, 683-690 (2009).
- Doherty, G. J., McMahon, H. T. Mechanisms of endocytosis. Annu. Rev. Biochem. 78, 857-902 (2009).
- Vercauteren, D., Vandenbroucke, R. E., Jones, A. T., Rejman, J., Demeester, J., De Smedt, S. C., Sanders, N. N., Braeckmans, K. The use of inhibitors to study endocytic pathways of gene carriers: optimization and pitfalls. Mol. Ther. 18, 561-569 (2010).
- Di Marzio, L., Marianecci, C., Cinque, B., Nazzarri, M., Cimini, A. M., Cristiano, L., Cifone, M. G., Alhaique, F., Carafa, M. pH-sensitive non-phospholipid vesicle and macrophage-like cells: binding, uptake and endocytotic pathway. Biochim. Biophys. Acta. 1778, 2749-2756 (2008).
- Torres, M. P., Vogel, B. M., Narasimhan, B., Mallapragada, S. K. Synthesis and characterization of novel polyanhydrides with tailored erosion mechanisms. J. Biomed. Mater. Res. A. 76, 102-110 (2006).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon