È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Analizzando internalizzazione cellulare di nanoparticelle e batteri da Multi-spettrale Citometria a Flusso Imaging
In questo articolo
Riepilogo
In questo articolo descriviamo un metodo che utilizza multi-spettrale di citometria a flusso di imaging per quantificare l'interiorizzazione delle nanoparticelle polianidride o batteri da parte delle cellule RAW 264.7.
Abstract
Sistemi di nanoparticelle sono emersi come strumenti preziosi nella consegna vaccino attraverso la loro capacità di fornire in modo efficiente merci, ivi compresi le proteine, le cellule presentanti l'antigene 1-5. Internalizzazione di nanoparticelle (NP) da cellule presentanti l'antigene è un punto critico nel generare una risposta immunitaria all'antigene incapsulato. Per determinare come le alterazioni della funzionalità impatto formulazione di nanoparticelle, abbiamo cercato di sviluppare un elevato throughput, il protocollo sperimentale quantitativa che fosse compatibile con rilevare le nanoparticelle internalizzate così come batteri. Ad oggi, due tecniche indipendenti, microscopia e citometria di flusso, sono stati i metodi utilizzati per studiare la fagocitosi di nanoparticelle. La natura throughput elevato della citometria a flusso genera solidi dati statistici. Tuttavia, a causa della bassa risoluzione, non riesce a quantificare con precisione internalizzati contro cellule nanoparticelle associati. Microscopia genera immagini ad alta risoluzione spaziale; hazione sul mercato, è in termini di tempo e coinvolge campioni di piccole dimensioni 6-8. Multi-spettrale citometria a flusso di immagini (MIFC) è una nuova tecnologia che incorpora aspetti di microscopia e di citometria a flusso che esegue multi-color immagini spettrali di campo fluorescenza e luminoso simultaneamente attraverso un nucleo laminare. Questa funzionalità fornisce un'analisi accurata di intensità di segnale fluorescenti e le relazioni spaziali tra le diverse strutture e funzioni cellulari ad alta velocità.
Qui, descriviamo un metodo che utilizza MIFC per caratterizzare le popolazioni di cellule che hanno interiorizzato nanoparticelle polianidride o Salmonella enterica sierotipo Typhimurium. Si descrivono anche la preparazione di sospensioni di nanoparticelle, etichettatura cellulare, su un sistema di acquisizione ImageStream X e l'analisi dei dati utilizzando l'applicazione IDEE. Abbiamo inoltre dimostrato l'applicazione di una tecnica che può essere utilizzata per differenziare il p internalizzazioneathways per nanoparticelle e batteri utilizzando citocalasina-D come un inibitore di actina-mediata fagocitosi.
Protocollo
1. RAW 264,7 Colture Cellulari
- Harvest RAW 264.7 cellule dai loro fiaschi quando raggiungano la confluenza raschiando delicatamente con una spatola delle cellule. Conte e la piastra in un 24-ben piatto di coltura delle cellule ad una densità di 5 x 10 5 cellule / pozzetto in 0,5 mL completa Medio Dulbeccòs Modified Eagle (cDMEM; 10% inattivato al calore siero fetale bovino (FBS), 2 mM Glutamax, e HEPES 10 mM) e incubare durante la notte a 37 ° C in 5% CO 2 incubatore.
2. Patogeno Salmonella enterica sierotipo Typhimurium 14.028 Trasformazione e Cultura
- Introdurre plasmidi ricombinanti esprimenti la proteina fluorescente verde (GFP) a S. Typhimurium mediante elettroporazione. In primo luogo, preparare cultura di Salmonella enterica sierotipo Typhimurium (ATCC 14028) in brodo LB ed incubare una notte a 37 ° C con aerazione.
- Il giorno seguente, pellet 1 mL di batteri a velocità massima in una microcentrifuga. Lavare le cellule four volte con 1 ml di MOPS / freddi soluzione di glicerolo (1 mM 3 - (N-morfolino) propansolfonico (MOPS) tampone in 20% glicerolo) delicatamente risospendere il pellet con una pipetta tra ogni lavaggio 9. Dopo l'ultimo lavaggio, risospendere il pellet in 50 microlitri di MOPS / glicerolo in cui ~ 0,5 ug di pAKgfp1 o pAKgfplux1 DNA plasmidico è stato aggiunto 10.
- Trasferire la sospensione cellulare in un pre-refrigerati 1 cuvetta elettroporazione gap mm e elettroporare utilizzando le impostazioni: 1800 V, 200 W, 25 mF. Subito dopo la consegna del polso, aggiungere 1 ml di brodo LB e trasferire la sospensione cellulare in un nuovo tubo di 15 ml. Lasciare le cellule di recuperare a 37 ° C con aerazione. Dopo un'ora, concentrare le cellule per centrifugazione e risospendere in ~ 100 pl di surnatante rimanente.
- Infine, le cellule piastra su piastre LB contenenti 100 pg / mL di ampicillina e incubare durante la notte a 37 ° C. Singole colonie sono purificati da restreaking con selezione antibiotica ed espressione di GFP is confermato da illuminazione con uno standard di isotiocianato di fluoresceina (FITC), set di filtri.
- Inoculare 10 ml di brodo LB contenente 25 mg / ml di ampicillina (o antibiotico appropriato) con un'ansata di Salmonella che è stata trasformata in modo stabile GFP espresso.
- Crescere i batteri notte a 37 ° C.
- Diluire 1:10 Salmonella in una provetta di coltura fresco contenente 10 ml di LB supplementato con ampicillina e incubare per 5 ore a 37 ° C.
- Misurare l'assorbanza della coltura a 600 nm e calcolare la concentrazione della Salmonella per mL usando una curva di crescita precedentemente stabilire.
- Diluire la Salmonella in cDMEM (0,5 mL / pozzetto) per ottenere una molteplicità di infezione (MOI) di 100 per RAW 264,7 cella.
3. Preparazione della sospensione di nanoparticelle
- Realizzare 1% FITC-caricati nanoparticelle come descritto in precedenza 11. Brevemente, le particelle sono fabbricati da polianidrideanti-solvente nanoencapsulation, in cui il polimero è disciolto in cloruro di metilene (4 ° C ad una concentrazione di 25 mg / mL) e si precipita in pentano (-30 ° C ad un rapporto 1:200 cloruro di metilene: pentano). Recuperare nanoparticelle mediante filtrazione sotto vuoto. Dopo evaporazione qualsiasi solvente residuo, pesare il polianidride secca utilizzando nanoparticelle sterilizzato peso della carta.
- Aggiungere 5 mg di nanoparticelle di 0,5 ml di fosfato freddo soluzione salina tamponata (PBS, calcio e magnesio libero, pH 7,4) in una provetta da microcentrifuga da 1,5 ml e mantenere in ghiaccio fino le nanoparticelle sono aggiunti alle cellule RAW 264.7.
- Sonicare la sospensione nanoparticelle (mantenendo su ghiaccio) utilizzando un processore ultrasuoni liquido dotato di micropunte per circa 25 s a 4 a 6 joule.
4. Fagocitosi Assay
- Pretrattare un sottoinsieme di cellule RAW 264.7 con 5 pg / mL citocalasina D-1 ora prima dell'introduzione di NP o Salmonella aspirando medie e replacing con cDMEM fresco integrato con l'inibitore. Riportare le culture di 37 ° C incubatore.
- Dopo l'incubazione 1 h con l'inibitore, rimuovere le piastre dal termostato, vortex la sospensione delle nanoparticelle, e aggiungere 10 microlitri nei rispettivi pozzetti.
- Vortex sulla Salmonella e infettare le cellule RAW 264.7 con una MOI di 100 aggiungendo i batteri nei rispettivi pozzetti.
- Toccare i tempi piastra poche per miscelare e incubare a 37 ° C o 4 ° C (controllo) per 45 min.
- Rimuovere le piastre dal termostato o il frigorifero e il luogo su ghiaccio. Lavare le cellule due volte con PBS ghiacciato (senza Ca 2 + e Mg 2 +) aspirando e scartando il vecchio mezzo per rimuovere le particelle non legato, Salmonella, e morti o distaccato cellule RAW 264.7.
- Per raccogliere le cellule RAW 264.7 dopo il secondo lavaggio, si aggiungano 250 microlitri del freddo PBS e raschiare delicatamente i pozzetti.
- Pipettare le cellule raccolte in chiaro-view scatto micro capcentrifugare i tubi e tenerli su ghiaccio.
- Lavare le cellule aggiungendo 1 ml di tampone di lavaggio a freddo (2% FBS inattivato al calore, azide di sodio allo 0,1% in PBS) e centrifugare a 250 g per 10 min a 4 ° C.
- Eliminare il surnatante e rimuovere i residui di tampone toccando il tubo invertito microcentrifuga su carta assorbente. Risospendere il pellet di cellule delicatamente rastrellando la provetta in un rack provetta.
- Fissare le cellule RAW 264.7 aggiungendo 100 pl del 4% paraformaldeide (PFA) in PBS e consentire alle cellule riposare per 15 min a temperatura ambiente (RT).
- Lavare le cellule RAW 264.7 aggiungendo 1 ml di Perm / tampone di lavaggio (BD Biosciences) e centrifugare a 250 g per 10 min a 4 ° C.
- Ripetere il punto 4.9.
- Colorare le cellule RAW 264.7 per actina aggiungendo 100 microlitri di Perm / Wash tampone contenente phalloidin Alexa Fluor 660 (AF 660, 1:150 diluizione) per 15 min a RT. Ripetere i passaggi 4.11 e 4.12.
- Risospendere le cellule RAW 264.7 in 50 pl diPBS contenente l'1% PFA e conservarli al buio a 4 ° C fino acquisizione.
Consigli & note:
- Singoli campioni di fluorescenza di colore devono essere preparati a questo passaggio per essere utilizzati come controlli di compensazione durante l'impostazione dello strumento ImageStream X. La compensazione controlli inclusi in questo esperimento sono stati: RAW 264.7 cellule (non etichettati per actina) incubate con nanoparticelle, le cellule RAW 264.7 (non etichettati per actina) incubate con Salmonella; actina solo etichettati cellule RAW 264.7.
- Per gli studi inibitore, è importante comprendere il controllo del veicolo in cui l'inibitore è stato sciolto come controllo. In questo esperimento, citocalasina-D è stato disciolto in 100% DMSO. Noi, pertanto, le cellule incubate in cDMEM contenente DMSO e quindi valutato l'interiorizzazione delle nanoparticelle o Salmonella. Non abbiamo rilevato alcuna differenza significativa tra il gruppo medio e DMSO controllo (dati non mostrati).
- La saguida per la preparazione servite ad esempio disponibile sul sito web Amnis ( https://www.amnis.com/images/1__Sample_Prep_Guide_3L_-_0418081.pdf ) è una buona risorsa per verificare la compatibilità di fluorofori con la X ImageStream. Per accedere a questo documento, gli utenti devono prima registrarsi (free-of-charge) con Amis.
5. Acquisizione campione sulla X ImageStream
- Accendere ImageStream X e il lancio INSPIRE.
- Inizializzare Fluidics. Alla fine di questo SpeedBeads script devono essere in esecuzione.
- Nel menu file, scegliere Carica modello predefinito.
- Nel menu Visualizza Image Gallery, selezionare ALL e premere Setup Run per avviare l'imaging le perline.
- Regolare il Nucleo di monitoraggio per le immagini del centro lateralmente (se necessario).
- Selezionare la (BF) Brightfield canale e fare clic su Imposta intensità.
- Attendere che il CV velocità di flusso è costantemente less dello 0,2%.
- Calibrare lo strumento quotidianamente. Nella scheda ASSIST, fare clic su Start tutto per eseguire calibrazioni e test e verificare che tutti hanno passato.
- Premere Blocco Flush and Load (FLL) per caricare il primo campione. Caricare il più luminoso esempio nella esperimento che reagisce con ogni fluorocromo utilizzato. E 'fondamentale che si esegue questo primo campione per stabilire le impostazioni dello strumento e poi non li cambierà per l'intero esperimento.
- Accendere ogni laser utilizzato per l'esperimento e impostare la potenza del laser in modo che ogni fluorocromo ha i valori dei pixel massimi compresi tra 100 e 4000 conteggi, misurati nei grafici a dispersione.
- Impostare i criteri di classificazione delle cellule, per eliminare la raccolta di oggetti indesiderati. Per la raccolta di dati da solo le celle, selezionare limitare l'area inferiore nel canale BF a 50 micron. Gli oggetti con superficie inferiore a 50 pm saranno considerati detriti e non saranno acquisiti. Selezionare i canali da raccogliere.
- Immettere il nome del file, cartella di destinazione, impostare Sequence# 1 e il numero di eventi da acquisire.
- Fare clic su Esegui Acquisizione di raccogliere e salvare il primo file di dati dell'esperimento.
- Clicca FLL ed eseguire l'esempio successivo sperimentale. Ripetere fino a quando tutti i campioni sperimentali sono stati raccolti.
- Fare clic su Impostazioni Comp (si spegne chiaro e laser scatter e consente la raccolta di tutti i canali) e raccogliere 500 cellule positive di ogni singolo campione colorato per ogni fluoroforo nell'esperimento di sviluppare una matrice di compensazione.
Riassumendo, i campioni possono essere eseguito nel seguente ordine:
- Brightest primo campione.
- Campioni rimanenti.
- Compensazione controlla contenente fluorescenza singolo colore.
Consigli & note:
- Un modello INSPIRE può essere salvato e ricaricato per impostare le impostazioni dello strumento. Una volta che i modelli vengono salvati, il campionatore può essere utilizzato per il funzionamento incustodito per eseguire i campioni sperimentali con un modello e il compensation controlla con un modello per ogni singolo controllo del colore.
- La configurazione ImageStream X per questi esperimenti: 488 e 658 nm di eccitazione laser, laser scatter 785 nm; 40X (0,75 NA) obiettivo, 6 sistema di canali con una pila standard di ISX filtro. L'opzione autocampionatore è stato usato per permettere l'acquisizione automatica dei campioni.
- Le seguenti impostazioni sono stati utilizzati per l'esperimento. Le nanoparticelle sono state ripreso con il laser 488 nm impostato a 10 mW, il laser 658 nm impostato a 50 mW, il laser 785 nm impostata a 2 mW e BF nel canale 1. Cellule infettate da Salmonella sono stati ripresi con il laser 488 nm impostato a 20 mW, laser 658 nm impostato a 50 mW, il laser 785 nm impostato su 2 mW, e BF nel canale 1. I controlli di compensazione sono stati raccolti in assenza di BF e 785. Almeno 5000 eventi (ossia, cellule) sono state ripreso per ciascuno dei campioni di prova.
6. Image Analysis
- Lanciare idee e fare doppio clic sulla procedura guidata di internalizzazione e il carico sule del campione di prova. file rif.
- Creare una matrice di compensazione cliccando su 'Matrix Nuovo' nella fase 2. La procedura guidata di compensazione è lanciato. Aggiungere i file per i controlli dei colori singoli in esperimento. Fare clic su Avanti la procedura guidata seguendo le indicazioni fino a quando il file di matrice di compensazione viene salvato e caricato nel box in fase 2 della procedura guidata internalizzazione.
- Fare clic su Avanti e seguire le indicazioni fino ad una. Daf file viene generato.
- Impostare le proprietà di visualizzazione delle immagini, selezionando i canali di immagine utilizzati durante l'acquisizione. Clicca su Ch 2 (FITC) e Ch 5 (AF 660). (BF e SSC sono selezionato di default).
- Selezionare il canale immagine per rendere il confine cella (CH01) e il canale in cui sono stati raccolti nanoparticelle o batteri (CH02).
- Un grafico a dispersione di Brightfield Area rispetto Aspect Ratio Brightfield di tutte le cellule viene generato. Definire la popolazione singola cella facendo clic su singoli punti e gating intorno canottiere. Singole cellule hanno un rapporto di unround 1 e doppiette circa 0,5 (Figura 1A).
- Un istogramma del Gradient Brightfield Root Mean Square (RMS) dell'immagine brightfield viene generata e la vista della popolazione nella galleria di immagini è impostato su bin selezionato (Figura 1B). Clicca sui bidoni per determinare dove le cellule iniziano a migliore messa a fuoco e disegnare una linea di regione per le cellule cancello mirati. Più alto è il gradiente RMS, meglio focalizzato. Salta il passo successivo a meno che non ci sono altre macchie che si desidera cancello.
- Un grafico a dispersione nuovo Intensità del Canale 2 sul rispetto all'asse x-Max Pixel del Ch2 sulla y è generato (Figura 1C). Clicca sui punti e visualizzare le immagini per aiutarvi a disegnare la zona intorno alle cellule che sono positive per le nanoparticelle o batteri.
- Un istogramma della funzione di internalizzazione viene generato con una regione che inizia a 0, che dovrebbe essere regolato osservando immagini (Figura 1D). La caratteristica internalizzazione è un rapporto dellaintensità all'interno della cellula per l'intensità della cellula intera. Si scalato in modo tale che ad un valore di 0 circa la metà dell'intensità è all'interno. Il wizard ha creato una regione per designare l'interno facendo una maschera che ha utilizzato l'immagine in ingresso cella da passaggio 5 per trovare la superficie cellulare e erosa questo 4 pixel. Si noti che questa maschera può essere regolata manualmente per i diversi tipi di cellule in caso di necessità. In questo esperimento, si regolato manualmente la funzionalità creando una maschera oggetto sull'immagine brightfield prima e eroso questo 4 pixel. La funzionalità internalizzazione è stato quindi calcolato sulla base di questa maschera 4 Oggetto pixel erosa. Questa caratteristica permette di distinguere particelle interiorizzati e batteri, che hanno la maggior parte del loro segnale di fluorescenza all'interno del perimetro maschera, da particelle superficiali legati e batteri, che hanno la maggior parte del loro segnale di fluorescenza di fuori del confine maschera (Figura 2).
- Creare un nuovo istogramma con l'internalizzazione nuova featura basata sulla maschera Object eroso. Disegnare una regione a porta sulle cellule interiorizzate visualizzando le immagini in modalità bin selezionato. Nei nostri esperimenti, abbiamo impostato questa porta a 0,3. Le cellule con un punteggio inferiore a 0,3 sono stati considerati le cellule di superficie delle particelle legate positivi.
- Infine, per eliminare le cellule con l'etichettatura di fondo e di individuare specifiche nanoparticelle interiorizzate o batteri, Spot IDEAS funzionalità Conteggio è stato utilizzato. Numero di spot è una funzione che conta il numero di componenti collegati o maschere di piccole dimensioni di un'immagine. Le funzioni della maschera, Spot, Peak e intensità sono stati utilizzati per definire i punti. La maschera trova posto i dettagli luminosi in un'immagine che hanno un raggio specificato dall'utente e soglia al di sopra della realtà locale, la maschera è ingentilita da sbriciolarsi intensità elevate in singoli punti utilizzando la funzione Peak e poi macchie di sopra di un intensità di 200 conteggi sono stati inclusi . Consultare la guida Amnis mascheramento spot per ulteriori informazioni (Figura 1E). A re le statistiche modello di porto è stato generato, includendo le varie funzioni del menu Report.
- Questo file è stato salvato come file di modello da utilizzare per l'analisi di tutti i file in batch sperimentali.
- Nel software IDEAS, fare clic su Strumenti → dati batch file e inserire tutti i file con rif. Aggiungere il file matrice di compensazione (. CTM) e il file template (. Ast) nelle sezioni corrispondenti. Invia il lotto per l'elaborazione. Dopo la fase di trattamento, tutti i file vengono analizzati e rif. File daf vengono generati per ciascuno dei singoli file grezzi. Un file di relazione finale viene generato con le statistiche per tutti i campioni.
Consigli & note:
- Analisi delle immagini è stata effettuata utilizzando il software IDEAS versione 4.0 e la procedura guidata internalizzazione con alcune modifiche. La procedura guidata è auto-istruttivo e. Ouptput file di daf può essere generato seguendo le istruzioni della procedura guidata.
7. Risultati rappresentativi
"> Le immagini rappresentative in Figura 2 dimostrano che MIFC può essere utilizzato con successo per distinguere tra internalizzato (pannello a sinistra) rispetto superficie legata (pannello destro) NP (Figura 2A) o Salmonella (Figura 2B). Internalizzazione del NP e Salmonella sono stati ridotti sia inibendo actina e abbassando la temperatura a 4 ° C (Fig. 3A). La percentuale di cellule positive per superficie vincolata NP aumentata da circa 8% a 37 ° C a più del 35% dopo o-citocalasina D o 4 ° C trattamento ( Figura 3B). In contrasto, la percentuale di cellule con Salmonella superficie legato è stato ridotto dal 35% al 15% dopo citocalasina D-trattamento. incubazione di cellule RAW 264.7 con Salmonella a 4 ° C diminuito internalizzazione senza un evidente aumento della quantità di superficie vincolata batteri rispetto al 37 ° di controllo C. Insieme, questi dati dimostrano che Salmonella e NP vengono interiorizzate da un processo simile cellulare che richiede actina e dipende dalla temperatura. Inoltre, i dati indicano che l'attaccamento sostenuto di Salmonella di macrofagi richiede polimerizzazione actina.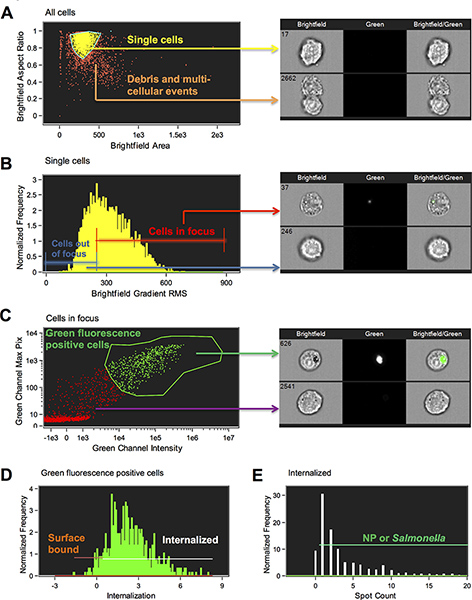
Figura 1. Schematica della strategia di gating utilizzato per determinare internalizzati rispetto nanoparticelle di superficie associati e Salmonella. (A) Per limitare l'analisi di cellule singole, è importante eliminare detriti e multi-cellulari eventi. Celle singole e farsetti erano separati da aggregati multicellulari che utilizzano l'area IDEAS caratteristiche e proporzioni dell'immagine brightfield (M01). Area è la dimensione dell'immagine in micron quadrati e rapporto di aspetto è l'asse minore diviso per l'asse maggiore e quindi una misura di circolarità (un cerchio perfetto avrà un rapporto di 1; doppietti tipicamente hanno asperapporti ct di circa 0.5 e aggregati multicellulari sono in genere inferiore a 0,5). Una regione è stata attirata da cancello su eventi singola cellula (Punto 6.6). (B) al cancello su cellule in-focus, le idee sono dotate di Gradient RMS dell'immagine brightfield è tracciata in un istogramma. La funzione gradiente RMS misura la qualità nitidezza di un'immagine rilevando le variazioni di valori di pixel nell'immagine. Un valore più elevato gradiente RMS indica un'immagine più mirata (Step 6.7). (C) di fluorescenza verde cellule positive sono state selezionate da gating sulle cellule con alti valori di pixel Max e intensità nel canale fluorescenza verde (Step 6.8). (D) Cellule con NP interiorizzata o Salmonella sono stati selezionati scegliendo la popolazione cellulare con un punteggio interiorizzazione uguale o superiore a 0,3. Le cellule che ricevono un punteggio inferiore a 0,3 sono stati considerati in superficie vincolata (Step 6.9). (E) celle del cancello "internalizzato" sono stati ulteriormente caratterizzati in base al numero di punti (NP o STM) perché There sono alcune cellule con colorazione di fondo che sono stati contati come internalizzata ma aveva un valore di punto zero (step 6,11). Immagini rappresentative sono presentate per ciascuna porta. Clicca qui per ingrandire la figura .
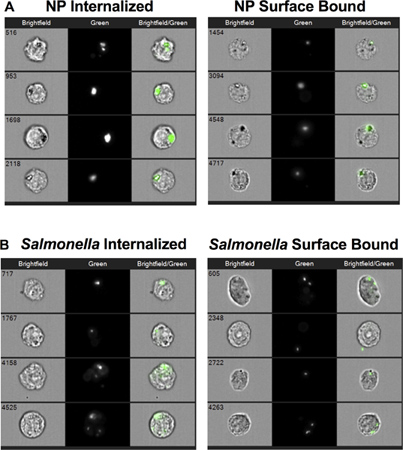
Figura 2. Immagini cellulari di nanoparticelle legate interiorizzate e di superficie (NP) o Salmonella. (A) immagini RAW 264.7 rappresentativi di cellule che interiorizzata NP (pannello a sinistra) e celle a cui NP erano legati alla loro superficie, ma non interiorizzata (pannello di destra). (B) rappresentativi di immagini RAW 264.7 cellule che Salmonella internalizzato (pannello sinistro) e cellule di Salmonella che sono stati legati alla loro superficie, ma non internalizzato (pannello destro).
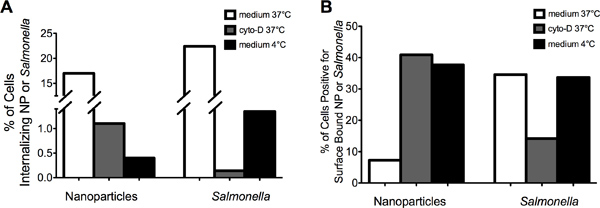
Figura 3. Cytochalail trattamento sin-D delle cellule inibito interiorizzazione di NP e Salmonella. (A) Pretrattamento di RAW 264.7 cellule con citocalasina-D o incubazione a 4 ° C riduce l'incidenza di internalizzazione NP o Salmonella rispetto al RAW 264.7 cellule incubate a 37 ° C in mezzo. RAW 264.7 cellule incubate in mezzo contenente DMSO (cioè, controllo del veicolo) hanno mostrato livelli internalizzazione simili per NP e Salmonella rispetto al medium da solo (dati non mostrati). (B) pretrattamento dei RAW 264.7 cellule con citocalasina-S è aumentata la percentuale di cellule con la superficie vincolata NP mentre diminuisce la percentuale di cellule con Salmonella superficie legato.
Discussione
Studi hanno dimostrato che le nanoparticelle biodegradabili a base di poli (lattico-co-glicolico (PLGA) o polianidridi possono essere usati per fornire antigeni incapsulati o farmaci alle cellule bersaglio. Assorbimento di tali nanoparticelle di cellule fagocitiche è importante per la loro efficacia, rendendo quantitativa . analisi di interiorizzazione critica nella progettazione di nuovi sistemi di consegna delle nanoparticelle Utilizzando questo metodo, assorbimento differenziale di nanoparticelle di vari tipi di cel...
Divulgazioni
Sherree amico L. è impiegato da Amnis Corporation, che produce il sistema X ImageStream.
Riconoscimenti
Gli autori desiderano ringraziare il ONR-MURI Award (NN00014-06-1-1176) e l'US Army Medical Research e Materiel Command (numeri di sovvenzione W81XWH-09-1-0386 e W81XWH-10-1-0806) per finanziarie sostenere.
Materiali
| Name | Company | Catalog Number | Comments |
| Nome del reattivo | Azienda | Numero di catalogo | Comments |
| RAW 264,7 linea cellulare | Type Culture Collection (ATCC) | TIB-71 | |
| Dulbeccòs Modified Eaglès Medium (DMEM) | Cellgro | 10-013-CV | |
| Siero fetale bovino | Atlanta Biologicals | S 11150 | Premium Grade |
| Glutamax | Gibco | 35050-061 | |
| HEPES | Gibco | 15630-080 | |
| Piastra da 24 pozzetti | TPP | 92024 | |
| Coltura cellulare Boccette | TPP | 90151 | |
| Cella scraper | TPP | 99002 | 24 cm |
| Salmonella enterica sierotipo Typhimurium | ATCC | 14028 | |
| BTX ECM630 Manipolatore cella Electro | BTX Harvard Apparatus | ||
| MOPS | Fisher Scientific | BP308 | |
| Soluzione salina tamponata (PBS) | Cellgro | 21-040-CV | |
| Ultrasuoni processore liquido | Misonix | S-4000 | |
| Citocalasina D- | Sigma-Aldrich, | C8273 | |
| Formaldeide | Polysciences | 04018 | |
| Tampone di lavaggio | 2% di FBS inattivato, sodio azide allo 0,1% in PBS. | ||
| Perm / tampone di lavaggio | BD Biosciences | 554714 | |
| Clear-visualizzare microprovette tappo a scatto | Sigma | T4816 | |
| Alexa Fluor 660 phalloidin | Invitrogen | A22285 | |
| ImageStream X | Amnis Corporazione | 100200 | Opzioni: 658nm laser, autocampionatore |
| Sodio azide | Fisher Scientific | S 227I-500 |
Riferimenti
- Ulery, B. D., Kumar, D., Ramer-Tait, A. E., Metzger, D. W., Wannemuehler, M. J., Narasimhan, B. Design of a protective single-dose intranasal nanoparticle-based vaccine platform for respiratory infectious diseases. PLoS One. 6, e17642 (2011).
- Kasturi, S. P., Skountzou, I., Albrecht, R. A., Koutsonanos, D., Hua, T., Nakaya, H. I., Ravindran, R., Stewart, S., Alam, M., Kwissa, M., Villinger, F., Murthy, N., Steel, J., Jacob, J., Hogan, R. J., García-Sastre, A., Compans, R., Pulendran, B. Programming the magnitude and persistence of antibody responses with innate immunity. Nature. 470, 543-547 (2011).
- Rice-Ficht, A. C., Arenas-Gamboa, A. M., Kahl-McDonagh, M. M., Ficht, T. A. Polymeric particles in vaccine delivery. Curr. Opin. Microbiol. 13, 106-112 (2010).
- Jain, J. P., Chitkara, D., Kumar, N. Polyanhydrides as localized drug delivery carrier: an update. Expert. Opin. Drug. Deliv. 5, 889-907 (2008).
- Pfeifer, B. A., Burdick, J. A., Little, S. R., Langer, R. Poly(ester-anhydride):poly(beta-amino ester) micro- and nanospheres: DNA encapsulation and cellular transfection. Int. J. Pharm. 304, 210-219 (2005).
- Ahmed, F., Friend, S., George, T. C., Barteneva, N., Lieberman, J. Numbers matter: quantitative and dynamic analysis of the formation of an immunological synapse using imaging flow cytometry. J. Immunol. Methods. 347, 79-86 (2009).
- Hampton, M. B., Winterbourn, C. C. Methods for quantifying phagocytosis and bacterial killing by human neutrophils. J. Immunol. Methods. 232, 15-22 (1999).
- Rieger, A. M., Hall, B. E., Barreda, D. R. Macrophage activation differentially modulates particle binding, phagocytosis and downstream antimicrobial mechanisms. Dev. Comp. Immunol. 34, 1144-1159 (2010).
- Murphy, K. C., Campellone, K. G. Lambda Red-mediated recombinogenic engineering of enterohemorrhagic and enteropathogenic E. coli. BMC. Mol. Biol. 4, 11 (2003).
- Karsi, A., Lawrence, M. L. Broad host range fluorescence and bioluminescence expression vectors for Gram-negative bacteria. Plasmid. 57, 286-295 (2007).
- Ulery, B. D., Phanse, Y., Sinha, A., Wannemuehler, M. J., Narasimhan, B., Bellaire, B. H. Polymer chemistry influences monocytic uptake of polyanhydride nanospheres. Pharm. Res. 26, 683-690 (2009).
- Doherty, G. J., McMahon, H. T. Mechanisms of endocytosis. Annu. Rev. Biochem. 78, 857-902 (2009).
- Vercauteren, D., Vandenbroucke, R. E., Jones, A. T., Rejman, J., Demeester, J., De Smedt, S. C., Sanders, N. N., Braeckmans, K. The use of inhibitors to study endocytic pathways of gene carriers: optimization and pitfalls. Mol. Ther. 18, 561-569 (2010).
- Di Marzio, L., Marianecci, C., Cinque, B., Nazzarri, M., Cimini, A. M., Cristiano, L., Cifone, M. G., Alhaique, F., Carafa, M. pH-sensitive non-phospholipid vesicle and macrophage-like cells: binding, uptake and endocytotic pathway. Biochim. Biophys. Acta. 1778, 2749-2756 (2008).
- Torres, M. P., Vogel, B. M., Narasimhan, B., Mallapragada, S. K. Synthesis and characterization of novel polyanhydrides with tailored erosion mechanisms. J. Biomed. Mater. Res. A. 76, 102-110 (2006).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon