Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Étiquetage spécifique de la séquence des acides nucléiques et des protéines avec Methyltransferases et le cofacteur Analogues
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
ADN et les protéines sont-séquence spécifiquement étiquetés avec une affinité ou groupes rapporteurs fluorescents à l'aide ADN ou de protéines méthyltransférases et analogues de cofacteur synthétiques. En fonction de la spécificité du cofacteur des enzymes, aziridine ou doubles analogues de cofacteurs activés sont utilisés pour l'étiquetage d'une ou deux étapes.
Résumé
-Adenosyl S-L-méthionine (SAM ou AdoMet) méthyltransférases dépendantes (MTAse) catalysent le transfert du groupe méthyle de l'AdoMet activé à des positions spécifiques dans l'ADN, l'ARN, les protéines et les petites biomolécules. Cette réaction de méthylation naturel peut être étendue à une grande variété de réactions d'alkylation utilisant des analogues de synthèse de cofacteurs. Remplacement du centre de sulfonium de AdoMet réactif avec un cycle aziridine conduit à des co-facteurs qui peuvent être couplés avec de l'ADN par diverses MTases d'ADN. Ces cofacteurs aziridine peuvent être équipés avec des groupes rapporteurs à différentes positions de la fraction de l'adénine et utilisés pour S spécifique M equence ethyltransferase- je nduced L abel ING de l'ADN (DNA Sourire). Comme exemple typique, nous donnons un protocole de biotinylation de l'ADN de plasmide pBR322 à la séquence 5'-ATCG A T-3 'avec l'ADN MTase M.BseCI et le cofacteur aziridine dans 6BAzune étape. Extension du groupe méthyle activé avec insaturés groupes alkyle résultats dans une autre classe d'analogues AdoMet qui sont utilisés pour m ethyltransferase dirigée de Transfert d'un G roupes ctivated (MTAG). Étant donné que les chaînes latérales longues sont activés par le centre de sulfonium et de la liaison insaturée, ces co-facteurs sont appelés analogues AdoMet double-activées. Ces analogues, non seulement fonction de cofacteurs de l'ADN, comme MTases les co-facteurs d'aziridine, mais aussi pour un ARN, de protéines et de petites molécules. MTases Ils sont généralement utilisés pour la modification enzymatique de substrats MTAse uniques avec des groupes fonctionnels qui sont marqués avec des groupes rapporteurs dans une deuxième étape chimique. Ceci est illustré dans un protocole de marquage par fluorescence de la protéine histone H3. Un petit groupe propargyle est transférée du cofacteur SeAdoYn analogue à la protéine par l'histone H3 lysine 4 (H3K4) MTase Set7 / 9 suivie d'étiquetage du clicalkynylated histone H3 avec TAMRA azoture. Étiquetage MTase médiation avec des analogues de cofacteur est une technologie habilitante pour de nombreuses applications intéressantes, dont l'identification et l'étude fonctionnelle des substrats MTASE ainsi que l'ADN de génotypage et de détection de la méthylation.
Introduction
Étiquetage spécifique d'acides nucléiques et de protéines 1,2 3,4 est d'un intérêt majeur pour les caractérisations fonctionnelles, le diagnostic médical et (nano) la biotechnologie. Nous présentons ici une méthode de marquage enzymatique pour ces biopolymères qui est basé sur S -adenosyl-L-méthionine (SAM ou AdoMet) méthyltransférases dépendantes (de MTases). Cette classe d'enzymes (EC 2.1.1.) Cible nucléophiles positions individuelles (azote, d'oxygène, de soufre et des atomes de carbone) à l'intérieur des résidus spécifiques d'acides nucléiques et de protéines et transfère le groupe méthyle activé de l'AdoMet cofacteur (figure 1A) 5 naturellement. En outre, MTases peuvent utiliser des analogues synthétiques de cofacteurs pour un étiquetage spécifique avec des étiquettes d'affinité, des fluorophores ou d'autres étiquettes (figure 1B) 6. Deux classes d'analogues AdoMet ont été développés: cofacteurs aziridine pour S equence spécifique M ethyltransferase- I Nduced L abel ING (Sourire) 7 et doubles analogues AdoMet activés pour m ethyltransferase dirigée de Transfert d'un G roupes ctivated (MTAG) 8.

Figure 1: Réactions catalysées par des méthyltransférases (MTases) de transfert de groupe A. méthyle à partir du cofacteur naturel AdoMet (SAM) sur divers substrats, y compris l'ADN, l'ARN, les protéines et les petites biomolécules B. Identification / fonctionnalisation des acides nucléiques et des protéines (NNNNN =.. paires de bases de l'ADN, des nucleotides de l'ARN et des acides aminés pour les protéines; XXXXX = séquence de reconnaissance de la MTase résidu avec de cible en vert) avec des analogues de synthèse de cofacteurs. Cofacteurs aziridine contenant un groupe rapporteur (sphère bleue)fixé au noyau adenine sont séquence spécifique couplé avec le résidu cible (à gauche) et analogues doubles activé AdoMet conduit au transfert de chaînes alkyle étendues portant un reporter chimique Y (à droite) qui peut être marqué par bioorthogonal clic réaction dans une seconde étape. Se il vous plaît cliquer ici pour voir une version plus grande de cette figure.
Cofacteurs aziridine fonctionnent mieux avec des ADN MTases. Ils contiennent un cycle à trois à un atome d'azote 9 (ou un N de moutarde 10,11) à la place du centre de sulfonium en tant que groupe réactif. La protonation de cet atome d'azote active le cycle aziridine pour une attaque nucléophile par le nucléotide cible qui entraîne le couplage covalent de l'ensemble avec de l'ADN cofacteur. En attachant des groupes rapporteurs à l'anneau de l'adénine les cofacteurs d'aziridine peuvent être utilisés en combinaison avec de l'ADN pour marquer MTases ADN en une étape ( g> Figure 1B, de gauche) 7,12. Cela est démontré en détail pour la biotinylation de l'ADN avec 6BAz 13-15 (cofacteur aziridine avec de la biotine attachée à la position 6 du cycle adénine) et l'ADN spécifique à l'adénine MTase de Bacillus stearothermophilus (M.BseCI) 16 (Figure 2, voir la section 2 du protocole: étiquetage en une seule étape de l'ADN via aziridine cofacteurs). En plus de M.BseCI («séquence de reconnaissance, l'ADN MTases à partir de Thermus aquaticus (M.TaqI, 5'-TCG A -3 5'-ATCG Un T-3) '), à partir de Haemophilus heamolyticus (M.HhaI, 5 »-G C GC-3 '), et de Spiroplasma (M.SssI, 5'-C G-3') ont été utilisés avec succès pour biotinyler ADN avec 6BAz 17. En outre, des cofacteurs aziridine peuvent être utilisés pour une étape de marquage d'ADN par fluorescence 18,19.
ontenu "fo: keep-together.within-page =" always ">
Figure 2:. Séquence spécifique à une étape de biotinylation avec M.BseCI ADN et l'ADN MTase 6BAz M.BseCI reconnaît la séquence d'ADN double brin 5'-ATCG A T-3 'et méthylation du groupe amino de la seconde adénine naturellement résidu (vert) en utilisant AdoMet. Avec le cofacteur aziridine 6BAz le cours de la réaction est changé et M.BseCI conduit à séquencer biotinylation spécifique d'ADN en couplant l'ensemble cofacteur y compris biotine (bleu) avec l'adénine cible. Se il vous plaît cliquer ici pour voir une version plus grande de cette figure.
Double analogues activés AdoMet contient étendues de chaînes latérales insaturées au lieu d'un groupe méthyle au centre de sulfonium (figure 1B , droite) 20. Le double ou triple liaison insaturée dans β-position pour le centre de sulfonium compense électroniquement effets stériques défavorables au sein de l'état de transition par la stabilisation de conjugaison. Étant donné que le centre de sulfonium et activent la liaison insaturée de la chaîne latérale pour le transfert enzymatique, ces co-facteurs ont été nommés analogues AdoMet double-activées. Typiquement, ils sont utilisés pour transférer des chaînes latérales avec des groupes uniques chimiques (reporters chimiques), comme les groupes amino, alcyne et azoture, par chimio-sélective étiquetage dans une deuxième étape 8,21. En général, les analogues doubles activé AdoMet peut non seulement fonctionner comme cofacteurs pour l'ADN MTases 8,20,21 mais aussi pour l'ARN MTases 22,23 et MTases de protéines de 24 à 28 permettant étiquetage supplémentaire de l'ARN et des protéines. Cependant, les longues chaînes latérales sont stériquement plus exigeant que un groupe méthyle et en élargissant les sites actifs MTASE par ingénierie des protéines est souventfr nécessaire pour obtenir des taux de transfert efficaces. Une autre solution à ce problème est d'utiliser un analogue AdoMet avec un petit groupe de propargyle (trois carbones) où l'alcyne terminale sert deux fonctions: 1. La stabilisation de l'état de transition pendant le transfert enzymatique et 2. poignée réactif pour la suite de modifications chimiques de cuivre catalysée cycloaddition azoture-alcyne (CuAAC) click chemistry. Il se est avéré que le propargylique résultant AdoMet analogique 29 est assez instable dans des conditions neutres ou légèrement basiques et seulement d'une utilité limitée. Cet inconvénient peut être résolu en remplaçant l'atome de soufre par le sélénium. Le cofacteur résultant 5 '- [(Se) [(3 S) -3-amino-3-carboxypropyle] prop-2-ynylselenonio] -5'-désoxyadénosine (SeAdoYn, figure 3) est acceptée par l'ADN de type sauvage, l'ARN MTases et de protéines de 30 à 32 qui abroge la nécessité d'ingénierie des protéines dans de nombreux cas. Ceci est illustré par fluorescence pro étiquetage protéine avec l'histone H3 lysine 4 (H3K4) MTase Set7 / 9 33 (Figure 3, voir la section de protocole 3: Deux étapes étiquetage de protéine via doubles cofacteurs activés).
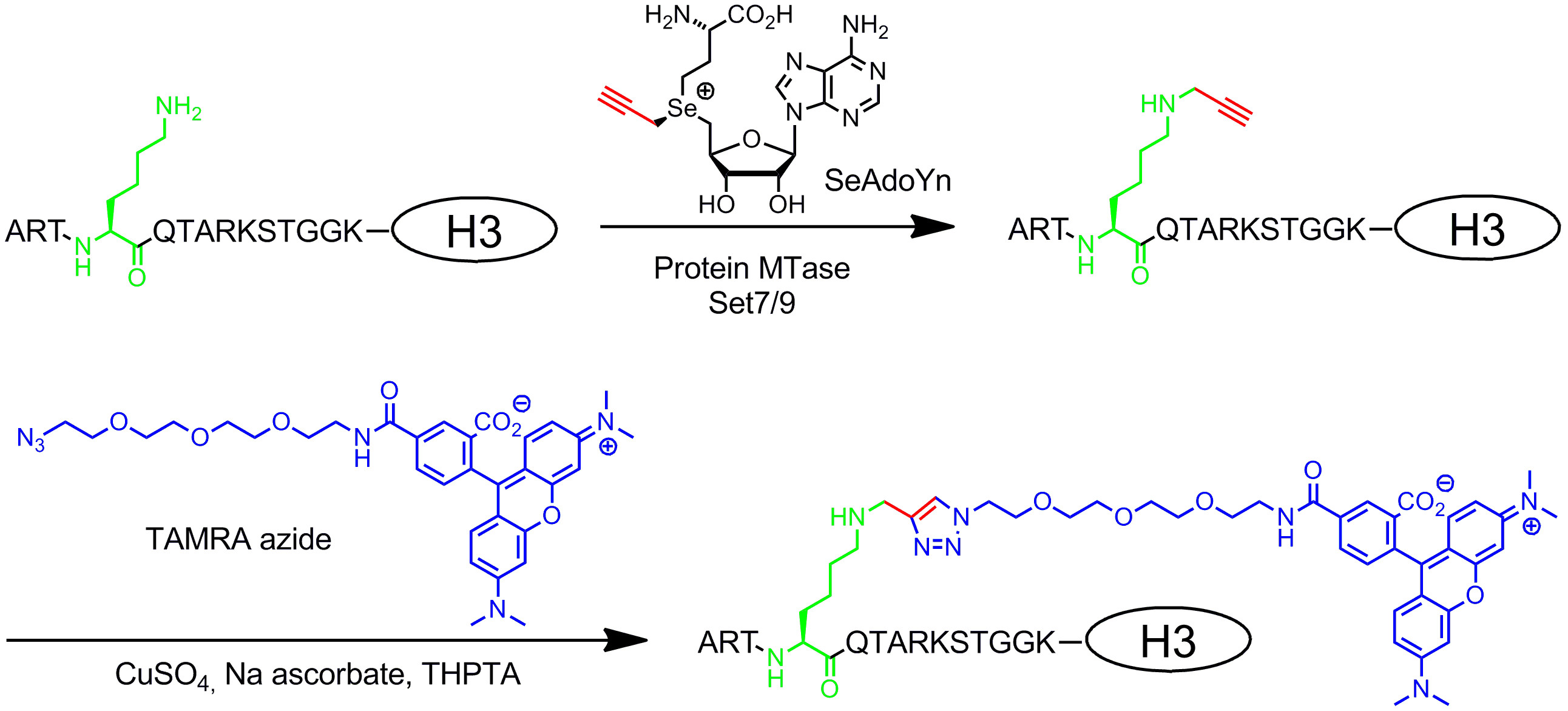
Figure 3:. Spécifique de séquence à deux étapes marquage par fluorescence de l'histone H3 avec Set7 / 9, SeAdoYn et TAMRA azide La protéine MTase Set7 / 9 méthylation naturellement le groupe amino de la lysine 4 de l'histone H3 (H3K4, vert) à l'aide AdoMet. Avec le cofacteur double-activé SeAdoYn l'MTase transfère un petit groupe propargyle (rouge) au résidu de lysine. Le triple liaison terminal connecté est alors modifiée sélectivement en un clic réaction bioorthogonal (cuivre-catalysée azoture-alcyne cycloaddition, CuAAC) avec l'azoture-dérivé TAMRA (tétraméthylrhodamine, bleu) fluorophore.charge / 52014 / 52014fig3highres.jpg "target =" _ blank "> Se il vous plaît, cliquez ici pour voir une version plus grande de cette figure.
Protocole
1. Instructions générales
- aziridine de magasin cofacteur 6BAz (dans du DMSO) et de la protéine MTase Set7 / 9 à -80 ° C et tous les autres réactifs, y compris cofacteur double-activé et SeAdoYn ADN MTase M.BseCI (dans 50% de glycerol) à -20 ° C.
- Déterminer la concentration de 6BAz et SeAdoYn par spectroscopie UV / VIS à l'aide de 269nm (6BAz) = 16,000 cm -1 M -1 et ε 260nm (SeAdoYn) = 15,400 cm -1 M de coefficients d'extinction -1 dans l'eau déminéralisée. Déterminer la concentration de MTases par le dosage de Bradford ou, si le coefficient d'extinction est disponible, par absorption directe à 280 nm.
- Essayez d'éviter de créer des bulles par la pipette ou vortex intensive pour éviter la perte de l'activité enzymatique. Au lieu de cela, mélanger en pipetant doucement monter et descendre.
- Lors de l'ajout de cofacteurs aziridine partir de solutions mères dans du DMSO se assurer que la concentration finale de DMSO dans le dosage est moinsà 5%. Toujours inclure des ions de magnésium 10 mM dans le tampon d'essai pour empêcher des réactions non spécifiques avec l'ADN.
- Lors de l'ajout doubles cofacteurs activés partir de solutions mères acides utiliser de petits volumes (solutions mères très concentrés) pour éviter les changements de pH et de se assurer que le pH de la solution de dosage ne change pas de façon significative. Eviter les thiols, par exemple, β-mercaptoéthanol ou le dithiothréitol (DTT), dans le tampon de dosage, car ils peuvent interférer avec la réaction clic par complexation des ions de cuivre est requise.
2. Une étape marquage de l'ADN via AZIRIDINE cofacteurs
- Methyltransferase induite label ING spécifiques de séquence (Sourire) de l'ADN plasmidique avec M.BseCI ADN MTase et aziridine cofacteur 6BAz.
- Décongeler la solution de cofacteurs à 20 ° C et préparer les mélanges réactionnels sur de la glace.
- En plus du dosage effectuer un contrôle "cofacteur", pour visualiser les modifications non spécifiques, et un & #8220; enzyme "le contrôle, pour se assurer que la préparation MTase est libre de la AdoMet cofacteur naturel.
- Pour le dosage mélange 2 pi de 10 x tampon de modification (contenant 100 mM de Tris-HCl, 100 mM de MgCl2, 20 mM de β-mercaptoéthanol, pH 7,4), 2 ul de pBR322 (0,5 ug / ul), 10 éq. M.BseCI par séquence de reconnaissance sur l'ADN (séquence de reconnaissance dans une pBR322) et le cofacteur aziridine 6BAz à une concentration finale de 60 uM dans un volume total de 20 ul. Ajouter cofacteur et l'ADN MTase dernière.
REMARQUE: β-mercaptoéthanol est toxique, corrosif et dommageables pour l'environnement. - Pour le contrôle "cofacteur" ajouter de l'eau déminéralisée à la place de M.BseCI et pour le contrôle "de l'enzyme" ajouter de l'eau déminéralisée à la place de 6BAz.
- Mélanger les solutions en pipetant doucement monter et descendre.
- Incuber les tubes à 55 ° C pendant 1 heure.
- Centrifuger brièvement pour collecter tout liquide au fond des tubes.
- Test de restriction-modification de vérifier modification de l'ADN.
- Préparer une solution en mélangeant 10 pi de tampon 10x R.TaqI (contenant 100 mM de Tris-HCl, 50 mM de MgCl2, 1 M de NaCl, 1 mg / ml d'albumine de sérum bovin, pH 8,0), 80 pi d'eau désionisée et 3,3 ul de la endonucléase de restriction (Rease) à partir de Thermus aquaticus (R.TaqI, 10 U / pl). Assurez-vous d'ajouter le Rease dans la dernière étape.
- A chaque tube de 2.1.7 ajouter 2 ul de tampon 10X Taq I et R. 28 ul de la solution ci-dessus à partir de (2.2.1).
- Mélanger les solutions en pipetant doucement monter et descendre.
- Incuber les tubes à 65 ° C pendant 30 min.
- Centrifuger brièvement pour collecter tout liquide au fond des tubes.
- Électromobilité dosage de décalage (EMSA) avec de la streptavidine pour vérifier modification fonctionnelle.
- Retirer 25 pi de chaque tube (2.2.5) et ajouter 2,4 ul d'une solution de streptavidine (1 mM par rapport à la streptavidine monomer dans un tampon de streptavidine contenant 100 mM de Na 2 HPO 4, NaCl 100 mM, pH 7,5; 4 équivalents de biotine total). Ajouter 2,4 pi de tampon de streptavidine dans les tubes restants.
- Incuber tous les tubes à 37 ° C pendant 1 heure.
- L'analyse par électrophorèse sur gel d'agarose.
- Ajouter 5 ul de tampon 6x de chargement (0,25% de bleu de bromophénol, 30% de glycerol) à chaque tube.
- Mélanger délicatement les solutions.
- Charge 10 pi de chaque échantillon dans les puits d'un gel d'agarose (1% d'agarose dans un tampon 0,5x TBE contient 1x GelRed partir d'une solution 10.000 fois de stock).
- Exécutez le gel dans du tampon TBE 0,5x avec 80 V env. 1 h.
- Visualiser les bandes d'ADN sur une table UV (312 nm) avec une caméra CCD équipé d'un filtre (540 ± 50 nm).
REMARQUE: la lumière UV est dommageable pour les yeux et la peau.
3. Deux étapes Protein Labeling via cofacteurs Double Activé
- MethyltransfTransfert de groupes activés (MTAG) avec Set7 / 9 et le cofacteur double-SeAdoYn activé pour histone H3 lysine 4 étiquetage (étape de modification) l'effacement Réalisé.
- Décongeler les composants et préparer les mélanges de réaction sur de la glace. REMARQUE: Toujours garder SeAdoYn refroidi pour éviter la dégradation.
- En plus du dosage effectuer un contrôle "cofacteur", pour visualiser les modifications non spécifiques, et une commande "enzyme", d'exclure les réactions non-spécifiques de la sonde fluorescente.
- Préparer une solution d'essai (20 ul) contenant du tampon de modification (50 mM Tris-HCl, 5% de glycerol, pH 8,5), 10 uM d'histone H3, 10 uM Set7 / 9 et 600 uM SeAdoYn (mélange des deux épimères en sélénium). Dans les dernières étapes ajouter cofacteur puis MTase.
- Pour le contrôle "cofacteur" préparer une solution de dosage en 3.1.3 et ajouter 60 mM AdoMet de rivaliser avec le cofacteur synthétique. Pour le contrôle "de l'enzyme" ajouter de l'eau déminéralisée à la place de SeAdoYn.
- Mélanger les solutions en pipettant doucement de haut en bas. Vérifier le pH en ajoutant 1 ul de chaque solution dans le domaine supérieur d'une bande de pH (plage de pH 5-10).
- Incuber à 37 ° C pendant 2 heures.
- En attendant, le gel préparer un gel à 12% de polyacrylamide-SDS (course: Bis-Tris 357 mM, pH 6.5 à 6.8, 0,1% (p / v) APS, 0,04% (v / v) de TEMED et 12% d'acrylamide / bisacrylamide 37,5: 1 ; gel de chargement: 357 mM de Bis-Tris, pH 6.5 à 6.8, 0,1% (p / v) APS, 0,04% (v / v) de TEMED et 5% d'acrylamide / bisacrylamide 37,5: 1).
NOTE: L'acrylamide / bisacrylamide est toxique et dangereux pour la santé. Porter des gants lors de cette procédure.
- Marquage chimique de la lysine dans alkinylated 4 histone H3 par cycloaddition de l'azide-alcyne (CuAAC) (étape de marquage) catalysée par le cuivre.
- Juste avant la fin de la réaction de modification préparer un clic 5x mélange contenant 3 mM de CuSO 4, 3 mM de tris (3-hydroxypropyl-triazolylméthyl) amine (THPTA), l'ascorbate de sodium 250 mM et 6 mM d'azide avec un TAMRAvolume total de 20 ul.
- Ajouter 5 ul de la 5x clic mélange fraîchement préparé dans chaque tube pour démarrer le CuAAC et éteindre la réaction de modification.
- Mélanger doucement en pipetant.
- Protéger tous les tubes avec une feuille d'aluminium de la lumière pour éviter photo-blanchiment du fluorophore.
- Incuber à 20 ° C pendant 1 heure.
- La précipitation des protéines pour enlever l'excès de la libre fluorophore TAMRA.
- Pour éviter resplendissement de l'histone H3 marqué fluorescent par intensive fluorescence dans-gel de la libre fluorophore TAMRA, enlever l'excès de fluorophore par précipitation des protéines (3.3.2 - 3.3.4) 34.
- Ajouter 75 ul méthanol, 18,8 pi chloroforme et 50 pi d'eau déminéralisée à chaque tube et vortex brièvement après chaque addition. Centrifuger à 16 000 g pendant 5 min. Retirer la phase supérieure sans perturber la couche d'interface, qui contient la protéine.
- Ajouter 56,3 pi méthanol à la phase restante in chaque tube vortex et centrifuger à 16 000 xg pendant 5 min pour sédimenter les protéines. Eliminer le surnageant. Répétez cette étape pour laver le culot.
- Couvrir les tubes ouverts avec un tissu non pelucheux et laissez-les sécher pendant 15 à 30 min.
- Analyse par SDS PAGE.
- Dissoudre les protéines précipitées du 3.3.4 dans 20 ul de tampon de charge SDS (50 mM de Tris-HCl, 2,5% (p / v) de SDS, 10% (v / v) de glycerol, 320 mM de β-mercaptoéthanol et 0,05% (p / v) de bleu de bromophénol, pH 6,8). Assurez-vous de dissoudre complètement la pastille en rinçant les parois des tubes avec une pipette.
- Incuber les échantillons à 95 ° C pendant 10 min et laisser refroidir à 20 ° C.
- Centrifuger brièvement pour collecter tout liquide au fond des tubes.
- Charger la totalité du montant de chaque échantillon dans les puits d'un gel de polyacrylamide SDS (3.1.7). Utiliser 50 mM MOPS, 50 mM de Tris-X (Tris-base), EDTA 5 mM, 0,1% (p / v) de SDS comme tampon pour électrophorèse.
- Exécutez le gel avec 120 V pendant env. 90 min.
- Visualiser la fluorescence dans le gel sur une table UV (312 nm) avec une caméra CCD équipé d'un filtre (540 nm ± 50 nm).
REMARQUE: la lumière UV est dommageable pour les yeux et la peau.
Résultats
Une étape marquage de l'ADN via AZIRIDINE cofacteurs
Cet exemple réaction est effectuée avec l'ADN MTase M.BseCI, qui modifie le second résidu d'adénine dans le A T-3 'la séquence à double brin 5'-ATCG et a un site de reconnaissance sur le plasmide pBR322 (figure 4A). Pour tester l'étiquetage du plasmide pBR322 est contestée par l'endonucléase de restriction (Rease) R.TaqI (5'-TCGA-3 '). R.TaqI possède ...
Discussion
Étiquetage en une seule étape de l'ADN avec MTases d'ADN et des cofacteurs d'aziridine (ADN Sourire) est une méthode robuste mais certains aspects doivent être considérés lors de la planification de l'expérience.
Cofacteur aziridine: La concentration 6BAz pour l'étiquetage d'ADN avec M.BseCI était de 60 uM. Lors de l'utilisation d'autres MTases ADN la concentration de cofacteur doit être optimisée, par exemple des concentrations au...
Déclarations de divulgation
The authors disclose the following competing financial interest: E.W. is inventor on related patents.
Remerciements
The authors thank Kerstin Glensk for preparing the MTases M.BseCI and Set7/9 and gratefully acknowledge funding by the Excellence Initiative of the German Federal and State Governments and RWTH Aachen University. The authors are happy to provide 6BAz and SeAdoYn or other cofactor analogues for collaborative research.
matériels
| Name | Company | Catalog Number | Comments |
| 6BAz | Synthesized according to Weinhold et al., Patent number US 8,129,106, published March 6, 2012. | ||
| β-Mercaptoethanol | Serva | 28625 | |
| Acetic acid | Fisher Scientific | 10304980 | |
| Acrylamide/Bis Solution, 37.5:1 | Serva | 10688 | |
| UltraPure Agarose | Invitrogen | 16500100 | |
| Ammonium persulfate (APS) | Serva | 13375 | |
| Bis-Tris | Gerbu | 1304 | |
| Boric acid | Gerbu | 1115 | |
| Bromophenol blue Na salt | Serva | 15375 | |
| Copper(II) sulfate | Aldrich | C1297 | |
| Chloroform | Fisher Scientific | 10020090 | |
| Coomassie Brilliant Blue | Serva | 17525 | |
| EDTA disodium salt | Gerbu | 1034 | |
| Ethanol | Merck | 100983 | |
| GelRed (10,000x in water) | Biotium | 41003 | |
| Glycerol (99.5%) | Gerbu | 2006 | |
| FastRuler Low Range DNA Ladder | Thermo Scientific | SM1103 | |
| Histone H3 | Expression plasmid obtained from Dr. Philipp Voigt and Prof. Danny Reinberg; expression and isolation according to T. J. Richmond et al., J. Mol. Biol. 1997, 272, 301-311. | ||
| M.BseCI | Expression plasmid obtained from Dr. Michael Kokkinidis; expression and isolation according to Kapetaniou et al., Acta Cryst. 2006, F63, 12-14. | ||
| Methanol | Fisher Scientific | 10675112 | |
| Magnesiumchloride hexahydrate | J.T. Baker | 4003 | |
| MOPS | Gerbu | 1081 | |
| Sodium chloride | Gerbu | 1112 | |
| pH strip (Neutralit) | Merck | 1,095,330,001 | |
| pBR322 | Thermo Scientific | SD0041 | |
| R.TaqI (10 u/µl) | Thermo Scientific | ER0671 | |
| SeAdoYn | Synthesized according to Willnow et al., ChemBioChem 2012, 13, 1167-1173. | ||
| Set7/9 | Expression plasmid obtained from Prof. Danny Reinberg, expression and isolation according to D. Reinberg et al., Genes Dev.2002, 16, 479-489. | ||
| Streptavidin | Gerbu | 3058 | |
| (+)-Sodium L-ascorbate | Sigma Life Science | A7631 | |
| SDS Granular | Gerbu | 1833 | |
| di-Sodium hydrogenphosphate | Merck | 106,586 | |
| TAMRA azide | Synthesized according to reference 30: Willnow et al., ChemBioChem 2012, 13, 1167-1173. | ||
| TaqI buffer (10x) | Thermo Scientific | B28 | |
| N,N,N',N'-Tetramethylethylenediamine (TEMED) | Acros Organics | 42058 | |
| Tris-HCl | Gerbu | 1028 | |
| Tris-X (TRIS-base) | Gerbu | 1018 | |
| Tris(3-hydroxypropyltriazolyl-methyl)amine (THPTA) | Sigma-Aldrich | 762342 |
Références
- Gottfried, A., Weinhold, E. Sequence-specific covalent labelling of DNA. Biochem. Soc. Trans. 39, 623-628 (2011).
- Zohar, H., Muller, S. J. Labeling DNA for single-molecule experiments: methods of labeling internal specific sequences on double-stranded DNA. Nanoscale. 3, 3027-3039 (2011).
- Hinner, M. J., Johnsson, K. How to obtain labeled proteins and what to do with them. Curr. Opin. Biotechnol. 21, 766-776 (2010).
- Wua, Y. -. W., Goody, R. S. Probing protein function by chemical modification. J. Pept. Sci. 16, 514-523 (2010).
- Struck, A. -. W., Thompson, M. L., Wong, L. S., Micklefield, J. S-Adenosyl-methionine-dependent methyltransferases: Highly versatile enzymes in biocatalysis, biosynthesis and other biotechnological applications. ChemBioChem. 13, 2642-2655 (2012).
- Klimasauskas, S., Weinhold, E. A new tool for biotechnology: AdoMet-dependent methyltransferases. Trends Biotechnol. 25, 99-104 (2007).
- Pljevaljcic, G., Schmidt, F., Weinhold, E. Sequence-specific Methyltransferase-Induced Labeling of DNA (SMILing DNA). ChemBioChem. 5, 265-269 (2004).
- Lukinavicius, G., Lapiene, V., Stasevskij, Z., Dalhoff, C., Weinhold, E., Klimasauskas, S. Targeted labeling of DNA by methyltransferase-directed Transfer of Activated Groups (mTAG). J. Am. Chem. Soc. 129, 2758-2759 (1021).
- Pignot, M., Siethoff, C., Linscheid, M., Weinhold, E. Coupling of a nucleoside with DNA by a methyltransferase. Angew. Chem. Int. Ed. 37, 2888-2891 (1998).
- Weller, R. L., Rajski, S. R. Design, synthesis, and preliminary biological evaluation of a DNA methyltransferase-directed alkylating agent. ChemBioChem. 7, 243-245 (2006).
- Du, Y., Hendrick, C. E., Frye, K. S., Comstock, L. R. Fluorescent DNA Labeling by N-Mustard Analogues of S-adenosyl-l-methionine. ChemBioChem. 13, 2225-2233 (2012).
- Pljevaljcic, G., Schmidt, F., Scheidig, A. J., Lurz, R., Weinhold, E. Quantitative labeling of long plasmid DNA with nanometer precision. ChemBioChem. 8, 1516-1519 (1002).
- Wilkinson, S., et al. Molecular scale architecture: engineered three- and four-way junctions. Bioconjugate Chem. 19, 470-475 (2008).
- Braun, G., et al. Enzyme-directed positioning of nanoparticles on large DNA templates. Bioconjugate Chem. 19, 476-479 (2008).
- Kim, S., et al. Enzymatically incorporated genomic tags for optical mapping of DNA binding proteins. Chem. Int. Ed. 51, 3578-3581 (2012).
- Rina, M., Bouriotis, V. Cloning purification and characterization of the BseCI DNA methyltransferase from Bacillus stearothermophilus. Gene. 133, 91-94 (1993).
- Weinhold, E., Meier, T., Düfel, H., Markert-Hahn, C., Schmuck, R. Sequence-specific detection of methylation in biomolecules. US Patent. , (2012).
- Pljevaljcic, G., Pignot, M., Weinhold, E. Design of a new fluorescent cofactor for DNA methyltransferases and sequence-specific labeling of DNA. J. Am. Chem. Soc. 125, 3492-3410 (2003).
- Schmidt, F. H. -. G., Hüben, M., Gider, B., Renault, F., Teulade-Fichou, M. -. P., Weinhold, E. Sequence-specific Methyltransferase-Induced Labelling (SMILing) of plasmid DNA for studying cell transfection. Bioorg. Med. Chem. 16, 40-48 (2008).
- Dalhoff, C., Lukinavicius, G., Klimasauskas, S., Weinhold, E. Direct transfer of extended groups from synthetic cofactors by DNA methyltransferases. Nat. Chem. Biol. 2, 31-32 (2006).
- Lukinavicius, G., Tomkuviene, M., Masevicius, V., Klimasauskas, S. Enhanced chemical stability of AdoMet analogues for improved methyltransferase-directed labeling of DNA. ACS Chem. Biol. 8, 1134-1139 (2013).
- Motorin, Y., et al. Expanding the chemical scope of RNA:methyltransferases to site-specific alkynylation of RNA for click labeling. Nucleic Acids Res. 39, 1943-1952 (1943).
- Schulz, D., Holstein, J. M., Rentmeister, A. A chemo-enzymatic approach for site-specific modification of the RNA cap. Angew. Chem. Int. Ed. 52, 7874-7878 (2013).
- Peters, W., et al. Enzymatic site-specific functionalization of protein methyltransferase substrates with alkynes for click labeling. Angew. Chem. Int. Ed. 49, 5170-5173 (2010).
- Islam, K., Zheng, W., Yu, H., Deng, H., Luo, M. Expanding cofactor repertoire of protein lysine methyltransferase for substrate labeling. ACS Chem. Biol. 6, 679-684 (2011).
- Wang, R., Zheng, W., Yu, H., Deng, H., Luo, M. Labeling substrates of protein arginine methyltransferase with engineered enzymes and matched S-adenosyl-l-methionine analogues. J. Am. Chem. Soc. 133, 7648-7651 (2011).
- Islam, K., et al. Bioorthogonal profiling of protein methylation using azido derivative of S-adenosyl-l-methionine. J. Am. Chem. Soc. 134, 5909-5915 (2012).
- Islam, K., et al. Defining efficient enzyme-cofactor pairs for bioorthogonal profiling of protein methylation. Proc. Natl. Acad. Sci. U.S.A. 110, 16778-16783 (2013).
- Binda, O., Boyce, M., Rush, J. S., Palaniappan, K. K., Bertozzi, C. R., Gozani, O. A chemical method for labeling lysine methyltransferase substrates. ChemBioChem. 12, 330-334 (2011).
- Willnow, S., Martin, M., Lüscher, B., Weinhold, E. A selenium-based click AdoMet analogue for versatile substrate labeling with wild-type protein methyltransferases. ChemBioChem. 13, 1167-1173 (2012).
- Bothwell, I. R., et al. Se-Adenosyl-l-selenomethionine cofactor analogue as a reporter of protein methylation. J. Am. Chem. Soc. 134, 14905-14912 (2012).
- Tomkuviene, M., Clouet-d’Orval, B., Cerniauskas, I., Weinhold, E., Klimasauskas, S. Programmable sequence-specific click-labeling of RNA using archaeal box C/D RNP methyltransferases. Nucleic Acids Res. 40, 6765-6773 (2012).
- Nishioka, K., et al. Set9, a novel histone H3 methyltransferase that facilitates transcription by precluding histone tail modifications required for heterochromatin formation. Genes Dev. 16, 479-489 (2002).
- Clark, P. M., et al. Direct in-gel fluorescence detection and cellular imaging of O-GlcNAc-modified proteins. J. Am. Chem. Soc. 130, (2008).
- Lukinavicius, G., Lapinaite, A., Urbanaviciute, G., Gerasimaite, R., Klimasauskas, S. Engineering the DNA cytosine-5 methyltransferase reaction for sequence-specific labeling of DNA. Nucleic Acids Res. 40, 11594-11602 (2012).
- Neely, R. K., Dedecker, P., Hotta, J., Urbanaviciute, G., Klimasauskas, S., Hofkens, J. DNA fluorocode: A single molecule, optical map of DNA with nanometre resolution. Chem. Sci. 1, 453-460 (2010).
- Roberts, R. J., Vincze, T., Posfai, J., Macelis, D. REBASE-a database for DNA restriction and modification: enzymes, genes and genomes. Nucleic Acids Res. 38, 234-236 (2010).
- Petrossian, T. C., Clarke, S. G. Uncovering the human methyltransferasome. Mol. Cell. Proteomics. 10, 1-12 (2011).
- Kriukiene, E., et al. DNA unmethylome profiling by covalent capture of CpG sites. Nat. Commun. 4, 2190 (2013).
- Wang, R., et al. Profiling genome-wide chromatin methylation with engineered posttranslation apparatus within living cells. J. Am. Chem. Soc. 135, 1048-1056 (2013).
- Zhang, C., Weller, R. L., Thorson, J. S., Rajski, S. R. Natural product diversification using a non-natural cofactor analogue of S-adenosyl-l-methionine. J. Am. Chem. Soc. 128, 2760-2761 (2006).
- Stecher, H., et al. Biocatalytic Fiedel-Crafts alkylation using non-natural cofactors. Angew. Chem. Int. Ed. 48, 9546-9548 (2009).
- Lee, B. W. K., Sun, H. G., Zang, T., Kim, B. J., Alfaro, J. F., Zhou, Z. S. Enzyme-catalyzed transfer of a ketone group from an S-adenosylmethionine analogue: A tool for the functional analysis of methyltransferases. J. Am. Chem. Soc. 132, 3642-3643 (2010).
- Winter, J. M., et al. Expanding the structural diversity of polyketides by exploring the cofactor tolerance of an inline methyltransferase domain. Org. Lett. 15, 3774-3777 (2013).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon