Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Mesure de la stabilité des protéines dans le salon embryons de poissons zèbres en utilisant la fluorescence Decay Après Photoconversion (FDAP)
Dans cet article
Résumé
Les teneurs en protéines dans les cellules et les tissus sont souvent étroitement réglementées par l'équilibre de la production de protéines et de dégagement. En utilisant la fluorescence Decay Après Photoconversion (FDAP), la cinétique de dégagement de protéines peuvent être mesurées expérimentalement in vivo.
Résumé
la stabilité des protéines influence de nombreux aspects de la biologie, et de mesurer la cinétique de libération de protéines peut fournir des informations importantes sur les systèmes biologiques. Dans les expériences FDAP, la clairance de protéines dans les organismes vivants peut être mesurée. Une protéine d'intérêt est marquée avec une protéine fluorescente photoconvertible, exprimée in vivo et photoconverted, et la diminution du signal dans le temps photoconverted est surveillée. Les données sont ensuite muni d'un modèle d'autorisation approprié pour déterminer la demi-vie de la protéine. Fait important, la cinétique de libération des populations de protéines dans les différents compartiments de l'organisme peuvent être examinées séparément en appliquant des masques à compartiments. Cette approche a été utilisée pour déterminer les demi-vies intra- et extracellulaires de protéines de signalisation sécrétées pendant le développement du poisson zèbre. Ici, nous décrivons un protocole pour des expériences FDAP dans embryons de poisson zèbre. Il devrait être possible d'utiliser FDAP pour déterminer la cinétique de libération detoute protéine tagable dans tout organisme optiquement accessible.
Introduction
Les niveaux de protéines dans des cellules et des organismes sont déterminés par leurs taux de production et de dégagement. Protéines demi-vie peut varier de quelques minutes à 1-4 jours. Dans de nombreux systèmes biologiques, la stabilisation ou la clairance de protéines clés a des effets importants sur l'activité cellulaire. La modulation de stabilité de la protéine intracellulaire est nécessaire pour la progression du cycle cellulaire de 5,6, la signalisation du développement 7-9, 10 l'apoptose, et la fonction normale et le maintien des neurones 11,12. La stabilité des protéines extracellulaires affecte la distribution et la disponibilité des protéines sécrétées, telles que 13,14 morphogènes, dans un tissu.
Au cours des dernières décennies, la stabilité des protéines a été évaluée principalement dans la culture de cellule à l'aide d'impulsions marquage radioactif ou d'expériences de chasse cycloheximide 15. Dans ces expériences pulse-chase, les cellules sont soit transitoirement exposées à un «pouls» de aminés radioactifsprécurseurs acides qui sont incorporés dans des protéines nouvellement synthétisées, ou ils sont exposés à la cycloheximide, qui inhibe la synthèse des protéines. Les cellules cultivées sont ensuite recueillies à différents points dans le temps, ainsi que d'une immunoprécipitation suivie d'une autoradiographie (en radioactives expériences pulse-chase) ou western blot (dans des expériences de cycloheximide) est utilisé pour quantifier la clairance de la protéine au fil du temps.
Classiques tests de stabilité des protéines présentent plusieurs inconvénients. Tout d'abord, les protéines dans ces dosages sont souvent pas exprimées dans leurs environnements endogènes, mais plutôt dans une culture tissulaire et parfois dans des cellules provenant d'espèces différentes. Pour les protéines dont la stabilité dépend du contexte, cette approche est problématique. Deuxièmement, il ne est pas possible de suivre la clairance de la protéine dans des cellules ou des organismes individuels au cours du temps, et les données provenant de ces essais reflète une moyenne de différentes populations de cellules à différents points dans le temps. Depuis cellules individuelles peuvent avoir commencéavec différentes quantités de protéines, peut avoir pris l'étiquette ou cycloheximide radioactifs à des moments différents, ou peuvent avoir différents cinétique d'élimination, ces données agrégées peuvent être trompeuses. Enfin, dans le cas d'expériences de chasse cycloheximide, l'addition de l'inhibiteur de la synthèse des protéines peut avoir des effets physiologiques non désirées qui pourraient modifier artificiellement la stabilité des protéines de 16 à 18. Ces lacunes peuvent être évités en utilisant la fluorescence Decay Après Photoconversion (FDAP), une technique qui utilise des protéines photoconvertible pour mesurer la clairance de la protéine dynamiquement dans les organismes vivant 19-25 (voir Discussion des limitations de la technique FDAP).
Photoconvertible protéines sont des protéines fluorescentes dont excitation et d'émission propriétés changer après une exposition à des longueurs d'onde spécifiques de la lumière 26. Une variante couramment utilisé est Dendra2, une protéine "vert au rouge» photoconvertible qui a initialement excitations et d'émission des propriétés similaires aux protéines fluorescentes vertes, mais après une exposition à "la photoconversion" de -son excitation UV / propriétés d'émission deviennent semblables à celles des protéines fluorescentes rouges 23,27. Surtout, nouvelle protéine produite après photoconversion ne aura pas les mêmes propriétés d'excitation / émission que la protéine photoconverted, permettant découplage de la production et de la clairance à la photoconversion et l'observation de seulement une piscine de protéines photoconverted. Marquage des protéines d'intérêt avec des protéines photoconvertible fournit ainsi un moyen pratique d'impulsion-étiquette protéines dans les organismes vivants intacts, optiquement accessibles.
Dans les dosages de FDAP (Figure 1A), une protéine d'intérêt est marquée avec une protéine photoconvertible et exprimé dans un organisme vivant (figure 1B). La protéine de fusion est photoconverted, et la diminution du signal photoconverted cours du temps est suivie par fluorescenMicroscopie CE (figure 1C). Les données sont ensuite équipé d'un modèle approprié pour déterminer la demi-vie de la protéine de fusion (Figure 1D).
Le dosage FDAP décrit ici a été conçu pour déterminer les demi-vies extracellulaires de protéines de signalisation sécrétées dans embryons de poisson zèbre au début de l'embryogenèse 19. Cependant, cette approche peut être adapté à tout organisme modèle transparent qui tolère l'imagerie en direct, et pourrait être utilisé pour surveiller le jeu de toute protéine intracellulaire ou extracellulaire tagable. Variations de la technique décrite ici ont été réalisées dans des cellules cultivées 20,23 et 22 drosophile et la souris 21 embryons.
Protocole
1. Création d'un Fusion photoconvertible construire et d'embryons de poissons zèbres Injecter dechorionated
- Générer une construction fonctionnelle contenant la protéine d'intérêt fusionnée à une protéine du vert au rouge photoconvertible (voir Discussion), puis utilisez la transcription in vitro pour générer plafonné ARNm codant pour la protéine de fusion comme dans Müller et al., 2012 19.
- Utilisez pronase pour enlever les chorions d'environ 30 embryons de poisson zèbre au stade unicellulaire. Alternativement, dechorionate manuellement embryons en utilisant une pince 28.
Remarque: Les embryons doivent être dechorionated pour l'imagerie ultérieure. Si on le souhaite, les embryons peuvent être injectés à travers le chorion et dechorionated plus tard, juste avant l'imagerie.- Faire un ml de solution 5 mg / stock de pronase de Streptomyces griseus dans le milieu de embryon de poisson zèbre standard de 19. Roche doucement la solution à température ambiante pendant 10 min pour permettre à la protéase de se dissoudre. Aliquote 2 mL dans des microtubes et le gel à -20 ° C.
- Transfert des embryons au stade de cellule unique à un verre d'un diamètre de 5 cm ou boîte de Petri en matière plastique revêtue d'agarose contenant ~ 8 ml de milieu d'embryon. Ajouter 2 ml de solution décongelé pronase boursier à l'antenne et incuber à température ambiante pendant 5 à 10 min.
- Évitez d'exposer les embryons à l'air ou en plastique, que le contact avec soit provoquera embryons dechorionated la rupture. Remplissez un récipient en verre de 200 ml avec le milieu de l'embryon. Transférer les embryons dans le bécher en inclinant la boîte de Pétri en plongeant dans le milieu.
- Après les embryons se sont installés au fond du gobelet, versez plus de la moyenne de l'embryon, puis versez moyen d'embryons frais dans le bécher. Le tourbillonnant légère du milieu verser dans le bécher provoque embryons perdent leurs chorions affaibli.
- Répétez l'étape 1.2.4.
- Transférer les embryons dechorionated dans un plat d'injection agarose revêtu 29 en utilisant une pipette Pasteur en verre avec une pointe flammé. Flamboyantla pointe de la pipette empêche les contours crénelés des embryons blessant.
- Co-injecter l'ARNm et 3 kDa Alexa488-dextran conjugué 29,30 (figure 1B; voir discussion pour suggéré ARNm et les montants Alexa488-dextran). Injecter directement dans le centre de la cellule (et non le jaune) afin d'assurer une distribution uniforme de l'ARNm et une fois colorant fluorescent clivage commence.
Remarque: Le signal Alexa488 sera utilisé lors de l'analyse des données afin de générer des masques compartimentés afin de distinguer entre la fluorescence intracellulaire et extracellulaire. - Transfert embryons injectés à un puits d'agarose revêtues 1-2% d'un plat à six puits en plastique rempli avec le milieu de l'embryon. Incuber dans l'obscurité à 28 ° C jusqu'à ce que les embryons ont atteint le stade de la sphère fin 31 (environ après la fécondation de 5 h). Vérifiez embryons chacun de deux heures sous un stéréomicroscope et enlever tous les débris générés par les embryons sont morts.
2. Montage embryons de poissons zèbres Photoconversion et imagerie sur un microscope confocal inversé
- Utilisez un stéréomicroscope pour identifier une à cinq embryons sains, et d'utiliser une pipette Pasteur en verre avec une pointe flammé pour les retirer de l'antenne.
- Éjecter délicatement les embryons dans un tube à centrifuger contenant ~ 1 ml de fondu 1% agarose bas point de fusion dans un milieu d'embryon de 1x Danieau (voir list des matériaux) (figure 2A).
Remarque: Agarose doit avoir une température comprise entre 40 et 42 ° C; des températures plus élevées pourraient endommager les embryons. - Dessiner les embryons de retour dans la pipette avec quelques agarose. Éjecter délicatement l'agarose et les embryons sur la vitre du couvercle d'un plat à fond de verre (figure 2B). Se assurer que l'épaisseur de la vitre de protection est compatible avec l'objectif de microscope confocal.
- Re-utiliser la pipette de verre, si désiré. Pour nettoyer l'agarose résiduel hors de la pipette et éviter le colmatage, rapidement milieu pipette d'embryons de haut en bas.Placer un tube de 15 ml remplie de ~ 5 ml de milieu d'embryon à côté du stéréomicroscope à cet effet.
- Utilisation d'une sonde en métal pour positionner les embryons de sorte que le pôle animal (de blastoderme) fait face à la vitre de protection. Travaillez rapidement depuis l'agarose se solidifie en 20-30 sec. Utilisez le stéréomicroscope pour surveiller les positions des embryons et réajuster si nécessaire jusqu'à ce que l'agarose durcit.
- Répétez les étapes 2.1 à 2.4 jusqu'à ce que le nombre souhaité d'embryons a été monté.
Remarque: Dans une expérience typique, quatre gouttes agarose contenant quatre ou cinq embryons chacun se adaptera facilement sur le couvercle en verre. Environ 16 embryons peuvent être visualisés au cours d'une seule expérience idéale (figure 2C). - Lorsque la gélose a solidifié, remplir la cuve à fond de verre avec du milieu de l'embryon de 1x Danieau.
3. Photoconverting et mesure de la diminution de la Signal Photoconverted
Un objectif 25X ou 40X eau is approprié à la taille et l'indice de réfraction d'embryons de poisson zèbre. Il est préférable d'utiliser de l'huile d'immersion avec le même indice de réfraction que l'eau plutôt que de l'eau réelle, car l'eau va se évaporer au cours de l'expérience de cinq heures. Se assurer que l'huile à immersion est conçu pour être utilisé avec une eau (pas d'huile) objectif.
- Placez une grande goutte d'huile d'immersion sur l'objectif de se assurer que le film d'huile entre l'objectif et le verre de couverture ne se cassera pas que la scène se déplace vers différentes positions de l'embryon au cours de l'imagerie. Fixer solidement le plat à fond de verre sur la scène de telle sorte que le plat ne passeront pas si les coups de théâtre. Si possible, utilisez un stade chauffé à 28 ° C, la température optimale pour le développement du poisson zèbre.
- Définir la position de chaque embryon dans le logiciel du microscope confocal. Réglez la profondeur z pour chaque embryon, et tenter de cibler à peu près le même plan dans chaque embryon.
Remarque: Environ 30 pm du pôle animal est un allerod profondeur depuis à cette profondeur de la couche enveloppante de l'embryon peut être évitée, zone d'imagerie est maximisée, et diffusion de la lumière est minime. Une tranche optique unique avec une épaisseur de 3,3 um ~ fournit suffisamment de données; il ne est pas nécessaire d'acquérir un z-pile (voir la section 5). - Recueillir deux signaux au cours de l'expérience: le signal "vert" de l'Alexa488-dextrane conjugué, qui sera utilisé lors de l'analyse des données d'isoler extracellulaire et intracellulaire par fluorescence et le signal "rouge" de la protéine de fusion après son photoconverted.
- Excite Alexa488 utilisant un laser de 488 nm, et de recueillir fluorescence émise entre ~ 500 et 540 nm. Remarque: Après photoconversion, beaucoup vert au rouge protéines photoconvertible (par exemple, Dendra2) peut être excité avec un laser 543 nm et émettent fluorescence entre ~ 550 et 650 nm. Réglez si nécessaire en fonction de la protéine photoconvertible utilisé.
- Acquérir "pré-photoconversion"Images, et de configurer le logiciel du microscope confocal à l'image de chacune des positions précédemment définies (de l'étape 3.2) avec les conditions d'imagerie appropriés tous les 10 ou 20 minutes pour un cours de temps de cinq heures (voir la section 5 et Discussion).
- Pour photoconvert la protéine de fusion, passer à un objectif 10X et d'exposer des groupes d'embryons à la lumière UV provenant d'une lampe à vapeur de mercure avec un filtre ~ 300-400 nm d'excitation à la sortie de 100% pendant 2 min. Détourner l'attention long de l'axe z pour promouvoir photoconversion uniforme (voir la section 5). Assurez-vous que l'huile d'immersion ne coule pas sur l'objectif 10x lors de photoconversion.
Remarque: Le déplacement de mise au point pendant photoconversion pourrait être automatisé afin d'éviter la variabilité des expérimentateurs. - Revenez à l'objectif 25X ou 40X immédiatement après photoconversion. Veiller à ce que les positions définies précédemment de l'étape 3.2 sont toujours exactes. Si le plat déplacé pendant photoconversion, re-définir les positions des embryons.
- Démarrez le programme créé à l'étape 3.4 et permet l'imagerie de continuer pendant 5 heures. Noter le temps écoulé entre le début et photoconversion de formation d'image pour chaque embryon.
- Vérifiez de temps sur l'expérience. Surveiller le niveau de milieu de Danieau et ajouter plus si nécessaire. Redémarrez le logiciel se il est en panne.
- Afin de déterminer les valeurs de fluorescence de fond qui seront utilisés au cours de l'analyse des données pour estimer l'asymptote un modèle de décroissance exponentielle, inclure des embryons qui ont été injectés avec Alexa488-dextrane mais non l'ARNm dans l'expérience. Afin de déterminer le bruit de l'instrument, qui sera également utilisé au cours de l'analyse des données ultérieur, acquérir une image en l'absence d'un échantillon.
4. Analyser les données en utilisant PyFDAP
- Inspecter visuellement le temps des ensembles de données de cours de chaque embryon. Jeter ensembles de données à partir d'embryons morts durant imagING, qui a déplacé de manière significative, qui ont de très faibles niveaux de signaux photoconverted, ou qui contiennent des régions de cellules qui ressemblent inhabituel et ont cessé de bouger et en divisant (typique d'embryons blessés ou malades).
Remarque: Parfois, des bulles dans l'huile d'immersion ou d'autres artefacts apparaissent dans une ou deux images dans un ensemble de données autrement utilisable. Notez toutes les images qui contiennent des artefacts; ils seront rejetées plus tard, et les points de temps restants de cet ensemble de données peuvent être analysées. - Utilisez le logiciel PyFDAP en Python pour analyser les données FDAP. PyFDAP calcule la demi-vie en déterminant l'intensité de fluorescence intracellulaire et extracellulaire rouge moyen dans chaque image et d'ajustement des données avec une fonction décroissante de façon exponentielle 56 (figure 3).
- Télécharger PyFDAP (voir Liste des matériaux).
- Utilisation PyFDAP pour séparer le signal intracellulaire et extracellulaire photoconverted (figure 3A, B). Nouse le signal Alexa488, qui est strictement intracellulaire, pour créer un masque intracellulaire. Appliquez ce masque à l'image du canal de rouge correspondant à éviter pixels intracellulaires d'être considérés lors du calcul de l'intensité moyenne extracellulaire. Pour mesurer l'intensité intracellulaire moyenne, inverser le masque.
- Dans PyFDAP, afficher les images masqués générés à l'étape 4.2.2. Inspecter visuellement ces images et jeter des ensembles de données dans laquelle masques ne distinguent pas exactement intracellulaire de l'espace extracellulaire (ce devrait être rare; noter que les membranes cellulaires sont inclus dans des images où l'espace extracellulaire a été masqués, mais ils pourraient être supprimés en modifiant le seuil algorithme ou en introduisant un masque de membrane (par exemple, en utilisant une membrane-PCP)). Écartent également toute simples images contenant des objets (par exemple, des bulles dans l'huile d'immersion) identifiés dans l'étape 4.1.
- Utilisez PyFDAP pour calculer les intensités de fluorescence extracellulaires et intracellulaires moyennes pour each image. PyFDAP ces moyennes calcule en additionnant les intensités des pixels qui tombent à l'extérieur du masque et en divisant par le nombre total de pixels additionnés.
- Monter les données de fluorescence (figure 3C) avec la fonction exponentielle suivante:
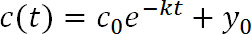
où t est le temps post-photoconversion, c (t) est l'intensité à une valeur donnée de t, c 0 est l'intensité à l'instant t = 0, k est le taux de clairance constante, et y 0 est l'asymptote que la fonction se approche en tant que fluorescence diminue (figure 1D). y 0 peut être limitée en fonction des mesures de l'étape 19 3,8. - Utilisation PyFDAP pour calculer les protéines extracellulaires et intracellulaires des demi-vies (τ) à partir des constantes de vitesse de dégagement (k) à l'aide de la relation suivante:

5. Les expériences de contrôle pour évaluer Photoblanchiment, par inadvertance Photoconversion et Photoconversion Uniformité
- Photoblanchiment évaluer
Remarque: Le photoblanchiment peut provoquer une diminution artéfactuelle l'intensité de fluorescence qui reflète les propriétés de blanchiment de la protéine fluorescente, en plus de la clairance de la protéine d'intérêt.- Pour évaluer d'éventuelles photoblanchiment, effectuer une série d'expériences FDAP avec des intervalles de 10 min entre l'imagerie et un deuxième ensemble avec des intervalles de 20 min entre imagerie (figure 4). Analyser les données des deux séries d'expériences utilisant PyFDAP comme décrit à la section 4.
- Comparer les demi-vies des expériences 10 et 20 de l'intervalle min. Plus longue demi-vie de 20 min d'intervalle expériences indiquent photoblanchiment significative. Si les demi-vies des deux expériences sont identiques, photobleaching ne est pas une préoccupation importante.
- Alternativement, d'évaluer photoblanchiment en acquérant une série de ~ 30 images immédiatement après photoconversion. Une diminution significative de l'intensité de fluorescence indique photoblanchiment significative.
- Si photoblanchiment est détectée, utiliser la puissance du laser inférieure, diminuer le temps d'imagerie, ou envisager d'utiliser une protéine photoconvertible plus photostable 32.
- Évaluation photoconversion par inadvertance.
Remarque: Dendra2 peut être photoconverted utilisant 488 nm éclairage 27. Lorsque excitant Alexa488 avec le laser 488 nm comme décrit dans l'étape 3.3.1, photoconversion par inadvertance et donc une augmentation artéfactuelle dans la demi-vie apparente de la protéine d'intérêt est possible. Cependant, nous et d'autres avons constaté que 33 488 nm éclairage est une méthode inefficace de photoconversion dans embryons de poisson zèbre.- Utilisez l'expérience de contrôle décrit à l'étape 5.1.1 pour détecter photoconversion par inadvertance. Comparer les demi-vies des expériences 10 et 20 de l'intervalle min. Courtes demi-vies de 20 min d'intervalle expériences indiquent photoconversion inadvertance significative. Si les demi-vies des deux expériences sont identiques, photoconversion par inadvertance ne est pas une préoccupation importante.
- Si par inadvertance photoconversion est détectée, utiliser une puissance laser de 488 nm inférieure et la réduction des temps d'imagerie pour éviter par inadvertance photoconverting Dendra2.
- Évaluation photoconversion uniformité.
Remarque: Si photoconversion est sollicité vers le pôle animal de l'embryon, la diminution de la fluorescence sera influencée par la diffusion de la protéine ou le mouvement des cellules dans des plans plus profondes (Figure 5A).- Pour déterminer si photoconversion est uniforme, exprimer une protéine sécrétée photoconvertible (pour des expériences avec des protéines de fusion extracellulaires) ou une protéine cytoplasmique photoconvertible (pour des expériences avec des protéines de fusion intracellulaires). Photoconvert comme d'habitude, tpoule acquièrent un z-stack englobant la plupart des blastoderme toutes les 20 minutes pendant 80 min.
- Si photoconversion est sollicité vers le pôle animal, l'intensité de fluorescence dans des plans profonds va augmenter au cours du temps due à la diffusion ou le mouvement des cellules (figure 5B). Si non uniforme photoconversion est détecté, se concentrer plus profondément dans les embryons au cours photoconversion.
Résultats
FDAP a été utilisé pour déterminer la demi-vie de protéines de signalisation extracellulaires dans 19 embryons de poisson zèbre. Une de ces protéines, strabisme, induit l'expression de gènes de 34 mesendodermal cours de l'embryogenèse. Squint-Dendra2 active l'expression des gènes de mesendodermal à des niveaux similaires à untagged Squint, comme démontré par qRT-PCR et l'hybridation in situ 19 essais. Les embryons sont co-injectés avec Alexa488-dex...
Discussion
Le succès d'une expérience FDAP repose sur la génération d'une protéine de fusion photoconvertible fonctionnel. Une protéine de marquage peut affecter son activité biologique et / ou les propriétés biophysiques, y compris sa localisation, la solubilité, la stabilité et 36 à 41. Soyez prêt à tester l'activité de plusieurs différentes constructions de fusion afin de trouver celui qui est actif. Nous avons trouvé que la modification de la position de la protéine photoconvertible par...
Déclarations de divulgation
Les auteurs ne ont aucun conflit de divulguer.
Remerciements
Les auteurs tiennent à remercier Jeffrey Farrell, James Gagnon, et Jennifer Bergmann des commentaires sur le manuscrit. KWR a été soutenu par le Programme National Science Foundation Graduate Research Fellowship lors de l'élaboration de l'essai FDAP. Ce travail a été soutenu par des subventions du NIH à AFS et par des subventions de la Fondation allemande pour la recherche (programme Emmy Noether), la Société Max Planck, et le Programme Human Frontier Science (Career Development Award) à PM.
matériels
| Name | Company | Catalog Number | Comments |
| PyFDAP (download from the following website: http://people.tuebingen.mpg.de/mueller-lab) | Install and operate using the instructions provided on the PyFDAP website; PyFDAP is compatible with Linux, Mac, and Windows operating systems. | ||
| mMessage mMachine Sp6 Transcription Kit | Life Technologies | AM1340 | To generate capped mRNA for injection into embryos |
| Alexa488-dextran conjugate, 3 kDa | Life Technologies | D34682 | Co-inject with mRNA to create intracellular and extracellular masks |
| 6-well plastic dish | BD Falcon | Incubate embryos in agarose-coated wells until ready for mounting | |
| Embryo medium | 250 mg/L Instant Ocean salt, 1 mg/L methylene blue in reverse osmosis water adjusted to pH 7 with NaHCO3 | ||
| Protease from Streptomyces griseus | Sigma | P5147 | Make a 5 mg/ml stock and use at 1 mg/ml to dechorionate embryos at the one-cell stage |
| 5 cm diameter glass Petri dish | For embryo dechorionation | ||
| 200 ml glass beaker | For embryo dechorionation | ||
| Microinjection apparatus | For injection of mRNA and dye into embryos at the one-cell stage | ||
| Stereomicroscope | For injecting and mounting embryos | ||
| 1x Danieau's medium | Dilute low melting point agarose and perform imaging in this medium; recipe: 0.2 mm filtered solution of 58 mM NaCl, 0.7 mM KCl, 0.4 mM MgSO4, 0.3 mM CaCl2, 5 mM HEPES pH 7.2 | ||
| UltraPure low melting point agarose | Invitrogen | 16520-100 | For mounting embryos; use at a concentration of 1% in Danieau's medium: add 200 mg to 20 ml Danieau's medium, microwave until dissolved, then aliquot 1 ml into microcentrifuge tubes; aliquots can be stored at 4 °C, re-melted at 70 °C, and cooled to 40–42 °C when ready to use |
| Glass Pasteur pipette | Kimble Chase (via Fisher) | 63A53WT | For mounting embryos; flame the tip to prevent jagged edges from injuring embryos |
| Metal probe | For positioning embryos during mounting | ||
| Glass bottom dishes | MatTek | P35G-1.5-14-C | Use the appropriate cover glass thickness for your objective; part number listed here is for cover glass No. 1.5 |
| 15 ml tube filled with ~5 ml embryo medium | BD Falcon | For rinsing residual agarose from the Pasteur pipette | |
| Inverted laser scanning confocal microscope | A mercury arc lamp, 488 nm laser, 543 nm laser, and the appropriate filter sets are required | ||
| Heated stage | To maintain embryos at the optimal temperature of 28 °C during the experiment | ||
| Confocal software capable of time-lapse imaging | Must be able to define multiple positions and automatically image them at defined intervals | ||
| 25X or 40X water objective | Objective for imaging | ||
| 10X air objective | Objective for photoconversion | ||
| Immersion oil | Immersion oil with the same refractive index as water |
Références
- Schwanhäusser, B., et al. Global quantification of mammalian gene expression control. Nature. 473 (7347), 337-342 (2011).
- Boisvert, F. M., et al. A Quantitative Spatial Proteomics Analysis of Proteome Turnover in Human Cells. Molecular & Cellular Proteomics. 11 (3), 011429 (2012).
- Belle, A., Tanay, A., Bitincka, L., Shamir, R., O'Shea, E. K. Quantification of protein half-lives in the budding yeast proteome. Proceedings of the National Academy of Sciences of the United States of America. 103 (35), 13004-13009 (2006).
- Eden, E., et al. Proteome Half-Life Dynamics in Living Human Cells. Science. 331 (6018), 764-768 (2011).
- Parry, D. H., O'Farrell, P. H. The schedule of destruction of three mitotic cyclins can dictate the timing of events during exit from mitosis. Current Biology. 11 (9), 671-683 (2001).
- Holloway, S. L., Glotzer, M., King, R. W., Murray, A. W. Anaphase is initiated by proteolysis rather than by the inactivation of maturation-promoting factor. Cell. 73 (7), 1393-1402 (1993).
- Dharmasiri, N., Estelle, M. Auxin signaling and regulated protein degradation. Trends Plant Sci. 9 (6), 302-308 (2004).
- MacDonald, B. T., Tamai, K., He, X. Wnt/beta-catenin signaling: components, mechanisms, and diseases. Developmental Cell. 17 (1), 9-26 (2009).
- Chen, X., et al. Processing and turnover of the Hedgehog protein in the endoplasmic reticulum. The Journal of Cell Biology. 192 (5), 825-838 (2011).
- Elmore, S. Apoptosis: A Review of Programmed Cell Death. Toxicologic Pathology. 35 (4), 495-516 (2007).
- Yi, J. J., Ehlers, M. D. Emerging Roles for Ubiquitin and Protein Degradation in Neuronal Function. Pharmacological Reviews. 59 (1), 14-39 (2007).
- Rubinsztein, D. C. The roles of intracellular protein-degradation pathways in neurodegeneration. Nature. 443 (7113), 780-786 (2006).
- Müller, P., Schier, A. F. Extracellular Movement of Signaling Molecules. Developmental Cell. 21 (1), 145-158 (2011).
- Eldar, A., Rosin, D., Shilo, B. -. Z., Barkai, N. Self-enhanced ligand degradation underlies robustness of morphogen gradients. Developmental Cell. 5 (4), 635-646 (2003).
- Zhou, P. Determining protein half-lives. Methods In Molecular Biology. 284, 67-77 (2004).
- Woodside, K. H. Effects of cycloheximide on protein degradation and gluconeogenesis in the perfused rat liver. Biochim Biophys Acta. 421 (1), 70-79 (1976).
- Schimke, R. T., Doyle, D. Control of enzyme levels in animal tissues. Annual Review of Biochemistry. 39, 929-976 (1970).
- Kenney, F. T. Turnover of rat liver tyrosine transaminase: stabilization after inhibition of protein synthesis. Science. 156 (3774), 525-528 (1967).
- Müller, P., et al. Differential Diffusivity of Nodal and Lefty Underlies a Reaction-Diffusion Patterning System. Science. 336 (6082), 721-724 (2012).
- Kiuchi, T., Nagai, T., Ohashi, K., Mizuno, K. Measurements of spatiotemporal changes in G-actin concentration reveal its effect on stimulus-induced actin assembly and lamellipodium extension. The Journal of Cell Biology. 193 (2), 365-380 (2011).
- Plachta, N., Bollenbach, T., Pease, S., Fraser, S. E., Pantazis, P. Oct4 kinetics predict cell lineage patterning in the early mammalian embryo. Nature Cell Biology. 13 (2), 17-123 (2011).
- Drocco, J. A., Grimm, O., Tank, D. W., Wieschaus, E. Measurement and Perturbation of Morphogen Lifetime: Effects on Gradient Shape. Biophys J. 101 (8), 1807-1815 (2011).
- Zhang, L., et al. Method for real-time monitoring of protein degradation at the single cell level. BioTechniques. 42 (4), 446-450 (2007).
- Miyawaki, A. Proteins on the move: insights gained from fluorescent protein technologies. Nat Rev Mol Cell Biol. 12 (10), 636-668 (2011).
- Pantazis, P., Supatto, W. Advances in whole-embryo imaging: a quantitative transition is underway. Nat Rev Mol Cell Biol. 15 (5), 327-339 (2014).
- Lukyanov, K. A., Chudakov, D. M., Lukyanov, S., Verkhusha, V. V. Innovation: Photoactivatable fluorescent proteins. Nat Rev Mol Cell Biol. 6 (11), 885-891 (2005).
- Gurskaya, N. G., et al. Engineering of a monomeric green-to-red photoactivatable fluorescent protein induced by blue light. Nature Biotechnology. 24 (4), 461-465 (2006).
- Zou, J., Wei, X. Transplantation of GFP-expressing Blastomeres for Live Imaging of Retinal and Brain Development in Chimeric Zebrafish Embryos. Journal of Visualized Experiments. (41), (2010).
- Yuan, S., Sun, Z. Microinjection of mRNA and Morpholino Antisense Oligonucleotides in Zebrafish Embryos. Journal of Visualized Experiments. (27), (2009).
- Rosen, J. N., Sweeney, M. F., Mably, J. D. Microinjection of Zebrafish Embryos to Analyze Gene Function. Journal of Visualized Experiments. (25), (2009).
- Kimmel, C. B., Ballard, W. W., Kimmel, S. R., Ullmann, B., Schilling, T. F. Stages of embryonic development of the zebrafish. Developmental Dynamics. 203 (3), (1995).
- McKinney, S. A., Murphy, C. S., Hazelwood, K. L., Davidson, M. W., Looger, L. L. A bright and photostable photoconvertible fluorescent protein. Nature Methods. 6 (2), 131-133 (2009).
- Dempsey, W. P., Qin, H., Pantazis, P. In Vivo Cell Tracking Using PhOTO Zebrafish. Methods in Molecular Biology. 1148, 217-228 (2014).
- Schier, A. F. Nodal Morphogens). Cold Spring Harbor Perspectives in Biology. 1 (5), a003459-a003459 (2009).
- Otsu, N. A threshold selection method from gray-level histograms. IEEE Trans Syst Man Cybern. 9 (1), 62-66 (1979).
- Pédelacq, J. -. D., Cabantous, S., Tran, T., Terwilliger, T. C., Waldo, G. S. Engineering and characterization of a superfolder green fluorescent protein. Nature Biotechnology. 24 (1), 79-88 (2005).
- Swulius, M. T., Jensen, G. J. The Helical MreB Cytoskeleton in Escherichia coli MC1000/pLE7 Is an Artifact of the N-Terminal Yellow Fluorescent Protein Tag. Journal of Bacteriology. 194 (23), 6382-6386 (2012).
- Landgraf, D., Okumus, B., Chien, P., Baker, T. A., Paulsson, J. Segregation of molecules at cell division reveals native protein localization. Nature Methods. 9 (5), 480-482 (2012).
- Stadler, C., et al. Immunofluorescence and fluorescent-protein tagging show high correlation for protein localization in mammalian cells. Nature Methods. 10 (4), 315-323 (2013).
- Quattrocchio, F. M., Spelt, C., Koes, R. Transgenes and protein localization: myths and legends. Trends Plant Sci. 18 (9), 473-476 (2013).
- Morimoto, Y. V., Kojima, S., Namba, K., Minamino, T. M153R Mutation in a pH-Sensitive Green Fluorescent Protein Stabilizes Its Fusion Proteins. PLoS ONE. 6 (5), e19598 (2011).
- Shaner, N. C., Steinbach, P. A., Tsien, R. Y. A guide to choosing fluorescent proteins. Nature Methods. 2 (12), 905-909 (2005).
- Waters, J. C. Accuracy and precision in quantitative fluorescence microscopy. The Journal of Cell Biology. 185 (7), 1135-1148 (2009).
- Moll, U. M., Petrenko, O. The MDM2-p53 interaction. Mol Cancer Res. 1 (14), 1001-1008 (2003).
- Auer, T. O., Duroure, K., De Cian, A., Concordet, J. P., Del Bene, F. Highly efficient CRISPR/Cas9-mediated knock-in in zebrafish by homology-independent DNA repair. Genome Research. 24 (1), 142-153 (2014).
- Bedell, V. M., et al. In vivo genome editing using a high-efficiency TALEN system. Nature. 491 (7422), 114-118 (2012).
- Hruscha, A., et al. Efficient CRISPR/Cas9 genome editing with low off-target effects in zebrafish. Development. 140 (24), 4982-2987 (2013).
- Hwang, W. Y., et al. Heritable and Precise Zebrafish Genome Editing Using a CRISPR-Cas System. PLoS ONE. 8 (7), 68708 (2013).
- Hwang, W. Y., et al. Efficient genome editing in zebrafish using a CRISPR-Cas system. Nature Biotechnology. 31 (3), 227-229 (2013).
- Zu, Y., et al. TALEN-mediated precise genome modification by homologous recombination in zebrafish. Nature Methods. 10 (4), 329-331 (2013).
- Keller, P. J., Schmidt, A. D., Wittbrodt, J., Stelzer, E. H. K. Reconstruction of Zebrafish Early Embryonic Development by Scanned Light Sheet Microscopy. Science. 322 (5904), 1064-1069 (2008).
- Blanchet, M. H., et al. Cripto Localizes Nodal at the Limiting Membrane of Early Endosomes. Science Signaling. 1 (45), ra13-ra13 (2008).
- Jullien, J., Gurdon, J. Morphogen gradient interpretation by a regulated trafficking step during ligand-receptor transduction. Genes & Development. 19 (22), 2682-2694 (2005).
- Incardona, J. P., et al. Receptor-mediated endocytosis of soluble and membrane-tethered Sonic hedgehog by Patched-1. Proceedings of the National Academy of Sciences of the United States of America. 97 (22), 12044-12049 (2000).
- Scholpp, S., Brand, M. Endocytosis Controls Spreading and Effective Signaling Range of Fgf8 Protein. Current Biology. 14 (20), 1834-1841 (2004).
- Bläßle, A., Müller, P. . PyFDAP: Automated analysis of Fluorescence Decay After Photoconversion (FDAP) experiments. Bioinformatics. In Press, (2014).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon