Method Article
Rapid Neuronal Différenciation des Induced cellules souches pluripotentes pour mesurer l'activité réseau sur Arrays Micro-électrode
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Nous modifions et mettre en œuvre un protocole précédemment publié décrivant la différenciation rapide, reproductible et efficace des humains induite par cellules souches pluripotentes (de hiPSCs) dans les neurones corticaux excitateurs 12. Plus précisément, notre modification permet un contrôle de la densité des cellules neuronales et l'utilisation sur des réseaux de micro-électrodes pour mesurer les propriétés électrophysiologiques au niveau du réseau.
Résumé
Neurones dérivés des humains induite par cellules souches pluripotentes (hiPSCs) fournissent un nouvel outil prometteur pour l'étude des troubles neurologiques. Dans la dernière décennie, de nombreux protocoles de différenciation hiPSCs dans les neurones ont été développés. Cependant, ces protocoles sont souvent lents avec une forte variabilité, une faible reproductibilité, et une faible efficacité. En outre, les neurones obtenus avec ces protocoles sont souvent immatures et dépourvus d'activité fonctionnelle adéquate tant au niveau unicellulaires et réseau à moins que les neurones sont cultivés pendant plusieurs mois. En partie à cause de ces limitations, les propriétés fonctionnelles des réseaux neuronaux hiPSC dérivés ne sont pas encore bien caractérisés. Ici, on adapte un protocole publié récemment qui décrit la production de neurones humains provenant hiPSCs par l' expression forcée du facteur de transcription neurogénine 2-12. Ce protocole est rapide (produisant les neurones matures dans les 3 semaines) et efficace, avec près de 100% l'efficacité de conversion TRANSDUCcellules ed (> 95% des cellules DAPI-positives sont MAP2 positive). En outre, le protocole donne une population homogène de neurones excitateurs qui permettraient à l'enquête sur les contributions spécifiques de type cellulaire à des troubles neurologiques. Nous avons modifié le protocole original en générant des cellules hiPSC transduites de manière stable, nous donnant un contrôle explicite sur le nombre total de neurones. Ces cellules sont ensuite utilisées pour produire des réseaux de neurones hiPSC dérivées sur des réseaux de micro-électrodes. De cette manière, l'activité électrophysiologique spontanée des réseaux de neurones hiPSC dérivés peut être mesuré et caractérisé, tout en conservant la cohérence interexperimental en fonction de la densité cellulaire. Le protocole présenté est largement applicable, en particulier pour des études mécanistiques et pharmacologiques sur les réseaux neuronaux humains.
Introduction
Le développement de l' origine humaine cellules souches pluripotentes (hiPSCs) protocoles de différenciation pour générer des neurones humains in vitro a fourni un nouvel outil puissant pour l' étude des troubles neurologiques. Jusqu'à récemment, l'étude de ces troubles a été sévèrement entravée par l'absence de systèmes modèles utilisant les neurones humains. Bien que les rongeurs peuvent être utilisés pour étudier les troubles neurologiques, les résultats de ces études ne peuvent pas être convertis facilement à l' homme 1. Compte tenu de ces limitations, les neurones hiPSC dérivés sont un modèle alternative prometteuse qui peut être utilisé pour élucider les mécanismes moléculaires sous - jacents et les troubles neurologiques in vitro pour le dépistage de la drogue.
Dans la dernière décennie, plusieurs protocoles pour convertir hiPSCs dans les neurones ont été développés 2-8. Cependant, ces protocoles sont encore limités à bien des égards. Tout d' abord, les protocoles sont souvent beaucoup de temps: les neurones électrogènes à maturation adéquate (c. -à- synapformation se) et l' activité fonctionnelle nécessite des mois de procédures de culture, ce qui rend difficiles études 9 à grande échelle. En outre, l'efficacité de conversion-hiPSC à neurone est faible. Les protocoles donnent souvent une population hétérogène de neurones, et donc ne permettent pas des études sur des sous-ensembles spécifiques de cellules neuronales. En outre, les protocoles ne sont pas reproductibles, ce qui donne des résultats différents pour les différentes lignes iPSC 10,11. Enfin, le stade de maturation et les propriétés fonctionnelles des neurones résultant 10 sont également variables.
Pour résoudre ces problèmes, Zhang et al. (2013) 12 ont développé un protocole qui génère rapidement et de manière reproductible à partir de neurones humains hiPSCs en surexprimant le facteur de transcription neurogénine-2. Tel que rapporté par les auteurs, la différenciation se produit relativement rapidement (seulement 2 - 3 semaines après l'induction de l'expression de neurogénine-2), le protocole est reproductible (propriétés neuronales sont indépendantes des sepuis ligne hiPSC), et la conversion-hiPSC à neurone est très efficace (près de 100%). La population de neurones générés avec leur protocole est homogène (ressemblant à la couche supérieure des neurones corticaux), permettant l'examen des contributions spécifiques du type cellulaire à des troubles neuronaux. En outre, leurs neurones hiPSC dérivés présentaient des propriétés matures (par exemple, la capacité de former des synapses et de l' activité fonctionnelle robuste) après seulement 20 d.
Caractériser les propriétés électrophysiologiques des neurones hiPSC dérivés au niveau du réseau est une condition préalable importante avant que la technologie hiPSC peut être exploitée pour l'étude des maladies humaines. Pour cette raison, de nombreux groupes de recherche ont récemment commencé à enquêter sur les neurones de cellules souches dérivées au niveau du réseau à l' aide de réseau de micro-électrode (MEA) périphériques (systèmes multicanaux, Reutlingen, Allemagne) 13-16. Les électrodes d'un MEA sont noyées dans un substrat sur lequel les cellules neuronales peuvent être cultivées.AME peuvent être utilisés pour explorer les propriétés électrophysiologiques des réseaux neuronaux et le développement in vitro de leur activité. À l'heure actuelle, les AME sont utilisés uniquement en combinaison avec des protocoles de différenciation qui prennent plusieurs mois pour obtenir des réseaux matures. Par conséquent, la combinaison AME avec un protocole de différenciation rapide devrait faciliter l'utilisation de cette technologie dans des études à grande échelle des troubles neurologiques.
Ici, nous présentons une modification de la Zhang et al. (2013) 12 protocole et l' adapter pour une utilisation sur les AME. En particulier, plutôt que de compter sur une lentiviral transduction aiguë, nous avons créé à la place des lignes hiPSC exprimant de manière stable rtTA / Ngn2 avant induction de la différenciation. Nous l' avons fait surtout d'avoir un contrôle reproductible sur la densité des cellules neuronales, puisque la densité des cellules neuronales est critique pour la formation de réseau neuronal, et un bon contact entre les neurones et les électrodes de la MEA 17,18. although Zhang et al. protocole est très efficace en ce qui concerne la conversion des hiPSCs transduites, il est intrinsèquement variable par rapport au rendement final des neurones à partir du nombre de hiPSCs plaqué initialement (voir la figure 2E dans Zhang et al.) 12. Avec une ligne stable, nous éliminons de nombreux problèmes qui causent la variabilité, comme la toxicité lentiviral et l'efficacité de l'infection. Nous avons ensuite optimisé les paramètres qui produisent de manière fiable des réseaux neuronaux hiPSC dérivés sur les AME, l' obtention de la maturation du réseau (par exemple, les événements synchrones impliquant une majorité des canaux) de la troisième semaine. Ce protocole rapide et fiable devrait permettre des comparaisons directes entre les neurones dérivés de différentes (c. -à- spécifique au patient) lignes de hiPSC ainsi que de fournir une cohérence solide pour les études pharmacologiques.
Protocole
Toutes les expériences sur les animaux ont été effectuées en conformité avec les soins des animaux et de l'utilisation des lignes directrices approuvées du Comité de protection des animaux, Centre médical de l'Université Radboud, aux Pays-Bas, (RU-DEC-2011-021, numéro de protocole: 77073).
1. Glia cellulaire Isolement et culture
NOTE: Le protocole présenté ici est basé sur les travaux de McCarthy et de Vellis 19, et un protocole détaillé très similaire pour les astrocytes de souris est disponible 20. Pour générer des cultures primaires d'astrocytes corticaux de embryonnaires (E18) le cerveau de rat, un rat enceinte doit être sacrifié, les embryons doivent être récoltées à partir de l'utérus, et les cerveaux ont besoin d'être isolé à partir des embryons. Pour remplir un flacon T75, les corticales de 2 cerveaux embryonnaires doivent être combinées. En variante, les astrocytes purifiés et congelés disponibles dans le commerce peuvent être achetés.
- Préparer le flacon de culture T75
- Poly diluer-D-lysine (PDL) en, Eau ultrapure stérile jusqu'à une concentration finale de 10 pg / ml. Ajouter 5 ml de PDL dilué dans le flacon de culture T75. Bruissement autour doucement pour mouiller la surface de croissance complet. Placer le ballon dans un humidifiée incubateur 37 ° C pendant 3 h.
- Aspirer le PDL du ballon. Rincer les 3x flacon avec 5 ml d'eau stérile pour éliminer PDL non lié. Aspirer l'eau complètement. Laisser le ballon à sécher dans une hotte à flux laminaire ou utilisé immédiatement.
- Dissection des corticales
- Préparer 50 ml de milieu de dissection: L-15 du milieu de Lebovitz avec 2% (v / v) B-27 supplément. Gardez sur la glace.
- Anesthetize le rat profondément avec de l'isoflurane dans une chambre d'induction (petits plexiglas boîte) jusqu'à ce que la respiration cesse (~ 5-8 min). Enlever le rat à partir de la chambre d'admission et immédiatement euthanasiés par dislocation cervicale.
- Vaporiser l'abdomen du rat avec 70% EtOH et essuyez l'excédent. Exposer et enlever l'utérus de la mère par césarienne sectià l' aide d' une paire de ciseaux 21.
- Couper les embryons individuels de leurs sacs amniotiques avec des ciseaux, transférer à une boîte de Pétri stérile rempli de milieu de dissection froide et garder sur la glace.
- Transfert d'embryons à nouveau à une nouvelle, stérile 6 cm boîte de Pétri remplie de milieu de dissection froide. Extraire les cerveaux des embryons sous un microscope stéréo. Pour exposer le cerveau, peler doucement la peau et le crâne en utilisant une pince. évider délicatement l'ensemble du cerveau et de transférer à un plat de 35 mm de Pétri avec un milieu de dissection frais, froid.
NOTE: cerveaux entiers disséqués à partir d' embryons peuvent être stockés dans un milieu de dissection sur la glace pendant de nombreuses heures sans perdre la viabilité cellulaire. - Séparer les deux hémisphères de chaque cerveau en coupant la ligne médiane avec des ciseaux à ressort à pointe fine ou un scalpel. dénuder délicatement les méninges avec une pince à pointe fine droite.
NOTE: Il est très important d'enlever les méninges complètement. thla contamination des fibroblastes empêche de la culture des astrocytes. Fibroblastes se divisent rapidement et les cellules seront éventuellement déplacer les autres cellules. - Retirer le mésencéphale / striatum et le bulbe olfactif avec des ciseaux à ressort ou un scalpel. Assurez-vous également de supprimer l'hippocampe (structure en forme de C qui est perimedian et caudale par rapport au cortex) avec des ciseaux à ressort ou un scalpel. Ramassez les hémisphères corticaux dans un tube de 15 ml de centrifugeuse rempli de 5 milieu de dissection mL. Gardez sur la glace.
- Dissociation des corticales
- Préparer 2 ml de Ca 2+ / Mg 2+ -free solution saline équilibrée de Hank (HBSS) avec 0,25% de trypsine (milieu de dissociation). Préparer 50 ml de glucose à haute milieu de Eagle modifié par Dulbecco (DMEM) avec 15% (v / v) de sérum bovin fœtal (FBS) et 1% (v / v) de pénicilline / streptomycine (milieu de culture) et stériliser par filtration.
- Laisser le tissu se déposent au fond du tube de centrifugation. soigneusement commepirate autant du milieu de dissection que possible au-dessus du tissu. Laver le tissu avec 5 ml de Ca 2+ / Mg 2+ -free HBSS (sans trypsine) et permettre au tissu de se déposer au fond du tube.
- Aspirer soigneusement le HBSS. Ajouter 2 ml milieu de dissociation et secouez doucement le tube pour mélanger l'enzyme autour du tissu. Incuber dans un bain d'eau à 37 ° C pendant 5 à 10 min. Flick le tube à quelques reprises pendant l'incubation pour agiter le tissu.
- Immédiatement triturer le tissu à l'aide d'une pipette de 1000 pi. Réglez le volume de pipetage à environ 800 pi. Aspirer les pièces et éjecte de force sur le côté du tube, directement au-dessus de la ligne de fluide. Cependant, essayez de réduire au minimum les bulles ou de mousse. Répétez jusqu'à ce que le tissu est suffisamment dissocié, environ 15 - 20x. Ajouter 8 ml de milieu de culture pour inactiver la trypsine. Mélanger doucement en inversant le tube plusieurs fois.
- Faire passer la suspension de cellules à travers un tamis cellulaire de 70 pm placée au-dessus d'un 50 mL tube de centrifugation. Rincer le tube 15 ml de milieu de culture et filtrer le milieu à travers le tamis cellulaire pour recueillir le fluide dans le tube de 50 ml de la suspension cellulaire. Rincer la crépine cellulaire quelques fois avec un milieu de culture. Après rinçage, le volume final doit être d'environ 20 à 25 ml.
- Sédimenter les cellules à 200 xg pendant 10 min. Aspirer soigneusement autant moyen que possible, sans toucher le culot cellulaire. Resuspendre les cellules dans 1 ml de milieu de culture en utilisant une pipette de 1000. Ajouter 11 ml de milieu de culture préchauffée et mélanger doucement (pour éviter les bulles) à l'aide d'une pipette de 10 ml.
- Rincer le flacon T75 PDL revêtu une fois avec 5 ml de milieu de culture. Aspirer le milieu et à transférer la suspension cellulaire dans le ballon. Toutes les cellules dans la suspension sont étalés et nous trouvent généralement pas nécessaire de les compter, étant donné que les astrocytes ne peuvent pas être différenciés des autres cellules dans la suspension. Placer le ballon dans un humidifiée à 37 ° C incubateur avec une atmosphère de 5% de CO 2 fou deux jours.
- L' expansion et le maintien des astrocytes
- Remplacez la totalité du support pour le moment 2 premiers jours après l'étalement initial. Remplacez la totalité du support après chaque 3 d. Toujours préchauffer le milieu frais à 37 ° C avant l'addition aux cellules.
NOTE: Les astrocytes nécessitent 7-10 d pour atteindre environ 90% de confluence (les astrocytes apparaissent comme une monocouche tessellated dense, avec les microglies et les oligodendrocytes se trouvant sur le dessus et entremêlés). - Lorsque les astrocytes atteignent environ 90% de confluence, agiter le flacon pour éliminer les cellules gliales contaminantes:
- Retirer le flacon de l'incubateur et serrer le bouchon (phénolique) ou couvrir le port (filtrée). Pour supprimer microglie, agiter le flacon sur une plate-forme orbitale à 180 rpm pendant 1 h. Aspirer le milieu. Rincer une fois avec 5 ml préchauffé milieu de culture, aspirée et le remplacer par 12 ml de milieu de culture.
- Retirerles oligodendrocytes, retournent le ballon à la plate-forme orbitale et agiter à 250 tours par minute, 37 ° C pendant un minimum de 7 h, mais de préférence O / N.
- Aspirer le milieu. Rincer une fois avec 5 ml préchauffé milieu de culture, aspirée et le remplacer par 12 ml de milieu de culture. Remettre le ballon à l'incubateur.
- Lorsque 100% confluentes, diviser les astrocytes à l'aide des modes opératoires standard à un rapport de 1: 3 à 1: 2 avec 0,05% de trypsine-acide éthylènediaminetétra-acétique (EDTA). Un flacon T75 à 100% de confluence donne habituellement d' environ 4,0 x 10 6 cellules au total. En vertu de ce programme, les cultures peuvent généralement être divisés une fois par semaine.
NOTE: Lorsque les astrocytes atteignent la confluence, elles peuvent être récoltées et utilisées pour la différenciation hiPSC comme décrit ci - dessous dans le protocole étape 3.4. Les astrocytes peuvent être divisés au moins une fois sans perte notable de viabilité. Ils peuvent être conservés pendant jusqu'à 2 mois en culture. De l'expérience, primaire jour embryonnaire 18 astrocytes de rat progrdevenir essively différenciation terminale et / ou perdre leur viabilité après la séparation répétée. Bien qu'il soit possible de geler les astrocytes pour une utilisation future, nous préférons isoler les astrocytes de cerveaux embryonnaires frais en cas de besoin.
- Remplacez la totalité du support pour le moment 2 premiers jours après l'étalement initial. Remplacez la totalité du support après chaque 3 d. Toujours préchauffer le milieu frais à 37 ° C avant l'addition aux cellules.
2. Génération de rtTA / Ngn2 -positifs hiPSCs
NOTE: Les hiPSCs utilisées pour nos expériences ont été générées en interne par lentiviral transduction des fibroblastes humains avec les facteurs de reprogrammation cmyc, SOX2, OCT4 et KLF4.
NOTE: Pour la génération de rtTA / Ngn2 hiPSCs -positif, vecteurs lentiviraux sont utilisés pour intégrer de manière stable les transgènes dans le génome des hiPSCs. Le protocole pour la production de lentivirus a été publiée précédemment 22. Les détails des vecteurs d'encapsidation lentiviral qui sont utilisés pour produire les particules de lentivirus rtTA et Ngn2 sont prévus dans la Table des matières / équipement. Le vecteur de transfert utilisé pour le lentivirus rtTA est pLVX-EF1α- (Tet-On-Advanced) IRES-G418 (R); à- dire ce vecteur code pour un Tet-On transactivateur avancée sous le contrôle d'un promoteur EF1 constitutive et confère une résistance à l'antibiotique G418. Le vecteur de transfert utilisé pour le lentivirus Ngn2 est pLVX- (TRE-thight) - (SOURIS) Ngn2-PGK-puromycine (R); -à- dire ce vecteur code pour le gène de la souris neurogénine-2 sous le contrôle d'un promoteur Tet-commandé et le gène de résistance à la puromycine sous le contrôle d'un promoteur constitutif PGK. Par conséquent, en utilisant ces deux vecteurs de transfert, une ligne de hiPSC peut être créée pour lequel l'expression d'anticorps murins neurogénine-2 peut être induite par supplémenter le milieu avec de la doxycycline. Pour la transduction des hiPSCs, le surnageant avec les particules de lentivirus est utilisé (dénommé «suspension lentivirus» dans la suite du texte), à savoir l' esprithout concentration des particules en utilisant une ultracentrifugation.
- Plate les hiPSCs (jour 1)
NOTE: Les volumes qui sont mentionnés dans ce protocole supposent que les hiPSCs sont cultivées dans une plaque à 6 puits et que les cellules d'un puits sont récoltés. En outre, on suppose que les cellules sont étalées par la suite dans 12 puits d'une plaque à 12 puits.- Préparer 10 ml DMEM froid / F12 avec 1% (v / v) Sous-sol Membrane Matrix (BMM) pour obtenir dilué BMM. Ajouter 800 ul dilué BMM par puits d'une plaque à 12 puits. Laisser incuber pendant au moins 1 h dans un humidifiée à 37 ° C incubateur avec une atmosphère de 5% de CO 2. Avant utilisation, incuber la plaque pendant 1 h à température ambiante.
- Chaud 15 ml Essential 8 (E8) milieu avec 1% (v / v) de pénicilline / streptomycine, 9 ml de DMEM / F12 et 1 ml d'une solution de détachement cellulaire (CDS) à la température ambiante. Supplément milieu E8 avec la protéine kinase Rho-associé (ROCK) inhibiteur.
- Aspirer le milieu usé des hiPSCs und ajouter 1 mL CDS aux hiPSCs. Incuber pendant 3 à 5 min dans un humidifiée à 37 ° C incubateur avec une atmosphère de 5% de CO 2. Vérifier sous le microscope, si les cellules sont détachent les unes des autres.
- Ajouter 2 mL de DMEM / F12 dans le puits, suspendre doucement les cellules avec une pipette de 1 000 et de transférer les cellules à un tube de 15 ml. Ajouter 7 ml de DMEM / F12 à la suspension cellulaire. Faites tourner les cellules à 200 xg pendant 5 min.
- Aspirer le surnageant et ajoute 2 ml de milieu préparé à E8. Obtenir une suspension cellulaire dans laquelle sont dissociées les hiPSCs (ne forment pas des amas cellulaires) en plaçant l'extrémité d'une pipette de 1000 ul contre le côté du tube de 15 ml et remise en suspension en douceur les cellules. Vérifiez sous le microscope si les cellules sont dissociées.
- Déterminer le nombre de cellules (cellules / ml) en utilisant une chambre d'hémocytomètre.
NOTE: Une plaque à 6 puits et à 80 - 90% de confluence donne habituellement 3,0 - 4,0 x 10 6 cellules au total. - Aspirerle BMM dilué dans les puits de la plaque à 12 puits. Diluer les cellules pour obtenir une suspension cellulaire de 3,0 x 10 4 cellules / mL. Planche 1 ml de la suspension cellulaire par puits de la plaque à 12 puits. Placez le puits plaque O / N 12 dans un humidifié 37 ° C incubateur avec une atmosphère de 5% de CO 2.
- Transduire les cellules iPS avec rtTA et Ngn2 lentivirus (jour 2)
- 12 ml de milieu chaud E8 avec 1% (v / v) de pénicilline / streptomycine à température ambiante. Compléter le milieu E8 avec un inhibiteur de la roche et du polybrène à une concentration finale de 8 pg / ml dans le milieu E8.
- Décongeler les aliquotes avec suspension lentivirus. Ajouter du polybrène à une concentration finale de 8 pg / ml à la suspension lentivirus. Aspirer le milieu usé et ajouter 1 mL du milieu E8 préparé à chaque puits.
- Effectuer la transduction avec des quantités différentes de la rtTA - et les suspensions Ngn2 de. Par exemple, transduÇe les hiPSCs en ajoutant 100 pi à la fois du -lentivirus rtTA et Ngn2 -lentivirus suspension à un puits de la plaque à 12 puits. Pour les autres puits, utiliser 200 pi, 300 pi, 400 pi et 500 pi suspension lentivirus au lieu de 100 pi. Les hiPSCs de deux puits de la plaque 12 puits ne doivent pas être transduites; ils serviront de contrôles lors de la sélection.
REMARQUE: Les transductions sont de préférence effectuées en double, de sorte que l'efficacité de transduction peut être évaluée de façon plus précise après le début de la sélection (voir protocole étape 2.2.4). La quantité de suspension de lentivirus qui est nécessaire pour transduire efficacement la plupart des hiPSCs dépend du titre de la suspension lentivirus et la ligne hiPSC qui est utilisé. Dans cette étude, nous utilisons habituellement 100 - 500 ul de lentivirus suspension pour transduire les hiPSCs. - Placer la plaque à 12 puits dans un humidifié 37 ° C incubateur avec une atmosphère de 5% de CO2 pendant 6 h. Avant la fin de la période d'incubation de 6 heures, 12 ml de milieu chaud E8 avec 1% (v / v) de pénicilline / streptomycine et de 12 tampon phosphate salin de Dulbecco mL (DPBS) à température ambiante. Supplément moyen E8 avec un inhibiteur de ROCK.
- Aspirer le milieu E8 passé. Laver chaque puits avec 1 ml DPBS. Ajouter 1 mL du milieu E8 préparé à chaque puits. Placez le puits plaque O / N 12 dans un humidifié 37 ° C incubateur avec une atmosphère de 5% de CO 2.
- Actualiser le moyen E8 (jour 3)
- 12 ml de milieu chaud E8 avec 1% (v / v) de pénicilline / streptomycine à TA. Aspirer le milieu usé des puits de la plaque à 12 puits et ajouter 1 mL du milieu E8 préparé à chaque puits. Placez le puits plaque O / N 12 dans un humidifié 37 ° C incubateur avec une atmosphère de 5% de CO 2.
- Effectuez la sélection avec la puromycine et G418 (jour 4-8)
NOTE: En fonction du taux de la hanche de la division cellulaireligne de SC et de l'efficacité de la transduction de lentivirus, les cellules peuvent atteindre 70 - 80% de confluence au cours de la période de sélection, à quel point les cultures doivent être divisés. Parce que le moment de la séparation ne peut être prédit à l'avance, il ne sera pas mentionnée dans le protocole. Cependant, au lieu de rafraîchir le milieu E8 complété avec les concentrations mentionnées de puromycine et G418, on peut diviser la culture hiPSC comme une culture normale de hiPSC (y compris l'étalement des cellules sur des plaques revêtues de vitronectine). La seule exception est que le milieu E8 devrait être complété par les concentrations mentionnées des antibiotiques pour continuer la sélection.- 12 ml de milieu chaud E8 avec 1% (v / v) de pénicilline / streptomycine à température ambiante. Ajouter puromycine et G418 pour la sélection; différentes quantités des antibiotiques sont ajoutés au cours de la période de sélection (tableau 1).
- Estimer l'efficacité de la transduction en estimant le pourcentage de la puromycine et G418-cellules résistantes. Pour estimer le pourcentage de cellules résistantes, estimer le pourcentage de cellules mortes (cellules non résistantes) pour les différentes conditions (cultures transduites avec différentes quantités de suspension lentivirus) et pour les cellules nontransduced (les cellules qui servent de commande de sélection). Calculer le pourcentage de cellules résistantes à [100% - (pourcentage de cellules mortes)].
Remarque: si les transductions avec les différentes quantités de suspension lentivirus ont été réalisées en double exemplaire, l'efficacité de transduction peut être évaluée de façon plus précise. L'état des cellules non transduites sert de contrôle de sélection; les pourcentages de cellules mortes pour des cultures transduites avec différentes quantités de suspension lentivirus doivent être inférieurs. Le pourcentage estimé de cellules résistantes est utilisé pour choisir les hiPSCs qui sont susceptibles positif pour les deux transgènes. D'une manière générale, on choisit les hiPSCs de l'état de transduction étaient> 90% de la cellules survivre à la période de sélection de 5 d. - Aspirer le milieu usé des hiPSCs et ajouter 1 mL du milieu E8 préparé aux puits. Placez le puits plaque O / N 12 dans un humidifié 37 ° C incubateur avec une atmosphère de 5% de CO 2.
| La concentration finale de G418 | La concentration finale de la puromycine | |
| jour 4 | 250 pg / ml | 2 pg / ml |
| jour 5 | 250 pg / ml | 2 pg / ml |
| jour 6 | 250 pg / ml | 1 pg / ml |
| jour 7 | 250 pg / ml | 1 pg / ml |
| jour 8 | 250 pg / ml | 1 pg / ml |
Tableau 1: Concentrations d'antibiotiques au cours de la période de sélection. Les concentrations de la puromycine et G418 lors de la 5 j de la période de sélection.
- Arrêtez la sélection et commencer la culture régulière (jour 9 et plus tard)
- Après la 5d période de sélection, la culture Les hiPSCs -positif rtTA / Ngn2 comme hiPSCs normaux, à l'exception que le milieu E8 des cellules est complétée par du G418 à une concentration finale de 50 pg / ml et avec la puromycine à une concentration finale de 0,5 pg / ml.
NOTE: Les cellules peuvent maintenant être congelés (selon les protocoles standards pour la cryoconservation des cellules) pour servir de sauvegarde. Ceci est une étape importante pour la reproductibilité du protocole de différenciation, car il permet l'utilisation d'un même lot de rtTA / Ngn2 hiPSCs -positif pour de nombreuses expériences futures de différenciation.
- Après la 5d période de sélection, la culture Les hiPSCs -positif rtTA / Ngn2 comme hiPSCs normaux, à l'exception que le milieu E8 des cellules est complétée par du G418 à une concentration finale de 50 pg / ml et avec la puromycine à une concentration finale de 0,5 pg / ml.
3. Différenciation des rtTA / Ngn2 hiPSCs -positifs à Neurones sur 6 puitsAME et verre Lamelles
NOTE: Dans ce protocole, les détails sont fournis pour différencier rtTA / Ngn2 hiPSCs -positifs sur deux substrats différents, à savoir AME 6 puits (dispositifs composés de 6 puits indépendants avec 9 enregistrement et 1 référence microélectrodes incorporés par puits) et des lamelles de verre en les puits d'une plaque à 24 puits. Les protocoles, cependant, peuvent facilement être adaptées à des substrats plus grands (par exemple, pour les puits de plaques 12 ou 6 puits), par extrapolation des valeurs mentionnées en fonction de la surface.
- Préparer les AME ou des lamelles de verre (jour 0 et le jour 1)
- Le jour avant le début de la différenciation, stériliser les AME selon les recommandations du fabricant.
- Diluer la protéine d'adhésion poly-L-ornithine (OLP) dans de l'eau ultrapure stérile jusqu'à une concentration finale de 50 pg / ml. Enduire la surface de l'électrode active des AME 6 puits en plaçant un 100 pidéposer de l'OLP dilué dans chaque puits. Placez les lamelles dans les puits de la plaque de 24 puits en utilisant des pinces stériles. Ajouter 800 pi de l'OLP dilué dans chaque puits. Empêcher les lamelles de flotter en les poussant vers le bas avec la pointe de la pipette de 1000 pi.
- Incuber les AME 6 puits et 24 puits plaque O / N dans un humidifié 37 ° C incubateur avec une atmosphère de 5% de CO 2. Le lendemain, aspirer l'OLP diluée. Laver les surfaces de verre des AME 6 puits et les lamelles deux fois avec de l'eau ultrapure stérile.
- Diluer la laminine dans le froid du DMEM / F12 à une concentration finale de 20 pg / mL (pour les AME 6 puits) et 10 pg / mL (pour les lamelles de verre). Immédiatement recouvrir la surface d'électrode active de la MEA à 6 puits en plaçant une goutte de 100 pl dans chaque puits. De même, ajouter 400 ul de la laminine dilué dans chaque puits de la plaque de 24 puits pour enrober les lamelles. Empêcher les lamelles de flotter en les poussant vers le bas avec la pointe de la pipette de 1000 pi.
- Incuber les AME 6 puits et plaque à 24 puits dans un humidifié 37 ° C incubateur avec une atmosphère de 5% de CO 2 pendant au moins 2 h.
- Plate les hiPSCs (jour 1)
NOTE: Les volumes qui sont mentionnés dans les étapes 3.2.1 - 3.2.4 supposent que les hiPSCs -positifs rtTA / Ngn2 sont cultivées dans une plaque de 6 puits et que les cellules d'un puits sont récoltés. Les volumes requis pour l'étalement des cellules sur les AME 6 puits et / ou des lamelles couvre-objet dépend du nombre des AME 6 puits et / ou le nombre de lamelles qui sont utilisés dans l'expérience; les chiffres indiqués dans les étapes 3.2.6 - 3.2.8 permettent l'extension aux différentes tailles d'expérience.- Chaud DMEM / F12, CDS et moyen E8 avec 1% (v / v) pénicilline / streptomycine à R / T. Ajouter la doxycycline à une concentration finale de 4 ug / mL et un inhibiteur de ROCK au milieu E8.
- Aspirer le milieu usé des hiPSCs -positifs rtTA / Ngn2 et ajouter 1 mL CDS aux hiPSCs. Incuber pendant 3 à 5 min dans un humidifiée à 37 ° C incubateur avec une atmosphère de 5% de CO 2. Vérifier sous le microscope, si les cellules sont détachent les unes des autres.
- Ajouter 2 mL de DMEM / F12 dans le puits, suspendre doucement les cellules avec une pipette de 1 000 et de transférer les cellules à un tube de 15 ml. Ajouter 7 ml de DMEM / F12 à la suspension cellulaire. Faites tourner les cellules à 200 xg pendant 5 min.
- Aspirer le surnageant et ajoute 2 ml de milieu préparé à E8. Dissocier les hiPSCs en mettant la pointe d'une pipette de 1000 pi contre le côté du tube de 15 ml et la remise en suspension doucement les cellules. Vérifiez sous le microscope si les cellules sont dissociées.
- Déterminer le nombre de cellules (cellules / ml) en utilisant une chambre d'hémocytomètre.
NOTE: Une plaque de 6 puits et à 80 - 90% de confluence donne habituellement 3,0 - 4,0 x 10 6 cellules au total. - Aspirer le laminine dilué. Pour les AME 6 puits, diluer les cellules pour obtenir un CEll suspension de 7,5 x 10 5 cellules / ml. Plaquer les cellules en ajoutant une goutte de 100 pl de la suspension cellulaire sur la surface de l'électrode active dans chaque puits des AME 6 puits. Les lamelles couvre -objet , on dilue les cellules pour obtenir une suspension de cellules de 4,0 x 10 4 cellules / mL. Plaquer les cellules par addition de 500 ul de la suspension cellulaire dans les puits de la plaque à 24 puits.
NOTE: La densité cellulaire finale sur les AME est plus élevé que sur les lamelles (figure 1A et B). Nous avons constaté que cette densité cellulaire élevée était nécessaire pour un enregistrement correct de l'activité du réseau. Dans le protocole, les chiffres sont fournis qui avéré être optimal pour les dosages. - Placez les AME 6 puits et plaque à 24 puits dans un humidifié 37 ° C incubateur avec une atmosphère de 5% de CO 2 pendant 2 h (AME) ou O / N (plaque à 24 puits).
- Après 2 h, ajouter avec précaution 500 ul du milieu E8 préparé à chaque puits des AME 6 puits. Placer le 6-nousll AME O / N dans un humidifié 37 ° C incubateur avec une atmosphère de 5% de CO 2.
- Changer le support (jour 2)
- Le lendemain, la préparation du DMEM / F12 avec 1% (v / v), N-2 supplément, 1% (v / v) d'acides aminés non essentiels et 1% (v / v) de pénicilline / streptomycine. Ajouter recombinante neurotrophine-3 humaine (NT-3) à une concentration finale de 10 ng / ml, le facteur neurotrophique humain recombinant dérivé du cerveau (BDNF) à une concentration finale de 10 ng / ml, et la doxycycline à une concentration finale de 4 pg / mL. Chauffer le milieu à 37 ° C.
- Ajouter la laminine à une concentration finale de 0,2 pg / ml dans le milieu. Filtrer le milieu résultant. Aspirer le milieu usé des puits des AME 6 puits et la plaque 24 puits et le remplacer par le milieu préparé. Incuber les AME 6 puits et 24 puits plaque O / N dans un humidifié 37 ° C incubateur avec une atmosphère de 5% de CO 2.
- Ajouter astrocytes de rat (jour 3)
NOTE: Les volumes qui sont mentionnés dans ce protocole supposent que les astrocytes de rat sont cultivées dans des flacons de culture T75. Il est essentiel que les astrocytes de rat qui sont ajoutés aux cultures sont de bonne qualité. Nous utilisons deux critères pour vérifier si les astrocytes de rat sont de bonne qualité. Tout d'abord, la culture d'astrocytes de rat devrait être en mesure de croître confluentes dans les dix jours après l'isolement des rats cerveaux embryonnaires. En second lieu , après avoir fendu la culture d'astrocytes de rat, les astrocytes de rats doivent être capables de former un confluentes monocouche tesselé (figure 1C). Si la culture d'astrocytes de rat ne remplit pas ces deux critères, nous vous conseillons de ne pas utiliser cette culture pour des expériences de différenciation.- Chaud de trypsine-EDTA à 0,05% à la température ambiante. Chauffer le DPBS et DMEM / F12 avec 1% (v / v) pénicilline / streptomycine à 37 ° C.
- Aspirer le milieu usé de la culture d'astrocytes de rat. Laver la culture en ajoutant 5 ml DPBS et bruire autour doucement.
- Aspirmangé le DPBS et ajouter 5 ml 0,05% de trypsine-EDTA. Swish la trypsine-EDTA autour doucement. Incuber dans un humidifiée à 37 ° C incubateur avec une atmosphère de 5% de CO 2 pendant 5 - 10 min.
- Vérifiez sous le microscope si les cellules sont détachées. Détachez les dernières cellules en frappant le ballon à quelques reprises.
- Ajouter 5 ml de DMEM / F12 dans le ballon. Triturer les cellules doucement à l'intérieur du flacon avec une pipette de 10 ml. Recueillir la suspension cellulaire dans un tube de 15 ml. Spin le tube à 200 xg pendant 8 min.
- Aspirer le surnageant et remettre en suspension les cellules dans 1 ml de DMEM / F12. Déterminer le nombre de cellules (cellules / ml) en utilisant une chambre d'hémocytomètre.
- Ajouter 7,5 x 10 4 astrocytes par puits des AME 6 puits. Ajouter 2,0 x 10 4 astrocytes par puits de la plaque de 24 puits. Incuber les AME et la plaque O 24 puits / N dans un humidifié 37 ° C incubateur avec une atmosphère de 5% de CO 2.
- Changer le support (jour 4)
- préparer le milieu Neurobasal avec 2% (v / v) B-27 supplément, 1% (v / v) de L-alanyl-L-glutamine et 1% (v / v) de pénicilline / streptomycine. Ajouter le NT-3 à une concentration finale de 10 ng / ml de BDNF à une concentration finale de 10 ng / ml, et la doxycycline à une concentration finale de 4 ug / ml. En outre, ajouter la cytosine β-D arabinofuranoside à une concentration de 2 uM.
REMARQUE: la cytosine β-D arabinofuranoside est ajouté au milieu pour inhiber la prolifération des astrocytes et de tuer les hiPSCs restants qui ne sont pas différencier en neurones. - Filtrez le moyen et chaud à 37 ° C. Aspirer le milieu usé des puits des AME 6 puits et la plaque 24 puits et le remplacer par le milieu préparé. Maintenir les AME 6 puits et la plaque de 24 puits dans un humidifié 37 ° C incubateur avec une atmosphère de 5% de CO 2.
- préparer le milieu Neurobasal avec 2% (v / v) B-27 supplément, 1% (v / v) de L-alanyl-L-glutamine et 1% (v / v) de pénicilline / streptomycine. Ajouter le NT-3 à une concentration finale de 10 ng / ml de BDNF à une concentration finale de 10 ng / ml, et la doxycycline à une concentration finale de 4 ug / ml. En outre, ajouter la cytosine β-D arabinofuranoside à une concentration de 2 uM.
- Actualiser le moyen (jour 6-28)
NOTE: A partir de 6 jours, rafraîchir la moitiédu milieu tous les deux jours. De 10 jours à compter, le milieu est complété avec du FBS pour soutenir la viabilité des astrocytes.- Préparer le milieu Neurobasal avec 2% (v / v) B-27 supplément, 1% (v / v) de L-alanyl-L-glutamine et 1% (v / v) de pénicilline / streptomycine. Ajouter le NT-3 à une concentration finale de 10 ng / ml de BDNF à une concentration finale de 10 ng / ml, et la doxycycline à une concentration finale de 4 ug / ml. A partir du jour 10 avant, également compléter le milieu avec 2,5% (v / v) de FBS. Filtrer le milieu résultant et chaud à 37 ° C.
- Retirer la moitié du milieu usé des puits des AME 6 puits et la plaque de 24 puits en utilisant une pipette de 1000 et le remplacer par le milieu préparé. Maintenir les AME 6 puits et la plaque de 24 puits dans un humidifié 37 ° C incubateur avec une atmosphère de 5% de CO 2.
4. Établir le profil neurophysiologique de Neurones hiPSC dérivés
NOTE: Deux à trois semaines après l'enla production de la différenciation, les neurones hiPSC dérivés peuvent être utilisés pour différentes analyses en aval. Dans cette section, des exemples de certaines analyses en aval sont donnés qui peuvent être effectuées pour établir le profil neurophysiologique des neurones hiPSC dérivés.
- Caractériser l'activité de réseau neuronal utilisant AME
- Enregistrement 20 min de l'activité électrophysiologique des neurones hiPSC dérivés de culture sur les AME. Pendant l'enregistrement, maintenir la température à 37 ° C, et d' empêcher l' évaporation et les changements de pH du milieu en gonflant une constante, faible débit de gaz humidifié (5% de CO 2, 20% de O 2, 75% N 2) sur la MEA .
- Après 1200X amplification (MEA 1060, MCS), échantillonner le signal à 10 kHz en utilisant la carte d'acquisition de données MCS. Analyser les données (pointes et de détection rafale) à l' aide d' un logiciel personnalisé 23.
- Caractériser l'activité électrophysiologique unicellulaire
- Transférez les lamelles contenant les cultures neuronales hiPSC dérivées à une chambre d'enregistrement à platine fixe immergé dans un microscope droit. Enregistrement 20 min de l' action spontanée des courants postsynaptiques potentiels évoqués (sEPSC) 24. Détecter l'événement synaptique en utilisant le programme neuroscientifique.
- Caractériser la morphologie neuronale et d' expression synapsine
- Fix et tacher les neurones hiPSC dérivés pour MAP2, synapsine-1/2, et PSD-95 22, 24, 25. Quantifier le nombre de puncta de Synapsin-1/2 et PSD-95 en utilisant un logiciel d'analyse d'image.
Résultats
Ici , nous avons réussi à modifier un protocole dans lequel hiPSCs sont différenciés directement dans les neurones corticaux par surexpression du facteur de transcription neurogénine-2 12 et nous l' avons adapté pour l'utilisation des AME. Cette approche est rapide et efficace qui nous permet d'obtenir des neurones fonctionnels et l'activité du réseau déjà au cours de la troisième semaine après l'induction de la différenciation.
Au cours du protocole de différenciation, les cellules ont commencé à ressembler morphologiquement neurones: les petits procédés ont été formés et les neurones ont commencé à relier les uns aux autres (figure 1A). Nous avons établi un profil neurophysiologique des neurones dérivés d'une lignée de hiPSC saine contrôle, en mesurant leur morphologie neuronale et les propriétés synaptiques au cours du développement. neurones hiPSC dérivés ont été colorées pour MAP2 et synapsine-1/2 à différents jours après le débutde différenciation (figure 2A). Les neurones dérivés montrent la morphologie des neurones matures déjà 3 semaines après l'induction de la différenciation. Le nombre de synapsine-1/2 lacrymaux (une mesure du nombre de synapses) a été quantifiée sur la base / 2 colorations immunocytochimie 1 synapsine. Le nombre de synapsine-1/2 lacrymaux a augmenté au fil du temps, ce qui suggère que le niveau de connectivité neuronale augmente également (figure 2B). Le nombre de synapsine-1/2 lacrymaux 23 jours après l'induction de la différenciation était similaire dans les deux lignées indépendantes IPS (figure 2C). A 23 DIV plus synapsin1 / 2 puncta ont été juxtaposée à PSD-95 lacrymaux, qui est indicatif de synapses fonctionnelles (Figure 2D).
En accord avec les résultats décrits par Zhang et al., Nous avons généré une population de neurones corticaux couche supérieure excitateurs, confirmée par m corticales spécifiques de sous - type pan-neuronale et arkers tels que BRN2 et SATB2 (couche II / III). Nous n'avons pas observé les neurones positifs pour les neurones de la couche profonde CTIP2 (couche V) ou FOXP2 (couche VI) (Figure 2E et F)
Pour caractériser l'activité électrophysiologique des neurones hiPSC dérivés, nous avons utilisé en cours de cellules entières et des enregistrements de serrage de tension, à savoir les propriétés intrinsèques et entrée excitatrice sur ces neurones ont été mesurés au cours du développement. Les neurones ont été capables de générer des potentiels d'action déjà une semaine après la différenciation et le pourcentage d'ensemencement des cellules a été augmenté au cours du temps (figure 2G et H). En outre, les neurones ont reçu entrée synaptique excitatrice déjà une semaine après l'induction de la différenciation: à la fois la fréquence et l' amplitude de l'entrée synaptique excitatrice a augmenté au cours du développement (figure 2I - K).
nt "fo: keep-together.within-page =" 1 "> Pour mieux comprendre comment l' activité cellule unique combine pour former des fonctions au niveau du réseau, il est essentiel d'étudier comment les neurones travaillent de concert in vitro réseaux neuronaux cultivés sur les AME. constituent un modèle expérimental précieux pour l' étude de la dynamique neuronales. Nous avons enregistré 20 min de l' activité réseau électrophysiologique des neurones dérivés d'une lignée de hiPSC saine maîtrise en culture sur 6 puits AME (Figure 2M). Quelques semaines après l'induction de la différenciation, les neurones dérivé de hiPSCs sain de contrôle formé des réseaux neuronaux fonctionnellement actifs, montrant des événements spontanés (0,62 ± 0,05 pic / s; Figure 2N). A ce stade de développement (soit 16 jours après le début de la différenciation) pas d' événements synchrones impliquant tous les canaux . des AME sont détectés (Figure 2O) le niveau d'activité du réseau a augmenté au cours du développement: au cours de la quatrième semaine après t il induction de la différenciation, les réseaux de neurones a montré un niveau élevé de l' activité spontanée (2,5 ± 0,1 pic / s; Figure 2N) dans tous les puits du dispositif. Les réseaux également exposés rafales synchrones de réseau (4,1 ± 0,1 rafale / min, Figure 2O) avec longue durée (2.100 ± 500 ms).
Figure 1: hiPSC Différenciation dans Neurones. R. Trois points de temps de différenciation en neurones hiPSCs sur les lamelles couvre -objet . B. Placage des hiPSCs sur les AME. C. astrocytes à 100% de confluence dans T75 flacon ( à noter que les cellules forment une monocouche tessellated). Barres d'échelle: 150 um. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
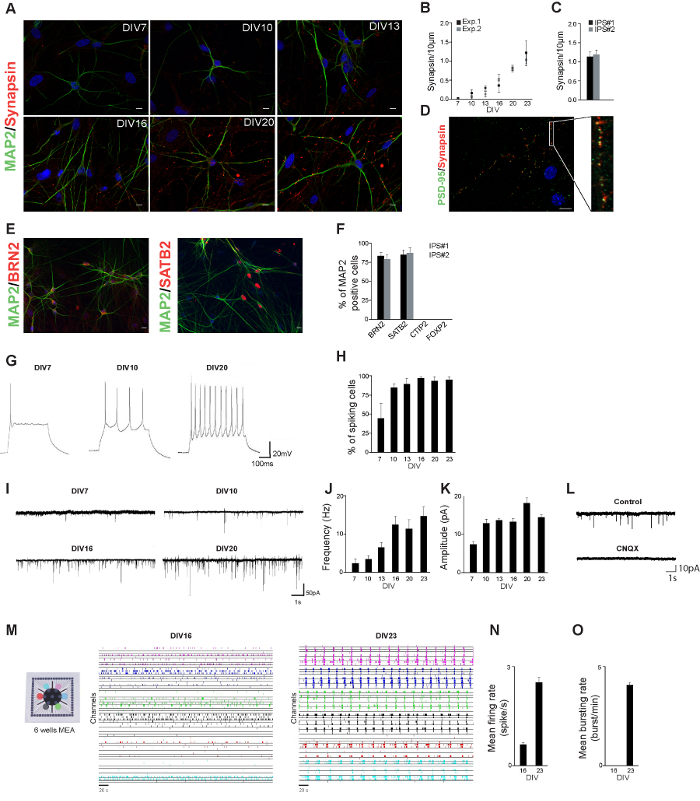
Figure 2. hiPSC dérivées Neurones Caractérisation. Neurones A. hiPSC dérivés ont été colorées pour MAP2 (vert) et synapsine-1/2 (rouge) à différents jours après le début de la différenciation. Barre d'échelle: 10 um. B. Quantification de la synapsine lacrymaux dans deux expériences indépendantes. Dans chacune des expériences d'au moins 10 cellules ont été analysées. C. Quantification de puncta synapsine à DIV23 dans les neurones dérivés de deux lignes IPS indépendants. Neurones D. hiPSC dérivées ont été colorées pour PSD-95 (vert) et Synapsin-1/2 (rouge) 23 jours après le début de la différenciation. puncta Synapsin sont juxtaposées à PSD-95 lacrymaux. Neurones E. hiPSC dérivés ont été colorées pour MAP2 (vert) et BRN2 (rouge) ou SATB2 (rouge) 23 jours après le début de la différenciation. F. Pourcentage de cellules positives que MAP2étaient positifs pour les marqueurs indiqués. G. représentatifs des enregistrements de serrage actuelle montrant que les potentiels d'action peuvent être générés dès 7 jours après le début de la différenciation. H. Pourcentage de cellules à différents jours après l'induction de la différenciation qui montrent un ou plusieurs potentiels d'action. I. traces représentatifs de courants postsynaptiques excitateurs (de EPSCs) reçues par les neurones hiPSC dérivés à différents jours après différenciation. J. Fréquence des courants postsynaptiques excitateurs au cours du développement. K. Amplitude des courants postsynaptiques excitateurs au cours du développement. L. Représentant des traces d'enregistrements RPEC sans (témoin) et avec CNQX (CNQX). M. Neurones dérivée d'une ligne hiPSC ont été cultivées sur un MEA à 6 puits et l' activité du réseau est représenté pour les réseaux neuronaux hiPSC dérivés 16 et 23 jours après l'induction de la différenciation. L'activité enregistrée à partir dechaque (taux d'échantillonnage de 10 kHz) et est indiqué par une couleur différente (5 min de la 20 min d'enregistrement sont représentés). N. taux de tir 16 et 23 jours après l'induction de la différenciation. O. taux Débordant 16 et 23 jours après l'induction de la différenciation. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
Compte tenu de ces résultats, la qualité des neurones hiPSC dérivé résultant peut être évaluée en faisant un profil neurophysiologique des cellules. Autrement dit, trois à quatre semaines après le début de la différenciation, de la morphologie, synapsine-1/2 expression et électrophysiologique des neurones peuvent être évalués. A ce point du temps, les neurones hiPSC dérivés devraient montrer une morphologie neuronale-like, pour être MAP2, synapsine / PSD-95 immunocytochimie lors de l'exécution positive et à Exhibit l'activité électrophysiologique spontanée (les deux à la seule cellule et au niveau du réseau).
Discussion
Ici , nous avons mis en place un protocole hiPSC-différenciation efficace publiée par Zhang et al. (2013) 12 pour mesurer l'activité de réseau des réseaux neuronaux hiPSC dérivés sur les AME. Nous avons adapté le protocole original en créant une ligne de hiPSC -positifs rtTA / Ngn2 avant induction de la différenciation neuronale. Cette étape supplémentaire nous permet de contrôler la densité des cellules neuronales de la MEA. Le contrôle de la densité neuronale était une condition préalable importante pour adapter le protocole aux AME et d'assurer la cohérence. Pour mesurer l'activité des réseaux de neurones en utilisant les AME, les neurones ont besoin pour former des réseaux denses directement au - dessus des électrodes de MEA 17,18. Cela exige nécessairement un contrôle serré sur la densité de placage des neurones. La ligne de hiPSC -positifs rtTA / Ngn2 permet un contrôle de la densité des neurones , car cette tactique ne repose pas sur transductions lentiviraux aiguës de hiPSCs avant la différenciation;par conséquent , la ligne de hiPSC -positif rtTA / Ngn2 élimine pratiquement toute variation du rendement final en raison, par exemple, la toxicité et l' efficacité lentiviral d'infection variable.
Une autre étape critique du procédé expérimental est le nombre d'astrocytes de rat qui sont co-cultivées avec les hiPSCs différenciant. Les astrocytes contribuent activement à l'affinement de développer des circuits neuronaux en contrôlant la formation des synapses, la maintenance et l'élimination, qui sont tous des processus importants pour le fonctionnement neuronal. Le protocole présenté dans ce document est très astrocyte dépendante: à pleine maturité et forment des synapses fonctionnelles, les neurones ont besoin de soutien des astrocytes. Nous avons connu que le nombre d'astrocytes devrait être à peu près égal au nombre de neurones hiPSC dérivés pour soutenir la maturation des neurones et la formation de réseaux de neurones présentant une activité spontanée. Depuis nos astrocytes protocole rendements cul de cellules primairestures ayant une durée de vie limitée, l'isolement des astrocytes de rat doit être effectué régulièrement.
Notre adaptation du protocole publié par Zhang et al. (2013) 12 pour une utilisation avec la technologie MEA sera probablement améliorer considérablement notre capacité à étudier l'activité de réseau de réseaux hiPSC dérivés. Auparavant, les protocoles utilisés pour l' étude des réseaux neuronaux hiPSC dérivés des AME comptaient sur les procédures de différenciation chronophages 13-16. Le protocole de Zhang et al. (2013) fournit une alternative rapide et notre modification supprime une source de variabilité, ce qui rend maintenant plus possible d'utiliser des neurones hiPSC dérivés en combinaison avec la technologie de la MEA, en particulier dans un haut débit ou d'études pharmacologiques. En outre, parce que la méthode publiée par Zhang et al. (2013) 12 cède une population homogène de la couche supérieure des neurones corticaux, notre protocole adapté rend possible des études ciblées dans le réseau unctivité de ce sous-ensemble neuronal particulier.
Néanmoins, cette approche présente plusieurs limites. Tout d' abord, l'homogénéité des cultures peut également être considéré comme un inconvénient, parce que les cultures sont moins susceptibles de ressembler à des réseaux in vivo, où les différentes classes de neurones (c. -à- neurones excitateurs et inhibiteurs) constituent un réseau hétérogène. Pour améliorer encore l'utilisation des neurones hiPSC dérivés avec la technologie MEA, il sera important d'élaborer des protocoles de différenciation rapide (basée transgène) pour d'autres populations de cellules neuronales. Si les protocoles deviennent disponibles, les réseaux in vitro pourraient imiter plus étroitement les réseaux in vivo. En second lieu , à l' heure actuelle des astrocytes de rat doit être ajouté aux neurones hiPSC dérivés d'aide à la croissance, et par conséquent le réseau neuronal résultant est pas un réseau de neurones stricto sensu humaine. protocoles fiables pour différencier en astrocytes hiPSCs peut dans le sol avenirve ce problème 26. Troisièmement, les réseaux de neurones bidimensionnels, tels que décrits ici, sont un modèle d'étude limitée des réseaux complexes tridimensionnelles in vivo des neurones. Heureusement, des protocoles décrivant des cultures tridimensionnelles de neurones primaires de rat en combinaison avec la technologie de la MEA sont déjà disponibles 27,28. Prospectivement, la combinaison de protocoles de différenciation rapides pour obtenir des neurones et des astrocytes hiPSC dérivés avec des techniques de culture en trois dimensions et de la technologie de MEA devrait fournir un nouvel aperçu des mécanismes biologiques sous-jacents des troubles neurologiques.
Déclarations de divulgation
The authors have nothing to disclose.
Remerciements
The authors thank Jessica Classen for performing the whole-cell patch-clamp experiments. The hiPSCs used in our experiments were kindly provided by Huiqing Zhou and Willem van den Akker from the Radboud University Nijmegen. The transfer vectors used in this protocol were kindly provided by Oliver Brüstle, Philipp Koch and Julia Ladewig from the University of Bonn Medical Centre.
matériels
| Name | Company | Catalog Number | Comments |
| Lebovitz's L-15 medium | Gibco | 11415-064 | |
| B-27 supplement | Gibco | 0080085SA | |
| Poly-D-Lysine (PDL) | Sigma-Aldrich | P6407 | |
| Ca2+/Mg2+-free HBSS | Gibco | 14175-095 | |
| 0.05% Trypsin-EDTA | Gibco | 25300-054 | |
| 2.5% Trypsin | Gibco | 15090-046 | |
| High-glucose DMEM | Gibco | 11965-092 | |
| FBS (Fetal Bovine Serum) | Sigma-Aldrich | F2442-500ML | |
| Penicillin/Streptomycin | Sigma-Aldrich | P4333 | |
| 70 µm cell strainer | BD Falcon | 352350 | |
| DPBS | Gibco | 14190-094 | |
| psPAX2 lentiviral packaging vector | Addgene | Plasmid #12260 | |
| pMD2.G lentiviral packaging vector | Addgene | Plasmid #12259 | |
| Basement membrane matrix | Gibco | A1413201 | |
| DMEM/F12 | Gibco | 11320-074 | |
| Cell detachment solution | Sigma-Aldrich | A6964 | |
| E8 medium | Gibco | A1517001 | |
| ROCK inhibitor | Gibco | A2644501 | Alternatively, ROCK inhibitors like thiazovivin can be used. |
| Polybrene | Sigma-Aldrich | H9268-5G | |
| G418 | Sigma-Aldrich | G8168-10ML | |
| Puromycin | Sigma-Aldrich | P9620-10ML | |
| Vitronectin | Gibco | A14700 | |
| 6-well MEAs | Multi Channel Systems | 60-6wellMEA200/30iR-Ti-tcr | |
| Glass coverslips | VWR | 631-0899 | |
| Poly-L-Ornithine (PLO) | Sigma-Aldrich | P3655-10MG | |
| Laminin | Sigma-Aldrich | L2020-1MG | |
| Doxycyclin | Sigma-Aldrich | D9891-5G | |
| N-2 supplement | Gibco | 17502-048 | |
| Non-essential amino acids | Sigma-Aldrich | M7145 | |
| NT-3, human recombinant | Promokine | C66425 | |
| BDNF, human recombinant | Promokine | C66212 | |
| Trypsin-EDTA | Gibco | 25300-054 | |
| L-alanyl-L-glutamine | Gibco | 35050-038 | |
| Neurobasal medium | Gibco | 21103-049 | |
| Cytosine β-D-arabinofuranoside | Sigma-Aldrich | C1768-100MG | |
| Straight fine-tipped forceps | Fine Science Tools | 11251 | |
| Fine-tipped spring scissors | Fine Science Tools | 91500-09 |
Références
- Ahfeldt, T., Litterman, N. K., Rubin, L. Studying human disease using human neurons. Brain Res. , (2016).
- Zhang, S. C., Wernig, M., Duncan, I. D., Brustle, O., Thomson, J. A. In vitro differentiation of transplantable neural precursors from human embryonic stem cells. Nat Biotechnol. 19 (12), 1129-1133 (2001).
- Perrier, A. L., et al. Derivation of midbrain dopamine neurons from human embryonic stem cells. Proc Natl Acad Sci USA. 101 (34), 12543-12548 (2004).
- Wu, H., et al. Integrative genomic and functional analyses reveal neuronal subtype differentiation bias in human embryonic stem cell lines. Proc Natl Acad Sci USA. 104 (34), 13821-13826 (2007).
- Chambers, S. M., Fasano, C. A., Papapetrou, E. P., Tomishima, M., Sadelain, M., Studer, L. Highly efficient neural conversion of human ES and iPS cells by dual inhibition of SMAD signaling. Nat Biotechnol. 27 (3), 275-280 (2009).
- Shi, Y., Kirwan, P., Livesey, F. J. Directed differentiation of human pluripotent stem cells to cerebral cortex neurons and neural networks. Nat Protoc. 7, 1836-1846 (2012).
- Espuny-Camacho, I., et al. Pyramidal neurons derived from human pluripotent stem cells integrate efficiently into mouse brain circuits in vivo. Neuron. 77 (3), 440-456 (2013).
- Maroof, A. M., et al. Directed differentiation and functional maturation of cortical interneurons from human embryonic stem cells. Cell Stem Cell. 12 (5), 559-572 (2013).
- Johnson, M. A., Weick, J. P., Pearce, R. A., Zhang, S. C. Functional neural development from human embryonic stem cells: accelerated synaptic activity via astrocyte coculture. J Neurosci. 27 (12), 3069-3077 (2007).
- Hu, B. Y., et al. Neural differentiation of human induced pluripotent stem cells follows developmental principles but with variable potency. Proc Natl Acad Sci USA. 107 (9), 4335-4340 (2010).
- Kim, H., et al. miR-371-3 expression predicts neural differentiation propensity in human pluripotent stem cells. Cell Stem Cell. 8 (6), 695-706 (2011).
- Zhang, Y., et al. Rapid single-step induction of functional neurons from human pluripotent stem cells. Neuron. 78 (5), 785-798 (2013).
- Odawara, A., Saitoh, Y., Alhebshi, A. H., Gotoh, M., Suzuki, I. Long-term electrophysiological activity and pharmacological response of a human induced pluripotent stem cell-derived neuron and astrocyte co-culture. Biochem Biophys Res Commun. 443 (4), 1176-1181 (2014).
- Odawara, A., Katoh, H., Matsuda, N., Suzuki, I. Physiological maturation and drug responses of human induced pluripotent stem cell-derived cortical neuronal networks in long-term culture. Sci Rep. 6, 26181 (2016).
- Amin, H., Maccione, A., Marinaro, F., Zordan, S., Nieus, T., Berdondini, L. Electrical Responses and Spontaneous Activity of Human iPS-Derived Neuronal Networks Characterized for 3-month Culture with 4096-Electrode Arrays. Front Neurosci. 10, (2016).
- Heikkila, T. J., et al. Human embryonic stem cell-derived neuronal cells form spontaneously active neuronal networks in vitro. Exp Neurol. 218 (1), 109-116 (2009).
- Massobrio, P., Massobrio, G., Martinoia, S. Multi-program approach for simulating recorded extracellular signals generated by neurons coupled to microelectrode arrays. Neurocomputing. 70, 2467-2476 (2007).
- Wang, L., Riss, M., Buitrago, J. O., Claverol-Tinturé, E. Biophysics of microchannel-enabled neuron-electrode interfaces. J Neural Eng. 9 (2), (2012).
- McCarthy, K. D., de Vellis, J. Preparation of separate astroglial and oligodendroglial cell cultures from rat cerebral tissue. J Cell Biol. 85, 890-902 (1980).
- Schildge, S., Bohrer, C., Beck, K., Schachtrup, C. Isolation and culture of mouse cortical astrocytes. J. Vis. Exp. (71), e50079 (2013).
- Pacifici, M., Peruzzi, F. Isolation and culture of rat embryonic neural cells: a quick protocol. J Vis Exp. (63), e3965 (2012).
- Ba, W., et al. ARHGAP12 functions as a developmental brake on excitatory synapse function. Cell Rep. 14 (6), 1355-1368 (2016).
- Bologna, L. L., et al. Investigating neuronal activity by SPYCODE multi-channel data analyzer. Neural Netw. 23 (6), 685-697 (2010).
- Ba, W., et al. TRIO loss of function is associated with mild intellectual disability and affects dendritic branching and synapse function. Hum Mol Genet. 25 (5), 892-902 (2016).
- Benevento, M., et al. Histone methylation by the Kleefstra syndrome protein EHMT1 mediates homeostatic synaptic scaling. Neuron. 91 (2), 341-355 (2016).
- Krencik, R., Weick, J. P., Liu, Y., Zhang, Z. -. J., Zhang, S. -. C. Specification of transplantable astroglial subtypes from human pluripotent stem cells. Nature biotechnology. 29 (6), 528-534 (2011).
- Frega, M., Tedesco, M., Massobrio, P., Pesce, M., Martinoia, S. Network dynamics of 3D engineered neuronal cultures: a new experimental model for in-vitro electrophysiology. Sci Rep. 4, 5489 (2014).
- Tedesco, M., Frega, M., Martinoia, S., Pesce, M., Massobrio, P. Interfacing 3D engineered neuronal cultures to micro-electrode arrays: an innovative in vitro experimental model. J Vis Exp. (104), e53080 (2015).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon