Method Article
Rapid neuronale differenziazione delle cellule staminali pluripotenti indotte per misurare l'attività di rete su array Micro-elettrodo
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Modifichiamo e implementare un protocollo precedentemente pubblicato che descrive la differenziazione rapida, riproducibile ed efficiente di indotto umani cellule staminali pluripotenti (hiPSCs) in eccitatorio neuroni corticali 12. In particolare, la nostra modifica permette il controllo della densità delle cellule neuronali e uso su array micro-elettrodi per misurare le proprietà elettrofisiologiche a livello di rete.
Abstract
I neuroni derivati da cellule staminali pluripotenti indotte umane (hiPSCs) forniscono un nuovo strumento promettente per lo studio disturbi neurologici. Negli ultimi dieci anni, sono stati sviluppati molti protocolli per differenziare hiPSCs in neuroni. Tuttavia, questi protocolli sono spesso lenti con elevata variabilità, bassa riproducibilità, e bassa efficienza. Inoltre, i neuroni ottenuti con questi protocolli sono spesso immature e mancano di adeguata attività funzionale a livello sia unicellulari e di rete a meno che i neuroni sono coltivati per parecchi mesi. In parte a causa di queste limitazioni, le proprietà funzionali delle reti neuronali hiPSC-derivati non sono ancora ben caratterizzati. Qui, adattiamo un protocollo recentemente pubblicato che descrive la produzione di neuroni umani da hiPSCs dall'espressione forzata del fattore di trascrizione neurogenina-2 12. Questo protocollo è rapido (dando neuroni maturi entro 3 settimane) e efficiente, con efficienza di conversione quasi il 100% di transduccellule ED (> 95% delle cellule DAPI-positive sono MAP2 positivo). Inoltre, il protocollo produce una popolazione omogenea di neuroni eccitatori che permetterebbero l'indagine di cellule di tipo contributi specifici disturbi neurologici. Abbiamo modificato il protocollo originale generando cellule hiPSC stabilmente trasdotte, dandoci il controllo esplicito rispetto al numero totale di neuroni. Queste cellule vengono poi utilizzati per generare reti neuronali hiPSC-derivati su array di micro-elettrodi. In questo modo, l'attività elettrofisiologica spontanea delle reti neuronali hiPSC-derivati può essere misurata e caratterizzata, pur mantenendo la coerenza interexperimental in termini di densità cellulare. Il protocollo presentato è ampiamente applicabile, in particolare per gli studi meccanicistici e farmacologici sulle reti neuronali umane.
Introduzione
Lo sviluppo di indotto umani cellule staminali pluripotenti (hiPSCs) protocolli di differenziazione per generare neuroni umani in vitro ha fornito un nuovo e potente strumento per lo studio delle malattie neurologiche. Fino a poco tempo, lo studio di questi disturbi è stato gravemente ostacolato dalla mancanza di sistemi modello utilizzando neuroni umani. Sebbene roditori possono essere utilizzati per studiare i disturbi neurologici, i risultati di tali studi non possono essere tradotti facilmente agli esseri umani 1. Date queste limitazioni, neuroni hiPSC derivati sono un modello alternativo promettente che può essere utilizzato per chiarire i meccanismi molecolari disturbi neurologici sottostanti e in vitro screening di farmaci.
Negli ultimi dieci anni, diversi protocolli per convertire hiPSCs in neuroni sono stati sviluppati 2-8. Tuttavia, questi protocolli sono ancora limitate in molti modi. In primo luogo, i protocolli sono spesso in termini di tempo: la generazione di neuroni con un'adeguata maturazione (es synapformazione SE) e l'attività funzionale richiede mesi di procedure di coltura, che rende gli studi su larga scala difficili 9. Inoltre, hiPSC-to-neurone efficienza di conversione è bassa. Protocolli spesso producono una popolazione eterogenea di neuroni, e quindi non consentono di studi specifici sottoinsiemi di cellule neuronali. Inoltre, i protocolli non sono riproducibili, ottenendo risultati diversi per le diverse linee IPSC 10,11. Infine, la fase di maturazione e proprietà funzionali dei neuroni risultanti sono variabili 10.
Per affrontare questi problemi, Zhang et al. (2013) 12 ha sviluppato un protocollo che rapidamente e riproducibile genera neuroni umani da hiPSCs dalla iperespressione del fattore di trascrizione neurogenina-2. Come riportato dagli autori, la differenziazione si verifica in tempi relativamente brevi (solo 2 - 3 settimane dopo l'induzione dell'espressione di neurogenina-2), il protocollo è riproducibile (le proprietà neuronali sono indipendenti dalle starting linea hiPSC), e la conversione hiPSC-to-neurone è molto efficace (quasi il 100%). La popolazione di neuroni generati con il loro protocollo è omogeneo (simile livello superiore neuroni corticali), che consente l'indagine di cellule di tipo contributi specifici per i disturbi neuronali. Inoltre, i loro neuroni hiPSC-derivati hanno mostrato proprietà maturi (ad esempio, la capacità di formare sinapsi e robusta attività funzionale) dopo solo 20 d.
Che caratterizzano le proprietà elettrofisiologiche di neuroni hiPSC derivati a livello di rete è un requisito importante prima tecnologia hiPSC può essere sfruttata per lo studio di malattie umane. Per questo motivo, molti gruppi di ricerca hanno recentemente iniziato a studiare i neuroni cellule staminali derivate a livello di rete che utilizza la matrice di micro-elettrodi (MEA) dispositivi (multicanale Systems, Reutlingen, Germania) 13-16. Gli elettrodi di un MEA sono incorporati in un substrato su cui le cellule neuronali possono essere coltivate.MEA possono essere usati per esplorare le proprietà elettrofisiologiche delle reti neuronali e lo sviluppo in vitro della loro attività. Attualmente, MEA vengono utilizzati solo in combinazione con i protocolli di differenziazione che richiedono diversi mesi per produrre reti maturi. Quindi, combinando MEA con un protocollo di differenziazione rapida dovrebbe facilitare l'uso di questa tecnologia in studi su larga scala di disturbi neurologici.
Qui, presentiamo una modifica del Zhang et al. (2013) 12 del protocollo e adattarlo per l'uso su MEA. In particolare, piuttosto che basarsi su una trasduzione lentivirale acuta, abbiamo invece creato linee hiPSC esprimono stabilmente rtTA / Ngn2 prima di indurre la differenziazione. Abbiamo fatto questo principalmente per avere il controllo riproducibile sulla densità cellulare neuronale, poiché la densità delle cellule neuronali è critica per la formazione della rete neuronale, e per un buon contatto tra i neuroni e gli elettrodi del MEA 17,18. although Zhang et al. protocollo è molto efficiente rispetto alla conversione del hiPSCs trasdotte, è intrinsecamente variabile rispetto alla resa finale dei neuroni dal numero di hiPSCs placcati inizialmente (vedere Figura 2E in Zhang et al.) 12. Con una linea stabile, si eliminano molti problemi che causano variabilità, quali la tossicità lentivirali e l'efficienza di infezione. Abbiamo quindi ottimizzato i parametri che producono in modo affidabile le reti neuronali hiPSC-derivati su MEA, ottenendo la maturazione di rete (ad esempio, gli eventi sincroni che coinvolgono la maggioranza dei canali) per la terza settimana. Questo protocollo rapido ed affidabile dovrebbe consentire un confronto diretto tra i neuroni derivati da diverse linee hiPSC (cioè paziente-specifici), nonché garantire la coerenza robusto per studi farmacologici.
Protocollo
Tutti gli esperimenti sugli animali sono stati eseguiti in conformità con le linee guida per la cura degli animali e nell'uso approvato Comitato Animal Care, Radboud University Medical Center, in Olanda, (RU-DEC-2011-021, numero di protocollo: 77073).
1. Glia cella di isolamento e cultura
NOTA: Il protocollo presentato qui è basato sul lavoro di McCarthy e de Vellis 19, e una molto simile protocollo dettagliato per gli astrociti del mouse è disponibile 20. Per generare colture primarie di astrociti corticali da embrionali cervello di ratto (E18), un topo in stato di gravidanza deve essere sacrificato, gli embrioni devono essere raccolti dall'utero, e il cervello devono essere isolati dagli embrioni. Per riempire un pallone T75, le cortecce da 2 cervelli embrionali devono essere combinati. In alternativa, sono disponibili commercialmente astrociti purificati e congelati possono essere acquistati.
- Preparare il pallone di coltura T75
- Diluire Poly-D-lisina (PDL) insterile, ultrapura acqua ad una concentrazione finale di 10 mg / mL. Aggiungere 5 ml del Pdl diluito al pallone di coltura T75. Swish intorno delicatamente per bagnare tutta la superficie di crescita. Porre il pallone in un umidificata 37 ° C incubatore per 3 ore.
- Aspirare il PDL dal pallone. Sciacquare i 3x pallone con 5 ml di acqua sterile per rimuovere PDL non legato. Aspirare completamente l'acqua. Lasciare la muffola asciugare in una cappa a flusso laminare o utilizzato immediatamente.
- La dissezione delle cortecce
- Preparare 50 ml di media dissezione: L-15 di media di Lebovitz con il 2% (v / v) B-27 supplemento. Tenere in ghiaccio.
- Anestetizzare il topo profondamente con isoflurano in una camera di induzione (scatola di piccole dimensioni plexiglas) fino a quando la respirazione cessa (~ 5 - 8 min). Rimuovere ratto dalla camera di induzione e subito eutanasia mediante dislocazione cervicale.
- Spruzzare l'addome del topo con il 70% EtOH e spazzare via l'eccesso. Esporre e rimuovere l'utero dalla diga via secti cesareosull'utilizzo di un paio di forbici 21.
- Tagliare i singoli embrioni da loro sacche amniotiche con le forbici, trasferimento in una capsula di Petri sterile riempito di media dissezione a freddo, e tenere in ghiaccio.
- Trasferire gli embrioni di nuovo per un nuovo, sterile, sei centimetri Petri piatto pieno di media dissezione a freddo. Estrarre cervelli di embrioni in un microscopio stereo. Per esporre il cervello, delicatamente togliete la pelle e il cranio con pinze. Delicatamente scavare tutto il cervello e il trasferimento di 35 mm piastra di Petri con fresco, di media dissezione a freddo.
NOTA: cervelli interi sezionato da embrioni possono essere memorizzati in medium dissezione in ghiaccio per molte ore senza perdere la vitalità cellulare. - Separare i due emisferi del cervello ogni tagliando attraverso la linea mediana con le forbici a molla a punta fine o un bisturi. striscia con cautela le meningi con pinze diritte a punta fine.
NOTA: E 'molto importante per rimuovere completamente le meningi. thè la contaminazione dei fibroblasti impedisce della cultura astrociti. I fibroblasti sono cellule in rapida divisione e tendono a spostare le altre cellule. - Rimuovere il mesencefalo / striato e del bulbo olfattivo con le forbici a molla o un bisturi. Inoltre, assicurarsi di rimuovere l'ippocampo (struttura a forma di C che è perimedian e caudali rispetto alla corteccia) con le forbici a molla o un bisturi. Raccogliere gli emisferi corticali in un tubo da 15 ml centrifuga piena di 5 ml di media dissezione. Tenere in ghiaccio.
- La dissociazione delle cortecce
- Preparare 2 ml di Ca 2+ / Mg 2+-free soluzione salina bilanciata di Hank (HBSS) con 0,25% tripsina (medie di dissociazione). Preparare 50 mL di alta glucosio Dulbecco Modified mezzo di Eagle (DMEM) con il 15% (v / v) siero fetale bovino (FBS) e 1% (v / v) di penicillina / streptomicina (media cultura) e filtro sterilizzare.
- Sia il tessuto si depositano sul fondo della provetta. attentamentepirata tanto del mezzo dissezione possibile dall'alto tessuto. Lavare il tessuto con 5 mL Ca 2+ / Mg 2+ -free HBSS (senza tripsina) e lasciare il tessuto di stabilirsi sul fondo della provetta.
- aspirare accuratamente la HBSS. Aggiungere 2 ml di media dissociazione e flick delicatamente il tubo per miscelare l'enzima in tutto il tessuto. Incubare a bagnomaria a 37 ° C per 5-10 min. Flick il tubo di un paio di volte durante l'incubazione per agitare il tessuto.
- Immediatamente triturare il tessuto con un puntale di 1.000 ml. Impostare il volume pipetter a circa 800 ml. Aspirare i pezzi ed espellere con forza sul lato del tubo, direttamente sopra la linea del fluido. Tuttavia, cercare di ridurre al minimo la formazione di schiuma o bolle. Ripetere fino a quando il tessuto è sufficientemente dissociato, circa 15 - 20x. Aggiungere 8 ml terreno di coltura per inattivare la tripsina. Mescolare delicatamente invertendo la provetta diverse volte.
- Passare la sospensione cellulare attraverso un filtro cella 70 micron posta sopra un 50 mL provetta da centrifuga. Risciacquare il tubo 15 ml con terreno di coltura e filtrare il mezzo attraverso il filtro cella per raccogliere il fluido nel tubo 50 ml con la sospensione cellulare. Risciacquare il filtro cella un paio di volte con terreno di coltura. Dopo il risciacquo, il volume finale deve essere di circa 20 - 25 mL.
- Agglomerare le cellule a 200 xg per 10 min. attenzione aspirare quanto mezzo possibile, senza toccare il pellet di cellule. Risospendere le cellule in 1 ml di terreno di coltura utilizzando una pipetta microlitri 1000. Aggiungere 11 ml di terreno di coltura preriscaldata e mescolare delicatamente (per evitare bolle) con una pipetta 10 ml.
- Lavare il pallone T75 PDL rivestite una volta con 5 ml di terreno di coltura. Aspirare il mezzo e trasferire la sospensione cellulare al pallone. Tutte le cellule sono in sospensione sono placcati, e lo troviamo generalmente inutile di contarli, dal momento che gli astrociti non possono essere differenziati da altre cellule in sospensione. Porre il pallone in un umidificata 37 ° C incubatore con atmosfera al 5% di CO 2 fo due giorni.
- Espansione e manutenzione degli astrociti
- Sostituire l'intero mezzo per la prima volta 2 d dopo la placcatura iniziale. Sostituire l'intero medio successivamente ogni 3 d. preriscaldare sempre il mezzo fresco a 37 ° C prima di aggiungere alle cellule.
NOTA: Gli astrociti richiedono 7-10 d per raggiungere circa il 90% di confluenza (gli astrociti appaiono come un monostrato mosaico densamente, con microglia e oligodendrociti sdraiato sulla parte superiore e mescolati). - Quando gli astrociti raggiungono circa il 90% di confluenza, agitare il pallone per rimuovere le cellule gliali contaminanti:
- Togliere il pallone dal termostato e stringere il tappo (fenolico) o coprire la porta (filtrata). Per rimuovere microglia, scuotere il pallone su una piattaforma orbitale a 180 giri al minuto per 1 ora. Aspirare il mezzo. Risciacquare una volta con 5 ml di pre-riscaldato terreno di coltura, aspirare e sostituirlo con 12 ml di terreno di coltura.
- Rimuovereoligodendrociti, restituiscono il pallone piattaforma orbitale e agitare a 250 rpm, 37 ° C per un minimo di 7 h, preferibilmente O / N.
- Aspirare il mezzo. Risciacquare una volta con 5 ml di pre-riscaldato terreno di coltura, aspirare e sostituirlo con 12 ml di terreno di coltura. Riportare il pallone per l'incubatore.
- Quando 100% confluenti, suddiviso astrociti utilizzando procedure standard con un rapporto di 1: 3 a 1: 2 con acido etilendiamminotetraacetico tripsina 0,05% (EDTA). Un pallone T75 al 100% di confluenza in genere produrranno circa 4.0 x 10 6 cellule in totale. Nell'ambito di questo programma, le culture in genere può essere suddiviso una volta alla settimana.
NOTA: Quando gli astrociti raggiungono la confluenza, possono essere raccolti e utilizzati per la differenziazione hiPSC come descritto di seguito nel passaggio protocollo 3.4. Gli astrociti possono essere divisi almeno una volta senza una notevole perdita di vitalità. Essi possono essere mantenuti fino a 2 mesi in coltura. Per esperienza, primaria giorno embrionale 18 astrociti di ratto progressively diventare terminalmente differenziate e / o perdere la vitalità dopo ripetuti divisione. Anche se è possibile congelare astrociti per uso futuro, preferiamo isolare i astrociti da cervelli freschi embrionali quando richiesto.
- Sostituire l'intero mezzo per la prima volta 2 d dopo la placcatura iniziale. Sostituire l'intero medio successivamente ogni 3 d. preriscaldare sempre il mezzo fresco a 37 ° C prima di aggiungere alle cellule.
2. Generazione di rtTA / Ngn2 -positivo hiPSCs
NOTA: Le hiPSCs utilizzati per i nostri esperimenti sono stati generati in-house da lentivirale trasduzione di fibroblasti umani con la riprogrammazione fattori cMyc, SOX2, OCT4 e Klf4.
NOTA: Per la generazione di rtTA / NGN2 hiPSCs -positive, vettori lentivirali sono utilizzati per integrare stabilmente i transgeni nel genoma dei hiPSCs. Il protocollo per la produzione del lentivirus è stato pubblicato in precedenza 22. I dettagli dei vettori lentivirali di confezionamento che vengono utilizzati per produrre le particelle lentivirali rtTA e NGN2 sono forniti nel Tabella dei materiali / attrezzature. Il vettore di trasferimento utilizzato per il lentivirus rtTA è pLVX-EF1α- (Tet-On-Advanced) -IRES-G418 (R); Per esempio questo vettore codifica una Tet-On transattivatore avanzata sotto il controllo di un promotore costitutivo EF1α e conferisce resistenza al G418 antibiotico. Il vettore di trasferimento utilizzato per il lentivirus Ngn2 è pLVX- (TRE-thight) - (MOUSE) Ngn2-PGK-puromicina (R); Per esempio questo vettore codifica il gene per murine neurogenina-2 sotto il controllo di un promotore Tet-controllata e il gene di resistenza puromicina sotto il controllo di un promotore PGK costitutiva. Quindi, utilizzando questi due vettori di trasferimento, può essere creata una linea hiPSC per cui l'espressione di murine neurogenina-2 possono essere indotte completando il mezzo con doxiciclina. Per la trasduzione dei hiPSCs, viene utilizzato il surnatante con le particelle lentivirali (denominato 'sospensione lentivirus' nel resto del testo), ossia ingegnoHout concentrare le particelle mediante ultracentrifugazione.
- Piastra le hiPSCs (giorno 1)
NOTA: I volumi che sono menzionati in questo protocollo si presuppone che i hiPSCs sono coltivate in una piastra 6 bene e che le cellule di un bene vengono raccolte. Inoltre, si assume che le cellule sono placcati successivamente in 12 pozzetti di una piastra ben 12.- Preparare 10 ml DMEM freddo / F12 con 1% (v / v) matrice di membrana basale (BMM) per ottenere diluito BMM. Aggiungere 800 ml diluiti BMM per pozzetto di una piastra ben 12. Incubare per almeno 1 h in un umidificata 37 ° C incubatore con atmosfera al 5% di CO 2. Prima dell'uso, incubare la piastra per 1 ora a RT.
- Essential 8 (E8) medium caldo 15 mL con 1% (v / v) di penicillina / streptomicina, 9 ml DMEM / F12 e 1 soluzione distacco cellulare mL (CDS) a temperatura ambiente. Supplemento medio E8 con Rho-associata proteina chinasi (ROCK) inibitore.
- Aspirare il medium speso dei hiPSCs und aggiungere 1 CDS mL ai hiPSCs. Incubare 3 - 5 min in un umidificata 37 ° C incubatore con atmosfera al 5% di CO 2. Controllare al microscopio se le cellule sono staccano l'uno dall'altro.
- Aggiungere 2 ml DMEM / F12 nel pozzo, sospendere delicatamente le cellule con una pipetta microlitri 1.000 e trasferire le cellule in una provetta da 15 ml. Aggiungere 7 ml DMEM / F12 alla sospensione cellulare. Spin le cellule a 200 xg per 5 min.
- Aspirare il surnatante e aggiungere 2 ml di mezzo E8 preparati. Ottenere una sospensione di cellule in cui sono dissociate le hiPSCs (non formare grumi di cellule) mettendo la punta di una pipetta 1000 microlitri contro il lato del tubo 15 mL e risospendere le cellule delicatamente. Controllare al microscopio se le cellule sono dissociate.
- Determinare il numero di cellule (cellule / ml) utilizzando una camera di emocitometro.
NOTA: Una piastra 6 bene bene a 80 - 90% di confluenza in genere produrrà 3,0 - 4,0 x 10 6 cellule in totale. - Aspirarela diluito BMM dai pozzi del 12 pozzetti. Diluire le cellule per ottenere una sospensione cellulare di 3,0 x 10 4 cellule / ml. Tavola 1 ml di sospensione cellulare per pozzetto della piastra ben 12. Posizionare la piastra ben 12 O / N in un umidificata 37 ° C incubatore con atmosfera al 5% di CO 2.
- Trasdurre le cellule iPS con rtTA e Ngn2 lentivirus (giorno 2)
- media Warm 12 mL E8 con 1% (v / v) di penicillina / streptomicina a temperatura ambiente. Supplemento medio E8 con inibitore ROCK e polibrene ad una concentrazione finale di 8 mg / ml al mezzo E8.
- Scongelare le aliquote con sospensione lentivirus. Aggiungere polibrene ad una concentrazione finale di 8 mg / mL di sospensione lentivirus. Aspirare il medio speso e aggiungere 1 ml di mezzo E8 preparata in ciascun pozzetto.
- Eseguire la trasduzione con diverse quantità di rtTA - e sospensioni NGN2 -lentivirus. Ad esempio, transduCe le hiPSCs aggiungendo 100 ml di entrambe le -lentivirus rtTA e sospensioni -lentivirus Ngn2 ad un pozzetto della piastra di 12 bene. Per gli altri pozzi, usare 200 ml, 300 ml, 400 ml e 500 ml di sospensione lentivirus invece di 100 ml. Le hiPSCs di due pozzetti della piastra 12 e non dovrebbero essere trasdotte; serviranno come controlli durante la selezione.
NOTA: I trasduzione sono preferibilmente eseguite in duplicato, in modo che l'efficienza di trasduzione può stimare più accuratamente dopo l'inizio della selezione (vedere fase protocollo 2.2.4). La quantità di sospensione lentivirus che è necessario per trasdurre efficientemente la maggioranza dei hiPSCs dipende dal titolo della sospensione lentivirus e la linea hiPSC utilizzato. In questo studio, usiamo di solito 100 - 500 ml di sospensione lentivirus di trasdurre i hiPSCs. - Posizionare la piastra ben 12 in un umidificata 37 ° C incubatore con atmosfera al 5% di CO2 per 6 h. Prima della fine del periodo di incubazione 6 h, caldo media 12 mL E8 con 1% (v / v) di penicillina / streptomicina e 12 di mL Dulbecco Phosphate-Buffered Saline (DPBS) per RT. Supplemento medio E8 con inibitore ROCK.
- Aspirare il mezzo di E8 speso. Lavare ogni pozzetto con 1 ml DPBS. Aggiungere 1 ml del mezzo E8 preparata in ciascun pozzetto. Posizionare la piastra ben 12 O / N in un umidificata 37 ° C incubatore con atmosfera al 5% di CO 2.
- Aggiornare la media E8 (giorno 3)
- media Warm 12 mL E8 con 1% (v / v) di penicillina / streptomicina a RT. Aspirare il medio speso dai pozzi del 12 piastra bene e aggiungere 1 ml di mezzo E8 pronto a ciascun pozzetto. Posizionare la piastra ben 12 O / N in un umidificata 37 ° C incubatore con atmosfera al 5% di CO 2.
- Eseguire la selezione con puromicina e G418 (giorno 4-8)
NOTA: A seconda del tasso di divisione cellulare dell'ancaLinea SC e l'efficienza della trasduzione lentivirale, le cellule possono raggiungere il 70 - 80% di confluenza durante il periodo di selezione, a quel punto le culture devono essere raggruppati. Poiché i tempi del frazionamento non può essere previsto in anticipo, non sarà menzionato nel protocollo. Tuttavia, invece di rinfrescare il mezzo E8 integrato con le concentrazioni indicate di puromicina e G418, si può dividere la cultura hiPSC come cultura hiPSC normale (comprese placcatura le cellule su piastre vitronectina rivestite). L'unica eccezione è che il mezzo E8 dovrebbe essere integrato con le concentrazioni menzionate degli antibiotici per continuare la selezione.- media Warm 12 mL E8 con 1% (v / v) di penicillina / streptomicina a temperatura ambiente. Aggiungere puromicina e G418 per la selezione; diverse quantità di antibiotici sono aggiunti durante il periodo di selezione (Tabella 1).
- Stimare l'efficienza della trasduzione stimando la percentuale di G418- e puromicinacellule resistenti all'azione. Per stimare la percentuale di cellule resistenti, stimare la percentuale di cellule morte (cellule nonresistant) per le diverse condizioni (le culture trasdotte con diverse quantità di sospensione lentivirus) e per le celle nontransduced (le cellule che servono come controllo di selezione). Calcolare la percentuale di cellule resistenti come [100% - (percentuale di cellule morte)].
NOTA: Se le trasduzioni con diverse quantità di sospensione lentivirus sono stati eseguiti in duplicato, l'efficienza di trasduzione può stimare più accuratamente. La condizione con le cellule non trasdotte serve come un controllo di selezione; le percentuali di cellule morte per le colture trasdotte con diverse quantità di sospensione lentivirus dovrebbe essere inferiore. La percentuale stimata di cellule resistenti è utilizzato per scegliere i hiPSCs che possono positivi per entrambe le transgeni. In generale, abbiamo scelto le hiPSCs dalla condizione trasduzione erano> 90% della cellas sopravvivere il periodo di selezione 5 d. - Aspirare il medium speso dei hiPSCs e aggiungere 1 ml di mezzo E8 pronto a pozzetti. Posizionare la piastra ben 12 O / N in un umidificata 37 ° C incubatore con atmosfera al 5% di CO 2.
| Concentrazione finale di G418 | Concentrazione finale di puromicina | |
| 4 ° giorno | 250 mg / ml | 2 mg / mL |
| giorno 5 | 250 mg / ml | 2 mg / mL |
| giorno 6 | 250 mg / ml | 1 mg / mL |
| giorno 7 | 250 mg / ml | 1 mg / mL |
| giorno 8 | 250 mg / ml | 1 mg / mL |
Tabella 1: Le concentrazioni di antibiotici durante il Periodo di selezione. Concentrazioni della puromicina e G418 durante la 5 D del periodo di selezione.
- Arrestare la selezione e avviare la coltura regolare (giorno 9 e versioni successive)
- Dopo il 5 d periodo di selezione, cultura i hiPSCs -positive rtTA / NGN2 come hiPSCs normali, con l'eccezione che il mezzo E8 delle cellule è completato con G418 ad una concentrazione finale di 50 mg / ml e con puromicina ad una concentrazione finale di 0,5 mg / mL.
NOTA: Le cellule possono essere congelati (secondo protocolli standard per la crioconservazione di cellule) per servire come backup. Questo è un passo importante per la riproducibilità del protocollo differenziazione, perché permette l'uso dello stesso lotto di rtTA / NGN2 hiPSCs -positive per vari esperimenti differenziazione futuri.
- Dopo il 5 d periodo di selezione, cultura i hiPSCs -positive rtTA / NGN2 come hiPSCs normali, con l'eccezione che il mezzo E8 delle cellule è completato con G418 ad una concentrazione finale di 50 mg / ml e con puromicina ad una concentrazione finale di 0,5 mg / mL.
3. Differenziazione delle rtTA / NGN2 hiPSCs -positive ai neuroni, il 6-beneMEA e vetrini di vetro
NOTA: In questo protocollo, i dettagli sono forniti per differenziare rtTA / NGN2 hiPSCs -positive su due substrati diversi, vale a dire MEA 6 pozzetti (dispositivi composti da 6 pozzi indipendenti con 9 registrazione e 1 di riferimento microelettrodi integrati per bene) e vetrini in i pozzetti di una piastra da 24 pozzetti. I protocolli, tuttavia, possono essere facilmente adattati per substrati grandi (ad esempio, per i pozzetti di piastre da 12 o 6 pozzetti), scalando i valori indicati in base alla superficie.
- Preparare i MEA o vetrini (giorno 0 e 1 giorno)
- Il giorno prima dell'inizio della differenziazione, sterilizzare i MEA in base alle raccomandazioni del produttore.
- Diluire la proteina di adesione Poly-L-ornitina (PLO) in acqua ultrapura sterile ad una concentrazione finale di 50 mg / mL. Rivestire la zona elettrodo attivo di 6 pozzetti MEA mettendo un 100 microlitririlasciare dell'OLP diluita in ogni pozzetto. Posizionare i vetrini nei pozzetti della piastra da 24 pozzetti con una pinzetta sterile. Aggiungere 800 ml di dell'OLP diluita in ogni pozzetto. Evitare che i coprioggetti da tasso variabile, spingendoli verso il basso con la punta della pipetta 1000 ml.
- Incubare le MEA 6 pozzetti e 24 pozzetti O / N in un umidificata 37 ° C incubatore con atmosfera al 5% di CO 2. Il giorno successivo, aspirare l'OLP diluito. Lavare le superfici vetrate delle MEA 6 pozzetti e i coprioggetti due volte con acqua ultrapura sterile.
- Diluire laminina in freddo DMEM / F12 ad una concentrazione finale di 20 mg / ml (per i MEA 6 pozzetti) e 10 mg / ml (per i vetrini). Subito rivestire la superficie dell'elettrodo attivo dei MEA 6 pozzetti mettendo una goccia 100 microlitri in ciascun pozzetto. Allo stesso modo, aggiungere 400 microlitri della laminina diluita in ciascun pozzetto della piastra 24 pozzetti per rivestire i coprioggetti. Evitare che i coprioggetti da tasso variabile, spingendoli verso il basso con la punta della pipetta 1000 microlitri.
- Incubare le MEA 6 pozzetti e 24 pozzetti in un umidificata 37 ° C incubatore con atmosfera al 5% di CO 2 per almeno 2 h.
- Piastra le hiPSCs (giorno 1)
NOTA: I volumi che sono menzionati nei passaggi 3.2.1 - 3.2.4 per scontato che i hiPSCs -positive rtTA / NGN2 sono coltivate in un 6-bene e che le cellule di un bene vengono raccolte. I volumi che sono necessari per la placcatura cellule sulle MEA 6 pozzetti e / o i coprioggetti dipende dal numero di 6 pozzetti MEA e / o il numero di coprioggetto utilizzati nell'esperimento; i numeri specificati nei passaggi 3.2.6 - 3.2.8 consentono il ridimensionamento di diverse dimensioni esperimento.- DMEM caldi / F12, CDS e medie E8 con 1% (v / v) di penicillina / streptomicina per R / T. Aggiungere doxiciclina a una concentrazione finale di 4 mg / ml e inibitore ROCK al mezzo E8.
- Aspirare il medium speso dei hiPSCs -positive rtTA / NGN2 e aggiungere 1 mL CDS ai hiPSCs. Incubare 3 - 5 min in un umidificata 37 ° C incubatore con atmosfera al 5% di CO 2. Controllare al microscopio se le cellule sono staccano l'uno dall'altro.
- Aggiungere 2 ml DMEM / F12 nel pozzo, sospendere delicatamente le cellule con una pipetta microlitri 1.000 e trasferire le cellule in una provetta da 15 ml. Aggiungere 7 ml DMEM / F12 alla sospensione cellulare. Spin le cellule a 200 xg per 5 min.
- Aspirare il surnatante e aggiungere 2 ml di mezzo E8 preparati. Dissociare il hiPSCs mettendo la punta di una pipetta 1000 microlitri contro il lato del tubo 15 mL e risospendere le cellule delicatamente. Controllare al microscopio se le cellule sono dissociate.
- Determinare il numero di cellule (cellule / ml) utilizzando una camera di emocitometro.
NOTA: A 6 pozzetti bene a 80-90% di confluenza in genere produrrà 3,0 - 4,0 x 10 6 cellule in totale. - Aspirare il laminina diluito. Per i MEA 6 pozzetti, diluire le cellule per ottenere un cell sospensione di 7,5 x 10 5 cellule / ml. Piastra le cellule aggiungendo una goccia di 100 microlitri della sospensione cellulare sull'area elettrodo attivo in ciascun pozzetto di MEA 6 pozzetti. Per i coprioggetti, diluire le cellule per ottenere una sospensione cellulare di 4,0 x 10 4 cellule / ml. Piastra le cellule aggiungendo 500 ml di sospensione cellulare nei pozzetti della piastra da 24 pozzetti.
NOTA: La densità cellulare finale sulle MEA è maggiore rispetto coprioggetto (Figura 1A e B). Abbiamo scoperto che questo ad alta densità cellulare è stato richiesto per una corretta registrazione dell'attività di rete. Nel protocollo, i numeri sono a condizione che si è rivelata ottimale per i saggi. - Posizionare i MEA 6 pozzetti e 24 pozzetti in un umidificata 37 ° C incubatore con atmosfera al 5% di CO 2 per 2 ore (MEA) o O / N (24 pozzetti).
- Dopo 2 h, aggiungere con cautela 500 ml di mezzo E8 preparati a ciascun pozzetto di MEA 6 pozzetti. Posizionare il 6-noill MEA O / N in un umidificata 37 ° C incubatore con atmosfera al 5% di CO 2.
- Cambiare il medio (giorno 2)
- Il giorno successivo, preparare DMEM / F12 con 1% (v / v) N-2 integratore, 1% (v / v) gli amminoacidi non essenziali e 1% (v / v) di penicillina / streptomicina. Aggiungere umana ricombinante neurotrofina-3 (NT-3) ad una concentrazione finale di 10 ng / mL, fattore di derivazione cerebrale umana ricombinante neurotrofico (BDNF) ad una concentrazione finale di 10 ng / mL, e doxiciclina ad una concentrazione finale di 4 mg / mL. Riscaldare il supporto a 37 ° C.
- Aggiungere laminina ad una concentrazione finale di 0,2 mg / mL al mezzo. Filtrare la media risultante. Aspirare il mezzo trascorso dai pozzi dei MEA 6 pozzetti e la piastra 24 pozzetti e sostituirlo con il mezzo preparato. Incubare le MEA 6 pozzetti e 24 pozzetti O / N in un umidificata 37 ° C incubatore con atmosfera al 5% di CO 2.
- Aggiungere astrociti di ratto (giorno 3)
Nota: I volumi che sono menzionati in questo protocollo si presuppone che i astrociti di ratto sono coltivate in fiasche di coltura T75. E 'fondamentale che gli astrociti di ratto che vengono aggiunti alle culture sono di buona qualità. Usiamo due criteri per verificare se gli astrociti di ratto sono di buona qualità. In primo luogo, la cultura astrociti di ratto dovrebbe essere in grado di crescere confluenti entro dieci giorni dopo l'isolamento dal ratto cervelli embrionali. In secondo luogo, dopo aver diviso la cultura astrociti di ratto, astrociti di ratto dovrebbero essere in grado di formare un confluenti monostrato tessellated (Figura 1C). Se la cultura del ratto astrociti non soddisfa questi due criteri, si consiglia di non utilizzare questa cultura per gli esperimenti di differenziazione.- Warm 0,05% tripsina-EDTA a RT. Riscaldare la DPBS e DMEM / F12 con 1% (v / v) di penicillina / streptomicina a 37 ° C.
- Aspirare il medium speso della cultura topo astrociti. Lavare la cultura con l'aggiunta di 5 ml di DPBS e fruscio intorno delicatamente.
- Aspirmangiato il DPBS e aggiungere 5 ml 0,05% tripsina-EDTA. Swish la tripsina-EDTA intorno delicatamente. Incubare in un umidificata 37 ° C incubatore con atmosfera al 5% di CO 2 per 5 - 10 min.
- Controllare al microscopio se le cellule sono staccati. Staccare le ultime cellule colpendo il pallone un paio di volte.
- Aggiungere 5 ml di DMEM / F12 al pallone. Triturare le cellule delicatamente dentro il pallone con una pipetta 10 ml. Raccogliere sospensione di cellule in una provetta da 15 ml. Spin la provetta a 200 xg per 8 minuti.
- Aspirare il surnatante e risospendere le cellule in 1 ml di DMEM / F12. Determinare il numero di cellule (cellule / ml) utilizzando una camera di emocitometro.
- Aggiungere 7,5 x 10 4 astrociti per pozzetto dei MEA 6 pozzetti. Aggiungere 2,0 x 10 4 astrociti per pozzetto della piastra da 24 pozzetti. Incubare le MEA e la piastra 24 pozzetti O / N in un umidificata 37 ° C incubatore con atmosfera al 5% di CO 2.
- Cambiare il medio (giorno 4)
- Preparati medio Neurobasal con 2% (v / v) B-27 supplemento, 1% (v / v) L-alanil-L-glutammina e 1% (v / v) di penicillina / streptomicina. Aggiungere NT-3 ad una concentrazione finale di 10 ng / mL, BDNF ad una concentrazione finale di 10 ng / mL, e doxiciclina ad una concentrazione finale di 4 mg / mL. Inoltre, aggiungere citosina β-D-arabinofuranoside ad una concentrazione di 2 mM.
NOTA: Cytosine β-D-arabinofuranoside viene aggiunto al mezzo di inibire la proliferazione astrociti e uccidere i rimanenti hiPSCs che non sono differenziarsi in neuroni. - Filtrare il mezzo e riscaldare a 37 ° C. Aspirare il mezzo trascorso dai pozzi dei MEA 6 pozzetti e la piastra 24 pozzetti e sostituirlo con il mezzo preparato. Mantenere i MEA 6 pozzetti e la piastra di 24 pozzetti in un umidificata 37 ° C incubatore con atmosfera al 5% di CO 2.
- Preparati medio Neurobasal con 2% (v / v) B-27 supplemento, 1% (v / v) L-alanil-L-glutammina e 1% (v / v) di penicillina / streptomicina. Aggiungere NT-3 ad una concentrazione finale di 10 ng / mL, BDNF ad una concentrazione finale di 10 ng / mL, e doxiciclina ad una concentrazione finale di 4 mg / mL. Inoltre, aggiungere citosina β-D-arabinofuranoside ad una concentrazione di 2 mM.
- Aggiornare la media (giorno 6-28)
NOTA: A partire dal giorno 6, aggiornare la metàdel mezzo ogni due giorni. Dal giorno 10 in poi, il mezzo è completato con FBS per sostenere la vitalità astrociti.- Preparare medio Neurobasal con 2% (v / v) B-27 supplemento, 1% (v / v) L-alanil-L-glutammina e 1% (v / v) di penicillina / streptomicina. Aggiungere NT-3 ad una concentrazione finale di 10 ng / mL, BDNF ad una concentrazione finale di 10 ng / mL, e doxiciclina ad una concentrazione finale di 4 mg / mL. Dal giorno 10 in poi, integrare anche il mezzo con 2,5% (v / v) di FBS. Filtrare la media risultante e calda a 37 ° C.
- Rimuovere la metà del mezzo trascorso dai pozzi dei MEA 6 pozzetti e la piastra a 24 pozzetti utilizzando una pipetta microlitri 1000 e sostituirlo con il mezzo preparato. Mantenere i MEA 6 pozzetti e la piastra di 24 pozzetti in un umidificata 37 ° C incubatore con atmosfera al 5% di CO 2.
4. Stabilire la neurofisiologico Profilo di neuroni hiPSC derivati
NOTA: due o tre settimane dopo l'inproduzione di differenziazione, i neuroni hiPSC derivati possono essere utilizzati per diverse analisi a valle. In questa sezione, esempi di alcune analisi a valle sono dati che può essere eseguita per stabilire il profilo neurofisiologico dei neuroni hiPSC derivati.
- Caratterizzare l'attività di rete neuronale utilizzando MEA
- Record 20 minuti di attività elettrofisiologica dei neuroni hiPSC derivati coltivate su MEA. Durante la registrazione, mantenere la temperatura a 37 ° C, e prevenire evaporazione e pH variazioni del mezzo gonfiando una costante, lento flusso di gas umidificato (5% CO 2, 20% O 2, 75% N 2) sul MEA .
- Dopo 1200X amplificazione (MEA 1060, MCS), campionare il segnale a 10 kHz utilizzando la scheda di acquisizione dati MCS. Analizzare i dati (base a punta e rilevamento scoppio) con un pacchetto software personalizzato 23.
- Caratterizzano l'attività elettrofisiologica cella singola
- Trasferire i vetrini contenenti le colture neuronali hiPSC di derivazione ad una camera di registrazione stadi fisso immerso in un microscopio in posizione verticale. Record 20 min di azione spontanea potenziali evocati correnti post-sinaptiche (sEPSC) 24. Rilevare l'evento sinaptica utilizzando il programma neuroscientifica.
- Caratterizzare la morfologia neuronale e l'espressione sinapsina
- Fissare e macchiare i neuroni hiPSC-derivati per MAP2, synapsin-1/2, e PSD-95 22, 24, 25. Quantificare il numero della sinapsina-1/2 e PSD-95 puncta utilizzando il software di analisi delle immagini.
Risultati
Qui abbiamo modificato con successo un protocollo in cui hiPSCs si differenziano direttamente in neuroni corticali esprimendo over-the fattore di trascrizione neurogenina-2 12 e abbiamo adattato per l'uso di MEA. Questo approccio è veloce ed efficiente che permette di ottenere neuroni funzionali e attività di rete già durante la terza settimana dopo l'induzione della differenziazione.
Nel corso del protocollo differenziazione, le cellule morfologicamente cominciato ad assomigliare neuroni: sono formate piccoli processi e neuroni cominciato collegando tra loro (Figura 1A). Abbiamo stabilito un profilo neurofisiologico dei neuroni derivati da una linea hiPSC sano di controllo, misurando la loro morfologia dei neuroni e delle proprietà sinaptiche durante lo sviluppo. neuroni hiPSC-derivati sono state colorate per MAP2 e synapsin-1/2 a diversi giorni dopo l'iniziodi differenziazione (Figura 2A). I neuroni derivati mostrano matura morfologia neuronale già 3 settimane dopo l'induzione della differenziazione. Il numero di synapsin-1/2 puncta (una misura del numero di sinapsi) è stato quantificato sulla base synapsin-1/2 colorazioni immunocitochimica. Il numero di synapsin-1/2 puncta aumentata nel tempo, suggerendo che il livello di connettività neuronale è in aumento (Figura 2B). Il numero di synapsin-1/2 puncta 23 giorni dopo l'induzione della differenziazione era simile in due linee IPS indipendenti (Figura 2C). Al 23 DIV più synapsin1 / 2 puncta sono stati giustapposti a PSD-95 puncta, che è indicativo di sinapsi funzionali (Figura 2D).
In linea con i risultati descritti da Zhang et al., Abbiamo generato una popolazione di eccitatori di livello superiore neuroni corticali, confermata da pan-neuronale e sottotipo specifico m corticale arkers come BRN2 e SATB2 (strato II / III). Non abbiamo osservato che i neuroni positivi per i neuroni strato profondo CTIP2 (livello V) o Foxp2 (strato VI) (Figura 2E e F)
A caratterizzare l'attività elettrofisiologica dei neuroni hiPSC-derivati, abbiamo usato corrente a cellula intera e le registrazioni morsetto di tensione, vale a dire le proprietà intrinseche e l'input eccitatori su questi neuroni sono stati misurati durante lo sviluppo. I neuroni sono stati in grado di generare potenziali d'azione già una settimana dopo la differenziazione e la percentuale di spiking cellule aumentava nel tempo (figura 2G e H). Inoltre, i neuroni ricevuti eccitatorio ingresso sinaptica già una settimana dopo l'induzione della differenziazione: sia frequenza e ampiezza dell'ingresso sinaptica eccitatoria aumentate durante lo sviluppo (Figura 2I - K).
nt "fo: keep-together.within-page =" 1 "> Per capire meglio come l'attività monocellulari combina per formare le funzioni a livello di rete, è essenziale per studiare come i neuroni funzionano in concerto a vitro reti neuronali coltivate su MEA. costituiscono un modello sperimentale utile per lo studio della dinamica neuronale. Abbiamo registrato 20 minuti di attività di rete elettrofisiologica dei neuroni derivati da una linea hiPSC sano controllo coltivati su 6 pozzetti MEA (Figura 2M). Poche settimane dopo l'induzione della differenziazione, i neuroni derivato da hiPSCs sani di controllo formata reti neuronali funzionalmente attivi, mostrando eventi spontanei (0.62 ± 0.05 picco / s; Figura 2N). in questa fase di sviluppo (cioè 16 d dopo l'inizio della differenziazione) eventi sincroni coinvolgono tutti i canali . dei MEA vengono rilevati (Figura 2O) il livello di attività di rete è aumentata durante lo sviluppo: durante la quarta settimana dopo il t che l'induzione della differenziazione, le reti neuronali mostrato alto livello di attività spontanea (2,5 ± 0,1 picco / s; Figura 2N) in tutti i pozzetti del dispositivo. Le reti anche esposte scoppia sincroni di rete (4.1 ± 0.1 scoppio / min, Figura 2O) con lunga durata (2.100 ± 500 ms).
Figura 1: hiPSC differenziazione in neuroni. A. Tre punti di tempo di differenziazione in neuroni hiPSCs su vetrini. B. placcatura di hiPSCs su MEA. C. Gli astrociti al 100% di confluenza nel pallone T75 (notare che le cellule formano un monostrato mosaico). Barre di scala: 150 micron. Clicca qui per vedere una versione più grande di questa figura.
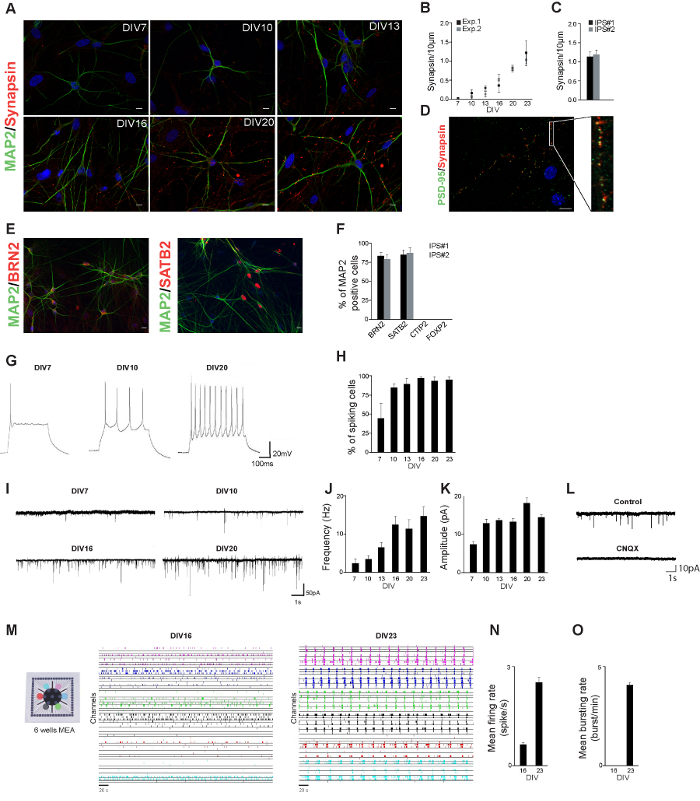
Figura 2. hiPSC-derivato neuroni Caratterizzazione. Neuroni A. hiPSC-derivati sono state colorate per MAP2 (verde) e synapsin-1/2 (rosso) a diversi giorni dopo l'inizio della differenziazione. barra della scala: 10 micron. B. La quantificazione di sinapsina puncta in due esperimenti indipendenti. In ogni esperimenti sono stati analizzati almeno 10 cellule. C. La quantificazione del puncta synapsin a DIV23 nei neuroni derivati da due linee IPS indipendenti. Neuroni D. hiPSC-derivati sono state colorate per PSD-95 (verde) e synapsin-1/2 (rosso) 23 giorni dopo l'inizio della differenziazione. puncta sinapsina sono giustapposti a PSD-95 puncta. Neuroni E. hiPSC-derivati sono state colorate per MAP2 (verde) e BRN2 (rosso) o SATB2 (rosso) 23 d dopo l'inizio della differenziazione. F. percentuale di cellule positive MAP2 chesono risultati positivi per i marcatori indicati. G. Rappresentante registrazioni clamp corrente mostrano che i potenziali d'azione possono essere generati come già 7 d dopo l'inizio della differenziazione. H. Percentuale di cellule a diversi giorni dopo l'induzione della differenziazione che mostrano uno o più potenziali d'azione. I. tracce rappresentativi delle correnti post-sinaptici eccitatori (EPSCs) ricevuti dai neuroni hiPSC derivati a diversi giorni dopo la differenziazione. J. frequenza delle correnti post-sinaptici eccitatori durante lo sviluppo. K. ampiezza delle correnti post-sinaptici eccitatori durante lo sviluppo. L. Rappresentante tracce delle registrazioni EPSC senza (controllo) e con CNQX (CNQX). M. neuroni derivata da una linea hiPSC sono state coltivate su un 6-ben MEA e attività di rete è indicata per le reti neuronali hiPSC-derivati 16 e 23 d dopo l'induzione della differenziazione. L'attività registrata dalciascuno (frequenza di campionamento di 10 kHz) e viene indicata con un colore diverso (5 min del 20 min di registrazione sono presenti). N. gittata 16 e 23 d dopo l'induzione della differenziazione. O. tasso di scoppio 16 e 23 d dopo l'induzione della differenziazione. Clicca qui per vedere una versione più grande di questa figura.
Visti i risultati, la qualità dei neuroni hiPSC derivato risultante può essere valutata facendo un profilo neurofisiologico delle cellule. Cioè, tre o quattro settimane dopo l'inizio della differenziazione, la morfologia, synapsin-1/2 espressione e elettrofisiologia dei neuroni possono essere valutate. A quel punto il tempo, i neuroni hiPSC-derivati sono tenuti a dimostrare una morfologia neuronale simile, per essere MAP2, synapsin / PSD-95 positivo immunocitochimica durante l'esecuzione, e di mostret spontanea attività elettrofisiologica (sia a singola cellula e livello di rete).
Discussione
Qui abbiamo implementato un efficiente protocollo hiPSC-differenziazione pubblicato da Zhang et al. (2013) 12 per la misurazione della attività di rete delle reti neuronali hiPSC-derivati su MEA. Abbiamo adattato il protocollo originale con la creazione di una linea hiPSC -positivo rtTA / Ngn2 prima di indurre la differenziazione neuronale. Questo ulteriore passo ci permette di controllare la densità delle cellule neuronali sul MEA. Il controllo della densità neuronale è stato un importante pre-requisito per adattare il protocollo di MEA e per garantire la coerenza. Per misurare l'attività delle reti neuronali con MEA, i neuroni devono formare reti dense direttamente sulla parte superiore degli elettrodi MEA 17,18. Ciò richiede necessariamente uno stretto controllo sulla densità di placcatura dei neuroni. La linea hiPSC -positivo rtTA / Ngn2 consente il controllo della densità neuronale perché questa tattica non si basa su trasduzione lentivirali acuti di hiPSCs prima di differenziazione;la linea -positive hiPSC rtTA / Ngn2 quindi elimina quasi qualsiasi variazione della resa finale a causa, per esempio, la tossicità lentivirale ed efficienza dell'infezione variabile.
Un altro punto critico della procedura sperimentale è il numero degli astrociti di ratto che sono co-coltivate con le hiPSCs differenziazione. Gli astrociti contribuiscano attivamente al perfezionamento di sviluppare circuiti neurali controllando la formazione delle sinapsi, la manutenzione, e l'eliminazione, che sono tutti processi importanti per il funzionamento neuronale. Il protocollo presentato in questo documento è fortemente astrociti-dipendente: a piena maturità e formano sinapsi funzionali, i neuroni richiedono il supporto dagli astrociti. Abbiamo sperimentato che il numero di astrociti dovrebbe essere all'incirca uguale al numero di neuroni hiPSC derivati per sostenere la maturazione dei neuroni e la formazione di reti neuronali che presentano attività spontanea. Dal momento che il nostro protocollo astrociti rendimenti cul cellula primariature con una durata di vita limitata, l'isolamento degli astrociti di ratto deve essere eseguita regolarmente.
Il nostro adattamento del protocollo pubblicato da Zhang et al. (2013) 12 per l'utilizzo con la tecnologia MEA sarà probabilmente migliorare significativamente la nostra capacità di studiare l'attività di rete delle reti hiPSC-derivati. In precedenza, i protocolli utilizzati per lo studio di reti neuronali hiPSC-derivati con MEA affidamento sulle procedure di differenziazione in termini di tempo 13-16. Il protocollo da Zhang et al. (2013) fornisce una rapida alternativa, e la nostra modifica rimuove una fonte di variabilità, che rende ora più possibile l'uso neuroni hiPSC-derivati in combinazione con la tecnologia MEA, soprattutto in alta produttività o studi farmacologici. Inoltre, poiché il metodo pubblicato da Zhang et al. (2013) 12 produce una popolazione omogenea di livello superiore neuroni corticali, il nostro protocollo adattato rende possibili studi mirati in rete unctivity di questo particolare sottoinsieme neuronale.
Tuttavia, questo approccio ha anche diversi limiti. In primo luogo, l'omogeneità delle culture può anche essere considerato uno svantaggio, perché le culture sono meno probabilità di assomigliare in reti vivo, dove diverse classi di neuroni (cioè neuroni inibitori ed eccitatori) costituiscono una rete eterogenea. Per migliorare ulteriormente l'uso dei neuroni hiPSC-derivati con tecnologia MEA, sarà importante sviluppare protocolli di differenziazione (transgene) basato rapidi per altre popolazioni di cellule neuronali. Se i protocolli saranno disponibili, le reti in vitro sarebbero imitare più da vicino le reti in vivo. In secondo luogo, attualmente astrociti di ratto devono essere aggiunti ai neuroni hiPSC-derivati per sostenere la crescita, e quindi la rete neuronale risultante non è un neuronale stricto sensu rete umana. protocolli affidabili per differenziare hiPSCs in astrociti potrebbero in futuro solve questo problema 26. Terzo, le reti neuronali bidimensionali, come descritto qui, sono un modello limitato per lo studio in vivo complesse reti neuronali tridimensionali. Fortunatamente, protocolli descrivono culture tridimensionali di neuroni primari di ratto in combinazione con la tecnologia MEA sono già disponibili 27,28. In prospettiva, la combinazione di protocolli di differenziazione rapidi per l'ottenimento di neuroni e astrociti hiPSC-derivati con tecniche di coltura tridimensionale e la tecnologia MEA dovrebbe fornire una visione nuova dei meccanismi biologici sottostanti disturbi neurologici.
Divulgazioni
The authors have nothing to disclose.
Riconoscimenti
The authors thank Jessica Classen for performing the whole-cell patch-clamp experiments. The hiPSCs used in our experiments were kindly provided by Huiqing Zhou and Willem van den Akker from the Radboud University Nijmegen. The transfer vectors used in this protocol were kindly provided by Oliver Brüstle, Philipp Koch and Julia Ladewig from the University of Bonn Medical Centre.
Materiali
| Name | Company | Catalog Number | Comments |
| Lebovitz's L-15 medium | Gibco | 11415-064 | |
| B-27 supplement | Gibco | 0080085SA | |
| Poly-D-Lysine (PDL) | Sigma-Aldrich | P6407 | |
| Ca2+/Mg2+-free HBSS | Gibco | 14175-095 | |
| 0.05% Trypsin-EDTA | Gibco | 25300-054 | |
| 2.5% Trypsin | Gibco | 15090-046 | |
| High-glucose DMEM | Gibco | 11965-092 | |
| FBS (Fetal Bovine Serum) | Sigma-Aldrich | F2442-500ML | |
| Penicillin/Streptomycin | Sigma-Aldrich | P4333 | |
| 70 µm cell strainer | BD Falcon | 352350 | |
| DPBS | Gibco | 14190-094 | |
| psPAX2 lentiviral packaging vector | Addgene | Plasmid #12260 | |
| pMD2.G lentiviral packaging vector | Addgene | Plasmid #12259 | |
| Basement membrane matrix | Gibco | A1413201 | |
| DMEM/F12 | Gibco | 11320-074 | |
| Cell detachment solution | Sigma-Aldrich | A6964 | |
| E8 medium | Gibco | A1517001 | |
| ROCK inhibitor | Gibco | A2644501 | Alternatively, ROCK inhibitors like thiazovivin can be used. |
| Polybrene | Sigma-Aldrich | H9268-5G | |
| G418 | Sigma-Aldrich | G8168-10ML | |
| Puromycin | Sigma-Aldrich | P9620-10ML | |
| Vitronectin | Gibco | A14700 | |
| 6-well MEAs | Multi Channel Systems | 60-6wellMEA200/30iR-Ti-tcr | |
| Glass coverslips | VWR | 631-0899 | |
| Poly-L-Ornithine (PLO) | Sigma-Aldrich | P3655-10MG | |
| Laminin | Sigma-Aldrich | L2020-1MG | |
| Doxycyclin | Sigma-Aldrich | D9891-5G | |
| N-2 supplement | Gibco | 17502-048 | |
| Non-essential amino acids | Sigma-Aldrich | M7145 | |
| NT-3, human recombinant | Promokine | C66425 | |
| BDNF, human recombinant | Promokine | C66212 | |
| Trypsin-EDTA | Gibco | 25300-054 | |
| L-alanyl-L-glutamine | Gibco | 35050-038 | |
| Neurobasal medium | Gibco | 21103-049 | |
| Cytosine β-D-arabinofuranoside | Sigma-Aldrich | C1768-100MG | |
| Straight fine-tipped forceps | Fine Science Tools | 11251 | |
| Fine-tipped spring scissors | Fine Science Tools | 91500-09 |
Riferimenti
- Ahfeldt, T., Litterman, N. K., Rubin, L. Studying human disease using human neurons. Brain Res. , (2016).
- Zhang, S. C., Wernig, M., Duncan, I. D., Brustle, O., Thomson, J. A. In vitro differentiation of transplantable neural precursors from human embryonic stem cells. Nat Biotechnol. 19 (12), 1129-1133 (2001).
- Perrier, A. L., et al. Derivation of midbrain dopamine neurons from human embryonic stem cells. Proc Natl Acad Sci USA. 101 (34), 12543-12548 (2004).
- Wu, H., et al. Integrative genomic and functional analyses reveal neuronal subtype differentiation bias in human embryonic stem cell lines. Proc Natl Acad Sci USA. 104 (34), 13821-13826 (2007).
- Chambers, S. M., Fasano, C. A., Papapetrou, E. P., Tomishima, M., Sadelain, M., Studer, L. Highly efficient neural conversion of human ES and iPS cells by dual inhibition of SMAD signaling. Nat Biotechnol. 27 (3), 275-280 (2009).
- Shi, Y., Kirwan, P., Livesey, F. J. Directed differentiation of human pluripotent stem cells to cerebral cortex neurons and neural networks. Nat Protoc. 7, 1836-1846 (2012).
- Espuny-Camacho, I., et al. Pyramidal neurons derived from human pluripotent stem cells integrate efficiently into mouse brain circuits in vivo. Neuron. 77 (3), 440-456 (2013).
- Maroof, A. M., et al. Directed differentiation and functional maturation of cortical interneurons from human embryonic stem cells. Cell Stem Cell. 12 (5), 559-572 (2013).
- Johnson, M. A., Weick, J. P., Pearce, R. A., Zhang, S. C. Functional neural development from human embryonic stem cells: accelerated synaptic activity via astrocyte coculture. J Neurosci. 27 (12), 3069-3077 (2007).
- Hu, B. Y., et al. Neural differentiation of human induced pluripotent stem cells follows developmental principles but with variable potency. Proc Natl Acad Sci USA. 107 (9), 4335-4340 (2010).
- Kim, H., et al. miR-371-3 expression predicts neural differentiation propensity in human pluripotent stem cells. Cell Stem Cell. 8 (6), 695-706 (2011).
- Zhang, Y., et al. Rapid single-step induction of functional neurons from human pluripotent stem cells. Neuron. 78 (5), 785-798 (2013).
- Odawara, A., Saitoh, Y., Alhebshi, A. H., Gotoh, M., Suzuki, I. Long-term electrophysiological activity and pharmacological response of a human induced pluripotent stem cell-derived neuron and astrocyte co-culture. Biochem Biophys Res Commun. 443 (4), 1176-1181 (2014).
- Odawara, A., Katoh, H., Matsuda, N., Suzuki, I. Physiological maturation and drug responses of human induced pluripotent stem cell-derived cortical neuronal networks in long-term culture. Sci Rep. 6, 26181 (2016).
- Amin, H., Maccione, A., Marinaro, F., Zordan, S., Nieus, T., Berdondini, L. Electrical Responses and Spontaneous Activity of Human iPS-Derived Neuronal Networks Characterized for 3-month Culture with 4096-Electrode Arrays. Front Neurosci. 10, (2016).
- Heikkila, T. J., et al. Human embryonic stem cell-derived neuronal cells form spontaneously active neuronal networks in vitro. Exp Neurol. 218 (1), 109-116 (2009).
- Massobrio, P., Massobrio, G., Martinoia, S. Multi-program approach for simulating recorded extracellular signals generated by neurons coupled to microelectrode arrays. Neurocomputing. 70, 2467-2476 (2007).
- Wang, L., Riss, M., Buitrago, J. O., Claverol-Tinturé, E. Biophysics of microchannel-enabled neuron-electrode interfaces. J Neural Eng. 9 (2), (2012).
- McCarthy, K. D., de Vellis, J. Preparation of separate astroglial and oligodendroglial cell cultures from rat cerebral tissue. J Cell Biol. 85, 890-902 (1980).
- Schildge, S., Bohrer, C., Beck, K., Schachtrup, C. Isolation and culture of mouse cortical astrocytes. J. Vis. Exp. (71), e50079 (2013).
- Pacifici, M., Peruzzi, F. Isolation and culture of rat embryonic neural cells: a quick protocol. J Vis Exp. (63), e3965 (2012).
- Ba, W., et al. ARHGAP12 functions as a developmental brake on excitatory synapse function. Cell Rep. 14 (6), 1355-1368 (2016).
- Bologna, L. L., et al. Investigating neuronal activity by SPYCODE multi-channel data analyzer. Neural Netw. 23 (6), 685-697 (2010).
- Ba, W., et al. TRIO loss of function is associated with mild intellectual disability and affects dendritic branching and synapse function. Hum Mol Genet. 25 (5), 892-902 (2016).
- Benevento, M., et al. Histone methylation by the Kleefstra syndrome protein EHMT1 mediates homeostatic synaptic scaling. Neuron. 91 (2), 341-355 (2016).
- Krencik, R., Weick, J. P., Liu, Y., Zhang, Z. -. J., Zhang, S. -. C. Specification of transplantable astroglial subtypes from human pluripotent stem cells. Nature biotechnology. 29 (6), 528-534 (2011).
- Frega, M., Tedesco, M., Massobrio, P., Pesce, M., Martinoia, S. Network dynamics of 3D engineered neuronal cultures: a new experimental model for in-vitro electrophysiology. Sci Rep. 4, 5489 (2014).
- Tedesco, M., Frega, M., Martinoia, S., Pesce, M., Massobrio, P. Interfacing 3D engineered neuronal cultures to micro-electrode arrays: an innovative in vitro experimental model. J Vis Exp. (104), e53080 (2015).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon